репликация ДНК

В молекулярной биологии , [1] [2] [3] Репликация ДНК — это биологический процесс создания двух идентичных реплик ДНК из одной исходной молекулы ДНК . [4] Репликация ДНК происходит во всех живых организмах и является наиболее важной частью биологической наследственности . Это важно для деления клеток во время роста и восстановления поврежденных тканей, а также гарантирует, что каждая новая клетка получит свою собственную копию ДНК. [5] Клетка обладает отличительным свойством деления, что делает необходимым репликацию ДНК.
ДНК состоит из двойной спирали двух комплементарных цепей . Двойная спираль описывает внешний вид двухцепочечной ДНК, которая, таким образом, состоит из двух линейных нитей, которые идут напротив друг друга и скручиваются вместе, образуя форму. [6] Во время репликации эти цепи разделяются. Каждая цепь исходной молекулы ДНК затем служит матрицей для производства своей копии — процесс, называемый полуконсервативной репликацией . В результате полуконсервативной репликации новая спираль будет состоять из исходной цепи ДНК, а также из вновь синтезированной цепи. [7] Механизмы клеточной корректуры и проверки ошибок обеспечивают почти идеальную точность репликации ДНК. [8] [9]
В клетке репликация ДНК начинается в определенных местах или источниках репликации . [10] в геноме [11] содержащий генетический материал организма. [12] Раскручивание ДНК в месте начала и синтез новых цепей, поддерживаемых ферментом, известным как хеликаза , приводит к тому, что репликационные вилки растут в двух направлениях от начала. Ряд белков связан с репликационной вилкой, помогая инициировать и продолжать синтез ДНК . Прежде всего, ДНК-полимераза синтезирует новые цепи, добавляя нуклеотиды , которые дополняют каждую (матрицу) цепь. Репликация ДНК происходит во время S-стадии интерфазы . [13]
Репликация ДНК (амплификация ДНК) также может осуществляться in vitro (искусственно, вне клетки). [14] ДНК-полимеразы, выделенные из клеток, и искусственные ДНК-праймеры можно использовать для запуска синтеза ДНК на известных последовательностях в матричной молекуле ДНК. полимеразная цепная реакция (ПЦР), лигазная цепная реакция (ЛЦР) и опосредованная транскрипцией амплификация Примерами являются (ТМА). В марте 2021 года исследователи сообщили о доказательствах, свидетельствующих о том, что предварительной формой транспортной РНК , необходимым компонентом трансляции , биологического синтеза новых белков в соответствии с генетическим кодом , могла быть сама молекула-репликатор на самых ранних стадиях развития жизни. или абиогенез . [15] [16]
Структура ДНК
[ редактировать ]
ДНК существует в виде двухцепочечной структуры, где обе нити скручены вместе, образуя характерную двойную спираль . Каждая отдельная нить ДНК представляет собой цепочку из четырех типов нуклеотидов . Нуклеотиды в ДНК содержат сахар дезоксирибозу , фосфат и азотистое основание . Четыре типа нуклеотидов соответствуют четырем нуклеиновым основаниям аденину , цитозину , гуанину и тимину , обычно обозначаемым сокращенно A, C, G и T. Аденин и гуанин представляют собой пурины. [17] основаниями, а цитозин и тимин являются пиримидинами . Эти нуклеотиды образуют фосфодиэфирные связи , создавая фосфатно-дезоксирибозный остов двойной спирали ДНК с нуклеиновыми основаниями, направленными внутрь (т. е. в сторону противоположной цепи). Нуклеиновые основания соединяются между цепями посредством водородных связей с образованием пар оснований . Аденин образует пару с тимином (две водородные связи), а гуанин — с цитозином (три водородные связи ). [18]
Нити ДНК имеют направленность , и разные концы одной цепи называются «3' (трехштриховым) концом» и «5' (пятиштриховым) концом». По соглашению, если дана последовательность оснований одной цепи ДНК, левый конец последовательности - это 5'-конец, а правый конец последовательности - это 3'-конец. Нити двойной спирали антипараллельны: одна из них расположена от 5’ к 3’, а противоположная – от 3’ к 5’. Эти термины относятся к атому углерода в дезоксирибозе, к которому присоединяется следующий фосфат в цепи. Направленность имеет последствия при синтезе ДНК, поскольку ДНК-полимераза может синтезировать ДНК только в одном направлении, добавляя нуклеотиды к 3'-концу цепи ДНК.
Спаривание комплементарных оснований в ДНК (посредством водородных связей ) означает, что информация, содержащаяся внутри каждой цепи, является избыточной. Фосфодиэфирные (внутрицепочечные) связи прочнее водородных (межцепочечных). Фактическая работа фосфодиэфирных связей заключается в том, что в полимерах ДНК 5'-атом углерода одного нуклеотида соединяется с 3'-атомом углерода другого нуклеотида, в то время как водородные связи стабилизируют двойные спирали ДНК поперек оси спирали, а не в направлении оси спирали. ось. [19] Это дает возможность отделить пряди друг от друга. Таким образом, нуклеотиды одной цепи можно использовать для реконструкции нуклеотидов вновь синтезированной партнерской цепи. [20]
ДНК-полимераза
[ редактировать ]
ДНК-полимеразы — это семейство ферментов , которые осуществляют все формы репликации ДНК. [22] ДНК-полимеразы, как правило, не могут инициировать синтез новых цепей, а могут только удлинять существующую цепь ДНК или РНК, соединенную с цепью матрицы. Чтобы начать синтез, необходимо создать короткий фрагмент РНК, называемый праймером , и соединить его с цепью ДНК-матрицы.
ДНК-полимераза добавляет новую цепь ДНК, удлиняя 3'-конец существующей нуклеотидной цепи, добавляя по одному новые нуклеотиды , соответствующие шаблонной цепи, посредством создания фосфодиэфирных связей . Энергия для этого процесса полимеризации ДНК поступает от гидролиза высокоэнергетических фосфатных (фосфоангидридных) связей между тремя фосфатами, прикрепленными к каждому невключенному основанию . Свободные основания с присоединенными к ним фосфатными группами называются нуклеотидами ; в частности, основания с тремя присоединенными фосфатными группами называются нуклеозидтрифосфатами . При присоединении нуклеотида к растущей цепи ДНК образование фосфодиэфирной связи между проксимальным фосфатом нуклеотида к растущей цепи сопровождается гидролизом высокоэнергетической фосфатной связи с высвобождением двух дистальных фосфатных групп в виде пирофосфат . Ферментативный гидролиз полученного пирофосфата в неорганический фосфат потребляет вторую высокоэнергетическую фосфатную связь и делает реакцию практически необратимой. [Примечание 1]
В целом ДНК-полимеразы очень точны: частота ошибок составляет менее одной ошибки на каждые 10. 7 добавлены нуклеотиды. [23] Некоторые ДНК-полимеразы могут также удалять нуклеотиды с конца развивающейся цепи, чтобы исправить несовпадающие основания. Это известно как корректура. Наконец, механизмы пострепликационной репарации несоответствий отслеживают ДНК на наличие ошибок, способные отличать несоответствия во вновь синтезированной цепи ДНК от исходной последовательности цепи. Вместе эти три этапа дискриминации обеспечивают точность репликации менее одной ошибки на каждые 10 9 добавлены нуклеотиды. [23]
Скорость репликации ДНК в живой клетке была впервые измерена как скорость удлинения ДНК фага Т4 в инфицированной фагом E. coli . [24] В период экспоненциального роста ДНК при 37°С скорость составляла 749 нуклеотидов в секунду. Частота мутаций на пару оснований за репликацию во время синтеза ДНК фага Т4 составляет 1,7 на 10. 8 . [25]
Процесс репликации
[ редактировать ]

Репликация ДНК, как и все процессы биологической полимеризации, протекает в три ферментативно катализируемых и скоординированных этапа: инициация, элонгация и терминация.
Инициация
[ редактировать ]
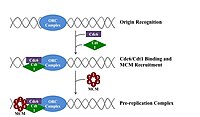
Чтобы клетка могла делиться , она должна сначала реплицировать свою ДНК. [26] Репликация ДНК — это процесс по принципу «все или ничего»; как только репликация начинается, она завершается. После завершения репликации она больше не повторяется в том же клеточном цикле. Это становится возможным благодаря разделению инициации пререпликационного комплекса .
Пререпликационный комплекс
[ редактировать ]В позднем митозе и ранней фазе G1 большой комплекс белков-инициаторов собирается в пререпликационный комплекс в определенных точках ДНК, известных как « оригиналы ». [11] [10] В E. coli основным белком-инициатором является ДНК А ; у дрожжей это комплекс распознавания происхождения . [27] Последовательности, используемые белками-инициаторами, имеют тенденцию быть «богатыми АТ» (богатыми основаниями аденина и тимина), поскольку пары оснований АТ имеют две водородные связи (а не три, образующиеся в паре CG), и, следовательно, их легче разделить. [28] У эукариот комплекс распознавания ориджина катализирует сборку белков-инициаторов в пререпликационный комплекс. Кроме того, недавний отчет предполагает, что ORC почкующихся дрожжей димеризуется в зависимости от клеточного цикла, чтобы контролировать лицензирование. [29] [30] В свою очередь, процесс димеризации ORC опосредован зависимым от клеточного цикла циклом димеризации Noc3p in vivo, и эта роль Noc3p отделена от его роли в биогенезе рибосом. Существенный цикл димеризации Noc3p опосредует образование двойного гексамера ORC при лицензировании репликации. ORC и Noc3p постоянно связаны с хроматином на протяжении всего клеточного цикла. [31] Cdc6 и Cdt1 затем связываются со связанным комплексом распознавания источника в источнике, чтобы сформировать более крупный комплекс, необходимый для загрузки комплекса Mcm в ДНК. У эукариот комплекс Mcm представляет собой хеликазу, которая расщепляет спираль ДНК в вилках и началах репликации. Комплекс Mcm рекрутируется на поздней фазе G1 и загружается комплексом ORC-Cdc6-Cdt1 в ДНК посредством АТФ-зависимого ремоделирования белка. Загрузка комплекса Mcm на исходную ДНК знаменует завершение формирования пререпликационного комплекса. [32]
Если условия окружающей среды подходят для поздней фазы G1, циклин G1 и G1/S - Cdk активируются комплексы , которые стимулируют экспрессию генов, кодирующих компоненты синтетического механизма ДНК. Активация G1/S-Cdk также способствует экспрессии и активации комплексов S-Cdk, которые могут играть роль в активации начала репликации в зависимости от вида и типа клеток. Контроль над этими Cdk варьируется в зависимости от типа клеток и стадии развития. Эту регуляцию лучше всего понять у почкующихся дрожжей , где S-циклины Clb5 и Clb6 в первую очередь ответственны за репликацию ДНК. [33] Комплексы Clb5,6-Cdk1 непосредственно запускают активацию начала репликации и, следовательно, необходимы на протяжении S-фазы для прямой активации каждого начала репликации. [32]
Аналогичным образом Cdc7 также необходим в фазе S для активации начала репликации. Cdc7 не активен на протяжении всего клеточного цикла, и его активация строго рассчитана во избежание преждевременного начала репликации ДНК. В конце G1 активность Cdc7 резко возрастает в результате ассоциации с регуляторной субъединицей DBF4 , которая напрямую связывает Cdc7 и способствует его протеинкиназной активности. Было обнаружено, что Cdc7 является лимитирующим регулятором активности происхождения. Вместе G1/S-Cdks и/или S-Cdks и Cdc7 взаимодействуют, напрямую активируя точки начала репликации, что приводит к инициации синтеза ДНК. [32]
Прединициационный комплекс
[ редактировать ]На ранней S-фазе активация S-Cdk и Cdc7 приводит к сборке преинициативного комплекса, массивного белкового комплекса, образующегося в начале. Формирование преинициативного комплекса вытесняет Cdc6 и Cdt1 из исходного репликационного комплекса, инактивируя и разбирая пререпликационный комплекс. Загрузка преинициативного комплекса в ориджин активирует хеликазу Mcm, вызывая раскручивание спирали ДНК. Преинициативный комплекс также загружает α-примазу и другие ДНК-полимеразы. в ДНК [32]
После того, как α-примаза синтезирует первые праймеры, соединения праймер-матрица взаимодействуют с загрузчиком зажима, который загружает скользящий зажим на ДНК, чтобы начать синтез ДНК. Компоненты преинициативного комплекса остаются связанными с репликационными вилками по мере их удаления от источника. [32]
Удлинение
[ редактировать ]ДНК-полимераза обладает 5’–3’-активностью.Все известные системы репликации ДНК требуют свободной 3'- гидроксильной группы, прежде чем можно будет начать синтез (примечание: матрица ДНК считывается в направлении от 3' к 5', тогда как новая цепь синтезируется в направлении от 5' к 3' - это часто смущенный). Выделяют четыре различных механизма синтеза ДНК:
- Все клеточные формы жизни и многие ДНК- вирусы , фаги и плазмиды используют примазу для синтеза короткого праймера РНК со свободной 3'-ОН-группой, которая впоследствии удлиняется ДНК-полимеразой.
- Ретроэлементы (включая ретровирусы ) используют транспортную РНК, которая запускает репликацию ДНК, предоставляя свободный 3'-ОН, который используется для элонгации обратной транскриптазой .
- В аденовирусах семейства φ29 и бактериофагах 3'-ОН-группа представлена боковой цепью аминокислоты белка, прикрепленного к геному (концевого белка), к которому ДНК-полимераза добавляет нуклеотиды для образования новой цепи.
- В одноцепочечных ДНК-вирусах (группа, в которую входят цирковирусы , геминивирусы , парвовирусы и другие), а также во многих фагах и плазмидах , использующих механизм репликации по катящемуся кругу (RCR), эндонуклеаза RCR создает разрыв в цепи генома. (одноцепочечные вирусы) или одной из цепей ДНК (плазмиды). 5'-конец разорванной цепи переносится на тирозиновый остаток нуклеазы, а свободная 3'-ОН-группа затем используется ДНК-полимеразой для синтеза новой цепи.
Клеточные организмы используют первый из этих путей, поскольку он наиболее известен. В этом механизме, как только две цепи разделены, примаза добавляет праймеры РНК к нитям матрицы. Ведущая цепь получает один праймер РНК, а отстающая цепь — несколько. Ведущая цепь непрерывно удлиняется от праймера с помощью ДНК-полимеразы с высокой процессивностью , тогда как отстающая цепь удлиняется прерывисто от каждого праймера, образуя фрагменты Оказаки . РНКаза удаляет фрагменты РНК праймера, и для заполнения пробелов входит ДНК-полимераза с низкой процессивностью, отличная от репликативной полимеразы. Когда это будет завершено, можно будет обнаружить одну зазубрину на ведущей нити и несколько зазубрин на отстающей нити. Лигаза заполняет эти разрывы, завершая таким образом новую реплицированную молекулу ДНК.
Примаза, используемая в этом процессе, существенно различается у бактерий и архей / эукариот . Бактерии используют примазу, принадлежащую к суперсемейству белков DnaG , которая содержит каталитический домен типа складки TOPRIM. [34] Складка TOPRIM содержит ядро α/β с четырьмя консервативными цепями в топологии , подобной Россману . Эта структура также обнаружена в каталитических доменах топоизомеразы Ia, топоизомеразы II, нуклеаз OLD-семейства и белков репарации ДНК, родственных белку RecR.
Примаза, используемая архей и эукариотами, напротив, содержит высокоразвитую версию мотива распознавания РНК (RRM). Эта примаза структурно сходна со многими вирусными РНК-зависимыми РНК-полимеразами, обратными транскриптазами, циклазами, генерирующими циклические нуклеотиды, и ДНК-полимеразами семейств A/B/Y, которые участвуют в репликации и репарации ДНК. При репликации эукариот примаза образует комплекс с Pol α. [35]
Множественные ДНК-полимеразы играют разные роли в процессе репликации ДНК. В E. coli ДНК Pol III является ферментом полимеразой, главным образом ответственным за репликацию ДНК. Он собирается в репликационный комплекс на репликационной вилке, который демонстрирует чрезвычайно высокую процессивность и остается неизменным на протяжении всего цикла репликации. Напротив, DNA Pol I является ферментом, ответственным за замену праймеров РНК на ДНК. активностью от 5' до 3' ДНК Pol I обладает экзонуклеазной в дополнение к своей полимеразной активности и использует свою экзонуклеазную активность для разрушения праймеров РНК перед ней, поскольку она удлиняет цепь ДНК позади нее в процессе, называемом трансляцией ника . Pol I гораздо менее процессивен, чем Pol III, поскольку его основная функция при репликации ДНК заключается в создании множества коротких участков ДНК, а не нескольких очень длинных участков.
У эукариот фермент с низкой процессивностью Pol α помогает инициировать репликацию, поскольку он образует комплекс с примазой. [36] Считается, что у эукариот синтез ведущей цепи осуществляется Pol ε; однако эта точка зрения недавно была оспорена, предполагая роль Pol δ. [37] Удаление праймера завершено Pol δ [38] а репарация ДНК при репликации завершается Pol ε.
По мере продолжения синтеза ДНК исходные нити ДНК продолжают раскручиваться с каждой стороны пузыря, образуя репликационную вилку с двумя зубцами. У бактерий, которые имеют единственный источник репликации на кольцевой хромосоме, этот процесс создает « тета-структуру » (напоминающую греческую букву тета: θ). Напротив, эукариоты имеют более длинные линейные хромосомы и инициируют репликацию в нескольких источниках внутри них. [39]
Репликационная вилка
[ редактировать ]
a: матрица, b: ведущая цепь, c: отстающая цепь, d: репликационная вилка, e: праймер, f: фрагменты Оказаки

Репликационная вилка — это структура, которая образуется внутри длинной спирали ДНК во время репликации ДНК. Его производят ферменты, называемые хеликазами, которые разрывают водородные связи, удерживающие цепи ДНК вместе в спирали. Полученная структура имеет два ветвящихся «зубца», каждый из которых состоит из одной цепи ДНК. Эти две цепи служат матрицей для ведущей и отстающей цепей, которые будут создаваться, когда ДНК-полимераза сопоставляет комплементарные нуклеотиды с матрицами; шаблоны можно правильно назвать шаблоном ведущей цепи и шаблоном отстающей цепи.
ДНК считывается ДНК-полимеразой в направлении от 3’ к 5’, что означает, что новая цепь синтезируется в направлении от 5’ к 3’. Поскольку матрицы ведущей и отстающей цепей в репликационной вилке ориентированы в противоположных направлениях, основной вопрос заключается в том, как добиться синтеза новой отстающей цепи ДНК, направление синтеза которой противоположно направлению растущей репликационной вилки.
Ведущая нить
[ редактировать ]Ведущая цепь — это цепь новой ДНК, которая синтезируется в том же направлении, что и растущая репликационная вилка. Этот вид репликации ДНК является непрерывным.
Отстающая прядь
[ редактировать ]Отстающая цепь — это цепь новой ДНК, направление синтеза которой противоположно направлению растущей репликационной вилки. Из-за своей ориентации репликация отстающей цепи более сложна по сравнению с репликацией ведущей цепи. Как следствие, ДНК-полимераза на этой цепи «отстает» от другой цепи.
Отстающая цепь синтезируется короткими отдельными сегментами. отстающей цепи На матрице примаза « читает» ДНК-матрицу и инициирует синтез короткого комплементарного праймера РНК . ДНК-полимераза удлиняет загрунтованные сегменты, образуя фрагменты Оказаки . Затем праймеры РНК удаляются и заменяются ДНК, а фрагменты ДНК соединяются ДНК-лигазой .
Динамика на репликационной вилке
[ редактировать ]
Во всех случаях геликаза состоит из шести полипептидов, которые охватывают только одну цепь реплицируемой ДНК. Две полимеразы связаны с гексамером геликазы. У эукариот геликаза обвивает ведущую цепь, а у прокариот – отстающую. [40]
Когда хеликаза раскручивает ДНК в репликационной вилке, ДНК, находящаяся впереди, вынуждена вращаться. Этот процесс приводит к накоплению искривлений в ДНК впереди. [41] Это накопление создает скручивающую нагрузку, которая в конечном итоге остановит репликационную вилку. Топоизомеразы — это ферменты, которые временно разрывают нити ДНК, снимая напряжение, вызванное раскручиванием двух нитей спирали ДНК; топоизомеразы (включая ДНК-гиразу ) достигают этого путем добавления отрицательных суперспиралей к спирали ДНК. [42]
Голая одноцепочечная ДНК имеет тенденцию сворачиваться, образуя вторичные структуры ; эти структуры могут мешать движению ДНК-полимеразы. Чтобы предотвратить это, одноцепочечные связывающие белки связываются с ДНК до тех пор, пока не будет синтезирована вторая цепь, предотвращая образование вторичной структуры. [43]
Двухцепочечная ДНК намотана вокруг гистонов , которые играют важную роль в регуляции экспрессии генов, поэтому реплицируемая ДНК должна быть намотана вокруг гистонов в тех же местах, что и исходная ДНК. [44] Чтобы обеспечить это, гистоновые шапероны разбирают хроматин перед его репликацией и заменяют гистоны в нужном месте. Некоторые шаги в этой сборке носят несколько умозрительный характер. [45]
Белки-зажимы действуют как скользящий зажим на ДНК, позволяя ДНК-полимеразе связываться с ее матрицей и способствуя процессивности. Внутренняя поверхность зажима позволяет пропустить через него ДНК. Как только полимераза достигает конца матрицы или обнаруживает двухцепочечную ДНК, скользящий зажим претерпевает конформационные изменения, которые высвобождают ДНК-полимеразу. Белки, загружающие зажим, используются для начальной загрузки зажима, распознавая соединение между матрицей и праймерами РНК. [9] :274-5
белки репликации ДНК
[ редактировать ]В репликационной вилке многие ферменты репликации собираются на ДНК в сложную молекулярную машину, называемую реплисомой . Ниже приводится список основных ферментов репликации ДНК, которые участвуют в реплисоме: [46]
| Фермент | Функция репликации ДНК |
|---|---|
| ДНК-геликаза | Также известен как фермент, дестабилизирующий спираль. Хеликаза разделяет две цепи ДНК в репликационной вилке позади топоизомеразы. |
| ДНК-полимераза | Фермент, ответственный за катализацию добавления нуклеотидных субстратов к ДНК в направлении от 5’ к 3’ во время репликации ДНК. Также осуществляет корректуру и исправление ошибок. Существует множество различных типов ДНК-полимеразы, каждый из которых выполняет разные функции в разных типах клеток. |
| зажим ДНК | Белок, который предотвращает диссоциацию удлиненных ДНК-полимераз от родительской цепи ДНК. |
| Одноцепочечный ДНК-связывающий белок | Связывается с оцДНК и предотвращает повторное отжиг двойной спирали ДНК после того, как ДНК-хеликаза ее раскручивает, тем самым поддерживая разделение цепей и облегчая синтез новой цепи. |
| топоизомераза | Освобождает ДНК от ее суперскрученной природы. |
| ДНК-гираза | Снимает напряжение раскручивания ДНК-хеликазой; это особый тип топоизомеразы |
| ДНК-лигаза | Повторно отжигает полуконсервативные цепи и соединяет фрагменты Оказаки отстающей цепи. |
| Примаза | Обеспечивает отправную точку РНК (или ДНК) для ДНК-полимеразы, чтобы начать синтез новой цепи ДНК. |
| Теломераза | Удлиняет теломерную ДНК путем добавления повторяющихся нуклеотидных последовательностей к концам хромосом эукариот . Это позволяет зародышевым и стволовым клеткам избежать ограничения Хейфлика на деление клеток. [47] |
in vitro Эксперименты на одной молекуле (с использованием оптического пинцета и магнитного пинцета ) обнаружили синергетические взаимодействия между ферментами реплисомы ( геликаза , полимераза и одноцепочечный ДНК-связывающий белок ) и с вилкой репликации ДНК, усиливающими раскручивание ДНК и репликацию ДНК. . [14] Эти результаты приводят к разработке кинетических моделей, учитывающих синергетические взаимодействия и их устойчивость. [14]
Репликационное оборудование
[ редактировать ]
Механизмы репликации состоят из факторов, участвующих в репликации ДНК и появляющихся на матрицах оцДНК. Механизмы репликации включают примозоторы — ферменты репликации; ДНК-полимераза, ДНК-хеликазы, ДНК-зажимы и ДНК-топоизомеразы, а также белки репликации; например, одноцепочечные ДНК-связывающие белки (SSB). В механизмах репликации эти компоненты координируются. У большинства бактерий все факторы, участвующие в репликации ДНК, расположены на репликационных вилках, и комплексы остаются на вилках во время репликации ДНК. Механизмы репликации также называются реплисомами или системами репликации ДНК. Эти термины являются общими терминами для белков, расположенных на репликационных вилках. В эукариотических и некоторых бактериальных клетках реплисомы не образуются.
In an alternative figure, DNA factories are similar to projectors and DNAs are like as cinematic films passing constantly into the projectors. In the replication factory model, after both DNA helicases for leading strands and lagging strands are loaded on the template DNAs, the helicases run along the DNAs into each other. The helicases remain associated for the remainder of replication process. Peter Meister et al. observed directly replication sites in budding yeast by monitoring green fluorescent protein (GFP)-tagged DNA polymerases α. They detected DNA replication of pairs of the tagged loci spaced apart symmetrically from a replication origin and found that the distance between the pairs decreased markedly by time.[48] This finding suggests that the mechanism of DNA replication goes with DNA factories. That is, couples of replication factories are loaded on replication origins and the factories associated with each other. Also, template DNAs move into the factories, which bring extrusion of the template ssDNAs and new DNAs. Meister's finding is the first direct evidence of replication factory model. Subsequent research has shown that DNA helicases form dimers in many eukaryotic cells and bacterial replication machineries stay in single intranuclear location during DNA synthesis.[49]
Фабрики репликации распутывают сестринские хроматиды. Распутывание необходимо для распределения хроматид по дочерним клеткам после репликации ДНК. Поскольку сестринские хроматиды после репликации ДНК удерживают друг друга кольцами когезина , существует единственный шанс на распутывание при репликации ДНК. Установка механизмов репликации в качестве фабрик репликации может повысить вероятность успеха репликации ДНК. Если репликационные вилки свободно перемещаются в хромосомах, сцепление ядер усугубляется и препятствует митотической сегрегации. [48]
Прекращение действия
[ редактировать ]Эукариоты инициируют репликацию ДНК во многих точках хромосомы, поэтому репликационные вилки встречаются и заканчиваются во многих точках хромосомы. Поскольку эукариоты имеют линейные хромосомы, репликация ДНК не может достичь самого конца хромосом. Из-за этой проблемы ДНК теряется в каждом цикле репликации с конца хромосомы. Теломеры — это участки повторяющейся ДНК, расположенные вблизи концов, и помогают предотвратить потерю генов из-за этого укорочения. Укорочение теломер — нормальный процесс в соматических клетках . Это укорачивает теломеры дочерней ДНК-хромосомы. В результате клетки могут делиться только определенное количество раз, прежде чем потеря ДНК предотвратит дальнейшее деление. (Это известно как предел Хейфлика .) Внутри линии зародышевых клеток , которая передает ДНК следующему поколению, теломераза удлиняет повторяющиеся последовательности теломерной области, чтобы предотвратить деградацию. Теломераза может ошибочно активироваться в соматических клетках, что иногда приводит к образованию рака . Повышенная теломеразная активность является одним из признаков рака.
Для завершения требуется, чтобы ход репликационной вилки ДНК был остановлен или заблокирован. Терминация в определенном локусе, когда она происходит, включает взаимодействие между двумя компонентами: (1) последовательностью сайта терминации в ДНК и (2) белком, который связывается с этой последовательностью, чтобы физически остановить репликацию ДНК. У различных видов бактерий он называется белком, связывающим сайт терминального узла репликации ДНК, или белком Ter .
Поскольку бактерии имеют кольцевые хромосомы, прекращение репликации происходит, когда две репликационные вилки встречаются на противоположном конце родительской хромосомы. E. coli регулирует этот процесс с помощью терминирующих последовательностей, которые при связывании с белком Tus позволяют проходить только одному направлению репликационной вилки. В результате репликационные вилки вынуждены всегда встречаться в области терминации хромосомы. [50]
Регулирование
[ редактировать ]
Эукариоты
[ редактировать ]У эукариот репликация ДНК контролируется в рамках клеточного цикла . По мере того как клетка растет и делится, она проходит этапы клеточного цикла; Репликация ДНК происходит во время S-фазы (фазы синтеза). Прогресс эукариотической клетки в цикле контролируется контрольными точками клеточного цикла . Прохождение контрольных точек контролируется посредством сложных взаимодействий между различными белками, включая циклины и циклин-зависимые киназы . [51] В отличие от бактерий, эукариотическая ДНК реплицируется в пределах ядра. [52]
Контрольная точка G1/S (контрольная точка рестрикции) регулирует, вступают ли эукариотические клетки в процесс репликации ДНК и последующего деления. Клетки, не прошедшие эту контрольную точку, остаются на стадии G0 и не реплицируют свою ДНК.
После того, как ДНК прошла тест «G1/S», ее можно скопировать только один раз в каждом клеточном цикле. Когда комплекс Mcm удаляется от источника, пререпликационный комплекс демонтируется. Поскольку новый комплекс Mcm не может быть загружен в точку начала репликации до тех пор, пока субъединицы до репликации не будут повторно активированы, одна точка начала репликации не может использоваться дважды в одном и том же клеточном цикле. [32]
Активация S-Cdks на ранней S-фазе способствует разрушению или ингибированию отдельных компонентов пререпликационного комплекса, предотвращая немедленную повторную сборку. S и M-Cdks продолжают блокировать сборку пререпликационного комплекса даже после завершения фазы S, гарантируя, что сборка не может произойти снова, пока вся активность Cdk не снизится в позднем митозе. [32]
У почкующихся дрожжей ингибирование сборки вызвано Cdk-зависимым фосфорилированием компонентов пререпликационного комплекса. В начале S-фазы фосфорилирование Cdc6 с помощью Cdk1 вызывает связывание Cdc6 с SCF убиквитинпротеинлигазой , что вызывает протеолитическое разрушение Cdc6. Cdk-зависимое фосфорилирование белков Mcm способствует их экспорту из ядра вместе с Cdt1 во время S-фазы, предотвращая загрузку новых комплексов Mcm в источниках в течение одного клеточного цикла. Фосфорилирование Cdk репликационного комплекса также ингибирует сборку пререпликационного комплекса. Индивидуального присутствия любого из этих трех механизмов достаточно, чтобы ингибировать сборку пререпликационного комплекса. Однако мутации всех трех белков в одной и той же клетке вызывают повторную инициацию репликации во многих источниках в пределах одного клеточного цикла. [32] [53]
В клетках животных белок геминин является ключевым ингибитором сборки пререпликационного комплекса. Геминин связывает Cdt1, предотвращая его связывание с комплексом распознавания источника. В G1 уровни геминина поддерживаются на низком уровне с помощью APC, который убиквитинирует геминин, направляя его на деградацию. Когда геминин разрушается, Cdt1 высвобождается, что позволяет ему функционировать в сборке предрепликационного комплекса. В конце G1 APC инактивируется, позволяя геминину накапливаться и связываться с Cdt1. [32]
Репликация хлоропластного и митохондриального геномов происходит независимо от клеточного цикла посредством процесса репликации D-петли .
Фокус репликации
[ редактировать ]В клетках позвоночных сайты репликации концентрируются в положениях, называемых фокусами репликации . [48] Сайты репликации можно обнаружить путем иммуноокрашивания дочерних цепей и ферментов репликации, а также мониторинга факторов репликации, меченных GFP. С помощью этих методов обнаружено, что в S-фазе деления клеток появляются очаги репликации различного размера и положения, и их количество на ядро намного меньше числа репликационных вилок генома.
П. Хойн и др. , [48] (2001) отслеживали фокусы репликации, меченные GFP, в почкующихся дрожжевых клетках и обнаружили, что точки начала репликации постоянно перемещаются в фазе G1 и S, а динамика значительно снижается в фазе S. [48] Традиционно сайты репликации фиксировались на пространственной структуре хромосом с помощью ядерного матрикса или ламинов . Результаты Хойна опровергают традиционные концепции о том, что у почкующихся дрожжей нет ламинов, и подтверждают, что точки начала репликации самособираются и образуют фокусы репликации.
Путем активации источников репликации, контролируемых в пространстве и времени, регулируется образование очагов репликации. DA Jackson и др. (1998) обнаружили, что соседние источники активируются одновременно в клетках млекопитающих. [48] Пространственное сопоставление сайтов репликации приводит к кластеризации вилок репликации. Кластеризация спасает остановившиеся вилки репликации и способствует нормальному развитию вилок репликации. Развитие репликационных вилок тормозится многими факторами; столкновение с белками или с комплексами, прочно связывающимися с ДНК, дефицит dNTP, разрывы на матричных ДНК и так далее. Если вилки репликации застревают, а остальные последовательности из застрявших вилок не копируются, то дочерние цепи получают нереплицированные сайты. Нереплицированные сайты на одной родительской цепи удерживают вместе другую цепь, но не дочерние цепи. Следовательно, образующиеся сестринские хроматиды не могут отделиться друг от друга и не могут делиться на 2 дочерние клетки. Когда соседние источники срабатывают и вилка из одного источника останавливается, вилка из другого источника получает доступ в направлении, противоположном застопорившейся вилке, и дублирует нереплицированные сайты. Другим механизмом спасения является использование дремлющих источников репликации. что избыточные источники не активируются при нормальной репликации ДНК.
Бактерии
[ редактировать ]
Большинство бактерий не проходят четко определенного клеточного цикла, а вместо этого постоянно копируют свою ДНК; во время быстрого роста это может привести к одновременному выполнению нескольких раундов репликации. [54] У E. coli , наиболее изученных бактерий, репликация ДНК регулируется с помощью нескольких механизмов, в том числе: гемиметилирования и секвестрации исходной последовательности, соотношения аденозинтрифосфата (АТФ) и аденозиндифосфата (АДФ) , а также уровней белка. ДНКА. Все это контролирует связывание белков-инициаторов с исходными последовательностями. [55]
Поскольку E. coli метилирует последовательности ДНК GATC, синтез ДНК приводит к полуметилированным последовательностям. Эта полуметилированная ДНК распознается белком SeqA , который связывает и изолирует исходную последовательность; кроме того, ДНКА (необходимая для инициации репликации) хуже связывается с гемиметилированной ДНК. В результате вновь реплицированные источники не могут немедленно инициировать следующий раунд репликации ДНК. [56]
АТФ накапливается, когда клетка находится в богатой среде, запуская репликацию ДНК, как только клетка достигает определенного размера. АТФ конкурирует с АДФ за связывание с ДНКА, а комплекс ДНКА-АТФ способен инициировать репликацию. Определенное количество белков DnaA также требуется для репликации ДНК — каждый раз, когда копируется источник, количество сайтов связывания для DnaA удваивается, что требует синтеза большего количества DnaA, чтобы обеспечить новую инициацию репликации.
У быстрорастущих бактерий, таких как E. coli , репликация хромосом занимает больше времени, чем деление клетки. Бактерии решают эту проблему, инициируя новый раунд репликации до того, как предыдущий будет завершен. [57] Новый раунд репликации сформирует хромосому клетки, которая рождается через два поколения после делящейся клетки. Этот механизм создает перекрывающиеся циклы репликации.
Проблемы с репликацией ДНК
[ редактировать ]Этот раздел нуждается в расширении . Вы можете помочь, добавив к нему . ( май 2020 г. ) |
Существует множество событий, которые способствуют репликационному стрессу, в том числе: [58]
- Неправильное включение рибонуклеотидов
- Необычные структуры ДНК
- Конфликты между репликацией и транскрипцией
- Недостаточность основных факторов репликации
- Общие хрупкие сайты
- Сверхэкспрессия или конститутивная активация онкогенов
- хроматина Недоступность
Полимеразная цепная реакция
[ редактировать ]Исследователи обычно реплицируют ДНК in vitro, используя полимеразную цепную реакцию (ПЦР). ПЦР использует пару праймеров для охвата целевой области матричной ДНК, а затем полимеризует партнерские цепи в каждом направлении от этих праймеров с помощью термостабильной ДНК-полимеразы . Повторение этого процесса в течение нескольких циклов усиливает целевой участок ДНК. В начале каждого цикла смесь матрицы и праймеров нагревается, отделяя вновь синтезированную молекулу и матрицу. Затем, по мере остывания смеси, оба они становятся матрицами для отжига новых праймеров, и от них распространяется полимераза. В результате количество копий целевого региона удваивается каждый раунд, увеличиваясь в геометрической прогрессии . [59]
См. также
[ редактировать ]- Аутопоэзис
- Клетка (биология)
- Деление клеток
- Расхождение хромосом
- Устройство хранения данных
- Ген
- Экспрессия генов
- Эпигенетика
- Геном
- ДНК Хатимодзи
- Жизнь
- Репликация (вычисления)
- Самовоспроизведение
Примечания
[ редактировать ]- ^ Энергетика этого процесса также может помочь объяснить направленность синтеза: если бы ДНК синтезировалась в направлении от 3' к 5', энергия для процесса поступала бы от 5'-конца растущей цепи, а не от свободных нуклеотидов. Проблема в том, что если бы высокоэнергетические трифосфаты находились на растущей цепи, а не на свободных нуклеотидах, корректура путем удаления несовпадающего терминального нуклеотида была бы проблематичной: как только нуклеотид добавляется, трифосфат теряется, и на ней остается один фосфат. остов между новым нуклеотидом и остальной частью цепи. Если бы добавленный нуклеотид не совпадал, удаление привело бы к тому, что цепь ДНК оканчивалась бы монофосфатом на конце «растущей цепи», а не высокоэнергетическим трифосфатом. Таким образом, прядь застрянет и больше не сможет расти. На самом деле, высокоэнергетические трифосфаты, гидролизующиеся на каждом этапе, происходят из свободных нуклеотидов, а не из полимеризованной цепи, поэтому этой проблемы не существует.
Ссылки
[ редактировать ]- ^ О'Доннелл М., Лэнгстон Л., Стиллман Б. (июль 2013 г.). «Принципы и концепции репликации ДНК у бактерий, архей и эукариев» . Перспективы Колд-Спринг-Харбор в биологии . 5 (7): а010108. doi : 10.1101/cshperspect.a010108 . ПМЦ 3685895 . ПМИД 23818497 .
- ^ Приоло Миннесота, Макалпайн DM (август 2016 г.). «Истоки репликации ДНК – с чего начать?» . Гены и развитие . 30 (15): 1683–1697. дои : 10.1101/gad.285114.116 . ПМК 5002974 . ПМИД 27542827 .
- ^ Дьюар Дж. М., Уолтер Дж. К. (август 2017 г.). «Механизмы терминации репликации ДНК» . Обзоры природы. Молекулярно-клеточная биология . 18 (8): 507–516. дои : 10.1038/номер.2017.42 . ПМК 6386472 . ПМИД 28537574 .
- ^ Сабхадия А (01 марта 2022 г.). «Что такое репликация ДНК и ее этапы?» . Проверено 4 августа 2023 г.
- ^ «ГЕНЕТИКА/РЕПЛИКАЦИЯ ДНК (БАЗОВЫЙ) – Pathwayz» . pathz.org . Проверено 10 декабря 2020 г.
- ^ «двойная спираль» . Изучайте науку в Scitable . Природное образование . Проверено 10 декабря 2020 г.
- ^ Молись, Лос-Анджелес (2008). «Полуконсервативная репликация ДНК; Мезельсон и Шталь» . Природное образование . 1 (1): 98.
- ^ Несовершенная репликация ДНК приводит к мутациям . Берг Дж.М., Тимочко Дж.Л., Страйер Л., Кларк Н.Д. (2002). «Глава 27: Репликация, рекомбинация и репарация ДНК» . Биохимия . WH Фриман и компания. ISBN 0-7167-3051-0 . Архивировано из оригинала 26 марта 2020 г. Проверено 9 августа 2019 г.
- ^ Jump up to: а б Лодиш Х., Берк А., Зипурски С.Л. и др. (2000). «Репликация, репарация и рекомбинация ДНК» . Молекулярно-клеточная биология (4-е изд.). У. Х. Фриман. ISBN 0-7167-3136-3 .
- ^ Jump up to: а б Ху Ю, Стиллман Б (февраль 2023 г.). «Происхождение репликации ДНК у эукариот» . Молекулярная клетка . 83 (3): 352–372. doi : 10.1016/j.molcel.2022.12.024 . ПМЦ 9898300 . ПМИД 36640769 .
- ^ Jump up to: а б Берг Дж.М., Тимочко Дж.Л., Страйер Л., Кларк Н.Д. (2002). «Глава 27, Раздел 4: Репликация обеих цепей ДНК происходит быстро из определенных стартовых сайтов» . Биохимия . WH Фриман и компания. ISBN 0-7167-3051-0 . Архивировано из оригинала 26 марта 2020 г. Проверено 9 августа 2019 г.
- ^ «Что такое геном?» . ваш геном . Проверено 10 декабря 2020 г.
- ^ Чагин, Вадим О.; Стир, Джеффри Х.; Кардозо, М. Кристина (апрель 2010 г.). «Организация репликации ДНК» . Перспективы Колд-Спринг-Харбор в биологии . 2 (4): а000737. doi : 10.1101/cshperspect.a000737 . ISSN 1943-0264 . ПМЦ 2845211 . ПМИД 20452942 .
- ^ Jump up to: а б с Харильо Х., Ибарра Б., Као-Гарсия Ф.Д. (2021). «Репликация ДНК: in vitro анализ и модели данных манипуляций с одиночными молекулами » . Журнал вычислительной и структурной биотехнологии . 19 : 3765–3778. дои : 10.1016/j.csbj.2021.06.032 . ПМЦ 8267548 . ПМИД 34285777 .
- ^ Кюнляйн А., Ланцмих С.А., Браун Д. (март 2021 г.). «Последовательности тРНК могут собираться в репликатор» . электронная жизнь . 10 : е63431. doi : 10.7554/eLife.63431 . ПМЦ 7924937 . ПМИД 33648631 .
- ^ Максимилиан Л. (3 апреля 2021 г.). «Решение проблемы курицы и яйца – «на шаг ближе к реконструкции происхождения жизни» » . СайТехДейли . Проверено 3 апреля 2021 г.
- ^ Фридберг ЕС (январь 2003 г.). «Повреждение и восстановление ДНК» . Природа . 421 (6921): 436–440. Бибкод : 2003Natur.421..436F . дои : 10.1038/nature01408 . ПМИД 12540918 .
- ^ Сабхадия А (13 марта 2023 г.). «Пара оснований: определение, правила в ДНК и РНК» . Проверено 4 августа 2023 г.
- ^ «Функция и структура ДНК (со схемой) (статья)» . Ханская академия . Проверено 10 декабря 2020 г.
- ^ Альбертс Б., Джонсон А., Льюис Дж., Рафф М., Робертс К., Уолтер П. (2002). Молекулярная биология клетки (4-е изд.). Гирляндная наука. стр. 238–240. ISBN 0-8153-3218-1 .
- ^ Эллисон Л. (2007). Фундаментальная молекулярная биология . Издательство Блэквелл. п. 112. ИСБН 978-1-4051-0379-4 .
- ^ Берг Дж.М., Тимочко Дж.Л., Страйер Л., Кларк Н.Д. (2002). Биохимия . WH Фриман и компания. ISBN 0-7167-3051-0 . Глава 27, раздел 2: ДНК-полимеразы требуют матрицы и праймера
- ^ Jump up to: а б Маккалок С.Д., Кункель Т.А. (январь 2008 г.). «Правильность синтеза ДНК с помощью эукариотических полимераз репликативного и транслейкционного синтеза» . Клеточные исследования . 18 (1): 148–161. дои : 10.1038/cr.2008.4 . ПМЦ 3639319 . ПМИД 18166979 .
- ^ Маккарти Д., Миннер С., Бернштейн Х., Бернштейн С. (октябрь 1976 г.). «Скорость элонгации ДНК и распределение точек роста фага Т4 дикого типа и янтарного мутанта с задержкой ДНК». Журнал молекулярной биологии . 106 (4): 963–981. дои : 10.1016/0022-2836(76)90346-6 . ПМИД 789903 .
- ^ Дрейк JW (1970) Молекулярная основа мутации. Холден-Дэй, Сан-Франциско ISBN 0-8162-2450-1 ISBN 978-0-8162-2450-0
- ^ Альбертс Б., Джонсон А., Льюис Дж., Рафф М., Робертс К., Уолтер П. (2002). «Глава 5: Механизмы репликации ДНК» . Молекулярная биология клетки . Гирляндная наука. ISBN 0-8153-3218-1 .
- ^ Вайгель С., Шмидт А., Рюкерт Б., Лурц Р., Мессер В. (ноябрь 1997 г.). «Связывание белка DnaA с отдельными блоками DnaA в точке начала репликации Escherichia coli, oriC» . Журнал ЭМБО . 16 (21): 6574–6583. дои : 10.1093/emboj/16.21.6574 . ПМК 1170261 . ПМИД 9351837 .
- ^ Лодиш Х., Берк А., Зипурски Л.С., Мацудайра П., Балтимор Д., Дарнелл Дж. (2000). Молекулярно-клеточная биология . WH Фриман и компания. ISBN 0-7167-3136-3 . 12.1. Общие особенности хромосомной репликации: три общие черты происхождения репликации
- ^ Лин Ю.К., Прашант С.Г. (июль 2021 г.). «Инициация репликации: последствия для целостности генома» . Восстановление ДНК . 103 : 103131. doi : 10.1016/j.dnarep.2021.103131 . ПМЦ 8296962 . ПМИД 33992866 .
- ^ Амин А., Ву Р., Чунг М.Х., Скотт Дж.Ф., Ван З., Чжоу З. и др. (март 2020 г.). «Основной и зависимый от клеточного цикла цикл димеризации ORC регулирует репликацию эукариотической хромосомной ДНК» . Отчеты по ячейкам . 30 (10): 3323–3338.e6. дои : 10.1016/j.celrep.2020.02.046 . ПМИД 32160540 .
- ^ Чжан Ю, Ю З, Фу Х, Лян С (июнь 2002 г.). «Noc3p, белок bHLH, играет важную роль в инициации репликации ДНК у почкующихся дрожжей» . Клетка . 109 (7): 849–860. дои : 10.1016/s0092-8674(02)00805-x . ПМИД 12110182 .
- ^ Jump up to: а б с д и ж г час я Морган Д.О. (2007). Клеточный цикл: принципы управления . Лондон: New Science Press. стр. 64–75. ISBN 978-0-19-920610-0 . ОСЛК 70173205 .
- ^ Дональдсон А.Д., Рагураман М.К., Фридман К.Л., Кросс Ф.Р., Брюэр Б.Дж., Фэнгман В.Л. (август 1998 г.). «CLB5-зависимая активация начала поздней репликации у S. cerevisiae» . Молекулярная клетка . 2 (2): 173–182. дои : 10.1016/s1097-2765(00)80127-6 . ПМИД 9734354 .
- ^ Аравинд Л., Лейпе Д.Д., Кунин Е.В. (сентябрь 1998 г.). «Топрим - консервативный каталитический домен в топоизомеразах типа IA и II, примазах типа DnaG, нуклеазах семейства OLD и белках RecR» . Исследования нуклеиновых кислот . 26 (18): 4205–4213. дои : 10.1093/нар/26.18.4205 . ПМЦ 147817 . ПМИД 9722641 .
- ^ Фрик Д.Н., Ричардсон CC (июль 2001 г.). «Примасы ДНК». Ежегодный обзор биохимии . 70 : 39–80. doi : 10.1146/annurev.biochem.70.1.39 . ПМИД 11395402 . S2CID 33197061 .
- ^ Барри Э.Р., Белл С.Д. (декабрь 2006 г.). «Репликация ДНК у архей» . Обзоры микробиологии и молекулярной биологии . 70 (4): 876–887. дои : 10.1128/MMBR.00029-06 . ПМК 1698513 . ПМИД 17158702 .
- ^ Стиллман Б. (июль 2015 г.). «Пересмотр ДНК-полимеразы в репликационной вилке у эукариот» . Молекулярная клетка . 59 (2): 139–141. doi : 10.1016/j.molcel.2015.07.004 . ПМЦ 4636199 . ПМИД 26186286 .
- ^ Росси М.Л. (февраль 2009 г.). Различение путей удаления праймера во время созревания фрагмента Оказаки эукариот (кандидатская диссертация). Школа медицины и стоматологии Рочестерского университета. hdl : 1802/6537 .
- ^ Хуберман Дж. А., Риггс А. Д. (март 1968 г.). «О механизме репликации ДНК в хромосомах млекопитающих». Журнал молекулярной биологии . 32 (2): 327–341. дои : 10.1016/0022-2836(68)90013-2 . ПМИД 5689363 .
- ^ Гао Ю, Цуй Ю, Фокс Т, Линь С, Ван Х, де Валь Н и др. (февраль 2019 г.). «Структуры и принципы действия реплисомы» . Наука . 363 (6429): 835. doi : 10.1126/science.aav7003 . ПМК 6681829 . ПМИД 30679383 .
- ^ Альбертс Б., Джонсон А., Льюис Дж., Рафф М., Робертс К., Уолтер П. (2002). «Механизмы репликации ДНК: ДНК-топоизомеразы предотвращают спутывание ДНК во время репликации» . Молекулярная биология клетки . Гирляндная наука. ISBN 0-8153-3218-1 .
- ^ Рис Р.Дж., Максвелл А. (26 сентября 2008 г.). «ДНК-гираза: строение и функции». Критические обзоры по биохимии и молекулярной биологии . 26 (3–4): 335–375. дои : 10.3109/10409239109114072 . ПМИД 1657531 .
- ^ Альбертс Б., Джонсон А., Льюис Дж., Рафф М., Робертс К., Уолтер П. (2002). «Механизмы репликации ДНК: специальные белки помогают раскрыть двойную спираль ДНК перед репликационной вилкой» . Молекулярная биология клетки . Гирляндная наука. ISBN 0-8153-3218-1 .
- ^ Кунин Евгений Владимирович; Крупович, Март; Исино, Соноко; Исино, Ёсидзуми (09.06.2020). «Механизм репликации LUCA: общий источник репликации и транскрипции ДНК» . БМК Биология . 18 (1): 61. дои : 10.1186/s12915-020-00800-9 . ISSN 1741-7007 . ПМЦ 7281927 . ПМИД 32517760 .
- ^ Рэнсом М., Деннехи Б.К., Тайлер Дж.К. (январь 2010 г.). «Сопровождающие гистоны во время репликации и репарации ДНК» . Клетка . 140 (2): 183–195. дои : 10.1016/j.cell.2010.01.004 . ПМЦ 3433953 . ПМИД 20141833 .
- ^ Гриффитс А.Дж., Весслер С.Р., Левонтин Р.К., Кэрролл С.Б. (2008). Введение в генетический анализ . WH Фриман и компания. ISBN 978-0-7167-6887-6 . [Глава 7: ДНК: структура и репликация. стр. 283–290]
- ^ Кларк Дж (11 мая 2009 г.). «Помешает ли предел Хейфлика нам жить вечно?» . Как все работает . Проверено 20 января 2015 г.
- ^ Jump up to: а б с д и ж Мейстер П., Таддей А., Гассер С.М. (июнь 2006 г.). «Вход и выход из фабрики репликации» . Клетка . 125 (7): 1233–5. дои : 10.1016/j.cell.2006.06.014 . ПМИД 16814710 . S2CID 15397410 .
- ^ Уотсон Дж.Д., Бейкер Т.А., Белл С.П., Ганн А., Левин М., Лосик Р., Инглис CH (2008). Молекулярная биология гена (6-е изд.). Сан-Франциско: Пирсон/Бенджамин Каммингс. п. 237. ИСБН 978-0-8053-9592-1 .
- ^ Браун Т.А. (2002). «Глава 13.2.3. Прекращение репликации» . Геномы . Научные издательства БИОС . ISBN 1-85996-228-9 .
- ^ Альбертс Б., Джонсон А., Льюис Дж., Рафф М., Робертс К., Уолтер П. (2002). «Внутриклеточный контроль событий клеточного цикла: комплексы S-фазы циклин-Cdk (S-Cdks) инициируют репликацию ДНК один раз за цикл» . Молекулярная биология клетки . Гирляндная наука. ISBN 0-8153-3218-1 .
- ^ Браун Т.А. (2002). «Глава 13: Репликация генома» . Геномы (2-е изд.). Оксфорд: Вили-Лисс.
- ^ Нгуен VQ, Co C, Ли JJ (июнь 2001 г.). «Циклин-зависимые киназы предотвращают повторную репликацию ДНК с помощью нескольких механизмов». Природа . 411 (6841): 1068–1073. Бибкод : 2001Natur.411.1068N . дои : 10.1038/35082600 . ПМИД 11429609 . S2CID 4393812 .
- ^ Тобиасон Д.М., Зайферт Х.С. (июнь 2006 г.). «Облигатный человеческий патоген Neisseria gonorrhoeae является полиплоидным» . ПЛОС Биология . 4 (6): е185. doi : 10.1371/journal.pbio.0040185 . ПМК 1470461 . ПМИД 16719561 .
- ^ О'Доннелл, Майкл; Лэнгстон, Лэнс; Стиллман, Брюс (01 июля 2013 г.). «Принципы и концепции репликации ДНК у бактерий, архей и эукариев» . Перспективы Колд-Спринг-Харбор в биологии . 5 (7): а010108. doi : 10.1101/cshperspect.a010108 . ISSN 1943-0264 . ПМЦ 3685895 . ПМИД 23818497 .
- ^ Слейтер С., Уолд С., Лу М., Бой Э., Скарстад К., Клекнер Н. (сентябрь 1995 г.). «Белок SeqA E. coli связывает oriC в двух различных метилмодулированных реакциях, соответствующих его роли в инициации репликации ДНК и секвестрации начала» . Клетка . 82 (6): 927–936. дои : 10.1016/0092-8674(95)90272-4 . ПМИД 7553853 . S2CID 14652024 .
- ^ Купер С., Хелмстеттер CE (февраль 1968 г.). «Репликация хромосом и цикл деления Escherichia coli B/r». Журнал молекулярной биологии . 31 (3): 519–540. дои : 10.1016/0022-2836(68)90425-7 . ПМИД 4866337 .
- ^ Земан М.К., Цимприч К.А. (январь 2014 г.). «Причины и последствия репликационного стресса» . Природная клеточная биология . 16 (1): 2–9. дои : 10.1038/ncb2897 . ПМЦ 4354890 . ПМИД 24366029 .
- ^ Сайки Р.К., Гельфанд Д.Х., Стоффель С., Шарф С.Дж., Хигучи Р., Хорн Г.Т. и др. (январь 1988 г.). «Праймер-направленная ферментативная амплификация ДНК с помощью термостабильной ДНК-полимеразы». Наука . 239 (4839): 487–491. Бибкод : 1988Sci...239..487S . дои : 10.1126/science.239.4839.487 . ПМИД 2448875 .