Максимальное уникальное совпадение
Эта статья нуждается в дополнительных цитатах для проверки . ( май 2021 г. ) |
Максимальное уникальное совпадение , или MUM сокращенно , является частью ключевого шага. [1] в множественном выравнивании последовательностей геномов биологии в вычислительной . Идентификация MUM и других потенциальных якорей является первым шагом в более крупных системах согласования, таких как MUMmer . Якоря — это области между двумя геномами, где они очень похожи. Чтобы понять, что такое МАМА, каждое слово в аббревиатуре можно разобрать по отдельности. Совпадение подразумевает, что подстрока встречается в обеих выравниваемых последовательностях. Уникальность означает, что подстрока встречается в каждой последовательности только один раз. Наконец, «максимум» утверждает, что подстрока не является частью другой более крупной строки, которая удовлетворяет обоим предыдущим требованиям. Идея, лежащая в основе этого, заключается в том, что длинные последовательности, которые точно совпадают и встречаются только один раз в каждом геноме, почти наверняка являются частью глобального выравнивания.
Формальное определение
[ редактировать ]«Для двух геномов A и B подстрока максимального уникального совпадения (MUM) представляет собой общую подстроку A и B, длина которой превышает указанную минимальную длину d (по умолчанию d = 20), такую что
- он максимальный, то есть его нельзя расширить с любого конца, не вызывая несоответствия и
- оно уникально в обеих последовательностях" [2]
Алгоритм
[ редактировать ]Идентификация набора MUM в двух очень длинных последовательностях генома не является тривиальной с вычислительной точки зрения задачей. [1] Существует несколько алгоритмических способов идентификации MUM при множественном выравнивании последовательностей. Самый простой и медленный метод — использование грубой силы, при котором для каждого индекса i в геноме A и каждого индекса j в геноме B вы вычисляете самый длинный общий префикс (P) для A[i...n] и B[j... м] . Затем вы должны гарантировать, что длина P равна как минимум d , где d — указанный минимальный размер MUM. Наконец, вы должны убедиться, что P уникален в обоих геномах. Таким образом, сложность метода грубой силы составляет O(mn) . [2]
идентифицируются путем построения обобщенного суффиксного дерева для A и B. На самом деле MUM Затем создается список для всех внутренних узлов ровно с одним дочерним элементом каждой последовательности генома. Для каждого из этих узлов (мы будем идентифицировать детей из генома A как i и детей из генома B как j ) проверяем, что A[i-1] ≠ B[j-1] и для тех, где это условие выполняется, мы знаем, что это МАМА. В этом случае сложность снижается до O(m+n) . [2]
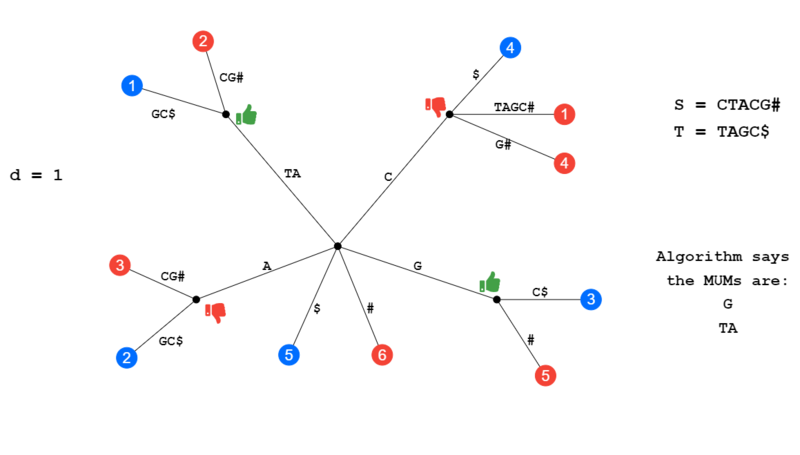
На рисунке ниже, учитывая начальные строки S и T и значение d, равное 1, MUM должны быть G и TA. Красный лист означает, что лист произошел из строки S, а синий лист обозначает T. строку Внутренний узел в A был отброшен, поскольку A[i-1] = B[j-1] означает, что символ, стоящий перед обоими A, идентичен (T), это условие, когда последовательности принадлежат к более крупной уникальной последовательности. Внутренний узел C отбрасывается, поскольку у него есть два потомка от S. В результате у нас остаются MUM G и TA.
Пример
[ редактировать ]Чтобы проиллюстрировать MUM, мы возьмем следующий пример. Допустим, d=3 и наши два генома выглядят следующим образом:
S = CTACTGTCATGCTGAAGTCATGCGGCTGGCTTAT#
T = CTGGTGTGATGCTAAGTCATGCGGTCTGGCTTAT$
Прорабатывая последовательность, наша первая MUM, удовлетворяющая условиям, происходит здесь:
S = CTAC TGT CATGCTGAAGTCATGCGGCTGGCTTAT#
T = CTGG TGT GATGCTAAGTCATGCGGTCTGGCTTAT$
TGT имеет длину не менее d, встречается только один раз в обеих последовательностях, а символы слева и справа различаются в разных геномах. Чтобы проиллюстрировать пример, когда расширение необходимо, чтобы гарантировать, что наша MUM не является частью более крупной последовательности и является уникальной, возьмем следующее:
S = CTAC TGT C ATG CTGAAGTCATGCGGCTGGCTTAT#
T = CTGG TGT G ATG CTAAGTCATGCGGTCTGGCTTAT$
В случае тестирования ATG как MUM возникают две проблемы, поскольку ATG не уникален и также является частью более крупной подпоследовательности, как показано здесь:
S = CTAC TGT C ATG CTGAAGTC ATGC GGCTGGCTTAT#
T = CTGG TGT G ATG CTAAGTC ATGC GGTCTGGCTTAT$
Следовательно, необходимо расширение этой последовательности в попытке удовлетворить условиям существования MUM, как показано здесь.
S = CTAC TGT C ATGCT GAAGTC ATGCG GCTGGCTTAT#
T = CTGG TGT G ATGCT AAGTC ATGCG GTCTGGCTTAT$
Использование этого метода итеративно создает конечный продукт, где каждый MUM идентифицируется для ясности, каждый MUM имеет цветовую маркировку:
S = CTAC TGT C ATGCT G AAGTCATGCGG CTGGCTTAT #
T = ACTT TGT G ATGCT AAGTCATGCGG T CTGGCTTAT $
Максимальное точное совпадение (MEM)
[ редактировать ]MUM являются подмножеством более крупного набора, называемого максимальными точными совпадениями или MEMS. В MEM условие уникальности MUM смягчено. MEM подобны локальному выравниванию , но в этом случае идентифицируют только последовательности, в которых нет пробелов.
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Jump up to: а б Делчер, Алабама; Касиф, С.; Флейшманн, РД; Петерсон, Дж.; Уайт, О.; Зальцберг, СЛ (1999). «Выравнивание целых геномов» . Исследования нуклеиновых кислот . 27 (11): 2369–2376. дои : 10.1093/нар/30.11.2478 . ПМЦ 117189 . ПМИД 10325427 .
- ^ Jump up to: а б с Винг-Кин, Сун (2010). Алгоритмы в биоинформатике: практическое введение (первое изд.). Бока-Ратон: Chapman & Hall/CRC Press. ISBN 978-1420070330 .