Совершенная филогения
Совершенная филогения — это термин, используемый в вычислительной филогенетике для обозначения филогенетического дерева , в котором все внутренние узлы могут быть помечены таким образом, что все признаки развиваются вниз по дереву без гомоплазии . То есть характеристики не соответствуют эволюционной конвергенции и не имеют аналогичных структур. Статистически это можно представить как предка, имеющего состояние «0» во всех характеристиках, где 0 означает отсутствие этой характеристики. Каждая из этих характеристик меняется от 0 до 1 ровно один раз и никогда не возвращается в состояние 0. Реальные данные редко соответствуют концепции идеальной филогении. [1] [2]
Здание
[ редактировать ]В общем, существует два разных типа данных, которые используются при построении филогенетического дерева. При вычислениях на основе расстояний филогенетическое дерево создается путем анализа взаимосвязей между расстоянием между видами и длинами ребер соответствующего дерева. При использовании подхода, основанного на признаках, состояния персонажей разных видов используются в качестве входных данных в попытке найти наиболее «идеальное» филогенетическое дерево. [3] [4]
Статистические компоненты идеального филогенетического дерева лучше всего можно описать следующим образом: [3]
Совершенная филогения для размера n x M матрицы состояний символов m представляет собой корневое дерево T с n листьями, удовлетворяющее:
я. Каждая строка M обозначает ровно один лист T.
ii. Каждый столбец M обозначает ровно одно ребро T.
iii. Каждое внутреннее ребро T помечено хотя бы одним столбцом M.iv. Символы, связанные с ребрами вдоль уникального пути от корня до листа v, точно определяют вектор символов v , т. е. вектор символов имеет запись 1 во всех столбцах, соответствующих символам, связанным с ребрами пути, и запись 0 в противном случае.
Стоит отметить, что очень редко можно найти реальные филогенетические данные, которые соответствуют концепциям и ограничениям, подробно описанным здесь. Поэтому часто бывает, что исследователи вынуждены идти на компромисс, разрабатывая деревья, которые просто пытаются минимизировать гомоплазию, находя набор совместимых символов максимальной мощности или создавая филогении, которые как можно ближе соответствуют разделам, подразумеваемым символами.
Пример
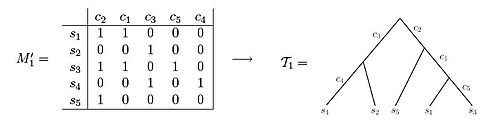
[ редактировать ]Оба этих набора данных иллюстрируют примеры матриц состояний символов . Используя матрицу M' 1, можно заметить, что результирующее филогенетическое дерево может быть создано так, что каждый из символов обозначает ровно одно ребро дерева. Напротив, наблюдая за матрицей M' 2 , можно увидеть, что невозможно построить филогенетическое дерево так, чтобы каждый символ обозначал только одну длину ребра. [3] Если образцы взяты из данных о частоте вариантов аллелей (VAF) популяции исследуемых клеток, записи в матрице символов представляют собой частоты мутаций и принимают значение от 0 до 1. А именно, если представляет позицию в геноме, то запись, соответствующая и образец будет хранить частоты геномов в образце с мутацией в положении . [5] [6] [7] [8] [9]
- Матрицы состояний символов
- Пример матрицы персонажей, которую можно изобразить как идеальную филогению.
Использование
[ редактировать ]Совершенная филогения — это теоретическая основа, которую также можно использовать в более практических методах. Одним из таких примеров является неполная направленная совершенная филогения. Эта концепция предполагает использование идеальных филогений с реальными и, следовательно, неполными и несовершенными наборами данных. Такой метод использует SINE для определения эволюционного сходства. Эти короткие вкрапленные элементы присутствуют во многих геномах и могут быть идентифицированы по их фланкирующим последовательностям. SINE предоставляют информацию о наследовании определенных признаков у разных видов. К сожалению, если SINE отсутствует, трудно узнать, присутствовали ли эти SINE до удаления. Используя алгоритмы, полученные на основе идеальных данных филогении, мы можем попытаться реконструировать филогенетическое дерево, несмотря на эти ограничения. [10]
Совершенная филогения также используется при построении карт гаплотипов . Используя концепции и алгоритмы, описанные в идеальной филогении, можно определить информацию об отсутствующих и недоступных данных гаплотипов. [11] Предполагая, что набор гаплотипов, полученных в результате картирования генотипов, соответствует и соответствует концепции идеальной филогении (а также другим предположениям, таким как идеальное менделевское наследование и тот факт, что на каждый SNP существует только одна мутация), можно сделать вывод: отсутствуют данные о гаплотипах. [12] [13] [14] [15]
Вывод о филогении на основе зашумленных данных VAF в рамках PPM является сложной проблемой. [5] Большинство инструментов вывода включают в себя некоторый эвристический этап, делающий вывод вычислительно управляемым. Примеры инструментов, которые выводят филогении на основе зашумленных данных VAF, включают AncesTree, Canopy, CITUP, EXACT и PhyloWGS. [5] [6] [7] [8] [9] В частности, EXACT выполняет точный вывод, используя графические процессоры для вычисления апостериорной вероятности для всех возможных деревьев для задач небольшого размера. Расширения PPM были сделаны с помощью сопутствующих инструментов. [16] [17] Например, такие инструменты, как MEDICC, TuMult и FISHtrees, позволяют как увеличивать, так и уменьшать количество копий данного генетического элемента, или плоидности, тем самым эффективно позволяя удалять мутации. [18] [19] [20]
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Фернандес-Бака Д. «Проблема идеальной филогении» (PDF) . Академическое издательство Kluwer . Проверено 30 сентября 2012 г.
- ^ Наклех Л., Ринге Д., Варноу Т. «Совершенные филогенетические сети: новая методология реконструкции эволюционной истории естественных языков» (PDF) . Проверено 1 октября 2012 года .
- ^ Перейти обратно: а б с Улер С. «В поисках идеальной филогении» (PDF) . Архивировано из оригинала (PDF) 4 марта 2016 года . Проверено 29 сентября 2012 г.
- ^ Никайдо М., Руни А.П., Окада Н. (август 1999 г.). «Филогенетические взаимоотношения среди китодактилей, основанные на вставках коротких и длинных вкраплений элементов: бегемоты — ближайшие современные родственники китов» . Труды Национальной академии наук Соединенных Штатов Америки . 96 (18): 10261–6. Бибкод : 1999PNAS...9610261N . дои : 10.1073/pnas.96.18.10261 . ЧВК 17876 . ПМИД 10468596 .
- ^ Перейти обратно: а б с Эль-Кебир М., Оспер Л., Ачесон-Филд Х., Рафаэль Б.Дж. (июнь 2015 г.). «Реконструкция клональных деревьев и состава опухолей на основе данных секвенирования нескольких образцов» . Биоинформатика . 31 (12): i62-70. doi : 10.1093/биоинформатика/btv261 . ПМЦ 4542783 . ПМИД 26072510 .
- ^ Перейти обратно: а б Сатас Дж., Рафаэль Б.Дж. (июль 2017 г.). «Выводы о филогении опухолей с использованием выборки по важности с ограничением по дереву» . Биоинформатика . 33 (14): i152–i160. doi : 10.1093/биоинформатика/btx270 . ПМК 5870673 . ПМИД 28882002 .
- ^ Перейти обратно: а б Маликич С., Макферсон А.В., Донмез Н., Сахинальп К.С. (май 2015 г.). «Вывод клональности в нескольких образцах опухолей с использованием филогении» . Биоинформатика . 31 (9): 1349–56. doi : 10.1093/биоинформатика/btv003 . ПМИД 25568283 .
- ^ Перейти обратно: а б Рэй С., Джиа Б., Сафави С., ван Опийнен Т., Исберг Р., Рош Дж., Бенто Дж. (22 августа 2019 г.). «Точный вывод в рамках идеальной модели филогении». arXiv : 1908.08623 [ q-bio.QM ].
- ^ Перейти обратно: а б Дешвар А.Г., Вембу С., Юнг К.К., Джанг Г.Х., Стейн Л., Моррис К. (февраль 2015 г.). «PhyloWGS: реконструкция субклонального состава и эволюции на основе полногеномного секвенирования опухолей» . Геномная биология . 16 (1): 35. дои : 10.1186/s13059-015-0602-8 . ПМЦ 4359439 . ПМИД 25786235 .
- ^ Пеер И., Пупко Т., Шамир Р., Шаран Р. «Неполная направленная совершенная филогения» . Тель-Авивский университет. Архивировано из оригинала 20 октября 2013 года . Проверено 30 октября 2012 г.
{{cite web}}: CS1 maint: bot: исходный статус URL неизвестен ( ссылка ) - ^ Эскин Э., Гальперин Э., Карп Р.М. (апрель 2003 г.). «Эффективная реконструкция структуры гаплотипов посредством идеальной филогении» (PDF) . Журнал биоинформатики и вычислительной биологии . 1 (1). Калифорнийский университет в Беркли: 1–20. дои : 10.1142/S0219720003000174 . ПМИД 15290779 . Проверено 30 октября 2012 г.
- ^ Гасфилд Д. «Обзор вычислительных методов вывода гаплотипов» (PDF) . Калифорнийский университет в Дэвисе . Проверено 18 ноября 2012 г.
- ^ Дин З., Фильков В., Гасфилд Д. «Алгоритм линейного времени для решения задачи гаплотипирования идеальной филогении» . Калифорнийский университет в Дэвисе . Проверено 18 ноября 2012 г.
- ^ Бафна В., Гасфилд Д., Лансия Г., Юзеф С. (2003). «Гаплотипирование как совершенная филогения: прямой подход». Журнал вычислительной биологии . 10 (3–4): 323–40. дои : 10.1089/10665270360688048 . ПМИД 12935331 .
- ^ Сеялиоглу Х. «Гаплотипирование как совершенная филогения» (PDF) . Архивировано из оригинала (PDF) 30 сентября 2011 года . Проверено 30 октября 2012 г.
- ^ Бониццони П., Каррьери А.П., Делла Ведова Дж., Трукко Дж. (октябрь 2014 г.). «Объяснение эволюции через ограниченную постоянную совершенную филогению» . БМК Геномика . 15 Приложение 6 (S6): S10. дои : 10.1186/1471-2164-15-S6-S10 . ПМК 4240218 . ПМИД 25572381 .
- ^ Хаджирасулиха И., Рафаэль Б.Дж. (2014), Браун Д., Моргенштерн Б. (ред.), «Реконструкция истории мутаций в множественных опухолях с использованием идеальных филогенных смесей», Алгоритмы в биоинформатике , Конспекты лекций по информатике, том. 8701, Springer Berlin Heidelberg, стр. 354–367, doi : 10.1007/978-3-662-44753-6_27 , ISBN 9783662447529
- ^ Шварц Р.Ф., Тринь А., Сипос Б., Брентон Дж.Д., Голдман Н., Марковец Ф. (апрель 2014 г.). Беренвинкель Н. (ред.). «Филогенетическая количественная оценка внутриопухолевой гетерогенности» . PLOS Вычислительная биология . 10 (4): e1003535. arXiv : 1306.1685 . Бибкод : 2014PLSCB..10E3535S . дои : 10.1371/journal.pcbi.1003535 . ПМЦ 3990475 . ПМИД 24743184 .
- ^ Летузе Э., Аллори Ю., Болле М.А., Радвани Ф., Гийон Ф. (2010). «Анализ профилей числа копий нескольких образцов опухолей от одного и того же пациента выявляет последовательные этапы онкогенеза» . Геномная биология . 11 (7): С76. дои : 10.1186/gb-2010-11-7-r76 . ПМЦ 2926787 . ПМИД 20649963 .
- ^ Герц Э.М., Чоудхури С.А., Ли В.Дж., Вангса Д., Хесельмейер-Хаддад К., Рид Т. и др. (30 июня 2016 г.). «FISHtrees 3.0: Филогенетика опухолей с использованием зонда плоидности» . ПЛОС ОДИН . 11 (6): e0158569. Бибкод : 2016PLoSO..1158569G . дои : 10.1371/journal.pone.0158569 . ПМЦ 4928784 . ПМИД 27362268 .
Внешние ссылки
[ редактировать ]- Одна из нескольких программ, доступных для анализа и создания филогенетических деревьев.
- Еще одна такая программа для анализа филогенетического дерева
- Дополнительная программа для анализа деревьев. Архивировано 14 марта 2018 г. на Wayback Machine.
- Статья, подробно описывающая пример того, как совершенную филогению можно использовать за пределами области генетики, например, в языковых ассоциациях.
- Github для «Алгоритма реконструкции клонального дерева на основе данных секвенирования рака из нескольких образцов» (AncesTree)
- Github за «Доступ к внутриопухолевой гетерогенности и отслеживание продольной и пространственной истории клональной эволюции с помощью секвенирования следующего поколения» (Canopy)
- Github для «Вывода клональности в опухолях с использованием филогении» (CITUP)
- Github для «Точного вывода в рамках идеальной модели филогении» (EXACT)
- Github: «Реконструкция субклонального состава и эволюции на основе полногеномного секвенирования опухолей» (PhyloWGS)