я замышляю
В вычислительной биологии график MA представляет собой применение графика Бланда-Альтмана для визуального представления геномных данных. График визуализирует различия между измерениями, выполненными в двух образцах, путем преобразования данных в шкалы M (логарифмическое соотношение) и A ( среднее среднее ), а затем нанесения этих значений на график. Хотя первоначально графики МА применялись в контексте данных об экспрессии генов на двухканальном ДНК-микрочипе , они также используются для визуализации высокопроизводительного анализа секвенирования. [1] [2]
Объяснение
[ редактировать ]Данные микрочипов часто нормализуются внутри массивов для контроля систематических отклонений в связывании красителей и эффективности гибридизации, а также других технических отклонений в зондах ДНК и печатающем наконечнике, используемом для определения массива. [3] Минимизируя эти систематические различия, можно обнаружить истинные биологические различия. Чтобы определить, нужна ли нормализация, можно построить график Cy5 интенсивностей (R) в зависимости от интенсивностей Cy3 (G) и посмотреть, составляет ли наклон линии около 1. Улучшенный метод, который по сути представляет собой масштабированное вращение на 45 градусов R против График G представляет собой график MA. [4] MA-график представляет собой график распределения соотношения интенсивностей красного и зеленого («M»), построенный по средней интенсивности («A»). M и A определяются следующими уравнениями.
Таким образом, M представляет собой двоичный логарифм отношения интенсивностей (или разницы между логарифмическими интенсивностями), а A — среднюю логарифмическую интенсивность для точки на графике. Затем графики MA используются для визуализации зависящего от интенсивности соотношения необработанных данных микрочипов (микрочипы обычно показывают здесь смещение: более высокое значение A приводит к более высокому |M |, т.е. чем ярче пятно, тем более вероятно наблюдаемое различие между образцом и контролем). На графике MA переменная M помещается на ось Y , а переменная A на ось X и дает быстрый обзор распределения данных .
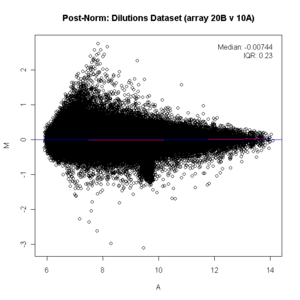
Во многих экспериментах по экспрессии генов на микрочипах в основе лежит предположение, что экспрессия большинства генов не изменится; следовательно, большинство точек на оси y ( M ) будут расположены в 0, поскольку log(1) равен 0. Если это не так, то метод нормализации , такой как LOESS. к данным перед этим следует применить статистический анализ. (На диаграмме ниже вы видите красную линию, идущую ниже нулевой отметки до нормализации, она должна быть прямой. Поскольку она не прямая, данные должны быть нормализованы. После нормализации красная линия проходит прямо на нулевой линии и отображается как розовый/черный.)
Пакеты
[ редактировать ]Несколько Bioconductor пакетов для программного обеспечения R предоставляют возможность создания графиков МА. К ним относятся affy (ma.plot, mva.pairs), limma (plotMA), marray (maPlot) и EdgeR(maPlot).
Подобные графики «RA» можно создать с помощью функции raPlot в Caroline CRAN R. пакете
Интерактивный график MA для фильтрации генов по значениям M, A и p, поиска по именам или с помощью лассо и сохранения выбранных генов доступен в виде R-Shiny кода Enhanced-MA-Plot .
Пример на языке программирования R
[ редактировать ]library(affy)
if (require(affydata))
{
data(Dilution)
}
y <- (exprs(Dilution)[, c("20B", "10A")])
x11()
ma.plot( rowMeans(log2(y)), log2(y[, 1])-log2(y[, 2]), cex=1 )
title("Dilutions Dataset (array 20B v 10A)")
library(preprocessCore)
#do a quantile normalization
x <- normalize.quantiles(y)
x11()
ma.plot( rowMeans(log2(x)), log2(x[, 1])-log2(x[, 2]), cex=1 )
title("Post Norm: Dilutions Dataset (array 20B v 10A)")
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Робинсон, доктор медицины; Маккарти, диджей; Смит, ГК (11 ноября 2009 г.). «edgeR: пакет Bioconductor для дифференциального анализа экспрессии цифровых данных об экспрессии генов» . Биоинформатика . 26 (1): 139–140. doi : 10.1093/биоинформатика/btp616 . ПМК 2796818 . ПМИД 19910308 .
- ^ С любовью, Михаил I; Хубер, Вольфганг; Андерс, Саймон (5 декабря 2014 г.). «Умеренная оценка кратности изменения и дисперсии данных секвенирования РНК с помощью DESeq2» . Геномная биология . 15 (12): 550. дои : 10.1186/s13059-014-0550-8 . ПМК 4302049 . ПМИД 25516281 .
- ^ YH Ян , С. Дудуа , П. Луу, Д.М. Лин, В. Пэн, Дж. Нгай, TP Speed . (2002). Нормализация данных микрочипов кДНК: надежный комплексный метод, учитывающий систематические вариации на одном и нескольких слайдах. Исследования нуклеиновых кислот, том. 30 (4) с. e15.
- ^ Дюдуа, С. , Ян, Ю.Х. , Кэллоу, М.Дж., Спид, Т.П. (2002). Статистические методы идентификации дифференциально экспрессируемых генов в экспериментах с реплицируемыми микрочипами кДНК. Стат. Грех. 12:1 111–139