секвенирование ДНК
| Часть серии о |
| Генетика |
|---|
 |
Секвенирование ДНК — это процесс определения последовательности нуклеиновой кислоты — порядка нуклеотидов в ДНК . Он включает в себя любой метод или технологию, которая используется для определения порядка четырех оснований: аденина , гуанина , цитозина и тимина . Появление методов быстрого секвенирования ДНК значительно ускорило биологические и медицинские исследования и открытия. [1] [2]
Знание последовательностей ДНК стало незаменимым для фундаментальных биологических исследований, ДНК-генографических проектов и во многих прикладных областях, таких как медицинская диагностика , биотехнология , судебная биология , вирусология и биологическая систематика . Сравнивая здоровые и мутировавшие последовательности ДНК, можно диагностировать различные заболевания, включая различные виды рака. [3] охарактеризовать репертуар антител, [4] и может использоваться для руководства лечением пациентов. [5] Наличие быстрого способа секвенирования ДНК позволяет оказывать более быструю и индивидуализированную медицинскую помощь, а также идентифицировать и каталогизировать больше организмов. [4]
Быстрая скорость секвенирования, достигаемая с помощью современной технологии секвенирования ДНК, сыграла важную роль в секвенировании полных последовательностей ДНК или геномов многочисленных типов и видов жизни, включая геном человека и другие полные последовательности ДНК многих животных, растений и микробов. разновидность.

Первые последовательности ДНК были получены в начале 1970-х годов академическими исследователями с помощью трудоемких методов, основанных на двумерной хроматографии . После разработки методов флуоресцентного секвенирования с помощью секвенатора ДНК , [6] Секвенирование ДНК стало проще и на порядки быстрее. [7] [8]
Приложения
[ редактировать ]Секвенирование ДНК может использоваться для определения последовательности отдельных генов , более крупных генетических областей (т.е. кластеров генов или оперонов ), полных хромосом или целых геномов любого организма. Секвенирование ДНК также является наиболее эффективным способом косвенного секвенирования РНК или белков (через их открытые рамки считывания ). Фактически, секвенирование ДНК стало ключевой технологией во многих областях биологии и других наук, таких как медицина, судебная экспертиза и антропология .
Молекулярная биология
[ редактировать ]Секвенирование используется в молекулярной биологии для изучения геномов и белков, которые они кодируют. Информация, полученная с помощью секвенирования, позволяет исследователям выявлять изменения в генах и некодирующей ДНК (включая регуляторные последовательности), ассоциации с заболеваниями и фенотипами, а также идентифицировать потенциальные мишени лекарств.
Эволюционная биология
[ редактировать ]Поскольку ДНК является информативной макромолекулой с точки зрения передачи от одного поколения к другому, секвенирование ДНК используется в эволюционной биологии для изучения того, как разные организмы связаны между собой и как они развивались. В феврале 2021 года ученые впервые сообщили о секвенировании ДНК останков животных , в данном случае мамонта , возрастом более миллиона лет, самой старой ДНК, секвенированной на сегодняшний день. [9] [10]
Метагеномика
[ редактировать ]Область метагеномики включает в себя идентификацию организмов, присутствующих в водоеме, сточных водах , грязи, мусоре, отфильтрованном из воздуха, или образцах мазков от организмов. Знание того, какие организмы присутствуют в конкретной среде, имеет решающее значение для исследований в области экологии , эпидемиологии , микробиологии и других областей. секвенирование позволяет исследователям определить, какие типы микробов могут присутствовать в микробиоме Например, .
Вирусология
[ редактировать ]Поскольку большинство вирусов слишком малы, чтобы их можно было увидеть в световой микроскоп, секвенирование является одним из основных инструментов в вирусологии для идентификации и изучения вируса. [11] Вирусные геномы могут быть основаны на ДНК или РНК. РНК-вирусы более чувствительны ко времени для секвенирования генома, поскольку они быстрее разлагаются в клинических образцах. [12] Традиционное секвенирование по Сэнгеру и секвенирование нового поколения используются для секвенирования вирусов в фундаментальных и клинических исследованиях, а также для диагностики новых вирусных инфекций, молекулярной эпидемиологии вирусных патогенов и тестирования на лекарственную устойчивость. хранится более 2,3 миллиона уникальных вирусных последовательностей В GenBank . [11] Недавно NGS превзошел традиционный метод Сэнгера как самый популярный подход к созданию вирусных геномов. [11]
Во время вспышки птичьего гриппа в 1990 году секвенирование вируса показало, что подтип гриппа возник в результате реассортации между перепелами и домашней птицей. Это привело к принятию в Гонконге закона , запрещающего продажу на рынке вместе живых перепелов и домашней птицы. Секвенирование вируса также можно использовать для оценки момента начала вирусной вспышки с помощью метода молекулярных часов . [12]
Лекарство
[ редактировать ]Медицинские техники могут секвенировать гены (или, теоретически, полные геномы) пациентов, чтобы определить, существует ли риск генетических заболеваний. Это форма генетического тестирования , хотя некоторые генетические тесты могут не включать секвенирование ДНК.
С 2013 года секвенирование ДНК все чаще используется для диагностики и лечения редких заболеваний. Поскольку выявляется все больше и больше генов, вызывающих редкие генетические заболевания, молекулярная диагностика пациентов становится все более распространенной. Секвенирование ДНК позволяет клиницистам выявлять генетические заболевания, улучшать лечение заболеваний, предоставлять репродуктивное консультирование и назначать более эффективные методы лечения. [13] Панели секвенирования генов используются для выявления множества потенциальных генетических причин предполагаемого заболевания. [14]
Кроме того, секвенирование ДНК может быть полезно для определения конкретных бактерий, чтобы обеспечить более точное лечение антибиотиками , тем самым снижая риск создания устойчивости к противомикробным препаратам в популяциях бактерий. [15] [16] [17] [18] [19] [20]
Судебное расследование
[ редактировать ]Секвенирование ДНК может использоваться наряду с методами профилирования ДНК для судебно-медицинской идентификации. [21] и тестирование на отцовство . Тестирование ДНК значительно изменилось за последние несколько десятилетий, чтобы в конечном итоге связать отпечаток ДНК с тем, что расследуется. Образцы ДНК в отпечатках пальцев, слюне, волосяных фолликулах и т. д. однозначно отличают каждый живой организм от другого. Тестирование ДНК — это метод, позволяющий обнаружить определенные геномы в цепи ДНК для создания уникального и индивидуального рисунка.
Четыре канонические основы
[ редактировать ]Каноническая структура ДНК имеет четыре основания: тимин (Т), аденин (А), цитозин (Ц) и гуанин (G). Секвенирование ДНК — это определение физического порядка этих оснований в молекуле ДНК. Однако существует множество других оснований, которые могут присутствовать в молекуле. У некоторых вирусов (в частности, бактериофага ) цитозин может быть заменен гидроксиметиловым или гидроксиметилглюкозоцитозиновым. [22] В ДНК млекопитающих варианты оснований с метильными группами или фосфосульфатом. могут быть обнаружены [23] [24] В зависимости от метода секвенирования конкретная модификация, например, распространенная у людей 5mC ( 5-метилцитозин ), может быть обнаружена или не обнаружена. [25]
Почти у всех организмов ДНК синтезируется in vivo с использованием только четырех канонических оснований; модификация, которая происходит после репликации, создает другие основания, такие как 5 метил С. Однако некоторые бактериофаги могут напрямую включать нестандартное основание. [26]
Помимо модификаций, ДНК постоянно подвергается воздействию факторов окружающей среды, таких как УФ-излучение и радикалы кислорода. В настоящее время наличие таких поврежденных оснований не обнаруживается большинством методов секвенирования ДНК, хотя PacBio опубликовала об этом публикации. [27]
История
[ редактировать ]Открытие структуры и функции ДНК
[ редактировать ]Дезоксирибонуклеиновая кислота ( ДНК ) была впервые обнаружена и выделена Фридрихом Мишером в 1869 году, но она оставалась недостаточно изученной в течение многих десятилетий, поскольку считалось, что белки, а не ДНК, поддерживают генетическую схему жизни. Эта ситуация изменилась после 1944 года в результате некоторых экспериментов Освальда Эйвери , Колина Маклауда и Маклина Маккарти, продемонстрировавших, что очищенная ДНК может превращать один штамм бактерий в другой. Впервые было показано, что ДНК способна изменять свойства клеток.
В 1953 году Джеймс Уотсон и Фрэнсис Крик выдвинули свою модель двойной спирали ДНК, основанную на кристаллизованных рентгеновских структурах, изучаемых Розалиндой Франклин . Согласно модели, ДНК состоит из двух нитей нуклеотидов, скрученных друг вокруг друга, связанных между собой водородными связями и идущих в противоположных направлениях. Каждая цепь состоит из четырех комплементарных нуклеотидов – аденина (А), цитозина (С), гуанина (G) и тимина (Т) – причем А на одной цепи всегда соединена с Т на другой, а С всегда в паре с G. Они предположили, что такая структура позволяет использовать каждую нить для реконструкции другой — идея, центральная для передачи наследственной информации между поколениями. [28]

Основа секвенирования белков была впервые заложена работой Фредерика Сэнгера , который к 1955 году завершил последовательность всех аминокислот в инсулине , небольшом белке, секретируемом поджелудочной железой. Это стало первым убедительным доказательством того, что белки представляют собой химические образования с определенной молекулярной структурой, а не случайную смесь веществ, взвешенных в жидкости. Успех Сэнгера в секвенировании инсулина вдохновил рентгеновских кристаллографов, в том числе Уотсона и Крика, которые к тому времени пытались понять, как ДНК управляет образованием белков внутри клетки. Вскоре после посещения серии лекций, прочитанных Фредериком Сэнгером в октябре 1954 года, Крик начал разрабатывать теорию, утверждавшую, что расположение нуклеотидов в ДНК определяет последовательность аминокислот в белках, что, в свою очередь, помогает определить функцию белка. Он опубликовал эту теорию в 1958 году. [29]
секвенирование РНК
[ редактировать ]Секвенирование РНК было одной из самых ранних форм секвенирования нуклеотидов. Главной вехой в секвенировании РНК является последовательность первого полного гена и полного генома бактериофага MS2 , идентифицированные и опубликованные Уолтером Фирсом и его коллегами из Гентского университета ( Гент , Бельгия ) в 1972 году. [30] и 1976 год. [31] Традиционные методы секвенирования РНК требуют создания молекулы кДНК , которую необходимо секвенировать. [32]
Ранние методы секвенирования ДНК
[ редактировать ]Первый метод определения последовательностей ДНК включал стратегию локализованного удлинения праймеров, разработанную Рэем Ву из Корнельского университета в 1970 году. [33] Катализ ДНК-полимеразы и специфическое мечение нуклеотидов, оба из которых занимают видное место в современных схемах секвенирования, использовались для секвенирования когезивных концов ДНК фага лямбда. [34] [35] [36] Между 1970 и 1973 годами Ву, Р. Падманабхан и его коллеги продемонстрировали, что этот метод можно использовать для определения любой последовательности ДНК с использованием синтетических праймеров, специфичных для местоположения. [37] [38] [8] Затем Фредерик Сэнгер применил эту стратегию удлинения праймера для разработки более быстрых методов секвенирования ДНК в MRC Центре в Кембридже , Великобритания, и в 1977 году опубликовал метод «секвенирования ДНК с ингибиторами обрыва цепи». [39] Уолтер Гилберт и Аллан Максам из Гарварда также разработали методы секвенирования, в том числе метод «секвенирования ДНК путем химической деградации». [40] [41] В 1973 году Гилберт и Максам сообщили о последовательности из 24 пар оснований, используя метод, известный как анализ блуждающих пятен. [42] Достижениям в секвенировании способствовало одновременное развитие технологии рекомбинантной ДНК , позволяющей изолировать образцы ДНК из источников, отличных от вирусов.
Секвенирование полных геномов
[ редактировать ]
Первым полным геномом ДНК, секвенированным в 1977 году, был геном бактериофага φX174 . [43] Ученые Совета медицинских исследований расшифровали полную последовательность ДНК вируса Эпштейна-Барра в 1984 году, обнаружив, что она содержит 172 282 нуклеотида. Завершение секвенирования стало важным поворотным моментом в секвенировании ДНК, поскольку оно было достигнуто без предварительного знания генетического профиля вируса. [44] [8]
Нерадиоактивный метод переноса молекул ДНК из реакционных смесей секвенирования на иммобилизирующую матрицу во время электрофореза был разработан Гербертом Полем и его сотрудниками в начале 1980-х годов. [45] [46] За этим последовала коммерциализация секвенатора ДНК «Direct-Blotting-Electrophoresis-System GATC 1500» компании GATC Biotech , который интенсивно использовался в рамках программы секвенирования генома ЕС, полная последовательность ДНК дрожжевых Saccharomyces cerevisiae хромосомы II. [47] Лероя Э. Худа Лаборатория в Калифорнийском технологическом институте объявила о выпуске первой полуавтоматической машины для секвенирования ДНК в 1986 году. [48] За этим последовал маркетинг Applied Biosystems первого полностью автоматизированного секвенатора ABI 370 в 1987 году и Dupont Genesis 2000. [49] в котором использовалась новая техника флуоресцентного мечения, позволяющая идентифицировать все четыре дидезоксинуклеотида на одной дорожке. К 1990 году Национальные институты здравоохранения США (NIH) начали крупномасштабные испытания по секвенированию Mycoplasma capricolum , Escherichia coli , Caenorhabditis elegans и Saccharomyces cerevisiae по цене 0,75 доллара США за основу. началось секвенирование последовательностей кДНК человека , называемых метками экспрессируемых последовательностей Тем временем в лаборатории Крейга Вентера , — попытка уловить кодирующую часть генома человека . [50] В 1995 году Вентер, Гамильтон Смит и коллеги из Института геномных исследований (TIGR) опубликовали первый полный геном свободноживущего организма, бактерии Haemophilus influenzae . Круговая хромосома содержит 1 830 137 оснований, и ее публикация в журнале Science [51] ознаменовало первое опубликованное использование полногеномного секвенирования, устраняющее необходимость в первоначальных усилиях по картированию.
К 2001 году методы дробового секвенирования были использованы для создания чернового варианта последовательности человеческого генома. [52] [53]
Методы высокопроизводительного секвенирования (HTS)
[ редактировать ]
Несколько новых методов секвенирования ДНК были разработаны в середине-конце 1990-х годов и к 2000 году были реализованы в коммерческих секвенаторах ДНК . Вместе они назывались методами секвенирования «следующего поколения» или «второго поколения» (NGS), чтобы различать их с помощью более ранних методов, включая секвенирование по Сэнгеру . В отличие от первого поколения секвенирования, технология NGS обычно характеризуется высокой масштабируемостью, позволяя секвенировать весь геном одновременно. Обычно это достигается путем фрагментации генома на небольшие части, случайной выборки фрагмента и его секвенирования с использованием одной из множества технологий, например описанных ниже. Целый геном возможен, потому что несколько фрагментов секвенируются одновременно (что дало этому название «массивно-параллельное» секвенирование) в автоматизированном процессе.
Технология NGS дала огромные возможности исследователям в поиске информации о здоровье, антропологам в изучении происхождения человека и стала катализатором движения « Персонализированной медицины ». Однако это также открыло больше возможностей для ошибок. Существует множество программных инструментов для проведения вычислительного анализа данных NGS, часто скомпилированных на онлайн-платформах, таких как CSI NGS Portal, каждый из которых имеет свой собственный алгоритм. Даже параметры в одном программном пакете могут изменить результат анализа. Кроме того, большие объемы данных, полученные с помощью секвенирования ДНК, также потребовали разработки новых методов и программ для анализа последовательностей. Для решения этих проблем было предпринято несколько попыток разработать стандарты в области NGS, большинство из которых были небольшими усилиями, предпринимаемыми отдельными лабораториями. Совсем недавно масштабная организованная работа, финансируемая FDA, завершилась созданием стандарта BioCompute .
26 октября 1990 года Роджер Цзиен , Пепи Росс, Маргарет Фэнсток и Аллан Дж. Джонстон подали патент, описывающий пошаговое («основание за основанием») секвенирование с использованием удаляемых 3'-блокаторов на массивах ДНК (блоты и отдельные молекулы ДНК). [55] В 1996 году Пол Нюрен и его студент Мостафа Ронаги из Королевского технологического института в Стокгольме опубликовали свой метод пиросеквенирования . [56]
1 апреля 1997 года Паскаль Майер и Лоран Фаринелли подали во Всемирную организацию интеллектуальной собственности патенты, описывающие секвенирование колоний ДНК. [57] Методы подготовки образцов ДНК и рандомизированной поверхностно- полимеразной цепной реакции (ПЦР), описанные в этом патенте, в сочетании с методом последовательного секвенирования Roger Tsien et al., теперь реализованы в Illumina. Hi-Seq компании секвенаторы генома.
В 1998 году Фил Грин и Брент Юинг из Вашингтонского университета описали свою оценку качества phred для анализа данных секвенатора: [58] метод ориентировочного анализа, получивший широкое распространение и до сих пор являющийся наиболее распространенным показателем для оценки точности платформы секвенирования. [59]
В 2000 году компания Lynx Therapeutics опубликовала и продала массовое параллельное сигнатурное секвенирование (MPSS). Этот метод включал в себя параллельную технологию секвенирования на основе шариков, опосредованную адаптером/лигированием, и стал первым коммерчески доступным методом секвенирования «следующего поколения», хотя Секвенаторы ДНК были проданы независимым лабораториям. [60]
Основные методы
[ редактировать ]Секвенирование Максама-Гилберта
[ редактировать ]Аллан Максам и Уолтер Гилберт в 1977 году опубликовали метод секвенирования ДНК, основанный на химической модификации ДНК и последующем расщеплении по определенным основаниям. [40] Этот метод, также известный как химическое секвенирование, позволял использовать очищенные образцы двухцепочечной ДНК без дальнейшего клонирования. Использование в этом методе радиоактивной маркировки и его техническая сложность препятствовали его широкому использованию после того, как методы Сэнгера были усовершенствованы.
Секвенирование Максама-Гилберта требует радиоактивной метки на одном 5'-конце ДНК и очистки фрагмента ДНК, подлежащего секвенированию. Затем химическая обработка приводит к разрывам небольшой части одного или двух из четырех нуклеотидных оснований в каждой из четырех реакций (G, A+G, C, C+T). Концентрацию модифицирующих химикатов контролируют так, чтобы в среднем внести одну модификацию на молекулу ДНК. Таким образом генерируется серия меченых фрагментов, от меченого радиоактивным изотопом конца до первого «разрезанного» сайта в каждой молекуле. Фрагменты в четырех реакциях подвергают электрофорезу рядом в денатурирующих акриламидных гелях для разделения по размерам. Для визуализации фрагментов гель подвергают рентгеновской пленке для авторадиографии, в результате чего образуется ряд темных полос, каждая из которых соответствует меченому радиоактивным изотопом фрагменту ДНК, по которому можно определить последовательность. [40]
Этот метод практически устарел по состоянию на 2023 год. [61]
Методы завершения цепи
[ редактировать ]Метод обрыва цепи, разработанный Фредериком Сэнгером и его коллегами в 1977 году, вскоре стал предпочтительным методом благодаря его относительной простоте и надежности. [39] [62] На момент изобретения метод терминатора цепи использовал меньше токсичных химикатов и меньшее количество радиоактивности, чем метод Максама и Гилберта. Из-за своей сравнительной простоты метод Сэнгера вскоре был автоматизирован и стал методом, использованным в первом поколении секвенаторов ДНК .
Секвенирование по Сэнгеру — это метод, который преобладал с 1980-х до середины 2000-х годов. За этот период в этой технике были достигнуты большие успехи, такие как флуоресцентное мечение, капиллярный электрофорез и общая автоматизация. Эти разработки позволили гораздо более эффективно секвенировать, что привело к снижению затрат. Метод Сэнгера в форме массового производства — это технология, которая позволила создать первый геном человека в 2001 году, ознаменовав начало эры геномики . Однако позже в том же десятилетии на рынок вышли радикально иные подходы, в результате чего стоимость генома снизилась со 100 миллионов долларов в 2001 году до 10 000 долларов в 2011 году. [63]
Секвенирование путем синтеза
[ редактировать ]Целью последовательного секвенирования путем синтеза (SBS) является определение последовательности образца ДНК путем обнаружения включения нуклеотида ДНК - полимеразой . Сконструированная полимераза используется для синтеза копии одной цепи ДНК и контролируется включение каждого нуклеотида. Принцип секвенирования в реальном времени путем синтеза был впервые описан в 1993 году. [64] с улучшениями, опубликованными несколько лет спустя. [65] Ключевые части очень схожи для всех вариантов SBS и включают (1) амплификацию ДНК (для усиления последующего сигнала) и прикрепление ДНК, подлежащей секвенированию, к твердой подложке, (2) генерацию одноцепочечной ДНК на твердой подложке. , (3) включение нуклеотидов с использованием сконструированной полимеразы и (4) обнаружение включения нуклеотида в реальном времени. Шаги 3-4 повторяются, и последовательность собирается из сигналов, полученных на этапе 4. Этот принцип в реальном времени Секвенирование-синтезом использовалось практически для всех параллельного секвенирования массовых инструментов , включая 454 , PacBio , IonTorrent , Illumina и MGI .
Крупномасштабное секвенирование и de novo . секвенирование
[ редактировать ]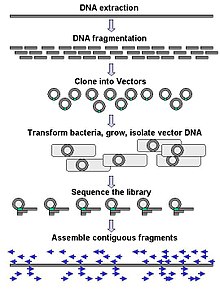
Крупномасштабное секвенирование часто направлено на секвенирование очень длинных фрагментов ДНК, таких как целые хромосомы , хотя крупномасштабное секвенирование также может использоваться для создания очень большого количества коротких последовательностей, например, обнаруженных при фаговом дисплее . Для более длинных мишеней, таких как хромосомы, общие подходы заключаются в разрезании (с помощью ферментов рестрикции ) или разрезании (с помощью механических сил) больших фрагментов ДНК на более короткие фрагменты ДНК. Фрагментированную ДНК можно затем клонировать в ДНК-вектор и амплифицировать в бактериальном хозяине, таком как Escherichia coli . Короткие фрагменты ДНК, выделенные из отдельных бактериальных колоний, индивидуально секвенируются и собираются электронным способом в одну длинную непрерывную последовательность. Исследования показали, что добавление этапа выбора размера для сбора фрагментов ДНК одинакового размера может повысить эффективность секвенирования и точность сборки генома. В этих исследованиях автоматическое определение размера оказалось более воспроизводимым и точным, чем ручное определение размера геля. [66] [67] [68]
Термин « секвенирование de novo » конкретно относится к методам, используемым для определения последовательности ДНК без ранее известной последовательности. De novo переводится с латыни как «с самого начала». Пробелы в собранной последовательности могут быть заполнены путем прохождения праймера . Различные стратегии имеют разные компромиссы в скорости и точности; Методы «дробовика» часто используются для секвенирования больших геномов, но их сборка сложна и трудна, особенно из-за повторов последовательности, часто вызывающих пробелы в сборке генома.
Большинство подходов к секвенированию используют этап клонирования in vitro для амплификации отдельных молекул ДНК, поскольку их методы молекулярного обнаружения недостаточно чувствительны для секвенирования одной молекулы. Эмульсионная ПЦР [69] изолирует отдельные молекулы ДНК вместе с шариками, покрытыми праймером, в водных каплях в масляной фазе. Затем в ходе полимеразной цепной реакции (ПЦР) каждая гранула покрывается клональными копиями молекулы ДНК с последующей иммобилизацией для последующего секвенирования. Эмульсионная ПЦР используется в методах, разработанных Marguilis et al. (коммерциализация осуществляется компанией 454 Life Sciences ), Shendure and Porreca et al. (также известное как « секвенирование полоний ») и секвенирование SOLiD (разработанное Agencourt , позже Applied Biosystems , теперь Life Technologies ). [70] [71] [72] Эмульсионный ПЦР также используется в платформах GemCode и Chromium, разработанных 10x Genomics . [73]
Последовательность дробовика
[ редактировать ]Секвенирование дробовиком — это метод секвенирования, предназначенный для анализа последовательностей ДНК длиной более 1000 пар оснований, включая целые хромосомы. Этот метод требует, чтобы целевая ДНК была разбита на случайные фрагменты. После секвенирования отдельных фрагментов с использованием метода терминации цепи последовательности можно собрать заново на основе их перекрывающихся участков. [74]
Высокопроизводительные методы
[ редактировать ]
Высокопроизводительное секвенирование, включающее методы секвенирования «короткого считывания» нового поколения и «длинного считывания» третьего поколения. [нет 1] применяется к секвенированию экзома , секвенированию генома, повторному секвенированию генома, профилированию транскриптома ( RNA-Seq ), взаимодействиям ДНК-белок ( ChIP-секвенирование ) и характеристике эпигенома . [75]
Высокий спрос на недорогое секвенирование привел к развитию технологий высокопроизводительного секвенирования, которые распараллеливают процесс секвенирования, создавая тысячи или миллионы последовательностей одновременно. [76] [77] [78] Технологии высокопроизводительного секвенирования призваны снизить стоимость секвенирования ДНК по сравнению с тем, что возможно при использовании стандартных методов красителя-терминатора. [79] При сверхвысокопроизводительном секвенировании можно параллельно выполнять до 500 000 операций секвенирования путем синтеза. [80] [81] [82] Такие технологии позволили секвенировать весь геном человека всего за один день. [83] По состоянию на 2019 год [update]Среди корпоративных лидеров в разработке продуктов для высокопроизводительного секвенирования были Illumina , Qiagen и ThermoFisher Scientific . [83]
| Метод | Длина чтения | Точность (однократное чтение, не консенсус) | Чтение за запуск | Время за прогон | Стоимость за 1 миллиард баз (в долларах США) | Преимущества | Недостатки |
|---|---|---|---|---|---|---|---|
| Секвенирование одиночных молекул в реальном времени (Pacific Biosciences) | 30000 п.о. ( N50 ); | Точность чтения 87 % [89] | 4 000 000 на ячейку SMRT Sequel 2, 100–200 гигабаз [86] [90] [91] | от 30 минут до 20 часов [86] [92] | $7.2-$43.3 | Быстрый. Обнаруживает 4мК, 5мК, 6мА. [93] | Умеренная пропускная способность. Оборудование может быть очень дорогим. |
| Ионный полупроводник (ионно-торрентное секвенирование) | до 600 б.п. [94] | 99.6% [95] | до 80 миллионов | 2 часа | $66.8-$950 | Менее дорогое оборудование. Быстрый. | Ошибки гомополимера. |
| Пиросеквенирование (454) | 700 б.п. | 99.9% | 1 миллион | 24 часа | $10,000 | Размер длинного чтения. Быстрый. | Пробежки стоят дорого. Ошибки гомополимера. |
| Секвенирование путем синтеза (Illumina) | MiniSeq, NextSeq: 75–300 п.н.; MiSeq: 50–600 п.о.; HiSeq 2500: 50–500 п.н.; HiSeq 3/4000: 50–300 п.о.; HiSeq X: 300 п.о. | 99,9% (Фред30) | MiniSeq/MiSeq: 1–25 миллионов; NextSeq: 130-00 миллионов; HiSeq 2500: 300 миллионов – 2 миллиарда; HiSeq 3/4000 2,5 миллиарда; HiSeq X: 3 миллиарда | От 1 до 11 дней, в зависимости от секвенатора и указанной длины чтения [96] | от 5 до 150 долларов | Потенциал для получения высокого выхода секвенирования в зависимости от модели секвенатора и желаемого применения. | Оборудование может быть очень дорогим. Требует высоких концентраций ДНК. |
| Комбинаторный синтез якоря зонда (cPAS-BGI/MGI) | BGISEQ-50: 35-50 п.н.; MGISEQ 200: 50-200 п.н.; BGISEQ-500, MGISEQ-2000: 50-300bp [97] | 99,9% (Фред30) | БГИСЭК-50: 160М; МГИСЭК 200: 300М; BGISEQ-500: 1300M на проточную кювету; MGISEQ-2000: проточная кювета 375M FCS, проточная кювета 1500M FCL на каждую проточную кювету. | От 1 до 9 дней в зависимости от прибора, длины считывания и количества проточных кювет, работающих одновременно. | $5– $120 | ||
| Секвенирование путем лигирования (SOLiD-секвенирование) | 50+35 или 50+50 б.п. | 99.9% | от 1,2 до 1,4 миллиарда | от 1 до 2 недель | $60–130 | Низкая стоимость за базу. | Медленнее, чем другие методы. Имеет проблемы с секвенированием палиндромных последовательностей. [98] |
| Нанопоровое секвенирование | Зависит от подготовки библиотеки, а не от устройства, поэтому пользователь выбирает длину чтения (сообщается до 2 272 580 п.н.). [99] ). | ~92–97% однократного чтения | зависит от длины чтения, выбранной пользователем | данные передаются в режиме реального времени. Выберите от 1 минуты до 48 часов | $7–100 | Самое длинное индивидуальное чтение. Доступное сообщество пользователей. Портативный (размером с ладонь). | Пропускная способность ниже, чем у других машин. Точность однократного считывания за 90 с. |
| Секвенирование GenapSys | Около 150 bp, односторонний | 99,9% (Фред30) | от 1 до 16 миллионов | Около 24 часов | $667 | Низкая стоимость инструмента (10 000 долларов США). | |
| Обрыв цепи (секвенирование по Сэнгеру) | от 400 до 900 пар оснований | 99.9% | Н/Д | от 20 минут до 3 часов | $2,400,000 | Полезно для многих приложений. | Более дорогой и непрактичный для крупных проектов секвенирования. Этот метод также требует трудоемкого этапа клонирования плазмиды или ПЦР. |
Методы секвенирования длительного чтения
[ редактировать ]Секвенирование одной молекулы в реальном времени (SMRT)
[ редактировать ]Секвенирование SMRT основано на подходе секвенирования путем синтеза. ДНК синтезируется в нулевых волноводах (ZMW) – небольших колодцеобразных контейнерах с инструментами для захвата, расположенными на дне колодца. Секвенирование проводят с использованием немодифицированной полимеразы (прикрепленной к нижней части ZMW) и флуоресцентно-меченных нуклеотидов, свободно перемещающихся в растворе. Лунки сконструированы таким образом, что детектируется только флуоресценция, возникающая на дне лунки. Флуоресцентная метка отделяется от нуклеотида при его включении в цепь ДНК, оставляя немодифицированную цепь ДНК. По данным компании Pacific Biosciences (PacBio), разработчика технологии SMRT, эта методология позволяет обнаруживать модификации нуклеотидов (например, метилирование цитозина). Это происходит путем наблюдения за кинетикой полимеразы. Этот подход позволяет читать 20 000 и более нуклеотидов со средней длиной чтения 5 килобаз. [90] [100] В 2015 году компания Pacific Biosciences объявила о выпуске нового инструмента для секвенирования под названием Sequel System с 1 миллионом ZMW по сравнению со 150 000 ZMW в приборе PacBio RS II. [101] [102] Секвенирование SMRT называется секвенированием « третьего поколения » или «длинным чтением».
Нанопоровое секвенирование ДНК
[ редактировать ]ДНК, проходя через нанопору, меняет свой ионный ток. Это изменение зависит от формы, размера и длины последовательности ДНК. Каждый тип нуклеотида блокирует поток ионов через пору на разный период времени. Метод не требует модифицированных нуклеотидов и выполняется в режиме реального времени. Секвенирование нанопор наряду с секвенированием SMRT называют секвенированием « третьего поколения » или «длинным чтением».
Ранние промышленные исследования этого метода были основаны на методе, называемом «экзонуклеазное секвенирование», при котором считывание электрических сигналов происходило, когда нуклеотиды проходили через поры альфа(α)-гемолизина, ковалентно связанные с циклодекстрином . [103] Однако последующий коммерческий метод, «секвенирование цепи», позволил секвенировать основания ДНК в неповрежденной цепи.
Двумя основными областями секвенирования нанопор, находящихся в разработке, являются твердотельное секвенирование нанопор и секвенирование нанопор на основе белков. Для секвенирования нанопор белков используются мембранные белковые комплексы, такие как α-гемолизин, MspA ( Mycobacterium smegmatis Porin A) или CssG, которые демонстрируют большие перспективы, учитывая их способность различать отдельные нуклеотиды и группы нуклеотидов. [104] Напротив, при секвенировании твердотельных нанопор используются синтетические материалы, такие как нитрид кремния и оксид алюминия, и он предпочтителен из-за его превосходных механических свойств, а также термической и химической стабильности. [105] Метод изготовления имеет важное значение для этого типа секвенирования, поскольку массив нанопор может содержать сотни пор диаметром менее восьми нанометров. [104]
Эта концепция возникла из идеи о том, что одноцепочечные молекулы ДНК или РНК могут быть электрофоретически пропущены в строгой линейной последовательности через биологическую пору, размер которой может составлять менее восьми нанометров, и могут быть обнаружены, поскольку молекулы выделяют ионный ток при движении через пора. Пора содержит область обнаружения, способную распознавать различные основания, причем каждое основание генерирует различные сигналы, зависящие от времени, соответствующие последовательности оснований, когда они пересекают пору, которые затем оцениваются. [105] Точный контроль над транспортом ДНК через поры имеет решающее значение для успеха. Различные ферменты, такие как экзонуклеазы и полимеразы, использовались для замедления этого процесса, располагая их возле входа в пору. [106]
Методы секвенирования короткого чтения
[ редактировать ]Массивно-параллельное секвенирование подписей (MPSS)
[ редактировать ]Первая из технологий высокопроизводительного секвенирования, массово-параллельное сигнатурное секвенирование (или MPSS, также называемое секвенированием следующего поколения), была разработана в 1990-х годах в компании Lynx Therapeutics, основанной в 1992 году Сиднеем Бреннером и Сэмом Элетром . MPSS представлял собой метод на основе шариков, в котором использовался сложный подход лигирования адаптера с последующим декодированием адаптера, считывающим последовательность с шагом в четыре нуклеотида. Этот метод сделал его чувствительным к систематической ошибке, связанной с конкретной последовательностью, или потере конкретных последовательностей. Поскольку технология была настолько сложной, MPSS выполнялась только «внутренними» компаниями Lynx Therapeutics, и никакие машины для секвенирования ДНК не продавались независимым лабораториям. В 2004 году компания Lynx Therapeutics объединилась с компанией Solexa (позже приобретенной Illumina ), что привело к развитию метода секвенирования путем синтеза, более простого подхода, приобретенного у Manteia Predictive Medicine , что сделало MPSS устаревшим. Однако основные свойства выходных данных MPSS были типичными для более поздних типов данных с высокой пропускной способностью, включая сотни тысяч коротких последовательностей ДНК. В случае MPSS их обычно использовали для секвенирования. кДНК для измерения уровней экспрессии генов . [60]
Секвенирование полонии
[ редактировать ]Метод секвенирования полоний , разработанный в лаборатории Джорджа М. Чёрча в Гарварде, был одной из первых высокопроизводительных систем секвенирования и использовался для секвенирования полного генома E. coli в 2005 году. [107] Он объединил библиотеку парных меток in vitro с эмульсионной ПЦР, автоматизированным микроскопом и химическим методом секвенирования на основе лигирования для секвенирования генома E. coli с точностью> 99,9999% и стоимостью примерно 1/9 от стоимости секвенирования по Сэнгеру. [107] Лицензия на технологию была передана компании Agencourt Biosciences, впоследствии передана в Agencourt Personal Genomics и в конечном итоге включена в платформу SOLiD Applied Biosystems . Позднее компания Applied Biosystems была приобретена компанией Life Technologies , ныне являющейся частью Thermo Fisher Scientific .
454 пиросеквенирование
[ редактировать ]Параллельная версия пиросеквенирования была разработана компанией 454 Life Sciences , которая впоследствии была приобретена Roche Diagnostics . Этот метод амплифицирует ДНК внутри капель воды в масляном растворе (эмульсионная ПЦР), при этом каждая капля содержит одну матрицу ДНК, прикрепленную к одной покрытой праймером бусине, которая затем образует клональную колонию. Секвенатор содержит множество лунок пиколитрового объема, каждая из которых содержит одну гранулу и секвенирующие ферменты. При пиросеквенировании используется люцифераза для генерации света для обнаружения отдельных нуклеотидов, добавленных к зарождающейся ДНК, а объединенные данные используются для генерации считываний последовательности . [70] Эта технология обеспечивает промежуточную длину считывания и цену за базу по сравнению с секвенированием Сэнгера, с одной стороны, и Solexa и SOLiD, с другой. [79]
Секвенирование Illumina (Solexa)
[ редактировать ]Компания Solexa , ныне являющаяся частью Illumina , была основана Шанкаром Баласубраманяном и Дэвидом Кленерманом в 1998 году и разработала метод секвенирования, основанный на технологии обратимых терминаторов красителя и сконструированных полимеразах. [108] Концепция обратимой терминированной химии была изобретена Бруно Канардом и Саймоном Сарфати в Институте Пастера в Париже. [109] [110] Он был разработан внутри компании Solexa лицами, указанными в соответствующих патентах. В 2004 году Solexa приобрела компанию Manteia Predictive Medicine , чтобы получить технологию массово-параллельного секвенирования, изобретенную в 1997 году Паскалем Майером и Лораном Фаринелли. [57] Он основан на «кластерах ДНК» или «колониях ДНК», что предполагает клональную амплификацию ДНК на поверхности. Кластерная технология была приобретена совместно с Lynx Therapeutics из Калифорнии. Позже Solexa Ltd. объединилась с Lynx и образовала Solexa Inc.


В этом методе молекулы ДНК и праймеры сначала прикрепляются к предметному стеклу или проточной ячейке и амплифицируются с помощью полимеразы, так что образуются локальные колонии клональной ДНК, позже получившие название «кластеры ДНК». Для определения последовательности добавляются четыре типа обратимых терминаторных оснований (RT-оснований) и отмываются невключенные нуклеотиды. Камера делает изображения флуоресцентно меченных нуклеотидов. Затем краситель вместе с блокатором концевого 3'-конца химически удаляется из ДНК, позволяя начать следующий цикл. В отличие от пиросеквенирования, цепи ДНК удлиняются по одному нуклеотиду за раз, и получение изображений может выполняться с задержкой, что позволяет захватывать очень большие массивы колоний ДНК с помощью последовательных изображений, полученных с одной камеры.

Разделение ферментативной реакции и захвата изображения обеспечивает оптимальную производительность и теоретически неограниченную мощность секвенирования. Таким образом, при оптимальной конфигурации в конечном итоге достижимая производительность прибора определяется исключительно скоростью аналого-цифрового преобразования камеры, умноженной на количество камер и разделенной на количество пикселей на колонию ДНК, необходимое для их оптимальной визуализации (приблизительно 10 пикселей/колония). В 2012 году с камерами, работающими на частоте аналого-цифрового преобразования более 10 МГц, и доступной оптикой, жидкостной и ферментативной аппаратурой, пропускная способность может быть кратна 1 миллиону нуклеотидов в секунду, что соответствует примерно 1 эквиваленту генома человека при 1x охвате в час на один инструмент. и 1 геном человека, повторно секвенированный (приблизительно 30 раз) в день на каждый инструмент (оснащенный одной камерой). [111]
Комбинаторный синтез якоря зонда (cPAS)
[ редактировать ]Этот метод представляет собой модернизированную модификацию технологии комбинаторного лигирования якоря зонда (cPAL), описанной Complete Genomics. [112] которая с тех пор стала частью китайской геномной компании BGI в 2013 году. [113] Обе компании усовершенствовали технологию, чтобы обеспечить большую длину считывания, сократить время реакции и ускорить получение результатов. Кроме того, данные теперь генерируются как последовательные полноразмерные чтения в стандартном формате файлов FASTQ и могут использоваться без изменений в большинстве конвейеров биоинформатического анализа на основе короткого чтения. [114] [ нужна ссылка ]
Две технологии, которые составляют основу этой высокопроизводительной технологии секвенирования, — это наношарики ДНК (DNB) и структурированные массивы для прикрепления наношариков к твердой поверхности. [112] Наношарики ДНК формируются путем денатурации двухцепочечных библиотек, лигированных с помощью адаптера, и лигирования прямой цепи только с шплинтовым олигонуклеотидом с образованием кольца оцДНК. Точные копии кругов, содержащих вставку ДНК, создаются с использованием амплификации по катящемуся кругу, которая генерирует примерно 300–500 копий. Длинная цепь оцДНК складывается сама по себе, образуя трехмерную структуру наношаров диаметром примерно 220 нм. Создание DNB заменяет необходимость создания ПЦР-копий библиотеки в проточной кювете и, как таковое, может устранить большую часть повторяющихся считываний, лигирования адаптер-адаптер и ошибок, вызванных ПЦР. [114] [ нужна ссылка ]

Узорчатый массив положительно заряженных пятен изготавливается с помощью методов фотолитографии и травления с последующей химической модификацией для создания проточной ячейки для секвенирования. Каждое пятно на проточной кювете имеет диаметр примерно 250 нм, разделено на 700 нм (от центра к центру) и позволяет легко прикрепить один отрицательно заряженный DNB к проточной кювете и, таким образом, уменьшить недостаточную или чрезмерную кластеризацию на проточной кювете. [112] [ нужна ссылка ]
Затем выполняется секвенирование путем добавления олигонуклеотидного зонда, который в комбинации прикрепляется к определенным сайтам в DNB. Зонд действует как якорь, который затем позволяет одному из четырех одиночных обратимо инактивированных меченых нуклеотидов связываться после прохождения через проточную кювету. Несвязанные нуклеотиды смываются перед лазерным возбуждением прикрепленных меток, затем излучают флуоресценцию, и сигнал улавливается камерами, который преобразуется в цифровой выход для определения оснований. Прикрепленное основание имеет терминатор и метку, химически расщепляемую по завершении цикла. Цикл повторяется с другим потоком свободных меченых нуклеотидов через проточную ячейку, чтобы позволить следующему нуклеотиду связаться и захватить его сигнал. Этот процесс выполняется несколько раз (обычно от 50 до 300 раз) для определения последовательности вставленного фрагмента ДНК со скоростью примерно 40 миллионов нуклеотидов в секунду по состоянию на 2018 год. [ нужна ссылка ]
SOLiD-секвенирование
[ редактировать ]

Технология SOLiD компании Applied Biosystems (теперь бренд Life Technologies ) использует секвенирование путем лигирования . Здесь пул всех возможных олигонуклеотидов фиксированной длины помечен в соответствии с положением секвенирования. Олигонуклеотиды отжигают и лигируют; предпочтительное лигирование ДНК-лигазой для совпадающих последовательностей приводит к получению сигнала, информативного о нуклеотиде в этом положении. Каждое основание в шаблоне секвенируется дважды, а полученные данные декодируются в соответствии со схемой кодирования двух оснований, используемой в этом методе. Перед секвенированием ДНК амплифицируют с помощью эмульсионной ПЦР. Полученные шарики, каждая из которых содержит отдельные копии одной и той же молекулы ДНК, наносятся на предметное стекло. [115] В результате получаются последовательности величин и длин, сравнимые с секвенированием Illumina. [79] что такое секвенирование методом лигирования вызывает некоторые проблемы с секвенированием палиндромных последовательностей. Сообщалось, [98]
Ионно-торрентное полупроводниковое секвенирование
[ редактировать ]Ion Torrent Systems Inc. (теперь принадлежащая Life Technologies ) разработала систему, основанную на использовании стандартной химии секвенирования, но с новой системой обнаружения на основе полупроводников. Этот метод секвенирования основан на обнаружении ионов водорода , которые высвобождаются во время , в полимеризации ДНК отличие от оптических методов, используемых в других системах секвенирования. Микролунка, содержащая матричную цепь ДНК, подлежащую секвенированию, заполняется нуклеотидом одного типа . Если введенный нуклеотид комплементарен ведущему матричному нуклеотиду, он включается в растущую комплементарную цепь. Это вызывает высвобождение ионов водорода, который запускает сверхчувствительный ионный датчик, указывающий на то, что реакция произошла. Если в матричной последовательности присутствуют гомополимерные повторы, в один цикл будут включены несколько нуклеотидов. Это приводит к соответствующему количеству высвободившихся атомов водорода и пропорционально более высокому электронному сигналу. [116]

Секвенирование ДНК наношариками
[ редактировать ]Секвенирование ДНК наношариками — это тип технологии высокопроизводительного секвенирования, используемый для определения всей геномной последовательности организма. Компания Complete Genomics использует эту технологию для секвенирования образцов, представленных независимыми исследователями. Этот метод использует репликацию по катящемуся кругу для амплификации небольших фрагментов геномной ДНК в наношарики ДНК. Затем для определения нуклеотидной последовательности используют несцепленное секвенирование путем лигирования. [117] Этот метод секвенирования ДНК позволяет секвенировать большое количество наношариков ДНК за один цикл при низких затратах на реагенты по сравнению с другими платформами для высокопроизводительного секвенирования. [118] Однако из каждого наношарика ДНК определяются только короткие последовательности ДНК, что затрудняет картирование коротких ридов с эталонным геномом . [117]
Секвенирование одной молекулы с помощью Heliscope
[ редактировать ]Секвенирование Heliscope — это метод секвенирования одиночных молекул, разработанный Helicos Biosciences . В нем используются фрагменты ДНК с добавленными хвостовыми адаптерами поли-А, которые прикрепляются к поверхности проточной кюветы. Следующие шаги включают секвенирование на основе удлинения с циклическими промывками проточной кюветы флуоресцентно меченными нуклеотидами (по одному типу нуклеотидов за раз, как в методе Сэнгера). Чтения выполняются секвенатором Heliscope. [119] [120] Чтения короткие, в среднем 35 п.н. [121] Что сделало эту технологию особенно новой, так это то, что она была первой в своем классе, которая секвенировала неамплифицированную ДНК, предотвращая тем самым любые ошибки чтения, связанные с этапами амплификации. [122] В 2009 году с помощью Heliscope секвенировали геном человека, однако в 2012 году компания обанкротилась. [123]
Микрофлюидные системы
[ редактировать ]Существует две основные микрофлюидные системы, которые используются для секвенирования ДНК; капельная микрофлюидика и цифровая микрофлюидика . Микрофлюидные устройства решают многие текущие ограничения существующих массивов секвенирования.
Абате и др. изучал использование микрофлюидных устройств на основе капель для секвенирования ДНК. [4] Эти устройства способны формировать и обрабатывать капли размером в пиколитр со скоростью тысячи в секунду. Устройства были созданы из полидиметилсилоксана (ПДМС) и использовали резонансный перенос энергии Форстера и анализы FRET для считывания последовательностей ДНК, содержащихся в каплях. Каждая позиция в массиве проверена на наличие определенной последовательности из 15 оснований. [4]
Фэйр и др. использовал цифровые микрофлюидные устройства для изучения пиросеквенирования ДНК . [124] Значительные преимущества включают портативность устройства, объем реагента, скорость анализа, возможности массового производства и высокую производительность. Это исследование предоставило доказательство концепции, показывающей, что цифровые устройства можно использовать для пиросеквенирования; исследование включало использование синтеза, который включает расширение ферментов и добавление меченых нуклеотидов. [124]
Болес и др. также изучал пиросеквенирование на цифровых микрофлюидных устройствах. [125] Они использовали электросмачивающее устройство для создания, смешивания и разделения капель. При секвенировании используется трехферментный протокол и шаблоны ДНК, закрепленные магнитными шариками. Устройство было протестировано с использованием двух протоколов и показало 100% точность на основе необработанных уровней пирограммы. Преимущества этих цифровых микрофлюидных устройств включают размер, стоимость и достижимый уровень функциональной интеграции. [125]
Исследования секвенирования ДНК с использованием микрофлюидики также могут быть применены к секвенированию РНК с использованием аналогичных капельных микрофлюидных методов, таких как метод inDrops. [126] Это показывает, что многие из этих методов секвенирования ДНК можно будет применять и дальше и использовать для лучшего понимания геномов и транскриптомов.
Методы в разработке
[ редактировать ]Методы секвенирования ДНК, которые в настоящее время разрабатываются, включают считывание последовательности при прохождении цепи ДНК через нанопоры (метод, который в настоящее время является коммерческим, но последующие поколения, такие как твердотельные нанопоры, все еще находятся в разработке), [127] [128] и методы на основе микроскопии, такие как атомно-силовая микроскопия или трансмиссионная электронная микроскопия , которые используются для идентификации положений отдельных нуклеотидов внутри длинных фрагментов ДНК (> 5000 п.н.) путем мечения нуклеотидов более тяжелыми элементами (например, галогенами) для визуального обнаружения и регистрации. . [129] [130] Технологии третьего поколения направлены на увеличение производительности и сокращение времени получения результата и затрат за счет устранения необходимости в чрезмерных реагентах и использования процессивности ДНК-полимеразы. [131]
Секвенирование ДНК туннельными токами
[ редактировать ]Другой подход использует измерения электрических туннельных токов в одноцепочечной ДНК при ее движении по каналу. В зависимости от своей электронной структуры каждая база по-разному влияет на туннельный ток. [132] позволяя различать разные базы. [133]
Использование туннельных токов может обеспечить секвенирование на несколько порядков быстрее, чем методы ионного тока, и секвенирование нескольких олигомеров ДНК и микроРНК уже достигнуто. [134]
Секвенирование путем гибридизации
[ редактировать ]Секвенирование путем гибридизации — это неферментативный метод, в котором используется микрочип ДНК . Один пул ДНК, последовательность которого необходимо определить, флуоресцентно метят и гибридизуют с массивом, содержащим известные последовательности. Сильные сигналы гибридизации из данного места на массиве идентифицируют его последовательность в секвенируемой ДНК. [135]
Этот метод секвенирования использует характеристики связывания библиотеки коротких одноцепочечных молекул ДНК (олигонуклеотидов), также называемых ДНК-зондами, для реконструкции целевой последовательности ДНК. Неспецифические гибриды удаляют промыванием и элюируют целевую ДНК. [136] Гибриды перестраиваются таким образом, что последовательность ДНК можно реконструировать. Преимущество этого типа секвенирования заключается в его способности захватывать большое количество целей с однородным покрытием. [137] Обычно требуется большое количество химикатов и исходной ДНК. Однако с появлением гибридизации на основе растворов требуется гораздо меньше оборудования и химикатов. [136]
Секвенирование с помощью масс-спектрометрии
[ редактировать ]Масс-спектрометрию можно использовать для определения последовательностей ДНК. Времяпролетная масс-спектрометрия с лазерной десорбцией и ионизацией с матрицей, или MALDI-TOF MS , специально исследовалась как альтернативный метод гель-электрофореза для визуализации фрагментов ДНК. С помощью этого метода фрагменты ДНК, полученные в результате реакций секвенирования обрыва цепи, сравниваются по массе, а не по размеру. Масса каждого нуклеотида отличается от других, и эту разницу можно обнаружить с помощью масс-спектрометрии. Однонуклеотидные мутации во фрагменте легче обнаружить с помощью МС, чем только с помощью гель-электрофореза. MALDI-TOF MS позволяет легче обнаруживать различия между фрагментами РНК, поэтому исследователи могут косвенно секвенировать ДНК с помощью методов MS, сначала преобразуя ее в РНК. [138]
Более высокое разрешение фрагментов ДНК, допускаемое методами на основе МС, представляет особый интерес для исследователей в области судебной медицины, поскольку они могут захотеть найти однонуклеотидные полиморфизмы в образцах ДНК человека для идентификации людей. Эти образцы могут быть сильно разложившимися, поэтому судебно-медицинские эксперты часто отдают предпочтение митохондриальной ДНК из-за ее более высокой стабильности и возможности применения для изучения происхождения. Методы секвенирования на основе MS использовались для сравнения последовательностей митохондриальной ДНК человека из образцов в Федерального бюро расследований. базе данных [139] и из костей, найденных в братских могилах солдат Первой мировой войны. [140]
Ранние методы обрыва цепи и TOF MS продемонстрировали длину чтения до 100 пар оснований. [141] Исследователи не смогли превысить этот средний размер чтения; Как и только секвенирование с обрывом цепи, секвенирование ДНК на основе MS может не подходить для крупных проектов секвенирования de novo . Несмотря на это, недавнее исследование использовало считывание коротких последовательностей и масс-спектроскопию для сравнения однонуклеотидных полиморфизмов в патогенных штаммах Streptococcus . [142]
Микрофлюидное секвенирование по Сэнгеру
[ редактировать ]При микрофлюидном секвенировании по Сэнгеру вся термоциклическая амплификация фрагментов ДНК, а также их разделение с помощью электрофореза выполняются на одной стеклянной пластине (диаметром примерно 10 см), что позволяет сократить использование реагентов, а также стоимость. [143] В некоторых случаях исследователи показали, что они могут увеличить производительность обычного секвенирования за счет использования микрочипов. [144] Для того, чтобы сделать такое использование технологий эффективным, все еще необходимо провести исследования.
Методы микроскопии
[ редактировать ]Этот подход напрямую визуализирует последовательность молекул ДНК с помощью электронной микроскопии. Первая идентификация пар оснований ДНК в интактных молекулах ДНК путем ферментативного включения модифицированных оснований, которые содержат атомы с увеличенным атомным номером, прямая визуализация и идентификация индивидуально меченных оснований в синтетической молекуле ДНК из 3272 пар оснований и вирусном геноме из 7249 пар оснований. было продемонстрировано. [145]
секвенирование РНКП
[ редактировать ]Этот метод основан на использовании РНК-полимеразы (РНКП), которая прикреплена к шарику полистирола . Один конец секвенируемой ДНК прикрепляют к другой бусине, причем обе бусины помещают в оптические ловушки. Движение РНКП во время транскрипции приближает шарики, и их относительное расстояние изменяется, что затем можно записать с разрешением в один нуклеотид. Последовательность выводится на основе четырех показаний с пониженными концентрациями каждого из четырех типов нуклеотидов, аналогично методу Сэнгера. [146] Проводят сравнение между областями и получают информацию о последовательностях путем сравнения известных областей последовательности с неизвестными областями последовательности. [147]
in vitro Высокопроизводительное секвенирование вирусов
[ редактировать ]Был разработан метод для анализа полных наборов белковых взаимодействий с использованием комбинации пиросеквенирования 454 и мРНК in vitro вирусной метода отображения . В частности, этот метод ковалентно связывает интересующие белки с кодирующими их мРНК, а затем обнаруживает фрагменты мРНК с помощью ПЦР с обратной транскрипцией . Затем мРНК можно амплифицировать и секвенировать. Комбинированный метод получил название IVV-HiTSeq и может быть реализован в бесклеточных условиях, хотя его результаты могут не быть репрезентативными для in vivo . условий [148]
Доля рынка
[ редактировать ]Хотя существует множество различных способов секвенирования ДНК, лишь немногие из них доминируют на рынке. В 2022 году у Illumina было около 80% рынка; остальную часть рынка занимают всего несколько игроков (PacBio, Oxford, 454, MGI) [149]
Подготовка проб
[ редактировать ]Успех любого протокола секвенирования ДНК зависит от экстракции и подготовки образцов ДНК или РНК из интересующего биологического материала.
- Успешная экстракция ДНК даст образец ДНК с длинными неразрушенными цепями.
- В результате успешной экстракции РНК будет получен образец РНК, который необходимо преобразовать в комплементарную ДНК (кДНК) с помощью обратной транскриптазы — ДНК-полимеразы, которая синтезирует комплементарную ДНК на основе существующих цепей РНК методом ПЦР. [150] Комплементарную ДНК затем можно обрабатывать так же, как геномную ДНК.
После выделения ДНК или РНК образцы могут потребовать дальнейшей подготовки в зависимости от метода секвенирования. Для секвенирования по Сэнгеру перед секвенированием требуются либо процедуры клонирования, либо ПЦР. В случае методов секвенирования нового поколения перед обработкой требуется подготовка библиотеки. [151] Оценка качества и количества нуклеиновых кислот как после экстракции, так и после подготовки библиотеки позволяет выявить деградированные, фрагментированные и низкочистые образцы и получить высококачественные данные секвенирования. [152]
Высокая производительность современных технологий секвенирования ДНК/РНК создала проблему для масштабирования метода подготовки проб. Для подготовки большего количества проб с меньшим общим временем работы используются несколько инструментов для работы с жидкостями:
| компания | Обработчики жидкостей / Автоматизация | low_mark_USD | Upper_mark_USD | Landing_url |
| Опентроны | OpenTrons OT-2 | $6,500 | $20,000 | https://www.opentrons.com/ |
| Гилсон | Гилсон Пипетмакс | $20,000 | $40,000 | https://gb.gilson.com/GBSV/system-pipetmax.html |
| Неотек | Неотек ЭзМейт | $25,000 | $45,000 | http://neotec.co.il/pipetting-device/ |
| Формулатрикс | Формулатрикс Богомол | $40,000 | $60,000 | https://formulatrix.com/liquid-handling-systems/mantis-liquid-handler/ |
| Хадсон Роботикс | Хадсон Роботикс СОЛО | $40,000 | $50,000 | https://hudsonrobotics.com/products/applications/automated-solutions-next-generation-sequencing-ngs/ |
| Гамильтон | Гамильтон Микролаб НИМБУС | $40,000 | $80,000 | https://www.hamiltoncompany.com/automated-liquid-handling/platforms/microlab-nimbus#specifications |
| ТТП Лабтех | TTP Labtech Mosquito HV Genomics | $45,000 | $80,000 | https://www.sptlabtech.com/products/liquid-handling/mosquito-hv-genomics/ |
| Бекман Коултер | Биомек 4000 | $50,000 | $65,000 | https://www.mybeckman.uk/liquid-handlers/biomek-4000/b22640 |
| Гамильтон | Hamilton Genomic STARlet | $50,000 | $100,000 | https://www.hamiltoncompany.com/automated-liquid-handling/assay-ready-workstations/genomic-starlet |
| Эппендорф | Эппендорф epMotion 5075t | $95,000 | $110,000 | https://www.eppendorf.com/epmotion/ |
| Бекман Коултер | Бекман Коултер Биомек i5 | $100,000 | $150,000 | https://www.beckman.com/liquid-handlers/biomek-i5 |
| Гамильтон | Гамильтон НГС ЗВЕЗДА | $100,000 | $200,000 | http://www.hamiltonrobotics.com/ |
| ПеркинЭлмер | Рабочая станция PerkinElmer Sciclone G3 NGS и NGSx | $150,000 | $220,000 | https://www.perkinelmer.com/uk/product/sciclone-g3-ngs-workstation-cls145321 |
| Аджилент | Аджилент Браво НГС | $170,000 | $290,000 | https://www.agilent.com/en/products/automated-liquid-handling/automated-liquid-handling-applications/bravo-ngs |
| Бекман Коултер | Бекман Коултер Биомек i7 | $200,000 | $250,000 | https://www.beckman.com/liquid-handlers/biomek-i7 |
| Лабцит Эхо 525 | Beckman Coulter Labcyte Echo 525 | $260,000 | $300,000 | https://www.labcyte.com/products/liquid-handling/echo-525-liquid-handler |
| Текан | Текан НГС | $270,000 | $350,000 | https://lifesciences.tecan.com/ngs-sample-preparation |
Инициативы развития
[ редактировать ]
В октябре 2006 года Фонд X Prize учредил инициативу по содействию развитию технологий полного секвенирования генома , названную Archon X Prize , намереваясь выделить 10 миллионов долларов «первой команде, которая сможет создать устройство и использовать его для секвенирования 100 человеческих геномов». в течение 10 дней или менее, с точностью не более одной ошибки на каждые 100 000 секвенированных оснований, с последовательностями, точно охватывающими не менее 98% генома, и с периодическими расходами не более 10 000 долларов США за геном». [153]
Каждый год Национальный институт исследования генома человека (NHGRI) выделяет гранты на новые исследования и разработки в области геномики . Гранты 2010 года и кандидаты 2011 года включают продолжающуюся работу в области микрофлюидных, полонических методологий и методологий секвенирования с тяжелыми основаниями. [154]
Вычислительные задачи
[ редактировать ]Описанные здесь технологии секвенирования производят необработанные данные, которые необходимо собрать в более длинные последовательности, такие как полные геномы ( сборка последовательностей ). Для достижения этой цели существует множество вычислительных задач, таких как оценка необработанных данных последовательности, которая выполняется с помощью программ и алгоритмов, таких как Phred и Phrap . Другие проблемы связаны с повторяющимися последовательностями, которые часто препятствуют полной сборке генома, поскольку встречаются во многих местах генома. Как следствие, многие последовательности не могут быть отнесены к конкретным хромосомам . Получение необработанных данных о последовательностях — это только начало детального биоинформационного анализа. [155] Тем не менее, были разработаны новые методы секвенирования и исправления ошибок секвенирования. [156]
Читать обрезку
[ редактировать ]Иногда необработанные чтения, произведенные секвенатором, являются правильными и точными только на части их длины. Использование всего чтения может привести к появлению артефактов в последующих анализах, таких как сборка генома, вызов SNP или оценка экспрессии генов. Были введены два класса программ обрезки, основанные на классах алгоритмов на основе окон или алгоритмов с текущей суммой. [157] Это неполный список доступных в настоящее время алгоритмов обрезки с указанием класса алгоритмов, к которому они принадлежат:
| Название алгоритма | Тип алгоритма | Связь |
|---|---|---|
| Cutadapt [158] | Текущая сумма | Cutadapt |
| КонДеТри [159] | На основе окон | КонДеТри |
| ЭРН ФИЛЬТР [160] | Текущая сумма | ЭРН ФИЛЬТР |
| Качественный триммер FASTX | На основе окон | Качественный триммер FASTX |
| ПРИНЦЕК [161] | На основе окон | ПРИНЦЕК |
| Триммоматик [162] | На основе окон | Триммоматик |
| СолексаQA [163] | На основе окон | СолексаQA |
| СолексаQA-BWA | Текущая сумма | СолексаQA-BWA |
| Серп | На основе окон | Серп |
Этические проблемы
[ редактировать ]Этот раздел нуждается в расширении . Вы можете помочь, добавив к нему . ( май 2015 г. ) |
Генетика человека была включена в область биоэтики с начала 1970-х годов. [164] а рост использования секвенирования ДНК (особенно высокопроизводительного секвенирования) породил ряд этических проблем. Одним из ключевых вопросов является право собственности на ДНК человека и данные, полученные при секвенировании этой ДНК. [165] Что касается самой молекулы ДНК, то ведущим судебным делом по этой теме было дело Мур против Регентов Калифорнийского университета (1990 г.), постановившее, что люди не имеют прав собственности на выброшенные клетки или любую прибыль, полученную с использованием этих клеток (например, в качестве запатентованного клеточная линия ). Однако люди имеют право на информированное согласие относительно удаления и использования клеток. Что касается данных, полученных с помощью секвенирования ДНК, Мур не дает человеку никаких прав на информацию, полученную из его ДНК. [165]
Поскольку секвенирование ДНК становится все более распространенным, хранение, безопасность и обмен геномными данными также становятся все более важными. [165] [166] Например, одна из проблем заключается в том, что страховщики могут использовать геномные данные человека для изменения своих расценок в зависимости от предполагаемого будущего здоровья человека на основе его ДНК. [166] [167] В мае 2008 года Закон о недискриминации генетической информации (GINA), запрещающий дискриминацию на основе генетической информации в отношении медицинского страхования и трудоустройства. в США был подписан [168] [169] В 2012 году Президентская комиссия США по изучению биоэтических проблем сообщила, что существующее законодательство о конфиденциальности данных секвенирования ДНК, такое как GINA и Закон о переносимости и подотчетности медицинского страхования, является недостаточным, отметив, что данные полногеномного секвенирования являются особенно конфиденциальными, поскольку они могут использоваться для идентификации не только человека, от которого были созданы данные, но и его родственников. [170] [171]
На большей части территории Соединенных Штатов «брошенная» ДНК, например найденная на облизанной марке или конверте, кофейной чашке, сигарете, жевательной резинке, домашнем мусоре или волосах, упавших на общественный тротуар, может быть законно собрана. и секвенированы кем угодно, включая полицию, частных детективов, политических оппонентов или людей, участвующих в спорах об отцовстве. По состоянию на 2013 год в одиннадцати штатах действуют законы, которые можно интерпретировать как запрещающие «кражу ДНК». [172]
Этические проблемы также возникли в связи с увеличением использования скрининга генетических вариаций как у новорожденных, так и у взрослых такими компаниями, как 23andMe . [173] [174] Утверждалось, что скрининг генетических вариаций может быть вредным, увеличивая беспокойство у людей, у которых обнаружен повышенный риск заболеваний. [175] Например, в одном случае, отмеченном в Time , врачи, проверяющие больного ребенка на наличие генетических вариантов, решили не информировать родителей о несвязанном варианте, связанном с деменцией, из-за вреда, который он может нанести родителям. [176] Однако исследование 2011 года, опубликованное в Медицинском журнале Новой Англии, показало, что люди, проходящие анализ риска заболеваний, не демонстрировали повышенного уровня тревоги. [175] Кроме того, развитие технологий секвенирования следующего поколения, таких как секвенирование на основе нанопор, также вызвало дополнительные этические проблемы. [177]
См. также
[ редактировать ]- Биоинформатика - вычислительный анализ больших и сложных наборов биологических данных.
- Секвенирование генома рака
- Круговое консенсусное секвенирование
- ДНК-вычисления - вычисления с использованием оборудования молекулярной биологии.
- Полевой транзистор ДНК - транзистор, который использует полевой эффект из-за частичных зарядов ДНК.
- Теория секвенирования ДНК - Биологическая теория
- Секвенатор ДНК - научный инструмент, используемый для автоматизации процесса секвенирования ДНК.
- Генографический проект - Проект гражданской науки
- Геномный проект - Научные усилия по определению полной последовательности генома организма.
- Секвенирование генома исчезающих видов – тестирование ДНК для оценки угрозы
- Геномный скимминг - метод секвенирования генома.
- IsoBase — функционально связанные белки в сетях PPI.
- Связанное-чтение последовательности
- Прыгающая библиотека
- Последовательность нуклеиновой кислоты - последовательность нуклеотидов в нуклеиновой кислоте.
- Мультиплексная амплификация зонда, зависимая от лигирования
- Персонализированная медицина - медицинская модель, которая адаптирует медицинскую практику к индивидуальному пациенту.
- Секвенирование белка - Секвенирование расположения аминокислот в белке.
- Последовательный майнинг
- Инструмент профилирования последовательностей
- Секвенирование путем гибридизации - метод определения составляющих нуклеотидов фиксированного размера в цепи ДНК.
- Секвенирование путем лигирования — метод секвенирования ДНК, в котором используется фермент ДНК-лигаза для идентификации нуклеотида, присутствующего в заданном положении в последовательности ДНК.
- TIARA (база данных) - База данных личной геномной информации.
- Секвенирование ДНК с помощью просвечивающей электронной микроскопии – технология секвенирования одиночных молекул
Примечания
[ редактировать ]- ^ «Следующее поколение» по-прежнему широко используется с 2019 года. Например, Стрейтон Дж., Фри Т., Сойер А., Мартин Дж. (февраль 2019 г.). «От секвенирования по Сэнгеру до баз данных геномов и не только» . БиоТехники . 66 (2): 60–63. дои : 10.2144/btn-2019-0011 . ПМИД 30744413 .
Технологии секвенирования нового поколения (NGS) произвели революцию в геномных исследованиях. (первое предложение статьи)
Ссылки
[ редактировать ]- ^ «Представляем «темную ДНК» — явление, которое может изменить наше представление об эволюции» . 24 августа 2017 г.
- ^ Бехджати С., Тарпей П.С. (декабрь 2013 г.). «Что такое секвенирование следующего поколения?» . Архив детских болезней: издание для образования и практики . 98 (6): 236–8. doi : 10.1136/archdischild-2013-304340 . ПМЦ 3841808 . ПМИД 23986538 .
- ^ Хмелецкий Дж, Мейерсон М (14 января 2014 г.). «Секвенирование ДНК рака: что мы узнали?» . Ежегодный обзор медицины . 65 (1): 63–79. doi : 10.1146/annurev-med-060712-200152 . ПМИД 24274178 .
- ^ Jump up to: а б с д Абате А.Р., Хунг Т., Сперлинг Р.А., Мэри П., Ротем А., Агрести Дж.Дж. и др. (декабрь 2013 г.). «Анализ последовательности ДНК с помощью капельной микрофлюидики» . Лаборатория на чипе . 13 (24): 4864–9. дои : 10.1039/c3lc50905b . ПМК 4090915 . ПМИД 24185402 .
- ^ Пекин Д., Схири Ю., Барет Дж.К., Ле Корр Д., Мазутис Л., Салем С.Б. и др. (июль 2011 г.). «Количественное и чувствительное обнаружение редких мутаций с использованием капельной микрофлюидики». Лаборатория на чипе . 11 (13): 2156–66. дои : 10.1039/c1lc20128j . ПМИД 21594292 .
- ^ Олсвик О, Уолберг Дж, Петтерсон Б, Улен М, Попович Т, Ваксмут И.К., Филдс П.И. (январь 1993 г.). «Использование автоматического секвенирования ампликонов, полученных в результате полимеразной цепной реакции, для идентификации трех типов субъединицы B холерного токсина в штаммах Vibrio cholerae O1» . Дж. Клин. Микробиол. 31 (1): 22–25. doi : 10.1128/JCM.31.1.22-25.1993 . ПМК 262614 . ПМИД 7678018 .

- ^ Петтерссон Э., Лундеберг Дж., Ахмадиан А. (февраль 2009 г.). «Поколения технологий секвенирования» . Геномика . 93 (2): 105–11. дои : 10.1016/j.ygeno.2008.10.003 . ПМИД 18992322 .
- ^ Jump up to: а б с Джей Э., Бамбара Р., Падманабхан Р., Ву Р. (март 1974 г.). «Анализ последовательности ДНК: общий, простой и быстрый метод секвенирования крупных фрагментов олигодезоксирибонуклеотидов путем картирования» . Исследования нуклеиновых кислот . 1 (3): 331–53. дои : 10.1093/нар/1.3.331 . ПМК 344020 . ПМИД 10793670 .
- ^ Хант, Кэти (17 февраля 2021 г.). «Самая старая в мире ДНК секвенирована у мамонта, жившего более миллиона лет назад» . CNN . Проверено 17 февраля 2021 г.
- ^ Каллауэй, Юэн (17 февраля 2021 г.). «Геномы мамонтов возрастом в миллион лет побили рекорд древнейшей древней ДНК — сохранившиеся в вечной мерзлоте зубы возрастом до 1,6 миллиона лет идентифицируют новый вид мамонтов в Сибири» . Природа . 590 (7847): 537–538. Бибкод : 2021Natur.590..537C . дои : 10.1038/d41586-021-00436-x . ПМИД 33597786 .
- ^ Jump up to: а б с Кастро, Кристина; Марин, Рэйчел; Рамос, Эдвард; Нг, Терри Фей Фан (2019). «Влияние вариантной интерференции на сборку de novo для глубокого секвенирования вирусов» . БМК Геномика . 21 (1): 421. bioRxiv 10.1101/815480 . дои : 10.1186/s12864-020-06801-w . ПМЦ 7306937 . ПМИД 32571214 .
- ^ Jump up to: а б Воль, Ширли; Шаффнер, Стивен Ф.; Сабети, Пардис К. (2016). «Геномный анализ вирусных вспышек» . Ежегодный обзор вирусологии . 3 (1): 173–195. doi : 10.1146/annurev-virology-110615-035747 . ПМК 5210220 . ПМИД 27501264 .
- ^ Бойкот, Ким М.; Ванстон, Меган Р.; Булман, Деннис Э.; Маккензи, Алекс Э. (октябрь 2013 г.). «Генетика редких заболеваний в эпоху секвенирования следующего поколения: от открытия к трансляции». Обзоры природы Генетика . 14 (10): 681–691. дои : 10.1038/nrg3555 . ПМИД 23999272 . S2CID 8496181 .
- ^ Бин, Лора; Функе, Биргит; Карлстон, Коллин М.; Гэннон, Дженнифер Л.; Кантарчи, Сибель; Крок, Брайан Л.; Чжан, Шулин; Байрак-Тойдемир, Пинар (март 2020 г.). «Диагностические панели секвенирования генов: от дизайна до отчета — технический стандарт Американского колледжа медицинской генетики и геномики (ACMG)» . Генетика в медицине . 22 (3): 453–461. дои : 10.1038/s41436-019-0666-z . ISSN 1098-3600 . ПМИД 31732716 .
- ^ Шлейзенер В., Кезер К.У., Беккерт П., Ниманн С., Фейрригель С. (2017). « Прогнозирование устойчивости микобактерий туберкулеза и классификация линий на основе секвенирования генома: сравнение инструментов автоматического анализа» . Научный представитель . 7 : 46327. Бибкод : 2017NatSR...746327S . дои : 10.1038/srep46327 . ПМЦ 7365310 . ПМИД 28425484 .
- ^ Маэ П., Эль Азами М., Барлас П., Турну М. (2019). «Крупномасштабная оценка TBProfiler и Mykrobe для прогнозирования устойчивости микобактерий туберкулеза к антибиотикам » . ПерДж . 7 : е6857. дои : 10.7717/peerj.6857 . ПМК 6500375 . ПМИД 31106066 .
- ^ Предиктор Mykrobe - Прогноз устойчивости к антибиотикам S. aureus и M. Tuberculosis на основе данных полногеномной последовательности.
- ^ Брэдли, Фелим; Гордон, Н. Клэр; Уокер, Тимоти М.; Данн, Лаура; Привет, Саймон; Хуанг, Билл; Эрл, Сара; Панкхерст, Луиза Дж.; Энсон, Люк; де Чезаре, Мариатереса; Пьяцца, Паоло; Вотинцева Антонина Александровна; Голубчик, Таня; Уилсон, Дэниел Дж.; Уилли, Дэвид Х.; Диль, Роланд; Ниманн, Стефан; Фейрригель, Силке; Коль, Томас А.; Исмаил, Назир; Омар, Шахид В.; Смит, Э. Грейс; Бак, Дэвид; Маквин, Гил; Уокер, А. Сара; Пето, Тим Э.А.; Крук, Деррик В.; Икбал, Замин (21 декабря 2015 г.). «Быстрый прогноз устойчивости к антибиотикам на основе данных о последовательностях генома золотистого стафилококка и микобактерии туберкулеза» . Природные коммуникации . 6 (1): 10063. Бибкод : 2015NatCo...610063B . дои : 10.1038/ncomms10063 . ПМК 4703848 . ПМИД 26686880 .
- ^ «Майкл Мосли против супербактерий» . Архивировано из оригинала 24 ноября 2020 года . Проверено 21 октября 2019 г.
- ^ Mykrobe , Mykrobe-tools, 24 декабря 2022 г. , дата обращения 2 января 2023 г.
- ^ Кертис С., Херевард Дж. (29 августа 2017 г.). «От места преступления до зала суда: путешествие образца ДНК» . Разговор .
- ^ Морера С., Ларивьер Л., Курцек Дж., Ашке-Сонненборн У., Фримонт П.С., Жанен Дж., Рюгер В. (август 2001 г.). «Кристаллические структуры бета-глюкозилтрансферазы фага Т4 высокого разрешения: индуцированное соответствие и эффект связывания субстрата и металла». Журнал молекулярной биологии . 311 (3): 569–77. дои : 10.1006/jmbi.2001.4905 . ПМИД 11493010 .
- ^ Эрлих М., Гама-Соса М.А., Хуанг Л.Х., Миджетт Р.М., Куо К.К., МакКьюн Р.А., Герке С. (апрель 1982 г.). «Количество и распределение 5-метилцитозина в ДНК человека из разных типов тканей клеток» . Исследования нуклеиновых кислот . 10 (8): 2709–21. дои : 10.1093/нар/10.8.2709 . ПМК 320645 . ПМИД 7079182 .
- ^ Эрлих М., Ван Р.Ю. (июнь 1981 г.). «5-Метилцитозин в эукариотической ДНК». Наука . 212 (4501): 1350–7. Бибкод : 1981Sci...212.1350E . дои : 10.1126/science.6262918 . ПМИД 6262918 .
- ^ Сонг CX, Кларк Т.А., Лу XY, Кислюк А., Дай Кью, Тернер С.В. и др. (ноябрь 2011 г.). «Чувствительное и специфическое секвенирование одиночных молекул 5-гидроксиметилцитозина» . Природные методы . 9 (1): 75–7. дои : 10.1038/nmeth.1779 . ПМЦ 3646335 . ПМИД 22101853 .
- ^ Чернецкий, Дариуш; Боном, Фредерик; Камински, Пьер-Александр; Деларю, Марк (5 августа 2021 г.). «Характеристика триады генов цианофага S-2L, достаточной для замены аденина на 2-аминоаденин в бактериальной ДНК» . Природные коммуникации . 12 (1): 4710. Бибкод : 2021NatCo..12.4710C . дои : 10.1038/s41467-021-25064-x . ПМЦ 8342488 . ПМИД 34354070 . S2CID 233745192 .
- ^ «Прямое обнаружение и секвенирование поврежденных оснований ДНК» . ПакБио . Проверено 31 июля 2024 г.
- ^ Уотсон Дж.Д., Крик Ф.Х. (1953). «Структура ДНК». Холодный источник Харб. Симп. Квант. Биол . 18 : 123–31. дои : 10.1101/SQB.1953.018.01.020 . ПМИД 13168976 .
- ^ Маркс, Л. «Путь к секвенированию ДНК: жизнь и работа Фредерика Сэнгера» . Что такое биотехнология? . Проверено 27 июня 2023 г.
- ^ Мин Джоу В., Хагеман Г., Изеберт М., Фирс В. (май 1972 г.). «Нуклеотидная последовательность гена, кодирующего белок оболочки бактериофага MS2». Природа . 237 (5350): 82–8. Бибкод : 1972Natur.237...82J . дои : 10.1038/237082a0 . ПМИД 4555447 . S2CID 4153893 .
- ^ Фирс В., Контрерас Р., Дюринк Ф., Хагеман Г., Изерентант Д., Меррегарт Дж., Мин Джоу В., Молеманс Ф., Раймакерс А., Ван ден Берге А., Волкарт Г., Изебарт М. (апрель 1976 г.). «Полная нуклеотидная последовательность РНК бактериофага MS2: первичная и вторичная структура гена репликазы». Природа . 260 (5551): 500–7. Бибкод : 1976Natur.260..500F . дои : 10.1038/260500a0 . ПМИД 1264203 . S2CID 4289674 .
- ^ Озсолак Ф., Милош П.М. (февраль 2011 г.). «Секвенирование РНК: достижения, проблемы и возможности» . Обзоры природы Генетика . 12 (2): 87–98. дои : 10.1038/nrg2934 . ПМК 3031867 . ПМИД 21191423 .
- ^ «Профиль факультета Рэя Ву» . Корнелльский университет. Архивировано из оригинала 4 марта 2009 года.
- ^ Падманабхан Р., Джей Э., Ву Р. (июнь 1974 г.). «Химический синтез праймера и его использование в анализе последовательности гена лизоцима бактериофага Т4» . Труды Национальной академии наук Соединенных Штатов Америки . 71 (6): 2510–4. Бибкод : 1974PNAS...71.2510P . дои : 10.1073/pnas.71.6.2510 . ПМЦ 388489 . ПМИД 4526223 .
- ^ Онага Л.А. (июнь 2014 г.). «Рэй Ву как пятое дело: демонстрация коллективной памяти в истории секвенирования ДНК». Исследования по истории и философии науки . Часть С. 46 : 1–14. дои : 10.1016/j.shpsc.2013.12.006 . ПМИД 24565976 .
- ^ Ву Р (1972). «Анализ нуклеотидной последовательности ДНК». Новая биология природы . 236 (68): 198–200. дои : 10.1038/newbio236198a0 . ПМИД 4553110 .
- ^ Падманабхан Р., Ву Р. (1972). «Анализ нуклеотидной последовательности ДНК. IX. Использование олигонуклеотидов определенной последовательности в качестве праймеров при анализе последовательности ДНК». Биохим. Биофиз. Рез. Коммун . 48 (5): 1295–302. дои : 10.1016/0006-291X(72)90852-2 . ПМИД 4560009 .
- ^ Ву Р, Ту CD, Падманабхан Р (1973). «Анализ нуклеотидной последовательности ДНК. XII. Химический синтез и анализ последовательности додекадезоксинуклеотида, который связывается с геном эндолизина бактериофага лямбда». Биохим. Биофиз. Рез. Коммун . 55 (4): 1092–99. дои : 10.1016/S0006-291X(73)80007-5 . ПМИД 4358929 .
- ^ Jump up to: а б Сэнгер Ф., Никлен С., Коулсон А.Р. (декабрь 1977 г.). «Секвенирование ДНК с ингибиторами обрыва цепи» . Учеб. Натл. акад. наук. США . 74 (12): 5463–77. Бибкод : 1977PNAS...74.5463S . дои : 10.1073/pnas.74.12.5463 . ПМК 431765 . ПМИД 271968 .
- ^ Jump up to: а б с Максам А.М., Гилберт В. (февраль 1977 г.). «Новый метод секвенирования ДНК» . Учеб. Натл. акад. наук. США . 74 (2): 560–64. Бибкод : 1977ПНАС...74..560М . дои : 10.1073/pnas.74.2.560 . ПМК 392330 . ПМИД 265521 .
- ^ Гилберт, В. Секвенирование ДНК и структура генов . Нобелевская лекция, 8 декабря 1980 г.
- ^ Гилберт В., Максам А. (декабрь 1973 г.). «Нуклеотидная последовательность оператора lac» . Учеб. Натл. акад. наук. США . 70 (12): 3581–84. Бибкод : 1973PNAS...70.3581G . дои : 10.1073/pnas.70.12.3581 . ПМК 427284 . ПМИД 4587255 .
- ^ Сэнгер Ф., генеральный директор Air, Баррелл Б.Г., Браун Н.Л., Коулсон А.Р., Фиддес К.А., Хатчисон К.А., Слокомб П.М., Смит М. (февраль 1977 г.). «Нуклеотидная последовательность ДНК бактериофага phi X174». Природа . 265 (5596): 687–95. Бибкод : 1977Natur.265..687S . дои : 10.1038/265687a0 . ПМИД 870828 . S2CID 4206886 .
- ^ Маркс, Л. «Следующий рубеж: человеческие вирусы» . Что такое биотехнология? . Проверено 27 июня 2023 г.
- ^ Бек С., Поль FM (1984). «Секвенирование ДНК методом прямого блоттинг-электрофореза» . ЭМБО Дж . 3 (12): 2905–09. дои : 10.1002/j.1460-2075.1984.tb02230.x . ПМК 557787 . ПМИД 6396083 .
- ^ Патент США 4631122 (1986).
- ^ Фельдманн Х. и др. (1994). «Полная последовательность ДНК дрожжевой хромосомы II» . ЭМБО Дж . 13 (24): 5795–809. дои : 10.1002/j.1460-2075.1994.tb06923.x . ПМЦ 395553 . ПМИД 7813418 .
- ^ Смит Л.М., Сандерс Дж.З., Кайзер Р.Дж., Хьюз П., Додд С., Коннелл С.Р., Хайнер С., Кент С.Б., Худ Л.Е. (12 июня 1986 г.). «Обнаружение флуоресценции при автоматизированном анализе последовательностей ДНК». Природа . 321 (6071): 674–79. Бибкод : 1986Natur.321..674S . дои : 10.1038/321674a0 . ПМИД 3713851 . S2CID 27800972 .
- ^ Пробер Дж.М., Трейнор Г.Л., Дам Р.Дж., Хоббс Ф.В., Робертсон К.В., Загурски Р.Дж., Кокуцца А.Дж., Дженсен М.А., Баумайстер К. (16 октября 1987 г.). «Система для быстрого секвенирования ДНК с флуоресцентными дидезоксинуклеотидами на конце цепи». Наука . 238 (4825): 336–41. Бибкод : 1987Sci...238..336P . дои : 10.1126/science.2443975 . ПМИД 2443975 .
- ^ Адамс М.Д., Келли Дж.М., Гокейн Дж.Д., Дубник М., Полимеропулос М.Х., Сяо Х., Меррил Ч.Р., Ву А., Олде Б., Морено Р.Ф. (июнь 1991 г.). «Комплементарное секвенирование ДНК: метки экспрессируемых последовательностей и проект генома человека». Наука . 252 (5013): 1651–56. Бибкод : 1991Sci...252.1651A . дои : 10.1126/science.2047873 . ПМИД 2047873 . S2CID 13436211 .
- ^ Флейшманн Р.Д., Адамс М.Д., Уайт О., Клейтон Р.А., Киркнесс Э.Ф., Керлаваж А.Р., Балт С.Дж., Томб Дж.Ф., Догерти Б.А., Меррик Дж.М. (июль 1995 г.). «Полногеномное случайное секвенирование и сборка Haemophilus influenzae Rd ». Наука . 269 (5223): 496–512. Бибкод : 1995Sci...269..496F . дои : 10.1126/science.7542800 . ПМИД 7542800 .
- ^ Ландер Э.С., Линтон Л.М., Биррен Б., Нусбаум С., Зоди М.К. и др. (февраль 2001 г.). «Первичное секвенирование и анализ генома человека» (PDF) . Природа . 409 (6822): 860–921. Бибкод : 2001Natur.409..860L . дои : 10.1038/35057062 . ПМИД 11237011 .
- ^ Вентер Дж.К., Адамс, доктор медицинских наук и др. (февраль 2001 г.). «Последовательность генома человека» . Наука . 291 (5507): 1304–51. Бибкод : 2001Sci...291.1304V . дои : 10.1126/science.1058040 . ПМИД 11181995 .
- ^ Ян, Аймин; Чжан, Вэй; Ван, Цзяхао; Ян, Кэ; Хан, Ян; Чжан, Лимин (2020). «Обзор применения алгоритмов машинного обучения для анализа последовательностей ДНК» . Границы биоинженерии и биотехнологии . 8 : 1032. дои : 10.3389/fbioe.2020.01032 . ПМЦ 7498545 . ПМИД 33015010 .
- ^ «Эспейснет – Библиографические данные» . world.espacenet.com .
- ^ Ронаги М., Карамохамед С., Петтерссон Б., Улен М., Найрен П. (1996). «Секвенирование ДНК в реальном времени с использованием обнаружения высвобождения пирофосфата». Аналитическая биохимия . 242 (1): 84–89. дои : 10.1006/abio.1996.0432 . ПМИД 8923969 .
- ^ Jump up to: а б Кавасима, Эрик Х.; Лоран Фаринелли; Паскаль Майер (12 мая 2005 г.). «Патент: Способ амплификации нуклеиновых кислот» . Архивировано из оригинала 22 февраля 2013 года . Проверено 22 декабря 2012 г.
- ^ Юинг Б., Грин П. (март 1998 г.). «Базовый вызов трассировок автоматического секвенатора с использованием phred. II. Вероятности ошибок» . Геном Рез . 8 (3): 186–94. дои : 10.1101/гр.8.3.186 . ПМИД 9521922 .
- ^ «Показатели качества секвенирования следующего поколения» (PDF) . Иллюмина . 31 октября 2011 года . Проверено 8 мая 2018 г.
- ^ Jump up to: а б Бреннер С., Джонсон М., Бриджем Дж., Голда Дж., Ллойд Д.Х., Джонсон Д., Луо С., МакКарди С., Фой М., Юэн М., Рот Р., Джордж Д., Элетр С., Альбрехт Дж., Вермаас Е., Уильямс С.Р., Мун К. , Берчем Т., Паллас М., Дюбридж Р.Б., Киршнер Дж., Фирон К., Мао Дж., Коркоран К. (2000). «Анализ экспрессии генов с помощью массово-параллельного сигнатурного секвенирования (MPSS) на массивах микрогранул». Природная биотехнология . 18 (6): 630–34. дои : 10.1038/76469 . ПМИД 10835600 . S2CID 13884154 .
- ^ «Секвенирование Максама Гилберта» . ПабМед .
- ^ Сэнгер Ф., Коулсон А.Р. (май 1975 г.). «Быстрый метод определения последовательностей ДНК путем синтеза с помощью ДНК-полимеразы». Дж. Мол. Биол . 94 (3): 441–48. дои : 10.1016/0022-2836(75)90213-2 . ПМИД 1100841 .
- ^ Веттерстранд, Крис. «Затраты на секвенирование ДНК: данные Программы секвенирования генома NHGRI (GSP)» . Национальный институт исследования генома человека . Проверено 30 мая 2013 г.
- ^ Найрен, П.; Петтерссон, Б.; Улен, М. (январь 1993 г.). «Твердофазное мини-секвенирование ДНК с помощью ферментативного люминометрического анализа неорганического пирофосфата». Аналитическая биохимия . 208 (1): 171–175. дои : 10.1006/abio.1993.1024 . ПМИД 8382019 .
- ^ Ронаги, Мостафа; Улен, Матиас; Нюрен, Пол (17 июля 1998 г.). «Метод секвенирования на основе пирофосфата реального времени». Наука . 281 (5375): 363–365. дои : 10.1126/science.281.5375.363 . ПМИД 9705713 . S2CID 26331871 .
- ^ Перепел М.А., Гу Ю, Свердлов Х., Мэйхо М. (2012). «Оценка и оптимизация препаративных полуавтоматических систем электрофореза для подготовки библиотеки Illumina». Электрофорез . 33 (23): 3521–28. дои : 10.1002/elps.201200128 . ПМИД 23147856 . S2CID 39818212 .
- ^ Дюхайм М.Б., Дэн Л., Пулос Б.Т., Салливан М.Б. (2012). «На пути к количественной метагеномике диких вирусов и других образцов ДНК со сверхнизкими концентрациями: строгая оценка и оптимизация метода линкерной амплификации» . Окружающая среда. Микробиол . 14 (9): 2526–37. Бибкод : 2012EnvMi..14.2526D . дои : 10.1111/j.1462-2920.2012.02791.x . ПМК 3466414 . ПМИД 22713159 .
- ^ Петерсон Б.К., Вебер Дж.Н., Кей Э.Х., Фишер Х.С., Хукстра Х.Э. (2012). «Двойной дайджест RADseq: недорогой метод обнаружения и генотипирования SNP de novo у модельных и немодельных видов» . ПЛОС ОДИН . 7 (5): e37135. Бибкод : 2012PLoSO...737135P . дои : 10.1371/journal.pone.0037135 . ПМК 3365034 . ПМИД 22675423 .
- ^ Уильямс Р., Пейсайович С.Г., Миллер О.Дж., Магдасси С., Тауфик Д.С., Гриффитс А.Д. (2006). «Амплификация сложных библиотек генов методом эмульсионной ПЦР». Природные методы . 3 (7): 545–50. дои : 10.1038/nmeth896 . ПМИД 16791213 . S2CID 27459628 .
- ^ Jump up to: а б Маргулис М., Эгхольм М. и др. (сентябрь 2005 г.). «Секвенирование генома в открытых микрофабрикатах пиколитровых реакторов высокой плотности» . Природа . 437 (7057): 376–80. Бибкод : 2005Natur.437..376M . дои : 10.1038/nature03959 . ПМЦ 1464427 . ПМИД 16056220 .
- ^ Шендуре Дж., Поррека Г.Дж., Реппас Н.Б., Лин X, Маккатчеон Дж.П., Розенбаум А.М., Ван М.Д., Чжан К., Митра Р.Д., Черч ГМ (2005). «Точное мультиплексное секвенирование полонии эволюционировавшего бактериального генома» . Наука . 309 (5741): 1728–32. Бибкод : 2005Sci...309.1728S . дои : 10.1126/science.1117389 . ПМИД 16081699 . S2CID 11405973 .
- ^ «Прикладные биосистемы — файл не найден (ошибка 404)» . 16 мая 2008 г. Архивировано из оригинала 16 мая 2008 г.
- ^ Гудвин С., Макферсон Джей.Д., МакКомби В.Р. (май 2016 г.). «Взросление: десять лет технологий секвенирования нового поколения» . Обзоры природы Генетика . 17 (6): 333–51. дои : 10.1038/nrg.2016.49 . ПМЦ 10373632 . ПМИД 27184599 . S2CID 8295541 .
- ^ Стаден Р. (11 июня 1979 г.). «Стратегия секвенирования ДНК с использованием компьютерных программ» . Исследования нуклеиновых кислот . 6 (7): 2601–10. дои : 10.1093/нар/6.7.2601 . ПМК 327874 . ПМИД 461197 .
- ^ де Магальяйнс Дж.П., Финч С.Э., Янссенс Г. (2010). «Секвенирование нового поколения в исследованиях старения: новые приложения, проблемы, подводные камни и возможные решения» . Обзоры исследований старения . 9 (3): 315–23. doi : 10.1016/j.arr.2009.10.006 . ПМЦ 2878865 . ПМИД 19900591 .
- ^ Града А (август 2013 г.). «Секвенирование нового поколения: методология и применение» . Джей Инвест Дерматол . 133 (8): е11. дои : 10.1038/jid.2013.248 . ПМИД 23856935 .
- ^ Зал N (май 2007 г.). «Передовые технологии секвенирования и их более широкое влияние в микробиологии» . Дж. Эксп. Биол. 210 (Часть 9): 1518–25. дои : 10.1242/jeb.001370 . ПМИД 17449817 .

- ^ Черч ГМ (январь 2006 г.). «Геномы для всех». наук. Являюсь. 294 (1): 46–54. Бибкод : 2006SciAm.294a..46C . doi : 10.1038/scientificamerican0106-46 . ПМИД 16468433 . S2CID 28769137 . (требуется подписка)
- ^ Jump up to: а б с Шустер СК (январь 2008 г.). «Секвенирование нового поколения меняет современную биологию». Нат. Методы . 5 (1): 16–18. дои : 10.1038/nmeth1156 . ПМИД 18165802 . S2CID 1465786 .
- ^ Калб, Гилберт; Моксли, Роберт (1992). Массовые параллельные, оптические и нейронные вычисления в США . IOS Пресс . ISBN 978-90-5199-097-3 . [ нужна страница ]
- ^ Тен Бош-младший, Гроди В.В. (2008). «Идти в ногу со следующим поколением» . Журнал молекулярной диагностики . 10 (6): 484–92. дои : 10.2353/jmoldx.2008.080027 . ПМК 2570630 . ПМИД 18832462 .

- ^ Такер Т., Марра М., Фридман Дж. М. (2009). «Массовое параллельное секвенирование: следующий большой шаг в генетической медицине» . Американский журнал генетики человека . 85 (2): 142–54. дои : 10.1016/j.ajhg.2009.06.022 . ПМЦ 2725244 . ПМИД 19679224 .

- ^ Jump up to: а б Стрейтон Дж., Фри Т., Сойер А., Мартин Дж. (февраль 2019 г.). «От секвенирования по Сэнгеру до баз данных геномов и не только» . БиоТехники . 66 (2). Наука будущего: 60–63. дои : 10.2144/btn-2019-0011 . ПМИД 30744413 .
- ^ Куэйл М.А., Смит М., Коупленд П., Отто Т.Д., Харрис С.Р., Коннор Т.Р., Бертони А., Свердлов Х.П., Га Ю (1 января 2012 г.). «Рассказ о трех платформах секвенирования следующего поколения: сравнение секвенаторов Ion Torrent, Pacific Biosciences и Ilumina MiSeq» . БМК Геномика . 13 (1): 341. дои : 10.1186/1471-2164-13-341 . ПМЦ 3431227 . ПМИД 22827831 .

- ^ Лю Л, Ли Й, Ли С, Ху Н, Хэ Й, Понг Р, Лин Д, Лу Л, Лоу М (1 января 2012 г.). «Сравнение систем секвенирования следующего поколения» . Журнал биомедицины и биотехнологии . 2012 : 251364. дои : 10.1155/2012/251364 . ПМЦ 3398667 . ПМИД 22829749 .

- ^ Jump up to: а б с «Новое программное обеспечение, полимераза для повышения производительности и доступности системы Sequel - PacBio» . 7 марта 2018 г.
- ^ «После года испытаний двое первых клиентов PacBio ожидают более регулярного использования секвенатора RS в 2012 году» . ГеномВеб. 10 января 2012г. ( требуется регистрация )
- ^ «Pacific Biosciences представляет новую химию с большей длиной считывания для обнаружения новых особенностей в последовательностях ДНК и перспективных исследований генома крупных организмов» (пресс-релиз). 2013.
- ^ Чин К.С., Александр Д.Х., Маркс П., Кламмер А.А., Дрейк Дж., Хайнер С., Клам А., Коупленд А., Хаддлстон Дж., Эйхлер Э.Э., Тернер С.В., Корлах Дж. (2013). «Негибридные готовые сборки микробного генома на основе давно прочитанных данных секвенирования SMRT». Нат. Методы . 10 (6): 563–69. дои : 10.1038/nmeth.2474 . ПМИД 23644548 . S2CID 205421576 .
- ^ Jump up to: а б «Сборка бактериального генома de novo: решенная проблема?» . 5 июля 2013 г.
- ^ Раско Д.А., Вебстер Д.Р., Саль Дж.В., Башир А., Бойзен Н., Шойц Ф., Паксинос Е.Е., Себра Р., Чин К.С., Илиопулос Д., Кламмер А., Пелусо П., Ли Л., Кислюк А.О., Буллард Дж., Касарскис А., Ван С. , Ид Дж., Ранг D, Редман Дж.К., Стерд С.Р., Фримодт-Мёллер Дж., Струве С., Петерсен А.М., Крогфельт К.А., Натаро Дж.П., Шадт Э.Э., Вальдор М.К. (25 августа 2011 г.). «Происхождение штамма, вызвавшего вспышку гемолитико-уремического синдрома в Германии» . N Engl J Med . 365 (8): 709–17. дои : 10.1056/NEJMoa1106920 . ПМК 3168948 . ПМИД 21793740 .

- ^ Тран Б, Браун А.М., Бедард П.Л., Винквист Э., Госс Г.Д., Хотте С.Дж., Уэлч С.А., Хирте Х.В., Чжан Т., Стейн Л.Д., Ферретти В., Уотт С. , Цзяо В., Нг К., Гай С., Шоу П., Петрочелли Т. , Хадсон Т.Дж. , Нил Б.Г., Онетто Н., Сиу Л.Л., Макферсон Дж.Д., Камел-Рид С., Дэнси Дж.Э. (1 января 2012 г.). «Возможность секвенирования раковых генов нового поколения в реальном времени, связанного с реакцией на лекарства: результаты клинических испытаний» . Межд. Дж. Рак . 132 (7): 1547–55. дои : 10.1002/ijc.27817 . ПМИД 22948899 . S2CID 72705 . (требуется подписка)
- ^ Мюррей И.А., Кларк Т.А., Морган Р.Д., Бойтано М., Антон Б.П., Луонг К., Фоменков А., Тернер С.В., Корлах Дж., Робертс Р.Дж. (2 октября 2012 г.). «Метиломы шести бактерий» . Исследования нуклеиновых кислот . 40 (22): 11450–62. дои : 10.1093/nar/gks891 . ПМК 3526280 . ПМИД 23034806 .
- ^ «Комплект-Chef Ion 520 и Ion 530 ExT – Thermo Fisher Scientific» . thermofisher.com .
- ^ «Чистая точность» . Архивировано из оригинала 30 марта 2018 года . Проверено 29 марта 2018 г.
- ^ ван Влит АХ (1 января 2010 г.). «Секвенирование микробных транскриптомов нового поколения: проблемы и возможности» . Письма FEMS по микробиологии . 302 (1): 1–7. дои : 10.1111/j.1574-6968.2009.01767.x . ПМИД 19735299 .

- ^ «БГИ и МГИСЭК» . ru.mgitech.cn . Проверено 5 июля 2018 г.
- ^ Jump up to: а б Хуан Ю.Ф., Чен С.С., Чан Ю.С., Чен Т.Х., Чиу К.П. (2012). «Палиндромная последовательность препятствует механизму секвенирования путем лигирования» . Системная биология BMC . 6 (Дополнение 2): S10. дои : 10.1186/1752-0509-6-S2-S10 . ПМК 3521181 . ПМИД 23281822 .
- ^ Свободный, Мэтью; Ракян, Вардхман; Холмс, Надин; Пейн, Александр (3 мая 2018 г.). «Наблюдение за китами с помощью BulkVis: графический просмотрщик оптовых файлов Oxford Nanopore fast5». bioRxiv 10.1101/312256 .
- ^ «Продажи PacBio начинают расти по мере того, как компания совершенствует свою продукцию» . 12 февраля 2013 г.
- ^ «Мир БиоИТ» . bio-itworld.com . Архивировано из оригинала 29 июля 2020 года . Проверено 16 ноября 2015 г.
- ^ «PacBio представляет высокопроизводительную и недорогую систему секвенирования одиночных молекул» . Октябрь 2015.
- ^ Кларк Дж., Ву Х.К., Джаясингхе Л., Патель А., Рид С., Бэйли Х. (апрель 2009 г.). «Непрерывная идентификация оснований для секвенирования ДНК нанопор одной молекулы». Природные нанотехнологии . 4 (4): 265–70. Бибкод : 2009NatNa...4..265C . дои : 10.1038/nnano.2009.12 . ПМИД 19350039 .
- ^ Jump up to: а б Дела Торре Р., Ларкин Дж., Сингер А., Меллер А. (2012). «Изготовление и характеристика твердотельных массивов нанопор для высокопроизводительного секвенирования ДНК» . Нанотехнологии . 23 (38): 385308. Бибкод : 2012Nanot..23L5308D . дои : 10.1088/0957-4484/23/38/385308 . ПМЦ 3557807 . ПМИД 22948520 .
- ^ Jump up to: а б Патак Б., Лофас Х., Прасонгкит Дж., Григорьев А., Ахуджа Р., Шайхер Р.Х. (2012). «Двойные функциональные золотые электроды со встроенными нанопорами для быстрого секвенирования ДНК» . Письма по прикладной физике . 100 (2): 023701. Бибкод : 2012ApPhL.100b3701P . дои : 10.1063/1.3673335 .
- ^ Корлах Дж., Маркс П.Дж., Цицерон Р.Л., Грей Дж.Дж., Мерфи Д.Л., Ройтман Д.Б., Фам Т.Т., Отто Г.А., Фоке М., Тернер С.В. (2008). «Селективная пассивация алюминием для направленной иммобилизации одиночных молекул ДНК-полимеразы в волноводных наноструктурах с нулевой модой» . Труды Национальной академии наук . 105 (4): 1176–81. Бибкод : 2008PNAS..105.1176K . дои : 10.1073/pnas.0710982105 . ПМК 2234111 . ПМИД 18216253 .
- ^ Jump up to: а б Шендуре Дж., Поррека Г.Дж., Реппас Н.Б., Лин X, Маккатчеон Дж.П., Розенбаум А.М., Ван М.Д., Чжан К., Митра Р.Д., Черч ГМ (9 сентября 2005 г.). «Точное мультиплексное секвенирование полоний эволюционировавшего бактериального генома» . Наука . 309 (5741): 1728–32. Бибкод : 2005Sci...309.1728S . дои : 10.1126/science.1117389 . ПМИД 16081699 . S2CID 11405973 .
- ^ Бентли Д.Р., Баласубраманиан С. и др. (2008). «Точное секвенирование всего генома человека с использованием обратимой химии терминаторов» . Природа . 456 (7218): 53–59. Бибкод : 2008Natur.456...53B . дои : 10.1038/nature07517 . ПМК 2581791 . ПМИД 18987734 .
- ^ Канард Б., Сарфати С. (13 октября 1994 г.), Новые производные, используемые для секвенирования нуклеиновых кислот , получено 9 марта 2016 г.
- ^ Канард Б., Сарфати Р.С. (октябрь 1994 г.). «Флуоресцентные субстраты ДНК-полимеразы с обратимыми 3'-метками». Джин . 148 (1): 1–6. дои : 10.1016/0378-1119(94)90226-7 . ПМИД 7523248 .
- ^ Мардис ЭР (2008). «Методы секвенирования ДНК нового поколения». Анну Рев Геном Хум Генет . 9 : 387–402. дои : 10.1146/annurev.genom.9.081307.164359 . ПМИД 18576944 .
- ^ Jump up to: а б с Дрманак Р., Спаркс А.Б., Кэллоу М.Дж., Халперн А.Л., Бернс Н.Л., Кермани Б.Г. и др. (январь 2010 г.). «Секвенирование генома человека с использованием чтения несцепленных оснований на самособирающихся наноматрицах ДНК» . Наука . 327 (5961): 78–81. Бибкод : 2010Sci...327...78D . дои : 10.1126/science.1181498 . ПМИД 19892942 . S2CID 17309571 .
- ^ брендонвд. «О нас – Полная геномика» . Полная геномика . Проверено 2 июля 2018 г.
- ^ Jump up to: а б Хуан Дж., Лян Х., Сюань Ю., Гэн С., Ли Ю., Лу Х. и др. (май 2017 г.). «Эталонный набор данных генома человека секвенатора BGISEQ-500» . ГигаСайенс . 6 (5): 1–9. doi : 10.1093/gigascience/gix024 . ПМК 5467036 . ПМИД 28379488 .
- ^ Валуев А., Итикава Дж., Тонхат Т., Стюарт Дж., Ранаде С., Пекхэм Х., Зенг К., Малек Дж.А., Коста Г., МакКернан К., Сидоу А., Файр А., Джонсон С.М. (июль 2008 г.). «Карта положения нуклеосом C. elegans с высоким разрешением показывает отсутствие универсального позиционирования, диктуемого последовательностью» . Геном Рез . 18 (7): 1051–63. дои : 10.1101/гр.076463.108 . ПМЦ 2493394 . ПМИД 18477713 .
- ^ Раск Н. (2011). «Потоки последовательности» . Нат-методы . 8 (1): 44. doi : 10.1038/nmeth.f.330 . S2CID 41040192 .
- ^ Jump up to: а б Дрманак Р., Спаркс А.Б. и др. (2010). «Секвенирование генома человека с использованием считываний несцепленных оснований в самособирающихся наноматрицах ДНК» . Наука . 327 (5961): 78–81. Бибкод : 2010Sci...327...78D . дои : 10.1126/science.1181498 . ПМИД 19892942 . S2CID 17309571 .
- ^ Поррека Дж.Дж. (2010). «Секвенирование генома на наношариках». Природная биотехнология . 28 (1): 43–44. дои : 10.1038/nbt0110-43 . ПМИД 20062041 . S2CID 54557996 .
- ^ «Система секвенирования генов / генетического анализатора HeliScope: Helicos BioSciences» . 2 ноября 2009 г. Архивировано из оригинала 2 ноября 2009 г.
- ^ Томпсон Дж. Ф., Штайнманн К. Э. (октябрь 2010 г.). «Секвенирование одной молекулы с помощью системы генетического анализа HeliScope» . Современные протоколы молекулярной биологии . Глава 7: Раздел 7.10. дои : 10.1002/0471142727.mb0710s92 . ПМЦ 2954431 . ПМИД 20890904 .
- ^ «Техническое объяснение tSMS SeqLL» . Продолжение. Архивировано из оригинала 8 августа 2014 года . Проверено 9 августа 2015 г.
- ^ Хизер, Джеймс М.; Чейн, Бенджамин (январь 2016 г.). «Последовательность секвенаторов: История секвенирования ДНК» . Геномика . 107 (1): 1–8. дои : 10.1016/j.ygeno.2015.11.003 . ПМЦ 4727787 . ПМИД 26554401 .
- ^ Сара Эль-Метвалли; Усама М. Уда; Мохамед Хелми (2014). «Новые горизонты секвенирования следующего поколения». Технологии секвенирования следующего поколения и проблемы сборки последовательностей . SpringerBriefs по системной биологии. Том. 7. Технологии секвенирования следующего поколения и проблемы сборки последовательностей, Springer Briefs in Systems Biology, том 7. стр. 51–59. дои : 10.1007/978-1-4939-0715-1_6 . ISBN 978-1-4939-0714-4 .
- ^ Jump up to: а б Фэйр РБ, Хлыстов А, Тейлор ТД, Иванов В, Эванс РД, Шринивасан В, Памула ВК, Поллак М.Г., Гриффин П.Б., Чжоу Дж (январь 2007 г.). «Химические и биологические применения цифровых микрофлюидных устройств». IEEE Проектирование и тестирование компьютеров . 24 (1): 10–24. CiteSeerX 10.1.1.559.1440 . дои : 10.1109/MDT.2007.8 . hdl : 10161/6987 . S2CID 10122940 .
- ^ Jump up to: а б Болес Д.Д., Бентон Дж.Л., Сью Г.Дж., Леви М.Х., Твар П.К., Сандал М.А. и др. (ноябрь 2011 г.). «Капельное пиросеквенирование с использованием цифровой микрофлюидики» . Аналитическая химия . 83 (22): 8439–47. дои : 10.1021/ac201416j . ПМК 3690483 . ПМИД 21932784 .
- ^ Зилионис Р., Найнис Дж., Верес А., Савова В., Земмур Д., Кляйн А.М., Мазутис Л. (январь 2017 г.). «Одноклеточное штрих-кодирование и секвенирование с использованием капельной микрофлюидики». Протоколы природы . 12 (1): 44–73. дои : 10.1038/nprot.2016.154 . ПМИД 27929523 . S2CID 767782 .
- ^ «Гарвардская группа нанопоров» . Mcb.harvard.edu. Архивировано из оригинала 21 февраля 2002 года . Проверено 15 ноября 2009 г.
- ^ «Секвенирование нанопор может сократить затраты на анализ ДНК» .
- ^ Патент США 20060029957 , ZS Genetics, «Системы и методы анализа полимеров нуклеиновых кислот и родственных компонентов», выдан 14 июля 2005 г.
- ^ Сюй М., Фудзита Д., Ханагата Н. (декабрь 2009 г.). «Перспективы и проблемы новых технологий секвенирования одномолекулярной ДНК». Маленький . 5 (23): 2638–49. дои : 10.1002/smll.200900976 . ПМИД 19904762 .
- ^ Шадт Э.Э., Тернер С., Касарскис А. (2010). «Окно в секвенирование третьего поколения» . Молекулярная генетика человека . 19 (Р2): 227–40 Рэндов. дои : 10.1093/hmg/ddq416 . ПМИД 20858600 .
- ^ Сюй М., Эндрес Р.Г., Аракава Ю. (2007). «Электронные свойства оснований ДНК» . Маленький . 3 (9): 1539–43. дои : 10.1002/smll.200600732 . ПМИД 17786897 .
- ^ Ди Вентра М (2013). «Быстрое секвенирование ДНК с помощью электрических средств стало на несколько дюймов ближе» . Нанотехнологии . 24 (34): 342501. Бибкод : 2013Nanot..24H2501D . дои : 10.1088/0957-4484/24/34/342501 . ПМИД 23899780 . S2CID 140101884 .
- ^ Оширо Т., Мацубара К., Цуцуи М., Фурухаши М., Танигучи М., Каваи Т. (2012). «Одномолекулярное электрическое случайное повторное секвенирование ДНК и РНК» . Научный представитель . 2 : 501. Бибкод : 2012NatSR...2E.501O . дои : 10.1038/srep00501 . ПМЦ 3392642 . ПМИД 22787559 .
- ^ Ханна Г.Дж., Джонсон В.А., Куритцкес Д.Р. , Ричман Д.Д., Мартинес-Пикадо Дж., Саттон Л., Хейзелвуд Дж.Д., Д'Акуила Р.Т. (1 июля 2000 г.). «Сравнение секвенирования путем гибридизации и циклического секвенирования для генотипирования обратной транскриптазы вируса иммунодефицита человека типа 1» . Дж. Клин. Микробиол . 38 (7): 2715–21. doi : 10.1128/JCM.38.7.2715-2721.2000 . ПМК 87006 . ПМИД 10878069 .
- ^ Jump up to: а б Мори М., Фернандес-Мармисс А., Кастинейрас Д., Фрага Х.М., Косе М.Л., Кочо Х.А. (2013). «Взгляд на прошлое, настоящее и будущее секвенирования ДНК». Молекулярная генетика и обмен веществ . 110 (1–2): 3–24. дои : 10.1016/j.ymgme.2013.04.024 . ПМИД 23742747 .
- ^ Цинь Ю, Шнайдер Т.М., Бреннер, член парламента (2012). Гибас С. (ред.). «Секвенирование путем гибридизации длинных мишеней» . ПЛОС ОДИН . 7 (5): e35819. Бибкод : 2012PLoSO...735819Q . дои : 10.1371/journal.pone.0035819 . ПМЦ 3344849 . ПМИД 22574124 .
- ^ Эдвардс-младший, Рупарел Х, Джу Дж (2005). «Масс-спектрометрическое секвенирование ДНК». Мутационные исследования . 573 (1–2): 3–12. дои : 10.1016/j.mrfmmm.2004.07.021 . ПМИД 15829234 .
- ^ Холл Т.А., Будоул Б., Цзян Й., Блин Л., Эшу М., Саннес-Лоури К.А., Сампат Р., Дрейдер Дж.Дж., Ханнис Дж.К., Харрелл П., Самант В., Уайт Н., Экер DJ, Хофстадлер С.А. (2005). «Анализ базового состава митохондриальной ДНК человека с использованием масс-спектрометрии с ионизацией электрораспылением: новый инструмент для идентификации и дифференциации человека». Аналитическая биохимия . 344 (1): 53–69. дои : 10.1016/j.ab.2005.05.028 . ПМИД 16054106 .
- ^ Ховард Р., Энчева В., Томсон Дж., Бач К., Чан Ю.Т., Коуэн С., Дебенхэм П., Диксон А., Краузе Ю., Кришан Е., Мур Д., Мур В., Охо М., Родригес С., Стоукс П., Уокер Дж., Циммерманн В. , Бараллон Р. (15 июня 2011 г.). «Сравнительный анализ митохондриальной ДНК человека из образцов костей времен Первой мировой войны с помощью секвенирования ДНК и масс-спектрометрии ESI-TOF» . Международная судебно-медицинская экспертиза: Генетика . 7 (1): 1–9. дои : 10.1016/j.fsigen.2011.05.009 . ПМИД 21683667 .
- ^ Монфорте Х.А., Беккер CH (1 марта 1997 г.). «Высокопроизводительный анализ ДНК методом времяпролетной масс-спектрометрии». Природная медицина . 3 (3): 360–62. дои : 10.1038/nm0397-360 . ПМИД 9055869 . S2CID 28386145 .
- ^ Берес С.Б., Кэрролл Р.К., Ши П.Р., Ситкевич И., Мартинес-Гутьеррес Х.К., Лоу Д.Э., МакГир А., Уилли Б.М., Грин К., Тиррелл Дж.Дж., Голдман Т.Д., Фельдгарден М., Биррен Б.В., Фофанов Ю., Боос Дж., Уитон В.Д., Хониш С., Массер Дж. М. (8 февраля 2010 г.). «Молекулярная сложность последовательных бактериальных эпидемий, раскрытая с помощью сравнительной патогеномики» . Труды Национальной академии наук . 107 (9): 4371–76. Бибкод : 2010PNAS..107.4371B . дои : 10.1073/pnas.0911295107 . ПМК 2840111 . ПМИД 20142485 .
- ^ Кан К.В., Фредлейк С.П., Доэрти Э.А., Бэррон А.Е. (1 ноября 2004 г.). «Секвенирование ДНК и генотипирование в миниатюрных системах электрофореза». Электрофорез . 25 (21–22): 3564–88. дои : 10.1002/elps.200406161 . ПМИД 15565709 . S2CID 4851728 .
- ^ Чен Ю.Дж., Роллер Э.Э., Хуан Икс (2010). «Секвенирование ДНК путем денатурации: экспериментальное подтверждение концепции с помощью встроенного жидкостного устройства» . Лаборатория на чипе . 10 (9): 1153–59. дои : 10.1039/b921417h . ПМЦ 2881221 . ПМИД 20390134 .
- ^ Белл, округ Колумбия, Томас В.К., Мурта К.М., Дионн К.А., Грэм А.С., Андерсон Дж.Э., Гловер В.Р. (9 октября 2012 г.). «Идентификация оснований ДНК методом электронной микроскопии». Микроскопия и микроанализ . 18 (5): 1049–53. Бибкод : 2012MiMic..18.1049B . дои : 10.1017/S1431927612012615 . ПМИД 23046798 . S2CID 25713635 .
- ^ Парик К.С., Смочинский Р., Третин А. (ноябрь 2011 г.). «Технологии секвенирования и секвенирование генома» . Журнал прикладной генетики . 52 (4): 413–35. дои : 10.1007/s13353-011-0057-x . ПМК 3189340 . ПМИД 21698376 .
- ^ Парик К.С., Смочинский Р., Третин А. (2011). «Технологии секвенирования и секвенирование генома» . Журнал прикладной генетики . 52 (4): 413–35. дои : 10.1007/s13353-011-0057-x . ПМК 3189340 . ПМИД 21698376 .
- ^ Фухимори С., Хираи Н., Охаси Х., Масуока К., Нисикими А., Фукуи Ю., Васио Т., Осикубо Т., Ямасита Т., Миямото-Сато Е (2012). «Секвенирование нового поколения в сочетании с технологией бесклеточного дисплея для высокопроизводительного получения надежных данных интерактома» . Научные отчеты . 2 : 691. Бибкод : 2012NatSR...2E.691F . дои : 10.1038/srep00691 . ПМЦ 3466446 . ПМИД 23056904 .
- ^ «Доля рынка секвенирования в 2022 году – такая же, какой была всегда (на данный момент)» . 25 июня 2023 г.
- ^ Харберс М. (2008). «Текущий статус клонирования кДНК» . Геномика . 91 (3): 232–42. дои : 10.1016/j.ygeno.2007.11.004 . ПМИД 18222633 .
- ^ Альберти А., Бельсер С., Энгелен С., Бертран Л., Орвен С., Бринас Л., Круо С. и др. (2014). «Сравнение методов подготовки библиотек показывает их влияние на интерпретацию метатранскриптомных данных» . БМК Геномика . 15 (1): 912–12. дои : 10.1186/1471-2164-15-912 . ПМК 4213505 . ПМИД 25331572 .
- ^ «Масштабируемые оценки качества нуклеиновых кислот для подготовки библиотеки секвенирования следующего поколения Illumina» (PDF) . Проверено 27 декабря 2017 г.
- ^ «Архонт Геномика XPRIZE» . Archon Genomics XPRIZE . Архивировано из оригинала 17 июня 2013 года . Проверено 9 августа 2007 г.
- ^ «Информация о гранте» . Национальный институт исследования генома человека (NHGRI) .
- ^ Северин Дж., Лицио М., Харшбаргер Дж., Каваджи Х., Дауб К.О., Хаяшизаки Ю., Бертин Н., Форрест А.Р. (2014). «Интерактивная визуализация и анализ крупномасштабных наборов данных секвенирования с использованием ZENBU». Нат. Биотехнология . 32 (3): 217–19. дои : 10.1038/nbt.2840 . ПМИД 24727769 . S2CID 26575621 .
- ^ Шмилович А, Бен-Гал I (2007). «Использование модели VOM для реконструкции потенциальных кодирующих областей в последовательностях EST» (PDF) . Вычислительная статистика . 22 (1): 49–69. дои : 10.1007/s00180-007-0021-8 . S2CID 2737235 . Архивировано из оригинала (PDF) 31 мая 2020 года . Проверено 10 января 2014 г.
- ^ Дель Фаббро С., Скалабрин С., Морганте М., Джорджи Ф.М. (2013). «Обширная оценка влияния обрезки чтения на анализ данных Illumina NGS» . ПЛОС ОДИН . 8 (12): е85024. Бибкод : 2013PLoSO...885024D . дои : 10.1371/journal.pone.0085024 . ПМЦ 3871669 . ПМИД 24376861 .
- ^ Мартин, Марсель (2 мая 2011 г.). «Cutadapt удаляет адаптерные последовательности из операций чтения высокопроизводительного секвенирования» . EMBnet.журнал . 17 (1): 10. doi : 10.14806/ej.17.1.200 .
- ^ Смедс Л., Кюнстнер А (19 октября 2011 г.). «ConDeTri — триммер чтения данных Illumina, зависящий от содержимого» . ПЛОС ОДИН . 6 (10): e26314. Бибкод : 2011PLoSO...626314S . дои : 10.1371/journal.pone.0026314 . ПМК 3198461 . ПМИД 22039460 .
- ^ Прецца Н., Дель Фаббро С., Вецци Ф., Де Паоли Э., Поликрити А. (2012). «Эрнэ-Бс5». Материалы конференции ACM по биоинформатике, вычислительной биологии и биомедицине . Том 12. стр. 12–19. дои : 10.1145/2382936.2382938 . ISBN 9781450316705 . S2CID 5673753 .
- ^ Шмидер Р., Эдвардс Р. (март 2011 г.). «Контроль качества и предварительная обработка наборов метагеномных данных» . Биоинформатика . 27 (6): 863–4. doi : 10.1093/биоинформатика/btr026 . ПМК 3051327 . ПМИД 21278185 .
- ^ Болджер А.М., Лозе М., Усадель Б. (август 2014 г.). «Trimmomatic: гибкий триммер для данных последовательности Illumina» . Биоинформатика . 30 (15): 2114–20. doi : 10.1093/биоинформатика/btu170 . ПМК 4103590 . ПМИД 24695404 .
- ^ Член парламента Кокса, Петерсон Д.А., Биггс П.Дж. (сентябрь 2010 г.). «SolexaQA: Краткая оценка качества данных секвенирования второго поколения Illumina» . БМК Биоинформатика . 11 (1): 485. дои : 10.1186/1471-2105-11-485 . ПМЦ 2956736 . ПМИД 20875133 .
- ^ Мюррей TH (январь 1991 г.). «Этические проблемы исследования генома человека» . Журнал ФАСЭБ . 5 (1): 55–60. дои : 10.1096/fasebj.5.1.1825074 . ПМИД 1825074 . S2CID 20009748 .
- ^ Jump up to: а б с Робертсон Дж. А. (август 2003 г.). «Геном за 1000 долларов: этические и юридические проблемы полногеномного секвенирования людей». Американский журнал биоэтики . 3 (3): W–IF1. дои : 10.1162/152651603322874762 . ПМИД 14735880 . S2CID 15357657 .
- ^ Jump up to: а б Хендерсон, Марк (9 сентября 2013 г.). «Секвенирование генома человека: настоящие этические дилеммы» . Хранитель . Проверено 20 мая 2015 г.
- ^ Хармон, Эми (24 февраля 2008 г.). «Страхи страховки заставляют многих избегать тестов ДНК» . Нью-Йорк Таймс . Проверено 20 мая 2015 г.
- ↑ Заявление о политике администрации , Административно-бюджетное управление администрации президента, 27 апреля 2007 г.
- ^ Национальный институт исследования генома человека (21 мая 2008 г.). «Президент Буш подписывает Закон о недискриминации генетической информации 2008 года» . Проверено 17 февраля 2014 г.
- ^ Бейкер, Моня (11 октября 2012 г.). «Отчеты комиссии по этике США о секвенировании ДНК и конфиденциальности» . Блог новостей природы .
- ^ «Конфиденциальность и прогресс в полногеномном секвенировании» (PDF) . Президентская комиссия по изучению вопросов биоэтики. Архивировано из оригинала (PDF) 12 июня 2015 года . Проверено 20 мая 2015 г.
- ^ Хартнетт, Кевин (12 мая 2013 г.). «ДНК в вашем мусоре: открыта для всех» . Бостон Глобус . Проверено 2 января 2023 г.
- ^ Гольденберг А.Дж., Шарп Р.Р. (февраль 2012 г.). «Этические опасности и программные проблемы геномного скрининга новорожденных» . ДЖАМА . 307 (5): 461–2. дои : 10.1001/jama.2012.68 . ПМЦ 3868436 . ПМИД 22298675 .
- ^ Хьюз, Вирджиния (7 января 2013 г.). «Пришло время перестать зацикливаться на опасностях генетической информации» . Журнал «Сланец» . Проверено 22 мая 2015 г.
- ^ Jump up to: а б Блосс К.С., Шорк, Нью-Джерси, Тополь Э.Дж. (февраль 2011 г.). «Эффект полногеномного профилирования непосредственно к потребителю для оценки риска заболеваний» . Медицинский журнал Новой Англии . 364 (6): 524–34. дои : 10.1056/NEJMoa1011893 . ПМЦ 3786730 . ПМИД 21226570 .
- ^ Рочман, Бонни (25 октября 2012 г.). «Что ваш врач не говорит вам о вашей ДНК» . Время.com . Проверено 22 мая 2015 г.
- ^ Саджир П., Мухаммад (4 мая 2023 г.). «Прорывная технология: изучение этических, юридических, политических и социальных последствий технологии секвенирования нанопор» . Отчеты ЭМБО . 24 (5): e56619. дои : 10.15252/эмбр.202256619 . ПМЦ 10157308 . ПМИД 36988424 . S2CID 257803254 .