ЧИП-экзо
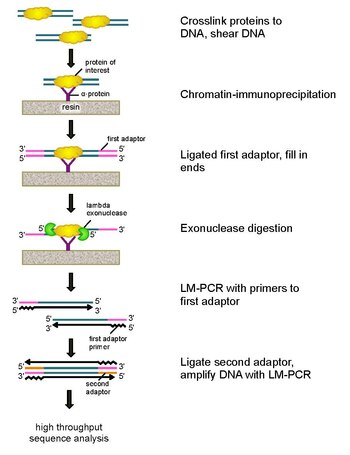
ChIP-exo — это метод, основанный на иммунопреципитации хроматина, для картирования мест, в которых интересующий белок ( фактор транскрипции ) связывается с геномом. Это модификация протокола ChIP-seq , улучшающая разрешение сайтов связывания с сотен пар оснований почти до одной пары оснований. Он использует экзонуклеазы для разрушения нитей связанной с белком ДНК в направлении 5'-3' с точностью до небольшого числа нуклеотидов от места связывания белка. Нуклеотиды обработанных экзонуклеазой концов определяются с использованием некоторой комбинации секвенирования ДНК , микрочипов и ПЦР . Затем эти последовательности наносятся на геном, чтобы определить участки генома, с которыми связывается белок.
Теория
[ редактировать ]Методы иммунопреципитации хроматина ( ChIP ) используются с 1984 года. [1] для обнаружения взаимодействий белок-ДНК. Было много вариаций ChIP для улучшения качества результатов. Одно из таких усовершенствований, ChIP-on-chip (ChIP-chip), сочетает в себе ChIP с технологией микрочипов. Этот метод имеет ограниченную чувствительность и специфичность, особенно in vivo, где микрочипы ограничены тысячами белков, присутствующих в ядерном компартменте, что приводит к высокому уровню ложноположительных результатов. [2] Далее последовало ChIP-секвенирование (ChIP-seq), которое сочетает в себе ChIP с высокопроизводительным секвенированием. [3] Однако гетерогенная природа расщепленных фрагментов ДНК отображает сайты связывания с точностью до ± 300 пар оснований, что ограничивает специфичность. Во-вторых, загрязнение ДНК представляет собой серьезную проблему, поскольку с интересующим белком перекрестно связаны очень немногие генетические локусы, что делает любую неспецифическую геномную ДНК значительным источником фонового шума. [4]
Чтобы решить эти проблемы, Ри и Пью пересмотрели классический анализ защиты от нуклеазы и разработали ChIP-exo. [5] Этот новый метод ChIP основан на лямбда -экзонуклеазе , которая разрушает только и всю несвязанную двухцепочечную ДНК в направлении 5'-3'. Вкратце, интересующий белок (создание белка с эпитопной меткой может быть полезно для иммунопреципитации) перекрестно сшивается in vivo с его естественными местами связывания по всему геному с помощью формальдегида.
Затем клетки собирают, разламывают, а хроматин разрезают и солюбилизируют ультразвуком . Затем антитело используется для иммунопреципитации интересующего белка вместе со сшитой ДНК. Затем к концам лигируют адаптеры ДНК-ПЦР, которые служат точкой запуска для синтеза второй цепи ДНК после расщепления экзонуклеазой. Затем лямбда-экзонуклеаза переваривает двойные цепи ДНК с 5'-конца до тех пор, пока переваривание не блокируется на границе ковалентного взаимодействия белок-ДНК. Большая часть загрязняющей ДНК разрушается за счет добавления второй одноцепочечной специфичной экзонуклеазы. После обратного сшивания праймеры к адаптерам ПЦР удлиняются с образованием двухцепочечной ДНК, а второй адаптер лигируется к 5'-концам, чтобы определить точное место прекращения расщепления экзонуклеазой. Затем библиотеку амплифицируют с помощью ПЦР, а продукты идентифицируют с помощью высокопроизводительного секвенирования . Этот метод позволяет получить разрешение до одной пары оснований для любого сайта связывания белка в любом геноме, что является гораздо более высоким разрешением, чем ChIP-чип или ChIP-seq.
Преимущества
[ редактировать ]Было показано, что ChIP-exo дает разрешение до одной пары оснований при определении мест связывания белка. В этом отличие от ChIP-seq, который может определить место связывания белка только с точностью до ± 300 пар оснований. [4]
Загрязнение несвязанных с белками фрагментов ДНК может привести к высокому уровню ложноположительных и отрицательных результатов в экспериментах ChIP. Добавление экзонуклеаз в процесс не только улучшает разрешение вызова сайта связывания, но и удаляет загрязняющую ДНК из раствора перед секвенированием. [4]
Белки, которые неэффективно связаны с нуклеотидным фрагментом, с большей вероятностью будут обнаружены с помощью ChIP-exo. Это позволило, например, распознать больше сайтов связывания транскрипционных факторов CTCF, чем было обнаружено ранее. [5]
Из-за более высокого разрешения и меньшего фона при использовании ChIP-exo требуется меньшая глубина покрытия секвенирования. [4]
Ограничения
[ редактировать ]Если комплекс белок-ДНК имеет несколько мест перекрестных связей в рамках одного события связывания, то может показаться, что существует несколько различных событий связывания. Вероятно, это является результатом того, что эти белки денатурируются и сшиваются в одном из доступных сайтов связывания в рамках одного и того же события. Затем экзонуклеаза остановится на одном из связанных сайтов, в зависимости от того, с каким сайтом сшит белок. [5]
Как и в случае любого метода на основе ChIP, для использования этого метода необходимо наличие подходящего антитела к интересующему белку.
Приложения
[ редактировать ]Ри и Пью представили ChIP-exo, проведя анализ небольшой коллекции факторов транскрипции: Reb1, Gal4, Phd1, Rap1 у дрожжей и CTCF у человека. Сайты Reb1 часто обнаруживались в кластерах, и заполненность этих кластеров была примерно в 10 раз выше, чем ожидалось. Вторичные сайты в кластерах были обнаружены на расстоянии ~40 п.н. от первичного сайта связывания. Связывающие мотивы Gal4 показали сильное предпочтение трем из четырех нуклеотидов, что указывает на негативное взаимодействие между Gal4 и исключенным нуклеотидом. Phd1 распознает три разных мотива, что объясняет предыдущие сообщения о неоднозначности мотива связывания Phd1. Было обнаружено, что Rap1 распознает четыре мотива.
Гены рибосомальных белков, связанные с этим белком, имели тенденцию использовать определенный мотив с более сильной консенсусной последовательностью. Другие гены часто использовали кластеры более слабых консенсусных мотивов, возможно, для достижения аналогичной занятости. Связывающие мотивы CTCF используют четыре «модуля». Половина связанных сайтов CTCF использовала модули 1 и 2, а остальные использовали некоторую комбинацию из четырех. Считается, что CTCF использует свои цинковые пальцы для распознавания различных комбинаций этих модулей. [5]
Ри и Пью проанализировали структуру и организацию преинициативного комплекса (PIC) в геномах Saccharomyces . Используя ChIP-exo, они смогли, среди других открытий, точно идентифицировать ТАТА-подобные особенности в промоторах, которые, как сообщается, не содержат ТАТА. [6]
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Гилмор, Д.С.; Джей Ти Лис (1983). «Обнаружение взаимодействий белок-ДНК in vivo : распределение РНК-полимеразы по конкретным бактериальным генам» . Труды Национальной академии наук . 81 (14): 4275–4279. дои : 10.1073/pnas.81.14.4275 . ПМК 345570 . ПМИД 6379641 .
- ^ Альберт, я; Т. Н. Маврич; ЛП Томшо; Дж Ци; С. Дж. Зантон; СК Шустер; Б. Ф. Пью (2007). «Настройки трансляции и вращения нуклеосом H2A.Z пересекают геном Saccharomyces cerevisiae ». Природа . 446 (7135): 572–576. Бибкод : 2007Natur.446..572A . дои : 10.1038/nature05632 . ПМИД 17392789 . S2CID 4416890 .
- ^ Рен, Б; Ф Роберт; Джей Джей Уайрик; О Апарисио! Э.Г. Дженнингс; Я Саймон; Дж. Цайтлингер; Дж. Шрайбер; Н. Ханнетт; Э Кан; и др. (2000). «Полногеномное расположение и функция ДНК-связывающих белков». Наука . 290 (5500): 2306–2309. Бибкод : 2000Sci...290.2306R . CiteSeerX 10.1.1.123.6772 . дои : 10.1126/science.290.5500.2306 . ПМИД 11125145 .
- ^ Jump up to: а б с д Пью, Бенджамин. «Методы, системы и наборы для обнаружения белок-нуклеиновых кислотных взаимодействий» . Публикация заявки в США . Патенты США . Проверено 17 февраля 2012 г.
- ^ Jump up to: а б с д Ри, Хо Сун; Би Джей Пью (2011). «Комплексные полногеномные взаимодействия белка и ДНК, обнаруженные при разрешении одного нуклеотида» . Клетка . 147 (6): 1408–1419. дои : 10.1016/j.cell.2011.11.013 . ПМЦ 3243364 . ПМИД 22153082 .
- ^ Ри, Хо Сун; Би Джей Пью (2012). «Полногеномная структура и организация прединициаторных комплексов эукариот» . Природа . 483 (7389): 295–301. Бибкод : 2012Natur.483..295R . дои : 10.1038/nature10799 . ПМК 3306527 . ПМИД 22258509 .
Внешние ссылки
[ редактировать ]- ДНК-белковые взаимодействия в высоком разрешении
- Разрешение связывания фактора транскрипции
- Иммунопреципитация хроматина высокого разрешения
- Важные белки, регулирующие гены, выявлены новым методом
- CexoR: пакет R/Bioconductor для выявления взаимодействий белок-ДНК с высоким разрешением в репликатах ChIP-exo
- Пеконик Геномика