Кинемаг

Кинемаге (сокращение от кинетического изображения ) — это интерактивная графическая научная иллюстрация. Его часто используют для визуализации молекул , особенно белков, хотя он также может представлять другие типы трехмерных данных (например, геометрические фигуры, социальные сети, [1] или тетраэдры базового состава РНК ). Система kinemage разработана для оптимизации простоты использования, интерактивности, а также восприятия и передачи подробной трехмерной информации. Информация о киноизображении хранится в текстовом файле, читаемом человеком и машиной, который описывает иерархию объектов отображения и их свойства и включает дополнительный пояснительный текст. Формат kinemage представляет собой определенный химический MIME-тип «chemical/x-kinemage» с расширением файла «.kin».
Ранняя история
[ редактировать ]Kinemages был впервые разработан Дэвидом Ричардсоном в Медицинской школе Университета Дьюка Society для журнала Protein , премьера которого состоялась в январе 1992 года. [2] В течение первых пяти лет (1992–1996) каждый выпуск журнала Protein Science включал на дискете приложение с интерактивной трехмерной компьютерной графикой kinemage для иллюстрации многих статей, а также программное обеспечение Mage ( кроссплатформенное , бесплатное, с открытым исходным кодом). ), чтобы отобразить их; [3] Дополнительные материалы kinemage по-прежнему доступны на веб-сайте журнала. Маг и РасМол [4] были первыми широко используемыми программами макромолекулярной графики для поддержки интерактивного отображения на персональных компьютерах . Кинемаги используются для обучения, [5] [6] и для дополнений к учебникам, [7] [8] индивидуальное исследование и анализ макромолекулярных структур.

Исследовательское использование
[ редактировать ]Совсем недавно, с появлением гораздо более широкого разнообразия других инструментов молекулярной графики , использование кинематиков для презентаций уступило место широкому спектру исследовательских целей, одновременно с появлением новых функций отображения и разработкой программного обеспечения, которое создает выходные данные в формате кинеизображений из другие виды молекулярных расчетов. Полноатомный контактный анализ [9] добавляет и оптимизирует явные атомы водорода, [10] а затем использует участки точечной поверхности для отображения водородной связи , Ван-дер-Ваальса и стерических столкновений между атомами. Результаты можно использовать визуально (в киноизображениях) и количественно для анализа детальных взаимодействий между молекулярными поверхностями. [11] [12] наиболее широко с целью проверки и улучшения молекулярных моделей на основе экспериментальных данных рентгеновской кристаллографии . [13] [14] [15] [16] И Mage, и KiNG (см. ниже) были улучшены для отображения данных в кинематографе более чем в трех измерениях (перемещение между представлениями в различных трехмерных проекциях, раскрашивание и выбор возможных кластеров точек данных, а также переключение на представление параллельных координат ). например, для определения кластеров благоприятных конформаций основной цепи РНК в 7-мерном пространстве двугранных углов основной цепи между одной рибозой и следующей. [17]
Интернет-использование в Интернете
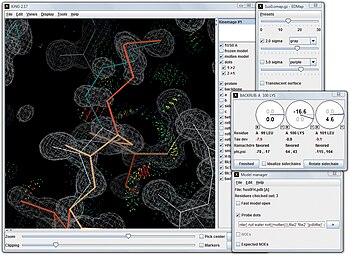
[ редактировать ]KiNG — это программа просмотра изображений с открытым исходным кодом, написанная на языке программирования Java Яном Дэвисом и Винсентом Ченом. [18] который может работать в интерактивном режиме либо автономно на пользовательском компьютере без подключения к сети, либо как веб-сервис на веб-странице . Интерактивная природа кинемажей — их основная цель и атрибут. Чтобы оценить их характер, демонстрация KiNG в браузере содержит два примера, которые можно перемещать в 3D, а также инструкции по встраиванию кинематографа на веб-страницу. [19] На рисунке ниже показано, как KiNG используется для ремоделирования боковой цепи лизина в кристаллической структуре высокого разрешения. KiNG является одним из средств просмотра, представленных на каждой структурной странице сайта банка данных белков. [20] и отображает результаты проверки в 3D на сайте MolProbity. [21] [22] [23] Kinemages также можно показывать в иммерсивных системах виртуальной реальности с помощью программного обеспечения KinImmerse с открытым исходным кодом. [24] Все программное обеспечение для отображения Kinemage и полноатомных контактов доступно бесплатно и с открытым исходным кодом на веб-сайте Kinemage .
См. также
[ редактировать ]- Молекулярная графика
- Ленточная диаграмма
- Сравнение программного обеспечения для моделирования молекулярной механики
Ссылки
[ редактировать ]- ^ Фриман, LC; иерархия; и др. (1998). «Изучение социальной структуры с помощью динамических трехмерных цветных изображений» (PDF) . Социальные сети . 20 (2): 109–118. дои : 10.1016/S0378-8733(97)00016-6 .
- ^ Ричардсон, округ Колумбия ; Дж. С. Ричардсон (январь 1992 г.). «Кинемаг: инструмент научной коммуникации» . Белковая наука . 1 (1): 3–9. дои : 10.1002/pro.5560010102 . ПМК 2142077 . ПМИД 1304880 .
- ^ Нейрат, Х. (1992). «Редакционная статья. Кинемаг: инструмент для научной иллюстрации» . Белковая наука . 5 (11): 2147. doi : 10.1002/pro.5560051101 . ПМК 2143300 .
- ^ Сэйл, Р. (1992). Материалы 10-й конференции Eurographics UK, 1992 г. Абингдон Пресс, Йорк.
- ^ Ричардсон, округ Колумбия; Дж. С. Ричардсон (1994). «Кинемаги - простая макромолекулярная графика для интерактивного обучения и публикации». Тенденции биохимических наук . 19 (3): 135–138. дои : 10.1016/0968-0004(94)90207-0 . ПМИД 8203021 .
- ^ Ричардсон, округ Колумбия; Дж. С. Ричардсон (2002). «Обучение молекулярной 3D-грамотности» . Образование в области биохимии и молекулярной биологии . 30 : 21–26. дои : 10.1002/bmb.2002.494030010005 .
- ^ Воэт, Д.; Дж. Г. Воэт; К.В. Пратт (1999). Основы биохимии . Джон Уайли и сыновья, Нью-Йорк.
- ^ Бранден, Калифорния; Дж. Туз (1999). Введение в структуру белка (2-е изд.). Garland Publishing, Inc., Нью-Йорк.
- ^ Слово, Дж. М.; и др. (1999). «Визуализация и количественная оценка молекулярного согласия: контактные точки небольшого зонда с явными атомами водорода». Журнал молекулярной биологии . 285 (4): 1711–1733. CiteSeerX 10.1.1.119.6173 . дои : 10.1006/jmbi.1998.2400 . ПМИД 9917407 .
- ^ Слово, Дж. М.; и др. (1999). «Аспарагин и глютамин: использование контактов атомов водорода при выборе ориентации амида боковой цепи». Журнал молекулярной биологии . 285 (4): 1735–1747. CiteSeerX 10.1.1.323.6971 . дои : 10.1006/jmbi.1998.2401 . ПМИД 9917408 .
- ^ Слово, Дж. М.; и др. (2000). «Изучение стерических ограничений белковых мутаций с использованием MAGE/PROBE» . Белковая наука . 9 (11): 2251–2259. дои : 10.1110/пс.9.11.2251 . ПМК 2144501 . ПМИД 11152136 .
- ^ Ричардсон, Дж.С .; Ричардсон, округ Колумбия (2002). «Природные белки β-листов используют негативный дизайн, чтобы избежать агрегации от края до края» . Учеб. Натл. акад. наук. США . 99 (5): 2754–2759. Бибкод : 2002PNAS...99.2754R . дои : 10.1073/pnas.052706099 . ПМК 122420 . ПМИД 11880627 .
- ^ Ричардсон, округ Колумбия; Ричардсон, Дж. С. (2001). «МАГ, ЗОНД и Кинемаги». Международные таблицы по кристаллографии . F, глава 25.2.8: 727–730.
- ^ Ричардсон, Джейн С.; и др. (2003). «Новые инструменты и данные для улучшения структур с использованием полноатомных контактов». Макромолекулярная кристаллография, Часть D. Методы энзимологии. Том. 374. стр. 385–412. дои : 10.1016/S0076-6879(03)74018-X . ISBN 978-0-12-182777-9 . ПМИД 14696383 .
- ^ Хигман, Вирджиния; и др. (2004). «Конформации боковой цепи аспарагина и глютамина в растворе и кристалле: сравнение лизоцима куриного яичного белка с использованием остаточных диполярных связей». Журнал биомолекулярного ЯМР . 30 (3): 327–346. дои : 10.1007/s10858-004-3218-y . ПМИД 15754058 . S2CID 26047711 .
- ^ Арендал III, Всемирный банк; и др. (2005). «Тест повышения точности модели в высокопроизводительной кристаллографии». Журнал структурной и функциональной геномики . 6 (1): 1–11. дои : 10.1007/s10969-005-3138-4 . ПМИД 15965733 . S2CID 2790812 .
- ^ Ричардсон, Дж. С.; и др. (2008). «Основная цепь РНК: консенсусные всеугловые конформеры и модульная номенклатура строк (вклад Консорциума онтологии РНК)» . РНК . 14 (3): 465–481. дои : 10.1261/rna.657708 . ПМК 2248255 . ПМИД 18192612 .
- ^ Чен, В.Б.; и др. (2009). «KING (Kinemage, Next Generation): универсальная интерактивная программа молекулярной и научной визуализации» . Белковая наука . 18 (11): 2403–2409. дои : 10.1002/pro.250 . ПМК 2788294 . ПМИД 19768809 .
- ^ KiNG в браузере
- ^ «Банк данных о белках» . Архивировано из оригинала 28 августа 2008 г. Проверено 7 декабря 2016 г.
- ^ МолПробити
- ^ Дэвис, штат Айова; и др. (2007). «MolProbity: полноатомные контакты и проверка структуры белков и нуклеиновых кислот» . Исследования нуклеиновых кислот . 35 (проблема с веб-сервером): W375–W383. дои : 10.1093/нар/gkm216 . ЧВК 1933162 . ПМИД 17452350 .
- ^ Чен, В.Б.; и др. (2010). «MolProbity: проверка полноатомной структуры для макромолекулярной кристаллографии» . Акта Кристаллографика . Д 66 (Часть 1): 12–21. дои : 10.1107/S0907444909042073 . ПМК 2803126 . ПМИД 20057044 .
- ^ Блок, Дж. Н.; и др. (2009). «KinImmerse: Макромолекулярная виртуальная реальность для ансамблей ЯМР» . Исходный код по биологии и медицине . 4 :3. дои : 10.1186/1751-0473-4-3 . ПМК 2650690 . ПМИД 19222844 .
Внешние ссылки
[ редактировать ]- Официальный сайт , оригинал Университета Дьюка, с примерами и программным обеспечением.
- пример кинемажа в браузере
- Банк данных белков RCSB
- MolProbity: проверка структуры с помощью онлайн-кинемажей KiNG