Неполная сортировка родословной
Неполная сортировка родословной , [ 1 ] [ 2 ] [ 3 ] также называемое гемиплазией, глубоким слиянием , сохранением предкового полиморфизма или межвидовым полиморфизмом, описывает явление в популяционной генетике, когда копии предковых генов не могут объединиться (оглядываясь назад во времени) в общую предковую копию до тех пор, пока они не станут глубже, чем предыдущие события видообразования. Это вызвано сортировкой генетических полиморфизмов, которые сохранялись в последовательных узлах древа вида. [ 4 ] Другими словами, дерево, созданное одним геном, отличается от дерева уровня популяции или вида, образуя несогласованное дерево. Каким бы ни был механизм, в результате созданное дерево видового уровня может различаться в зависимости от выбранных генов, используемых для оценки. [ 5 ] [ 6 ] Это контрастирует с полной сортировкой по линии , где дерево, созданное геном, такое же, как дерево уровня популяции или вида. Оба являются обычными результатами филогенетического анализа, хотя это зависит от гена, организма и метода отбора проб.
Концепция
[ редактировать ]
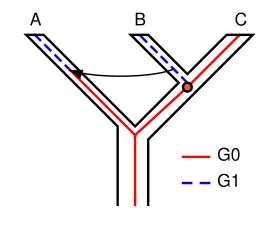
Концепция неполной сортировки линий имеет некоторые важные последствия для филогенетических методов. Сохранение полиморфизмов в различных событиях видообразования может привести к неполной сортировке линий. Предположим, происходят два последующих события видообразования, когда вид-предок дает начало сначала виду A, а затем видам B и C. При изучении одного гена он может иметь несколько версий ( аллелей ), вызывающих появление разных признаков (полиморфизмов). В примере, показанном на рисунке 1, ген G имеет две версии (аллели): G0 и G1. У предка A, B и C изначально была только одна версия гена G, G0. В какой-то момент произошла мутация, и предковая популяция стала полиморфной: у некоторых людей был G0, а у других — G1. Когда вид A отделился, он сохранил только G1, тогда как предок B и C остался полиморфным. Когда B и C разошлись, B сохранил только G1, а C только G0; ни один из них теперь не был полиморфным по G. Дерево гена G показывает A и B как сестер, тогда как дерево видов показывает B и C как сестер. Если филогения этих видов основана на гене G, она не будет отражать реальные взаимоотношения между видами. Другими словами, наиболее родственные виды не обязательно унаследуют наиболее родственные гены. Это, конечно, упрощенный пример неполной сортировки линий, и в реальных исследованиях он обычно более сложен и содержит больше генов и видов. [ 7 ] [ 8 ]
Однако к такому же кажущемуся несоответствию могут привести и другие механизмы, например, аллели могут перемещаться через границы видов посредством гибридизации, а ДНК может передаваться между видами с помощью вирусов. [ 9 ] Это проиллюстрировано на рисунке 2. Здесь предок A, B и C, а также предок B и C имели только версию G0 гена G. Мутация произошла при расхождении B и C, и B приобрел мутированную версия, G1. Спустя некоторое время стрелка показывает, что G1 был перенесен из B в A каким-то образом (например, гибридизацией или горизонтальным переносом гена). Изучение только конечных состояний G у трех видов показывает, что A и B являются сестрами, а не B и C, как на рисунке 1, но на рисунке 2 это не вызвано неполной сортировкой линий.
Подразумеваемое
[ редактировать ]Неполная сортировка линий имеет важные последствия для филогенетических исследований. Существует вероятность того, что при создании филогенетического дерева оно может не напоминать реальные отношения из-за неполной сортировки линий. Однако поток генов между линиями путем гибридизации или горизонтального переноса генов может привести к появлению одного и того же противоречивого филогенетического дерева. Различение этих различных процессов может показаться трудным, но разрабатывается множество исследований и различных статистических подходов, чтобы лучше понять эту эволюционную динамику. [ 10 ] Одним из способов уменьшить последствия неполной сортировки линий является использование нескольких генов для создания филогении видов или популяций. Чем больше генов используется, тем надежнее становится филогения. [ 8 ]
У диплоидных организмов
[ редактировать ]Неполная сортировка по происхождению обычно происходит при половом размножении , поскольку вид не может быть прослежен до одного человека или гнездящейся пары. Когда популяции племен организмов велики (т.е. тысячи), каждый ген имеет некоторое разнообразие, и дерево генов состоит из других ранее существовавших линий. Если население больше, эти наследственные линии будут сохраняться дольше. Когда вы объединяете большие наследственные популяции с близко приуроченными событиями видообразования, эти разные части ДНК сохраняют противоречивую принадлежность. Это затрудняет определение общего предка или точек ветвления. [ 5 ]
В эволюции приматов
[ редактировать ]При изучении приматов шимпанзе и бонобо более родственны друг другу, чем любые другие таксоны, и, таким образом, являются сестринскими таксонами . Тем не менее, для 1,6% генома бонобо последовательности более тесно связаны с гомологами человека, чем с шимпанзе, что, вероятно, является результатом неполной сортировки линий. [ 5 ] Исследование более 23 000 выравниваний последовательностей ДНК в семействе Hominidae (человекообразных обезьян, включая человека) показало, что около 23% не подтверждают известные сестринские отношения шимпанзе и человека. [ 9 ]
В эволюции человека
[ редактировать ]В эволюции человека неполная сортировка линий используется для диаграммы линий гомининов, которые, возможно, не удалось разобраться в то же время, когда видообразование происходило в доисторические времена. [ 11 ] Благодаря появлению генетического тестирования и секвенирования генома исследователи обнаружили, что генетические отношения между линиями гомининов могут противоречить предыдущим представлениям об их родстве, основанным на физических характеристиках. [ 11 ] Более того, дивергенция последнего общего предка (LCA) не обязательно может происходить одновременно с видообразованием. [ 12 ] Сортировка по происхождению — это метод, который позволяет палеоантропологам исследовать генетические связи и различия, которые могут не соответствовать их предыдущим моделям видообразования, основанным только на филогении . [ 11 ]
Неполная сортировка генеалогического древа человека представляет собой область большого интереса. Существует ряд неизвестных при рассмотрении как перехода от архаичного человека к современному человеку, так и отклонения других человекообразных обезьян от линии гомининов. [ 13 ]
Обезьяна и человекообразный / человеческое расхождение
[ редактировать ]Неполная сортировка линий означает, что среднее время дивергенции между генами может отличаться от времени дивергенции между видами. Модели предполагают, что среднее время расхождения между генами в геноме человека и шимпанзе старше, чем время расхождения между людьми и гориллами. Это означает, что общий предок человека и шимпанзе оставил следы генетического материала, который присутствовал у общего предка человека, шимпанзе и гориллы. [ 12 ] Однако генетическое древо несколько отличается от древа вида или филогении. [ 14 ] В филогенном дереве, когда мы смотрим на эволюционные отношения между человеком, шимпанзе бонобо и гориллой, результаты показывают, что разделение бонобо и шимпанзе произошло в непосредственной близости во времени от общего предка бонобо-шимпанзе и человека. , [ 12 ] что указывает на то, что люди и шимпанзе имели общего предка в течение нескольких миллионов лет после отделения от горилл. Это создает явление неполной сортировки родословной. Сегодня исследователи полагаются на фрагменты ДНК для изучения эволюционных взаимоотношений между людьми и их собратьями в надежде, что они предоставят информацию о видообразовании и наследственных процессах на основе геномов разных типов людей. [ 15 ]
В вирусах
[ редактировать ]
Неполная сортировка линий является общей чертой вирусной филодинамики , где филогения, представленная передачей заболевания от одного человека к другому, то есть дерево уровня популяции, часто не соответствует дереву, созданному на основе генетического анализа из-за узким местам населения , которые являются неотъемлемой особенностью вирусной передачи заболеваний. Рисунок 3 иллюстрирует, как это может произойти. Это имеет отношение к криминальной передаче ВИЧ , где в некоторых уголовных делах для вывода о передаче использовался филогенетический анализ одного или двух генов штаммов обвиняемого и жертвы; однако общность неполной сортировки по линии означает, что передачу нельзя сделать вывод исключительно на основе такого базового анализа. [ 16 ]
В лингвистике
[ редактировать ]Жак и Лист (2019) [ 17 ] показывают, что концепция неполной сортировки линий может быть применена для объяснения недревовидных явлений в эволюции языка. Калян и Франсуа (2019), сторонники метода исторической глоттометрии , модели, оспаривающей применимость древовидной модели в исторической лингвистике, соглашаются, что «Историческая глоттометрия не бросает вызов модели генеалогического древа, если принять во внимание неполную сортировку по происхождению. " [ 18 ]
См. также
[ редактировать ]- Гиббон – последний общий предок человека.
- Лаггарский сокол
- Коалесцентная теория
- Многовидовой процесс слияния
- Родословная (эволюция)
Ссылки
[ редактировать ]- ^ Симпсон, Майкл Дж. (19 июля 2010 г.). Систематика растений . Академическая пресса. ISBN 9780080922089 .
- ^ Курицын А; Кишка, Т; Шмитц, Дж; Чураков, Г (2016). «Неполная статистика сортировки линий и гибридизации для крупномасштабных данных о вставке ретропозонов» . PLOS Вычислительная биология . 12 (3): e1004812. Бибкод : 2016PLSCB..12E4812K . дои : 10.1371/journal.pcbi.1004812 . ПМЦ 4788455 . ПМИД 26967525 .
- ^ Сух, А; Смедс, Л; Эллегрен, Х (2015). «Динамика неполной сортировки линий в древней адаптивной радиации неофианских птиц» . ПЛОС Биология . 13 (8): e1002224. дои : 10.1371/journal.pbio.1002224 . ПМК 4540587 . ПМИД 26284513 .
- ^ Мэддисон, Уэйн П. (1 сентября 1997 г.). Винс, Джон Дж. (ред.). «Генные деревья в деревьях видов» . Систематическая биология . 46 (3). Издательство Оксфордского университета (OUP): 523–536. дои : 10.1093/sysbio/46.3.523 . ISSN 1076-836X .
- ^ Jump up to: а б с Роджерс, Джеффри; Гиббс, Ричард А. (01 мая 2014 г.). «Сравнительная геномика приматов: новые закономерности содержания и динамики генома» . Обзоры природы Генетика . 15 (5): 347–359. дои : 10.1038/nrg3707 . ПМЦ 4113315 . ПМИД 24709753 .
- ^ Шен, Син-Син; Хиттингер, Крис Тодд; Рокас, Антонис (2017). «Спорные отношения в филогеномных исследованиях могут быть вызваны несколькими генами» . Экология и эволюция природы . 1 (5): 126. дои : 10.1038/s41559-017-0126 . ISSN 2397-334X . ПМК 5560076 . ПМИД 28812701 .
- ^ Копетти, Дарио; Буркес, Альберто; Бустаманте, Энрикена; Шарбоно, Джозеф Л.М.; Чайлдс, Кевин Л.; Эгиарте, Луис Э.; Ли, Сынхи; Лю, Тиффани Л.; МакМахон, Мишель М.; Уайтмен, Ной К.; Винг, Род А.; Войцеховский, Мартин Ф. и Сандерсон, Майкл Дж. (07.11.2017). «Обширное несоответствие генного дерева и гемиплазия сформировали геномы колонновидных кактусов Северной Америки». Труды Национальной академии наук . 114 (45): 12003–12008. Бибкод : 2017PNAS..11412003C . дои : 10.1073/pnas.1706367114 . ПМЦ 5692538 . ПМИД 29078296 .
- ^ Jump up to: а б Футуйма, Дуглас Дж. (15 июля 2013 г.). Эволюция (3-е изд.). Сандерленд, Массачусетс, США ISBN 9781605351155 . OCLC 824532153 .
{{cite book}}: CS1 maint: отсутствует местоположение издателя ( ссылка ) - ^ Jump up to: а б Авис, Джон К. и Робинсон, Теренс Дж. (2008). «Гемиплазия: новый термин в лексиконе филогенетики». Систематическая биология . 57 (3): 503–507. дои : 10.1080/10635150802164587 . ПМИД 18570042 .
- ^ Варноу, Тэнди; Байзид, доктор медицины Шамсуццоха; Мирараб, Сиаваш (01 мая 2016 г.). «Оценка суммарных методов для оценки многолокусного дерева видов при наличии неполной сортировки по происхождению» . Систематическая биология . 65 (3): 366–380. дои : 10.1093/sysbio/syu063 . ISSN 1063-5157 . ПМИД 25164915 .
- ^ Jump up to: а б с Мэддисон, Уэйн П. (1 сентября 1997 г.). «Генные деревья в деревьях видов» . Систематическая биология . 46 (3): 523–536. дои : 10.1093/sysbio/46.3.523 . ISSN 1076-836X .
- ^ Jump up to: а б с Майлунд, Томас; Мунк, Каспер; Шируп, Миккель Хайде (23 ноября 2014 г.). «Сортировка линий у обезьян». Ежегодный обзор генетики . 48 (1): 519–535. doi : 10.1146/annurev-genet-120213-092532 . ISSN 0066-4197 . ПМИД 25251849 .
- ^ Николс, Ричард (июль 2001 г.). «Генные деревья и деревья видов — это не одно и то же». Тенденции в экологии и эволюции . 16 (7): 358–364. дои : 10.1016/s0169-5347(01)02203-0 . ISSN 0169-5347 . ПМИД 11403868 .
- ^ «Видообразование приматов: пример африканских обезьян | Изучайте науку в Scitable» . www.nature.com . Проверено 30 мая 2020 г.
- ^ Пейрень, Стефан; Бойл, Майкл Джеймс; Даннеманн, Майкл; Прюфер, Кей (сентябрь 2017 г.). «Обнаружение древнего положительного отбора у людей с использованием расширенной сортировки линий» . Геномные исследования . 27 (9): 1563–1572. дои : 10.1101/гр.219493.116 . ISSN 1088-9051 . ПМК 5580715 . ПМИД 28720580 .
- ^ Лейтнер, Томас (май 2019 г.). «Филогенетика передачи ВИЧ: учет разнообразия внутри хозяина» . Современное мнение о ВИЧ и СПИДе . 14 (3): 181–187. дои : 10.1097/COH.0000000000000536 . ISSN 1746-630X . ПМК 6449181 . ПМИД 30920395 .
- ^ Жак, Гийом ; Список, Иоганн-Маттис (2019). «Почему нам нужны древовидные модели в лингвистической реконструкции (и когда их следует применять)». Журнал исторической лингвистики . 9 (1): 128–167. дои : 10.1075/jhl.17008.mat . hdl : 21.11116/0000-0004-4D2E-4 . ISSN 2210-2116 . S2CID 52220491 .
- ^ Кальян, Шива; Франсуа, Александр (2019). «Когда волны встречаются с деревьями». Журнал исторической лингвистики . 9 (1): 168–177. дои : 10.1075/jhl.18019.kal . ISSN 2210-2116 . S2CID 198707375 .
Внешние ссылки
[ редактировать ]- Венема, Д. (01 августа 2013 г.). «Основы эволюции: неполная сортировка по происхождению и размеры популяции предков» . Биологос . Проверено 29 июня 2018 г.
- Мэддисон, Уэйн П.; Ноулз, Л. Лейси; Коллинз, Тим (2006). «Выводы о филогении, несмотря на неполную сортировку линий» . Систематическая биология . 55 (1): 21–30. дои : 10.1080/10635150500354928 . ISSN 1076-836X . ПМИД 16507521 .
- Жоли, Саймон; Макленачан, Патрисия А.; Локхарт, Питер Дж. (2009). «Статистический подход к различению гибридизации и неполной сортировки по происхождению». Американский натуралист . 174 (2): Е54–Е70. дои : 10.1086/600082 . ПМИД 19519219 . S2CID 205991354 .
- Карстенс, Брайан С.; Ноулз, Л. Лейси; Коллинз, Тим (2007). «Оценка филогении видов на основе вероятностей генного дерева, несмотря на неполную сортировку линий: пример кузнечиков Melanoplus». Систематическая биология . 56 (3): 400–411. дои : 10.1080/10635150701405560 . ISSN 1076-836X . ПМИД 17520504 . S2CID 11591644 .
- Скорнавакка, К.; Галтье, Н. (2017). «Неполная сортировка линий в филогеномике млекопитающих» . Систематическая биология . 66 (1): 112–120. doi : 10.1093/sysbio/syw082 . ПМИД 28173480 .