Миграция филиалов
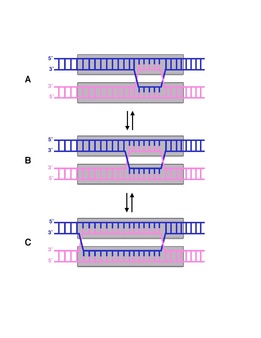
Миграция ветвей — это процесс, при котором пары оснований в гомологичных цепях ДНК последовательно заменяются в месте соединения Холлидея , перемещая точку ветвления вверх или вниз по последовательности ДНК. [1] Миграция ветвей — это второй этап генетической рекомбинации , следующий за обменом двумя одиночными нитями ДНК между двумя гомологичными хромосомами. [2] Процесс носит случайный характер, и точка ветвления может смещаться на цепи в любом направлении, влияя на степень обмена генетического материала. [1] Миграцию ветвей также можно увидеть при ДНК репарации и репликации , при заполнении пробелов в последовательности. Это также можно увидеть, когда чужеродный фрагмент ДНК проникает в цепь. [2]
Механизм
[ редактировать ]Механизм миграции ветвей у прокариот и эукариот различен . [2]
Прокариоты
[ редактировать ]
Механизм миграции прокариотических ветвей неоднократно изучался на Escherichia coli . [2] В E. coli белки RuvA и RuvB объединяются и образуют комплекс, который облегчает этот процесс несколькими способами. RuvA представляет собой тетрамер и связывается с ДНК в месте соединения Холлидея, когда она находится в открытой X-форме. Белок связывается таким образом, что ДНК, входящая/выходящая из соединения, по-прежнему может свободно вращаться и скользить через него. RuvA имеет домен с кислыми аминокислотными остатками, которые мешают парам оснований в центре соединения. Это заставляет пары оснований расходиться, так что они могут повторно отжигаться с парами оснований на гомологичных цепях. [3]
Чтобы произошла миграция, RuvA должен быть связан с RuvB и ATP . RuvB обладает способностью гидролизовать АТФ, приводя в движение точку ветвления. RuvB представляет собой гексамер с хеликазной активностью, также связывающий ДНК. Поскольку АТФ гидролизуется, RuvB вращает рекомбинированные цепи, вытягивая их из соединения, но не разделяет цепи, как это сделала бы геликаза. [3]
Последний этап миграции ветвей называется разрешением и требует белка RuvC . Белок представляет собой димер и связывается с соединением Холлидея, когда принимает сложную X-форму. Белок обладает эндонуклеазной активностью и расщепляет нити ровно в одно и то же время. Расщепление является симметричным и дает две рекомбинированные молекулы ДНК с одноцепочечными разрывами. [4]
Эукариоты
[ редактировать ]Эукариотический механизм гораздо более сложен и включает в себя различные и дополнительные белки, но следует тому же общему пути. [2] Сообщается, что Rad54 , высококонсервативный эукариотический белок, олигомеризуется на соединениях Холлидея, способствуя миграции ветвей. [5]
Архея
[ редактировать ]Хеликаза (обозначенная Saci-0814), выделенная из термофильной кренархеи Sulfolobus acidocaldarius , диссоциировала структуры соединения Холлидея ДНК и продемонстрировала активность миграции ветвей in vitro . [6] В штамме S. acidocaldarius, удаленном по Saci-0814, частота гомологичной рекомбинации была снижена в пять раз по сравнению с родительским штаммом, что указывает на то, что Saci-0814 участвует в гомологичной рекомбинации in vivo . На основании этих данных оказывается, что Saci-0814 используется в гомологичной рекомбинации у S. acidocaldarius и действует как хеликаза миграции ветвей. [6] Гомологичная рекомбинация, по-видимому, является важной адаптацией у гипертермофилов, таких как S. acidocaldarius , для эффективного восстановления повреждений ДНК. Хеликаза Saci-0814 классифицируется как aLhr1 (архейная длинная геликаза, родственная 1) в суперсемействе 2 геликаз, и ее гомологи консервативны среди архей.
Контроль
[ редактировать ]
Скорость миграции ветвей зависит от количества двухвалентных ионов, в частности ионов магния (Mg 2+ ), присутствующий во время рекомбинации. [1] Ионы определяют, какую структуру примет переход Холлидея, поскольку они играют стабилизирующую роль. Когда ионы отсутствуют, основные цепи отталкиваются друг от друга, и соединение приобретает открытую X-структуру. [7] В этом состоянии миграция оптимальна и место соединения будет свободно перемещаться вверх и вниз по прядям. [3] Когда ионы присутствуют, они нейтрализуют отрицательно заряженную основу. Это позволяет нитям сближаться друг с другом, и соединение принимает сложную X-структуру. [7] Именно в этом состоянии разрешение будет оптимальным, позволяя RuvC привязаться к соединению. [3]
Ссылки
[ редактировать ]- ^ Jump up to: а б с Лилли, Дэвид М.Дж. (1 мая 2000 г.). «Структуры спиральных соединений нуклеиновых кислот». Ежеквартальные обзоры биофизики . 33 (2): 109–159. дои : 10.1017/s0033583500003590 . ISSN 1469-8994 . ПМИД 11131562 . S2CID 40501795 .
- ^ Jump up to: а б с д и «Генетическая рекомбинация | Изучайте науку в Scitable» . www.nature.com . Проверено 13 ноября 2015 г.
- ^ Jump up to: а б с д Ямада, Казухиро; Ариеси, Марико; Морикава, Косукэ (1 апреля 2004 г.). «Трехмерные структурные представления о миграции и разрешении ветвей при гомологичной рекомбинации ДНК». Современное мнение в области структурной биологии . 14 (2): 130–137. дои : 10.1016/j.sbi.2004.03.005 . ПМИД 15093826 .
- ^ Гурекка, КМ; Коморовска, В.; Новотны, М. (2013). «Кристаллическая структура резольвазы RuvC в комплексе с субстратом соединения Холлидея» . Исследования нуклеиновых кислот . 41 (21): 9945–9955. дои : 10.1093/nar/gkt769 . ПМЦ 3834835 . ПМИД 23980027 .
- ^ Гоял Н., Росси М.Дж., Мазина О.М., Чи Ю, Мориц Р.Л., Клерман Б.Е., Мазин А.В. (2018). «N-концевой домен RAD54 представляет собой сенсор ДНК, который связывает гидролиз АТФ с миграцией ветвей соединений Холлидея» . Нат Коммун . 9 (1): 34. Бибкод : 2018NatCo...9...34G . дои : 10.1038/s41467-017-02497-x . ПМК 5750232 . ПМИД 29295984 .
- ^ Jump up to: а б Сузуки, Сёдзи; Куросава, Норио; Ямагами, Такеши; Мацумото, Сюнсукэ; Нумата, Томоюки; Исино, Соноко; Исино, Ёсидзуми (2021). «Генетические и биохимические характеристики геликазы aLhr1 у термофильных Crenarchaeon Sulfolobus acidocaldarius» . Катализаторы . 12:34 . дои : 10.3390/catal12010034 .
- ^ Jump up to: а б Клегг, Р.М. (1 января 1993 г.). «Структура четырехстороннего соединения в ДНК». Ежегодный обзор биофизики и биомолекулярной структуры . 22 (1): 299–328. дои : 10.1146/annurev.bb.22.060193.001503 . ПМИД 8347993 .