Цифровая патология
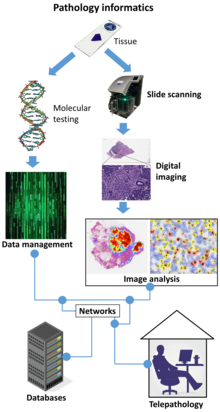
Цифровая патология является подполе патологии , которая фокусируется на управлении и анализе информации, полученной из оцифрованных слайдов образца. Он использует компьютерную технологию и виртуальную микроскопию для просмотра, управления, обмена и анализа цифровых слайдов на компьютерных мониторах. [ 1 ] Эта область имеет применение в диагностической медицине и направлена на достижение более эффективных и экономичных диагнозов , прогнозов и прогнозов заболеваний посредством достижения в области машинного обучения и искусственного интеллекта в здравоохранении . [ 2 ] [ 3 ]
История
[ редактировать ]Корни цифровой патологии отслеживают в 1960 -х годах с ранними телепатологическими экспериментами. Концепция виртуальной микроскопии появилась в 1990 -х годах в различных областях исследований в области наук о жизни [ 4 ] Полем На рубеже веков научное сообщество все больше и больше соглашалось с термином «цифровой патологии», чтобы обозначить усилия по оцифровке в патологии. Однако в 2000 году технические требования (сканер, хранение, сеть) по -прежнему были ограничивающим фактором для широкого распространения концепций цифровой патологии. Это изменилось как новая мощная и доступная технология сканера, а также технологии массового / облачного хранения появились на рынке. Область радиологии подверглась цифровой трансформации почти 15 лет назад, не потому, что радиология более продвинута, но существуют фундаментальные различия между цифровыми изображениями в радиологии и цифровой патологии: источник изображения в радиологии является (живым) пациентом и сегодня в В большинстве случаев изображение даже в основном фиксируется в цифровом формате. В патологии сканирование выполняется из сохранившихся и обработанных образцов, для ретроспективных исследований даже из слайдов, хранящихся в биобанке . Помимо этой разницы в преаналитике и содержании метаданных , необходимое хранилище в цифровой патологии на два-три порядка выше, чем в радиологии. Тем не менее, преимущества, ожидаемые с помощью цифровой патологии, аналогичны преимуществам радиологии:
- Возможность быстро передавать цифровые слайды на расстояниях, что позволяет телепатологическим сценариям.
- Возможность получить доступ к прошлому образцу от тех же пациентов и аналогичных случаев для сравнения и обзора, с гораздо меньшими усилиями, чем извлечение слайдов с архивных полков.
- Возможность сравнивать различные области нескольких слайдов одновременно (слайд в режиме слайда) с помощью виртуального микроскопа.
- Возможность аннотировать области непосредственно в слайде и делиться этим для обучения и исследований.
Цифровая патология сегодня широко используется в образовательных целях [ 5 ] в телепатологии и телеконсультации , а также в исследовательских проектах. Цифровая патология позволяет делиться и аннотировать слайды гораздо проще, а для загрузки аннотированных наборов лекций генерирует новые возможности для электронного обучения и обмена знаниями в патологии. Цифровая патология в диагностике является новой и будущей областью.
Среда
[ редактировать ]Сканирование
[ редактировать ]

Цифровые слайды создаются из стеклянных слайдов с использованием специализированных сканирующих машин. Все высококачественные сканирования должны быть свободны от пыли, царапин и других препятствий. Существует два общих метода для сканирования цифрового слайда, сканирования на основе плитки и сканирования на основе линий. [ 6 ] Обе технологии используют интегрированную камеру и моторизованную стадию, чтобы перемещать слайд вокруг, пока визуализируются части ткани. Сканеры плиток захватывают квадратные изображения поля зрения, покрывающие всю область ткани на слайде, в то время как линейные сканеры захватывают изображения ткани в длинных, непрерывных полосах, а не плитки. В обоих случаях программное обеспечение, связанное с сканером, сшивает плитки или линии вместе в одно бесшовное изображение.
Z-stacking is the scanning of a slide at multiple focal planes along the vertical z-axis.[7]
View
[edit]Digital slides are accessible for viewing via a computer monitor and viewing software either locally or remotely via the Internet. An example of an open-source, web-based viewer for this purpose implemented in pure JavaScript, for desktop and mobile, is the OpenSeadragon[8] viewer. QuPath[9] is another such open source software, which is often used for digital pathology applications because it offers a powerful set of tools for working with whole slide images. OpenSlide,[10] on the other hand is a C library (Python and Java bindings are also available) that provides a simple interface to read and view whole-slide images.
Manage
[edit]Digital slides are maintained in an information management system that allows for archival and intelligent retrieval.
Network
[edit]Digital slides are often stored and delivered over the Internet or private networks, for viewing and consultation.
Analyze
[edit]Image analysis tools are used to derive objective quantification measures from digital slides. Image segmentation and classification algorithms, often implemented using deep learning neural networks, are used to identify medically significant regions and objects on digital slides. A GPU acceleration software for pathology imaging analysis, cross-comparing spatial boundaries of a huge amount of segmented micro-anatomic objects has been developed.[11] The core algorithm of PixelBox in this software has been adopted in Fixstars' Geometric Performance Primitives (GPP) library[12] as a part of NVIDIA Developer, which is a production geometry engine for advanced graphical information systems, electronic design automation, computer vision and motion planning solutions.[13]
-
Tissue segmentation for digital calculation of bone marrow cellularity in QuPath: The system is trained on the appearance of immune cells versus other tissue, and uses this to give an overall percentage of each type.
-
Breast cancer prediction by AI.[15]
Integrate
[edit]Digital pathology workflow is integrated into the institution's overall operational environment. Slide digitization is expected to reduce the number of routine, manually reviewed slides, maximizing workload efficiency.
Sharing
[edit]Digital pathology also allows internet information sharing for education, diagnostics, publication and research. This may take the form of publicly available datasets or open source access to machine learning algorithms.
Challenges
[edit]

Digital pathology has been approved by the FDA for primary diagnosis.[16] The approval was based on a multi-center study of 1,992 cases in which whole-slide imaging (WSI) was shown to be non-inferior to microscopy across a wide range of surgical pathology specimens, sample types and stains.[17] While there are advantages to WSI when creating digital data from glass slides, when it comes to real-time telepathology applications, WSI is not a strong choice for discussion and collaboration between multiple remote pathologists.[18] Furthermore, unlike digital radiology where the elimination of film made return on investment (ROI) clear, the ROI on digital pathology equipment is less obvious. The strongest ROI justification includes improved quality of healthcare, increased efficiency for pathologists, and reduced costs in handling glass slides.[19]
Validation
[edit]Validation of a digital microscopy workflow in a specific environment (see above) is important to ensure high diagnostic performance of pathologists when evaluating digital whole-slide images. There are different methods that can be used for this validation process.[20] The College of American Pathologists has published a guideline with minimal requirements for validation of whole slide imaging systems for diagnostic purposes in human pathology.[21]
Potential
[edit]Trained pathologists traditionally view tissue slides under a microscope. These tissue slides may be stained to highlight cellular structures. When slides are digitized, they are able to be shared through tele-pathology and are numerically analyzed using computer algorithms. Algorithms can be used to automate the manual counting of structures, or for classifying the condition of tissue such as is used in grading tumors. They can additionally be used for feature detection of mitotic figures, epithelial cells, or tissue specific structures such as lung cancer nodules, glomeruli, or vessels, or estimation of molecular biomarkers such as mutated genes, tumor mutational burden, or transcriptional changes.[22][23][24] This has the potential to reduce human error and improve accuracy of diagnoses. Digital slides can be easily shared, increasing the potential for data usage in education as well as in consultations between expert pathologists. Multiplexed imaging (staining multiple markers on the same slide) allows pathologists to understand finer distribution of cell-types and their relative locations.[25] An understanding of the spatial distribution of cell-types or markers and pathways they express, can allow for prescription of targeted drugs or build combinational therapies in a personalized manner.
See also
[edit]References
[edit]- ^ Pantanowitz L (2018). "Twenty Years of Digital Pathology: An Overview of the Road Travelled, What is on the Horizon, and the Emergence of Vendor-Neutral Archives". Journal of Pathology Informatics. 9: 40. doi:10.4103/jpi.jpi_69_18. PMC 6289005. PMID 30607307.
- ^ "Whole Slide Imaging | MBF Bioscience". www.mbfbioscience.com. Retrieved 2019-12-02.
- ^ Saillard, Charlie; Dubois, Rémy; Tchita, Oussama; Loiseau, Nicolas; Garcia, Thierry; Adriansen, Aurélie; Carpentier, Séverine; Reyre, Joelle; Enea, Diana; von Loga, Katharina; Kamoun, Aurélie; Rossat, Stéphane; Wiscart, Corentin; Sefta, Meriem; Auffret, Michaël (2023-11-06). "Validation of MSIntuit as an AI-based pre-screening tool for MSI detection from colorectal cancer histology slides". Nature Communications. 14 (1): 6695. doi:10.1038/s41467-023-42453-6. ISSN 2041-1723. PMC 10628260. PMID 37932267.
- ^ Ferreira, R; Moon, J; Humphries, J; Sussman, A; Saltz, J; Miller, R; Demarzo, A (1997). "The virtual microscope". Romanian Journal of Morphology and Embryology. 45: 449–453. PMC 2233368. PMID 9357666.
- ^ Hamilton, Peter W.; Wang, Yinhai; McCullough, Stephen J.; Sussman (2012). "Virtual microscopy and digital pathology in training and education". APMIS. 120 (4): 305–315. doi:10.1111/j.1600-0463.2011.02869.x. PMID 22429213. S2CID 20599493.
- ^ "Informatics, digital & computational pathology".
- ^ Evans AJ, Salama ME, Henricks WH, Pantanowitz L (2017). "Implementation of Whole Slide Imaging for Clinical Purposes: Issues to Consider From the Perspective of Early Adopters". Arch Pathol Lab Med. 141 (7): 944–959. doi:10.5858/arpa.2016-0074-OA. PMID 28440660.
- ^ OpenSeadragon on GitHub
- ^ QuPath on GitHub
- ^ OpenSlide website
- ^ Kaibo Wang; Yin Huai; Rubao Lee; Fusheng Wang; Xiaodong Zhang; Joel H. Saltz (2012). "Accelerating Pathology Image Data Cross-Comparison on CPU-GPU Hybrid Systems". Proceedings VLDB Endowment. Proceedings of the VLDB Endowment. International Conference on Very Large Data Bases. Vol. 5, no. 11. pp. 1543–1554. PMC 3553551. PMID 23355955.
- ^ "Geometric Performance Primitives (GPP)". NVIDIA Developer.
- ^ "Letter to Kaibo Wang" (PDF).
- ^ Lourenço BC, Guimarães-Teixeira C, Flores BCT, Miranda-Gonçalves V, Guimarães R, Cantante M, et al. (2022). "Ki67 and LSD1 Expression in Testicular Germ Cell Tumors Is Not Associated with Patient Outcome: Investigation Using a Digital Pathology Algorithm". Life. 12 (2): 264. doi:10.3390/life12020264. PMC 8875543. PMID 35207551.
- Figure 2 - available via license: Creative Commons Attribution 4.0 International - ^ Cruz-Roa A, Gilmore H, Basavanhally A, Feldman M, Ganesan S, Shih NNC, et al. (2017). "Accurate and reproducible invasive breast cancer detection in whole-slide images: A Deep Learning approach for quantifying tumor extent". Sci Rep. 7: 46450. doi:10.1038/srep46450. PMC 5394452. PMID 28418027.
"This work is licensed under a Creative Commons Attribution 4.0 International License." - ^ "FDA allows marketing of first whole slide imaging system for digital pathology" (Press release). FDA. April 12, 2017. Retrieved May 24, 2017.
- ^ Mukhopadhyay, Sanjay; Feldman, Michael; Abels, Esther (2017). "Whole slide imaging versus microscopy for primary diagnosis in surgical pathology: a multicenter randomized blinded noninferiority study of 1992 cases (pivotal study)". American Journal of Surgical Pathology. 42 (1): 39–52. doi:10.1097/PAS.0000000000000948. PMC 5737464. PMID 28961557.
- ^ Siegel, Gabriel; Regelman, Dan; Maronpot, Robert; Rosenstock, Moti; Hayashi, Shim-mo; Nyska, Abraham (Oct 2018). "Utilizing novel telepathology system in preclinical studies and peer review". Journal of Toxicologic Pathology. 31 (4): 315–319. doi:10.1293/tox.2018-0032. PMC 6206289. PMID 30393436.
- ^ "How to Build a Business Case to Justify the Investment in Digital Pathology". Sectra Medical Systems. Retrieved April 26, 2015.
- ^ Бертрам, Кристоф А; Статоникос, Николас; Донован, Тарин А; Бартель, Александр; Fuchs-Baumgartinger, Andrea; Липник, Каролин; Может удивляться, Пол Дж; Bonsembiante, Federico; Клопфлейш, Роберт (сентябрь 2021 г.). «Проверка цифровой микроскопии: обзор методов проверки и источников смещения» . Ветеринарная патология . 59 (1): 26–38. doi : 10.1177/03009858211040476 . PMC 8761960 . PMID 34433345 .
- ^ Эванс, Эндрю Дж; Браун, Ричард; Буй, Мэрилин (2021). «Проверка систем визуализации целых слайдов для диагностических целей в патологии: обновление руководства от Колледжа американских патологов в сотрудничестве с Американским обществом по клинической патологии и Ассоциации патологической информатики» . Arch Pathol Lab Med . 146 (4): 440–450. doi : 10.5858/arpa.2020-0723-cp . PMID 34003251 .
- ^ Аэффнер, Фамке; Зарелла, Марк Д.; Бухбиндер, Натан; Буй, Мэрилин М.; Гудман, Мэтью Р.; Хартман, Дуглас Дж.; Луджан, Джованни М.; Молани, Мариам А.; Parwani, Anil v.; Лиллард, Кейт; Тернер, Оливер С. (2019-03-08). «Введение в цифровой анализ изображений в визуализации всего слайда: белый документ из ассоциации цифровой патологии» . Журнал патологии информатики . 10 : 9. doi : 10.4103/jpi.jpi_82_18 . ISSN 2229-5089 . PMC 6437786 . PMID 30984469 .
- ^ Jain, Mika S.; Массуд, Тарик Ф. (2020). «Прогнозирование мутационной нагрузки опухоли от гистопатологических изображений с использованием многомасштабного глубокого обучения» . Интеллект природы . 2 (6): 356–362. doi : 10.1038/s42256-020-0190-5 . ISSN 2522-5839 . S2CID 220510782 .
- ^ Сераг, Ахмед; Ионо-маргинану, Адриан; Куреши, Хаммад; Макмиллан, Райан; Святой Мартин, Мари-Джудит; Даймонд, Джим; О'Рейли, Пол; Гамильтон, Питер (2019). «Трансляционное ИИ и глубокое обучение в диагностической патологии» . Границы в медицине . 6 : 185. doi : 10.3389/fmed.2019.00185 . ISSN 2296-858X . PMC 6779702 . PMID 31632973 .
- ^ Нирмал, Аджит Дж.; Малига, Золтан; Валлиус, Туулия; Quattrochi, Брайан; Чен, Алис А.; Джейкобсон, Коннор А.; Pelletier, Roxanne J.; Япп, Кларенс; Arias-Camison, Raquel; Чен, Ю-Ан; Лиан, Кристина Г. (2022-04-11). «Пространственный ландшафт прогрессирования и иммуноэдината при первичной меланоме при разрешении одноклеточных» . Открытие рака . 12 (6): 1518–1541. doi : 10.1158/2159-8290.CD-21-1357 . ISSN 2159-8290 . PMC 9167783 . PMID 35404441 .
Дальнейшее чтение
[ редактировать ]- Кайзер, K; Кайзер, G; Radziszowski, D; Oehmann, A (1999). «От телепатологии до института виртуальной патологии: новый мир цифровой патологии» (PDF) . Румынский журнал морфологии и эмбриологии . 45 : 3–9. PMID 15847374 .
- МакКаллоу, Брюс; Инг, Xiaoyou; Монтичелло, Томас; Bonnefoi, Marc (2004). «Цифровая микроскопия визуализация и новые подходы в токсикологической патологии» . Токсикологическая патология . 32 (5): 49–58. doi : 10.1080/01926230490451734 . PMID 15503664 .
- Шланген, Дэвид; Стед, Манфред; Bontas, Elena Paslaru (2004). «Кормление совы: извлечение и представление содержания отчетов о патологии» . NLPXML '04 Труды семинара по NLP и XML . NLPXML '04: 43–50.
- Круз-Роа, Ангел; Диас, Глория; Ромеро, Эдуардо; Гонсалес, Фабио (2011). «Автоматическая аннотация гистопатологических изображений с использованием скрытой тематической модели на основе неотрицательной факторизации матрицы» . Журнал патологии информатики . 2 (4): 4. doi : 10.4103/2153-3539.92031 . PMC 3312710 . PMID 22811960 .
- «Электронное здоровье и телемедицина». Международный журнал компьютерной радиологии и хирургии . 1 (Дополнение 1): 119–35. 2006. DOI : 10.1007/S11548-006-0012-1 . S2CID 5642084 .
- Прекрасно, Джеффри Л.; Грибики, Дана М.; Silowash, Russell; Хо, Джонхан; Гилбертсон, Джон Р.; Энтони, Лесли; Уилсон, Робб; Parwani, Anil v.; и др. (2008). «Оценка интерпретации иммуногистохимии с изображением цельного слайда в оспаривании биопсии иглы предстательной железы». Человеческая патология . 39 (4): 564–72. doi : 10.1016/j.humpath.2007.08.007 . PMID 18234276 .
- Кайзер, Клаус; Кайзер, Джан; Radziszowski, Dominik; Oehmann, Alexander (2004). «Новые разработки в области цифровой патологии: от телепатологии до лаборатории виртуальной патологии» . В Дуплаге, Мариус; Zieliński, Krzysztof; Ингрэм, Дэвид (ред.). Преобразование здравоохранения с помощью информационных технологий . Исследования по технологиям здравоохранения и информатике. Тол. 105. IOS Press. С. 61–9. ISBN 978-1-58603-438-2 Полем ISSN 0926-9630 . PMID 15718595 .
- Толксдорф, Роберт; Bontas, Elena Paslaru (2004). «Организация знаний в семантической паутине для патологии». Объектно-ориентированные и интернет-технологии . Заметки лекции в информатике. Тол. 3263. С. 115–56. doi : 10.1007/978-3-540-30196-7_4 . ISBN 978-3-540-23201-8 Полем S2CID 18006838 .
- Поттс, Стивен Дж. (2009). «Цифровая патология в обнаружении и развитии лекарств: многосайтовая интеграция». Drug Discovery сегодня . 14 (19–20): 935–41. doi : 10.1016/j.drudis.2009.06.013 . PMID 19596461 .
- Поттс, Стивен Дж.; Янг Г. Дэвид; Воэлькер, Фрэнк А. (2010). «Роль и влияние количественной патологии открытия». Drug Discovery сегодня . 15 (21–22): 943–50. doi : 10.1016/j.drudis.2010.09.001 . PMID 20946967 .
- Zwonitzer, R; Калински, т; Хофманн, ч; Росснер, а; Бернардинг, Дж. (2007). «Цифровая патология: Dicom-Conform Draft, Testdle и первые результаты». Компьютерные методы и программы в биомедицине . 87 (3): 181–8. doi : 10.1016/j.cmpb.2007.05.010 . PMID 17618703 .
![Расчет окрашивания KI67 с помощью Qupath в чистой семиноме, которая дает измерение скорости пролиферации опухоли. Цвета представляют собой интенсивность выражения: выражение синего не, желто-низкое, оранжево-умеренное и красное выражение. [14]](http://upload.wikimedia.org/wikipedia/commons/thumb/e/eb/Ki67_calculation_by_QuPath_in_a_pure_seminoma.png/295px-Ki67_calculation_by_QuPath_in_a_pure_seminoma.png)

![Прогноз рака молочной железы по ai. [15]](http://upload.wikimedia.org/wikipedia/commons/thumb/4/4f/Histopathology_of_tumor_identification_by_AI.png/278px-Histopathology_of_tumor_identification_by_AI.png)