ГЕНКОД
| Содержание | |
|---|---|
| Описание | Энциклопедия генов и вариантов генов |
| Типы данных захвачен | Все особенности генов в геноме человека и мыши |
| Контакт | |
| Исследовательский центр | Wellcome Trust Sanger Institute |
| Авторы | Харроу Дж. и др. [1] |
| Первичное цитирование | ПМИД 22955987 |
| Дата выпуска | Сентябрь 2012 г. |
| Доступ | |
| Веб-сайт | Генкод веб-сайта |
| Инструменты | |
| Интернет | Браузер генома UCSC: http://genome.cse.ucsc.edu/encode/ |
| Разнообразный | |
| Лицензия | Открытый доступ |
| Выпуск данных частота | Человек - Ежеквартально Мышь - раз в полгода |
| Версия | Человек – выпуск 37 (февраль 2021 г.) Мышь — выпуск M26 (февраль 2021 г.) |
GENCODE — это научный проект по исследованию генома , являющийся частью ENCODE масштабного проекта (ENCyclopedia Of DNA Elements).
Консорциум GENCODE изначально был сформирован в рамках пилотного этапа проекта ENCODE для идентификации и картирования всех генов, кодирующих белки, в регионах ENCODE (около 1% генома человека). [2] Учитывая первоначальный успех проекта, GENCODE теперь стремится создать «Энциклопедию генов и вариантов генов». [2]
Результатом будет набор аннотаций, включающий все локусы , кодирующие белок, с альтернативно транскрибируемыми вариантами . [3] некодирующие локусы [4] с транскриптами и псевдогенами . [5]
Текущий прогресс
[ редактировать ]В настоящее время GENCODE продвигается к достижению своих целей на втором этапе проекта. [6]
Последним выпуском аннотаций генетического набора человека является Gencode 36 с датой заморозки в декабре 2020 года. В этом выпуске используется новейшая сборка эталонного генома человека GRCh38 . [7]
Последней версией аннотаций генетического набора мышей является Gencode M25, также с датой заморозки в декабре 2020 года. [7]
С сентября 2009 года GENCODE представляет собой набор генов человека, используемый в проекте Ensembl , и каждая новая версия GENCODE соответствует выпуску Ensembl. [8]
История
[ редактировать ]
2003 сентябрь
Проект был разработан в три этапа: пилотный проект, этап разработки технологии и этап производства. [9] Пилотный этап проекта ENCODE был направлен на глубокое исследование, вычислительным и экспериментальным путем, 44 областей общей последовательностью 30 МБ, представляющих примерно 1% генома человека. В рамках этого этапа был сформирован консорциум GENCODE для идентификации и картирования всех генов, кодирующих белки, в регионах ENCODE. [2] Предполагалось, что результаты первых двух этапов будут использованы для определения наилучшего пути анализа оставшихся 99% генома человека на экономически эффективном и комплексном этапе производства. [9]
2005 апрель
Первый выпуск аннотаций 44 регионов ENCODE был заморожен 29 апреля 2005 г. и использовался на первом семинаре проекта оценки аннотаций генома ENCODE (E-GASP). [2] Версия 1 GENCODE содержала 416 известных локусов, 26 новых (кодирующих последовательность ДНК) локусов CDS, 82 новых локуса транскриптов, 78 предполагаемых локусов, 104 процессированных псевдогенов и 66 непроцессированных псевдогенов.
2005 октябрь
Вторая версия (выпуск 02) была заморожена 14 октября 2005 г. и содержала обновления после открытий, полученных в результате экспериментальных проверок с использованием методов RACE и RT-PCR . [2] Версия 2 GENCODE содержала 411 известных локусов, 30 новых локусов CDS, 81 новый локус транскриптов, 83 предполагаемых локуса, 104 процессированных псевдогена и 66 непроцессированных псевдогенов.
2007 июнь
Выводы пилотного проекта были опубликованы в июне 2007 года. [10] Полученные результаты подчеркнули успех пилотного проекта по созданию реальной платформы и новых технологий для характеристики функциональных элементов генома человека, что открывает путь к открытию полногеномных исследований.
2007 октябрь
Новое финансирование было частью усилий NHGRI по расширению проекта ENCODE до этапа производства всего генома наряду с дополнительными пилотными исследованиями.
2012 сентябрь
В сентябре 2012 года консорциум GENCODE опубликовал крупный документ, в котором обсуждаются результаты основного выпуска — GENCODE Release 7, который был заморожен в декабре 2011 года. [11]
2018
В 2018 году одним из последних дополнений к проекту GENCODE стал трек CRISPR/Cas9 на сборках человека и модельных организмов. CRISPR — это метод редактирования генома, в котором используются последовательности РНК, которые успешно связываются с отредактированной областью с высокой специфичностью. Новый трек был разработан, чтобы помочь в поиске подходящих направляющих предложений путем перечисления потенциальных сайтов связывания комплекса CRISPR/Cas9, которые находятся рядом с транскрибируемыми областями или в пределах 200 п.н. от них. Для каждого сайта трек предоставляет возможные направляющие последовательности, а также набор прогнозируемых показателей эффективности и специфичности для этих направляющих последовательностей. Он также предоставляет информацию о потенциальных отклонениях от цели, сгруппированную по количеству несоответствий между отклонением от цели и ориентиром. [11]
2020
Среди других достижений: завершена первая ручная аннотация эталонного генома мыши, начато сотрудничество с базами данных эталонных аннотаций RefSeq и Uniprot для достижения конвергенции аннотаций, а аннотация днРНК была улучшена за счет открытия новых локусов. и новые транскрипты в существующих локусах. Кроме того, учитывая пандемию COVID-19 в 2020 году, возникла необходимость поддержать исследования, реагирующие на ситуацию, поэтому GENCODE рассмотрел и улучшил аннотацию для набора генов, кодирующих белки, связанных с инфекцией SARSCoV-2. [12]
Ключевые участники
[ редактировать ]Ключевые участники проекта GENCODE оставались относительно последовательными на всех его этапах, а теперь общий проект возглавляет Институт Wellcome Trust Sanger.
Краткое описание ключевых учреждений, участвующих на каждом этапе, перечислено ниже: [6] [13]
| ГЕНКОД, этап 2 (текущий) | Этап масштабирования GENCODE | Пилотный этап GENCODE | |
|---|---|---|---|
| Wellcome Sanger Institute , Кембридж, Великобритания | Wellcome Sanger Institute, Кембридж, Великобритания | Wellcome Trust Sanger Institute, Кембридж, Великобритания
| |
| Центр геномного регулирования, Барселона, Каталония, Испания | Центр геномного регулирования, Барселона, Каталония, Испания | Муниципальный институт медицинских исследований (IMIM), Барселона, Каталония, Испания | |
| Университет Лозанны , Швейцария | Университет Лозанны, Швейцария | Женевский университет , Швейцария | |
| Калифорнийский университет, Санта-Круз , Калифорния, США | Калифорнийский университет, Санта-Круз, США | Вашингтонский университет в Сент-Луисе, США | |
| Массачусетский технологический институт , Бостон, США | Массачусетский технологический институт, Бостон, США | Калифорнийский университет, Беркли, США | |
| Йельский университет , Нью-Хейвен, США | Йельский университет, Нью-Хейвен, США | Европейский институт биоинформатики , Хинкстон, Великобритания | |
| Испанский национальный онкологический исследовательский центр (CNIO), Мадрид, Испания | Испанский национальный онкологический исследовательский центр, Мадрид, Испания | ||
| Вашингтонский университет в Сент-Луисе , США |
Участники, ИП и СО-ИП
[ редактировать ]Источник: [8]
- Пол Фличек (ведущий исследователь), Европейский институт биоинформатики EMBL, Кембридж, Великобритания
- Родерик Гиго (PI), Центр геномного регулирования (CRG), Барселона, Каталония, Испания
- Манолис Келлис (PI), Массачусетский технологический институт (MIT), Бостон, США
- Марк Б. Герштейн (PI), Йельский университет, Нью-Хейвен, США
- Бенедикт Пейтен (PI), Калифорнийский университет, Санта-Круз, Калифорния, США
- Майкл Тресс, Испанский национальный онкологический исследовательский центр (CNIO), Мадрид, Испания
- Джиоти Чоудхари, Институт исследования рака (ICR), Лондон, Великобритания
Ключевая статистика
[ редактировать ]С момента своего создания GENCODE выпустила 36 версий аннотаций набора генов человека (исключая незначительные обновления).
Ключевые сводные статистические данные самой последней аннотации набора генов человека GENCODE ( выпуск 36, заморозка в декабре 2020 г. ) показаны ниже: [14]
| Категории | Общий | Категории | Общий |
|---|---|---|---|
| Общее количество генов | 60,660 | Общее количество транскриптов | 232,117 |
| Гены, кодирующие белки | 19,962 | Транскрипты, кодирующие белок | 85,269 |
| Гены длинных некодирующих РНК | 17,958 | - полноразмерное кодирование белка: | 59,269 |
| Малые некодирующие РНК-гены | 7,569 | - кодирование белков частичной длины: | 26,000 |
| Псевдогены | 14,761 | Бессмысленные транскрипты распада, опосредованные | 17,378 |
| - обработанные псевдогены: | 10,669 | Транскрипты длинных некодирующих локусов РНК | 48,734 |
| - необработанные псевдогены: | 3,554 | ||
| - унитарные псевдогены: | 236 | ||
| - полиморфные псевдогены: | 48 | ||
| - псевдогены: | 18 | ||
| Сегменты гена иммуноглобулина/рецептора Т-клеток | 645 | Общее количество различных переводов | 63,058 |
| - сегменты, кодирующие белок: | 409 | Гены, имеющие более чем одну различную трансляцию | 13,685 |
| - псевдогены: | 236 |
Благодаря достижениям в технологиях секвенирования (таких как RT-PCR-seq), расширению охвата ручных аннотаций (группа HAVANA) и усовершенствованию алгоритмов автоматического аннотирования с использованием Ensembl, точность и полнота аннотаций GENCODE постоянно совершенствуются посредством итерации выпусков. .
Ниже показано сравнение ключевых статистических данных трех основных выпусков GENCODE до 2014 года. [14] Очевидно, что хотя охват по общему числу обнаруженных генов неуклонно растет, количество генов, кодирующих белки, фактически уменьшается. В основном это связано с новыми экспериментальными данными, полученными с использованием кластеров экспрессии генов Cap Analysis (CAGE) , аннотированных сайтов PolyA и совпадений пептидов . [11]
- Версия 7 (заморозка в декабре 2010 г., ГРЧ37) - Ансамбль 62
- Версия 10 (заморозка в июле 2011 г., ГРЧ37) - Ансамбль 65
- Версия 20 (заморозка в апреле 2014 г., ГРЧ38) - Ансамбль 76
- Сравнение версий GENCODE Human (расшифровки)
- Сравнение человеческих версий GENCODE (Гены)
- Сравнение версий GENCODE Human (переводы)
Методология
[ редактировать ]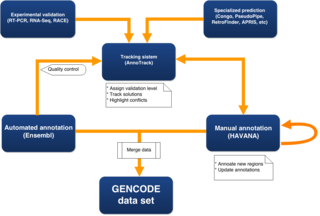
Предполагаемые локусы можно проверить с помощью экспериментов в мокрой лаборатории, а компьютерные прогнозы анализируются вручную. [15] В настоящее время, чтобы гарантировать, что набор аннотаций охватывает весь геном, а не только области, которые были аннотированы вручную, создается объединенный набор данных с использованием ручных аннотаций из HAVANA вместе с автоматическими аннотациями из автоматически аннотируемого набора генов Ensembl. Этот процесс также добавляет уникальные полноразмерные предсказания CDS из набора кодирующих белков Ensembl в гены, аннотированные вручную, чтобы обеспечить наиболее полную и актуальную аннотацию генома. [16]
Автоматическое аннотирование (Ансамбль)
[ редактировать ]Транскрипты Ensembl представляют собой продукты автоматической системы аннотаций генов Ensembl (набор конвейеров аннотаций генов), называемой сборкой генов Ensembl. Все транскрипты Ensembl основаны на экспериментальных данных, поэтому автоматизированный конвейер опирается на мРНК и белковые последовательности, депонированные в общедоступные базы данных научного сообщества. [17]
Ручная аннотация (группа HAVANA)
[ редактировать ]В консорциуме GENCODE есть несколько аналитических групп, которые управляют конвейерами, помогающими ручным аннотаторам создавать модели в неаннотированных регионах и выявлять потенциально пропущенные или неправильные ручные аннотации, включая полностью отсутствующие локусы, отсутствующие альтернативные изоформы, неправильные сайты сплайсинга и неправильные биотипы. Они передаются аннотаторам, работающим вручную, с помощью системы отслеживания AnnoTrack. [18] Некоторые из этих конвейеров используют данные из других подгрупп ENCODE, включая данные RNASeq, модификации гистонов, а также данные CAGE и Ditag. Данные RNAseq являются важным новым источником данных, но создание на их основе полных моделей генов является сложной проблемой. В рамках GENCODE было проведено соревнование по оценке качества прогнозов, полученных с помощью различных конвейеров прогнозирования RNAseq (см. RGASP ниже). Для подтверждения неопределенных моделей GENCODE также имеет конвейер экспериментальной проверки с использованием секвенирования РНК и RACE. [16]
Оценка качества
[ редактировать ]Для GENCODE 7 моделям транскриптов присваивается высокий или низкий уровень поддержки на основе нового метода, разработанного для оценки качества транскриптов. [2]
Использование/Доступ
[ редактировать ]Текущая версия набора генов человека GENCODE (выпуск 20 GENCODE) включает файлы аннотаций (в форматах GTF и GFF3), файлы FASTA и файлы METADATA, связанные с аннотацией GENCODE во всех геномных регионах (эталонные хромосомы/патчи/каркасы/гаплотипы). Данные аннотаций передаются на эталонные хромосомы и хранятся в отдельных файлах, которые включают: аннотацию генов, признаки PolyA, аннотированные HAVANA, (ретротранспонированные) псевдогены, предсказанные конвейерами Йельского университета и UCSC, но не HAVANA, длинные некодирующие РНК и тРНК. структуры, предсказанные с помощью тРНК-сканирования.Некоторые примеры строк в формате GTF показаны ниже:
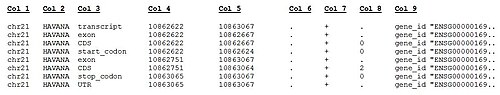
Столбцы в форматах файлов GENCODE GTF описаны ниже.
Описание формата файла GENCODE GTF. Стандартные столбцы GTF, разделенные TAB
| Номер столбца | Содержание | Значения/формат |
|---|---|---|
| 1 | название хромосомы | chr{1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,X,Y, М} |
| 2 | источник аннотации | {ENSEMBL, ГАВАНА} |
| 3 | тип объекта | {ген, транскрипт, экзон, CDS, UTR, стартовый_кодон, стоп_кодон, селеноцистеин} |
| 4 | начальное местоположение генома | целочисленное значение (отсчет от 1) |
| 5 | конечное местоположение генома | целочисленное значение |
| 6 | оценка (не используется) | . |
| 7 | геномная цепь | {+,-} |
| 8 | геномная фаза (для функций CDS) | {0,1,2,.} |
| 9 | дополнительная информация в виде пар ключ-значение | См. объяснение в таблице ниже. |
Описание пар ключ-значение в 9-м столбце файла GENCODE GTF (формат: ключ «значение»)
| Имя ключа | Формат значения |
|---|---|
| ген_ид | ENSGXXXXXXXXXXX |
| расшифровка_id | ЭНСХХХХХХХХХ |
| генный_тип | список биотипов |
| генный_статус | {ИЗВЕСТНО, РОМАНА, ПРЕДПОЛАГАЕМОЕ} |
| имя_гена | нить |
| тип_расшифровки | список биотипов |
| транскрипт_статус | {ИЗВЕСТНО, РОМАНА, ПРЕДПОЛАГАЕМОЕ} |
| имя_расшифровки | нить |
| экзон_номер | указывает биологическое положение экзона в транскрипте |
| exon_id | ЭНСЕКХХХХХХХХХ |
| уровень |
|
Браузер генома биодальянса
[ редактировать ]Кроме того, веб-сайт GENCODE содержит браузер генома для человека и мыши, где вы можете получить доступ к любой области генома, указав номер хромосомы и начальную и конечную позицию (например, 22:30,700,000..30,900,000), а также идентификатор транскрипта ENS (с/ без версии), идентификатор гена ENS (с версией/без версии) и имя гена. Браузер работает на платформе Biodalliance. [19]
Проблемы
[ редактировать ]Определение «гена»
[ редактировать ]Определение «гена» никогда не было тривиальным вопросом: за годы, прошедшие после открытия генома человека, было предложено множество определений и понятий. Сначала гены были задуманы в 1900-х годах как отдельные единицы наследственности, затем их считали основой синтеза белка, а в более поздние времена их стали определять как генетический код, транскрибируемый в РНК. Хотя определение гена сильно изменилось за последнее столетие, оно остается сложной и противоречивой темой для многих исследователей. С появлением проекта ENCODE/GENCODE были обнаружены еще более проблематичные аспекты определения, включая альтернативный сплайсинг (когда ряд экзонов разделены интронами), межгенную транскрипцию и сложные модели дисперсной регуляции, а также не -генная консервативность и обилие некодирующих генов РНК. Поскольку GENCODE пытается создать энциклопедию генов и вариантов генов, эти проблемы представляют собой растущую задачу для проекта GENCODE по разработке обновленного понятия гена. [20]
Проект «Геном человека»
[ редактировать ]Проект «Геном человека» представлял собой международную исследовательскую попытку определить последовательность генома человека и идентифицировать содержащиеся в нем гены. Проект координировали Национальные институты здравоохранения и Министерство энергетики США. Среди дополнительных участников были университеты США и международные партнеры в Великобритании, Франции, Германии, Японии и Китае. Проект «Геном человека» официально начался в 1990 году и был завершен в 2003 году, на два года раньше первоначального графика. [21]
Подпроекты
[ редактировать ]Вместе
[ редактировать ]Ensembl является частью проекта GENCODE. [22]
Дизайн микрочипа для экспрессии днРНК
[ редактировать ]Ключевой областью исследований проекта GENCODE было изучение биологической значимости длинных некодирующих РНК (днРНК). Чтобы лучше понять экспрессию днРНК у людей, GENCODE создал подпроект по разработке индивидуальных платформ микрочипов, способных количественно определять транскрипты в аннотации днРНК GENCODE. [4] Ряд проектов был создан с использованием системы Agilent Technologies eArray, и эти проекты доступны в стандартном пользовательском формате Agilent. [4]
РГАСП
[ редактировать ]Проект оценки аннотаций генома RNA-seq (RGASP) предназначен для оценки эффективности различных вычислительных методов для высококачественного анализа данных последовательностей РНК. Основными целями RGASP являются предоставление объективной оценки программного обеспечения для выравнивания РНК-секвенирования, характеристики транскриптов (обнаружение, реконструкция и количественная оценка), а также определение возможности автоматизированных аннотаций генома на основе секвенирования транскриптома. [23]
RGASP организован в рамках консорциума, созданного по образцу семинара по прогнозированию генов EGASP (ENCODE Genome Annotation Assessment Project), и были проведены два раунда семинаров, посвященных различным аспектам анализа секвенирования РНК, а также изменению технологий и форматов секвенирования. Одним из главных открытий, сделанных в ходе 1 и 2 раундов проекта, стала важность согласованности прочтений для качества прогнозов генов. Таким образом, в настоящее время (в 2014 г.) проводится третий раунд семинара RGASP, который сосредоточится в первую очередь на сопоставлении чтения с геномом. [23]
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Харроу Дж., Фрэнкиш А., Гонсалес Дж.М., Тапанари Э., Диханс М., Кокочински Ф. и др. (сентябрь 2012 г.). «GENCODE: эталонная аннотация генома человека для проекта ENCODE» (PDF) . Геномные исследования . 22 (9): 1760–74. дои : 10.1101/гр.135350.111 . ПМЦ 3431492 . ПМИД 22955987 .
- ^ Jump up to: а б с д и ж Харроу Дж., Дено Ф., Фрэнкиш А., Реймонд А., Чен С.К., Краст Дж. и др. (2006). «GENCODE: создание справочной аннотации для ENCODE» . Геномная биология . 7 (Приложение 1): С4.1–9. дои : 10.1186/gb-2006-7-s1-s4 . ПМК 1810553 . ПМИД 16925838 .
- ^ Фрэнкиш А., Мадж Дж. М., Томас М., Харроу Дж. (2012). «Важность выявления альтернативного сплайсинга в аннотации генома позвоночных» . База данных . 2012 : bas014. дои : 10.1093/база данных/bas014 . ПМК 3308168 . ПМИД 22434846 .
- ^ Jump up to: а б с Дерриен Т., Джонсон Р., Буссотти Г., Танзер А., Джебали С., Тилгнер Х. и др. (сентябрь 2012 г.). «Каталог длинных некодирующих РНК человека GENCODE v7: анализ их генной структуры, эволюции и экспрессии» . Геномные исследования . 22 (9): 1775–89. дои : 10.1101/гр.132159.111 . ПМЦ 3431493 . ПМИД 22955988 .
- ^ Пей Б., Сису С., Франкиш А., Ховальд С., Хабеггер Л., Му XJ и др. (сентябрь 2012 г.). «Ресурс псевдогена GENCODE» . Геномная биология . 13 (9): С51. дои : 10.1186/gb-2012-13-9-r51 . ПМЦ 3491395 . ПМИД 22951037 .
- ^ Jump up to: а б «GENCODE — Домашняя страница» . 20 декабря 2020 г.
- ^ Jump up to: а б «ГЕНКОД – Данные» . ГЕНКОД . Wellcome Trust Sanger Institute. Сентябрь 2019 года . Проверено 14 октября 2019 г.
- ^ Jump up to: а б «ГЕНКОД» . Wellcome Trust Sanger Institute. п. Проект GENCODE: Энциклопедия генов и вариантов генов . Проверено 20 декабря 2020 г.
- ^ Jump up to: а б Консорциум проекта ENCODE (октябрь 2004 г.). «Проект ENCODE (ЭНЦИКЛОПЕДИЯ ЭЛЕМЕНТОВ ДНК)» . Наука . 306 (5696): 636–40. Бибкод : 2004Sci...306..636E . дои : 10.1126/science.1105136 . ПМИД 15499007 . S2CID 22837649 .
- ^ Бирни Э., Стаматояннопулос Х.А., Дутта А., Гиго Р., Гингерас Т.Р., Маргулис Э.Х. и др. (июнь 2007 г.). «Идентификация и анализ функциональных элементов в 1% генома человека в рамках пилотного проекта ENCODE» . Природа . 447 (7146): 799–816. Бибкод : 2007Natur.447..799B . дои : 10.1038/nature05874 . ПМК 2212820 . ПМИД 17571346 .
- ^ Jump up to: а б с Каспер Дж., Цвейг А.С., Вильярреал С., Тайнер С., Спейр М.Л., Розенблум К.Р. и др. (январь 2018 г.). «База данных браузера генома UCSC: обновление 2018 г.» . Исследования нуклеиновых кислот . 46 (Д1): Д762–Д769. дои : 10.1093/нар/gkx1020 . ПМЦ 5753355 . ПМИД 29106570 .
- ^ Франкиш А., Дикханс М., Юнгрейс И., Лагард Дж., Лавленд Дж.Э., Мадж Дж.М. и др. (декабрь 2020 г.). «ГЕНКОД 2021» . Исследования нуклеиновых кислот . 49 (Д1): Д916–Д923. дои : 10.1093/nar/gkaa1087 . ПМЦ 7778937 . ПМИД 33270111 . S2CID 227260109 .
- ^ «Участники проекта GENCODE» . Лаборатория геномных исследований биоинформатики . в. 2005 . Проверено 8 сентября 2014 г.
- ^ Jump up to: а б «ГЕНКОД – Статистика» . ГЕНКОД . Wellcome Trust Sanger Institute. в. 2014. Архивировано из оригинала 19 июня 2018 года . Проверено 20 декабря 2020 г.
{{cite web}}: CS1 maint: bot: исходный статус URL неизвестен ( ссылка ) - ^ «ГЕНКОД – Цели» . ГЕНКОД . Wellcome Trust Sanger Institute. в. 2013 . Проверено 5 сентября 2014 г.
- ^ Jump up to: а б Сирл С., Фрэнкиш А., Бигнелл А., Акен Б., Дерриен Т., Дикханс М. и др. (2010). «Набор генов человека GENCODE» . Геномная биология . 11 (Приложение 1): 36. doi : 10.1186/gb-2010-11-S1-P36 . ПМК 3026266 .
- ^ «Ансамбль - Домашняя страница» . Ансамбль . Август 2014 года . Проверено 6 сентября 2014 г.
- ^ Кокочински Ф., Харроу Дж., Хаббард Т. (октябрь 2010 г.). «AnnoTrack — система отслеживания аннотаций генома» . БМК Геномика . 11 : 538. дои : 10.1186/1471-2164-11-538 . ПМК 3091687 . ПМИД 20923551 .
- ^ «Биодальянс - Домашняя страница» . 20 декабря 2020 г.
- ^ Герштейн М.Б., Брюс С., Розовский Дж.С., Чжэн Д., Ду Дж., Корбель Дж.О. и др. (июнь 2007 г.). «Что такое ген пост-КОДИРОВАНИЕ? История и обновленное определение» . Геномные исследования . 17 (6): 669–81. дои : 10.1101/гр.6339607 . ПМИД 17567988 .
- ^ «Проект «Геном человека» — Домашняя страница» . 20 декабря 2020 г.
- ^ «КОДИРОВАТЬ данные в Ensembl» . Ансамбль . Август 2014. Архивировано из оригинала 9 ноября 2017 года . Проверено 7 сентября 2014 г.
- ^ Jump up to: а б Стейгер Т., Абриль Дж. Ф., Энгстрем П.Г., Кокочински Ф., Хаббард Т.Дж., Гиго Р. и др. (декабрь 2013 г.). «Оценка методов реконструкции транскрипта для РНК-сек» . Природные методы . 10 (12): 1177–84. дои : 10.1038/nmeth.2714 . ПМЦ 3851240 . ПМИД 24185837 .


