Альтернативный сплайсинг

Альтернативный сплайсинг , или альтернативный сплайсинг РНК , или дифференциальный сплайсинг — это альтернативный процесс сплайсинга во время экспрессии генов, который позволяет одному гену производить различные варианты сплайсинга. Например, некоторые экзоны гена могут быть включены в конечный РНК-продукт гена или исключены из него. [1] Это означает, что экзоны соединяются в разных комбинациях, что приводит к разным вариантам сплайсинга. В случае генов, кодирующих белки, белки, транслированные из этих вариантов сплайсинга, могут содержать различия в аминокислотной последовательности и биологических функциях (см. Рисунок).
Биологически значимый альтернативный сплайсинг происходит как нормальное явление у эукариот , где он увеличивает количество белков, которые могут кодироваться геномом. [1] Широко распространено мнение, что у людей около 95% мультиэкзонных генов подвергаются альтернативному сплайсингу с образованием функциональных альтернативных продуктов из одного и того же гена. [2] но многие ученые полагают, что большинство наблюдаемых вариантов сплайсинга происходят из-за ошибок сплайсинга, а фактическое количество биологически значимых альтернативно сплайсированных генов намного ниже. [3] [4]
Открытие [ править ]
Альтернативный сплайсинг впервые был обнаружен в 1977 году. [5] [6] Аденовирус производит пять первичных транскриптов в начале своего инфекционного цикла, до репликации вирусной ДНК, и еще один позже, после начала репликации ДНК. Ранние первичные транскрипты продолжают производиться после начала репликации ДНК. Дополнительный первичный транскрипт, образующийся на поздних стадиях инфекции, имеет большой размер и составляет 5/6 генома аденовируса размером 32 т.п.н. Это намного больше, чем любая из отдельных мРНК аденовируса, присутствующих в инфицированных клетках. Исследователи обнаружили, что первичный транскрипт РНК, продуцируемый аденовирусом типа 2 на поздней стадии, подвергался сплайсингу разными способами, в результате чего образовывались мРНК, кодирующие различные вирусные белки. Кроме того, первичный транскрипт содержал несколько сайтов полиаденилирования , что давало разные 3'-концы процессированным мРНК. [7] [8] [9]
первый пример альтернативного сплайсинга транскрипта нормального эндогенного гена . В 1981 году был охарактеризован [7] ген, кодирующий щитовидной железы гормон кальцитонин, Было обнаружено, что альтернативно сплайсируется в клетках млекопитающих. Первичный транскрипт этого гена содержит 6 экзонов; мРНК кальцитонина содержит экзоны 1–4 и заканчивается после сайта полиаденилирования в экзоне 4. Другая мРНК образуется из этой пре-мРНК путем пропуска экзона 4 и включает экзоны 1–3, 5 и 6. Она кодирует белок, известный как CGRP ( пептид, родственный гену кальцитонина ). [10] [11] Примеры альтернативного сплайсинга транскриптов генов иммуноглобина у млекопитающих наблюдались также в начале 1980-х годов. [7] [12]
С тех пор у эукариот было обнаружено множество других примеров биологически значимого альтернативного сплайсинга. [1] «Рекордсменом» по альтернативному сплайсингу является ген D. melanogaster под названием Dscam , который потенциально может иметь 38 016 вариантов сплайсинга. [13]
В 2021 году было обнаружено, что геном аденовируса типа 2, аденовируса, в котором впервые был идентифицирован альтернативный сплайсинг, способен производить гораздо большее разнообразие вариантов сплайсинга, чем считалось ранее. [14] Используя технологию секвенирования нового поколения, исследователи смогли обновить транскриптом аденовируса человека типа 2 и задокументировать наличие 904 вариантов сплайсинга, продуцируемых вирусом, посредством сложной схемы альтернативного сплайсинга. Было показано, что очень немногие из этих вариантов сращивания функциональны, и авторы поднимают этот вопрос в своей статье.
- «Нерешенный вопрос заключается в том, какую роль играет зверинец новых РНК, или же они являются ложными молекулами, созданными перегруженным механизмом сплайсинга». [14]
Режимы [ править ]

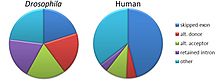
Общепризнаны пять основных способов альтернативного сплайсинга. [1] [2] [16] [15]
- Пропуск экзона или кассетный экзон : в этом случае экзон может быть сплайсирован из первичного транскрипта или сохранен. Это наиболее распространенный тип пре-мРНК млекопитающих . [15]
- Взаимоисключающие экзоны : после сплайсинга в мРНК сохраняется один из двух экзонов, но не оба.
- Альтернативный донорский сайт : используется альтернативный 5'-конец сплайсинга (донорский сайт), изменяющий 3'-границу верхнего экзона.
- Альтернативный акцепторный сайт : используется альтернативный 3'-сайт сплайсинга (акцепторный сайт), изменяющий 5'-границу нижестоящего экзона.
- Сохранение интрона . Последовательность может быть вырезана как интрон или просто сохранена. Это отличается от пропуска экзона, поскольку сохраненная последовательность не окружена интронами . Если сохраненный интрон находится в кодирующей области, он должен кодировать аминокислоты в рамке с соседними экзонами, иначе стоп-кодон или сдвиг рамки считывания приведут к тому, что белок станет нефункциональным. Это самый редкий способ у млекопитающих, но наиболее распространенный у растений. [15] [17]
Помимо этих основных способов альтернативного сплайсинга, существуют два других основных механизма, с помощью которых разные мРНК могут генерироваться из одного и того же гена; несколько промоторов и несколько полиаденилирования сайтов . Использование нескольких промоторов правильно описывается как механизм регуляции транскрипции, а не как альтернативный сплайсинг; начиная транскрипцию в разных точках, можно генерировать транскрипты с разными 5'-экзонами. На другом конце несколько сайтов полиаденилирования обеспечивают разные 3'-конечные точки транскрипта. Оба этих механизма обнаруживаются в сочетании с альтернативным сплайсингом и обеспечивают дополнительное разнообразие мРНК, полученных из гена. [1] [16]

Эти режимы описывают основные механизмы сплайсинга, но могут оказаться недостаточными для описания сложных событий сплайсинга. Например, на рисунке справа показаны 3 сплайс-формы гена мышиной гиалуронидазы 3. Сравнение экзонной структуры, показанной в первой строке (зеленый), со структурой во второй строке (желтый), показывает сохранение интрона, тогда как сравнение второй и третьей сплайс-формы (желтый и синий) демонстрирует пропуск экзона. Недавно была предложена модельная номенклатура для однозначного обозначения всех возможных схем сплайсинга. [15]
Механизмы [ править ]
Общий механизм сращивания [ править ]

Когда пре-мРНК транскрибируется с ДНК , она включает несколько интронов и экзонов . (У нематод среднее число составляет 4–5 экзонов и интронов; у плодовой мухи Drosophila в одной транскрибируемой пре-мРНК может быть более 100 интронов и экзонов.) Экзоны, которые должны сохраниться в мРНК , определяются в процессе сплайсинга. . Регуляция и выбор сайтов сплайсинга осуществляются транс-действующими белками-активаторами сплайсинга и белками-репрессорами сплайсинга, а также цис-действующими элементами внутри самой пре-мРНК, такими как экзонные усилители сплайсинга и экзонные сайленсеры сплайсинга.
Типичный эукариотический ядерный интрон имеет консенсусные последовательности, определяющие важные области. Каждый интрон имеет последовательность GU на своем 5'-конце. Около 3'-конца имеется участок разветвления. Нуклеотид в точке ветвления всегда представляет собой букву А; консенсус вокруг этой последовательности несколько различается. У людей консенсусная последовательность сайта ветвления представляет собой yUnAy. [18] За местом ветвления следует серия пиримидинов – полипиримидиновый тракт – затем AG на 3'-конце. [16]
Сплайсинг мРНК осуществляется комплексом РНК и белка, известным как сплайсосома , содержащим мяРНП , обозначенные U1, U2 , U4, U5 и U6 (U3 не участвует в сплайсинге мРНК). [19] U1 связывается с 5'-GU, а U2 с помощью белковых факторов U2AF связывается с точкой ветвления А внутри сайта ветвления. Комплекс на этой стадии известен как комплекс сплайсосомы А. Формирование комплекса A обычно является ключевым шагом в определении концов интрона, который необходимо отсоединить, и определения концов экзона, который необходимо сохранить. [16] (Номенклатура U обусловлена высоким содержанием уридина).
Комплекс U4,U5,U6 связывается, и U6 заменяет позицию U1. U1 и U4 уходят. Оставшийся комплекс затем осуществляет две реакции переэтерификации . При первой переэтерификации 5'-конец интрона отщепляется от верхнего экзона и соединяется с сайтом ветвления А посредством 2',5'- фосфодиэфирной связи. При второй переэтерификации 3'-конец интрона отщепляется от нижестоящего экзона, и два экзона соединяются фосфодиэфирной связью. Затем интрон высвобождается в лариатной форме и разрушается. [1]
и Регуляторные белки элементы

Сплайсинг регулируется транс-действующими белками (репрессорами и активаторами) и соответствующими цис-действующими регуляторными сайтами (сайленсерами и энхансерами) на пре-мРНК. Однако, учитывая сложность альтернативного сплайсинга, следует отметить, что эффекты фактора сплайсинга часто зависят от положения. То есть фактор сплайсинга, который служит активатором сплайсинга при связывании с интронным энхансерным элементом, может служить репрессором при связывании со своим сплайсинговым элементом в контексте экзона, и наоборот. [20] Вторичная структура транскрипта пре-мРНК также играет роль в регуляции сплайсинга, например, путем объединения элементов сплайсинга или маскировки последовательности, которая в противном случае служила бы связывающим элементом для фактора сплайсинга. [21] [22] Вместе эти элементы образуют «код сплайсинга», который определяет, как будет происходить сплайсинг в различных клеточных условиях. [23] [24]
Существует два основных типа элементов последовательности цис-действующей РНК, присутствующих в пре-мРНК, и они имеют соответствующие транс-действующие РНК-связывающие белки . сплайсинга Сайленсеры - это сайты, с которыми связываются белки-репрессоры сплайсинга, что снижает вероятность того, что соседний сайт будет использоваться в качестве соединения сплайсинга. Они могут быть расположены в самом интроне (интронные сайленсеры сплайсинга, ISS) или в соседнем экзоне ( экзонные сайленсеры сплайсинга , ESS). Они различаются по последовательности, а также по типам белков, которые с ними связываются. Большинство репрессоров сплайсинга представляют собой гетерогенные ядерные рибонуклеопротеины (hnRNP), такие как hnRNPA1 и белок, связывающий полипиримидиновый тракт (PTB). [16] [23] сплайсинга Энхансеры — это сайты, с которыми связываются белки-активаторы сплайсинга, что увеличивает вероятность того, что близлежащий сайт будет использоваться в качестве соединения сплайсинга. Они также могут возникать в интроне (интронные энхансеры сплайсинга, ISE) или экзоне ( экзонные энхансеры сплайсинга , ESE). Большинство белков-активаторов, которые связываются с ISE и ESE, являются членами семейства белков SR . Такие белки содержат мотивы узнавания РНК и домены, богатые аргинином и серином (RS). [16] [23]
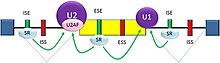
В общем, детерминанты сплайсинга работают взаимозависимо, в зависимости от контекста, так что правила, регулирующие сплайсинг, образуют код сплайсинга. [24] Присутствие определенного элемента последовательности цис-действующей РНК может увеличить вероятность сплайсинга близлежащего сайта в некоторых случаях, но уменьшить вероятность в других случаях, в зависимости от контекста. Контекст, в котором действуют регуляторные элементы, включает контекст цис-действия, который устанавливается наличием других особенностей последовательности РНК, и контекст транс-действия, который устанавливается клеточными условиями. Например, некоторые элементы последовательности цис-действующей РНК влияют на сплайсинг только в том случае, если несколько элементов присутствуют в одной и той же области, чтобы установить контекст. В качестве другого примера, цис-действующий элемент может оказывать противоположные эффекты на сплайсинг, в зависимости от того, какие белки экспрессируются в клетке (например, нейрональные или ненейрональные PTB). Адаптивная значимость сплайсинга сайленсеров и энхансеров подтверждена исследованиями, показывающими, что в генах человека существует сильный отбор против мутаций, которые производят новые сайленсеры или разрушают существующие энхансеры. [25] [26]
Примеры [ править ]
: Drosophila dsx экзона Пропуск
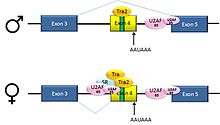
Пре-мРНК гена melanogaster dsx D. содержат 6 экзонов. У самцов экзоны 1,2,3,5 и 6 соединяются, образуя мРНК, которая кодирует белок, регулирующий транскрипцию, необходимый для развития самцов. У женщин экзоны 1,2,3 и 4 соединяются, и сигнал полиаденилирования в экзоне 4 вызывает расщепление мРНК в этой точке. Полученная мРНК представляет собой белок, регулирующий транскрипцию, необходимый для развития самок. [27]
Это пример пропуска экзона. Интрон выше экзона 4 имеет полипиримидиновый тракт , который плохо соответствует консенсусной последовательности , поэтому белки U2AF плохо связываются с ним без помощи активаторов сплайсинга. Поэтому этот 3'-акцепторный сайт сплайсинга не используется у самцов. Однако самки производят активатор сплайсинга Трансформатор (Тра) (см. ниже). Белок SR Tra2 вырабатывается у представителей обоих полов и связывается с ESE в экзоне 4; если Tra присутствует, он связывается с Tra2 и вместе с другим белком SR образует комплекс, который помогает белкам U2AF связываться со слабым полипиримидиновым трактом. U2 рекрутируется в соответствующую точку ветвления, и это приводит к включению экзона 4 в мРНК. [27] [28]
Альтернативные акцепторные сайты: дрозофила. Трансформатор [ править ]

Пре-мРНК гена трансформера (Tra) Drosophila melanogaster подвергаются альтернативному сплайсингу посредством режима альтернативного акцепторного сайта. Ген Tra кодирует белок, который экспрессируется только у женщин. Первичный транскрипт этого гена содержит интрон с двумя возможными акцепторными сайтами. У самцов используется верхний акцепторный сайт. Это приводит к включению в обрабатываемый транскрипт более длинной версии экзона 2, включая ранний стоп-кодон . Полученная мРНК кодирует усеченный белковый продукт, который неактивен. Самки производят главный белок определения пола Sex Lethal (Sxl). Белок Sxl представляет собой репрессор сплайсинга, который связывается с ISS в РНК транскрипта Tra вблизи вышестоящего акцепторного сайта, предотвращая связывание белка U2AF с полипиримидиновым трактом. Это предотвращает использование этого соединения, смещая связывание сплайсосомы к нижележащему акцепторному сайту. Сплайсинг в этой точке обходит стоп-кодон, который вырезается как часть интрона. Полученная мРНК кодирует активный белок Tra, который сам по себе является регулятором альтернативного сплайсинга других генов, связанных с полом (см. dsx выше). [1]
: рецептор Fas Определение экзона
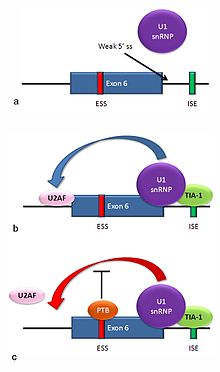
Множественные изоформы белка рецептора Fas производятся путем альтернативного сплайсинга. Две обычно встречающиеся у человека изоформы производятся по механизму пропуска экзона. мРНК, включающая экзон 6, кодирует мембраносвязанную форму рецептора Fas, которая способствует апоптозу или запрограммированной гибели клеток. Повышенная экспрессия рецептора Fas в клетках кожи, хронически подвергающихся воздействию солнца, и отсутствие экспрессии в клетках рака кожи позволяют предположить, что этот механизм может быть важен для устранения предраковых клеток у людей. [29] Если экзон 6 пропущен, полученная мРНК кодирует растворимый белок Fas, который не способствует апоптозу. Включение или пропуск экзона зависит от двух антагонистических белков: TIA-1 и белка, связывающего полипиримидиновый тракт (PTB).
- 5'-донорный сайт в интроне ниже экзона 6 в пре-мРНК имеет слабое согласие с консенсусной последовательностью и обычно не связывается с мяРНП U1. Если U1 не связывается, экзон пропускается (см. «а» на сопроводительном рисунке).
- Связывание белка TIA-1 с сайтом энхансера интронного сплайсинга стабилизирует связывание мяРНП U1. [16] Образующийся в результате комплекс 5'-донорного сайта способствует связыванию фактора сплайсинга U2AF с 3'-сайтом сплайсинга выше экзона посредством механизма, который еще не известен (см. b). [30]
- Экзон 6 содержит богатый пиримидином экзонный сайленсер сплайсинга, ure6 , с которым может связываться PTB. Если PTB связывается, он ингибирует влияние 5'-донорного комплекса на связывание U2AF с акцепторным сайтом, что приводит к пропуску экзона (см. c).
Этот механизм является примером определения экзонов при сплайсинге. Сплайсосома собирается на интроне, и субъединицы snRNP сворачивают РНК так, что 5'- и 3'-концы интрона соединяются. Однако недавно изученные примеры, подобные этому, показывают, что между концами экзона также существуют взаимодействия. В этом конкретном случае эти взаимодействия определения экзонов необходимы, чтобы обеспечить связывание основных факторов сплайсинга до сборки сплайсосом на двух фланкирующих интронах. [30]
активатора: ВИЧ экзон 2 - 1 Конкуренция репрессора - tat

ВИЧ , ретровирус , вызывающий СПИД у людей, продуцирует единственный первичный транскрипт РНК, который альтернативно сплайсируется несколькими способами с образованием более 40 различных мРНК. [31] Равновесие среди дифференциально сплайсированных транскриптов обеспечивает наличие множества мРНК, кодирующих разные продукты, необходимые для размножения вируса. [32] Один из транскриптов дифференциального сплайсинга содержит ген tat , в котором экзон 2 представляет собой кассетный экзон, который может быть пропущен или включен. Включение tat-экзона 2 в РНК регулируется конкуренцией между репрессором сплайсинга hnRNP A1 и SR-белком SC35. Внутри экзона 2 экзонная последовательность сайленсера сплайсинга (ESS) и экзонная последовательность энхансера сплайсинга (ESE) перекрываются. Если белок-репрессор А1 связывается с ESS, он инициирует совместное связывание нескольких молекул А1, распространяясь на 5'-донорный сайт выше экзона 2 и предотвращая связывание основного фактора сплайсинга U2AF35 с полипиримидиновым трактом. Если SC35 связывается с ESE, он предотвращает связывание A1 и поддерживает 5'-донорный сайт в доступном состоянии для сборки сплайсосомы. Конкуренция между активатором и репрессором обеспечивает продукцию обоих типов мРНК (с экзоном 2 и без него). [31]
значимость Адаптивная
Подлинный альтернативный сплайсинг происходит как в генах, кодирующих белок, так и в некодирующих генах, с образованием множества продуктов (белков или некодирующих РНК). Внешняя информация необходима для того, чтобы решить, какой продукт будет произведен, учитывая последовательность ДНК и исходный транскрипт. Поскольку методы регуляции наследуются, это открывает новые способы воздействия мутаций на экспрессию генов. [33]
Альтернативный сплайсинг может обеспечить эволюционную гибкость. Единственная точечная мутация может привести к тому, что данный экзон будет иногда исключен или включен в транскрипт во время сплайсинга, что позволит производить новую изоформу белка без потери исходного белка. [1] Исследования выявили внутренне неупорядоченные области (см. Внутренне неструктурированные белки ), обогащенные неконститутивными экзонами. [34] предполагая, что изоформы белка могут проявлять функциональное разнообразие из-за изменения функциональных модулей внутри этих областей. Такое функциональное разнообразие, достигаемое изоформами, отражается в моделях их экспрессии и может быть предсказано с помощью подходов машинного обучения. [35] [36] Сравнительные исследования показывают, что альтернативный сплайсинг предшествовал многоклеточному развитию в эволюции, и предполагают, что этот механизм мог быть использован для помощи в развитии многоклеточных организмов. [37]
Исследования, основанные на проекте «Геном человека» и других методах секвенирования генома, показали, что у человека лишь примерно на 30% больше генов, чем у круглого червя Caenorhabditis elegans , и лишь примерно в два раза больше, чем у мухи Drosophila melanogaster . Это открытие привело к предположению, что воспринимаемая большая сложность людей или позвоночных в целом может быть связана с более высокой скоростью альтернативного сплайсинга у людей, чем у беспозвоночных. [38] [39] Однако исследование образцов из 100 000 меток экспрессируемых последовательностей (EST) от человека, мыши, крысы, коровы, мухи ( D. melanogaster ), червя ( C. elegans ) и растения Arabidopsis thaliana не выявило больших различий в частоте альтернативно сплайсированные гены среди людей и любых других протестированных животных. [40] Другое исследование, однако, предположило, что эти результаты являются результатом разного количества EST, доступных для разных организмов. Когда они сравнили частоты альтернативного сплайсинга в случайных подмножествах генов каждого организма, авторы пришли к выводу, что у позвоночных действительно более высокие показатели альтернативного сплайсинга, чем у беспозвоночных. [41]
Болезнь [ править ]
Изменения в механизме процессинга РНК могут привести к неправильному сплайсингу нескольких транскриптов, в то время как однонуклеотидные изменения в сайтах сплайсинга или цис-действующих регуляторных сайтах сплайсинга могут привести к различиям в сплайсинге одного гена и, следовательно, в мРНК, продуцируемой из транскрипты мутантного гена. Исследование 2005 года, включающее вероятностный анализ, показало, что более 60% мутаций , вызывающих заболевания у человека , влияют на сплайсинг, а не непосредственно на кодирующие последовательности. [42] Более недавнее исследование показывает, что треть всех наследственных заболеваний, вероятно, имеют компонент сплайсинга. [20] Независимо от точного процента, существует ряд заболеваний, связанных со сплайсингом. [43] Как описано ниже, ярким примером заболеваний, связанных со сплайсингом, является рак.
Аномально сплайсированные мРНК также обнаруживаются в большом количестве раковых клеток. [44] [45] [46] Комбинированный анализ RNA-Seq и протеомный анализ выявил поразительную дифференциальную экспрессию.сплайсинговых изоформ ключевых белков важных путей развития рака. [47] Не всегда ясно, способствуют ли такие аберрантные модели сплайсинга раковому росту или являются просто следствием клеточных аномалий, связанных с раком. Было показано, что для некоторых типов рака, таких как колоректальный рак и рак простаты, количество ошибок сплайсинга на рак сильно различается в зависимости от конкретного рака; это явление называется нестабильностью транскриптома . [48] [49] Кроме того, было показано, что нестабильность транскриптома коррелирует со сниженным уровнем экспрессии генов факторов сплайсинга. мутация DNMT3A Было продемонстрировано, что способствует гематологическим злокачественным новообразованиям , и что DNMT3A клеточные линии с мутацией демонстрируют нестабильность транскриптома по сравнению с их изогенными аналогами дикого типа. [50]
Фактически, в раковых клетках действительно наблюдается снижение альтернативного сплайсинга по сравнению с нормальными, а типы сплайсинга различаются; например, раковые клетки демонстрируют более высокий уровень задержки интронов, чем нормальные клетки, но более низкий уровень пропуска экзонов. [51] Некоторые различия в сплайсинге раковых клеток могут быть связаны с высокой частотой соматических мутаций в генах факторов сплайсинга. [46] а некоторые могут быть результатом изменений в фосфорилировании транс-действующих факторов сплайсинга. [33] Другие могут быть вызваны изменениями в относительном количестве вырабатываемых факторов сплайсинга; например, было показано, что клетки рака молочной железы имеют повышенный уровень фактора сплайсинга SF2/ASF . [52] Одно исследование показало, что относительно небольшой процент (383 из более чем 26 000) альтернативных вариантов сплайсинга встречался значительно чаще в опухолевых клетках, чем в нормальных клетках, что позволяет предположить, что существует ограниченный набор генов, которые при неправильном сплайсинге способствуют развитию опухоли. разработка. [53] Однако считается, что вредные эффекты неправильного сплайсинга транскриптов обычно защищаются и устраняются с помощью клеточного механизма посттранскрипционного контроля качества, называемого нонсенс-опосредованным распадом мРНК [NMD]. [54]
Одним из примеров специфического варианта сплайсинга, связанного с раком, является один из генов DNMT человека . Три гена DNMT кодируют ферменты, которые добавляют к ДНК метильные группы — модификация, которая часто оказывает регуляторное воздействие. Несколько мРНК DNMT3B с аномальным сплайсингом обнаружены в опухолях и линиях раковых клеток. В двух отдельных исследованиях экспрессия двух из этих аномально сплайсированных мРНК в клетках млекопитающих вызвала изменения в паттернах метилирования ДНК в этих клетках. Клетки с одной из аномальных мРНК также росли в два раза быстрее, чем контрольные клетки, что указывает на прямой вклад этого продукта в развитие опухоли. [33]
Другой пример — Рон ( MST1R ) протоонкоген . Важным свойством раковых клеток является их способность перемещаться и проникать в нормальные ткани. Было обнаружено, что продукция аномально сплайсированного транскрипта Рона связана с повышенными уровнями SF2/ASF в клетках рака молочной железы. Аномальная изоформа белка Ron, кодируемая этой мРНК, приводит к подвижности клеток . [52]
Сверхэкспрессия укороченного сплайсингового варианта гена FOSB – ΔFosB – в определенной популяции нейронов прилежащего ядра была идентифицирована как причинный механизм, участвующий в индукции и поддержании зависимости от наркотиков и естественных вознаграждений . [55] [56] [57] [58]
Недавние провокационные исследования указывают на ключевую функцию структуры хроматина и модификаций гистонов в альтернативной регуляции сплайсинга. Эти открытия позволяют предположить, что эпигенетическая регуляция определяет не только то, какие части генома экспрессируются, но и то, как они соединяются. [59]
всетранскриптомный анализ ) Геномно-масштабный (
Транскриптомный анализ альтернативного сплайсинга обычно выполняется с помощью высокопроизводительного секвенирования РНК. Чаще всего с помощью секвенирования короткого считывания, например, с помощью приборов Illumina. Но еще более информативным является длительное секвенирование, например, с помощью инструментов Nanopore или PacBio. Анализ всего транскриптома может, например, использоваться для измерения количества отклонений альтернативного сплайсинга, например, в когорте рака. [60]
Технологии глубокого секвенирования использовались для проведения полногеномного анализа как необработанных, так и обработанных мРНК; тем самым давая представление об альтернативном сплайсинге. Например, результаты использования глубокого секвенирования показывают, что у людей примерно 95% транскриптов мультиэкзонных генов подвергаются альтернативному сплайсингу, при этом ряд транскриптов пре-мРНК сплайсируются тканеспецифичным образом. [2] Функциональная геномика и вычислительные подходы, основанные на множественном обучении , также были разработаны для интеграции данных секвенирования РНК для прогнозирования функций альтернативно сплайсированных изоформ. [36] Глубокое секвенирование также помогло обнаружить in vivo временные лариаты , которые высвобождаются во время сплайсинга, определить последовательности сайтов ветвления и крупномасштабное картирование точек ветвления в транскриптах пре-мРНК человека. [61]
Исторически альтернативно сплайсированные транскрипты были обнаружены путем сравнения последовательностей EST , но это требует секвенирования очень большого количества EST. Большинство EST-библиотек происходят из очень ограниченного числа тканей, поэтому в любом случае тканеспецифичные варианты сплайсинга, скорее всего, будут пропущены. Однако были разработаны высокопроизводительные подходы к исследованию сплайсинга, такие как анализ на основе микрочипов ДНК , анализы связывания РНК и глубокое секвенирование . Эти методы можно использовать для скрининга полиморфизмов или мутаций внутри или вокруг элементов сплайсинга, которые влияют на связывание белков. В сочетании с анализами сплайсинга, включая in vivo анализы репортерных генов , можно затем проанализировать функциональные эффекты полиморфизмов или мутаций на сплайсинг транскриптов пре-мРНК. [20] [23] [62]
массивы фрагментов ДНК, представляющие отдельные экзоны ( например, Affymetrix экзонный микрочип ) или границы экзонов/экзонов ( например, массивы от ExonHit или Jivan При микрочиповом анализе использовались ). Затем массив исследуют меченой кДНК из представляющих интерес тканей. кДНК зонда связываются с ДНК экзонов, которые включены в мРНК в ткани их происхождения, или с ДНК с границы, где соединяются два экзона. Это может выявить наличие определенных альтернативно сплайсированных мРНК. [63]
CLIP ( сшивка и иммунопреципитация ) использует УФ-излучение для связывания белков с молекулами РНК в ткани во время сплайсинга. Затем интересующий транс-действующий регуляторный белок сплайсинга осаждают с использованием специфических антител. Когда РНК, прикрепленная к этому белку, выделяется и клонируется, она обнаруживает целевые последовательности для этого белка. [64] Другим методом идентификации РНК-связывающих белков и картирования их связывания с транскриптами пре-мРНК является «Оценка геномных аптамеров на микрочипах по сдвигу (MEGAshift)».net [65] Этот метод включает в себя адаптацию метода «Систематической эволюции лигандов путем экспоненциального обогащения (SELEX)». [66] вместе со считыванием на основе микроматрицы. Использование метода MEGAshift позволило лучше понять регуляцию альтернативного сплайсинга, позволив идентифицировать последовательности в транскриптах пре-мРНК, окружающих альтернативно сплайсированные экзоны, которые опосредуют связывание с различными факторами сплайсинга, такими как ASF/SF2 и PTB. [67] Этот подход также использовался для определения взаимосвязи между вторичной структурой РНК и связыванием факторов сплайсинга. [22]
Использование репортерных анализов позволяет найти белки сплайсинга, участвующие в конкретном событии альтернативного сплайсинга, путем конструирования репортерных генов, которые будут экспрессировать один из двух различных флуоресцентных белков в зависимости от происходящей реакции сплайсинга. Этот метод использовался для выделения мутантов, влияющих на сплайсинг, и, таким образом, для идентификации новых белков-регуляторов сплайсинга, инактивированных в этих мутантах. [64]
Недавние достижения в предсказании структуры белков способствовали разработке новых инструментов для аннотации генома и анализа альтернативного сплайсинга. Например, isoform.io, платформа, основанная на предсказаниях структуры белков, оценила сотни тысяч изоформ генов, кодирующих человеческие белки, собранных в результате многочисленных экспериментов по секвенированию РНК в различных тканях человека. Этот всесторонний анализ привел к идентификации многочисленных изоформ с более достоверно предсказанной структурой и потенциально превосходящими функциями по сравнению с каноническими изоформами в последней базе данных генов человека. Интегрируя структурные предсказания с данными экспрессии и эволюции, этот подход продемонстрировал потенциал предсказания структуры белка как инструмента для уточнения аннотации генома человека. [68]
Базы данных [ править ]
Существует коллекция альтернативных баз данных сплайсинга. [69] [70] [71] Эти базы данных полезны для поиска генов, имеющих пре-мРНК, подвергающихся альтернативному сплайсингу и событиям альтернативного сплайсинга, или для изучения функционального воздействия альтернативного сплайсинга.
- АспикДБ База данных
- Интронатора База данных
- ПроСАС База данных
См. также [ править ]
Ссылки [ править ]
- ^ Jump up to: Перейти обратно: а б с д и ж г час Черный ДЛ (2003). «Механизмы альтернативного сплайсинга премессенджерной РНК» (PDF) . Ежегодный обзор биохимии . 72 (1): 291–336. doi : 10.1146/annurev.biochem.72.121801.161720 . ПМИД 12626338 . S2CID 23576288 .
- ^ Jump up to: Перейти обратно: а б с Пан Кью, Шай О, Ли Л.Дж., Фрей Б.Дж., Бленкоу Б.Дж. (декабрь 2008 г.). «Глубокое исследование сложности альтернативного сплайсинга транскриптома человека с помощью высокопроизводительного секвенирования». Природная генетика . 40 (12): 1413–5. дои : 10.1038/ng.259 . ПМИД 18978789 . S2CID 9228930 .
- ^ Бхуян С.А., Ли С., Фан М., Хантингтон Б., Хоган Э., Лю CC, Лю Дж., Павлидис П. (2018). «Систематическая оценка функции изоформы в литературных отчетах об альтернативном сплайсинге» . БМК Геномика . 19 (1): 637. doi : 10.1186/s12864-018-5013-2 . ПМК 6114036 . ПМИД 30153812 . S2CID 52113302 .
- ^ Тресс МЛ, Абаскаль Ф, Валенсия А (2017). «Альтернативный сплайсинг, возможно, не является ключом к сложности протеома» . Тенденции биохимических наук . 42 (2): 98–110. дои : 10.1016/j.tibs.2016.08.008 . ПМК 6526280 . ПМИД 27712956 .
- ^ Чоу Л.Т., Гелинас Р.Э., Брокер Т.Р., Робертс Р.Дж. (сентябрь 1977 г.). «Удивительное расположение последовательностей на 5'-концах информационной РНК аденовируса 2». Клетка . 12 (1): 1–8. дои : 10.1016/0092-8674(77)90180-5 . ПМИД 902310 . S2CID 2099968 .
- ^ Бергет С.М., Мур С., Шарп П.А. (август 1977 г.). «Сплайсированные сегменты на 5'-конце поздней мРНК аденовируса 2» . Труды Национальной академии наук Соединенных Штатов Америки . 74 (8): 3171–5. Бибкод : 1977PNAS...74.3171B . дои : 10.1073/pnas.74.8.3171 . ПМЦ 431482 . ПМИД 269380 .
- ^ Jump up to: Перейти обратно: а б с Лефф С.Е., Розенфельд М.Г., Эванс Р.М. (1986). «Сложные единицы транскрипции: разнообразие экспрессии генов за счет альтернативного процессинга РНК». Ежегодный обзор биохимии . 55 (1): 1091–117. дои : 10.1146/annurev.bi.55.070186.005303 . ПМИД 3017190 .
- ^ Чоу Л.Т., Брокер Т.Р. (октябрь 1978 г.). «Сплайсированные структуры волокна аденовируса 2 и других поздних мРНК». Клетка . 15 (2): 497–510. дои : 10.1016/0092-8674(78)90019-3 . ПМИД 719751 . S2CID 44642349 .
- ^ Невинс-младший, Дарнелл Дж.Э. (декабрь 1978 г.). «Этапы обработки мРНК Ad2: ядерные последовательности поли(А) + консервативны, а добавление поли(А) предшествует сплайсингу». Клетка . 15 (4): 1477–93. дои : 10.1016/0092-8674(78)90071-5 . ПМИД 729004 . S2CID 39704416 .
- ^ Розенфельд М.Г., Амара С.Г., Роос Б.А., Онг Э.С., Эванс Р.М. (март 1981 г.). «Измененная экспрессия гена кальцитонина, связанная с полиморфизмом РНК». Природа . 290 (5801): 63–5. Бибкод : 1981Natur.290...63R . дои : 10.1038/290063a0 . ПМИД 7207587 . S2CID 4318349 .
- ^ Розенфельд М.Г., Лин Ч.Р., Амара С.Г., Столарски Л., Роос Б.А., Онг Э.С., Эванс Р.М. (март 1982 г.). «Полиморфизм мРНК кальцитонина: переключение пептидов, связанное с событиями альтернативного сплайсинга РНК» . Труды Национальной академии наук Соединенных Штатов Америки . 79 (6): 1717–21. Бибкод : 1982PNAS...79.1717R . дои : 10.1073/pnas.79.6.1717 . ПМК 346051 . ПМИД 6952224 .
- ^ Маки Р., Редер В., Траунекер А., Сидман С., Вабл М., Рашке В., Тонегава С. (май 1981 г.). «Роль перестройки ДНК и альтернативного процессинга РНК в экспрессии дельта-генов иммуноглобулина». Клетка . 24 (2): 353–65. дои : 10.1016/0092-8674(81)90325-1 . ПМИД 6786756 . S2CID 13208589 .
- ^ Шмукер Д., Клеменс Дж.К., Шу Х., Уорби К.А., Сяо Дж., Муда М. и др. (июнь 2000 г.). «Drosophila Dscam — это рецептор направления аксонов, обладающий исключительным молекулярным разнообразием» . Клетка . 101 (6): 671–84. дои : 10.1016/S0092-8674(00)80878-8 . ПМИД 10892653 . S2CID 13829976 .
- ^ Jump up to: Перейти обратно: а б Вестергрен Якобссон А., Сегерман Б., Уоллерман О., Линд С.Б., Чжао Х., Рубин С.Дж. и др. (ноябрь 2020 г.). «Транскриптом аденовируса человека типа 2: удивительная сложность альтернативно сплайсированных мРНК» . Журнал вирусологии . 95 (4). дои : 10.1128/JVI.01869-20 . ПМЦ 7851563 . ПМИД 33239457 .
- ^ Jump up to: Перейти обратно: а б с д и Саммет М., Фуассак С., Гиго Р. (август 2008 г.). Брент М.Р. (ред.). «Общее определение и номенклатура альтернативных событий сплайсинга» . PLOS Вычислительная биология . 4 (8): e1000147. Бибкод : 2008PLSCB...4E0147S . дои : 10.1371/journal.pcbi.1000147 . ПМК 2467475 . ПМИД 18688268 .
- ^ Jump up to: Перейти обратно: а б с д и ж г час Мэтлин А.Дж., Кларк Ф., Смит К.В. (май 2005 г.). «Понимание альтернативного сплайсинга: к сотовому коду». Обзоры природы. Молекулярно-клеточная биология . 6 (5): 386–98. дои : 10.1038/nrm1645 . ПМИД 15956978 . S2CID 14883495 .
- ^ Нер-Гаон, Хадас; Халахми, Ронит; Савальди-Гольдштейн, Сигал; Рубин, Эйтан; Офир, Рон; Флюр, Роберт (2004). «Удержание интронов является основным явлением альтернативного сплайсинга у арабидопсиса» . Заводской журнал . 39 (6): 877–885. дои : 10.1111/j.1365-313X.2004.02172.x . ISSN 1365-313X . ПМИД 15341630 .
- ^ Гао К., Масуда А., Мацуура Т., Оно К. (апрель 2008 г.). «Консенсусная последовательность точки ветвления человека — yUnAy» . Исследования нуклеиновых кислот . 36 (7): 2257–67. дои : 10.1093/нар/gkn073 . ПМК 2367711 . ПМИД 18285363 .
- ^ Кларк Д. (2005). Молекулярная биология . Амстердам: Elsevier Academic Press. ISBN 978-0-12-175551-5 .
- ^ Jump up to: Перейти обратно: а б с Лим К.Х., Феррарис Л., Филлу М.Е., Рафаэль Б.Дж., Fairbrother WG (июль 2011 г.). «Использование позиционного распределения для идентификации элементов сплайсинга и прогнозирования дефектов обработки пре-мРНК в генах человека» . Труды Национальной академии наук Соединенных Штатов Америки . 108 (27): 11093–8. Бибкод : 2011PNAS..10811093H . дои : 10.1073/pnas.1101135108 . ПМК 3131313 . ПМИД 21685335 .
- ^ Варф М.Б., Берглунд Дж.А. (март 2010 г.). «Роль структуры РНК в регуляции сплайсинга пре-мРНК» . Тенденции биохимических наук . 35 (3): 169–78. дои : 10.1016/j.tibs.2009.10.004 . ПМЦ 2834840 . ПМИД 19959365 .
- ^ Jump up to: Перейти обратно: а б Рид, округ Колумбия, Чанг Б.Л., Гундерсон С.И., Альперт Л., Томпсон В.А., Фэйрбратер В.Г. (декабрь 2009 г.). «SELEX следующего поколения идентифицирует последовательность и структурные детерминанты связывания фактора сплайсинга в последовательности пре-мРНК человека» . РНК . 15 (12): 2385–97. дои : 10.1261/rna.1821809 . ПМЦ 2779669 . ПМИД 19861426 .
- ^ Jump up to: Перейти обратно: а б с д Ван З, Burge CB (май 2008 г.). «Регулирование сварки: от перечня регулирующих элементов к интегрированному кодексу сварки» (Бесплатный полный текст) . РНК . 14 (5): 802–13. дои : 10.1261/rna.876308 . ПМК 2327353 . ПМИД 18369186 .
- ^ Jump up to: Перейти обратно: а б Бараш Ю., Каларко Дж.А., Гао В., Пан К., Ван Х., Шай О. и др. (май 2010 г.). «Расшифровка кода сплайсинга». Природа . 465 (7294): 53–9. Бибкод : 2010Natur.465...53B . дои : 10.1038/nature09000 . ПМИД 20445623 . S2CID 2398858 .
- ^ Кэ С., Чжан XH, Часин Л.А. (апрель 2008 г.). «Позитивный отбор, действующий на мотивы сплайсинга, отражает компенсаторную эволюцию» . Геномные исследования . 18 (4): 533–43. дои : 10.1101/гр.070268.107 . ПМК 2279241 . ПМИД 18204002 .
- ^ Fairbrother WG, Holste D, Burge CB, Sharp PA (сентябрь 2004 г.). «Проверка экзонных энхансеров сплайсинга на основе однонуклеотидного полиморфизма» . ПЛОС Биология . 2 (9): Е268. дои : 10.1371/journal.pbio.0020268 . ПМК 514884 . ПМИД 15340491 .
- ^ Jump up to: Перейти обратно: а б Линч К.В., Маниатис Т. (август 1996 г.). «Сборка специфических белковых комплексов SR на различных регуляторных элементах энхансера двуполого сплайсинга дрозофилы» . Гены и развитие . 10 (16): 2089–101. дои : 10.1101/гад.16.10.2089 . ПМИД 8769651 .
- ^ Грейвли Б.Р., Хертель К.Дж., Маниатис Т. (июнь 2001 г.). «Роль U2AF35 и U2AF65 в энхансер-зависимом сплайсинге» . РНК . 7 (6): 806–18. дои : 10.1017/S1355838201010317 . ПМК 1370132 . ПМИД 11421359 .
- ^ Филипович Э., Адегбойега П., Санчес Р.Л., Гаталика З. (февраль 2002 г.). «Экспрессия CD95 (Fas) в подвергшейся воздействию солнца коже человека и кожных карциномах» . Рак . 94 (3): 814–9. дои : 10.1002/cncr.10277 . ПМИД 11857317 . S2CID 23772719 .
- ^ Jump up to: Перейти обратно: а б Искьердо Х.М., Майос Н., Боннал С., Мартинес С., Кастело Р., Гиго Р. и др. (август 2005 г.). «Регуляция альтернативного сплайсинга Fas за счет антагонистического воздействия TIA-1 и PTB на определение экзонов» . Молекулярная клетка . 19 (4): 475–84. doi : 10.1016/j.molcel.2005.06.015 . ПМИД 16109372 .
- ^ Jump up to: Перейти обратно: а б Залер А.М., Дамгаард К.К., Кьемс Дж., Капути М. (март 2004 г.). «SC35 и гетерогенные ядерные белки рибонуклеопротеина A/B связываются с расположенным рядом элементом экзонного энхансера сплайсинга/экзонного сайленсера сплайсинга, чтобы регулировать сплайсинг экзона 2 tat ВИЧ-1» . Журнал биологической химии . 279 (11): 10077–84. дои : 10.1074/jbc.M312743200 . ПМИД 14703516 .
- ^ Жакне С., Меро А., Билодо П.С., Дамье Л., Штольцфус С.М., Бранлан С. (ноябрь 2001 г.). «Второй глушитель сплайсинга экзона в составе tat-экзона 2 вируса иммунодефицита человека 1 подавляет сплайсинг мРНК Tat и связывает белок hnRNP H» . Журнал биологической химии . 276 (44): 40464–75. дои : 10.1074/jbc.M104070200 . ПМИД 11526107 .
- ^ Jump up to: Перейти обратно: а б с Факенталь Дж.Д., Годли Л.А. (2008). «Аберрантный сплайсинг РНК и его функциональные последствия в раковых клетках» (полный текст) . Модели и механизмы заболеваний . 1 (1): 37–42. дои : 10.1242/dmm.000331 . ПМК 2561970 . ПМИД 19048051 .
- ^ Ромеро П.Р., Заиди С., Фанг Ю.Ю., Уверский В.Н., Радивожак П., Олдфилд С.Дж. и др. (май 2006 г.). «Альтернативный сплайсинг в сочетании с внутренним нарушением белка обеспечивает увеличение функционального разнообразия в многоклеточных организмах» . Труды Национальной академии наук Соединенных Штатов Америки . 103 (22): 8390–5. Бибкод : 2006PNAS..103.8390R . дои : 10.1073/pnas.0507916103 . ПМЦ 1482503 . ПМИД 16717195 .
- ^ Ли Х.Д., Менон Р., Оменн Г.С., Гуань Ю (август 2014 г.). «Новая эра интеграции геномных данных для анализа функции изоформ сплайсинга» . Тенденции в генетике . 30 (8): 340–7. дои : 10.1016/j.tig.2014.05.005 . ПМЦ 4112133 . ПМИД 24951248 .
- ^ Jump up to: Перейти обратно: а б Экси Р., Ли Х.Д., Менон Р., Вэнь Ю., Оменн Г.С., Крецлер М., Гуань Ю. (ноябрь 2013 г.). «Систематическое дифференцирование функций альтернативно сплайсированных изоформ посредством интеграции данных секвенирования РНК» . PLOS Вычислительная биология . 9 (11): e1003314. Бибкод : 2013PLSCB...9E3314E . дои : 10.1371/journal.pcbi.1003314 . ПМЦ 3820534 . ПМИД 24244129 .
- ^ Иримия М, Руков Дж.Л., Пенни Д., Рой С.В. (октябрь 2007 г.). «Функциональный и эволюционный анализ альтернативно сплайсированных генов согласуется с ранним эукариотическим происхождением альтернативного сплайсинга» . Эволюционная биология BMC . 7 (1): 188. Бибкод : 2007BMCEE...7..188I . дои : 10.1186/1471-2148-7-188 . ПМК 2082043 . ПМИД 17916237 .
- ^ Юинг Б., Грин П. (июнь 2000 г.). «Анализ экспрессируемых меток последовательностей указывает на 35 000 генов человека». Природная генетика . 25 (2): 232–4. дои : 10.1038/76115 . ПМИД 10835644 . S2CID 19165121 .
- ^ Роест Кроллиус Х., Жайон О., Берно А., Дасильва С., Буно Л., Фишер С. и др. (июнь 2000 г.). «Оценка количества генов человека, полученная с помощью полногеномного анализа с использованием последовательности ДНК Tetraodon nigroviridis». Природная генетика . 25 (2): 235–8. дои : 10.1038/76118 . ПМИД 10835645 . S2CID 44052050 .
- ^ Бретт Д., Поспишил Х., Валькарсель Дж., Райх Дж., Борк П. (январь 2002 г.). «Альтернативный сплайсинг и сложность генома». Природная генетика . 30 (1): 29–30. дои : 10.1038/ng803 . ПМИД 11743582 . S2CID 2724843 .
- ^ Ким Э, Маген А, Аст Г (2006). «Различные уровни альтернативного сплайсинга у эукариот» . Исследования нуклеиновых кислот . 35 (1): 125–31. дои : 10.1093/нар/gkl924 . ПМК 1802581 . ПМИД 17158149 .
- ^ Лопес-Бигас Н., Аудит Б., Узунис С., Парра Г., Гиго Р. (март 2005 г.). «Являются ли сплайсинговые мутации наиболее частой причиной наследственных заболеваний?». Письма ФЭБС . 579 (9): 1900–3. дои : 10.1016/j.febslet.2005.02.047 . ПМИД 15792793 . S2CID 30174458 .
- ^ Уорд Эй Джей, Купер Т. А. (январь 2010 г.). «Патобиология сплайсинга» . Журнал патологии . 220 (2): 152–63. дои : 10.1002/путь.2649 . ПМЦ 2855871 . ПМИД 19918805 .
- ^ Скотхайм Р.И., Нис М. (2007). «Альтернативный сплайсинг при раке: шум, функциональный или систематический?». Международный журнал биохимии и клеточной биологии . 39 (7–8): 1432–49. doi : 10.1016/j.biocel.2007.02.016 . ПМИД 17416541 .
- ^ Хэ С, Чжоу Ф, Цзо З, Ченг Х, Чжоу Р (2009). Бауэр Дж.А. (ред.). «Глобальный взгляд на варианты транскриптов, специфичных для рака, посредством субтрактивного анализа всего транскриптома» . ПЛОС ОДИН . 4 (3): e4732. Бибкод : 2009PLoSO...4.4732H . дои : 10.1371/journal.pone.0004732 . ПМЦ 2648985 . ПМИД 19266097 .
- ^ Jump up to: Перейти обратно: а б Свин А., Килпинен С., Руусулехто А., Лоте Р.А., Скотхайм Р.И. (май 2016 г.). «Аберрантный сплайсинг РНК при раке; изменения экспрессии и драйверные мутации генов факторов сплайсинга» . Онкоген . 35 (19): 2413–27. дои : 10.1038/onc.2015.318 . ПМИД 26300000 . S2CID 22943729 .
- ^ Оменн Г.С., Гуань Ю, Менон Р. (июль 2014 г.). «Новый класс кандидатов в белковые биомаркеры рака: дифференциально экспрессируемые варианты сплайсинга ERBB2 (HER2/neu) и ERBB1 (EGFR) в клеточных линиях рака молочной железы» . Журнал протеомики . 107 : 103–12. дои : 10.1016/j.jprot.2014.04.012 . ПМК 4123867 . ПМИД 24802673 .
- ^ Свин А., Йоханнессен Б., Тейшейра М.Р., Лоте Р.А., Скотхайм Р.И. (август 2014 г.). «Нестабильность транскриптома как молекулярная характеристика панрака карцином» . БМК Геномика . 15 (1): 672. дои : 10.1186/1471-2164-15-672 . ПМК 3219073 . ПМИД 25109687 .
- ^ Свен А., Агесен Т.Х., Несбаккен А., Рогнум Т.О., Лоте Р.А., Скотхайм Р.И. (май 2011 г.). «Нестабильность транскриптома при колоректальном раке, выявленная с помощью экзонного микрочипового анализа: связь с уровнями экспрессии факторов сплайсинга и выживаемостью пациентов» . Геномная медицина . 3 (5): 32. дои : 10,1186/гм248 . ПМК 4137096 . ПМИД 21619627 .
- ^ Банасзак Л.Г., Джудис В., Чжао X, Ву З, Гао С., Хосокава К. и др. (март 2018 г.). «Аномальный сплайсинг РНК и нестабильность генома после индукции мутаций DNMT3A путем редактирования гена CRISPR/Cas9» . Клетки крови, молекулы и болезни . 69 : 10–22. дои : 10.1016/j.bcmd.2017.12.002 . ПМК 6728079 . ПМИД 29324392 .
- ^ Ким Э, Горен А, Аст Г (январь 2008 г.). «Взгляд на связь между раком и альтернативным сплайсингом». Тенденции в генетике . 24 (1): 7–10. дои : 10.1016/j.tig.2007.10.001 . ПМИД 18054115 .
- ^ Jump up to: Перейти обратно: а б Гигна С., Джордано С., Шен Х., Бенвенуто Ф., Кастильони Ф., Комольо П.М. и др. (декабрь 2005 г.). «Подвижность клеток контролируется SF2/ASF посредством альтернативного сплайсинга протоонкогена Ron» . Молекулярная клетка . 20 (6): 881–90. doi : 10.1016/j.molcel.2005.10.026 . ПМИД 16364913 .
- ^ Хуэй Л, Чжан Икс, Ву Икс, Линь З, Ван Ц, Ли Ю, Ху Г (апрель 2004 г.). «Идентификация альтернативно сплайсированных вариантов мРНК, связанных с раком, путем полногеномного выравнивания EST» . Онкоген . 23 (17): 3013–23. дои : 10.1038/sj.onc.1207362 . ПМИД 15048092 .
- ^ Данквардт С., Ной-Йилик Г., Терманн Р., Фреде У., Хентце М.В., Кулозик А.Е. (март 2002 г.). «Аномально сплайсированные мРНК бета-глобина: одна точечная мутация генерирует транскрипты, чувствительные и нечувствительные к нонсенс-опосредованному распаду мРНК» . Кровь . 99 (5): 1811–6. дои : 10.1182/blood.V99.5.1811 . ПМИД 11861299 . S2CID 17128174 .
- ^ Нестлер Э.Дж. (декабрь 2013 г.). «Клеточная основа памяти при наркомании» . Диалоги в клинической неврологии . 15 (4): 431–43. doi : 10.31887/DCNS.2013.15.4/enestler . ПМЦ 3898681 . ПМИД 24459410 .
НЕСМОТРЯ НА ЗНАЧИМОСТЬ МНОЖЕСТВЕННЫХ ПСИХОСОЦИАЛЬНЫХ ФАКТОРОВ, НАРКОТИЧЕСКАЯ ЗАВИСИМОСТЬ ПО СВОЕЙ ОСНОВЕ ВКЛЮЧАЕТ БИОЛОГИЧЕСКИЙ ПРОЦЕСС: способность многократного воздействия злоупотребляемого наркотика вызывать изменения в уязвимом мозге, которые приводят к компульсивному поиску и приему наркотиков и потере контроля. над употреблением наркотиков, которые определяют состояние наркомании. ... Большой объем литературы продемонстрировал, что такая индукция ΔFosB в нейронах NAc D1-типа увеличивает чувствительность животного к лекарству, а также к естественным вознаграждениям и способствует самостоятельному приему лекарства, предположительно через процесс положительного подкрепления.
- ^ Раффл Дж.К. (ноябрь 2014 г.). «Молекулярная нейробиология зависимости: о чем вообще (Δ)FosB?». Американский журнал о злоупотреблении наркотиками и алкоголем . 40 (6): 428–37. дои : 10.3109/00952990.2014.933840 . ПМИД 25083822 . S2CID 19157711 .
ΔFosB является важным фактором транскрипции, участвующим в молекулярных и поведенческих путях развития зависимости после неоднократного воздействия наркотиков. Образование ΔFosB во многих областях мозга и молекулярный путь, ведущий к образованию комплексов AP-1, хорошо изучены. Установление функционального назначения ΔFosB позволило продолжить определение некоторых ключевых аспектов его молекулярных каскадов, включая такие эффекторы, как GluR2 (87,88), Cdk5 (93) и NFkB (100). Более того, многие из этих выявленных молекулярных изменений теперь напрямую связаны со структурными, физиологическими и поведенческими изменениями, наблюдаемыми после хронического воздействия наркотиков (60,95,97,102). Новые горизонты исследований молекулярной роли ΔFosB были открыты благодаря эпигенетическим исследованиям, а недавние достижения проиллюстрировали роль ΔFosB, действующего на ДНК и гистоны, действительно как молекулярный переключатель (34). Благодаря нашему лучшему пониманию ΔFosB при зависимости, стало возможным оценить потенциал привыкания современных лекарств (119), а также использовать его в качестве биомаркера для оценки эффективности терапевтических вмешательств (121,122,124). Некоторые из этих предлагаемых мер имеют ограничения (125) или находятся в зачаточном состоянии (75). Однако есть надежда, что некоторые из этих предварительных результатов могут привести к созданию инновационных методов лечения, которые крайне необходимы при зависимости.
- ^ Билински П., Войтыла А., Капка-Скшипчак Л., Хведорович Р., Циранка М., Студзински Т. (2012). «Эпигенетическая регуляция при наркомании». Анналы сельскохозяйственной и экологической медицины . 19 (3): 491–6. ПМИД 23020045 .
По этим причинам ΔFosB считается основным и причинным фактором транскрипции в создании новых нейронных связей в центре вознаграждения, префронтальной коре и других областях лимбической системы. Это отражается на повышенном, стабильном и продолжительном уровне чувствительности к кокаину и другим наркотикам, а также на склонности к рецидивам даже после длительных периодов воздержания. Эти недавно построенные сети функционируют очень эффективно, используя новые пути, как только злоупотребляемые наркотиками продолжают приниматься.
- ^ Олсен CM (декабрь 2011 г.). «Естественные награды, нейропластичность и ненаркотическая зависимость» . Нейрофармакология . 61 (7): 1109–22. doi : 10.1016/j.neuropharm.2011.03.010 . ПМК 3139704 . ПМИД 21459101 .
- ^ Луко Р.Ф., Алло М., Шор И.Е., Корнблихтт А.Р., Мистели Т. (январь 2011 г.). «Эпигенетика альтернативного сплайсинга пре-мРНК» . Клетка . 144 (1): 16–26. дои : 10.1016/j.cell.2010.11.056 . ПМК 3038581 . ПМИД 21215366 .
- ^ Стрёмме Дж. М., Йоханнессен Б., Скотхайм Р.И. (2023 г.). «Отклоняющийся альтернативный сплайсинг как молекулярный подтип микросателлитно-стабильного колоректального рака» . JCO Клиническая информатика рака . 7 (7): e2200159. дои : 10.1200/CCI.22.00159 . hdl : 10852/108838 . ПМИД 36821799 .
- ^ Таггарт А.Дж., Дезимоун А.М., Ши Дж.С., Филлу М.Е., Fairbrother WG (июнь 2012 г.). «Крупномасштабное картирование точек ветвления в транскриптах пре-мРНК человека in vivo» . Структурная и молекулярная биология природы . 19 (7): 719–21. дои : 10.1038/nsmb.2327 . ПМЦ 3465671 . ПМИД 22705790 .
- ^ Fairbrother WG, Yeh RF, Sharp PA, Burge CB (август 2002 г.). «Прогностическая идентификация экзонных энхансеров сплайсинга в генах человека» . Наука . 297 (5583): 1007–13. Бибкод : 2002Sci...297.1007F . дои : 10.1126/science.1073774 . ПМИД 12114529 . S2CID 8689111 .
- ^ Пан К., Шай О., Мискитта С., Чжан В., Зальцман А.Л., Мохаммад Н. и др. (декабрь 2004 г.). «Выявление глобальных регуляторных особенностей альтернативного сплайсинга млекопитающих с использованием платформы количественного микрочипа» . Молекулярная клетка . 16 (6): 929–41. doi : 10.1016/j.molcel.2004.12.004 . ПМИД 15610736 .
- ^ Jump up to: Перейти обратно: а б Дэвид Си Джей, Мэнли Дж. Л. (февраль 2008 г.). «Поиск альтернативных регуляторов сплайсинга: новые подходы открывают путь к коду сплайсинга» . Гены и развитие . 22 (3): 279–85. дои : 10.1101/gad.1643108 . ПМЦ 2731647 . ПМИД 18245441 .
- ^ Уоткинс К.Х., Стюарт А., Фэйрбратер В. (декабрь 2009 г.). «Быстрый высокопроизводительный метод картирования рибонуклеопротеинов (РНП) на пре-мРНК человека» . Журнал визуализированных экспериментов . 34 (34): 1622. дои : 10.3791/1622 . ПМК 3152247 . ПМИД 19956082 .
- ^ Туерк C, Золото L (август 1990 г.). «Систематическая эволюция лигандов путем экспоненциального обогащения: лиганды РНК к ДНК-полимеразе бактериофага Т4». Наука . 249 (4968): 505–10. Бибкод : 1990Sci...249..505T . дои : 10.1126/science.2200121 . ПМИД 2200121 .
- ^ Чанг Б., Левин Дж., Томпсон В.А., Фэйрбратер В.Г. (март 2010 г.). «Высокопроизводительный анализ связывания определяет специфичность связывания ASF/SF2 с альтернативно сплайсированными пре-мРНК человека» . Комбинаторная химия и высокопроизводительный скрининг . 13 (3): 242–52. дои : 10.2174/138620710790980522 . ПМЦ 3427726 . ПМИД 20015017 .
- ^ Соммер, Маркус Дж.; Ча, Суён; Варабьев, Алесь; Ринкон, Наталья; Парк, Сукхван; Минкин, Илья; Пертеа, Михаэла; Штайнеггер, Мартин; Зальцберг, Стивен Л. (15 декабря 2022 г.). «Идентификация изоформ транскриптома человека на основе структуры» . электронная жизнь . 11 : е82556. doi : 10.7554/eLife.82556 . ПМЦ 9812405 . ПМИД 36519529 .
- ^ Тапиал Дж., Ха К.С., Стерн-Вейлер Т., Гор А., Брауншвейг У., Эрмосо-Пулидо А. и др. (октябрь 2017 г.). «Атлас альтернативных профилей сплайсинга и функциональных ассоциаций раскрывает новые регуляторные программы и гены, которые одновременно экспрессируют несколько основных изоформ» . Геномные исследования . 27 (10): 1759–1768. дои : 10.1101/гр.220962.117 . ПМК 5630039 . ПМИД 28855263 .
- ^ Лоуади З., Юань К., Гресс А., Цой О., Калинина О.В., Баумбах Дж. и др. (январь 2021 г.). «DIGGER: изучение функциональной роли альтернативного сплайсинга во взаимодействиях белков» . Исследования нуклеиновых кислот . 49 (Д1): Д309–Д318. дои : 10.1093/nar/gkaa768 . ПМЦ 7778957 . ПМИД 32976589 .
- ^ Родригес Х.М., Майетта П., Эзкурдиа И., Пьетрелли А., Весселинк Дж.Дж., Лопес Г. и др. (январь 2013 г.). «APPRIS: аннотация основных и альтернативных изоформ сплайсинга» . Исследования нуклеиновых кислот . 41 (Проблема с базой данных): D110-7. дои : 10.1093/нар/gks1058 . ПМЦ 3531113 . ПМИД 23161672 .
Внешние ссылки [ править ]
- Общее определение и номенклатура альтернативных событий сплайсинга на SciVee
- AStalavista (инструмент визуализации ландшафта альтернативного сращивания), метод исчерпывающей в вычислительном отношении классификации альтернативных структур сращивания. Архивировано 11 декабря 2009 г. на Wayback Machine.
- IsoPred: предсказанные с помощью вычислений функции изоформ.
- Stamms-lab.net: Исследовательская группа, занимающаяся альтернативными проблемами сплайсинга и неправильным сплайсингом при заболеваниях человека.
- Альтернативный сплайсинг ионных каналов головного мозга, связанный с психическими и неврологическими заболеваниями
- BIPASS: веб-сервисы в альтернативном сплайсинге