Маркеры ДНК, связанные с сайтом рестрикции
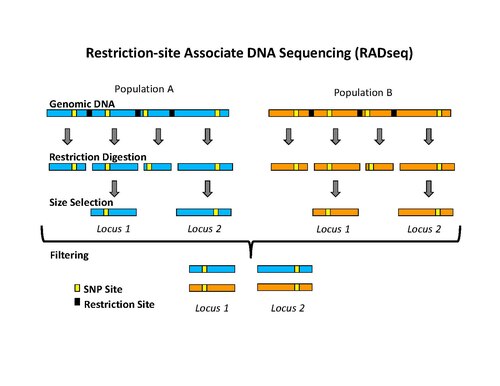
Маркеры ДНК, ассоциированные с сайтом рестрикции (RAD), представляют собой тип генетических маркеров , которые полезны для картирования ассоциаций, QTL-картирования , популяционной генетики , экологической генетики и эволюционной генетики. Использование маркеров RAD для генетического картирования часто называют RAD-картированием. Важным аспектом маркеров и картирования RAD является процесс выделения меток RAD, которые представляют собой последовательности ДНК, которые непосредственно фланкируют каждый экземпляр определенного сайта рестрикции фермента рестрикции по всему геному. [ 1 ] После выделения RAD-меток их можно использовать для идентификации и генотипирования полиморфизмов последовательностей ДНК, главным образом в форме однонуклеотидных полиморфизмов (SNP) . [ 1 ] Полиморфизмы, которые идентифицируются и генотипируются путем выделения и анализа меток RAD, называются маркерами RAD. Хотя генотипирование путем секвенирования представляет собой подход, аналогичный методу RAD-seq, они имеют некоторые существенные различия. [ 2 ] [ 3 ] [ 4 ]
Изоляция тегов RAD
[ редактировать ]Использование фланкирующих последовательностей ДНК вокруг каждого сайта рестрикции является важным аспектом меток RAD. [ 1 ] Плотность RAD-меток в геноме зависит от фермента рестрикции, используемого в процессе выделения. [ 5 ] Существуют и другие методы маркеров сайтов рестрикции, такие как RFLP или полиморфизм длин амплифицированных фрагментов (AFLP), которые используют полиморфизм длин фрагментов, вызванный различными сайтами рестрикции, для различия генетического полиморфизма. Использование фланкирующих последовательностей ДНК в методах RAD-меток называется методом пониженного представления. [ 2 ]
Первоначальная процедура выделения меток RAD включала расщепление ДНК определенным ферментом рестрикции, лигирование биотинилированных адаптеров к выступам, случайное разрезание ДНК на фрагменты, намного меньшие, чем среднее расстояние между сайтами рестрикции, и выделение биотинилированных фрагментов с использованием стрептавидиновых шариков. [ 1 ] Первоначально эта процедура использовалась для выделения меток RAD для анализа на микрочипах . [ 1 ] [ 6 ] [ 7 ] Совсем недавно процедура изоляции тегов RAD была модифицирована для использования с высокопроизводительным секвенированием на платформе Illumina , преимуществом которой является значительное снижение частоты необработанных ошибок и высокая пропускная способность. [ 5 ] Новая процедура включает в себя расщепление ДНК определенным ферментом рестрикции (например: SbfI, NsiI,…), лигирование первого адаптера, называемого P1, к выступам, случайное разрезание ДНК на фрагменты, намного меньшие, чем среднее расстояние между сайтами рестрикции. подготовка разрезанных концов к тупым , лигирование второго адаптера (P2) и использование ПЦР для специфической амплификации фрагментов, содержащих оба адаптера. Важно отметить, что первый адаптер содержит короткий штрих-код последовательности ДНК, называемый MID (молекулярный идентификатор), который используется в качестве маркера для идентификации различных образцов ДНК, которые объединяются вместе и секвенируются в одной реакции. [ 5 ] [ 8 ] Использование высокопроизводительного секвенирования для анализа RAD-тегов можно классифицировать как секвенирование с уменьшенным представлением, которое включает, среди прочего, RADSeq (RAD-Sequencing). [ 2 ]
Обнаружение и генотипирование маркеров RAD
[ редактировать ]После выделения RAD-меток их можно использовать для идентификации и генотипирования полиморфизмов последовательностей ДНК, таких как однонуклеотидные полиморфизмы (SNP). [ 1 ] [ 5 ] Эти полиморфные сайты называются маркерами RAD. Самый эффективный способ найти RAD-метки — это высокопроизводительное секвенирование ДНК . [ 5 ] [ 8 ] называется секвенированием тегов RAD, секвенированием RAD, RAD-Seq или RADSeq.
До разработки технологий высокопроизводительного секвенирования маркеры RAD идентифицировались путем гибридизации меток RAD с микрочипами. [ 1 ] [ 6 ] [ 7 ] Из-за низкой чувствительности микрочипов этот подход может обнаруживать только полиморфизмы последовательностей ДНК, которые нарушают сайты рестрикции и приводят к отсутствию меток RAD, или существенные полиморфизмы последовательностей ДНК, которые нарушают гибридизацию меток RAD. Следовательно, плотность генетических маркеров, которую можно достичь с помощью микрочипов, намного ниже, чем это возможно при высокопроизводительном секвенировании ДНК. [ 9 ]
История
[ редактировать ]Маркеры RAD были сначала реализованы с использованием микрочипов, а затем адаптированы для NGS (секвенирования следующего поколения). [ 9 ] Он был разработан совместно лабораториями Эрика Джонсона и Уильяма Креско в Университете Орегона примерно в 2006 году. Они подтвердили полезность маркеров RAD, определив точки останова рекомбинации у D. melanogaster и обнаружив QTL у трехиглой колюшки. [ 1 ]
ddRADseq
[ редактировать ]В 2012 году был предложен модифицированный метод маркировки RAD, названный двойным дайджестом RADseq (ddRADseq). [ 10 ] [ 11 ] Добавляя второй фермент рестрикции, заменяя случайное срезание и этап жесткого выбора размера ДНК, можно выполнить недорогое популяционное генотипирование. Это может быть особенно мощным инструментом для сканирования всего генома с целью отбора и дифференциации или адаптации популяции. [ 11 ]
В АРЕНДЕ
[ редактировать ]Исследование 2016 года представило новый метод под названием гибридизация RAD (hyRAD). [ 12 ] где биотинилированные фрагменты RAD, покрывающие случайную часть генома, используются в качестве приманок для захвата гомологичных фрагментов из библиотек геномного секвенирования . Фрагменты ДНК сначала генерируются с использованием протокола ddRADseq, применяемого к свежим образцам, и используются в качестве зондов гибридизационного захвата для обогащения библиотек дробовика интересующими фрагментами. Этот простой и экономически эффективный подход позволяет секвенировать ортологичные локусы даже из сильно деградированных образцов ДНК, открывая новые возможности исследований в области музеомики . Еще одним преимуществом метода является то, что он не зависит от наличия сайта рестрикции , что улучшает охват локусов в выборке. Методика была впервые опробована на музейных и свежих образцах Oedaleus Decorus , палеарктического вида кузнечиков, а затем применена у регента-медоеда . [ 13 ] членистоногие , [ 14 ] среди других видов. Был разработан лабораторный протокол для внедрения hyRAD у птиц. [ 15 ]
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Jump up to: а б с д и ж г час Миллер М.Р., Данэм Дж.П., Аморес А., Креско В.А., Джонсон Э.А. (февраль 2007 г.). «Быстрая и экономически эффективная идентификация и генотипирование полиморфизма с использованием маркеров ДНК, связанных с сайтом рестрикции (RAD)» . Геномные исследования . 17 (2): 240–248. дои : 10.1101/гр.5681207 . ПМК 1781356 . ПМИД 17189378 .
- ^ Jump up to: а б с Дэйви Дж.В., Гогенлоэ П.А., Эттер П.Д., Бун Дж.К., Кэтчен Дж.М., Блакстер М.Л. (июнь 2011 г.). «Обнаружение и генотипирование генетических маркеров по всему геному с использованием секвенирования нового поколения». Обзоры природы. Генетика . 12 (7): 499–510. дои : 10.1038/nrg3012 . ПМИД 21681211 . S2CID 15080731 .
- ^ Кэмпбелл Э.О., Брюне Б.М., Дюпюи-младший, Сперлинг Ф.А. (2018). «Может ли RRS под любым другим названием звучать как RAD?» . Методы экологии и эволюции . 9 (9): 1920–1927. дои : 10.1111/2041-210X.13038 .
- ^ Во Ф., Дютуа Л., Фрейзер К.И., Уотерс Дж.М. (2022). «Генотипирование путем секвенирования для биогеографии» . Журнал биогеографии . 50 (2): 262–281. дои : 10.1111/jbi.14516 .
- ^ Jump up to: а б с д и Бэрд Н.А., Эттер П.Д., Этвуд Т.С., Карри М.К., Шивер А.Л., Льюис З.А. и др. (2008). «Быстрое обнаружение SNP и генетическое картирование с использованием секвенированных маркеров RAD» . ПЛОС ОДИН . 3 (10): е3376. Бибкод : 2008PLoSO...3.3376B . дои : 10.1371/journal.pone.0003376 . ПМК 2557064 . ПМИД 18852878 .
- ^ Jump up to: а б Миллер М.Р., Этвуд Т.С., Имс Б.Ф., Эберхарт Дж.К., Ян Ю.Л., Постлтуэйт Дж.Х., Джонсон Э.А. (2007). «Микроматрицы RAD-маркеров позволяют быстро картировать мутации рыбок данио» . Геномная биология . 8 (6): 105 рандов. дои : 10.1186/gb-2007-8-6-r105 . ПМК 2394753 . ПМИД 17553171 .
- ^ Jump up to: а б Льюис З.А., Шивер А.Л., Стифлер Н., Миллер М.Р., Джонсон Э.А., Селкер ЕС (октябрь 2007 г.). «Высокоплотное обнаружение маркеров ДНК, связанных с сайтами рестрикции, для быстрого картирования мутировавших локусов в Neurospora» . Генетика . 177 (2): 1163–1171. doi : 10.1534/genetics.107.078147 . ПМК 2034621 . ПМИД 17660537 .
- ^ Jump up to: а б Гогенлоэ П.А., Башам С., Эттер П.Д., Стифлер Н., Джонсон Э.А., Креско В.А. (февраль 2010 г.). «Популяционная геномика параллельной адаптации трехиглой колюшки с использованием секвенированных меток RAD» . ПЛОС Генетика . 6 (2): e1000862. дои : 10.1371/journal.pgen.1000862 . ПМК 2829049 . ПМИД 20195501 .
- ^ Jump up to: а б Шендуре Дж., Джи Х (октябрь 2008 г.). «Секвенирование ДНК нового поколения». Природная биотехнология . 26 (10): 1135–1145. дои : 10.1038/nbt1486 . ПМИД 18846087 . S2CID 6384349 .
- ^ Петерсон Б.К., Вебер Дж.Н., Кей Э.Х., Фишер Х.С., Хукстра Х.Э. (2012). «Двойной дайджест RADseq: недорогой метод обнаружения и генотипирования SNP de novo у модельных и немодельных видов» . ПЛОС ОДИН . 7 (5): e37135. Бибкод : 2012PLoSO...737135P . дои : 10.1371/journal.pone.0037135 . ПМК 3365034 . ПМИД 22675423 .
- ^ Jump up to: а б Петерсон Б.К., Вебер Дж.Н., Кей Э.Х., Фишер Х.С., Хукстра Х.Э. (2012). «Двойной дайджест RADseq: недорогой метод обнаружения и генотипирования SNP de novo у модельных и немодельных видов» . ПЛОС ОДИН . 7 (5): e37135. Бибкод : 2012PLoSO...737135P . дои : 10.1371/journal.pone.0037135 . ПМК 3365034 . ПМИД 22675423 .
- ^ Сучан Т., Питтелуд С., Герасимова Н.С., Костикова А., Шмид С., Арриго Н. и др. (2016). Орландо Л. (ред.). «Захват гибридизации с использованием зондов RAD (hyRAD), нового инструмента для проведения геномного анализа коллекционных образцов» . ПЛОС ОДИН . 11 (3): e0151651. Бибкод : 2016PLoSO..1151651S . дои : 10.1371/journal.pone.0151651 . ПМК 4801390 . ПМИД 26999359 .
- ^ Кратес Р., Ола Г., Адамски М., Эйткен Н., Бэнкс С., Ингверсен Д. и др. (2019). Расселло М.А. (ред.). «Геномное влияние резкого сокращения популяции кочевых певчих птиц» . ПЛОС ОДИН . 14 (10): e0223953. Бибкод : 2019PLoSO..1423953C . дои : 10.1371/journal.pone.0223953 . ПМК 6812763 . PMID 31647830 .
- ^ Фэрклот, Брант К. (2017). Гилберт, М. (ред.). «Выявление консервативных геномных элементов и разработка универсальных наборов приманок для их обогащения» . Методы экологии и эволюции . 8 (9): 1103–1112. дои : 10.1111/2041-210X.12754 . ISSN 2041-210X . S2CID 51896564 .
- ^ Ола Г., Эйткен Н., Сухан Т. (25 января 2018 г.). «hyRAD для птиц v1» . Протоколы.io . doi : 10.17504/protocols.io.mt2c6qe .