GemIdent

GemIdent — это интерактивная программа распознавания изображений , которая определяет интересующие области на изображениях и фотографиях. Он специально разработан для изображений с небольшим количеством цветов, где интересующие объекты выглядят одинаково с небольшими вариациями. Например, сегментация цветного изображения :
- Апельсины с дерева
- Окрашенные клетки на микроскопических изображениях
GemIdent также включает в себя инструменты анализа данных для исследования пространственных отношений между идентифицированными объектами.
История [ править ]
GemIdent был разработан Адамом Капельнером в Стэнфордском университете с июня 2006 года по январь 2007 года в лаборатории доктора Питера Ли под руководством профессора Сьюзен Холмс. [1] Концепция была вдохновлена данными Kohrt et al. [2] которые проанализировали иммунные профили лимфатических узлов у больных раком молочной железы. Следовательно, GemIdent хорошо работает при идентификации клеток в ткани, окрашенной ИГХ , на изображениях с помощью автоматизированной световой микроскопии , когда ядерное фоновое пятно и мембранное/цитоплазматическое окрашивание четко определены. В 2008 году он был адаптирован для поддержки мультиспектральной визуализации . методов [3]
Методология [ править ]
GemIdent использует контролируемое обучение для автоматического выполнениявыявление областей интереса на изображениях. Следовательно, пользователю приходится проделать значительный объем работы, сначала предоставив соответствующие цвета, затем указав примеры самих объектов или регионов, а также негативов ( обучающего набора создание ).
Когда пользователь нажимает на пиксель, многие оценки генерируются с использованием информации об окружающем цвете посредством генерации атрибута Mahalanobis Ring Score (подробное описание см. в документе JSS). Эти оценки затем используются для создания случайного лесного классификатора машинного обучения, который затем будет классифицировать пиксели в любом заданном изображении.
После классификации могут быть ошибки. Пользователь может вернуться к обучению и указать на конкретные ошибки, а затем переклассифицировать их. Эти итерации обучения-классификации-переобучения-переклассификации (называемые интерактивным повышением ) могут привести к очень точной сегментации.
Недавние заявки [ править ]
В 2010 году Сетиади и др. [4] проанализировали гистологические срезы лимфатических узлов, оценивая пространственную плотность В- и Т-клеток. «Число клеток не отражает весь спектр информации, закодированной в тканях».
Исходный код [ править ]
Java Исходный код теперь открыт под лицензией GPL2 . [5]
Примеры [ править ]

Необработанная фотография (слева), наложенная маска, показывающая результаты классификации пикселей (в центре), и, наконец, фотография отмечена центроидами интересующего объекта — апельсинов (справа).
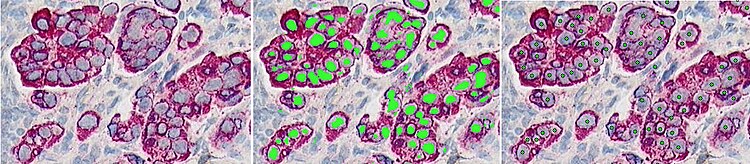
Необработанное микроскопическое изображение окрашенного лимфатического узла (слева) из исследования Корта. [2] наложенная маска, показывающая результаты классификации пикселей (в центре), и, наконец, изображение отмечено центроидами интересующего объекта - ядер рака (справа)

Этот пример иллюстрирует способность GemIdent находить несколько фенотипов на одном изображении: необработанное микроскопическое изображение окрашенного лимфатического узла (вверху слева) из исследования Корта, [2] наложенная маска, показывающая результаты классификации пикселей (вверху справа), и, наконец, изображение, отмеченное центроидами интересующих объектов - ядер рака (в зеленых звездах), Т-клеток (в желтых звездах) и неспецифических фоновые ядра (у голубых звезд).

Интерфейс анализа данных и визуализации командной строки в действии, анализирующий результаты классификации лимфатических узлов из исследования Корта. [2] Гистограмма . отображает распределение расстояний от Т-клеток до соседних раковых клеток Бинарное изображение раковой оболочки является результатом классификации только по пикселям. Открытый PDF- документ представляет собой автоматически созданный отчет об анализе, который включает в себя миниатюрное изображение всего лимфатического узла, подсчеты и коэффициенты ошибок I типа для всех фенотипов , а также расшифровку выполненных анализов.
Ссылки [ править ]
- ^ Капельнер, Адам; Питер П. Ли; Сьюзан Холмс (июль 2007 г.). «Интерактивная статистическая система сегментации и визуализации изображений» . Международная конференция по визуализации медицинской информации – BioMedical Visualization (MediVis 2007) . Компьютерное общество IEEE. стр. 81–86. дои : 10.1109/MEDIVIS.2007.5 . ISBN 978-0-7695-2904-2 . S2CID 16260264 . Архивировано из оригинала 15 апреля 2013 г.
{{cite book}}:|journal=игнорируется ( помогите ) - ^ Jump up to: Перейти обратно: а б с д Корт, Холбрук Э; Навид Нури; Кент Ноуэлс; Дениз Джонсон; Сьюзан Холмс; Питер П. Ли (сентябрь 2005 г.). «Профиль иммунных клеток в подмышечных лимфатических узлах предсказывает безрецидивное выживание при раке молочной железы» . ПЛОС Медицина . 2 (9): е284. doi : 10.1371/journal.pmed.0020284 . ISSN 1549-1676 . ПМК 1198041 . ПМИД 16124834 .
- ^ Холмс, Сьюзен; Адам Капельнер; Питер П. Ли (15 января 2009 г.). «Интерактивная система сегментации статистических изображений Java: GemIdent» . Журнал статистического программного обеспечения . 30 (10): 1–20. дои : 10.18637/jss.v030.i10 . ISSN 1548-7660 . ПМК 3100170 . ПМИД 21614138 .
- ^ Сетиади, Франческа; Нельсон К. Рэй; Холбрук Э. Корт; Адам Капельнер; Валерия Каркамо-Кавасос; Эдина Б. Левич; Сина Ядегариня; Крис М. ван дер Лоос; Эрих Дж. Шварц; Сьюзан Холмс; Питер П. Ли (25 августа 2010 г.). «Количественный, архитектурный анализ подмножеств иммунных клеток в опухолевых лимфатических узлах у больных раком молочной железы и здоровых лимфатических узлах» . ПЛОС ОДИН . 5 (8): е12420. Бибкод : 2010PLoSO...512420S . дои : 10.1371/journal.pone.0012420 . ПМЦ 2928294 . ПМИД 20811638 .
- ^ «Капельнер/ГемИдент» . Гитхаб . Апрель 2019.