чип для иммунопреципитации РНК
RIP-чип (чип иммунопреципитации РНК) — это метод молекулярной биологии , сочетающий иммунопреципитацию РНК с микрочипом . Целью этого метода является определение того, какие последовательности РНК взаимодействуют с конкретным РНК-связывающим белком интересующим in vivo . [1] [2] [3] [4] Его также можно использовать для определения относительных уровней экспрессии генов , для идентификации подмножеств РНК, которые могут совместно регулироваться, или для идентификации РНК, которые могут иметь родственные функции. [4] [5] Этот метод дает представление о посттранскрипционной регуляции генов, которая происходит между РНК и РНК-связывающими белками. [5]
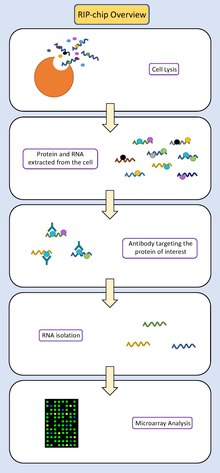
Процедурный обзор [6] [7] [8]
[ редактировать ]- Соберите и лизируйте интересующие клетки .
- Выделите из раствора все фрагменты РНК и связанные с ними белки.
- Иммунопреципитировать интересующий белок. Раствором, содержащим связанные с белком РНК, промывают шарики, конъюгированные с антителами . Эти антитела предназначены для связывания с интересующим белком. Они вытягивают белок (и любые фрагменты РНК, специфически связанные с ним) из раствора, содержащего остальное содержимое клетки.
- Диссоциируйте связанную с белком РНК от комплекса антитело-шарики . Затем используйте центрифугу , чтобы отделить связанную с белком РНК от более тяжелых комплексов антитело-шарики, сохранив связанную с белком РНК и выбросив шарики.
- Диссоциируйте РНК от интересующего белка .
- Изолируйте фрагменты РНК от белка с помощью центрифуги.
- Используйте ПЦР с обратной транскрипцией для преобразования фрагментов РНК в кДНК (ДНК, которая комплементарна фрагментам РНК).
- Флуоресцентно пометьте эти фрагменты кДНК.
- Подготовьте генный чип . Это небольшой чип, к которому в известных местах привязаны последовательности ДНК. Эти последовательности ДНК соответствуют всем известным генам в геноме организма, с которым работает исследователь (или подмножеству генов, которое интересует исследователя). Собранные последовательности кДНК будут комплементарны некоторым из этих последовательностей ДНК, поскольку кДНК представляют собой подмножество РНК, транскрибируемых из генома.
- Позвольте фрагментам кДНК конкурентно гибридизоваться с последовательностями ДНК, связанными с чипом .
- Обнаружение флуоресцентного сигнала от кДНК, связанной с чипом, сообщает исследователям, какие гены на чипе были гибридизированы с кДНК.
Гены, флуоресцентно идентифицированные с помощью чип-анализа, представляют собой гены, чья РНК взаимодействует с исходным интересующим белком. Сила флуоресцентного сигнала конкретного гена может указывать на то, сколько этой конкретной РНК присутствовало в исходном образце, что указывает на уровень экспрессии этого гена.
Разработка и подобные методы
[ редактировать ]Предыдущие методы, направленные на понимание взаимодействий белок-РНК, включали анализы сдвига электрофоретической подвижности РНК и УФ-сшивку с последующей RT-PCR, [9] однако такой селективный анализ нельзя использовать, если связанные РНК еще не известны. [1] Чтобы решить эту проблему, RIP-чип объединяет иммунопреципитацию РНК для выделения молекул РНК, взаимодействующих со специфическими белками, с помощью микрочипа, который может выяснить идентичность РНК, участвующих в этом взаимодействии. [5] [1] Альтернативы RIP-чипу включают:
- RIP-seq : включает секвенирование РНК, которые были извлечены, с использованием высокопроизводительного секвенирования, а не анализ их с помощью микрочипа. [5] Авторы Чжао и др., 2010. [10] объединил процедуру иммунопреципитации РНК с секвенированием РНК . Используя специфические антитела (α-Ezh2), они иммунопреципитировали ядерную РНК, выделенную из мышиных ES- клеток, а затем секвенировали полученную РНК с помощью платформы секвенирования нового поколения Illumina. [10]
- КЛИП : РНК-связывающий белок сшивается с РНК с помощью УФ-излучения перед лизисом, за которым следует фрагментация РНК, иммунопреципитация, промывка высоким содержанием соли, электрофорез в ДСН-ПААГ, мембранный перенос, расщепление протеиназой, подготовка библиотеки кДНК. и секвенирование для идентификации сайтов прямого связывания РНК. [11] CLIP впервые был объединен с высокопроизводительным секвенированием в HITS-CLIP для определения сайтов связывания Nova-РНК в мозге мыши. [12] и в iCLIP , который позволил амплифицировать укороченные кДНК и ввел использование UMI. [13]
- ChIP-on-chip : аналогичный метод, который обнаруживает связывание белков с геномной ДНК, а не с РНК. [14]
Ссылки
[ редактировать ]- ^ Jump up to: а б с Гальярди, Мириам; Матараццо, Мария Р. (2016), Ланцуоло, Кьяра; Бодега, Беатрис (ред.), «RIP: Иммунопреципитация РНК» , Белки группы Polycomb: методы и протоколы , Методы молекулярной биологии, том. 1480, Нью-Йорк, штат Нью-Йорк: Springer, стр. 73–86, doi : 10.1007/978-1-4939-6380-5_7 , ISBN. 978-1-4939-6380-5 , PMID 27659976 , получено 1 декабря 2020 г.
- ^ Таунли-Тилсон, WH Дэвин; Пендерграсс, Сара А.; Марзлафф, Уильям Ф.; Уитфилд, Майкл Л. (1 октября 2006 г.). «Полногеномный анализ мРНК, связанных с белком, связывающим стебель-петля гистонов» . РНК . 12 (10): 1853–1867. дои : 10.1261/rna.76006 . ISSN 1355-8382 . ПМК 1581977 . ПМИД 16931877 .
- ^ Халил, Ахмад М.; Гуттман, Митчелл; Уарте, Майте; Гарбер, Мануэль; Радж, Арджун; Моралес, Дианали Ривеа; Томас, Келли; Прессер, Авива; Бернштейн, Брэдли Э.; Ауденарден, Александр ван; Регев, Авив (14 июля 2009 г.). «Многие большие межгенные некодирующие РНК человека связываются с комплексами, модифицирующими хроматин, и влияют на экспрессию генов» . Труды Национальной академии наук . 106 (28): 11667–11672. Бибкод : 2009PNAS..10611667K . дои : 10.1073/pnas.0904715106 . ISSN 0027-8424 . ПМК 2704857 . ПМИД 19571010 .
- ^ Jump up to: а б Хендриксон, Дэвид Г.; Хоган, Дэниел Дж.; Хершлаг, Даниэль; Феррелл, Джеймс Э.; Браун, Патрик О. (07 мая 2008 г.). «Систематическая идентификация мРНК, рекрутированных в Argonaute 2 с помощью специфических микроРНК, и соответствующие изменения в количестве транскриптов» . ПЛОС ОДИН . 3 (5): е2126. Бибкод : 2008PLoSO...3.2126H . дои : 10.1371/journal.pone.0002126 . ISSN 1932-6203 . ПМК 2330160 . ПМИД 18461144 .
- ^ Jump up to: а б с д Джаясилан, Сабаринатх; Дойл, Фрэнсис; Тененбаум, Скотт А. (01 мая 2014 г.). «Профилирование посттранскрипционных сетевых подмножеств мРНК с использованием RIP-Chip и RIP-Seq» . Методы . Геномные подходы к изучению транскрипционных и посттранскрипционных процессов. 67 (1): 13–19. дои : 10.1016/j.ymeth.2013.11.001 . ISSN 1046-2023 . ПМК 4004666 . ПМИД 24257445 .
- ^ Барони, Тимоти Э.; Читтур, Шридар В.; Джордж, Аджиш Д.; Тененбаум, Скотт А. (2008), Вилуш, Джеффри (ред.), «Достижения в анализе RIP-чипов: профилирование иммунопреципитации и микрочипов РНК-связывающего белка» , Посттранскрипционная регуляция генов , Методы молекулярной биологии, том. 419, Тотова, Нью-Джерси: Humana Press, стр. 93–108, doi : 10.1007/978-1-59745-033-1_6 , ISBN. 978-1-59745-033-1 , PMID 18369977 , получено 1 декабря 2020 г.
- ^ Конрад, Николас К. (01 января 2008 г.), «Глава 15 - Методы совместной иммунопреципитации для оценки взаимодействий РНК и белка in vivo», в Макват, Линн Э.; Киледжян, Мегердич (ред.), «Обмен РНК у эукариот: анализ специализированных путей распада РНК и контроля качества» , «Методы в энзимологии», том. 449, Academic Press, стр. 317–342, номер документа : 10.1016/S0076-6879(08)02415-4 , ISBN. 9780123745842 , PMID 19215765 , получено 1 декабря 2020 г.
- ^ Ниранджанакумари, Сомаше; Ласда, Эрика; Бразас, Роберт; Гарсиа-Бланко, Мариано А. (1 февраля 2002 г.). «Обратимое сшивание в сочетании с иммунопреципитацией для изучения взаимодействий РНК и белка in vivo» . Методы . 26 (2): 182–190. дои : 10.1016/S1046-2023(02)00021-X . ISSN 1046-2023 . ПМИД 12054895 .
- ^ Томсон, Америка; Роджерс, Дж.; Уокер, Се; Стейтон, Дж. М.; Лидман, Пи Джей (1 ноября 1999 г.). «Оптимизированные анализы сдвига РНК в геле и перекрестного связывания УФ-излучением для характеристики взаимодействий цитоплазматической РНК-белка» . БиоТехники . 27 (5): 1032–1042. дои : 10.2144/99275rr03 . ISSN 0736-6205 . ПМИД 10572651 .
- ^ Jump up to: а б Чжао, Цзин; Осуми, Тоширо К.; Кунг, Джонни Т.; Огава, Юя; Грау, Дэниел Дж.; Сарма, Кавита; Сон, Джи Джун; Кингстон, Роберт Э.; Боровский, Марк; Ли, Джинни Т. (декабрь 2010 г.). «Полногеномная идентификация РНК, связанных с Polycomb, с помощью RIP-seq» . Молекулярная клетка . 40 (6): 939–953. doi : 10.1016/j.molcel.2010.12.011 . ISSN 1097-2765 . ПМК 3021903 . ПМИД 21172659 .
- ^ Уле, Джерней; Дженсен, Кирк Б.; Руджиу, Маттео; Меле, Альдо; Уле, Альяз; Дарнелл, Роберт Б. (14 ноября 2003 г.). «CLIP идентифицирует сети РНК, регулируемые Nova, в мозге» . Наука . 302 (5648): 1212–1215. Бибкод : 2003Sci...302.1212U . дои : 10.1126/science.1090095 . ISSN 1095-9203 . ПМИД 14615540 . S2CID 23420615 .
- ^ Ликаталоси, Донни Д.; Меле, Альдо; Фак, Джон Дж.; Уле, Джерней; Кайикчи, Мелис; Чи, Сон Ук; Кларк, Тайсон А.; Швейцер, Энтони К.; Блюм, Джон Э.; Ван, Сюнин; Дарнелл, Дженнифер С. (ноябрь 2008 г.). «HITS-CLIP дает общегеномное представление об альтернативной обработке РНК в мозге» . Природа . 456 (7221): 464–469. Бибкод : 2008Natur.456..464L . дои : 10.1038/nature07488 . ISSN 1476-4687 . ПМК 2597294 . ПМИД 18978773 .
- ^ Кениг, Джулиан; Зарнак, Кэти; Рот, Грегор; Курк, Томаз; Кайикчи, Мелис; Жупан, Блаз; Тернер, Дэниел Дж.; Ласкомб, Николас М.; Уле, Джерней (июль 2010 г.). «iCLIP раскрывает функцию частиц hnRNP при сплайсинге с разрешением отдельных нуклеотидов» . Структурная и молекулярная биология природы . 17 (7): 909–915. дои : 10.1038/nsmb.1838 . ISSN 1545-9985 . ПМК 3000544 . ПМИД 20601959 .
- ^ Ансорж, Вильгельм Дж. (01 января 2010 г.), Патринос, Джордж П.; Ансорж, Вильгельм Дж. (ред.), «Глава 24 - Новые методы секвенирования ДНК следующего поколения для сверхвысокопроизводительных приложений в биомедицине» , Молекулярная диагностика (второе издание) , Сан-Диего: Academic Press, стр. 365– 378, номер домена : 10.1016/b978-0-12-374537-8.00024-9 , ISBN 978-0-12-374537-8 , получено 1 декабря 2020 г.