ЧИА-ПЭТ
Анализ взаимодействия хроматина с помощью секвенирования парных концевых меток ( ChIA-PET или ChIA-PETS ) — это метод, который включает в себя обогащение на основе иммунопреципитации хроматина (ChIP), бесконтактное лигирование хроматина , метки с парными концами и высокопроизводительное секвенирование для определения de novo. дальнодействующие взаимодействия хроматина по всему геному. [1]
Гены могут регулироваться областями, удаленными от промотора, такими как регуляторные элементы, инсуляторы и пограничные элементы, а также сайты связывания транскрипционных факторов (TFBS) . Выявление взаимодействия между регуляторными областями и областями кодирования генов важно для понимания механизмов, управляющих регуляцией генов в здоровом состоянии и при заболеваниях (Maston et al., 2006). ChIA-PET можно использовать для идентификации уникальных функциональных хроматина взаимодействий между дистальными и проксимальными сайтами связывания регуляторных транскрипционных факторов и промоторами генов, с которыми они взаимодействуют.
ChIA-PET также можно использовать для раскрытия механизмов контроля генома во время таких процессов, как клеток дифференцировка , пролиферация и развитие . Создавая интерактома карты ChIA-PET для ДНК-связывающих регуляторных белков и промоторных областей, мы можем лучше идентифицировать уникальные цели для терапевтического вмешательства (Fullwood & Yijun, 2009).
Методология
[ редактировать ]Метод ЧИА-ПЭТ сочетает в себе методы на основе ЧИП, [2] и методы, основанные на захвате конформации хромосомы (3C), [3] расширить возможности обоих подходов. ChIP-секвенирование (ChIP-Seq) — популярный метод, используемый для идентификации TFBS, в то время как 3C используется для идентификации дальних взаимодействий хроматина. Независимо друг от друга оба страдают от ограничений в выявлении дальних взаимодействий de novo в масштабах всего генома. Хотя ChIP-Seq способен идентифицировать TFBS по всему геному, [4] [5] он предоставляет только линейную информацию о сайтах связывания белков вдоль хромосом (но не о взаимодействиях между ними) и может страдать от высокого геномного фонового шума (ложноположительных результатов). Хотя 3C способен анализировать нелинейные, дальнодействующие взаимодействия хроматина, его нельзя использовать в масштабах всего генома, и, как и ChIP-Seq, он также страдает от высокого уровня фонового шума. Поскольку шум увеличивается по мере увеличения расстояния между взаимодействующими областями (максимум 100 КБ), для точной характеристики взаимодействий хроматина требуются трудоемкие и утомительные меры контроля. [6] В отличие от 3C, который представляет собой метод профилирования локус-специфического взаимодействия, альтернативные методы, такие как Hi-C, для профилирования взаимодействий по всему геному. были созданы [7] Несмотря на методы профилирования всего генома как для TFBS, так и для дальнодействующих взаимодействий, сочетание подходов с методом ChIA-PET позволяет идентифицировать области генома, с которыми связан интересующий белок, а также область генома, с которой он взаимодействует. [8] [9]
Метод ChIA-PET успешно решает проблемы неспецифического взаимодействия шума, обнаруженные в ChIP-Seq, путем обработки ультразвуком фрагментов ChIP с целью отделения случайных вложений от конкретных комплексов взаимодействия. Следующий шаг, который называется обогащением, снижает сложность полногеномного анализа и добавляет специфичности взаимодействиям хроматина, связанным с заранее определенными ТФ (факторами транскрипции). Способность подходов 3C идентифицировать дальнодействующие взаимодействия основана на теории бесконтактного лигирования. Что касается взаимного лигирования ДНК, фрагменты, связанные общими белковыми комплексами, имеют большие кинетические преимущества в разбавленных условиях, чем фрагменты, свободно диффундирующие в растворе или закрепленные в различных комплексах. ChIA-PET использует эту концепцию путем включения линкерных последовательностей на свободные концы фрагментов ДНК, связанных с белковыми комплексами. Чтобы построить связность фрагментов, связанных регуляторными комплексами, линкерные последовательности лигируют во время лигирования по близости ядер. Таким образом, продукты лигирования, соединенного с линкером, можно анализировать с помощью сверхвысокопроизводительного секвенирования ПЭТ и сопоставлять с эталонный геном . Поскольку ChIA-PET не зависит от конкретных сайтов для обнаружения, как 3C и 4C, он позволяет беспристрастно, по всему геному обнаруживать de novo взаимодействия хроматина. [8] По сравнению с Hi-C использование антитела ограничивает количество секвенируемых фрагментов взаимодействиями хроматина, связанными с интересующим белком, что также может облегчить анализ данных.
Рабочий процесс
[ редактировать ]Мокрая лабораторная часть рабочего процесса:



- Формальдегид используется для сшивания комплексов ДНК-белок. Обработка ультразвуком используется для разрушения хроматина, а также для уменьшения неспецифических взаимодействий.
- Специфическое антитело выбора используется для обогащения фрагментов хроматина, связанных с интересующим белком. Материал ChIP, связанный антителом, используется для создания ChIA-PET.
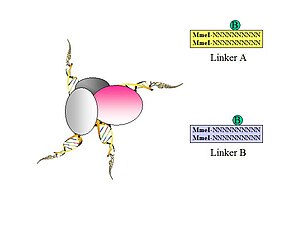
- Рисунок 1. Биотинилированные олигонуклеотидные полулинкеры, содержащие фланкирующие сайты MmeI, используются для соединения фрагментов ДНК, лигированных по близости. Создаются два разных линкера (A и B) со специфическими нуклеотидными штрих-кодами (CG или AT) для каждой из двух последовательностей линкера (это позволит идентифицировать химерный продукт лигирования, как описано на рисунке 5).
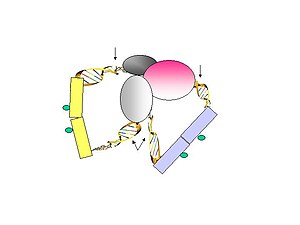
- Рисунок 2. Линкеры лигированы к привязанным фрагментам ДНК.
- Рисунок 3. Фрагменты линкера лигируются на шариках ChIP в разбавленных условиях. Очищенная ДНК затем переваривается MmeI, который разрезает ее на расстоянии от сайта узнавания, высвобождая структуру тег-линкер-метка.
- Рисунок 4. Биотинилированные ПЭТ затем иммобилизуют на магнитных шариках, конъюгированных со стрептавидином.
- Рисунок 5. Последовательности ПЭТ со штрих-кодом линкера AA (CG/CG) и BB (AT/AT) считаются возможными продуктами внутрикомплексного лигирования, тогда как последовательности ПЭТ с составом линкера AB (CG/AT) считаются образуются из химерных продуктов лигирования фрагментов ДНК, связанных в различные хроматиновые комплексы.
Часть рабочего процесса в сухой лаборатории:
ПЭТ-извлечение, картирование и статистический анализ
ПЭТ-метки извлекаются и сопоставляются с эталонным геномом человека in silico .
Идентификация пиков, обогащенных ChIP (сайтов связывания)
Самолигированные ПЭТ используются для идентификации сайтов, обогащенных ChIP, поскольку они обеспечивают наиболее надежное картирование (20 + 20 бит/с) эталонного генома.
Алгоритм поиска пиков обогащения ChIP
Вызванный пик считается сайтом связывания, если имеется несколько перекрывающихся самолигированных ПЭТ. Частота ложных обнаружений (FDR) определяется с помощью статистического моделирования для оценки случайного фона перекрытий виртуальных ДНК, полученных с помощью ПЭТ, и предполагаемого фонового шума.
Фильтрация повторяющейся ДНК (влияет на неспецифическое связывание)
Области-сателлиты и сайты связывания, присутствующие в регионах с серьезными структурными изменениями, удаляются.
счетчик обогащения чипа
Количество самолигированных и интерлигирующих ПЭТ (в пределах окна +250 п.н.) сообщается на каждом сайте. Общее количество самолигированных и интерлигированных ПЭТ в определенном месте называется количеством обогащения ChIP.
Рисунок 6. Классификация ПЭТ. Уникально выровненные последовательности ПЭТ можно классифицировать по тому, происходят ли они из одного фрагмента ДНК или из двух фрагментов ДНК.

- Самолигирующие ПЭТ
Если две метки ПЭТ картированы на одной и той же хромосоме с геномным диапазоном в диапазоне фрагментов ДНК ChIP (менее 3 т.п.н.), с ожидаемой ориентацией самолигирования и на одной и той же цепи, их считают полученными из самолигирование одного фрагмента ДНК ChIP и считается самолигированием ПЭТ.
- ПЭТ с интерлигацией
Если ПЭТ не соответствует этим критериям, то ПЭТ, скорее всего, является результатом лигирования двух фрагментов ДНК и называется ПЭТ между лигированием. Две метки ПЭТ с перевязкой не имеют фиксированной ориентации меток, могут не находиться на одних и тех же цепях, могут иметь какой-либо геномный диапазон и могут не соответствовать одной и той же хромосоме.
- ПЭТ с внутрихромосомными перевязками
Если две метки ПЭТ с перевязкой картированы в одной и той же хромосоме, но с размером > 3 КБ в любой ориентации, то эти ПЭТ называются ПЭТ с внутрихромосомными перевязками.
- ПЭТ с межхромосомными перевязками
ПЭТ, которые картированы на разных хромосомах, называются ПЭТ с межхромосомными перевязками.
Рисунок 7. Предлагаемый механизм, показывающий, как дистальные регуляторные элементы могут инициировать дальнодействующие взаимодействия хроматина с участием промоторных областей генов-мишеней.

Взаимодействия образуют структуры петель ДНК с множеством TFBS в центре закрепления. Маленькие петли могут упаковывать гены вблизи центра закрепления в плотный субкомпартмент, что может увеличивать локальную концентрацию регуляторных белков для усиления активации транскрипции. Этот механизм может также повысить эффективность транскрипции, позволяя РНК pol II циклически вращать плотные кольцевые матрицы генов. Большие петли взаимодействия с большей вероятностью связывают вместе удаленные гены на обоих концах петли, расположенные вблизи якорных сайтов, для скоординированной регуляции, или могут разделять гены в длинные петли, чтобы предотвратить их активацию. Адаптировано из Fullwood et al. (2009).
Сильные и слабые стороны
[ редактировать ]Преимущества метода ЧИА-ПЭТ
- ChIA-PET потенциально может стать беспристрастным полногеномным подходом de novo для анализа взаимодействия хроматина на больших расстояниях (Fullwood & Yijun, 2009).
- Эксперимент ChIA-PET способен предоставить два глобальных набора данных: сайты связывания белковых факторов (самолигированные ПЭТ); и взаимодействия между сайтами связывания (взаимосвязанными ПЭТ).
- ChIA-PET включает ChIP, чтобы упростить полногеномный анализ, и добавляет специфичности взаимодействиям хроматина, связанным с конкретными интересующими факторами.
- ChIA-PET совместим с подходами секвенирования нового поколения на основе меток, такими как пиросеквенирование Roche 454, Illumina GA, ABI SOLiD и Helicos.
- ChIA-PET применим ко многим различным белковым факторам, участвующим в регуляции транскрипции или структурной конформации хроматина.
- Анализ ChIA-PET можно применять к взаимодействиям хроматина, участвующим в определенном ядерном процессе. Используя общие ТФ, такие как РНК-полимераза II, можно будет идентифицировать все взаимодействия хроматина, участвующие в регуляции транскрипции. Кроме того, использование белковых факторов, участвующих в репликации ДНК или структуре хроматина, позволит идентифицировать все взаимодействия, обусловленные репликацией ДНК и структурной модификацией хроматина (Fullwood et al., 2009).
Слабые стороны
- Хорошо известно, что цис- и трансрегуляторные комплексы содержат уникальные комбинации белков, зависящие от клеточных и тканеспецифичных условий (Dekker et al., 2006). Хотя идентификация одиночного функционального TFBS является значительным достижением, использование ChIA-PET для идентификации отдельных белков в комплексе потребует догадок и множества экспериментов для идентификации каждого взаимодействующего белка. Это будет дорогостоящим и трудоемким мероприятием.
- ChIA-PET ограничен качеством, чистотой и специфичностью используемых антител (Fullwood et al., 2009).
- ChIA-PET зависит от идентификации последовательностей, которые могут быть сопоставлены с эталонной последовательностью (ссылка).
- ChIA-PET требует использования компьютерных алгоритмов пикового вызова для организации и сопоставления чтений ПЭТ с эталонным геномом. Из-за различий между программными платформами результаты могут различаться в зависимости от того, какая программа используется.
- Хотя повторяющиеся участки ДНК могут быть связаны с регуляцией генов (Polak & Domany, 2006), их необходимо удалить, поскольку они могут повлиять на данные (Fullwood et al., 2009).
- Обогащение из двух мест одновременно вносит предвзятость в отношении соседних регионов. [10]
История
[ редактировать ]Фуллвуд и др. (2009) использовали ChIA-PET для обнаружения и картирования сети взаимодействия хроматина, опосредованной рецептором эстрогена альфа (ER-альфа) в раковых клетках человека. Полученная в результате глобальная карта интерактома хроматина показала, что удаленные сайты связывания ER-альфа также были прикреплены к промоторам генов посредством дальнодействующих взаимодействий с хроматином, что позволяет предположить, что ER-альфа функционирует за счет обширного образования петель в хроматине, чтобы объединить гены для скоординированной регуляции транскрипции.
Анализ и программное обеспечение
[ редактировать ]| Программное обеспечение | Описание |
|---|---|
| C3PET | Программный комплекс для обработки данных ChIA-PET. Использует непараметрический байесовский подход для прогнозирования белковых комплексов, взаимодействующих с хроматином. [1] [2] |
| Инструмент ЧИА-ПЭТ | Программный комплекс для обработки данных ChIA-PET. [3] [4] |
| ЧИА-ПЭТ2 | Программный комплекс для обработки данных ChIA-PET. Поддерживает данные из различных протоколов и обеспечивает контроль качества анализа данных. [5] [6] |
| ЧИА-Сиг | Программный пакет для обработки данных ChIA-PET с использованием модели NCHG. Веб-сайт ЧИА-Сиг [7] [8] |
| ЭЛАНД | Сопоставляет фрагменты ДНК, обогащенные ChIP, с эталонным геномом человека. [9] |
| манго | Программный комплекс для обработки данных ChIA-PET. Выполняет все необходимые этапы обработки наборов данных ChIA-PET и предоставляет статистические оценки достоверности взаимодействий. [10] [11] |
| Моделирование Монте-Карло | Используется для оценки частоты ложных обнаружений. [11] |
| Геномные взаимодействия | Пакет R для обработки данных ChIA-PET или Hi-C. [12] [13] |
| ДАВАТЬ | Библиотека программирования для создания собственного браузера генома, совместимого с данными ChIA-PET или Hi-C. [14] [15] |
| Повтор Маскер | Маскировка повторяющихся элементов in-silico. [16] |
Альтернативы
[ редактировать ]
Оригинальный метод ChIP представляет собой технологию на основе антител, которая избирательно идентифицирует и связывает белки, чтобы предоставить информацию о состояниях хроматина и транскрипции генов . [17]
Этот метод устраняет ряд недостатков, связанных с методами на основе 3C, путем сбора трехмерных близостей между любым количеством геномных локусов . [12]
Распознавание взаимодействий с разделенным пулом по расширению тегов (SPRITE)
[ редактировать ]SPRITE — это метод картирования взаимодействий высшего порядка в ядре по всему геному. Этот подход обнаруживает взаимодействия, которые происходят на больших пространственных расстояниях, и позволяет обнаруживать по всему геному многочисленные взаимодействия РНК и ДНК , которые происходят одновременно. [13]
ЧИА-Дроп
[ редактировать ]ChIA-Drop — это простой метод анализа мультиплексных взаимодействий хроматина с использованием капельного секвенирования и секвенирования со штрих-кодом с точностью до одной молекулы. Предыдущие подходы на парном уровне популяции, такие как Hi-C и ChIA-PET, отличаются от этой технологии. [14] [15]
Ссылки
[ редактировать ]- ^ Фуллвуд, Мелисса Дж.; Жуань, Ицзюнь (01 мая 2009 г.). «Методы идентификации дальнодействующих хроматиновых взаимодействий на основе ChIP» . Журнал клеточной биохимии . 107 (1): 30–39. дои : 10.1002/jcb.22116 . ISSN 1097-4644 . ПМЦ 2748757 . ПМИД 19247990 .
- ^ Куо, Мин-Хао; Эллис, К.Дэвид (ноябрь 1999 г.). «Сшивание in vivo и иммунопреципитация для изучения динамических ассоциаций белок: ДНК в среде хроматина» . Методы . 19 (3): 425–433. дои : 10.1006/meth.1999.0879 . ПМИД 10579938 .
- ^ Деккер, Иов; Риппе, Карстен; Деккер, Мартин; Клекнер, Нэнси (15 февраля 2002 г.). «Захват конформации хромосомы» . Наука . 295 (5558): 1306–1311. Бибкод : 2002Sci...295.1306D . дои : 10.1126/science.1067799 . ISSN 0036-8075 . ПМИД 11847345 . S2CID 3561891 .
- ^ Барский, Артем; Кудапа, Суреш; Цуй, Кайронг; Ро, Тэ Ён; Шонес, Дастин Э.; Ван, Жибин; Вэй, Банда; Чепелев, Юрий; Чжао, Кеджи (май 2007 г.). «Профилирование метилирования гистонов в геноме человека с высоким разрешением» . Клетка . 129 (4): 823–837. дои : 10.1016/j.cell.2007.05.009 . ПМИД 17512414 . S2CID 6326093 .
- ^ Вэй, Цзя-Линь; Ву, Цян; Вега, Винсенсиус Б.; Чиу, Го Пин; Нг, Патрик; Чжан, Тао; Шахаб, Атиф; Ён, Хау Чунг; Фу, Ютао; Вэн, Чжипин; Лю, Цзянь Цзюнь (январь 2006 г.). «Глобальная карта сайтов связывания транскрипционных факторов p53 в геноме человека» . Клетка . 124 (1): 207–219. дои : 10.1016/j.cell.2005.10.043 . ПМИД 16413492 . S2CID 15698887 .
- ^ Деккер, Иов (январь 2006 г.). «Три буквы «С» захвата конформации хромосом: контроль, контроль, контроль» . Природные методы . 3 (1): 17–21. дои : 10.1038/nmeth823 . ISSN 1548-7091 . ПМИД 16369547 . S2CID 25821556 .
- ^ Либерман-Айден, Эрез; ван Беркум, Нинка Л.; Уильямс, Луиза; Имакаев Максим; Рагочи, Тобиас; Рассказываю, Агнес; Амит, Идо; Ладжуа, Брайан Р.; Сабо, Питер Дж.; Доршнер, Майкл О.; Сандстрем, Ричард (9 октября 2009 г.). «Комплексное картирование долгосрочных взаимодействий раскрывает принципы свертывания человеческого генома» . Наука . 326 (5950): 289–293. Бибкод : 2009Sci...326..289L . дои : 10.1126/science.1181369 . ISSN 0036-8075 . ПМЦ 2858594 . ПМИД 19815776 .
- ^ Jump up to: а б Фуллвуд, Мелисса Дж.; Лю, Мэй Хуэй; Пан, Ю Фу; Лю, Цзюнь; Сюй, Хань; Мохамед, Юсофф Бин; Орлов Юрий Л.; Велков, Стоян; Эй, Андреа; Мэй, По Хуай; Чу, Элейн Джи (ноябрь 2009 г.). «Интерактивный хроматин человека, связанный с эстрогеновым рецептором-α» . Природа . 462 (7269): 58–64. Бибкод : 2009Natur.462...58F . дои : 10.1038/nature08497 . ISSN 0028-0836 . ПМЦ 2774924 . ПМИД 19890323 .
- ^ Ли, Голян; Жуань, Сяоань; Ауэрбах, Раймонд К.; Сандху, Кулджит Сингх; Чжэн, Мэйчжэнь; Ван, Пин; Пох, Хуай Мэй; Гох, Юфен; Лим, Джоанн; Чжан, Цзинъяо; Сим, Хуэй Шань (20 января 2012 г.). «Обширные промотор-центрированные взаимодействия хроматина обеспечивают топологическую основу регуляции транскрипции» . Клетка . 148 (1): 84–98. дои : 10.1016/j.cell.2011.12.014 . ISSN 0092-8674 . ПМК 3339270 . ПМИД 22265404 .
- ^ Даунс, Дэмиен Дж.; Бигри, Роберт А.; Госден, Мэтью Э.; Телениус, Елена; Карпентер, Стефани Дж.; Нуссбаум, Леа; Де Орнеллас, Сара; Сержант Мартин; Эйсбоутс, Крис К.; Швессингер, Рон; Керри, Джон (22 января 2021 г.). «Целевой 3C-опрос организации цис-регуляторных элементов с высоким разрешением в полногеномном масштабе» . Природные коммуникации . 12 (1): 531. Бибкод : 2021NatCo..12..531D . дои : 10.1038/s41467-020-20809-6 . ISSN 2041-1723 . ПМЦ 7822813 . ПМИД 33483495 .
- ^ Метод Монте-Карло
- ^ Бигри, Роберт А.; Скиалдоне, Антонио; Шулер, Маркус; Кремер, Дороти, Калифорния; Чоталия, Мита; Се, Шейла К.; Барбьери, Мариано; де Сантьяго, Инес; Лавитас, Лирон-Марк; Бранко, Мигель Р.; Фрейзер, Джеймс (март 2017 г.). «Сложные контакты между несколькими энхансерами, зафиксированные с помощью картирования архитектуры генома» . Природа . 543 (7646): 519–524. Бибкод : 2017Natur.543..519B . дои : 10.1038/nature21411 . ISSN 1476-4687 . ПМК 5366070 . ПМИД 28273065 .
- ^ Кинодоз, София А.; Олликайнен, Ной; Табак, Барбара; Палла, Али; Шмидт, Ян Мартен; Детмар, Элизабет; Лай, Мейсон М.; Шишкин, Александр А.; Бхат, Прашант; Такей, Йодай; Трин, Вики (26 июля 2018 г.). «Межхромосомные центры высшего порядка формируют трехмерную организацию генома в ядре» . Клетка . 174 (3): 744–757.e24. дои : 10.1016/j.cell.2018.05.024 . ISSN 0092-8674 . ПМК 6548320 . ПМИД 29887377 . S2CID 47013513 .
- ^ Чжэн, Мэйчжэнь; Тянь, Саймон Чжунюань; Капурсо, Дэниел; Ким, Минджи; Маурья, Рахул; Ли, Бёнку; Пикуч, Эмали; Гун, Лян; Чжу, Жаклин Юфен; Ли, Чжихуэй; Вонг, Чи Хонг (февраль 2019 г.). «Мультиплексные взаимодействия хроматина с точностью до одной молекулы» . Природа . 566 (7745): 558–562. Бибкод : 2019Natur.566..558Z . дои : 10.1038/s41586-019-0949-1 . ISSN 1476-4687 . ПМК 7001875 . ПМИД 30778195 .
- ^ Кох, Линда (апрель 2019 г.). «Понимание взаимодействия хроматина» . Обзоры природы Генетика . 20 (4): 192–193. дои : 10.1038/s41576-019-0103-9 . ISSN 1471-0064 . ПМИД 30783222 . S2CID 67752274 .
- Барски и др., (2007). Профилирование метилирования гистонов в геноме человека с высоким разрешением. Клетка. (129); 823–37.
- Деккер, (2002). Фиксация конформации хромосом. Наука. (295); 1306–1311.
- Деккер, (2006). Три буквы «С» конформации хромосомы: контроль, контроль, контроль. Нат. Методы. (3); 17–21.
- Фуллвуд и др. (2009). Интерактивный хроматин человека, связанный с эстрогеновым рецептором-α. Природа. (462); 58–64.
- Фуллвуд и Иджун (2009). Методы идентификации дальнодействующих хроматиновых взаимодействий на основе ChIP. J Cell Biochem. 107(1); 30–39.
- Джонсон и др. (2007). Полногеномное картирование взаимодействий белка и ДНК in vivo. Наука. (316); 1497–502.
- Куо и Эллис (1999). Сшивание и иммунопреципитация in vivo для изучения динамических ассоциаций «Белок: ДНК» в среде хроматина. Методы. (19); 425–33.
- Ли, Г., Фуллвуд, М.Дж., Сюй, Х., Мулавади, Ф.Х., Велков, С., Вега, В., Арияратне, П.Н., Мохамед, Ю.Б., Оой, Х.С., Теннакун, К., Вэй, С.Л., Руан , Y. и Sung, WK Инструмент ChIA-PET для комплексного анализа взаимодействия хроматина с секвенированием парных концевых меток. Геном Биол, 11 (2). Р22.
- Мастон и др., (2006). Транскрипционные регуляторные элементы в геноме человека. Анну. Реверс: Геномика. Хм Жене. (7); 29–59.
- Полак и Домани (2006). Элементы Alu содержат множество сайтов связывания факторов транскрипции и могут играть роль в регуляции процессов развития. БМК Геномика. (7); 133.
- Вэй и др. (2006). Глобальная карта сайтов связывания транскрипционных факторов p53 в геноме человека. Клетка. (124); 207–19.
Внешние ссылки
[ редактировать ]- Браузер генома ChIA-PET . Этот браузер предназначен для просмотра данных Fullwood et al. (2009) и включает специальную программу просмотра взаимодействий всего генома, которая обеспечивает макроскопическую картину сайтов связывания и взаимодействий, а также ландшафт всего генома.