Сайт связывания ДНК
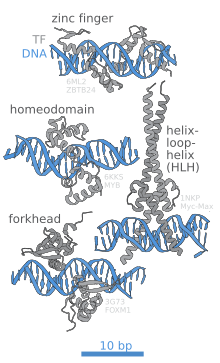
Сайты связывания ДНК — это тип сайта связывания , обнаруженный в ДНК , с которым могут связываться другие молекулы. Сайты связывания ДНК отличаются от других сайтов связывания тем, что (1) они являются частью последовательности ДНК (например, генома) и (2) они связаны с ДНК-связывающими белками . Сайты связывания ДНК часто связаны со специализированными белками, известными как факторы транскрипции , и, таким образом, связаны с регуляцией транскрипции . Сумма сайтов связывания ДНК конкретного фактора транскрипции называется его цистромом . Сайты связывания ДНК также охватывают мишени других белков, таких как ферменты рестрикции , сайт-специфические рекомбиназы (см. Сайт-специфическая рекомбинация ) и метилтрансферазы . [ 1 ]
Таким образом, сайты связывания ДНК можно определить как короткие последовательности ДНК (обычно длиной от 4 до 30 пар оснований, но до 200 пар оснований для сайтов рекомбинации), которые специфически связываются одним или несколькими ДНК-связывающими белками или белковыми комплексами. Сообщалось, что некоторые сайты связывания могут подвергаться быстрым эволюционным изменениям. [ 2 ]
Типы сайтов связывания ДНК
[ редактировать ]Сайты связывания ДНК можно классифицировать в соответствии с их биологической функцией. Таким образом, мы можем различать сайты связывания транскрипционных факторов, сайты рестрикции и сайты рекомбинации. Некоторые авторы предположили, что сайты связывания также можно классифицировать по наиболее удобному способу их представления. [ 3 ] С одной стороны, сайты рестрикции обычно могут быть представлены консенсусными последовательностями. Это связано с тем, что они нацелены в основном на идентичные последовательности, а эффективность рестрикции резко снижается для менее похожих последовательностей. С другой стороны, сайты связывания ДНК для данного фактора транскрипции обычно различны, с разной степенью сродства фактора транскрипции к различным сайтам связывания. Это затрудняет точное представление сайтов связывания транскрипционных факторов с использованием консенсусных последовательностей , и они обычно представляются с использованием позиционно-зависимых частотных матриц (PSFM), которые часто графически изображаются с использованием логотипов последовательностей . Однако этот аргумент отчасти произволен. Ферменты рестрикции, как и факторы транскрипции, обеспечивают постепенный, хотя и резкий, диапазон сродства к различным сайтам. [ 4 ] и, таким образом, они также лучше всего представлены PSFM. Аналогично, сайт-специфические рекомбиназы также демонстрируют различный диапазон сродства к различным сайтам-мишеням. [ 5 ] [ 6 ]
История и основные методики эксперимента.
[ редактировать ]Существование чего-то похожего на сайты связывания ДНК было заподозрено на основании экспериментов по биологии бактериофага лямбда. [ 7 ] и регуляция лакового оперона Escherichia coli . [ 8 ] Сайты связывания ДНК были окончательно подтверждены в обеих системах. [ 9 ] [ 10 ] [ 11 ] с появлением методов секвенирования ДНК . С тех пор с помощью множества экспериментальных методов были обнаружены сайты связывания ДНК для многих факторов транскрипции, ферментов рестрикции и сайт-специфических рекомбиназ. Исторически сложилось так, что экспериментальными методами обнаружения и анализа сайтов связывания ДНК были анализ ДНКазы и анализ электрофоретического сдвига подвижности (EMSA). Однако развитие микрочипов ДНК и методов быстрого секвенирования привело к появлению новых, массово параллельных методов идентификации сайтов связывания in vivo, таких как ChIP-чип и ChIP-Seq . [ 12 ] Для количественной оценки сродства связывания [ 13 ] белков и других молекул к специфическим сайтам связывания ДНК биофизический метод микромасштабный термофорез [ 14 ] используется.
Базы данных
[ редактировать ]Из-за разнообразия экспериментальных методов, используемых при определении сайтов связывания, а также неоднородного охвата большинства организмов и факторов транскрипции, не существует центральной базы данных (аналогично GenBank в Национальном центре биотехнологической информации ) для сайтов связывания ДНК. Несмотря на то, что NCBI предполагает аннотацию сайта связывания ДНК в своих эталонных последовательностях ( RefSeq ), в большинстве материалов эта информация опускается. Более того, из-за ограниченного успеха биоинформатики в создании эффективных инструментов прогнозирования сайтов связывания ДНК (большие уровни ложноположительных результатов часто связаны с методами обнаружения мотивов in silico/поиска сайтов), не предпринималось систематических усилий по компьютерному аннотированию этих функций в секвенированных геномы.
Однако существует несколько частных и общедоступных баз данных, посвященных компиляции экспериментально зарегистрированных, а иногда и предсказанных с помощью вычислений сайтов связывания для различных факторов транскрипции в разных организмах. Ниже приведена неполная таблица доступных баз данных:
| Имя | Организмы | Источник | Доступ | URL-адрес |
|---|---|---|---|---|
| ПлантРегКарта | 165 видов растений (например, Arabidopsis thaliana, Oryza sativa, Zea mays и т. д.) | Экспертное курирование и прогнозирование | Общественный | [1] |
| ДЖАСПАР | Позвоночные животные, растения, грибы, мухи и черви | Экспертное курирование с литературной поддержкой | Общественный | [2] |
| СНГ-BP | Все эукариоты | Экспериментально полученные мотивы и предсказания | Общественный | [3] |
| СобратьTF | Прокариоты | Литературное курирование | Общественный | [4] |
| РайтТочный | Прокариоты | Экспертное курирование | Общественный | [5] |
| РегТрансБаза | Прокариоты | Экспертное/литературное курирование | Общественный | [6] |
| РегулонДБ | кишечная палочка | Экспертное курирование | Общественный | [7] Архивировано 7 мая 2017 г. в Wayback Machine. |
| ПРОДОРИК | Прокариоты | Экспертное курирование | Общественный | [8] Архивировано 16 мая 2007 г. в Wayback Machine. |
| ТРАНСФАК | Млекопитающие | Экспертное/литературное курирование | Государственный/Частный | [9] Архивировано 23 октября 2008 г. в Wayback Machine. |
| ШАГ | Человек, Мышь, Крыса | Компьютерные прогнозы, ручное курирование | Общественный | [10] |
| ДБСД | Виды дрозофилы | Литература/Экспертное курирование | Общественный | [11] |
| ГОКОМОКО | Человек, Мышь | Литература/Экспертное курирование | Общественный | [12] , [13] |
| МетМотив | Человек, Мышь | Экспертное курирование | Общественный | [14] Архивировано 29 октября 2019 г. на Wayback Machine. |
Представление сайтов связывания ДНК
[ редактировать ]Совокупность сайтов связывания ДНК, обычно называемая мотивом связывания ДНК, может быть представлена консенсусной последовательностью . Такое представление имеет то преимущество, что оно компактно, но за счет игнорирования значительного объема информации. [ 15 ] Более точный способ представления сайтов связывания - это матрица частот, специфичная для позиции (PSFM). Эти матрицы дают информацию о частоте каждого основания в каждом положении мотива связывания ДНК. [ 3 ] PSFM обычно задумываются с неявным предположением о позиционной независимости (различные положения в сайте связывания ДНК независимо вносят вклад в функцию сайта), хотя это предположение оспаривается для некоторых сайтов связывания ДНК. [ 16 ] Информация о частоте в PSFM может быть формально интерпретирована в рамках теории информации . [ 17 ] что привело к его графическому представлению в виде логотипа последовательности .
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | |
| А | 1 | 0 | 1 | 5 | 32 | 5 | 35 | 23 | 34 | 14 | 43 | 13 | 34 | 4 | 52 | 3 |
| С | 50 | 1 | 0 | 1 | 5 | 6 | 0 | 4 | 4 | 13 | 3 | 8 | 17 | 51 | 2 | 0 |
| Г | 0 | 0 | 54 | 15 | 5 | 5 | 12 | 2 | 7 | 1 | 1 | 3 | 1 | 0 | 1 | 52 |
| Т | 5 | 55 | 1 | 35 | 14 | 40 | 9 | 27 | 11 | 28 | 9 | 32 | 4 | 1 | 1 | 1 |
| Сумма | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 | 56 |
PSFM для репрессора транскрипции LexA , полученного из 56 сайтов связывания LexA, хранящихся в Prodoric. Относительные частоты получаются путем деления отсчетов в каждой ячейке на общее количество (56)
Компьютерный поиск и обнаружение сайтов связывания
[ редактировать ]В биоинформатике можно выделить две отдельные проблемы, связанные с сайтами связывания ДНК: поиск дополнительных членов известного мотива связывания ДНК (проблема поиска сайта) и обнаружение новых мотивов связывания ДНК в коллекциях функционально связанных последовательностей ( мотива последовательности проблема обнаружения ). . [ 18 ] Для поиска сайтов связывания было предложено множество различных методов. Большинство из них опираются на принципы теории информации и имеют в наличии веб-серверы (Yellaboina)(Munch), тогда как другие авторы прибегают к методам машинного обучения , например искусственным нейронным сетям . [ 3 ] [ 19 ] [ 20 ] Также доступно множество алгоритмов для обнаружения мотивов последовательности . Эти методы основаны на гипотезе о том, что набор последовательностей имеет общий мотив связывания по функциональным причинам. Методы обнаружения связывающих мотивов можно условно разделить на перечислительные, детерминированные и стохастические. [ 21 ] МЕМ [ 22 ] и консенсус [ 23 ] являются классическими примерами детерминированной оптимизации, а сэмплер Гиббса [ 24 ] представляет собой традиционную реализацию чисто стохастического метода обнаружения мотивов связывания ДНК. Другим примером этого класса методов является SeSiMCMC. [ 25 ] который сосредоточен на слабых сайтах TFBS с симметрией. В то время как перечислительные методы часто прибегают к представлению сайтов связывания в регулярных выражениях , PSFM и их формальная обработка в рамках методов теории информации являются предпочтительным представлением как для детерминистических, так и для стохастических методов. Гибридные методы, например ChIPMunk [ 26 ] который сочетает в себе жадную оптимизацию с субдискретизацией, также используйте PSFM. Недавние достижения в секвенировании привели к внедрению подходов сравнительной геномики к обнаружению мотивов связывания ДНК, примером чего является PhyloGibbs. [ 27 ] [ 28 ]
Более сложные методы поиска сайтов связывания и обнаружения мотивов основаны на укладке оснований и других взаимодействиях между основаниями ДНК, но из-за небольших размеров выборки, обычно доступных для сайтов связывания в ДНК, их эффективность до сих пор не используется полностью. Примером такого инструмента является ULPB. [ 29 ]
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Хэлфорд ES; Марко Дж. Ф. (2004). «Как сайт-специфичные ДНК-связывающие белки находят свои цели?» . Исследования нуклеиновых кислот . 32 (10): 3040–3052. дои : 10.1093/nar/gkh624 . ПМЦ 434431 . ПМИД 15178741 .
- ^ Борнеман, Арканзас; Джаноулис, Т.А.; Чжан, З.Д.; Ю, Х.; Розовский Дж.; Серингхаус, MR; Ван, Л.Ю.; Герштейн М. и Снайдер М. (2007). «Расхождение сайтов связывания факторов транскрипции у родственных видов дрожжей». Наука . 317 (5839): 815–819. Бибкод : 2007Sci...317..815B . дои : 10.1126/science.1140748 . ПМИД 17690298 . S2CID 21535866 .
- ^ Jump up to: а б с Стормо Г.Д. (2000). «Сайты связывания ДНК: представление и открытие» . Биоинформатика . 16 (1): 16–23. дои : 10.1093/биоинформатика/16.1.16 . ПМИД 10812473 .
- ^ Пингуд А., Йелч А. (1997). «Распознавание и расщепление ДНК эндонуклеазами рестрикции типа II» . Европейский журнал биохимии . 246 (1): 1–22. дои : 10.1111/j.1432-1033.1997.t01-6-00001.x . ПМИД 9210460 .
- ^ Гьёда А., Комано Т (2000). «Очистка и характеристика шаффлон-специфической рекомбиназы R64» . Журнал бактериологии . 182 (10): 2787–2792. дои : 10.1128/JB.182.10.2787-2792.2000 . ПМК 101987 . ПМИД 10781547 .
- ^ Бирге, Э.А. (2006). «15: Рекомбинация, специфичная для сайта». Генетика бактерий и бактериофагов (5-е изд.). Спрингер. стр. 463–478. ISBN 978-0-387-23919-4 .
- ^ Кэмпбелл А. (1963). «Генетика тонкой структуры и ее связь с функцией». Ежегодный обзор микробиологии . 17 (1): 2787–2792. дои : 10.1146/annurev.mi.17.100163.000405 . ПМИД 14145311 .
- ^ Джейкоб Ф., Моно Дж. (1961). «Генетические регуляторные механизмы синтеза белков». Журнал молекулярной биологии . 3 (3): 318–356. дои : 10.1016/S0022-2836(61)80072-7 . ПМИД 13718526 . S2CID 19804795 .
- ^ Гилберт В., Максам А. (1973). «Нуклеотидная последовательность оператора lac» . Труды Национальной академии наук Соединенных Штатов Америки . 70 (12): 3581–3584. Бибкод : 1973PNAS...70.3581G . дои : 10.1073/pnas.70.12.3581 . ПМК 427284 . ПМИД 4587255 .
- ^ Маниатис Т., Пташне М., Баррелл Б.Г., Донельсон Дж. (1974). «Последовательность сайта связывания репрессора в ДНК бактериофага лямбда». Природа . 250 (465): 394–397. Бибкод : 1974Natur.250..394M . дои : 10.1038/250394a0 . ПМИД 4854243 . S2CID 4204720 .
- ^ Нэш Х.А. (1975). «Интегративная рекомбинация ДНК бактериофага лямбда in vitro» . Труды Национальной академии наук Соединенных Штатов Америки . 72 (3): 1072–1076. Бибкод : 1975PNAS...72.1072N . дои : 10.1073/pnas.72.3.1072 . ПМК 432468 . ПМИД 1055366 .
- ^ Ельницкий Л., Джин В.Х., Фарнхэм П.Дж., Джонс С.Дж. (2006). «Определение сайтов связывания транскрипционных факторов млекопитающих: обзор вычислительных и экспериментальных методов» . Геномные исследования . 16 (12): 1455–1464. дои : 10.1101/гр.4140006 . ПМИД 17053094 .
- ^ Бааске П., Венкен С.Дж., Райнек П., Дур С., Браун Д. (февраль 2010 г.). «Оптический термофорез количественно определяет буферную зависимость связывания аптамеров». Энджью. хим. Межд. Эд . 49 (12): 2238–41. дои : 10.1002/anie.200903998 . ПМИД 20186894 . S2CID 42489892 .
- «Горячий путь к новым лекарствам» . Физика.орг . 24 февраля 2010 г.
- ^ Винкен CJ; и др. (2010). «Анализ связывания белков в биологических жидкостях с использованием микромасштабного термофореза» . Природные коммуникации . 1 (7): 100. Бибкод : 2010NatCo...1..100W . дои : 10.1038/ncomms1093 . ПМИД 20981028 .
- ^ Шнайдер ТД (2002). «Последовательность консенсуса Дзен» . Прикладная биоинформатика . 1 (3): 111–119. ПМЦ 1852464 . ПМИД 15130839 .
- ^ Булык М.Л.; Джонсон PL; Черч ГМ (2002). «Нуклеотиды сайтов связывания транскрипционных факторов оказывают взаимозависимое влияние на аффинность связывания транскрипционных факторов» . Исследования нуклеиновых кислот . 30 (5): 1255–1261. дои : 10.1093/нар/30.5.1255 . ПМК 101241 . ПМИД 11861919 .
- ^ Шнайдер Т.Д., Стормо Г.Д., Голд Л., Эренфойхт А. (1986). «Информативность сайтов связывания на нуклеотидных последовательностях». Журнал молекулярной биологии . 188 (3): 415–431X. дои : 10.1016/0022-2836(86)90165-8 . ПМИД 3525846 .
- ^ Эрилл I; О'Нил MC (2009). «Пересмотр методов идентификации сайтов связывания ДНК, основанных на теории информации» . БМК Биоинформатика . 10 (1): 57. дои : 10.1186/1471-2105-10-57 . ПМК 2680408 . ПМИД 19210776 .
- ^ Бизант Д., Майзель Дж. (1995). «Идентификация сайтов связывания рибосом в Escherichia coli с использованием моделей нейронных сетей» . Исследования нуклеиновых кислот . 23 (9): 1632–1639. дои : 10.1093/нар/23.9.1632 . ПМК 306908 . ПМИД 7784221 .
- ^ О'Нил MC (1991). «Обучение нейронных сетей обратного распространения ошибки определению и обнаружению сайтов связывания ДНК» . Исследования нуклеиновых кислот . 19 (2): 133–318. дои : 10.1093/нар/19.2.313 . ПМЦ 333596 . ПМИД 2014171 .
- ^ Бэйли Т.Л. (2008). «Открытие мотивов последовательности». Биоинформатика (PDF) . Методы молекулярной биологии. Том. 452. стр. 231–251. дои : 10.1007/978-1-60327-159-2_12 . ISBN 978-1-58829-707-5 . ПМИД 18566768 .
- ^ Бэйли Т.Л. (2002). «Открытие новых мотивов последовательностей с помощью ЦЕМА». Современные протоколы в биоинформатике . 2 (4): 2.4.1–2.4.35. дои : 10.1002/0471250953.bi0204s00 . ПМИД 18792935 . S2CID 205157795 .
- ^ Стормо Г.Д., Хартцелл Г.В., третий (1989). «Идентификация сайтов связывания белков по невыровненным фрагментам ДНК» . Труды Национальной академии наук Соединенных Штатов Америки . 86 (4): 1183–1187. Бибкод : 1989PNAS...86.1183S . дои : 10.1073/pnas.86.4.1183 . ПМК 286650 . ПМИД 2919167 .
- ^ Лоуренс К.Э., Альтшул С.Ф. , Богуски М.С. , Лю Дж.С., Нойвальд А.Ф., Вуттон Дж.К. (1993). «Обнаружение тонких сигналов последовательности: стратегия выборки Гиббса для множественного выравнивания». Наука . 262 (5131): 208–214. Бибкод : 1993Sci...262..208L . дои : 10.1126/science.8211139 . ПМИД 8211139 . S2CID 3040614 .
- ^ Фаворов А.В.; М.С. Гельфанд; А.В. Герасимова; Д.А. Равчеев; А.А. Миронов; В.Я. Макеев (15 мая 2005 г.). «Пробоотборник Гиббса для идентификации симметрично структурированных, расположенных между собой мотивов ДНК с улучшенной оценкой длины сигнала» . Биоинформатика . 21 (10): 2240–2245. doi : 10.1093/биоинформатика/bti336 . ISSN 1367-4803 . ПМИД 15728117 .
- ^ Кулаковский, ИВ; В.А. Боева; А.В. Фаворов; В.Я. Макеев (24 августа 2010 г.). «Глубокий и широкий поиск мотивов связывания в данных ChIP-Seq» . Биоинформатика . 26 (20): 2622–3. doi : 10.1093/биоинформатика/btq488 . ISSN 1367-4811 . ПМИД 20736340 .
- ^ Дас МК, Дай Гонконг (2007). «Обзор алгоритмов поиска мотивов ДНК» . БМК Биоинформатика . 8 (Приложение 7): S21. дои : 10.1186/1471-2105-8-S7-S21 . ПМК 2099490 . ПМИД 18047721 .
- ^ Сиддхартхан Р., Сиггия Э.Д., ван Нимвеген Э. (2005). «PhyloGibbs: инструмент для поиска мотивов выборки Гиббса, учитывающий филогению» . ПЛОС Компьютерная Биол . 1 (7): е67. Бибкод : 2005PLSCB...1...67S . дои : 10.1371/journal.pcbi.0010067 . ПМК 1309704 . ПМИД 16477324 .
- ^ Салама Р.А., Стекел DJ (2010). «Включение взаимозависимостей соседних оснований существенно улучшает предсказание места связывания прокариотического транскрипционного фактора в масштабе всего генома» . Исследования нуклеиновых кислот . 38 (12): е135. дои : 10.1093/nar/gkq274 . ПМЦ 2896541 . ПМИД 20439311 .
Внешние ссылки
[ редактировать ]- ENCODE threads Explorer Мотивы факторов транскрипции в природе
- Отобранные вручную мотивы связывания TF для 157 видов растений. Архивировано 19 октября 2016 г. на Wayback Machine.