Транскрипционный фактор
| Глоссарий факторов транскрипции | |
|---|---|
| |

В молекулярной биологии фактор транскрипции ( TF ) (или фактор связывания ДНК, специфичный для последовательности ) представляет собой белок , который контролирует скорость транскрипции генетической информации из ДНК в информационную РНК путем связывания с определенной последовательностью ДНК . [1] [2] Функция ТФ заключается в регулировании – включении и выключении – генов, чтобы гарантировать, что они экспрессируются в нужных клетках в нужное время и в нужном количестве на протяжении всей жизни клетки и организма. Группы ТФ функционируют скоординировано, направляя деление клеток , рост клеток и их гибель на протяжении всей жизни; клеток миграция и организация ( план тела ) во время эмбрионального развития; и периодически в ответ на сигналы извне клетки, такие как гормон . насчитывается около 1600 ТФ В геноме человека . [3] [4] [5] Факторы транскрипции являются членами протеома , а также регулома .
ТФ работают отдельно или в комплексе с другими белками, способствуя (в качестве активатора ) или блокируя (в качестве репрессора ) рекрутирование РНК-полимеразы (фермента, который осуществляет транскрипцию генетической информации с ДНК на РНК) к конкретным генам. [6] [7] [8]
Определяющей особенностью ТФ является то, что они содержат по крайней мере один ДНК-связывающий домен (DBD), который прикрепляется к определенной последовательности ДНК, прилегающей к генам, которые они регулируют. [9] [10] ТФ группируются в классы на основе их DBD. [11] [12] Другие белки, такие как коактиваторы , ремоделеры хроматина , ацетилтрансферазы гистонов , деацетилазы гистонов , киназы и метилазы , также необходимы для регуляции генов, но не имеют ДНК-связывающих доменов и, следовательно, не являются ТФ. [13]
ТФ представляют интерес для медицины, поскольку мутации ТФ могут вызывать специфические заболевания, и потенциально против них могут быть нацелены лекарства.
Число
[ редактировать ]Факторы транскрипции необходимы для регуляции экспрессии генов и, как следствие, обнаружены во всех живых организмах. Количество факторов транскрипции, обнаруженных в организме, увеличивается с размером генома, а более крупные геномы, как правило, имеют больше факторов транскрипции на ген. [14]
имеется около 2800 белков В геноме человека , содержащих ДНК-связывающие домены, и предположительно 1600 из них функционируют как факторы транскрипции. [3] хотя другие исследования показывают, что это меньшее число. [15] Таким образом, примерно 10% генов в геноме кодируют факторы транскрипции, что делает это семейство самым крупным семейством белков человека. Более того, гены часто окружены несколькими сайтами связывания различных факторов транскрипции, и эффективная экспрессия каждого из этих генов требует совместного действия нескольких различных факторов транскрипции (см., например, ядерные факторы гепатоцитов ). Следовательно, комбинаторное использование подмножества примерно из 2000 факторов транскрипции человека легко объясняет уникальную регуляцию каждого гена в геноме человека во время развития . [13]
Механизм
[ редактировать ]Факторы транскрипции связываются либо с энхансерными , либо с промоторными областями ДНК, прилегающими к генам, которые они регулируют. В зависимости от фактора транскрипции транскрипция соседнего гена регулируется либо вверх, либо вниз . Факторы транскрипции используют различные механизмы регуляции экспрессии генов. [16] Эти механизмы включают в себя:
- стабилизировать или блокировать связывание РНК-полимеразы с ДНК [ нужна ссылка ]
- катализируют ацетилирование или деацетилирование белков- гистонов . Фактор транскрипции может либо делать это напрямую, либо рекрутировать другие белки с такой каталитической активностью. Многие факторы транскрипции используют тот или иной из двух противоположных механизмов регуляции транскрипции: [17]
- Активность гистон-ацетилтрансферазы (HAT) – ацетилирует белки гистонов , что ослабляет связь ДНК с гистонами , что делает ДНК более доступной для транскрипции, тем самым усиливая транскрипцию.
- Активность гистондеацетилазы (HDAC) – деацетилирует белки гистонов , что усиливает связь ДНК с гистонами, которые делают ДНК менее доступной для транскрипции, тем самым подавляя транскрипцию.
- рекрутировать белки -коактиваторы или корепрессоры в комплекс ДНК фактора транскрипции [18]
Функция
[ редактировать ]Факторы транскрипции — это одна из групп белков, которые читают и интерпретируют генетическую «схему» ДНК. Они связываются с ДНК и помогают запустить программу увеличения или уменьшения транскрипции генов. Таким образом, они жизненно важны для многих важных клеточных процессов. Ниже приведены некоторые важные функции и биологические роли факторов транскрипции:
Базальная регуляция транскрипции
[ редактировать ]У эукариот важный класс факторов транскрипции, называемый общими факторами транскрипции (GTF). для осуществления транскрипции необходим [19] [20] [21] Многие из этих GTF на самом деле не связываются с ДНК, а скорее являются частью большого комплекса преинициации транскрипции , который напрямую взаимодействует с РНК-полимеразой . Наиболее распространенными GTF являются TFIIA , TFIIB , TFIID (см. также TATA-связывающий белок ), TFIIE , TFIIF и TFIIH . [22] Преинициативный комплекс связывается с промоторными областями ДНК, расположенными выше гена, который они регулируют.
Дифференциальное усиление транскрипции
[ редактировать ]Другие факторы транскрипции по-разному регулируют экспрессию различных генов путем связывания с энхансерными областями ДНК, соседними с регулируемыми генами. Эти факторы транскрипции имеют решающее значение для обеспечения экспрессии генов в нужной клетке в нужное время и в нужном количестве, в зависимости от меняющихся потребностей организма. [ нужна ссылка ]
Разработка
[ редактировать ]Многие транскрипционные факторы у многоклеточных организмов участвуют в развитии. [23] В ответ на стимулы эти факторы транскрипции включают/выключают транскрипцию соответствующих генов, что, в свою очередь, позволяет изменять морфологию клеток или активность, необходимую для определения судьбы клеток и клеточной дифференцировки . Семейство транскрипционных факторов Hox , например, важно для формирования правильного строения тела у организмов, столь же разнообразных, как у плодовых мух и у людей. [24] [25] Другим примером является фактор транскрипции, кодируемый геном области Y, определяющим пол (SRY), который играет важную роль в определении пола у людей. [26]
Реакция на межклеточные сигналы
[ редактировать ]Клетки могут общаться друг с другом, высвобождая молекулы, которые создают сигнальные каскады внутри другой восприимчивой клетки. Если сигнал требует усиления или подавления генов в клетке-реципиенте, часто факторы транскрипции оказываются ниже по ходу сигнального каскада. [27] Передача сигналов эстрогена является примером довольно короткого сигнального каскада, в котором участвует фактор транскрипции рецептора эстрогена : эстроген секретируется такими тканями, как яичники и плацента , проникает через клеточную мембрану клетки-реципиента и связывается с рецептором эстрогена в клеточной мембране. цитоплазма . клетки Затем рецептор эстрогена попадает в ядро и связывается с ее участками связывания ДНК , изменяя регуляцию транскрипции связанных генов. [28]
Реакция на окружающую среду
[ редактировать ]Факторы транскрипции действуют не только после сигнальных каскадов, связанных с биологическими стимулами, но также могут находиться после сигнальных каскадов, участвующих в стимулах окружающей среды. Примеры включают фактор теплового шока (HSF), который активирует гены, необходимые для выживания при более высоких температурах, [29] фактор, индуцируемый гипоксией (HIF), который активирует гены, необходимые для выживания клеток в средах с низким содержанием кислорода, [30] и белок, связывающий регуляторные элементы стерола (SREBP), который помогает поддерживать правильный уровень липидов в клетке. [31]
Контроль клеточного цикла
[ редактировать ]Многие факторы транскрипции, особенно те, которые являются протоонкогенами или супрессорами опухолей , помогают регулировать клеточный цикл и, таким образом, определяют, насколько большой станет клетка и когда она сможет разделить на две дочерние клетки. [32] [33] Одним из примеров является онкоген Myc , который играет важную роль в росте клеток и апоптозе . [34]
Патогенез
[ редактировать ]Факторы транскрипции также можно использовать для изменения экспрессии генов в клетке-хозяине с целью стимулирования патогенеза. Хорошо изученным примером этого являются эффекторы, подобные активаторам транскрипции ( эффекторы TAL ), секретируемые бактериями Xanthomonas . При введении в растения эти белки могут проникать в ядро растительной клетки, связывать последовательности промотора растения и активировать транскрипцию генов растений, которые способствуют бактериальной инфекции. [35] Эффекторы TAL содержат центральную область повтора, в которой существует простая связь между идентичностью двух критических остатков в последовательных повторах и последовательных оснований ДНК в целевом сайте эффектора TAL. [36] [37] Это свойство, вероятно, облегчает эволюцию этих белков, чтобы лучше конкурировать с защитными механизмами клетки-хозяина. [38]
Регулирование
[ редактировать ]В биологии важные процессы часто имеют несколько уровней регулирования и контроля. Это также верно и в отношении факторов транскрипции: не только факторы транскрипции контролируют скорость транскрипции, регулируя количество генных продуктов (РНК и белка), доступных клетке, но и сами факторы транскрипции регулируются (часто другими факторами транскрипции). Ниже приводится краткий обзор некоторых способов регулирования активности транскрипционных факторов:
Синтез
[ редактировать ]Факторы транскрипции (как и все белки) транскрибируются с гена на хромосоме в РНК, а затем РНК транслируется в белок. Любой из этих этапов можно регулировать, чтобы повлиять на продукцию (и, следовательно, на активность) транскрипционного фактора. Следствием этого является то, что факторы транскрипции могут регулировать себя. Например, в петле отрицательной обратной связи фактор транскрипции действует как собственный репрессор: если белок фактора транскрипции связывает ДНК своего собственного гена, он подавляет выработку большего количества самого себя. Это один из механизмов поддержания низких уровней транскрипционного фактора в клетке. [39]
Ядерная локализация
[ редактировать ]У эукариот факторы транскрипции (как и большинство белков) транскрибируются в ядре клетки , но затем транслируются в цитоплазме . Многие белки, активные в ядре, содержат сигналы ядерной локализации , которые направляют их в ядро. Но для многих факторов транскрипции это ключевой момент в их регуляции. [40] Важные классы транскрипционных факторов, такие как некоторые ядерные рецепторы, должны сначала связать лиганд , находясь в цитоплазме, прежде чем они смогут переместиться в ядро. [40]
Активация
[ редактировать ]Факторы транскрипции могут быть активированы (или деактивированы) через их сигнально-чувствительный домен с помощью ряда механизмов, включая:
- связывание лиганда . Связывание лиганда не только способно влиять на расположение транскрипционного фактора внутри клетки, но и связывание лиганда может также влиять на то, находится ли транскрипционный фактор в активном состоянии и способен ли связывать ДНК или другие кофакторы (см., например, ядерный фактор). рецепторы ).
- фосфорилирование [41] [42] – Многие факторы транскрипции, такие как белки STAT, должны быть фосфорилированы , прежде чем они смогут связываться с ДНК.
- взаимодействие с другими факторами транскрипции ( например , гомо- или гетеродимеризация ) или корегуляторными белками [ нужна ссылка ]
Доступность сайта связывания ДНК
[ редактировать ]У эукариот ДНК организована с помощью гистонов в компактные частицы, называемые нуклеосомами , где последовательности примерно из 147 пар оснований ДНК делают ~1,65 оборота вокруг октамеров гистоновых белков. ДНК внутри нуклеосом недоступна для многих факторов транскрипции. Некоторые факторы транскрипции, так называемые пионерские факторы, все еще способны связывать свои сайты связывания с нуклеосомной ДНК. Для большинства других факторов транскрипции нуклеосома должна активно раскручиваться с помощью молекулярных моторов, таких как ремоделеры хроматина . [43] Альтернативно, нуклеосома может быть частично развернута за счет тепловых флуктуаций, обеспечивая временный доступ к сайту связывания транскрипционного фактора. Во многих случаях фактору транскрипции приходится конкурировать за связывание со своим участком связывания ДНК с другими факторами транскрипции и гистонами или негистоновыми белками хроматина. [44] Пары транскрипционных факторов и других белков могут играть антагонистическую роль (активатор или репрессор) в регуляции одного и того же гена . [ нужна ссылка ]
Наличие других кофакторов/факторов транскрипции
[ редактировать ]Большинство факторов транскрипции не работают в одиночку. Многие большие семейства ТФ образуют сложные гомотипические или гетеротипические взаимодействия посредством димеризации. [45] Для того чтобы произошла транскрипция гена, ряд факторов транскрипции должен связываться с регуляторными последовательностями ДНК. Этот набор факторов транскрипции, в свою очередь, рекрутирует промежуточные белки, такие как кофакторы , которые обеспечивают эффективное рекрутирование преинициативного комплекса и РНК-полимеразы . Таким образом, чтобы один фактор транскрипции инициировал транскрипцию, все эти другие белки также должны присутствовать, а фактор транскрипции должен находиться в состоянии, в котором он может связываться с ними при необходимости.Кофакторы — это белки, которые модулируют действие факторов транскрипции. Кофакторы взаимозаменяемы между промоторами конкретных генов; белковый комплекс, занимающий промоторную ДНК, и аминокислотная последовательность кофактора определяют его пространственную конформацию. Например, некоторые стероидные рецепторы могут обмениваться кофакторами с NF-κB , что является переключением между воспалением и клеточной дифференцировкой; таким образом, стероиды могут влиять на воспалительную реакцию и функцию определенных тканей. [46]
Взаимодействие с метилированным цитозином
[ редактировать ]Факторы транскрипции и метилированные цитозины в ДНК играют важную роль в регуляции экспрессии генов. (Метилирование цитозина в ДНК в первую очередь происходит там, где за цитозином следует гуанин в 5'-3'-последовательности ДНК, сайте CpG .) Метилирование сайтов CpG в промоторной области гена обычно подавляет транскрипцию гена. [47] в то время как метилирование CpG в теле гена увеличивает экспрессию. [48] Ферменты ТЕТ играют центральную роль в деметилировании метилированных цитозинов. Деметилирование CpG в промоторе гена под действием активности фермента ТЕТ увеличивает транскрипцию гена. [49]
Были оценены сайты связывания ДНК 519 факторов транскрипции. [50] Из них 169 транскрипционных факторов (33%) не имели CpG-динуклеотидов в своих сайтах связывания, а 33 транскрипционных фактора (6%) могли связываться с CpG-содержащим мотивом, но не отдавали предпочтение сайту связывания с метилированным мотивом. или неметилированный CpG. Было 117 факторов транскрипции (23%), которые ингибировали связывание со своей последовательностью связывания, если она содержала метилированный сайт CpG, 175 факторов транскрипции (34%), которые имели усиленное связывание, если их последовательность связывания имела метилированный сайт CpG, и 25 транскрипционных факторов факторы (5%) либо ингибировались, либо усиливали связывание в зависимости от того, где в последовательности связывания находился метилированный CpG. [ нужна ссылка ]
Ферменты ТЕТ не связываются специфически с метилцитозином, за исключением случаев его рекрутирования (см. Деметилирование ДНК ). Было показано , что множественные факторы транскрипции, важные для дифференциации клеток и спецификации линий, включая NANOG , SALL4 A, WT1 , EBF1 , PU.1 и E2A , рекрутируют ферменты TET в определенные геномные локусы (в первую очередь энхансеры) для воздействия на метилцитозин (mC). и превращают его в гидроксиметилцитозин hmC (и в большинстве случаев маркируют их для последующего полного деметилирования до цитозина). [51] Опосредованное ТЕТ преобразование mC в hmC, по-видимому, нарушает связывание 5mC-связывающих белков, включая белки MECP2 и MBD ( метил-CpG-связывающий домен ), способствуя ремоделированию нуклеосом и связыванию факторов транскрипции, тем самым активируя транскрипцию этих генов. EGR1 является важным транскрипционным фактором формирования памяти . Он играет важную роль в головного мозга перепрограммировании нейронов эпигенетическом . Транскрипционный фактор EGR1 рекрутирует белок TET1 , который инициирует путь деметилирования ДНК . [52] EGR1 вместе с TET1 используется для программирования распределения сайтов метилирования в ДНК мозга во время развития мозга и обучения (см. Эпигенетика в обучении и памяти ).
Структура
[ редактировать ]

Факторы транскрипции имеют модульную структуру и содержат следующие домены : [1]
- ДНК-связывающий домен ( DBD ), который прикрепляется к определенным последовательностям ДНК ( энхансер или промотор трансгена вектора . Необходимый компонент для всех векторов. Используется для управления транскрипцией последовательностей промотора ), прилегающих к регулируемым генам. Последовательности ДНК, которые связывают факторы транскрипции, часто называют элементами ответа .
- Домен активации ( AD ), который содержит сайты связывания для других белков, таких как корегуляторы транскрипции . Эти сайты связывания часто называют функциями активации ( AF ), доменом трансактивации ( TAD ) или транс-активирующим доменом TAD , не следует путать с топологически ассоциированным доменом ( TAD ). [53]
- Необязательный сигнал-чувствительный домен ( SSD ) ( например , лиганд-связывающий домен), который воспринимает внешние сигналы и в ответ передает эти сигналы остальной части транскрипционного комплекса, что приводит к повышению или понижению регуляции экспрессии генов. . Кроме того, DBD и сигнально-чувствительные домены могут располагаться на отдельных белках, которые связываются внутри транскрипционного комплекса для регулирования экспрессии генов.
ДНК-связывающий домен
[ редактировать ]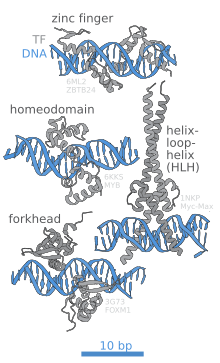
Часть ( домен ) транскрипционного фактора, который связывает ДНК, называется его ДНК-связывающим доменом. Ниже приведен неполный список некоторых основных семейств ДНК-связывающих доменов/факторов транскрипции:
Элементы реагирования
[ редактировать ]Последовательность ДНК, с которой связывается фактор транскрипции, называется сайтом связывания фактора транскрипции или элементом ответа . [62]
Факторы транскрипции взаимодействуют со своими сайтами связывания, используя комбинацию электростатических которых являются водородные связи ( особым случаем ) и сил Ван-дер-Ваальса . Из-за природы этих химических взаимодействий большинство факторов транскрипции связывают ДНК определенным образом. Однако не все основания в сайте связывания транскрипционного фактора могут фактически взаимодействовать с транскрипционным фактором. Кроме того, некоторые из этих взаимодействий могут быть слабее других. Таким образом, факторы транскрипции не связывают только одну последовательность, а способны связывать подмножество близкородственных последовательностей, каждая из которых имеет разную силу взаимодействия. [ нужна ссылка ]
Например, хотя консенсусным сайтом связывания ТАТА -связывающего белка (TBP) является TATAAAA, фактор транскрипции TBP также может связывать подобные последовательности, такие как TATATAT или TATATAA. [ нужна ссылка ]
Поскольку факторы транскрипции могут связывать набор родственных последовательностей, а эти последовательности, как правило, короткие, потенциальные сайты связывания факторов транскрипции могут возникнуть случайно, если последовательность ДНК достаточно длинная. Однако маловероятно, что фактор транскрипции свяжет все совместимые последовательности геноме клетки в . Другие ограничения, такие как доступность ДНК в клетке или наличие кофакторов, также могут помочь определить, где фактически будет связываться транскрипционный фактор. Таким образом, учитывая последовательность генома, все еще трудно предсказать, где именно транскрипционный фактор будет связываться в живой клетке.
Однако дополнительная специфичность распознавания может быть достигнута за счет использования более чем одного ДНК-связывающего домена (например, тандемных DBD в одном и том же факторе транскрипции или за счет димеризации двух факторов транскрипции), которые связываются с двумя или более соседними последовательностями ДНК.
Клиническое значение
[ редактировать ]Факторы транскрипции имеют клиническое значение как минимум по двум причинам: (1) мутации могут быть связаны с конкретными заболеваниями и (2) они могут быть мишенью лекарств.
расстройства
[ редактировать ]Из-за их важной роли в развитии, межклеточной передаче сигналов и клеточном цикле некоторые заболевания человека связаны с мутациями факторов транскрипции. [63]
Многие факторы транскрипции являются либо опухолевыми супрессорами , либо онкогенами , и, таким образом, их мутации или аберрантная регуляция связаны с раком. Известно, что три группы транскрипционных факторов играют важную роль при раке человека: (1) семейства NF-kappaB и AP-1 , (2) семейство STAT и (3) стероидные рецепторы . [64]
Ниже приведены несколько наиболее изученных примеров:
| Состояние | Описание | Локус |
|---|---|---|
| Прямой синдром | Мутации транскрипционного фактора MECP2 связаны с синдромом Ретта , расстройством нервного развития. [65] [66] | Xq28 |
| Диабет | Редкая форма диабета , называемая MODY (диабет зрелого возраста у молодых), может быть вызвана мутациями ядерных факторов гепатоцитов (HNF). [67] или фактор-1 промотора инсулина (IPF1/Pdx1). [68] | несколько |
| Вербальная диспраксия развития | Мутации транскрипционного фактора FOXP2 связаны с вербальной диспраксией развития — заболеванием, при котором люди не способны производить тонко скоординированные движения, необходимые для речи. [69] | 7q31 |
| Аутоиммунные заболевания | Мутации транскрипционного фактора FOXP3 вызывают редкую форму аутоиммунного заболевания, называемого IPEX . [70] | Xp11.23-q13.3 |
| Синдром Ли-Фраумени | Вызывается мутациями в опухолевом супрессоре р53 . [71] | 17п13.1 |
| Рак молочной железы | Семейство STAT имеет отношение к раку молочной железы . [72] | несколько |
| Множественные виды рака | Семья HOX участвует в развитии различных видов рака. [73] | несколько |
| Остеоартрит | Мутация или снижение активности SOX9 [74] |
Потенциальные мишени для наркотиков
[ редактировать ]Примерно 10% назначаемых в настоящее время лекарств напрямую воздействуют на класс ядерных рецепторов транскрипционных факторов. [75] Примеры включают тамоксифен и бикалутамид для лечения рака молочной железы и простаты соответственно, а также различные типы противовоспалительных и анаболических стероидов . [76] Кроме того, факторы транскрипции часто модулируются лекарственными средствами косвенно через сигнальные каскады . Возможно, с помощью лекарств можно напрямую воздействовать на другие, менее изученные факторы транскрипции, такие как NF-κB . [77] [78] [79] [80] Считается, что на факторы транскрипции, не входящие в семейство ядерных рецепторов, труднее воздействовать с помощью низкомолекулярной терапии, поскольку неясно, являются ли они «лекарственными», но в отношении Pax2 был достигнут прогресс. [81] [82] и путь с надрезом . [83]
Роль в эволюции
[ редактировать ]Дупликация генов сыграла решающую роль в эволюции видов. Это особенно относится к транскрипционным факторам. Как только они возникают в виде дубликатов, накопленные мутации, кодирующие одну копию, могут иметь место, не оказывая негативного влияния на регуляцию нижестоящих мишеней. изменения специфичности связывания ДНК однокопийного фактора транскрипции Leafy Однако недавно были выяснены , который встречается у большинства наземных растений. В этом отношении однокопийный транскрипционный фактор может претерпевать изменение специфичности за счет беспорядочного промежуточного соединения без потери функции. Подобные механизмы были предложены в контексте всех альтернативных филогенетических гипотез и роли факторов транскрипции в эволюции всех видов. [84] [85]
Роль в деятельности биоконтроля
[ редактировать ]Факторы транскрипции играют роль в активности устойчивости , что важно для успешной по биоконтролю деятельности . Устойчивость к окислительному стрессу и чувствительность к щелочному pH были обеспечены транскрипционным фактором Yap1 и Rim101 Papiliotrema terrestris LS28 в качестве молекулярных инструментов, которые позволили понять генетические механизмы, лежащие в основе активности биоконтроля, которая поддерживает программы управления заболеваниями , основанные на биологическом и комплексном контроле. [86]
Анализ
[ редактировать ]Существуют различные технологии для анализа факторов транскрипции. На геномном ДНК и исследование баз данных. секвенирование уровне обычно используются [87] Белковую версию транскрипционного фактора можно обнаружить с помощью специфических антител . Образец обнаруживается с помощью вестерн-блоттинга . Используя анализ электрофоретического сдвига подвижности (EMSA), [88] может быть обнаружен профиль активации транскрипционных факторов. Мультиплексный подход к профилированию активации представляет собой систему TF - чипов, в которой можно параллельно обнаружить несколько различных факторов транскрипции. [ нужна ссылка ]
Наиболее часто используемым методом идентификации сайтов связывания транскрипционных факторов является иммунопреципитация хроматина (ChIP). [89] Этот метод основан на химической фиксации хроматина формальдегидом с последующей совместной осаждением ДНК и интересующего транскрипционного фактора с использованием антитела , которое специфически нацелено на этот белок. Затем последовательности ДНК можно идентифицировать с помощью микрочипов или высокопроизводительного секвенирования ( ChIP-seq ) для определения сайтов связывания факторов транскрипции. Если антитела к интересующему белку отсутствуют, DamID . удобной альтернативой может быть [90]
Классы
[ редактировать ]Как более подробно описано ниже, транскрипционные факторы можно классифицировать по их (1) механизму действия, (2) регуляторной функции или (3) гомологии последовательностей (и, следовательно, структурному сходству) в их ДНК-связывающих доменах.
Механистический
[ редактировать ]Существует два механистических класса транскрипционных факторов:
- Общие факторы транскрипции участвуют в формировании преинициаторного комплекса . Наиболее распространенные сокращенно обозначаются как TFIIA , TFIIB , TFIID , TFIIE , TFIIF и TFIIH . Они распространены повсеместно и взаимодействуют с основной областью промотора, окружающей сайт(ы) начала транскрипции всех генов класса II . [91]
- Вышестоящие факторы транскрипции — это белки, которые связываются где-то выше сайта инициации, стимулируя или подавляя транскрипцию. Они являются примерно синонимами конкретных факторов транскрипции , поскольку они значительно различаются в зависимости от того, какие последовательности распознавания присутствуют поблизости от гена. [92]
| Примеры специфических факторов транскрипции [92] | |||
|---|---|---|---|
| Фактор | Конструктивный тип | Последовательность распознавания | Привязывается как |
| СП1 | Цинковый палец | 5'- ГГГКГГ- 3' | Мономер |
| АП-1 | Основная молния | 5'-ТГА(Г/Ц)ТСА-3' | Димер |
| С/EBP | Основная молния | 5'-ATTGGCCAAT-3' | Димер |
| Фактор теплового шока | Основная молния | 5'-XGAAX-3' | Тример |
| АТФ/КРЭБ | Основная молния | 5'-TGACGTCA-3' | Димер |
| c-Myc | Базовая спираль-петля-спираль | 5'-CACGTG-3' | Димер |
| 1 октября | Спираль-поворот-спираль | 5'-ATGCAAAT-3' | Мономер |
| НФ-1 | Роман | 5'-TTGGCXXXXXGCCAA-3' | Димер |
| (G/C) = G или C Икс знак равно А , Т , G или C | |||
Функциональный
[ редактировать ]Факторы транскрипции классифицируются в зависимости от их регуляторной функции: [13]
- I. конститутивно активен – всегда присутствует во всех клетках – общие факторы транскрипции , Sp1 , NF1 , CCAAT
- II. условно активный – требует активации
- II.A онтогенетический (клеточно-специфичный) – экспрессия строго контролируется, но после экспрессии не требует дополнительной активации – GATA , HNF , PIT-1 , MyoD , Myf5 , Hox , Winged Helix.
- II.B зависимый от сигнала – для активации требуется внешний сигнал
- II.B.1 внеклеточный лиганд ( эндокринный или паракринный )-зависимый – ядерные рецепторы
- II.B.2 внутриклеточный лиганд ( аутокринный )-зависимый – активируется небольшими внутриклеточными молекулами – SREBP , p53 , орфанные ядерные рецепторы
- II.B.3 рецептор-зависимый от клеточной мембраны – сигнальные каскады второго мессенджера, приводящие к фосфорилированию транскрипционного фактора
Структурный
[ редактировать ]Факторы транскрипции часто классифицируются на основе сходства последовательностей и, следовательно, третичной структуры их ДНК-связывающих доменов: [93] [12] [94] [11]
- 1 суперкласс: базовые домены
- 1.1 Класс: молнии Лейциновые факторы ( bZIP )
- 1.2 Класс: факторы спираль-петля-спираль ( bHLH )
- 1.2.1 Семейство: вездесущие (класс А) факторы
- 1.2.2 Семейство: миогенные факторы транскрипции ( MyoD )
- 1.2.3 Семья: Ахаэте-Скуте
- 1.2.4 Семья: Тал/Твист/Атонал/Хен
- 1.3 Класс: факторы спираль-петля-спираль/лейциновая молния ( bHLH-ZIP )
- 1.4 Класс: НФ-1
- 1,5 Класс: RF-X
- 1.6 Класс: БХШ
- 2 Суперкласс: цинк-координирующие ДНК-связывающие домены.
- 2.1 Класс: цинковый палец Cys4 типа ядерного рецептора .
- 2.1.1 Семейство: рецепторы стероидных гормонов.
- 2.1.2 Семейство: рецепторам гормонов щитовидной железы. факторы, подобные
- 2.2 Класс: разнообразные цинковые пальцы Cys4
- 2.2.1 Семейство: GATA-факторы
- 2.3 Класс: домен цинкового пальца Cys2His2
- 2.4 Класс: Цистеин-цинковый кластер Cys6
- 2.5 Класс: Цинковые пальцы чередующегося состава.
- 2.1 Класс: цинковый палец Cys4 типа ядерного рецептора .
- 3 Суперкласс: спираль-поворот-спираль
- 3.1 Класс: Домен Homeo
- 3.1.1 Семья: только домен Homeo; включает убкс
- 3.1.2 Семейство: факторы домена POU ; включает октябрь
- 3.1.3 Семья: домен Homeo с регионом LIM
- 3.1.4 Семейство: гомео-домен плюс мотивы из цинковых пальцев.
- 3.2 Класс: Парная коробка
- 3.2.1 Семья: парный плюс домен homeo
- 3.2.2 Семья: только парный домен
- 3.3 Класс: Вилочная головка / крылатая спираль
- 3.3.1 Семья: регуляторы развития; включает вилку
- 3.3.2 Семейство: тканеспецифические регуляторы
- 3.3.3 Семейство: факторы, контролирующие клеточный цикл
- 3.3.0 Семейство: Другие регулирующие органы
- 3.4 Класс: Факторы теплового шока
- 3.4.1 Семья: HSF
- 3.5 Класс: Кластеры триптофана.
- 3.5.1 Семья: Майб
- 3.5.2 Семейство: Ets-типа
- 3.5.3 Семья: факторы регуляции интерферона
- 3.6 Класс: Домен TEA (фактор усилителя транскрипции)
- 3.1 Класс: Домен Homeo
- 4 Суперкласс: факторы бета-каркаса с контактами с малыми канавками
- 4.1 Класс: RHR ( область гомологии Rel )
- 4.2 Класс: СТАТ
- 4.2.1 Семейство: СТАТ
- 4.3 Класс: стр53
- 4.3.1 Семья: стр. 53
- 4.4 Класс: коробка MADS
- 4.4.1. Семья: регуляторы дифференциации; включает ( Mef2 )
- 4.4.2 Семейство: Реагирующие на внешние сигналы, SRF ( сывороточный фактор ответа ) ( SRF )
- 4.4.3 Семейство: Метаболические регуляторы (ARG80).
- 4.5 Класс: факторы транскрипции альфа-спирали бета-баррель
- 4.6 Класс: ТАТА-связывающие белки
- 4.6.1 Семейство: ТБП
- 4.7 Класс: HMG-коробка
- 4.8 Класс: Гетеромерные факторы CCAAT
- 4.8.1 Семейство: гетеромерные факторы CCAAT
- 4.9 Класс: Грейнихед
- 4.9.1 Семья: Грейнихед
- 4.10 Класс: холодного шока Факторы
- 4.10.1 Семейство: csd
- 4.11 Класс: Коротышка
- 4.11.1 Семья: Коротышка
- 0 Суперкласс: другие факторы транскрипции
- 0.1 Класс: Белки медного кулака
- 0,2 Класс: HMGI(Y) ( HMGA1 )
- 0.2.1 Семейство: HMGI(Y)
- 0.3 Класс: Карманный домен
- 0.4 Класс: факторы, подобные E1A.
- 0.5 Класс: Факторы, связанные с AP2/EREBP
Базы данных транскрипционных факторов
[ редактировать ]Существует множество баз данных, каталогизирующих информацию о факторах транскрипции, но их объем и полезность сильно различаются. Некоторые могут содержать только информацию о реальных белках, некоторые — об их сайтах связывания или об их генах-мишенях. Примеры включают следующее:
- footprintDB — база метаданных из нескольких баз данных, включая JASPAR и другие.
- JASPAR : база данных сайтов связывания транскрипционных факторов эукариот.
- PlantTFD : база данных факторов транскрипции растений. [95]
- TcoF-DB : База данных кофакторов транскрипции и взаимодействий факторов транскрипции. [96]
- TFcheckpoint : база данных кандидатов на ТФ человека, мышей и крыс.
- transscriptionfactor.org (сейчас коммерческий, продает реагенты)
- MethMotif.org : интегративная клеточно-специфичная база данных мотивов связывания факторов транскрипции в сочетании с профилями метилирования ДНК. [97]
См. также
[ редактировать ]- Семейство белков Cdx
- ДНК-связывающий белок
- Ингибитор ДНК-связывающего белка
- Картограф(2)
- Ядерный рецептор , класс транскрипционных факторов, активируемых лигандом.
- Открытая база данных нормативных аннотаций
- Филогенетический след
- База данных ТРАНСФАК
- ЙетФаSCo
Ссылки
[ редактировать ]- ^ Jump up to: а б Латчман Д.С. (декабрь 1997 г.). «Факторы транскрипции: обзор» . Международный журнал биохимии и клеточной биологии . 29 (12): 1305–12. дои : 10.1016/S1357-2725(97)00085-X . ЧВК 2002184 . ПМИД 9570129 .
- ^ Карин М. (февраль 1990 г.). «Слишком много факторов транскрипции: положительные и отрицательные взаимодействия». Новый биолог . 2 (2): 126–31. ПМИД 2128034 .
- ^ Jump up to: а б Бабу М.М., Ласкомб Н.М., Аравинд Л., Герштейн М., Тейхманн С.А. (июнь 2004 г.). «Структура и эволюция сетей регуляции транскрипции» (PDF) . Современное мнение в области структурной биологии . 14 (3): 283–91. дои : 10.1016/j.sbi.2004.05.004 . ПМИД 15193307 . Архивировано из оригинала (PDF) 30 августа 2019 года . Проверено 25 октября 2017 г.
- ^ Как регулируются гены: факторы транскрипции на YouTube
- ^ Ламберт С., Джолма А., Кампителли Л., Пратюш З., Дас К., Инь Ю. и др. (2018). «Факторы транскрипции человека» . Клетка . 172 (4): 650–665. дои : 10.1016/j.cell.2018.01.029 . ПМИД 29425488 .
Окончательный подсчет включает 1639 известных или вероятных человеческих ФТ.
- ^ Редер Р.Г. (сентябрь 1996 г.). «Роль общих факторов инициации транскрипции РНК-полимеразой II». Тенденции биохимических наук . 21 (9): 327–35. дои : 10.1016/S0968-0004(96)10050-5 . ПМИД 8870495 .
- ^ Николов Д.Б., Берли С.К. (январь 1997 г.). «Инициация транскрипции РНК-полимеразы II: структурный взгляд» . Труды Национальной академии наук Соединенных Штатов Америки . 94 (1): 15–22. Бибкод : 1997ПНАС...94...15Н . дои : 10.1073/pnas.94.1.15 . ПМК 33652 . ПМИД 8990153 .
- ^ Ли Т.И., Молодой РА (2000). «Транскрипция генов, кодирующих эукариотические белки». Ежегодный обзор генетики . 34 : 77–137. дои : 10.1146/annurev.genet.34.1.77 . ПМИД 11092823 .
- ^ Митчелл П.Дж., Тцзян Р. (июль 1989 г.). «Регуляция транскрипции в клетках млекопитающих с помощью ДНК-связывающих белков, специфичных для последовательности». Наука . 245 (4916): 371–8. Бибкод : 1989Sci...245..371M . дои : 10.1126/science.2667136 . ПМИД 2667136 .
- ^ Пташне М., Ганн А. (апрель 1997 г.). «Транкрипционная активация путем рекрутирования». Природа . 386 (6625): 569–77. Бибкод : 1997Natur.386..569P . дои : 10.1038/386569a0 . ПМИД 9121580 . S2CID 6203915 .
- ^ Jump up to: а б Цзинь Дж, Чжан Х, Конг Л, Гао Г, Луо Дж (январь 2014 г.). «PlantTFDB 3.0: портал для функционального и эволюционного изучения факторов транскрипции растений» . Исследования нуклеиновых кислот . 42 (Проблема с базой данных): D1182-7. дои : 10.1093/нар/gkt1016 . ПМК 3965000 . ПМИД 24174544 .
- ^ Jump up to: а б Матис В., Кель-Маргулис О.В., Фрике Е., Либих И., Ланд С., Барре-Дирри А. и др. (январь 2006 г.). «ТРАНСФАК и его модуль ТРАНСКомпель: регуляция транскрипционных генов у эукариот» . Исследования нуклеиновых кислот . 34 (Проблема с базой данных): D108-10. дои : 10.1093/nar/gkj143 . ПМЦ 1347505 . ПМИД 16381825 .
- ^ Jump up to: а б с Бриванлу А.Х., Дарнелл Дж.Э. (февраль 2002 г.). «Передача сигнала и контроль экспрессии генов». Наука . 295 (5556): 813–8. Бибкод : 2002Sci...295..813B . дои : 10.1126/science.1066355 . ПМИД 11823631 . S2CID 14954195 .
- ^ ван Нимвеген Э. (сентябрь 2003 г.). «Законы масштабирования функционального содержания геномов». Тенденции в генетике . 19 (9): 479–84. arXiv : физика/0307001 . дои : 10.1016/S0168-9525(03)00203-8 . ПМИД 12957540 . S2CID 15887416 .
- ^ «Список всех факторов транскрипции человека» . biostars.org .
- ^ Джилл Дж. (2001). «Регуляция инициации эукариотической транскрипции». Очерки по биохимии . 37 : 33–43. дои : 10.1042/bse0370033 . ПМИД 11758455 .
- ^ Нарликар Г.Дж., Fan HY, Kingston RE (февраль 2002 г.). «Сотрудничество между комплексами, регулирующими структуру и транскрипцию хроматина» . Клетка . 108 (4): 475–87. дои : 10.1016/S0092-8674(02)00654-2 . ПМИД 11909519 . S2CID 14586791 .
- ^ Сюй Л, Гласс С.К., Розенфельд М.Г. (апрель 1999 г.). «Коактиваторные и корепрессорные комплексы в функции ядерных рецепторов». Текущее мнение в области генетики и развития . 9 (2): 140–7. дои : 10.1016/S0959-437X(99)80021-5 . ПМИД 10322133 .
- ^ Роберт О.Дж. Вайнцирл (1999). Механизмы экспрессии генов: структура, функции и эволюция базального транскрипционного аппарата . Мировое научное издательство. ISBN 1-86094-126-5 .
- ^ Риз Дж. К. (апрель 2003 г.). «Базальные факторы транскрипции». Текущее мнение в области генетики и развития . 13 (2): 114–8. дои : 10.1016/S0959-437X(03)00013-3 . ПМИД 12672487 .
- ^ Шилатифард А., Конэвей Р.К., Конэвей Дж.В. (2003). «Комплекс элонгации РНК-полимеразы II». Ежегодный обзор биохимии . 72 : 693–715. doi : 10.1146/annurev.biochem.72.121801.161551 . ПМИД 12676794 .
- ^ Томас MC, Чан CM (2006). «Общий механизм транскрипции и общие кофакторы». Критические обзоры по биохимии и молекулярной биологии . 41 (3): 105–78. дои : 10.1080/10409230600648736 . ПМИД 16858867 . S2CID 13073440 .
- ^ Лобе К.Г. (1992). Транскрипционные факторы и развитие млекопитающих . Текущие темы биологии развития. Том. 27. С. 351–83. дои : 10.1016/S0070-2153(08)60539-6 . ISBN 978-0-12-153127-0 . ПМИД 1424766 .
- ^ Лемонс Д., Макгиннис В. (сентябрь 2006 г.). «Геномная эволюция кластеров генов Hox». Наука . 313 (5795): 1918–22. Бибкод : 2006Sci...313.1918L . дои : 10.1126/science.1132040 . ПМИД 17008523 . S2CID 35650754 .
- ^ Моенс CB , Селлери Л (март 2006 г.). «Кофакторы Hox в развитии позвоночных» . Биология развития . 291 (2): 193–206. дои : 10.1016/j.ydbio.2005.10.032 . ПМИД 16515781 .
- ^ Оттоленги С., Уда М., Криспони Л., Омари С., Као А., Форабоско А. и др. (январь 2007 г.). «Определенность и стабильность пола». Биоэссе . 29 (1): 15–25. doi : 10.1002/bies.20515 . ПМИД 17187356 . S2CID 23824870 .
- ^ Поусон Т. (1993). «Передача сигнала - консервативный путь от мембраны к ядру». Генетика развития . 14 (5): 333–8. дои : 10.1002/dvg.1020140502 . ПМИД 8293575 .
- ^ Осборн С.К., Шифф Р., Фукуа С.А., Шоу Дж. (декабрь 2001 г.). «Рецептор эстрогена: современное понимание его активации и модуляции». Клинические исследования рака . 7 (12 дополнений): 4338–4342, обсуждение 4411–4412. ПМИД 11916222 .
- ^ Шамовский И., Нудлер Э. (март 2008 г.). «Новое понимание механизма активации реакции на тепловой шок» . Клеточные и молекулярные науки о жизни . 65 (6): 855–61. дои : 10.1007/s00018-008-7458-y . ПМЦ 11131843 . ПМИД 18239856 . S2CID 9912334 .
- ^ Бенизри Э., Жинувес А., Берра Э. (апрель 2008 г.). «Магия сигнального каскада гипоксии» . Клеточные и молекулярные науки о жизни . 65 (7–8): 1133–49. дои : 10.1007/s00018-008-7472-0 . ПМЦ 11131810 . ПМИД 18202826 . S2CID 44049779 .
- ^ Вебер Л.В., Болл М., Stampfl A (ноябрь 2004 г.). «Поддержание гомеостаза холестерина: белки, связывающие регуляторные элементы стерола» . Всемирный журнал гастроэнтерологии . 10 (21): 3081–7. дои : 10.3748/wjg.v10.i21.3081 . ПМЦ 4611246 . ПМИД 15457548 .
- ^ Уитон К., Атаджа П., Риабовол К. (1996). «Регуляция активности транскрипционных факторов во время клеточного старения». Биохимия и клеточная биология . 74 (4): 523–34. дои : 10.1139/o96-056 . ПМИД 8960358 .
- ^ Мейяппан М., Атаджа П.В., Риабовол К.Т. (1996). «Регуляция экспрессии генов и активности связывания транскрипционных факторов во время клеточного старения». Биологические сигналы . 5 (3): 130–8. дои : 10.1159/000109183 . ПМИД 8864058 .
- ^ Эван Дж., Харрингтон Э., Фаниди А., Лэнд Х., Амати Б., Беннетт М. (август 1994 г.). «Комплексный контроль пролиферации и гибели клеток с помощью онкогена c-myc». Философские труды Лондонского королевского общества. Серия Б, Биологические науки . 345 (1313): 269–75. Бибкод : 1994RSPTB.345..269E . дои : 10.1098/rstb.1994.0105 . ПМИД 7846125 .
- ^ Бох Дж., Бонас У (2010). «Эффекторы семейства Xanthomonas AvrBs3 типа III: открытие и функция». Ежегодный обзор фитопатологии . 48 : 419–36. doi : 10.1146/annurev-phyto-080508-081936 . ПМИД 19400638 .
- ^ Москва М.Ю., Богданов А.Ю. (декабрь 2009 г.). «Простой шифр управляет распознаванием ДНК эффекторами TAL». Наука . 326 (5959): 1501. Бибкод : 2009Sci...326.1501M . дои : 10.1126/science.1178817 . ПМИД 19933106 . S2CID 6648530 .
- ^ Бох Дж., Шольце Х., Шорнак С., Ландграф А., Хан С., Кей С. и др. (декабрь 2009 г.). «Взлом кода специфичности связывания ДНК эффекторов TAL-типа III». Наука . 326 (5959): 1509–12. Бибкод : 2009Sci...326.1509B . дои : 10.1126/science.1178811 . ПМИД 19933107 . S2CID 206522347 .
- ^ Войтас Д.Ф., Йонг Дж.К. (декабрь 2009 г.). «Наука о растениях. Связывание ДНК стало проще» . Наука . 326 (5959): 1491–2. Бибкод : 2009Sci...326.1491V . дои : 10.1126/science.1183604 . ПМЦ 7814878 . ПМИД 20007890 . S2CID 33257689 .
- ^ Пан Г, Ли Дж, Чжоу Ю, Чжэн Х, Пей Д (август 2006 г.). «Петля отрицательной обратной связи факторов транскрипции, которая контролирует плюрипотентность и самообновление стволовых клеток» . Журнал ФАСЭБ . 20 (10): 1730–2. doi : 10.1096/fj.05-5543fje . ПМИД 16790525 . S2CID 44783683 .
- ^ Jump up to: а б Уайтсайд С.Т., Гудборн С. (апрель 1993 г.). «Передача сигнала и ядерное нацеливание: регуляция активности транскрипционных факторов путем субклеточной локализации». Журнал клеточной науки . 104 (4): 949–55. дои : 10.1242/jcs.104.4.949 . ПМИД 8314906 .
- ^ Боманн Д. (ноябрь 1990 г.). «Фосфорилирование транскрипционного фактора: связь между передачей сигнала и регуляцией экспрессии генов». Раковые клетки . 2 (11): 337–44. ПМИД 2149275 .
- ^ Вайгель Н.Л., Мур Н.Л. (октябрь 2007 г.). «Фосфорилирование стероидных рецепторов: ключевой модулятор нескольких функций рецепторов» . Молекулярная эндокринология . 21 (10): 2311–9. дои : 10.1210/me.2007-0101 . ПМИД 17536004 .
- ^ Тейф В.Б., Риппе К. (сентябрь 2009 г.). «Предсказание положения нуклеосом в ДНК: сочетание предпочтений внутренней последовательности и активности ремоделера» . Исследования нуклеиновых кислот . 37 (17): 5641–55. дои : 10.1093/нар/gkp610 . ПМК 2761276 . ПМИД 19625488 .
- ^ Тейф В.Б., Риппе К. (октябрь 2010 г.). «Статистически-механические решетчатые модели связывания белка с ДНК в хроматине». Физический журнал: конденсированное вещество . 22 (41): 414105. arXiv : 1004.5514 . Бибкод : 2010JPCM...22O4105T . дои : 10.1088/0953-8984/22/41/414105 . ПМИД 21386588 . S2CID 103345 .
- ^ Амуциас Г.Д., Робертсон Д.Л., Ван де Пер Ю., Оливер С.Г. (май 2008 г.). «Выберите своих партнеров: димеризация эукариотических факторов транскрипции». Тенденции биохимических наук . 33 (5): 220–9. дои : 10.1016/j.tibs.2008.02.002 . ПМИД 18406148 .
- ^ Копленд Дж.А., Шеффилд-Мур М., Колджич-Зиванович Н., Джентри С., Лампроу Г., Цорцату-Статопулу Ф. и др. (июнь 2009 г.). «Рецепторы половых стероидов при дифференцировке скелета и эпителиальной неоплазии: возможно ли тканеспецифическое вмешательство?». Биоэссе . 31 (6): 629–41. doi : 10.1002/bies.200800138 . ПМИД 19382224 . S2CID 205469320 .
- ^ Вебер М., Хеллманн И., Стадлер М.Б., Рамос Л., Паабо С., Ребхан М. и др. (апрель 2007 г.). «Распространение, потенциал молчания и эволюционное влияние метилирования ДНК промотора в геноме человека». Нат. Жене . 39 (4): 457–66. дои : 10.1038/ng1990 . ПМИД 17334365 . S2CID 22446734 .
- ^ Ян X, Хан Х, Де Карвалью Д.Д., Лэй Ф.Д., Джонс П.А., Лян Г. (октябрь 2014 г.). «Метилирование тел генов может изменить экспрессию генов и является терапевтической мишенью при раке» . Раковая клетка . 26 (4): 577–90. дои : 10.1016/j.ccr.2014.07.028 . ПМК 4224113 . ПМИД 25263941 .
- ^ Маедер М.Л., Ангстман Дж.Ф., Ричардсон М.Е., Линдер С.Дж., Касио В.М., Цай С.К. и др. (декабрь 2013 г.). «Направленное деметилирование ДНК и активация эндогенных генов с использованием программируемых слитых белков TALE-TET1» . Нат. Биотехнология . 31 (12): 1137–42. дои : 10.1038/nbt.2726 . ПМЦ 3858462 . ПМИД 24108092 .
- ^ Инь Ю, Моргунова Е, Джолма А, Каасинен Е, Саху Б, Хунд-Сайед С и др. (май 2017 г.). «Влияние метилирования цитозина на специфичность связывания ДНК факторов транскрипции человека» . Наука . 356 (6337): eaaj2239. дои : 10.1126/science.aaj2239 . ПМК 8009048 . ПМИД 28473536 . S2CID 206653898 .
- ^ Лио CJ, Рао А (2019). «Ферменты ТЕТ и 5hmC в системах адаптивного и врожденного иммунитета» . Фронт Иммунол . 10 :210. дои : 10.3389/fimmu.2019.00210 . ПМК 6379312 . ПМИД 30809228 .
- ^ Сунь Z, Сюй X, Хэ Дж, Мюррей А, Сунь М.А., Вэй X, Ван X, Маккойг Е, Се Е, Цзян X, Ли Л, Чжу Дж, Чен Дж, Морозов А, Пикрелл А.М., Теус М.Х., Се H. EGR1 рекрутирует TET1 для формирования метилома мозга во время развития и при активности нейронов. Нац Коммун. 29 августа 2019 г.; 10 (1): 3892. дои: 10.1038/s41467-019-11905-3. ПМИД 31467272
- ^ Вернмарк А., Трейтер Э., Райт А.П., Густафссон Дж.А. (октябрь 2003 г.). «Функции активации 1 и 2 ядерных рецепторов: молекулярные стратегии активации транскрипции» . Молекулярная эндокринология . 17 (10): 1901–9. дои : 10.1210/me.2002-0384 . ПМИД 12893880 . S2CID 31314461 .
- ^ Литтлвуд Т.Д., Эван Дж.И. (1995). «Факторы транскрипции 2: спираль-петля-спираль». Белковый профиль . 2 (6): 621–702. ПМИД 7553065 .
- ^ Винсон С., Мякишев М., Ачарья А., Мир А.А., Молл Дж.Р., Бонович М. (сентябрь 2002 г.). «Классификация белков B-ZIP человека на основе свойств димеризации» . Молекулярная и клеточная биология . 22 (18): 6321–35. дои : 10.1128/MCB.22.18.6321-6335.2002 . ПМЦ 135624 . ПМИД 12192032 .
- ^ Винтдженс Р., Руман М. (сентябрь 1996 г.). «Структурная классификация ДНК-связывающих доменов HTH и режимы взаимодействия белок-ДНК». Журнал молекулярной биологии . 262 (2): 294–313. дои : 10.1006/jmbi.1996.0514 . ПМИД 8831795 .
- ^ Геринг В.Дж., Аффольтер М., Бурглин Т. (1994). «Гомеодоменные белки». Ежегодный обзор биохимии . 63 : 487–526. дои : 10.1146/annurev.bi.63.070194.002415 . ПМИД 7979246 .
- ^ Бурглин Т.Р., Аффольтер М (июнь 2016 г.). «Гомеодоменные белки: обновление» . Хромосома . 125 (3): 497–521. дои : 10.1007/s00412-015-0543-8 . ПМЦ 4901127 . ПМИД 26464018 .
- ^ Даль Э., Косеки Х., Баллинг Р. (сентябрь 1997 г.). «Гены Pax и органогенез». Биоэссе . 19 (9): 755–65. дои : 10.1002/bies.950190905 . ПМИД 9297966 . S2CID 23755557 .
- ^ Лэйти Дж. Х., Ли Б. М., Райт П. Е. (февраль 2001 г.). «Белки цинковых пальцев: новое понимание структурного и функционального разнообразия». Современное мнение в области структурной биологии . 11 (1): 39–46. дои : 10.1016/S0959-440X(00)00167-6 . ПМИД 11179890 .
- ^ Вулф С.А., Неклюдова Л., Пабо К.О. (2000). «Распознавание ДНК белками цинковых пальцев Cys2His2». Ежегодный обзор биофизики и биомолекулярной структуры . 29 : 183–212. doi : 10.1146/annurev.biophys.29.1.183 . ПМИД 10940247 .
- ^ Ван Дж.К. (март 2005 г.). «Поиск основных целей регуляторов транскрипции» . Клеточный цикл . 4 (3): 356–8. дои : 10.4161/cc.4.3.1521 . ПМИД 15711128 .
- ^ Семенца, Грегг Л. (1999). Транскрипционные факторы и болезни человека . Оксфорд [Оксфордшир]: Издательство Оксфордского университета. ISBN 978-0-19-511239-9 .
- ^ Либерманн Т.А., Зербини Л.Ф. (февраль 2006 г.). «Нацеливание на транскрипционные факторы для генной терапии рака». Современная генная терапия . 6 (1): 17–33. дои : 10.2174/156652306775515501 . ПМИД 16475943 .
- ^ Моретти П., Зогби Х.Ю. (июнь 2006 г.). «Дисфункция MeCP2 при синдроме Ретта и связанных с ним расстройствах». Текущее мнение в области генетики и развития . 16 (3): 276–81. дои : 10.1016/j.где.2006.04.009 . ПМИД 16647848 .
- ^ Чедвик Л.Х., Уэйд, Пенсильвания (апрель 2007 г.). «MeCP2 при синдроме Ретта: репрессор транскрипции или архитектурный белок хроматина?» . Текущее мнение в области генетики и развития . 17 (2): 121–5. дои : 10.1016/j.где.2007.02.003 . ПМИД 17317146 . Архивировано из оригинала 2 октября 2023 года — через Zenodo.
- ^ Маэстро М.А., Кардалда С., Бой С.Ф., Луко Р.Ф., Сервитья Х.М., Феррер Дж. (2007). «Отличительные роли HNF1 Β , HNF1 α и HNF4 α в регуляции развития поджелудочной железы, функции и роста Β-клеток» . Различная роль HNF1beta, HNF1alpha и HNF4alpha в регуляции развития поджелудочной железы, функции и роста бета-клеток . Эндокринное развитие. Том. 12. Каргерское медицинское и научное издательство. стр. 33–45. дои : 10.1159/000109603 . ISBN 978-3-8055-8385-5 . ПМИД 17923767 .
- ^ Аль-Куобейли Ф, Монтенарх М (апрель 2008 г.). «Гомеобокс-фактор-1 поджелудочной железы и двенадцатиперстной кишки и сахарный диабет 2-го типа (обзор)» . Международный журнал молекулярной медицины . 21 (4): 399–404. дои : 10.3892/ijmm.21.4.399 . ПМИД 18360684 . Архивировано из оригинала 2 октября 2023 года.
- ^ Леннон П.А., Купер М.Л., Пайффер Д.А., Гундерсон К.Л., Патель А., Питерс С. и др. (апрель 2007 г.). «Удаление 7q31.1 подтверждает участие FOXP2 в нарушениях речи: клинический отчет и обзор». Американский журнал медицинской генетики. Часть А. 143А (8): 791–8. дои : 10.1002/ajmg.a.31632 . ПМИД 17330859 . S2CID 22021740 .
- ^ ван дер Влит Х.Дж., Ньювенхейс Э.Э. (2007). «IPEX как результат мутации FOXP3» . Клиническая и развивающая иммунология . 2007 : 1–5. дои : 10.1155/2007/89017 . ПМК 2248278 . ПМИД 18317533 .
- ^ Ивакума Т., Лозано Г., Флорес Э.Р. (июль 2005 г.). «Синдром Ли-Фраумени: семейное дело p53» . Клеточный цикл . 4 (7): 865–7. дои : 10.4161/cc.4.7.1800 . ПМИД 15917654 .
- ^ Резюме Клевенджера (ноябрь 2004 г.). «Роль и регуляция факторов транскрипции семейства Stat при раке молочной железы человека» . Американский журнал патологии (обзор). 165 (5): 1449–1460. дои : 10.1016/S0002-9440(10)63403-7 . ПМК 1618660 . ПМИД 15509516 .
- ^ « Факторы транскрипции как мишени и маркеры рака». Семинар 2007 г.» . Архивировано из оригинала 25 мая 2012 года . Проверено 14 декабря 2009 г.
- ^ Говиндарадж К., Хендрикс Дж., Лидке Д.С., Карпериен М., Пост Дж.Н. (январь 2019 г.). «Изменения в восстановлении флуоресценции после фотообесцвечивания (FRAP) как индикатор активности транскрипционного фактора SOX9» . Biochimica et Biophysica Acta (BBA) - Механизмы регуляции генов . 1862 (1): 107–117. дои : 10.1016/j.bbagrm.2018.11.001 . PMID 30465885 .
- ^ Оверингтон Дж. П., Аль-Лазикани Б., Хопкинс А. Л. (декабрь 2006 г.). «Сколько существует целей по борьбе с наркотиками?». Обзоры природы. Открытие наркотиков . 5 (12): 993–6. дои : 10.1038/nrd2199 . ПМИД 17139284 . S2CID 11979420 .
- ^ Гронемейер Х., Густавссон Дж.А., Лаудет В. (ноябрь 2004 г.). «Принципы модуляции суперсемейства ядерных рецепторов». Обзоры природы. Открытие наркотиков . 3 (11): 950–64. дои : 10.1038/nrd1551 . ПМИД 15520817 . S2CID 205475111 .
- ^ Бастин С.А., Маккей И.А. (июнь 1994 г.). «Факторы транскрипции: мишени для новых дизайнерских лекарств». Британский журнал биомедицинской науки . 51 (2): 147–57. ПМИД 8049612 .
- ^ Батт Т.Р., Каратанасис С.К. (1995). «Факторы транскрипции как мишени для лекарств: возможности терапевтической селективности» . Экспрессия генов . 4 (6): 319–36. ПМК 6134363 . ПМИД 7549464 .
- ^ Папавассилиу А.Г. (август 1998 г.). «Агенты, модулирующие фактор транскрипции: точность и селективность при разработке лекарств». Молекулярная медицина сегодня . 4 (8): 358–66. дои : 10.1016/S1357-4310(98)01303-3 . ПМИД 9755455 .
- ^ Гош Д., Папавассилиу А.Г. (2005). «Терапия транскрипционными факторами: перспектива или магнит». Современная медицинская химия . 12 (6): 691–701. дои : 10.2174/0929867053202197 . ПМИД 15790306 .
- ^ Гримли Э., Ляо С., Рангини Э., Николовска-Колеска З., Дресслер Г. (2017). «Ингибирование активации транскрипции Pax2 с помощью небольшой молекулы, нацеленной на ДНК-связывающий домен» . АКС Химическая биология . 12 (3): 724–734. doi : 10.1021/acschembio.6b00782 . ПМК 5761330 . ПМИД 28094913 .
- ^ Гримли Э., Дресслер Г.Р. (2018). «Являются ли белки Pax потенциальными терапевтическими мишенями при заболеваниях почек и раке?» . Почки Интернешнл . 94 (2): 259–267. дои : 10.1016/j.kint.2018.01.025 . ПМК 6054895 . ПМИД 29685496 .
- ^ Мёллеринг Р.Э., Корнехо М., Дэвис Т.Н., Дель Бьянко С., Астер Дж.К., Блэклоу С.С. и др. (ноябрь 2009 г.). «Прямое ингибирование комплекса транскрипционных факторов NOTCH» . Природа . 462 (7270): 182–8. Бибкод : 2009Natur.462..182M . дои : 10.1038/nature08543 . ПМЦ 2951323 . ПМИД 19907488 .
- Разместить резюме в: Кэтрин Бэгли (11 ноября 2009 г.). «Новая мишень для лечения рака» . Ученый . Архивировано из оригинала 16 ноября 2009 года.
- ^ Саю С., Моннио М., Нанао М.Х., Мойруд Э., Брокингтон С.Ф., Тевенон Э. и др. (февраль 2014 г.). «Неразборчивое промежуточное соединение лежит в основе эволюции специфичности связывания ДНК LEAFY» . Наука . 343 (6171): 645–8. Бибкод : 2014Sci...343..645S . дои : 10.1126/science.1248229 . hdl : 1885/64773 . ПМИД 24436181 . S2CID 207778924 . [ постоянная мертвая ссылка ]
- ^ Цзинь Дж, Хэ К, Тан Икс, Ли З, Лу Л, Чжао Ю и др. (июль 2015 г.). «Карта регуляции транскрипции арабидопсиса раскрывает отличительные функциональные и эволюционные особенности новых факторов транскрипции» . Молекулярная биология и эволюция . 32 (7): 1767–73. дои : 10.1093/molbev/msv058 . ПМЦ 4476157 . ПМИД 25750178 .
- ^ Кастория Р., Микколи С., Бароне Г., Палмьери Д., Де Кертис Ф., Лима Г. и др. (март 2021 г.). Могу ли я (ред.). «Молекулярные инструменты для дрожжей Papiliotrema terrestris LS28 и идентификация Yap1 как фактора транскрипции, участвующего в активности биоконтроля» . Прикладная и экологическая микробиология . 87 (7). Бибкод : 2021ApEnM..87E2910C . дои : 10.1128/АЕМ.02910-20 . ПМК 8091616 . ПМИД 33452020 .
- ^ Грау Дж, Бен-Гал I, Пош С, Гросс I (июль 2006 г.). «VOMBAT: прогнозирование сайтов связывания транскрипционных факторов с использованием байесовских деревьев переменного порядка» (PDF) . Исследования нуклеиновых кислот . 34 (проблема с веб-сервером): W529-33. дои : 10.1093/нар/gkl212 . ПМЦ 1538886 . ПМИД 16845064 . Архивировано из оригинала (PDF) 30 сентября 2018 года . Проверено 10 января 2014 г.
- ^ Вента Н., Штраус Х., Мейер С., Винкемайер У. (июль 2008 г.). «Фосфорилирование тирозина регулирует разделение STAT1 между различными конформациями димера» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (27): 9238–43. Бибкод : 2008PNAS..105.9238W . дои : 10.1073/pnas.0802130105 . ПМЦ 2453697 . ПМИД 18591661 .
- ^ Фьюри Т.С. (декабрь 2012 г.). «ChIP-seq и не только: новые и улучшенные методологии обнаружения и характеристики взаимодействий белок-ДНК» . Обзоры природы. Генетика . 13 (12): 840–52. дои : 10.1038/nrg3306 . ПМЦ 3591838 . ПМИД 23090257 .
- ^ Оги Дж.Н., Саутхолл, Т.Д. (январь 2016 г.). «Черт, это хорошо! DamID профилирование взаимодействий белок-ДНК» . Междисциплинарные обзоры Wiley: Биология развития . 5 (1): 25–37. дои : 10.1002/wdev.205 . ПМЦ 4737221 . ПМИД 26383089 .
- ^ Орфанидес Г., Лагранж Т., Рейнберг Д. (ноябрь 1996 г.). «Общие факторы транскрипции РНК-полимеразы II» . Гены и развитие . 10 (21): 2657–83. дои : 10.1101/gad.10.21.2657 . ПМИД 8946909 .
- ^ Jump up to: а б Бор ВФ (2003). Медицинская физиология: клеточный и молекулярный подход . Эльзевир/Сондерс. стр. 125–126. ISBN 1-4160-2328-3 .
- ^ Стегмайер П., Кел А.Э., Вингендер Э. (2004). «Систематическая классификация факторов транскрипции ДНК-связывающих доменов» . Геномная информатика. Международная конференция по геномной информатике . 15 (2): 276–86. ПМИД 15706513 . Архивировано из оригинала 19 июня 2013 года.
- ^ «База данных ТРАНСФАК» . Проверено 5 августа 2007 г.
- ^ Цзинь Дж., Тянь Ф., Ян Д.К., Мэн Ю.К., Конг Л., Луо Дж. и др. (январь 2017 г.). «PlantTFDB 4.0: к центральному узлу факторов транскрипции и регуляторных взаимодействий в растениях» . Исследования нуклеиновых кислот . 45 (Д1): Д1040–Д1045. дои : 10.1093/nar/gkw982 . ПМК 5210657 . ПМИД 27924042 .
- ^ Шмайер С., Алам Т., Эссак М., Бажич В.Б. (январь 2017 г.). «TcoF-DB v2: обновление базы данных кофакторов транскрипции человека и мыши и взаимодействий факторов транскрипции» . Исследования нуклеиновых кислот . 45 (Д1): Д145–Д150. дои : 10.1093/нар/gkw1007 . ПМК 5210517 . ПМИД 27789689 .
- ^ Сюань Линь QX, Сиан С., Ан О, Тьеффри Д., Джа С., Бенукраф Т. (январь 2019 г.). «MethMotif: интегративная база данных, специфичная для клеток, мотивов связывания транскрипционных факторов в сочетании с профилями метилирования ДНК» . Исследования нуклеиновых кислот . 47 (Д1): Д145–Д154. дои : 10.1093/nar/gky1005 . ПМК 6323897 . ПМИД 30380113 .
Дальнейшее чтение
[ редактировать ]- Карретеро-Паулет, Лоренцо; Галстян, Анаит; Ройг-Вилланова, Ирма; Мартинес-Гарсия, Хайме Ф.; Бильбао-Кастро, Хосе Р. «Полногеномная классификация и эволюционный анализ семейства транскрипционных факторов bHLH у арабидопсиса, тополя, риса, мха и водорослей». Физиология растений , 153, 3, 2010-07, стр. 1398–1412. дои:10.1104/стр.110.153593 . ISSN 0032-0889
- Цзинь Дж, Хэ К, Тан Икс, Ли З, Лу Л, Чжао Ю и др. (2015). «Карта регуляции транскрипции арабидопсиса раскрывает отличительные функциональные и эволюционные особенности новых факторов транскрипции» . Молекулярная биология и эволюция . 32 (7): 1767–73. дои : 10.1093/molbev/msv058 . ПМЦ 4476157 . ПМИД 25750178 .
Внешние ссылки
[ редактировать ]- Транскрипция + факторы Национальной медицинской библиотеки США в медицинских предметных рубриках (MeSH)
- База данных факторов транскрипции. Архивировано 4 декабря 2008 г. на Wayback Machine.