Cre рекомбиназа
| Cre рекомбиназа | |||
|---|---|---|---|
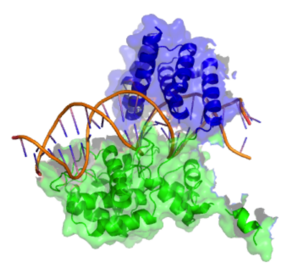
 Структура фермента рекомбиназы Cre (димера), связанного с ДНК-субстратом | |||
| Идентификаторы | |||
| Организм | |||
| Символ | кре | ||
| Входить | 2777477 | ||
| RefSeq (защита) | YP_006472.1 | ||
| ЮниПрот | P06956 | ||
| Другие данные | |||
| Номер ЕС | 2.7.7.- | ||
| хромосома | геном: 0 - 0 Мб | ||
| |||
Cre-рекомбиназа тирозинрекомбиназу, представляет собой фермент полученный из бактериофага P1 . Фермент использует механизм, подобный топоизомеразе I, для осуществления событий сайт-специфической рекомбинации . Фермент (38 кДа) является членом семейства интегразных сайт-специфических рекомбиназ, и известно, что он катализирует событие сайт-специфической рекомбинации между двумя сайтами узнавания ДНК ( сайты LoxP ). Этот сайт узнавания loxP длиной 34 пары оснований (bp) состоит из двух палиндромных последовательностей длиной 13 пар оснований , которые фланкируют спейсерную область длиной 8 bp. Продукты Cre-опосредованной рекомбинации по сайтам loxP зависят от местоположения и относительной ориентации сайтов loxP. Два отдельных вида ДНК, оба содержащие сайты loxP, могут подвергаться слиянию в результате рекомбинации, опосредованной Cre. Последовательности ДНК, обнаруженные между двумя сайтами loxP, называются « флоксированными ». В этом случае продукты рекомбинации, опосредованной Cre, зависят от ориентации сайтов loxP. ДНК, обнаруженная между двумя сайтами loxP, ориентированными в одном направлении, будет вырезана в виде кольцевой петли ДНК, в то время как промежуточная ДНК между двумя сайтами loxP, ориентированными в противоположном направлении, будет инвертирована. [1] Для своей функции фермент не требует дополнительных кофакторов (таких как АТФ ) или вспомогательных белков. [2]
Фермент играет важную роль в жизненном цикле бактериофага P1, например, в циклизации линейного генома и разрешении димерных хромосом , образующихся после репликации ДНК . [3]
Рекомбиназа Cre — широко используемый инструмент в области молекулярной биологии . Уникальная и специфическая система рекомбинации фермента используется для манипулирования генами и хромосомами в огромном диапазоне исследований, таких как нокаут генов или нокаут в исследованиях. Способность фермента эффективно работать в широком диапазоне клеточных сред (включая млекопитающих, растения, бактерии и дрожжи) позволяет использовать систему рекомбинации Cre-Lox в огромном количестве организмов, что делает ее особенно полезным инструментом в научных исследованиях. . [4]
Открытие
[ редактировать ]Исследования, проведенные в 1981 году Штернбергом и Гамильтоном, показали, что бактериофаг P1 обладает уникальной системой сайт-специфической рекомбинации. EcoRI- бактериофага P1 фрагменты генома были созданы и клонированы в лямбда-векторы . Было обнаружено, что фрагмент EcoRI размером 6,5 т.п.н. (фрагмент 7) обеспечивает эффективные события рекомбинации. [5] Известно, что механизм этих событий рекомбинации уникален, поскольку они происходят в отсутствие бактериальных белков RecA и RecBCD . Компоненты этой рекомбинационной системы были выяснены с помощью исследований делеционного мутагенеза . Эти исследования показали, что для возникновения эффективных событий рекомбинации необходимы как продукт гена P1, так и сайт рекомбинации. Продукт гена P1 был назван Cre ( вызывает рекомбинацию ) , а сайт рекомбинации — loxP ( локус кроссингинга ( x )over, P -1). [5] Белок Cre был очищен в 1983 году и оказался белком массой 35 000 Да. [2] не требуются высокоэнергетические кофакторы, такие как АТФ или вспомогательные белки. Для рекомбиназной активности очищенного белка [2] Ранние исследования также показали, что Cre связывается с неспецифическими последовательностями ДНК, обладая при этом в 20 раз более высоким сродством к последовательностям loxP, а результаты ранних исследований ДНК-следов также показали, что молекулы Cre связывают сайты loxP в виде димеров . [2]

| Члены семейства тирозинрекомбиназы [3] |
|---|
| S.cerevisiae Рекомбиназа Flp |
| Бактериальная рекомбиназа XerC |
| Бактериальная рекомбиназа XerD |
| белок λ-интегразы |
| Белок интегразы HP1 |
Структура
[ редактировать ]Рекомбиназа Cre состоит из 343 аминокислот , которые образуют два отдельных домена. Аминоконцевой сегментов , домен включает остатки 20–129 и содержит 5 альфа-спиральных связанных серией коротких петель. Спирали A и E участвуют в образовании тетрамера рекомбиназы, причем C-концевая область спирали E, как известно, образует контакты с C-концевым доменом соседних субъединиц. Спирали B и D образуют прямые контакты с большой бороздкой ДНК loxP. Считается, что эти две спирали осуществляют три прямых контакта с основаниями ДНК в сайте loxP. Карбокси -концевой домен фермента состоит из аминокислот 132–341 и содержит активный центр фермента. Общая структура этого домена имеет большое структурное сходство с каталитическим доменом других ферментов того же семейства, таких как интеграза λ и интеграза HP1. Этот домен имеет преимущественно спиральную структуру с 9 отдельными спиралями (F-N). Концевая спираль (N) выступает из основной части карбоксильного домена, и считается, что эта спираль играет роль в обеспечении взаимодействий с другими субъединицами. Кристаллические структуры демонстрируют, что эта концевая N-спираль зарывает свою гидрофобную поверхность в акцепторный карман соседней субъединицы Cre. [6]
Эффект двухдоменной структуры заключается в формировании С-образного зажима, захватывающего ДНК с противоположных сторон. [3]
Активный сайт
[ редактировать ]
Активный центр фермента Cre состоит из консервативных каталитической триады остатков Arg 173, His 289, Arg 292, а также консервативных нуклеофильных остатков Tyr 324 и Trp 315. В отличие от некоторых ферментов рекомбиназы, таких как рекомбиназа Flp, Cre не образует активный центр между отдельными субъединицами, и все остатки, входящие в активный центр, находятся на одной субъединице. Следовательно, когда две молекулы Cre связываются в одном сайте loxP, присутствуют два активных центра. Рекомбинация, опосредованная Cre, требует образования синапса, в котором два комплекса Cre-LoxP соединяются с образованием так называемого тетрамера синапса, в котором присутствуют 4 различных активных центра. [6] Tyr 324 действует как нуклеофил , образуя ковалентную 3'-фосфотирозиновую связь с ДНК-субстратом. Разделяющийся фосфат (фосфат , нацеленный на нуклеофильную атаку в сайте расщепления) координируется боковыми цепями трех аминокислотных остатков каталитической триады ( Arg 173, His 289 и Trp 315). Индольный с этим азот триптофана 315 также образует водородную связь разрезаемым фосфатом. (Примечание: Гистидин А занимает этот сайт у других членов семейства тирозинрекомбиназ и выполняет ту же функцию). Эта реакция расщепляет ДНК и освобождает 5'-гидроксильную группу. Этот процесс происходит в активном центре двух из четырех субъединиц рекомбиназы, присутствующих в тетрамере синапса. Если 5'-гидроксильные группы атакуют 3'-фосфотирозиновую связь, одна пара нитей ДНК поменяется с образованием промежуточного соединения Холлидея . [3]
Приложения
[ редактировать ]Роль в бактериофаге P1
[ редактировать ]Рекомбиназа Cre играет важную роль в жизненном цикле бактериофага P1 . При инфицировании клетки система Cre-loxP используется для вызова циркуляризации ДНК P1. В дополнение к этому Cre также используется для разделения димерной лизогенной ДНК P1, которая образуется во время клеточного деления фага. [7]
Использование в исследованиях
[ редактировать ]Индуцируемая активация Cre достигается с использованием варианта CreER (рецептора эстрогена), который активируется только после введения тамоксифена . [8] Это достигается за счет слияния мутированного лигандсвязывающего домена рецептора эстрогена с рекомбиназой Cre, в результате чего Cre специфически активируется тамоксифеном. В отсутствие тамоксифена CreER приведет к перемещению мутантной рекомбиназы в цитоплазму. Белок будет оставаться в этом месте в инактивированном состоянии до тех пор, пока не будет введен тамоксифен. После введения тамоксифена он метаболизируется в 4-гидрокситамоксифен, который затем связывается с ЭР и приводит к транслокации CreER в ядро, где он затем способен расщеплять lox-сайты. [9] Важно отметить, что иногда флуоресцентные репортеры могут активироваться в отсутствие тамоксифена из-за утечки нескольких молекул рекомбиназы Cre в ядро, что в сочетании с очень чувствительными репортерами приводит к непреднамеренному мечению клеток. [10] CreER(T2) был разработан для минимизации тамоксифен-независимой рекомбинации и максимизации чувствительности к тамоксифену.
Улучшения
[ редактировать ]В последние годы рекомбиназа Cre была улучшена за счет преобразования в предпочтительные кодоны млекопитающих , удаления известных загадочных сайтов сплайсинга , изменения стоп-кодона и снижения содержания CpG для снижения риска эпигенетического молчания у млекопитающих . [11] Также был идентифицирован ряд мутантов с повышенной точностью. [12]
См. также
[ редактировать ]- Кре-Локс рекомбинация
- Рекомбинация FLP-FRT
- Cre/loxP-System (на немецком языке)
Ссылки
[ редактировать ]- ^ Надь А. (февраль 2000 г.). «Cre-рекомбиназа: универсальный реагент для адаптации генома» . Бытие . 26 (2): 99–109. doi : 10.1002/(SICI)1526-968X(200002)26:2<99::AID-GENE1>3.0.CO;2-B . ПМИД 10686599 .
- ^ Jump up to: а б с д Абремски К., Хесс Р. (февраль 1984 г.). «Сайт-специфическая рекомбинация бактериофага P1. Очистка и свойства белка рекомбиназы Cre» . Журнал биологической химии . 259 (3): 1509–1514. дои : 10.1016/S0021-9258(17)43437-5 . ПМИД 6319400 .
- ^ Jump up to: а б с д Ван Дуйн Г.Д. (2001). «Структурный взгляд на сайт-специфическую рекомбинацию cre-loxp». Ежегодный обзор биофизики и биомолекулярной структуры . 30 : 87–104. doi : 10.1146/annurev.biophys.30.1.87 . ПМИД 11340053 .
- ^ Эннифар Э., Мейер Дж. Э., Бухгольц Ф., Стюарт А. Ф., Сак Д. (сентябрь 2003 г.). «Кристаллическая структура синапса Cre-рекомбиназа-loxP Cre дикого типа обнаруживает новую конформацию спейсера, предполагающую альтернативный механизм активации расщепления ДНК» . Исследования нуклеиновых кислот . 31 (18): 5449–5460. дои : 10.1093/нар/gkg732 . ПМК 203317 . ПМИД 12954782 .
- ^ Jump up to: а б Штернберг Н., Гамильтон Д. (август 1981 г.). «Сайт-специфическая рекомбинация бактериофага P1. I. Рекомбинация между сайтами loxP». Журнал молекулярной биологии . 150 (4): 467–486. дои : 10.1016/0022-2836(81)90375-2 . ПМИД 6276557 .
- ^ Jump up to: а б Го Ф., Гопаул Д.Н., ван Дуйн Г.Д. (сентябрь 1997 г.). «Структура рекомбиназы Cre в комплексе с ДНК в синапсе сайт-специфической рекомбинации». Природа . 389 (6646): 40–46. Бибкод : 1997Natur.389...40G . дои : 10.1038/37925 . ПМИД 9288963 . S2CID 4401434 .
- ^ Шейх А.С., Садовский П.Д. (февраль 1997 г.). «Рекомбиназа Cre расщепляет транс-сайт lox» . Журнал биологической химии . 272 (9): 5695–5702. дои : 10.1074/jbc.272.9.5695 . ПМИД 9038180 .
- ^ Уолрат Дж.К., Хоуз Дж.Дж., Ван Дайк Т., Рейли К.М. (2010). «Генетически-инженерные модели мышей в исследованиях рака» . Достижения в области исследований рака . 106 : 113–64. дои : 10.1016/S0065-230X(10)06004-5 . ISBN 9780123747716 . ПМЦ 3533445 . ПМИД 20399958 .
- ^ Кристианто Дж., Джонсон М.Г., Застроу Р.К., Рэдклифф А.Б., Бланк Р.Д. (июнь 2017 г.). «Спонтанная рекомбиназная активность Cre-ERT2 in vivo» . Трансгенные исследования . 26 (3): 411–417. дои : 10.1007/s11248-017-0018-1 . ПМЦ 9474299 . ПМИД 28409408 . S2CID 4377498 .
- ^ Альварес-Аснар А, Мартинес-Коррал И, Даубель Н, Бетсхольц К, Мякинен Т, Генгель К (февраль 2020 г.). «Линии Т2» . Трансгенные исследования . 29 (1): 53–68. дои : 10.1007/s11248-019-00177-8 . ПМЦ 7000517 . ПМИД 31641921 .
- ^ Шимшек Д.Р., Ким Дж., Хюбнер М.Р., Спергель Д.Д., Буххольц Ф., Казанова Э., Стюарт А.Ф., Зеебург П.Х., Шпренгель Р. (январь 2002 г.). «Улучшенная кодонами экспрессия рекомбиназы Cre (iCre) у мышей». Бытие . 32 (1): 19–26. дои : 10.1002/gen.10023 . ПМИД 11835670 . S2CID 46000513 .
- ^ Ерошенко Н., генеральный директор Церкви (сентябрь 2013 г.). «Мутанты рекомбиназы Cre с повышенной точностью» . Природные коммуникации . 4 : 2509. Бибкод : 2013NatCo...4.2509E . дои : 10.1038/ncomms3509 . ПМК 3972015 . ПМИД 24056590 .
Внешние ссылки
[ редактировать ]- Рекомбиназа Cre Национальной медицинской библиотеки США по медицинским предметным рубрикам (MeSH)