SNP-генотипирование
Генотипирование SNP — это измерение генетических вариаций однонуклеотидных полиморфизмов (SNP) между представителями одного вида. Это форма генотипирования , которая представляет собой измерение более общих генетических вариаций. SNP являются одним из наиболее распространенных типов генетических вариаций. SNP — это мутация одной пары оснований в определенном локусе , обычно состоящая из двух аллелей (где частота редких аллелей составляет > 1%). Обнаружено, что SNP участвуют в этиологии многих заболеваний человека и вызывают особый интерес в фармакогенетике . Поскольку SNP консервативны в ходе эволюции, они были предложены в качестве маркеров для использования в анализе локусов количественных признаков ( QTL ) и в исследованиях ассоциаций вместо микросателлитов . Использование SNP расширяется в проекте HapMap , целью которого является предоставление минимального набора SNP, необходимого для генотипирования генома человека. SNP также могут предоставить генетический отпечаток пальца для использования при проверке личности. [1] Рост интереса к SNP отразился в бурном развитии разнообразных методов генотипирования SNP.
Методы на основе гибридизации
[ редактировать ]Было разработано несколько приложений, которые опрашивают SNP путем гибридизации дополнительных зондов ДНК с сайтом SNP. Задача этого подхода заключается в уменьшении перекрестной гибридизации между аллель-специфичными зондами. Эту проблему обычно решают путем манипулирования условиями жесткости гибридизации. [1]
Динамическая аллель-специфическая гибридизация
[ редактировать ]
Генотипирование динамической аллель-специфической гибридизации (DASH) использует различия в температуре плавления ДНК, возникающие в результате нестабильности несовпадающих пар оснований. Этот процесс может быть значительно автоматизирован и включает в себя несколько простых принципов. [ нужна ссылка ]
На первом этапе геномный сегмент амплифицируется и прикрепляется к шарику посредством реакции ПЦР с биотинилированным праймером. На втором этапе амплифицированный продукт прикрепляют к колонке со стрептавидином и промывают NaOH для удаления небиотинилированной цепи. Затем добавляют аллель -специфичный олигонуклеотид в присутствии молекулы, которая флуоресцирует при связывании с двухцепочечной ДНК. Затем интенсивность измеряют по мере повышения температуры до тех пор, пока температура плавления (Tm) не будет определена . SNP приведет к более низкому, чем ожидалось, Tm. [2]
Поскольку генотипирование DASH измеряет количественные изменения Tm, оно позволяет измерять все типы мутаций, а не только SNP. Другие преимущества DASH включают возможность работы с датчиками без меток, а также простоту конструкции и условий эксплуатации. [ нужна ссылка ]
Молекулярные маяки
[ редактировать ]
Для обнаружения SNP с помощью молекулярных маяков используется специально разработанный одноцепочечный олигонуклеотидный зонд. Олигонуклеотид сконструирован таким образом, что на каждом конце имеются комплементарные области, а между ними расположена последовательность зонда. Такая конструкция позволяет зонду принимать структуру шпильки или петли-стержня в его естественном, изолированном состоянии. К одному концу зонда прикреплен флуорофор, а к другому - тушитель флуоресценции. Благодаря петлевой структуре зонда флуорофор находится близко к тушителю, что предотвращает излучение молекулой флуоресценции. Молекула также сконструирована таким образом, что только последовательность зонда комплементарна геномной ДНК, которая будет использоваться в анализе (Abravaya et al. 2003).
Если во время анализа последовательность зонда молекулярного маяка встретит целевую геномную ДНК, она отожжется и гибридизуется. Из-за длины последовательности зонда шпильчатый сегмент зонда будет денатурирован в пользу образования более длинного и стабильного гибрида зонд-мишень. Это конформационное изменение позволяет флуорофору и тушителю освободиться от их тесного соседства из-за ассоциации шпилек, что позволяет молекуле флуоресцировать.
Если, с другой стороны, последовательность зонда встречает целевую последовательность, содержащую всего лишь один некомплементарный нуклеотид, молекулярный маяк будет преимущественно оставаться в своем естественном состоянии шпильки, и флуоресценция не будет наблюдаться, поскольку флуорофор остается погашенным.
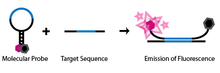
Уникальная конструкция этих молекулярных маяков позволяет с помощью простого диагностического анализа идентифицировать SNP в заданном месте. Если молекулярный маяк спроектирован так, чтобы соответствовать аллели дикого типа, а другой — для соответствия мутанту аллели, оба могут быть использованы для идентификации генотипа человека. Если во время анализа обнаруживается только длина волны флуорофора первого зонда, значит, человек гомозиготен по отношению к дикому типу. Если обнаруживается только длина волны второго зонда, значит, человек гомозиготен по мутантному аллелю. Наконец, если обнаружены обе длины волн, то оба молекулярных маяка должны гибридизоваться со своими комплементами, и, таким образом, человек должен содержать обе аллели и быть гетерозиготным.
SNP-микрочипы
[ редактировать ]В массивах SNP олигонуклеотидов высокой плотности сотни тысяч зондов расположены на небольшом чипе, что позволяет одновременно опрашивать множество SNP. [1] Поскольку аллели SNP различаются только одним нуклеотидом и трудно достичь оптимальных условий гибридизации для всех зондов в массиве, целевая ДНК может гибридизоваться с несовпадающими зондами. Эту проблему частично решают путем использования нескольких резервных зондов для опроса каждого SNP. Зонды предназначены для обнаружения сайта SNP в нескольких разных местах, а также для обнаружения несовпадений с аллелем SNP. Сравнивая разную степень гибридизации целевой ДНК с каждым из этих избыточных зондов, можно определить конкретные гомозиготные и гетерозиготные аллели. [1] Хотя олигонуклеотидные микрочипы имеют сравнительно более низкую специфичность и чувствительность, масштаб SNP, которые можно исследовать, является основным преимуществом. Генный чип Affymetrix Human SNP 5.0 выполняет полногеномный анализ, который позволяет генотипировать более 500 000 человеческих SNP (Affymetrix 2007).
Ферментные методы
[ редактировать ]Широкий спектр ферментов, включая ДНК-лигазу , ДНК-полимеразу и нуклеазы, использовался для создания высокоточных методов генотипирования SNP.
Полиморфизм длины рестрикционных фрагментов
[ редактировать ]Полиморфизм длины рестрикционных фрагментов (ПДРФ) считается самым простым и ранним методом обнаружения SNP. SNP-RFLP использует множество различных эндонуклеаз рестрикции и их высокое сродство к уникальным и специфическим сайтам рестрикции. Выполняя расщепление геномного образца и определяя длину фрагментов с помощью анализа в геле, можно установить, разрезают ли ферменты ожидаемые сайты рестрикции. Неспособность разрезать геномный образец приводит к получению заметно большего, чем ожидалось, фрагмента, что означает наличие мутации в месте сайта рестрикции, которая обеспечивает его защиту от активности нуклеазы.
Сочетание таких факторов, как высокая сложность большинства эукариотических геномов, потребность в специфических эндонуклеазах, тот факт, что точную мутацию не обязательно можно определить в одном эксперименте, а также медленный характер гелевых анализов, делают ПДРФ плохим выбором для высокопроизводительного анализа.
Методы на основе ПЦР
[ редактировать ]В системе ПЦР с устойчивыми к мутациям тетрапраймерной амплификации, или ARMS-PCR, используются две пары праймеров для амплификации двух аллелей в одной реакции ПЦР. Праймеры сконструированы таким образом, что две пары праймеров перекрываются в месте расположения SNP, но каждый из них идеально соответствует только одному из возможных SNP. В основе изобретения неожиданно оказалось, что олигонуклеотиды с несовпадающим 3'-остатком не будут функционировать в качестве праймеров в ПЦР в соответствующих условиях. [3] В результате, если в реакции ПЦР присутствует данный аллель, пара праймеров, специфичная для этого аллеля, будет производить продукт, но не для альтернативного аллеля с другим SNP. Две пары праймеров также сконструированы таким образом, что их продукты ПЦР имеют существенно разную длину, что позволяет легко различить полосы с помощью гель-электрофореза или анализа температуры плавления. [4] [5] При изучении результатов, если образец генома является гомозиготным, то полученные продукты ПЦР будут получены из праймера, соответствующего расположению SNP, и внешнего праймера противоположной цепи, а также из двух внешних праймеров. Если геномный образец является гетерозиготным, то продукты будут результатом праймера каждой аллели и их соответствующих аналогов внешнего праймера, а также внешних праймеров.
Альтернативная стратегия — провести несколько реакций qPCR с разными наборами праймеров, нацеленными на каждую аллель отдельно. Хорошо разработанные праймеры будут амплифицировать целевой SNP в гораздо более раннем цикле, чем другие SNP. Это позволяет различить более двух аллелей, хотя для каждого SNP требуется отдельная реакция qPCR. Для достижения достаточно высокой специфичности последовательность праймера может потребовать размещения искусственного несовпадения рядом с ее 3'-концом, что представляет собой подход, широко известный как Taq-MAMA. [6]
Лоскутная эндонуклеаза
[ редактировать ]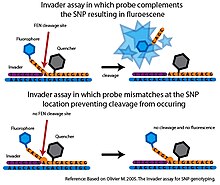
Эндонуклеаза лоскута (FEN) представляет собой эндонуклеазу, которая катализирует структурно-специфическое расщепление. Это расщепление очень чувствительно к несовпадениям и может использоваться для опроса SNP с высокой степенью специфичности. [7]
В базовом Invader анализе FEN, называемый расщепляющей, комбинируется с двумя специфическими олигонуклеотидными зондами, которые вместе с целевой ДНК могут образовывать трехчастную структуру, распознаваемую расщепляющей. [7] Первый зонд, названный олигонуклеотидом Invader , комплементарен 3'-концу целевой ДНК. Последнее основание олигонуклеотида Invader представляет собой несовпадающее основание, которое перекрывает нуклеотид SNP в целевой ДНК. Второй зонд представляет собой аллель-специфичный зонд, который комплементарен 5'-концу целевой ДНК, но также проходит за 3'-сторону нуклеотида SNP. Аллель-специфичный зонд будет содержать основание, комплементарное нуклеотиду SNP. Если целевая ДНК содержит желаемый аллель, зонды Invader и аллель-специфические связываются с целевой ДНК, образуя трехчастную структуру. Эта структура распознается расщепляющей кислотой, которая расщепляет и высвобождает 3'-конец аллель-специфического зонда. Если нуклеотид SNP в целевой ДНК не комплементарен аллель-специфичному зонду, правильная трехчастная структура не формируется и расщепления не происходит. Анализ Invader обычно сочетается с системой флуоресцентного резонансного переноса энергии (FRET) для обнаружения события расщепления. В этой схеме молекула-гаситель присоединяется к 3'-концу, а флуорофор - к 5'-концу аллель-специфического зонда. Если произойдет расщепление, флуорофор отделится от молекулы-гасителя, генерируя обнаруживаемый сигнал. [7]
При использовании несовпадающих зондов происходит лишь минимальное расщепление, что делает анализ Invader высокоспецифичным. Однако в исходном формате в каждом реакционном образце можно было опросить только один аллель SNP, и для генерации обнаружимого сигнала в разумные сроки требовалось большое количество целевой ДНК. [7] Несколько разработок расширили первоначальный анализ Invader . Проводя вторичные реакции расщепления FEN, реакция последовательного инвазивного усиления сигнала (SISAR) позволяет опрашивать оба аллеля SNP в одной реакции. Для анализа SISAR Invader также требуется меньше целевой ДНК, что повышает чувствительность оригинального анализа Invader . [7] Анализ также был адаптирован несколькими способами для использования в формате с высокой пропускной способностью. На одной платформе аллель-специфичные зонды прикреплены к микросферам. Когда расщепление FEN генерирует обнаруживаемый флуоресцентный сигнал, сигнал измеряют с помощью проточной цитометрии. Чувствительность проточной цитометрии исключает необходимость ПЦР-амплификации целевой ДНК (Rao et al. 2003). Эти высокопроизводительные платформы не продвинулись дальше стадии проверки принципа действия, и до сих пор система Invader не использовалась ни в каких крупномасштабных проектах генотипирования SNP. [7]
Расширение праймера
[ редактировать ]Расширение праймера — это двухэтапный процесс, который сначала включает гибридизацию зонда с основаниями, расположенными непосредственно перед нуклеотидом SNP, с последующей реакцией «мини-секвенирования», в которой ДНК-полимераза удлиняет гибридизованный праймер путем добавления основания, комплементарного нуклеотид SNP. Это включенное основание обнаруживается и определяет аллель SNP (Goelet et al. 1999; Syvanen 2001). Поскольку расширение праймера основано на использовании высокоточного фермента ДНК-полимеразы, этот метод, как правило, очень надежен. Расширение праймера позволяет генотипировать большинство SNP в очень похожих условиях реакции, что делает его также очень гибким. Метод расширения праймера используется во многих форматах анализа. В этих форматах используется широкий спектр методов обнаружения, включая масс-спектрометрию MALDI-TOF (см. Sequenom ) и методы, подобные ELISA . [1]
Как правило, существует два основных подхода, в которых используется включение либо флуоресцентно меченных дидезоксинуклеотидов (ddNTP), либо флуоресцентно меченных дезоксинуклеотидов (dNTP). При использовании ddNTP зонды гибридизуются с целевой ДНК непосредственно перед нуклеотидом SNP, и к 3'-концу зонда добавляется одиночный ddNTP, комплементарный аллели SNP (отсутствующий 3'-гидроксил в дидиоксинуклеотиде предотвращает добавление дополнительных нуклеотидов). ). Каждый ddNTP помечен различным флуоресцентным сигналом, что позволяет обнаружить все четыре аллели в одной реакции. В случае dNTP аллель-специфичные зонды имеют 3'-основания, которые комплементарны каждому из опрашиваемых аллелей SNP. Если целевая ДНК содержит аллель, комплементарную 3'-основанию зонда, целевая ДНК полностью гибридизуется с зондом, позволяя ДНК-полимеразе выходить из 3'-конца зонда. Это обнаруживается путем включения флуоресцентно меченных dNTP на конец зонда. Если целевая ДНК не содержит аллель, комплементарную 3'-основанию зонда, целевая ДНК будет образовывать несоответствие на 3'-конце зонда, и ДНК-полимераза не сможет выходить из 3'-конца зонда. Преимущество второго подхода заключается в том, что несколько меченых dNTP могут быть включены в растущую цепь, что позволяет усилить сигнал. Однако в некоторых редких случаях ДНК-полимераза может распространяться из несовпадающих 3'-зондов, что дает ложноположительный результат. [1]
Другой подход используется в Sequenom методе генотипирования SNP iPLEX компании , в котором используется масс-спектрометр MassARRAY. Удлинительные зонды сконструированы таким образом, что можно амплифицировать и анализировать 40 различных анализов SNP в ПЦР-коктейле. В реакции удлинения используются ddNTP, как указано выше, но обнаружение аллеля SNP зависит от фактической массы продукта удлинения, а не от флуоресцентной молекулы. Этот метод рассчитан на низкую и средневысокую производительность и не предназначен для сканирования всего генома.
Гибкость и специфичность удлинения праймера делают его пригодным для высокопроизводительного анализа. Зонды расширения праймера можно расположить на предметных стеклах, что позволяет одновременно генотипировать множество SNP. Эта технология, широко называемая расширением массивных праймеров (APEX), имеет ряд преимуществ по сравнению с методами, основанными на дифференциальной гибридизации зондов. Для сравнения, методы APEX обладают большей дискриминационной способностью, чем методы, использующие эту дифференциальную гибридизацию, поскольку часто невозможно получить оптимальные условия гибридизации для тысяч зондов на микрочипах ДНК (обычно это решается за счет наличия сильно избыточных зондов). Однако такая же плотность зондов не может быть достигнута в методах APEX, что приводит к снижению производительности за один цикл. [1]
Illumina компании Анализ Infinium Incorporated является примером конвейера полногеномного генотипирования, основанного на методе расширения праймеров. С помощью анализа Infinium можно генотипировать более 100 000 SNP. В анализе используются нуклеотиды, меченные гаптенами, в реакции удлинения праймера. Гаптеновая метка распознается антителами, которые, в свою очередь, связываются с обнаруживаемым сигналом (Gunderson et al. 2006).
APEX-2 представляет собой метод генотипирования с расширением праймеров, который позволяет параллельно идентифицировать сотни SNP или мутаций с использованием эффективной гомогенной мультиплексной ПЦР (до 640 плексов) и четырехцветного одноосновательного расширения на микрочипе. Для мультиплексной ПЦР требуется два олигонуклеотида на каждый SNP/ампликон, генерирующий мутацию, которые содержат тестируемую пару оснований. Те же олигонуклеотиды используются на следующем этапе в качестве иммобилизованных одноосновных праймеров удлинения на микрочипе (Крюцков и др., 2008).
5'-нуклеаза
[ редактировать ]5'-нуклеазная активность ДНК-полимеразы Taq используется в анализе TaqMan для генотипирования SNP. Анализ TaqMan выполняется одновременно с реакцией ПЦР, и результаты можно считывать в режиме реального времени по мере прохождения реакции ПЦР (McGuigan & Ralston 2002). Для анализа требуются праймеры для прямой и обратной ПЦР, которые будут амплифицировать область, включающую полиморфный сайт SNP. Распознавание аллелей достигается с использованием FRET в сочетании с одним или двумя аллель-специфичными зондами, которые гибридизуются с полиморфным сайтом SNP. Зонды будут иметь флуорофор, связанный с их 5'-концом, и молекулу-гаситель, связанную с их 3'-концом. Пока зонд не поврежден, тушитель будет оставаться в непосредственной близости от флуорофора, подавляя сигнал флуорофора. Если на этапе ПЦР-амплификации аллель-специфичный зонд полностью комплементарен аллелю SNP, он связывается с целевой цепью ДНК, а затем разрушается под действием 5'-нуклеазной активности Taq-полимеразы, поскольку он удлиняет ДНК из ПЦР. грунтовки. Деградация зонда приводит к отделению флуорофора от молекулы-гасителя, генерируя обнаруживаемый сигнал. Если аллель-специфичный зонд не является полностью комплементарным, он будет иметь более низкую температуру плавления и не будет столь эффективно связываться. Это предотвращает воздействие нуклеазы на зонд (McGuigan & Ralston 2002).
Поскольку анализ TaqMan основан на ПЦР, его относительно просто реализовать. Анализ TaqMan можно мультиплексировать, объединяя обнаружение до семи SNP в одной реакции. Однако, поскольку для каждого SNP требуется отдельный зонд, анализ TaqMan ограничен тем, насколько близко могут располагаться SNP. Масштаб анализа можно значительно увеличить, проводя множество одновременных реакций в микротитровальных планшетах. Как правило, TaqMan ограничен приложениями, которые включают опрос небольшого количества SNP, поскольку для каждого SNP необходимо разработать оптимальные зонды и условия реакции (Syvanen 2001).
Анализ лигирования олигонуклеотидов
[ редактировать ]ДНК-лигаза катализирует связывание 3'-конца фрагмента ДНК с 5'-концом непосредственно соседнего фрагмента ДНК. Этот механизм можно использовать для опроса SNP путем гибридизации двух зондов непосредственно над полиморфным сайтом SNP, при этом лигирование может происходить, если зонды идентичны целевой ДНК. В анализе олигонуклеотид-лигазы разработаны два зонда; аллель-специфичный зонд, который гибридизуется с целевой ДНК так, что его 3'-основание расположено непосредственно над нуклеотидом SNP, и второй зонд, который гибридизует матрицу выше (ниже в комплементарной цепи) полиморфного сайта SNP, обеспечивая 5'-конец для реакции лигирования. Если аллель-специфичный зонд соответствует целевой ДНК, он полностью гибридизуется с целевой ДНК, и может произойти лигирование. Лигирование обычно не происходит при наличии несовпадающего 3'-основания. Лигированные или нелигированные продукты можно обнаружить с помощью гель-электрофореза, масс-спектрометрии MALDI-TOF или капиллярного электрофореза для крупномасштабных применений. [1] При наличии соответствующих последовательностей и меток на олигонуклеотидах можно с высокой производительностью получать данные о последовательностях на основе продуктов лигирования и определения генотипов (Curry et al., 2012). Использование большого количества индексов образцов позволяет генерировать высокопроизводительные данные о последовательностях сотен SNP в тысячах образцов за небольшую часть высокопроизводительного секвенирования. Это массовое генотипирование с помощью технологии секвенирования (MGST). [ нужна ссылка ]
Другие методы постамплификации, основанные на физических свойствах ДНК.
[ редактировать ]Характерные свойства ДНК, такие как температура плавления и одноцепочечная конформация, использовались в нескольких приложениях для различения аллелей SNP. Эти методы очень часто достигают высокой специфичности, но требуют высокооптимизированных условий для получения наилучших возможных результатов.
Одноцепочечный конформационный полиморфизм
[ редактировать ]Одноцепочечная ДНК (оцДНК) сворачивается в третичную структуру. Конформация зависит от последовательности, и большинство мутаций одной пары оснований изменяют форму структуры. При нанесении на гель третичная форма будет определять подвижность оцДНК, обеспечивая механизм дифференциации аллелей SNP. Этот метод сначала включает ПЦР-амплификацию целевой ДНК. Двухцепочечные продукты ПЦР денатурируются с использованием тепла и формальдегида для получения оцДНК. ОцДНК наносят на неденатурирующий гель для электрофореза и позволяют сложиться в третичную структуру. Различия в последовательности ДНК изменят третичную конформацию и будут обнаружены как разница в подвижности цепей оцДНК (Costabile et al. 2006). Этот метод широко используется, поскольку он технически прост, относительно недорог и использует общедоступное оборудование. Однако по сравнению с другими методами генотипирования SNP чувствительность этого анализа ниже. Было обнаружено, что конформация оцДНК сильно зависит от температуры, и обычно неясно, какова идеальная температура. Очень часто анализ проводится с использованием нескольких разных температур. Также существует ограничение на длину фрагмента, поскольку чувствительность падает при использовании последовательностей длиной более 400 п.о. (Cosabile et al. 2006).
Гель-электрофорез в градиенте температуры
[ редактировать ]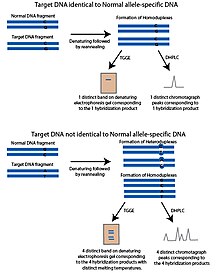
Метод гель-электрофореза в температурном градиенте (TGGE) или капиллярного электрофореза в температурном градиенте (TGCE) основан на том принципе, что частично денатурированная ДНК более ограничена и движется медленнее в пористом материале, таком как гель. Это свойство позволяет разделять ДНК по температуре плавления. Чтобы адаптировать эти методы для обнаружения SNP, используются два фрагмента; ДНК-мишень, которая содержит опрашиваемый полиморфный сайт SNP и аллель-специфическую последовательность ДНК, называемую нормальным фрагментом ДНК. Нормальный фрагмент идентичен целевой ДНК, за исключением потенциально полиморфного сайта SNP, который неизвестен в целевой ДНК. Фрагменты денатурируют, а затем повторно отжигают. Если целевая ДНК имеет тот же аллель, что и нормальный фрагмент, образуются гомодуплексы, которые будут иметь ту же температуру плавления. При работе на геле с градиентом температуры появится только одна полоса. Если целевая ДНК имеет отдельный аллель, после этапа повторного отжига образуются четыре продукта; гомодуплексы, состоящие из целевой ДНК, гомодуплексы, состоящие из нормальной ДНК, и два гетеродуплекса каждой цепи целевой ДНК, гибридизованной с нормальной цепью ДНК. Эти четыре продукта будут иметь разные температуры плавления и будут выглядеть в денатурирующем геле в виде четырех полос. [1]
Денатурирующая высокоэффективная жидкостная хроматография
[ редактировать ]Денатурирующая высокоэффективная жидкостная хроматография (ДВЭЖХ) использует обращенно-фазовую ВЭЖХ для исследования SNP. Ключом к ДВЭЖХ является твердая фаза, которая имеет различное сродство к одно- и двухцепочечной ДНК. При ДВЭЖХ фрагменты ДНК денатурируются путем нагревания, а затем повторно отжигаются. Температура плавления повторно отожженных фрагментов ДНК определяет продолжительность их удержания в колонке. [8] С помощью ПЦР генерируют два фрагмента; ДНК-мишень, содержащая полиморфный сайт SNP и аллель-специфическую последовательность ДНК, называемую нормальным фрагментом ДНК. Этот нормальный фрагмент идентичен целевой ДНК, за исключением потенциально полиморфного сайта SNP, который неизвестен в целевой ДНК. Фрагменты денатурируют, а затем постепенно отжигают. Реанналированные продукты добавляют в колонку ДВЭЖХ. Если аллель SNP в целевой ДНК соответствует нормальному фрагменту ДНК, на этапе повторного отжига будут образовываться только идентичные гомодуплексы. Если целевая ДНК содержит аллель SNP, отличный от нормального фрагмента ДНК, в дополнение к гомодуплексам будут образовываться гетеродуплексы целевой ДНК и нормальной ДНК, содержащей несовпадающий полиморфный сайт. Несовпадающие гетеродуплексы будут иметь другую температуру плавления, чем гомодуплексы, и не будут удерживаться в колонке так долго. Это создает хроматографическую картину, которая отличается от картины, которая была бы создана, если бы целевой фрагмент ДНК и нормальные фрагменты ДНК были идентичны. Элюированную ДНК обнаруживают по УФ-поглощению. [8]
ДВЭЖХ легко автоматизируется, поскольку не требуется маркировка или очистка фрагментов ДНК. Метод также относительно быстр и обладает высокой специфичностью. Одним из основных недостатков ДВЭЖХ является то, что температура колонки должна быть оптимизирована для каждой мишени, чтобы достичь нужной степени денатурации. [1]
Плавление всего ампликона с высоким разрешением
[ редактировать ]Анализ плавления с высоким разрешением — самый простой для понимания метод на основе ПЦР. По сути, здесь применимы те же термодинамические свойства, которые позволили работать гелевым методам, причем в режиме реального времени. Флуориметр контролирует денатурацию всего ампликона дцДНК после ПЦР. Вы создаете праймеры, специфичные для сайта, который хотите амплифицировать. Вы «окрашиваете» ампликон двухцепочечным красителем, включенным в смесь для ПЦР. Ds-специфичный краситель интегрируется в продукт ПЦР. По сути, весь ампликон становится зондом. Это открывает новые возможности для открытий. Либо вы размещаете праймеры очень близко к любой стороне рассматриваемого SNP (генотипирование малых ампликонов, Liew, 2004), либо амплифицируете более крупный участок (длиной 100-400 пар оснований) для целей сканирования. Для простого генотипирования SNP проще сделать ампликон небольшим, чтобы минимизировать вероятность того, что вы перепутаете один SNP с другим. Определяют температуру плавления (Tm) всего ампликона, и большинство гомозигот достаточно сильно отличаются (в лучших инструментах) по Tm от генотипа. Гетерозиготы еще легче дифференцировать, поскольку у них образуются гетеродуплексы (см. объяснения на основе геля), которые уширяют переход плавления и обычно дают два различимых пика. Описано плавление ампликона с использованием флуоресцентно-меченного праймера (Gundry et al., 2003), но оно менее практично, чем использование ds-специфичных красителей из-за стоимости флуорогенного праймера.
Сканирование более крупных ампликонов основано на тех же принципах, которые изложены выше. Однако информативными становятся температура плавления и общая форма кривой плавления. Для ампликонов >c.150bp часто имеется >2 пиков плавления, каждый из которых может варьироваться в зависимости от состава матрицы ДНК. Многим исследователям удалось успешно исключить большую часть секвенирования с помощью сканирования на основе плавления, что позволило провести точное локусное генотипирование большого количества людей. [9] Многие исследователи обнаружили, что сканирование мутаций с использованием плавления с высоким разрешением является жизнеспособным и практичным способом изучения целых генов.
Использование белков, связывающих несовпадения ДНК
[ редактировать ]Белки, связывающие несовпадения ДНК, могут различать несовпадения отдельных нуклеотидов и, таким образом, облегчать дифференциальный анализ SNP. Например, белок MutS из Thermus aquaticus связывает различные одиночные нуклеотидные несоответствия с разным сродством и может использоваться в капиллярном электрофорезе для дифференциации всех шести наборов несовпадений (Drabovich & Krylov 2006).
СНПлекс
[ редактировать ]SNPlex — это запатентованная платформа генотипирования, продаваемая Applied Biosystems .
Нуклеазный анализ Surveyor
[ редактировать ]Нуклеаза Surveyor представляет собой фермент эндонуклеазу ошибочного спаривания, который распознает все замены оснований и небольшие вставки/делеции (инделеции) и расщепляет 3'-сторону несовпадающих участков в обеих цепях ДНК.
Секвенирование
[ редактировать ]Технологии секвенирования следующего поколения, такие как пиросеквенирование последовательности менее 250 оснований за одно считывание, что ограничивает их способность секвенировать целые геномы. Однако их способность генерировать результаты в режиме реального времени и возможность масштабного масштабирования делают их жизнеспособным вариантом для секвенирования небольших регионов для выполнения генотипирования SNP. По сравнению с другими методами генотипирования SNP, секвенирование особенно подходит для идентификации нескольких SNP в небольшой области, такой как высокополиморфная область главного комплекса гистосовместимости генома. [1]
Ссылки
[ редактировать ]- ^ Jump up to: а б с д и ж г час я дж к Харброн С., Рэпли Р. (2004). Молекулярный анализ и открытие генома . John Wiley & Sons Ltd. Лондон: ISBN 978-0-471-49919-0 .
- ^ Хауэлл В.М., Джобс М., Джилленстен Ю., Брукс А.Дж. (январь 1999 г.). «Динамическая аллель-специфическая гибридизация. Новый метод оценки однонуклеотидных полиморфизмов». Природная биотехнология . 17 (1): 87–88. дои : 10.1038/5270 . ПМИД 9920276 . S2CID 37367067 .
- ^ Ньютон С.Р., Грэм А., Хептинсталл Л.Е., Пауэлл С.Дж., Саммерс С., Калшекер Н. и др. (апрель 1989 г.). «Анализ любой точечной мутации в ДНК. Система устойчивых к амплификации мутаций (ARMS)» . Исследования нуклеиновых кислот . 17 (7): 2503–2516. дои : 10.1093/нар/17.7.2503 . ПМК 317639 . ПМИД 2785681 .
- ^ Булдук Б.К., Килич Х.Б., Бекирджан-Курт С.Э., Халилоглу Г., Эрдем Оздамар С., Топалоглу Х., Коджафе Йч (март 2020 г.). «Новая стратегия амплификации-резистентной мутации и ПЦР для скрининга патогенных вариантов MT-TL1 в хранилищах пациентов». Генетическое тестирование и молекулярные биомаркеры . 24 (3): 165–170. дои : 10.1089/gtmb.2019.0079 . ПМИД 32167396 . S2CID 212693790 .
- ^ Бердселл Д.Н., Пирсон Т., Прайс Э.П., Хорнстра Х.М., Нера Р.Д., Стоун Н. и др. (2012). «Анализ расплава анализов мутаций амплификации несоответствия (Melt-MAMA): функциональное исследование экономичного анализа генотипирования SNP на бактериальных моделях» . ПЛОС ОДИН . 7 (3): e32866. дои : 10.1371/journal.pone.0032866.s002 . ПМЦ 3306377 . ПМИД 22438886 .
- ^ Ли Б, Кадура И, Фу DJ, Уотсон Д.Э. (февраль 2004 г.). «Генотипирование с помощью TaqMAMA». Геномика . 83 (2): 311–320. дои : 10.1016/j.ygeno.2003.08.005 . ПМИД 14706460 .
- ^ Jump up to: а б с д и ж Оливье М. (июнь 2005 г.). «Анализ Invader для генотипирования SNP» . Мутационные исследования . 573 (1–2): 103–110. дои : 10.1016/j.mrfmmm.2004.08.016 . ПМЦ 2771639 . ПМИД 15829241 .
- ^ Jump up to: а б Офнер П.Дж., Андерхилл, Пенсильвания (1995). «Сравнительное секвенирование ДНК методом денатурирующей высокоэффективной жидкостной хроматографии (ДВЭЖХ)». Ам Джей Хум Жене . 57 : 103–10.
- ^ Данг XD, Келлехер CT, Ховард-Уильямс Э., резюме Мида (сентябрь 2012 г.). «Быстрая идентификация гаплотипов хлоропластов с использованием анализа плавления с высоким разрешением» (PDF) . Ресурсы молекулярной экологии . 12 (5): 894–908. дои : 10.1111/j.1755-0998.2012.03164.x . ПМИД 22783911 . S2CID 8384592 .
Дальнейшее чтение
[ редактировать ]- Абравая К., Хафф Дж., Маршалл Р., Мерчант Б., Маллен С., Шнайдер Г., Робинсон Дж. (апрель 2003 г.). «Молекулярные маяки как диагностические инструменты: технология и применение». Клиническая химия и лабораторная медицина . 41 (4): 468–474. дои : 10.1515/CCLM.2003.070 . ПМИД 12747588 . S2CID 36220311 .
- Костабиле М., Куах А., Ферранте А. (декабрь 2006 г.). «Молекулярные подходы в диагностике первичных иммунодефицитных заболеваний» . Человеческая мутация . 27 (12): 1163–1173. дои : 10.1002/humu.20412 . ПМК 7165860 . ПМИД 16960849 .
- Драбович А.П., Крылов С.Н. (март 2006 г.). «Идентификация пар оснований в однонуклеотидных полиморфизмах с помощью капиллярного электрофореза, опосредованного белком MutS». Аналитическая химия . 78 (6): 2035–2038. дои : 10.1021/ac0520386 . ПМИД 16536443 .
- Гандри К.Н., Вандерстин Дж.Г., Рид Г.Х., Прайор Р.Дж., Чен Дж., Виттвер К.Т. (март 2003 г.). «Анализ плавления ампликонов с мечеными праймерами: метод закрытой пробирки для дифференциации гомозигот и гетерозигот» . Клиническая химия . 49 (3): 396–406. дои : 10.1373/49.3.396 . ПМИД 12600951 .
- Гундерсон К.Л., Стимерс Ф.Дж., Рен Х., Нг П., Чжоу Л., Цан С. и др. (2006). Полногеномное генотипирование . Методы энзимологии. Том. 410. стр. 359–376. дои : 10.1016/S0076-6879(06)10017-8 . ISBN 9780121828158 . ПМИД 16938560 .
- Лью М., Прайор Р., Пале Р., Медоуз С., Эрали М., Лион Э., Виттвер С. (июль 2004 г.). «Генотипирование однонуклеотидных полиморфизмов путем плавления небольших ампликонов с высоким разрешением» . Клиническая химия . 50 (7): 1156–1164. дои : 10.1373/clinchem.2004.032136 . ПМИД 15229148 .
- Макгиган Ф.Е., Ралстон С.Х. (сентябрь 2002 г.). «Обнаружение однонуклеотидного полиморфизма: аллельная дискриминация с использованием TaqMan». Психиатрическая генетика . 12 (3): 133–136. дои : 10.1097/00041444-200209000-00003 . ПМИД 12218656 .
- Сювенен AC (декабрь 2001 г.). «Доступ к генетическим вариациям: генотипирование однонуклеотидных полиморфизмов». Обзоры природы. Генетика . 2 (12): 930–942. дои : 10.1038/35103535 . ПМИД 11733746 . S2CID 15411761 .
Внешние ссылки
[ редактировать ]- Международный проект HapMap. Архивировано 16 апреля 2014 г. в Wayback Machine.
- Проект персонализированной медицины долины Делавэр использует SNP, чтобы сделать медицину индивидуальной
- Генотипирование с помощью анализа лигирования олигонуклеотидов (OLA)
- Зачем SNP тестирует ваших мышей?