Топологически связывающая область

( Топологически ассоциированный домен TAD) представляет собой самодействующую геномную область, а это означает, что последовательности ДНК внутри TAD физически взаимодействуют друг с другом чаще, чем с последовательностями вне TAD. [1] Средний размер TAD в клетках мышей составляет 880 т.п.н. , и они имеют аналогичные размеры у видов немлекопитающих. [2] Границы по обе стороны этих доменов консервативны между разными типами клеток млекопитающих и даже между видами. [2] и высоко обогащены CCCTC-связывающим фактором (CTCF) и когезином . [1] Кроме того, некоторые типы генов (например, гены транспортных РНК и гены «домашнего хозяйства» ) появляются вблизи границ TAD чаще, чем можно было бы ожидать случайно. [3] [4]
Функции TAD до конца не изучены и до сих пор являются предметом дискуссий. Большинство исследований показывают, что TAD регулируют экспрессию генов , ограничивая взаимодействие энхансер - промотор для каждого TAD; [5] однако недавнее исследование разъединяет организацию TAD и экспрессию генов. [6] Установлено, что нарушение границ TAD связано с широким спектром заболеваний, таких как рак , [7] [8] [9] различные пороки развития конечностей, такие как синполидактилия , синдром Кука и F-синдром, [10] и ряд заболеваний головного мозга, таких как гипопластическое мозолистое тело и демиелинизирующая лейкодистрофия у взрослых. [10]
ряд белковых комплексов Механизмы, лежащие в основе формирования TAD, также сложны и еще полностью не выяснены, хотя с границами TAD связан и элементов ДНК. Однако модель наручников и модель экструзии петли описывают образование TAD с помощью белков CTCF и когезина. [11] Более того, было высказано предположение, что жесткость границ TAD сама по себе может вызывать изоляцию доменов и образование TAD. [11]
Открытия и разнообразие
[ редактировать ]TAD определяются как области, последовательности ДНК которых преимущественно контактируют друг с другом. Они были обнаружены в 2012 году с использованием методов захвата конформации хромосом, включая Hi-C . [3] [12] [4] Было показано, что они присутствуют у нескольких видов, [13] в том числе плодовые мухи ( дрозофилы ), [14] мышь , [3] растения, грибы и человек [4] геномы. У бактерий они называются хромосомными взаимодействующими доменами (CID). [13]
Аналитические инструменты и базы данных
[ редактировать ]Местоположение TAD определяется путем применения алгоритма к данным Hi-C. Например, TAD часто называют по так называемому «индексу направленности». [4] Индекс направленности рассчитывается для отдельных бункеров по 40 КБ путем сбора операций чтения, попадающих в бункер, и наблюдения за тем, сопоставляются ли их парные чтения вверх или вниз по потоку от контейнера (пары чтения должны охватывать не более 2 МБ). Положительное значение указывает на то, что больше пар чтения находится в нисходящем направлении, чем в восходящем, а отрицательное значение указывает на обратное. Математически индекс направленности представляет собой знаковую статистику хи-квадрат.
Разработка специализированных геномных браузеров и инструментов визуализации. [15] такие как Juicebox, [16] HiGlass [17] /Цветочные люди, [18] Браузер 3D-генома, [19] 3DIV, [20] 3D-GNOME, [21] и ТАДКБ [22] позволили нам визуализировать TAD-организацию интересующих областей в разных типах клеток.
Механизмы формирования
[ редактировать ]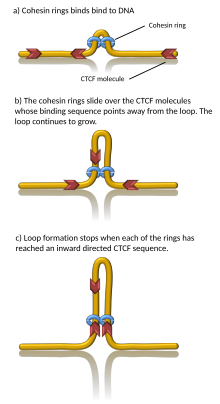
Известно, что ряд белков связан с образованием TAD, включая белок CTCF и белковый комплекс когезин . [1] Также неизвестно, какие компоненты требуются на границах ТОР; однако в клетках млекопитающих было показано, что эти пограничные области имеют сравнительно высокие уровни связывания CTCF. Кроме того, некоторые типы генов (например, гены транспортных РНК и гены «домашнего хозяйства» ) появляются вблизи границ TAD чаще, чем можно было бы ожидать случайно. [3] [4]
Компьютерное моделирование показало, что экструзия петли хроматина, управляемая когезиновыми моторами, может генерировать TAD. [23] [24] В модели экструзии петли когезин связывает хроматин, втягивает его и выдавливает хроматин, постепенно увеличивая петлю. Хроматин по обе стороны комплекса когезина выдавливается до тех пор, пока когезин не встретит связанный с хроматином белок CTCF, обычно расположенный на границе TAD. Таким образом, границы TAD могут быть объединены в качестве якорей петли хроматина. [25] Действительно, in vitro было обнаружено, что когезин процессивно выдавливает петли ДНК АТФ-зависимым образом. [26] [27] [28] и остановился в CTCF. [29] [30] Однако некоторые данные in vitro указывают на то, что наблюдаемые петли могут быть артефактами. [31] [32] Важно отметить, что, поскольку когезины могут динамически отделяться от хроматина, эта модель предполагает, что TAD (и связанные с ними петли хроматина) являются динамическими, временными структурами. [23] в соответствии с наблюдениями in vivo. [33] [34] [35] [36]
Были предложены другие механизмы формирования TAD. Например, некоторые моделирования показывают, что суперспирализация, генерируемая транскрипцией, может перемещать когезин к границам TAD. [37] [38] или что пассивно диффундирующие когезиновые «скользящие звенья» [39] [40] может генерировать TAD.
Характеристики
[ редактировать ]Сохранение
[ редактировать ]Сообщалось, что TAD относительно постоянны между различными типами клеток (например, в стволовых клетках и клетках крови) и даже между видами в определенных случаях. [4] [41] [42] [43]
Отношения с контактами промоутера-энхансера
[ редактировать ]Большинство наблюдаемых взаимодействий между промоторами и энхансерами не пересекают границы TAD. Удаление границы TAD (например, с помощью CRISPR для удаления соответствующего участка генома) может позволить сформироваться новым контактам промотор-энхансер. Это может повлиять на экспрессию близлежащих генов — такая неправильная регуляция, как было показано, вызывает пороки развития конечностей (например, полидактилию ) у людей и мышей. [42]
Компьютерное моделирование показало, что индуцированная транскрипцией сверхспирализация волокон хроматина может объяснить, как образуются TAD и как они могут обеспечивать очень эффективные взаимодействия между энхансерами и родственными им промоторами, расположенными в одном и том же TAD. [37]
Связь с другими структурными особенностями генома
[ редактировать ]Было показано, что домены времени репликации связаны с TAD, поскольку их граница локализована совместно с границами TAD, которые расположены по обе стороны компартментов. [44] Предполагается, что изолированные окрестности , петли ДНК, образованные областями, связанными с CTCF/когезином, функционально лежат в основе TAD. [45]
Роль в болезни
[ редактировать ]Нарушение границ TAD может повлиять на экспрессию близлежащих генов и вызвать заболевание. [46]
Например, сообщалось, что структурные варианты генома, которые нарушают границы TAD, вызывают нарушения развития, такие как пороки развития конечностей человека. [47] [48] [49] Кроме того, несколько исследований предоставили доказательства того, что нарушение или перестановка границ TAD может обеспечить преимущества роста некоторых видов рака, таких как острый Т-клеточный лимфобластный лейкоз (T-ALL), [50] глиомы, [51] и рак легких. [52]
Домены, связанные с пластинкой
[ редактировать ]
Ассоциированные с пластинкой домены (LAD) представляют собой части хроматина, которые тесно взаимодействуют с пластинкой, сетчатой структурой на внутренней мембране ядра . [53] LAD состоят в основном из транскрипционно молчащего хроматина, обогащенного триметилированным Lys27 на гистоне H3 (т.е. H3K27me3 ); Это обычная посттрансляционная гистоновая модификация гетерохроматина . [54] LAD имеют сайты связывания CTCF на своей периферии. [53]
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Jump up to: а б с Помбо А., Диллон Н. (апрель 2015 г.). «Трехмерная архитектура генома: игроки и механизмы». Обзоры природы. Молекулярно-клеточная биология . 16 (4): 245–257. дои : 10.1038/nrm3965 . ПМИД 25757416 . S2CID 6713103 .
- ^ Jump up to: а б Ю М, Рен Б (октябрь 2017 г.). «Трехмерная организация геномов млекопитающих» . Ежегодный обзор клеточной биологии и биологии развития . 33 : 265–289. doi : 10.1146/annurev-cellbio-100616-060531 . ПМЦ 5837811 . ПМИД 28783961 .
- ^ Jump up to: а б с д Нора Э.П., Ладжуа Б.Р., Шульц Э.Г., Джорджетти Л., Окамото И., Слуга Н. и др. (апрель 2012 г.). «Пространственное разделение регуляторного ландшафта центра Х-инактивации» . Природа . 485 (7398): 381–385. Бибкод : 2012Natur.485..381N . дои : 10.1038/nature11049 . ПМЦ 3555144 . ПМИД 22495304 .
- ^ Jump up to: а б с д и ж Диксон Дж.Р., Сельварадж С., Юэ Ф., Ким А., Ли Ю., Шен Ю. и др. (апрель 2012 г.). «Топологические домены в геномах млекопитающих, выявленные путем анализа взаимодействий хроматина» . Природа . 485 (7398): 376–380. Бибкод : 2012Natur.485..376D . дои : 10.1038/nature11082 . ПМЦ 3356448 . ПМИД 22495300 .
- ^ Крийгер П.Х., де Лаат В. (декабрь 2016 г.). «Регуляция экспрессии генов, связанных с заболеваниями, в 3D-геноме». Обзоры природы. Молекулярно-клеточная биология . 17 (12): 771–782. дои : 10.1038/номер.2016.138 . ПМИД 27826147 . S2CID 11484886 .
- ^ Гави-Хельм Ю; Янковский А; Мейерс С; Виалес РР; Корбель Ю.О .; Ферлонг Э.Э. (август 2019 г.). «Сильно перестроенные хромосомы обнаруживают разрыв между топологией генома и экспрессией генов» . Природная генетика . 51 (8): 1272–1282. дои : 10.1038/s41588-019-0462-3 . ПМК 7116017 . ПМИД 31308546 .
- ^ Корсес М.Р., Корсес В.Г. (февраль 2016 г.). «Трехмерный геном рака» . Текущее мнение в области генетики и развития . 36 : 1–7. дои : 10.1016/j.где.2016.01.002 . ПМЦ 4880523 . ПМИД 26855137 .
- ^ Валтон А.Л.; Деккер Дж. (февраль 2016 г.). «Нарушение TAD как фактор онкогенности» . Текущее мнение в области генетики и развития . 36 : 34–40. дои : 10.1016/j.где.2016.03.008 . ПМК 4880504 . ПМИД 27111891 .
- ^ Ачингер-Кавецка Дж., Кларк С.Дж. (январь 2017 г.). «Нарушение трехмерной схемы генома рака» . Эпигеномика . 9 (1): 47–55. дои : 10.2217/epi-2016-0111 . ПМИД 27936932 .
- ^ Jump up to: а б Шпильманн М., Лупианьес Д.Г., Мундлос С. (июль 2018 г.). «Структурные вариации 3D-генома». Обзоры природы. Генетика . 19 (7): 453–467. дои : 10.1038/s41576-018-0007-0 . hdl : 21.11116/0000-0003-610A-5 . ПМИД 29692413 . S2CID 22325904 .
- ^ Jump up to: а б Диксон-младший, Горкин Д.Ю., Рен Б. (июнь 2016 г.). «Хроматиновые домены: единицы хромосомной организации» . Молекулярная клетка . 62 (5): 668–680. doi : 10.1016/j.molcel.2016.05.018 . ПМК 5371509 . ПМИД 27259200 .
- ^ де Лаат В., Дюбул Д. (октябрь 2013 г.). «Топология усилителей развития млекопитающих и их регуляторный ландшафт». Природа . 502 (7472): 499–506. Бибкод : 2013Natur.502..499D . дои : 10.1038/nature12753 . ПМИД 24153303 . S2CID 4468533 .
- ^ Jump up to: а б Сабо К., Бантиньи Ф., Кавалли Дж. (апрель 2019 г.). «Принципы сворачивания генома в топологически ассоциированные домены» . Достижения науки . 5 (4): eaaw1668. Бибкод : 2019SciA....5.1668S . дои : 10.1126/sciadv.aaw1668 . ПМК 6457944 . ПМИД 30989119 .
- ^ Секстон Т., Яффе Е., Кенигсберг Е., Бантиньис Ф., Леблан Б., Хойхман М. и др. (февраль 2012 г.). «Принципы трехмерной складки и функциональной организации генома дрозофилы» . Клетка . 148 (3): 458–472. дои : 10.1016/j.cell.2012.01.010 . ПМИД 22265598 .
- ^ Инг-Симмонс Э., Вакерисас Дж. М. (сентябрь 2019 г.). «Визуализация трехмерной организации генома в двух измерениях» . Разработка . 146 (19): 99–101. дои : 10.1242/dev.177162 . ПМИД 31558569 .
- ^ Дюран, Северная Каролина; Робинсон Дж.Т.; Шамим М.С.; Махол I; Месиров Ж.П.; Лендер ES; Эйден Э.Л. (июль 2016 г.). «Juicebox предоставляет систему визуализации карт контактов Hi-C с неограниченным масштабированием» . Клеточные системы . 3 (1): 99–101. дои : 10.1016/j.cels.2015.07.012 . ПМК 5596920 . ПМИД 27467250 .
- ^ Керпеджиев П., Абденнур Н., Лекщас Ф., МакКаллум С., Динкла К., Стробелт Х. и др. (август 2018 г.). «HiGlass: визуальное исследование и анализ карт взаимодействия геномов через Интернет» . Геномная биология . 19 (1): 125. дои : 10.1186/s13059-018-1486-1 . ПМК 6109259 . ПМИД 30143029 .
- ^ Лекшас Ф., Бах Б., Керпеджиев П., Геленборг Н., Пфистер Х. (январь 2018 г.). «HiPiler: визуальное исследование матриц взаимодействия больших геномов с помощью интерактивных малых кратных» . Транзакции IEEE по визуализации и компьютерной графике . 24 (1): 522–531. дои : 10.1109/TVCG.2017.2745978 . ПМК 6038708 . ПМИД 28866592 .
- ^ Ван Ю, Сун Ф, Чжан Б, Чжан Л, Сюй Дж, Куанг Д и др. (октябрь 2018 г.). «3D-геномный браузер: веб-браузер для визуализации трехмерной организации генома и дальних взаимодействий хроматина» . Геномная биология . 19 (1): 151. дои : 10.1186/s13059-018-1519-9 . ПМК 6172833 . ПМИД 30286773 .
- ^ Ян Д., Чан И, Чхве Дж., Ким М.С., Ли А.Дж., Ким Х. и др. (январь 2018 г.). «3DIV: средство просмотра трехмерного взаимодействия генома и база данных» . Исследования нуклеиновых кислот . 46 (Д1): Д52–Д57. дои : 10.1093/нар/gkx1017 . ПМЦ 5753379 . ПМИД 29106613 .
- ^ Салай П., Михальски П.Дж., Врублевски П., Тан З., Кадлоф М., Маццокко Г. и др. (июль 2016 г.). «3D-GNOME: интегрированный веб-сервис для структурного моделирования 3D-генома» . Исследования нуклеиновых кислот . 44 (П1): W288–W293. дои : 10.1093/nar/gkw437 . ПМЦ 4987952 . ПМИД 27185892 .
- ^ Лю Т., Портер Дж., Чжао К. и др. TADKB: Семейная классификация и база знаний топологически связанных доменов. BMC Genomics 20, 217 (2019). https://doi.org/10.1186/s12864-019-5551-2
- ^ Jump up to: а б Фуденберг Г., Имакаев М., Лу С., Голобородько А., Абденнур Н., Мирный Л.А. (май 2016 г.). «Формирование хромосомных доменов путем экструзии петель» . Отчеты по ячейкам . 15 (9): 2038–2049. дои : 10.1016/j.celrep.2016.04.085 . ПМЦ 4889513 . ПМИД 27210764 .
- ^ Санборн А.Л.; Рао СС; Хуан СК; Дюран, Северная Каролина; Хантли М.Х.; Джуэтт А.И.; и др. (ноябрь 2015 г.). «Экструзия хроматина объясняет ключевые особенности образования петель и доменов в геномах дикого типа и сконструированных геномах» . Труды Национальной академии наук Соединенных Штатов Америки . 112 (47): Е6456–Е6465. Бибкод : 2015PNAS..112E6456S . дои : 10.1073/pnas.1518552112 . ПМЦ 4664323 . ПМИД 26499245 .
- ^ Яцкевич С; Роудс Дж; Нэсмит К. (декабрь 2019 г.). «Организация хромосомной ДНК комплексами ГМК» . Ежегодный обзор генетики . 53 (1): 445–482. doi : 10.1146/annurev-genet-112618-043633 . ПМИД 31577909 . S2CID 203653572 .
- ^ Гольфье С., Куэйл Т., Кимура Х., Брюге Ж. (май 2020 г.). Деккер, Струл К., Мирный Л.А., Мусаккио А., Марко Дж.Ф. (ред.). «Когезин и конденсин выдавливают петли ДНК в зависимости от клеточного цикла» . электронная жизнь . 9 : е53885. doi : 10.7554/eLife.53885 . ПМЦ 7316503 . ПМИД 32396063 .
- ^ Дэвидсон И.Ф., Бауэр Б., Гетц Д., Тан В., Вутц Г., Питерс Дж.М. (декабрь 2019 г.). «Экструзия петли ДНК человеческим когезином» . Наука . 366 (6471): 1338–1345. Бибкод : 2019Sci...366.1338D . дои : 10.1126/science.aaz3418 . ПМИД 31753851 . S2CID 208228309 .
- ^ Ким Ю, Ши З, Чжан Х, Финкельштейн И.Дж., Ю Х (декабрь 2019 г.). «Человеческий когезин уплотняет ДНК путем экструзии петли» . Наука . 366 (6471): 1345–1349. Бибкод : 2019Sci...366.1345K . дои : 10.1126/science.aaz4475 . ПМЦ 7387118 . ПМИД 31780627 .
- ^ Дэвидсон И.Ф., Барт Р., Зачек М., ван дер Торре Дж., Тан В., Нагасака К. и др. (09.09.2022). «CTCF представляет собой зависящий от напряжения ДНК барьер на пути когезин-опосредованной экструзии петли ДНК». bioRxiv 10.1101/2022.09.08.507093 .
- ^ Чжан Х, Ши З, Баниган Э.Дж., Ким Ю, Ю Х, Бай Х, Финкельштейн И.Дж. (07.10.2022). «CTCF и R-петли являются границами петель ДНК, опосредованных когезином». bioRxiv 10.1101/2022.09.15.508177 .
- ^ Ман, Чжоу (сентябрь 2022 г.). «Скольжение ДНК и образование петель комплексом SMC E. coli: MukBEF» . Отчеты по биохимии и биофизике . 31 : 101297. doi : 10.1016/j.bbrep.2022.101297 . ПМЦ 9234588 . ПМИД 35770038 .
- ^ Рю Дж.К., Бушу С., Лю Х.В., Ким Э., Минамино М., де Гроот Р., Катан А.Дж., Бонато А., Марендуццо Д., Микьелетто Д., Ульманн Ф. (февраль 2021 г.). «Мостиковое разделение фаз, индуцированное белковыми комплексами когезина SMC» . Достижения науки . 7 (7): eabe5905. Бибкод : 2021SciA....7.5905R . дои : 10.1126/sciadv.abe5905 . ПМЦ 7875533 . PMID 33568486 .
- ^ Габриэле М., Брандао Х.Б., Гросс-Хольц С., Джа А., Дейли Г.М., Каттоглио С. и др. (апрель 2022 г.). «Динамика образования петель хроматина, опосредованного CTCF и когезином, выявленная с помощью визуализации живых клеток» . Наука . 376 (6592): 496–501. Бибкод : 2022Sci...376..496G . дои : 10.1126/science.abn6583 . ПМК 9069445 . ПМИД 35420890 .
- ^ Беквит К.С., Эдегорд-Фогнер О., Мореро Н.Р., Бартон С., Шудер Ф., Тан В. и др. (02.05.2022). «Визуализация экструзии петли путем наномасштабного отслеживания ДНК в отдельных клетках человека». bioRxiv 10.1101/2021.04.12.439407 .
- ^ Мах П., Кос П.И., Жан Й., Крамард Дж., Годен С., Тюннерманн Дж. и др. (03.03.2022). «Визуализация живых клеток и физическое моделирование показывают контроль динамики сворачивания хромосом с помощью когезина и CTCF». bioRxiv 10.1101/2022.03.03.482826 .
- ^ Флямер И.М., Гасслер Дж., Имакаев М., Брандао Х.Б., Ульянов С.В., Абденнур Н. и др. (апрель 2017 г.). «Одноядерный Hi-C обнаруживает уникальную реорганизацию хроматина при переходе от ооцита к зиготе» . Природа . 544 (7648): 110–114. Бибкод : 2017Natur.544..110F . дои : 10.1038/nature21711 . ПМЦ 5639698 . ПМИД 28355183 .
- ^ Jump up to: а б Рако Д., Бенедетти Ф., Дорье Дж., Стасиак А. (январь 2019 г.). «Являются ли TAD суперспиральными?» . Исследования нуклеиновых кислот . 47 (2): 521–532. дои : 10.1093/nar/gky1091 . ПМК 6344874 . ПМИД 30395328 .
- ^ Рако Д., Бенедетти Ф., Дорье Дж., Стасиак А. (февраль 2018 г.). «Вызванная транскрипцией сверхспирализация как движущая сила экструзии петель хроматина во время образования TAD в интерфазных хромосомах» . Исследования нуклеиновых кислот . 46 (4): 1648–1660. дои : 10.1093/нар/gkx1123 . ПМЦ 5829651 . ПМИД 29140466 .
- ^ Брэкли К.А., Джонсон Дж., Микьелетто Д., Морозов А.Н., Никодеми М., Кук П.Р., Марендуццо Д. (сентябрь 2017 г.). «Неравновесное зацикливание хромосом посредством молекулярных скользящих связей». Письма о физических отзывах . 119 (13): 138101. arXiv : 1612.07256 . Бибкод : 2017PhRvL.119m8101B . doi : 10.1103/PhysRevLett.119.138101 . ПМИД 29341686 . S2CID 14706723 .
- ^ Ямамото Т., Шиссель Х. (сентябрь 2017 г.). «Осмотический механизм процесса экструзии петли». Физический обзор E . 96 (3–1): 030402. Бибкод : 2017PhRvE..96c0402Y . дои : 10.1103/PhysRevE.96.030402 . hdl : 1887/58394 . ПМИД 29346962 .
- ^ Виетри Рудан М., Баррингтон С., Хендерсон С., Эрнст С., Одом Д.Т., Танай А., Хаджур С. (март 2015 г.). «Сравнительный Hi-C показывает, что CTCF лежит в основе эволюции архитектуры хромосомных доменов» . Отчеты по ячейкам . 10 (8): 1297–1309. дои : 10.1016/j.celrep.2015.02.004 . ПМЦ 4542312 . ПМИД 25732821 .
- ^ Jump up to: а б Йост Д., Вайлант С., Мейстер П. (февраль 2017 г.). «Сочетание 1D-модификаций и 3D-ядерной организации: данные, модели и функции». Современное мнение в области клеточной биологии . 44 : 20–27. дои : 10.1016/j.ceb.2016.12.001 . ПМИД 28040646 .
- ^ Ян Ю, Чжан Ю, Рен Б, Диксон-младший, Ма Дж (июнь 2019 г.). «Сравнение трехмерной организации генома у нескольких видов с использованием Phylo-HMRF» . Клеточные системы . 8 (6): 494–505.e14. дои : 10.1016/j.cels.2019.05.011 . ПМК 6706282 . ПМИД 31229558 .
- ^ Маршал С., Сима Дж., Гилберт Д.М. (декабрь 2019 г.). «Контроль времени репликации ДНК в трехмерном геноме». Обзоры природы. Молекулярно-клеточная биология . 20 (12): 721–737. дои : 10.1038/s41580-019-0162-y . PMID 31477886 . S2CID 201714312 .
- ^ Джи Икс, Дадон Д.Б., Пауэлл Б.Е., Фан З.П., Борхес-Ривера Д., Шачар С. и др. (февраль 2016 г.). «Трехмерный ландшафт регуляции хромосом в плюрипотентных клетках человека» . Клеточная стволовая клетка . 18 (2): 262–275. дои : 10.1016/j.stem.2015.11.007 . ПМЦ 4848748 . ПМИД 26686465 .
- ^ Лупианьес Д.Г., Шпильманн М., Мундлос С. (апрель 2016 г.). «Нарушение TAD: как изменения доменов хроматина приводят к заболеванию». Тенденции в генетике . 32 (4): 225–237. дои : 10.1016/j.tig.2016.01.003 . hdl : 11858/00-001M-0000-002E-1D1D-D . ПМИД 26862051 .
- ^ Лупианьес Д.Г., Крафт К., Генрих В., Кравиц П., Бранкати Ф., Клопоцкий Е. и др. (май 2015 г.). «Нарушения топологических доменов хроматина вызывают патогенную перестройку взаимодействий гена и усилителя» . Клетка . 161 (5): 1012–1025. дои : 10.1016/j.cell.2015.04.004 . ПМЦ 4791538 . ПМИД 25959774 .
- ^ Анжер Н (9 января 2017 г.). «Общий дефект семьи проливает свет на геном человека» . Нью-Йорк Таймс .
- ^ Франке М., Ибрагим Д.М., Андрей Г., Шварцер В., Генрих В., Шёпфлин Р. и др. (октябрь 2016 г.). «Образование новых доменов хроматина определяет патогенность геномных дупликаций». Природа . 538 (7624): 265–269. Бибкод : 2016Natur.538..265F . дои : 10.1038/nature19800 . hdl : 11858/00-001M-0000-002C-010A-3 . ПМИД 27706140 . S2CID 4463482 .
- ^ Хниш Д., Вайнтрауб А.С., Дэй Д.С., Валтон А.Л., Бак Р.О., Ли Ч.Х. и др. (март 2016 г.). «Активация протоонкогенов путем разрушения окрестностей хромосом» . Наука . 351 (6280): 1454–1458. Бибкод : 2016Sci...351.1454H . дои : 10.1126/science.aad9024 . ПМЦ 4884612 . ПМИД 26940867 .
- ^ Флавахан В.А., Драйер Ю., Лиау Б.Б., Гиллеспи С.М., Вентайхер А.С., Стеммер-Рахамимов А.О. и др. (январь 2016 г.). «Инсуляторная дисфункция и активация онкогенов в глиомах с мутацией IDH» . Природа . 529 (7584): 110–114. Бибкод : 2016Natur.529..110F . дои : 10.1038/nature16490 . ПМЦ 4831574 . ПМИД 26700815 .
- ^ Вайшенфельдт Дж., Дубаш Т., Дринас А.П., Мардин Б.Р., Чен Ю., Штютц А.М. и др. (январь 2017 г.). «Панараковый анализ изменений числа соматических копий указывает на участие IRS4 и IGF2 в захвате энхансера» . Природная генетика . 49 (1): 65–74. дои : 10.1038/ng.3722 . ПМЦ 5791882 . ПМИД 27869826 .
- ^ Jump up to: а б Гонсалес-Сандовал А; Гассер С.М. (август 2016 г.). «О TAD и LAD: пространственный контроль над экспрессией генов». Тенденции в генетике . 32 (8): 485–495. дои : 10.1016/j.tig.2016.05.004 . ПМИД 27312344 .
- ^ Ли М., Лю Г.Х., Изписуа Бельмонте Дж.К. (июль 2012 г.). «Навигация по эпигенетическому ландшафту плюрипотентных стволовых клеток». Обзоры природы. Молекулярно-клеточная биология . 13 (8): 524–535. дои : 10.1038/nrm3393 . ПМИД 22820889 . S2CID 22524502 .