Анализ метилирования Illumina
на В анализе метилирования Illumina платформе Infinium I используется технология BeadChip. [ нужны разъяснения ] для создания комплексного полногеномного человека профилирования метилирования ДНК . Подобно бисульфитному секвенированию и пиросеквенированию , этот метод количественно определяет метилирования в различных локусах генома уровни . Этот анализ используется для зондов метилирования на чипе Illumina Infinium HumanMmethylation27 BeadChip (далее — массив 27k [метилирования]). Зонды целевых областей массива 27k генома человека для измерения уровней метилирования 27 578 динуклеотидов CpG в 14 495 генах. [ 1 ] В 2008 году компания Illumina выпустила массив Infinium HumanMmethylation450 BeadChip («массив 450 K»), который нацелен на более чем 450 000 сайтов метилирования. [ 2 ] В 2016 году был выпущен Infinium MmethylationEPIC BeadChip («EPIC»), который исследует более 850 000 сайтов метилирования в геноме человека. [ 3 ]
Фон
[ редактировать ]Метилирование ДНК играет значительную роль в эпигенетической регуляции структуры хроматина , которая в последнее десятилетие признана важной в регуляции экспрессии генов , развития и генетического импринтинга у позвоночных. [ 1 ] Было показано, что изменения в характере и уровне метилирования способствуют развитию рака и различных заболеваний развития. [ 4 ] Например, гиперметилирование промоторных CpG -островков гена-супрессора опухоли , что, в свою очередь, приводит к его молчанию , часто связано с опухолевым генезом . [ 4 ] Крупномасштабное измерение паттернов метилирования ДНК из широкого набора генов может позволить нам лучше понять взаимосвязь между эпигенетическими изменениями и генезисом различных заболеваний, а также лучше понять роль, которую эпигенетика играет в тканеспецифической дифференцировке .

Материал
[ редактировать ]Чип метилирования Illumina 27k содержит 27 578 отдельных сайтов CpG, распределенных по 14 495 генам. [ 1 ] Эти гены включают гены RefSeq из базы данных NCBI CCDS, раковые гены, которые демонстрируют дифференциальные закономерности метилирования в ходе их прогрессирования, а также промоторы микроРНК. [ 5 ] Маркеры, включенные в чип, сведены в таблицу 1. [ 5 ]
Метод
[ редактировать ]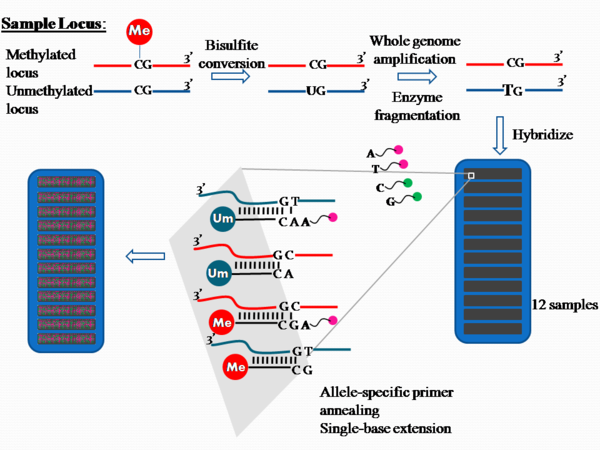
Процесс химического анализа Infinium I показан на рисунке 1.
Бисульфитная обработка
Приблизительно 1 мкг геномной ДНК используется при преобразовании бисульфита для превращения неметилированного цитозина в урацил . Продукт содержит непреобразованный цитозин там, где он был ранее метилирован, но цитозин превращается в урацил, если он ранее не был метилирован.
Амплификация цельногеномной ДНК
ДНК, обработанная бисульфитом, подвергается полногеномной амплификации с множественным смещением посредством случайного прайминга гексамера и ДНК-полимеразы Φ29 , которая обладает корректирующей активностью, приводящей к частоте ошибок в 100 раз ниже, чем у Taq-полимеразы . Затем продукты ферментативно фрагментируются. [ 1 ] очищенный от dNTP, праймеров и ферментов, и нанесенный на чип. [ 6 ]
Гибридизация и одноосновное расширение
На чипе имеется два типа шариков для каждого сайта CpG ( или «CG» , как показано на рисунке 1) на каждый локус. Каждый протестированный локус дифференцируется по разным типам шариков. [ 1 ] Оба типа шариков прикреплены к одноцепочечным 50-мерным ДНК- олигонуклеотидам , которые различаются по последовательности только на свободном конце; этот тип зонда известен как аллель-специфичный олигонуклеотид . Один из типов шариков будет соответствовать локусу метилированного цитозина, а другой будет соответствовать локусу неметилированного цитозина, который был преобразован в урацил во время обработки бисульфитом и позже амплифицирован в тимин во время полногеномной амплификации.
Продукты амплифицированной ДНК, преобразованные бисульфитом, денатурируются в одиночные цепи и гибридизуются с чипом посредством аллель-специфического отжига либо со специфичным для метилирования зондом, либо с зондом неметилирования. За гибридизацией следует удлинение одного основания с помощью дидезоксинуклеотидов, меченных гаптенами. ddCTP и ddGTP мечены биотином , а ddATP и ddUTP мечены 2,4-динитрофенолом (DNP). [ 7 ]
Флуоресцентное окрашивание и сканирование чипа
После включения этих меченных гаптенами ddNTP проводятся многоуровневые иммуногистохимические анализы путем повторных раундов окрашивания комбинацией антител для дифференциации двух типов. [ 7 ] После окрашивания чип сканируют, чтобы показать интенсивность неметилированных и метилированных типов гранул. (рис. 2). [ 1 ] Необработанные данные анализируются с помощью фирменного программного обеспечения и рассчитываются соотношения интенсивностей флуоресценции между двумя типами шариков. Для данного индивида в данном локусе значение отношения 0 соответствует неметилированию локуса ( т.е. гомозиготному неметилированному состоянию); соотношение 1 соответствует общему метилированию ( т.е. гомозиготному метилированию); а значение 0,5 означает, что одна копия метилирована, а другая нет ( т.е. гетерозиготность) в диплоидном геноме человека. [ 1 ] [ 5 ]

Анализ данных метилирования
Сканированные изображения данных метилирования с помощью микрочипа дополнительно анализируются системой, которая нормализует необработанные данные, чтобы уменьшить влияние экспериментальных отклонений, фоновой и средней нормализации, а также выполняет стандартные статистические тесты на результатах. [ 8 ] Затем данные можно объединить в несколько типов цифр для визуализации и анализа. Диаграммы рассеяния используются для корреляции данных по метилированию; гистограммы для визуализации относительных уровней метилирования в каждом тестируемом сайте; тепловые карты для кластеризации данных для сравнения профиля метилирования в тестируемых сайтах. На рисунке 2 показаны различные типы получаемых результатов.
Преимущества и недостатки
[ редактировать ]Преимущества
- не ПЦР требуется, а это означает, что не будет селективной предвзятости в сторону более коротких фрагментов. [ 5 ]
- Возможность исследования до 12 образцов на чип обеспечивает высокую производительность обработки. [ 5 ]
- Позволяет интегрировать данные между другими платформами, такими как экспрессия генов и профилирование микроРНК . [ 5 ]
- Метод рассматривает ~2 сайта CpG на каждый островок CpG, обеспечивая охват всего генома паттернов метилирования (850 тыс.?).
Недостатки
- Не каждый ген, аннотированный в базе данных NCBI, был включен в дизайн этого анализа. Первая версия охватывала 14 495 генов из 17 052 GeneID, существовавших на тот момент (сборка человека 36.3). [ 9 ]
- По данным Стаафа и др. (2008), [ 10 ] «Анализ Infinium II, похоже, имел смещение интенсивности красителя между двумя каналами, используемыми при обнаружении флуоресценции. Более того, это смещение не было устранено даже после того, как данные прошли алгоритмы нормализации, используемые в программном обеспечении BeadStudio». Эта проблема, хотя и справедлива для анализа метилирования GoldenGate, не относится к чипам HumanMmethylation27k, где оба зонда в паре вытягиваются и флуоресцируют в одном и том же канале. [ 10 ]
См. также
[ редактировать ]- MethDB База данных метилирования ДНК
Ссылки
[ редактировать ]- ^ Jump up to: а б с д и ж г час Вайзенбергер, диджей. и др. (2008)Комплексный анализ метилирования ДНК на аналитической платформе Illumina Infinium. [1]
- ^ Infinium® HumanMmethylation450 BeadChip
- ^ «Набор Infinium MmethylationEPIC | Массив для определения профилей метилирования для EWAS» . www.illumina.com . Проверено 10 января 2020 г.
- ^ Jump up to: а б Метилирование ДНК в развитии и заболеваниях человека. Гопалакришнан С., Ван Эмбург Б.О., Робертсон К.Д. Мутационные исследования. 1 декабря 2008 г.;647(1-2):30-8. Epub 2008, 20 августа. Обзор.
- ^ Jump up to: а б с д и ж Illumina: анализ метилирования ДНК: http://www.illumina.com/downloads/DNAMmethylationAnalysis_DataSheet.pdf [ постоянная мертвая ссылка ]
- ^ Гундерсон, КЛ. и др. Масштабируемый анализ генотипирования SNP по всему геному с использованием технологии микрочипов. Nature Genetics 37, 549–554 (2005).
- ^ Jump up to: а б Стимерс, Ф.Дж. и др. Полногеномное генотипирование с помощью анализа однооснования. Природные методы , Vol. 3, № 1, 31 – 33 января (2006).
- ^ Руководство пользователя модуля метилирования BeadStudio. Версия 3. Системы и программное обеспечение Illumina. 2006 г.
- ^ NCBI, проект Consensus CDS (CCDS)
- ^ Jump up to: а б Стааф Дж. и др. Нормализация данных SNP полного генома Illumina Infinium улучшает оценки числа копий и соотношения интенсивностей аллелей. BMC Биоинформатика 2008, 9:409. [2]
Внешние ссылки
[ редактировать ]- Аннотация к массивам метилирования 450k компании Illumina на сайте Bioconductor.
- Станция эпигенетического метилирования
- Nature Reviews: Коллекция метилирования ДНК в Nature Reviews
- Протоколы анализа ДНК/метилирования
- Illumina, метилирование инфиния
- OMICtools : образовательный каталог для анализа данных массива метилирования ДНК.