Одноклеточный анализ
В области клеточной биологии , анализа одиночных клеток и субклеточного анализа. [1] — это изучение геномики , транскриптомики , протеомики , метаболомики и межклеточных взаимодействий на уровне отдельных клеток. [2] [3] [4] Концепция анализа отдельных клеток возникла в 1970-х годах. До открытия гетерогенности анализ отдельных клеток в основном относился к анализу или манипулированию отдельной клеткой в основной популяции клеток в определенных условиях с использованием оптического или электронного микроскопа. [5] На сегодняшний день из-за гетерогенности, наблюдаемой как в популяциях эукариотических, так и в прокариотических клеток, анализ одной клетки позволяет обнаружить механизмы, не наблюдаемые при изучении массовой популяции клеток. [6] Такие технологии, как сортировка клеток, активируемая флуоресценцией (FACS), позволяют точно изолировать выбранные отдельные клетки из сложных образцов, а технологии высокопроизводительного разделения отдельных клеток [7] [8] [9] обеспечить одновременный молекулярный анализ сотен или тысяч отдельных несортированных клеток; это особенно полезно для анализа вариаций транскриптома в генотипически идентичных клетках, что позволяет определить подтипы клеток, которые иначе невозможно обнаружить. Развитие новых технологий расширяет наши возможности анализа генома и транскриптома отдельных клеток. [10] а также количественно оценить их протеом и метаболом . [11] [12] [13] Методы масс-спектрометрии стали важными аналитическими инструментами для протеомного и метаболомного анализа отдельных клеток. [14] [15] Последние достижения позволили количественно оценить тысячи белков в сотнях отдельных клеток. [16] и, таким образом, сделать возможными новые типы анализа. [17] [18] Секвенирование in situ и флуоресцентная гибридизация in situ (FISH) не требуют выделения клеток и все чаще используются для анализа тканей. [19]
Одноклеточная изоляция
[ редактировать ]Многие методы анализа отдельных клеток требуют изоляции отдельных клеток. В настоящее время для выделения отдельных клеток используются следующие методы: диэлектрофоретическая цифровая сортировка, ферментативное расщепление, FACS , гидродинамические ловушки, микродиссекция с лазерным захватом , ручной сбор, микрофлюидика , струйная печать или IJP, микроманипуляция , серийное разведение и рамановский пинцет.
Ручной сбор отдельных клеток — это метод, при котором клетки в суспензии просматриваются под микроскопом и индивидуально отбираются с помощью микропипетки . [20] [21] Рамановский пинцет — это метод, в котором рамановская спектроскопия сочетается с оптическим пинцетом , который использует лазерный луч для захвата клеток и манипулирования ими. [22]
В методе диэлектрофоретической цифровой сортировки используется полупроводниковый контролируемый массив электродов в микрофлюидном чипе для улавливания отдельных клеток в диэлектрофоретических (DEP) клетках. Идентификация клеток обеспечивается сочетанием флуоресцентных маркеров с наблюдением изображений. Точность доставки обеспечивается за счет контролируемого полупроводниковым движением клеток DEP в проточной кювете.
Я струйная печать [23] сочетает микрофлюидику с MEMS на CMOS-чипе, чтобы обеспечить индивидуальный контроль над большим количеством печатающих сопел, используя ту же технологию, что и домашняя струйная печать. IJP позволяет регулировать силу сдвига при выбросе образца, что значительно повышает выживаемость клеток. Этот подход в сочетании с оптическим контролем и распознаванием изображений на основе искусственного интеллекта не только гарантирует дозирование отдельных клеток в луночный планшет или другую среду, но также может оценить качество образца клеток, отбраковывая дефектные клетки, мусор и фрагменты.
Разработка микрофлюидных биочипов на основе гидродинамики с каждым годом растет. В этом методе клетки или частицы захватываются в определенной области для анализа одиночных клеток (SCA), как правило, без применения внешних силовых полей, таких как оптические, электрические, магнитные или акустические. Существует необходимость изучить понимание SCA в естественном состоянии клетки, и разработка этих методов крайне важна для этого исследования. Исследователи подчеркнули обширную потенциальную область, которую необходимо изучить для разработки устройств на основе биочипов, отвечающих требованиям рынка и исследователей. Гидродинамическая микрофлюидика облегчает разработку пассивных лабораторных приложений. Последний обзор дает отчет о последних достижениях в этой области, а также об их механизмах, методах и приложениях. [24]
Ассоциированные технологии
[ редактировать ]В методе диэлектрофоретической цифровой сортировки используется полупроводниковый контролируемый массив электродов в микрофлюидном чипе для улавливания отдельных клеток в диэлектрофоретических (DEP) клетках. Идентификация клеток обеспечивается сочетанием флуоресцентных маркеров с наблюдением изображений. Точность доставки обеспечивается за счет контролируемого полупроводниковым движением клеток DEP в проточной кювете.
Гидродинамические ловушки позволяют изолировать отдельную клетку в «ловушке» в один момент времени посредством пассивного микрофлюидного транспорта. Количество изолированных клеток можно манипулировать в зависимости от количества ловушек в системе.
В методе микродиссекции с лазерным захватом используется лазер для рассечения и отделения отдельных клеток или срезов из представляющих интерес образцов тканей. Эти методы включают наблюдение за клеткой под микроскопом, чтобы можно было идентифицировать и пометить участок для анализа, чтобы лазер мог разрезать клетку. Затем клетку можно извлечь для анализа.
Ручной сбор отдельных клеток — это метод, при котором клетки в суспензии просматриваются под микроскопом и индивидуально отбираются с помощью микропипетки .
Микрофлюидика позволяет изолировать отдельные клетки для дальнейшего анализа. Следующие принципы описывают различные микрофлюидные процессы разделения отдельных клеток: изоляция «капли в масле», пневматические мембранные клапаны и гидродинамические ловушки для клеток. В микрофлюидике на основе «капли в масле» используются заполненные маслом каналы для удержания разделенных капель воды. Это позволяет локализовать и изолировать одну клетку изнутри масляных каналов. Пневматические мембранные клапаны используют манипулирование давлением воздуха для изоляции отдельных клеток за счет отклонения мембраны. Манипулирование источником давления позволяет открывать или закрывать каналы в микрофлюидной сети. Обычно система требует присутствия оператора и ограничена в пропускной способности.
Техника рамановских пинцетов сочетает в себе использование рамановской спектроскопии и оптических пинцетов , которые используют лазерный луч для захвата клеток и манипулирования ими.
Разработка микрофлюидных биочипов на основе гидродинамики с каждым годом растет. В этом методе клетки улавливаются в определенной области для анализа отдельных клеток (SCA). Обычно это происходит без применения внешних силовых полей, таких как оптические, электрические, магнитные или акустические. Существует необходимость изучить возможности SCA в естественном состоянии клетки, и разработка этих методов крайне важна для этого исследования. Исследователи подчеркнули необходимость разработки устройств на основе биочипов, отвечающих требованиям рынка и исследователей. Гидродинамическая микрофлюидика облегчает разработку пассивных лабораторных приложений.
Геномика
[ редактировать ]Техники
[ редактировать ]Геномика одиночных клеток во многом зависит от увеличения количества копий ДНК, обнаруженных в клетке, чтобы их было достаточно для секвенирования. Это привело к разработке стратегий полногеномной амплификации (WGA). В настоящее время стратегии WGA можно сгруппировать в три категории:
- Контролируемое праймирование и ПЦР-амплификация: ПЦР WGA с адаптером-линкером
- Случайное праймирование и ПЦР-амплификация: DOP-PCR, MALBAC.
- Случайное праймирование и изотермическая амплификация: MDA
Во многих сравнительных исследованиях сообщается, что адаптер-линкер PCR WGA лучше всего подходит для анализа диплоидных мутаций отдельных клеток благодаря его очень низкому эффекту выпадения аллелей. [25] [26] [27] и для профилирования изменения количества копий из-за низкого шума как с aCGH, так и с низкочастотным секвенированием NGS. [28] [29] Этот метод применим только к клеткам человека, как фиксированным, так и нефиксированным.
Один из широко распространенных методов WGA называется полимеразной цепной реакцией с вырожденными олигонуклеотидами (DOP-PCR). Этот метод использует хорошо зарекомендовавший себя метод ПЦР амплификации ДНК, чтобы попытаться амплифицировать весь геном с использованием большого набора праймеров . Было показано, что, несмотря на простоту, этот метод имеет очень низкий охват генома. Улучшением DOP-PCR является множественная амплификация смещения (MDA), в которой используются случайные праймеры и высокоточный фермент , обычно ДНК-полимераза Φ29 , для достижения амплификации более крупных фрагментов и большего охвата генома, чем DOP-PCR. Несмотря на эти улучшения, MDA по-прежнему имеет смещение, зависящее от последовательности (определенные части генома амплифицируются больше, чем другие, из-за их последовательности). Метод, который, как было показано, в значительной степени позволяет избежать систематической ошибки, наблюдаемой в DOP-PCR и MDA, представляет собой циклы множественного отжига и амплификации на основе циклов (MALBAC). Смещение в этой системе уменьшается за счет копирования только исходной цепи ДНК, а не копирования копий. Основным недостатком использования MALBA является меньшая точность по сравнению с DOP-PCR и MDA из-за фермента, используемого для копирования ДНК. [11] После амплификации с использованием любого из вышеперечисленных методов ДНК можно секвенировать с использованием метода Сэнгера или секвенирования следующего поколения (NGS).
Цель
[ редактировать ]Есть два основных применения изучения генома на уровне отдельных клеток. Одним из приложений является отслеживание изменений, происходящих в бактериальных популяциях, где часто наблюдаются фенотипические различия. Эти различия не учитываются при массовом секвенировании популяции, но их можно наблюдать при секвенировании отдельных клеток. [30] Второе важное применение — изучение генетической эволюции рака. Поскольку раковые клетки постоянно мутируют, представляет большой интерес посмотреть, как рак развивается на генетическом уровне. Эти закономерности соматических мутаций и аберрации числа копий можно наблюдать с помощью секвенирования отдельных клеток. [2]
Транскриптомика
[ редактировать ]Техники
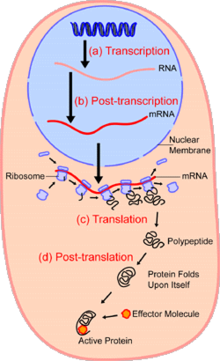
[ редактировать ]В транскриптомике отдельных клеток используются методы секвенирования, аналогичные геномике отдельных клеток, или прямое обнаружение с использованием флуоресцентной гибридизации in situ . Первым шагом в количественном определении транскриптома является преобразование РНК в кДНК с помощью обратной транскриптазы , чтобы содержимое клетки можно было секвенировать с использованием методов NGS, как это делается в геномике. После преобразования кДНК недостаточно для секвенирования, поэтому для обеспечения возможности секвенирования к кДНК применяются те же методы амплификации ДНК, которые обсуждаются в области геномики отдельных клеток. [2] С другой стороны, флуоресцентные соединения, прикрепленные к зондам гибридизации РНК, используются для идентификации конкретных последовательностей, а последовательное применение различных зондов РНК позволит создать комплексный транскриптом. [31] [32]
Цель
[ редактировать ]Цель транскриптомики отдельных клеток — определить, какие гены экспрессируются в каждой клетке. Транскриптом часто используется для количественной оценки экспрессии гена вместо протеома из-за трудностей, связанных в настоящее время с амплификацией уровней белка. [2]
Есть три основные причины, по которым экспрессию генов изучают с использованием этого метода: изучение динамики генов, сплайсинга РНК и типирования клеток. Динамику генов обычно изучают, чтобы определить, какие изменения в экспрессии генов влияют на различные характеристики клеток. Например, этот тип транскриптомного анализа часто используется для изучения эмбрионального развития. Исследования сплайсинга РНК сосредоточены на понимании регуляции различных изоформ транскриптов . Транскриптомика отдельных клеток также использовалась для типирования клеток, когда гены, экспрессируемые в клетке, используются для идентификации типов клеток. Основная цель типирования клеток — найти способ определить идентичность клеток, не имеющих известных генетических маркеров . [2]
Экспрессия РНК может служить показателем обилия белка. Однако численность белка регулируется сложным взаимодействием между экспрессией РНК и посттранскрипционными процессами. Хотя это технически сложнее, трансляцию можно контролировать с помощью профилирования рибосом в отдельных клетках. [33]
Протеомика
[ редактировать ]Техники
[ редактировать ]Существует три основных подхода к протеомике одиночных клеток: методы на основе антител, методы на основе флуоресцентных белков и методы на основе масс-спектроскопии. [34] [18]
Методы на основе антител
[ редактировать ]В методах, основанных на антителах, используются разработанные антитела для связывания с интересующими белками, что позволяет идентифицировать относительное количество нескольких отдельных мишеней с помощью одного из нескольких различных методов.
Визуализация: антитела могут быть связаны с флуоресцентными молекулами, такими как квантовые точки , или помечены органическими флуорофорами для обнаружения с помощью флуоресцентной микроскопии . Поскольку к каждому антителу прикреплены квантовые точки разного цвета или уникальные флуорофоры, можно идентифицировать несколько разных белков в одной клетке. Квантовые точки можно смыть с антител, не повреждая образец, что позволяет проводить несколько раундов количественного определения белка с использованием этого метода на одном и том же образце. [35] В методах, основанных на органических флуорофорах, флуоресцентные метки прикрепляются обратимой связью, такой как ДНК-гибрид (которая может плавиться/диссоциировать в условиях низкого содержания соли). [36] или химически инактивированные, [37] позволяя проводить несколько циклов анализа с количественной оценкой 3-5 целей за цикл. Эти подходы использовались для количественной оценки содержания белка в образцах биопсии пациентов (например, рака) для картирования вариабельной экспрессии белка в тканях и/или опухолях. [37] и для измерения изменений в экспрессии белка и передаче сигналов клетками в ответ на лечение рака. [36]
Массовая цитометрия : изотопы редких металлов, обычно не встречающиеся в клетках или тканях, могут быть прикреплены к отдельным антителам и обнаружены с помощью масс-спектрометрии для одновременной и чувствительной идентификации белков. [38] Эти методы могут быть сильно мультиплексированы для одновременной количественной оценки многих мишеней (панелей до 38 маркеров) в отдельных клетках. [39]
Количественная оценка ДНК-антител: другой метод на основе антител преобразует уровни белка в уровни ДНК. [34] Преобразование в ДНК позволяет повысить уровень белка и использовать NGS для количественного определения белков. В одном из таких подходов для каждого белка, необходимого для количественного определения, выбираются два антитела. Затем два антитела модифицируются, чтобы к ним была присоединена одноцепочечная ДНК, которая является комплементарной. Когда два антитела связываются с белком, комплементарные цепи отжигаются и образуют двухцепочечный сегмент ДНК, который затем можно амплифицировать с помощью ПЦР. Каждая пара антител, предназначенных для одного белка, помечена другой последовательностью ДНК. Затем ДНК, амплифицированную с помощью ПЦР, можно секвенировать и количественно определить уровни белка. [40]
Методы на основе масс-спектрометрии
[ редактировать ]В протеомике, основанной на масс-спектроскопии, для идентификации пептидов необходимы три основных этапа: подготовка проб, разделение пептидов и идентификация пептидов. Несколько групп сосредоточили свое внимание на ооцитах или клетках очень ранней стадии дробления, поскольку эти клетки необычно велики и предоставляют достаточно материала для анализа. [41] [42] [43] [44] Другой подход, протеомика одиночных клеток с помощью масс-спектрометрии (SCoPE-MS), позволил количественно оценить тысячи белков в клетках млекопитающих с типичными размерами клеток (диаметр 10-15 мкм) путем объединения клеток-носителей и одноклеточного штрих-кодирования. [45] [46] Второе поколение, SCoPE2, [47] [48] увеличена производительность за счет автоматизированной и миниатюрной подготовки проб; [49] Это также улучшило количественную надежность и охват протеома за счет оптимизации ЖХ-МС/МС на основе данных. [50] и идентификация пептидов. [51] Чувствительность и последовательность этих методов были дополнительно улучшены за счет определения приоритетов. [52] и массовая параллельная подготовка проб в каплях нанолитрового размера. [53] Другое направление анализа одноклеточных белков основано на масштабируемой системе мультиплексного независимого от данных сбора данных (plexDIA), которая позволяет экономить время за счет параллельного анализа как пептидных ионов, так и образцов белка, тем самым обеспечивая мультипликативный выигрыш в производительности. [54] [55] [56]
Разделение белков разного размера можно осуществить с помощью капиллярного электрофореза (КЭ) или жидкостной хроматографии (ЖХ) (использование жидкостной хроматографии с масс-спектроскопией также известно как ЖХ-МС). [42] [43] [44] [45] На этом этапе пептиды упорядочиваются перед количественным определением с использованием тандемной масс-спектроскопии (МС/МС). Основное различие между методами количественного определения заключается в том, что некоторые используют метки на пептидах, такие как тандемные массовые метки (TMT) или диметиловые метки , которые используются для определения того, из какой клетки произошел определенный белок (белки, поступающие из каждой клетки, имеют разные метки), в то время как другие используют а не метки (определите количество ячеек по отдельности). Данные масс-спектроскопии затем анализируются путем пропускания данных через базы данных, которые преобразуют информацию об идентифицированных пептидах в количественную оценку уровней белка. [42] [43] [44] [45] [57] Эти методы очень похожи на те, которые используются для количественного определения протеома объемных клеток , с модификациями, позволяющими использовать очень небольшой объем образца. [58]
Методы ионизации, используемые в анализе одиночных клеток на основе масс-спектрометрии.
[ редактировать ]Для анализа отдельных клеток можно использовать огромное разнообразие методов ионизации. Выбор метода ионизации имеет решающее значение для обнаружения аналита. Решающее значение может иметь то, какой тип соединений является ионизируемым и в каком состоянии они появляются, например, заряд и возможная фрагментация ионов. [59] Несколько примеров ионизации упомянуты в параграфах ниже.
Нано-ДЕЗИ
[ редактировать ]Одним из возможных способов измерения содержания одиночных клеток является нано-DESI (наноспрей-десорбция-ионизация электрораспылением). В отличие от десорбционной ионизации электрораспылением , которая представляет собой метод десорбции, nano-DESI представляет собой метод жидкостной экстракции , который позволяет брать образцы с небольших поверхностей и поэтому подходит для анализа отдельных клеток. В nano-DESI два капилляра из плавленого кварца расположены V-образной формы, образуя угол прибл. 85 градусов. Два капилляра соприкасаются, поэтому между ними может образоваться жидкостный мостик, позволяющий брать образцы с поверхностей размером до одной клетки. Первичный капилляр доставляет растворитель к поверхности образца, где происходит экстракция, а вторичный капилляр направляет растворитель с экстрагированными молекулами на вход МС. Масс-спектрометрия (МС) Nano-DESI позволяет проводить чувствительное молекулярное профилирование и количественную оценку эндогенных частиц размером всего в несколько сотен фмоль-с в отдельных клетках с более высокой производительностью. Ланекофф и др. идентифицировали 14 аминокислот, 6 метаболитов и несколько липидных молекул из одиночных клеток щеки с помощью nano-DESI MS. [60]
повредить
[ редактировать ]При лазерной абляции ионизации электрораспылением (LAESI) лазер используется для абляции поверхности образца, а испускаемые молекулы ионизируются в газовой фазе заряженными каплями электрораспыления. Подобно DESI, ионизация происходит в условиях окружающей среды . Андертон и др . использовали этот метод ионизации в сочетании с масс-спектрометром с преобразованием Фурье для анализа 200 отдельных клеток Allium cepa (красного лука) с высоким пространственным разрешением. [61]
ВИМС
[ редактировать ]Масс-спектрометрия вторичных ионов (ВИМС) — это метод, аналогичный DESI, но, хотя DESI является методом ионизации окружающей среды, SIMS происходит в вакууме . Поверхность твердого образца бомбардируется высокосфокусированным пучком первичных ионов . Когда они ударяются о поверхность, молекулы вылетают с поверхности и ионизируются. Выбор первичных ионов определяет размер пучка, а также степень ионизации и фрагментации. [62] Парик и др. выполнили метаболомику, чтобы проследить, как пурины синтезируются в пуриносомах , и использовали изотопную маркировку и SIMS-визуализацию, чтобы напрямую наблюдать горячие точки метаболической активности в замороженных клетках HeLa. [63]
МАЛЬДИ
[ редактировать ]При матричной лазерной десорбции и ионизации (MALDI) образец помещается в химическую матрицу , способную поглощать энергию лазера. Подобно ВИМС, ионизация происходит в вакууме. Лазерное облучение удаляет материал матрицы с поверхности и приводит к образованию заряженных частиц матрицы в газовой фазе, молекулы аналита ионизируются из этой заряженной химической матрицы. Лю и др. использовали MALDI-MS для обнаружения восьми фосфолипидов из отдельных клеток A549. [64] Визуализация MALDI MS может использоваться для пространственной метаболомики и анализа отдельных клеток. [65] [66]
Цель
[ редактировать ]Цель изучения протеома — лучше понять активность клеток на уровне отдельных клеток. Поскольку белки отвечают за определение того, как действует клетка, понимание протеома отдельной клетки дает лучшее понимание того, как работает клетка и как экспрессия генов меняется в клетке из-за различных стимулов окружающей среды. Хотя транскриптомика преследует ту же цель, что и протеомика, она не так точна при определении экспрессии генов в клетках, поскольку не учитывает посттранскрипционную регуляцию . [12] Транскриптомика по-прежнему важна, поскольку изучение разницы между уровнями РНК и белка может дать представление о том, какие гены регулируются посттранскрипционно.
Метаболомика
[ редактировать ]Техники
[ редактировать ]Существует четыре основных метода, используемых для количественной оценки метаболома отдельных клеток: обнаружение на основе флуоресценции, флуоресцентные биосенсоры, биосенсоры FRET и масс-спектроскопия. Первые три перечисленных метода используют флуоресцентную микроскопию для обнаружения молекул в клетке. Обычно в этих анализах используются небольшие флуоресцентные метки, прикрепленные к интересующим молекулам, однако было показано, что это слишком инвазивно для метаболомики отдельных клеток и изменяет активность метаболитов. Текущее решение этой проблемы заключается в использовании флуоресцентных белков, которые будут действовать как детекторы метаболитов, флуоресцируя всякий раз, когда они связываются с интересующим метаболитом. [67]
Масс-спектроскопия становится наиболее часто используемым методом метаболомики одиночных клеток. Его преимущества заключаются в том, что нет необходимости разрабатывать флуоресцентные белки для всех интересующих молекул, и он способен обнаруживать метаболиты в фемтомольном диапазоне. [15] Подобно методам, обсуждаемым в протеомике, также был достигнут успех в сочетании масс-спектроскопии с методами разделения, такими как капиллярный электрофорез, для количественного определения метаболитов. Этот метод также способен обнаруживать метаболиты, присутствующие в фемтомольных концентрациях. [67] Было продемонстрировано, что другой метод, использующий капиллярный микроотбор проб в сочетании с масс-спектрометрией с разделением по подвижности ионов, улучшает молекулярный охват и разделение ионов для метаболомики одиночных клеток. [21] [68] Исследователи пытаются разработать метод, который сможет реализовать то, чего не хватает современным методам: высокую производительность, более высокую чувствительность к метаболитам с более низким содержанием или низкой эффективностью ионизации, хорошую воспроизводимость и возможность количественного определения метаболитов. [69]
Цель
[ редактировать ]Целью метаболомики одиночных клеток является лучшее понимание на молекулярном уровне основных биологических тем, таких как: рак, стволовые клетки, старение, а также развитие устойчивости к лекарствам. В целом метаболомика фокусируется главным образом на понимании того, как клетки справляются со стрессами окружающей среды на молекулярном уровне, а также на более динамичном понимании клеточных функций. [67]
Реконструкция траекторий развития
[ редактировать ]Транскриптомные анализы отдельных клеток позволили определить траектории развития реконструкции. Ветвление этих траекторий описывает дифференцировку клеток. Были разработаны различные методы для реконструкции ветвящихся траекторий развития на основе транскриптомных данных одиночных клеток. [70] [71] [72] [73] [74] Они используют различные передовые математические концепции оптимальной транспортировки. [72] к основным графам. [73] Некоторые библиотеки программного обеспечения для реконструкции и визуализации траекторий дифференциации линий находятся в свободном доступе в Интернете. [75]
Межклеточное взаимодействие
[ редактировать ]Межклеточные взаимодействия характеризуются стабильными и преходящими взаимодействиями.
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Сюздак Г. (сентябрь 2023 г.). «Субклеточная количественная визуализация метаболитов на уровне органелл». Природный метаболизм . 5 (9): 1446–1448. дои : 10.1038/s42255-023-00882-z . ПМИД 37679555 . S2CID 261607846 .
- ^ Перейти обратно: а б с д и Ван Д., Бодовиц С. (июнь 2010 г.). «Анализ одиночных клеток: новый рубеж в «омике» » . Тенденции в биотехнологии . 28 (6): 281–90. дои : 10.1016/j.tibtech.2010.03.002 . ПМЦ 2876223 . ПМИД 20434785 .
- ^ Хабиби И., Чеонг Р., Липняцки Т., Левченко А., Эмамян Э.С., Абди А. (апрель 2017 г.). «Вычисление и измерение ошибок принятия решений ячейками с использованием данных одной ячейки» . PLOS Вычислительная биология . 13 (4): e1005436. Бибкод : 2017PLSCB..13E5436H . дои : 10.1371/journal.pcbi.1005436 . ПМК 5397092 . ПМИД 28379950 .
- ^ Меруан А., Рей-Вилламисар Н., Лу Ю., Лиади И., Ромен Г., Лу Дж. и др. (октябрь 2015 г.). «Автоматическое профилирование индивидуальных межклеточных взаимодействий с помощью высокопроизводительной покадровой микроскопии в сетках нанолунок (TIMING)» . Биоинформатика . 31 (19): 3189–97. doi : 10.1093/биоинформатика/btv355 . ПМК 4693004 . ПМИД 26059718 .
- ^ Лу Дж., Хо Х., Конг С., Ван Т., Хо Ю. (сентябрь 2019 г.). «Технологические достижения в многомасштабном анализе одиночных клеток в биомедицине». Продвинутые биосистемы . 3 (11): e1900138. дои : 10.1002/adbi.201900138 . ПМИД 32648696 . S2CID 203101696 .
- ^ Альтшулер С.Дж., Ву Л.Ф. (май 2010 г.). «Клеточная гетерогенность: имеют ли различия значение?» . Клетка . 141 (4): 559–63. дои : 10.1016/j.cell.2010.04.033 . ПМЦ 2918286 . ПМИД 20478246 .
- ^ Ху П., Чжан В., Синь Х., Дэн Г. (25 октября 2016 г.). «Выделение и анализ одиночных клеток» . Границы клеточной биологии и биологии развития . 4 : 116. дои : 10.3389/fcell.2016.00116 . ПМК 5078503 . ПМИД 27826548 .
- ^ Мора-Кастилья С., То С., Ваезеслами С., Мори Р., Сринивасан С., Дамди Дж. Н. и др. (август 2016 г.). «Технологии миниатюризации для эффективной подготовки одноклеточных библиотек для секвенирования следующего поколения» . Журнал автоматизации лабораторий . 21 (4): 557–67. дои : 10.1177/2211068216630741 . ПМЦ 4948133 . ПМИД 26891732 .
- ^ Чжэн Г.С., Терри Дж.М., Белградер П., Рывкин П., Бент З.В., Уилсон Р. и др. (январь 2017 г.). «Массовое параллельное цифровое транскрипционное профилирование отдельных клеток» . Природные коммуникации . 8 : 14049. Бибкод : 2017NatCo...814049Z . дои : 10.1038/ncomms14049 . ПМК 5241818 . ПМИД 28091601 .
- ^ Меркателли Д., Бальбони Н., Пальма А., Алео Э., Санна П.П., Перини Г. и др. (январь 2021 г.). «Анализ одноклеточной генной сети и транскрипционный ландшафт клеточных линий нейробластомы, амплифицированных MYCN» . Биомолекулы . 11 (2): 177. doi : 10.3390/biom11020177 . ПМЦ 7912277 . ПМИД 33525507 .
- ^ Перейти обратно: а б Хуан Л., Ма Ф., Чепмен А., Лу С., Се XS (2015). «Одноклеточная амплификация и секвенирование цельного генома: методология и приложения» . Ежегодный обзор геномики и генетики человека . 16 (1): 79–102. doi : 10.1146/annurev-genom-090413-025352 . ПМИД 26077818 . S2CID 12987987 .
- ^ Перейти обратно: а б Ву А.Р., Ван Дж., Стритс А.М., Хуан Ю (июнь 2017 г.). «Одноклеточный транскрипционный анализ». Ежегодный обзор аналитической химии . 10 (1): 439–462. doi : 10.1146/annurev-anchem-061516-045228 . ПМИД 28301747 . S2CID 40069109 .
- ^ Циорис К., Торрес А.Дж., Дус ТБ, Лав Дж.К. (2014). «Новый набор инструментов для оценки отдельных клеток» . Ежегодный обзор химической и биомолекулярной инженерии . 5 : 455–77. doi : 10.1146/annurev-chembioeng-060713-035958 . ПМК 4309009 . ПМИД 24910919 .
- ^ Коми Т.Дж., До Т.Д., Рубахин С.С., Свидлер СП (март 2017 г.). «Классификация клеток на основе их химического профиля: прогресс в одноклеточной масс-спектрометрии» . Журнал Американского химического общества . 139 (11): 3920–3929. дои : 10.1021/jacs.6b12822 . ПМЦ 5364434 . ПМИД 28135079 .
- ^ Перейти обратно: а б Чжан Л., Вертес А. (апрель 2018 г.). «Одноклеточные масс-спектрометрические подходы к исследованию клеточной гетерогенности» . Ангеванде Хеми . 57 (17): 4466–4477. дои : 10.1002/anie.201709719 . ПМИД 29218763 . S2CID 4928231 .
- ^ Славов Н. (июнь 2020 г.). «Анализ одноклеточных белков методом масс-спектрометрии» . Современное мнение в области химической биологии . 60 : 1–9. arXiv : 2004.02069 . дои : 10.1016/j.cbpa.2020.04.018 . ISSN 1367-5931 . ПМЦ 7767890 . ПМИД 32599342 . S2CID 219966629 .
- ^ Шпехт Х., Славов Н. (август 2018 г.). «Трансформационные возможности протеомики одноклеточных клеток» . Журнал исследований протеома . 17 (8): 2565–2571. doi : 10.1021/acs.jproteome.8b00257 . ПМК 6089608 . ПМИД 29945450 .
- ^ Перейти обратно: а б Славов Н. (январь 2020 г.). «Распаковка протеома в одиночных клетках» . Наука . 367 (6477): 512–513. Бибкод : 2020Sci...367..512S . дои : 10.1126/science.aaz6695 . ПМК 7029782 . ПМИД 32001644 .
- ^ Ли Дж. Х. (июль 2017 г.). «Реконструкция экспрессии генов De Novo в космосе» . Тенденции молекулярной медицины . 23 (7): 583–593. doi : 10.1016/j.molmed.2017.05.004 . ПМЦ 5514424 . ПМИД 28571832 .
- ^ Гросс А., Шендубе Дж., Циммерманн С., Стееб М., Ценгерле Р., Колтай П. (июль 2015 г.). «Технологии выделения одноклеточных клеток» . Международный журнал молекулярных наук . 16 (8): 16897–919. дои : 10.3390/ijms160816897 . ПМЦ 4581176 . ПМИД 26213926 .
- ^ Перейти обратно: а б Чжан Л., Вертес А. (октябрь 2015 г.). «Энергетический заряд, окислительно-восстановительное состояние и обмен метаболитов в отдельных гепатоцитах человека, выявленные с помощью масс-спектрометрии с капиллярным микроотбором» . Аналитическая химия . 87 (20): 10397–405. дои : 10.1021/acs.analchem.5b02502 . ПМИД 26398405 .
- ^ Фариа ЕС, Гарднер П. (январь 2012 г.). «Анализ одиночных эукариотических клеток с использованием рамановского пинцета». Линдстрем С., Андерссон-Сван Х. (ред.). Одноклеточный анализ . Методы молекулярной биологии. Том. 853. Хумана Пресс. стр. 151–67. дои : 10.1007/978-1-61779-567-1_12 . ISBN 978-1-61779-566-4 . ПМИД 22323146 .
- ^ «ММС» . ММС . Проверено 15 мая 2024 г.
- ^ Нараянамурти В., Нагараджан С., Самсури Ф., Шридхар Т.М. (30 июня 2017 г.). «Микрофлюидный гидродинамический захват для анализа одиночных клеток: механизмы, методы и применение» . Аналитические методы . 9 (25): 3751–3772. дои : 10.1039/C7AY00656J . ISSN 1759-9679 .
- ^ Бабаян А., Алави М., Гормли М., Мюллер В., Викман Х., Макмаллин Р.П. и др. (август 2017 г.). «Сравнительное исследование амплификации всего генома и эффективности секвенирования следующего поколения одиночных раковых клеток» . Онкотаргет . 8 (34): 56066–56080. дои : 10.18632/oncotarget.10701 . ПМЦ 5593545 . ПМИД 28915574 .
- ^ Биндер В., Бартенхаген С., Окпаньи В., Гомберт М., Молендик Б., Беренс Б. и др. (октябрь 2014 г.). «Новый рабочий процесс для полногеномного секвенирования отдельных клеток человека» . Человеческая мутация . 35 (10): 1260–70. дои : 10.1002/humu.22625 . ПМИД 25066732 . S2CID 27392899 .
- ^ Боргстрем Э., Патерлини М., Молд Дж.Э., Фризен Дж., Лундеберг Дж. (2017). «Сравнение методов амплификации всего генома для секвенирования экзома отдельных клеток человека» . ПЛОС ОДИН . 12 (2): e0171566. Бибкод : 2017PLoSO..1271566B . дои : 10.1371/journal.pone.0171566 . ПМК 5313163 . ПМИД 28207771 .
- ^ Норманд Э., Кдайсат С., Би В., Шоу С., Ван ден Вейвер И., Боде А. и др. (сентябрь 2016 г.). «Сравнение трех методов амплификации всего генома для обнаружения геномных аберраций в отдельных клетках». Пренатальная диагностика . 36 (9): 823–30. дои : 10.1002/pd.4866 . ПМИД 27368744 . S2CID 5537482 .
- ^ Вандер Плаетсен А.С., Делей Л., Корнелис С., Тиллеман Л., Ван Ньювербург Ф., Дефорс Д. (декабрь 2017 г.). «Профилирование STR и анализ вариаций числа копий на отдельных сохраненных клетках с использованием современных методов амплификации всего генома» . Научные отчеты . 7 (1): 17189. Бибкод : 2017НатСР...717189В . дои : 10.1038/s41598-017-17525-5 . ПМЦ 5719346 . ПМИД 29215049 .
- ^ Калиски Т., Quake SR (апрель 2011 г.). «Одноклеточная геномика». Природные методы . 8 (4): 311–4. дои : 10.1038/nmeth0411-311 . ПМИД 21451520 . S2CID 5601612 .
- ^ Любек Э., Джошкун А.Ф., Жиентаев Т., Ахмад М., Кай Л. (апрель 2014 г.). «Профилирование одноклеточной РНК in situ путем последовательной гибридизации» . Природные методы . 11 (4): 360–1. дои : 10.1038/nmeth.2892 . ПМЦ 4085791 . ПМИД 24681720 .
- ^ Чен К.Х., Беттигер А.Н., Моффитт-младший, Ван С., Чжуан X (апрель 2015 г.). «Визуализация РНК. Пространственно разрешенное, высокомультиплексированное профилирование РНК в отдельных клетках» (PDF) . Наука . 348 (6233): ааа6090. дои : 10.1126/science.aaa6090 . ПМЦ 4662681 . ПМИД 25858977 .
- ^ Озадам Х., Тонн Т., Хан С.М., Сегура А., Хоскинс И., Рао С. и др. (июнь 2023 г.). «Количественная оценка занятости рибосом в отдельных клетках на ранних стадиях развития мышей» . Природа . 618 (7967): 1057–1064. Бибкод : 2023Natur.618.1057O . дои : 10.1038/s41586-023-06228-9 . ПМЦ 10307641 . ПМИД 37344592 .
- ^ Перейти обратно: а б Леви Э, Славов Н (октябрь 2018 г.). «Анализ одноклеточных белков для системной биологии» . Очерки по биохимии . 62 (4): 595–605. дои : 10.1042/EBC20180014 . ПМК 6204083 . ПМИД 30072488 .
- ^ Зражевский П., Тру Л.Д., Гао X (октябрь 2013 г.). «Многоцветное многоцикловое молекулярное профилирование с помощью квантовых точек для анализа одиночных клеток» . Протоколы природы . 8 (10): 1852–69. дои : 10.1038/nprot.2013.112 . ПМЦ 4108347 . ПМИД 24008381 .
- ^ Перейти обратно: а б Гидт Р.Дж., Патания Д., Карлсон Дж.К., МакФарланд П.Дж., Дель Кастильо А.Ф., Джурик Д. и др. (октябрь 2018 г.). «Анализ одноклеточных штрих-кодов обеспечивает быстрое считывание клеточных сигнальных путей в клинических образцах» . Природные коммуникации . 9 (1): 4550. Бибкод : 2018NatCo...9.4550G . дои : 10.1038/s41467-018-07002-6 . ПМК 6208406 . ПМИД 30382095 .
- ^ Перейти обратно: а б Лин Дж.Р., Изар Б., Ван С., Япп С., Мэй С., Шах П.М. и др. (июль 2018 г.). Чакраборти А.К., Радж А., Марр С., Хорват П. (ред.). «Высокомультиплексная иммунофлуоресцентная визуализация тканей и опухолей человека с использованием t-CyCIF и обычных оптических микроскопов» . электронная жизнь . 7 : е31657. дои : 10.7554/eLife.31657 . ПМК 6075866 . ПМИД 29993362 .
- ^ Наир Н., Мэй Х.Э., Чен С.Ю., Хейл М., Нолан Г.П., Меккер Х.Т. и др. (май 2015 г.). «Массовая цитометрия как платформа для открытия клеточных биомаркеров для эффективного лечения ревматических заболеваний» . Исследования и терапия артрита . 17 (1): 127. doi : 10.1186/s13075-015-0644-z (неактивен 30 марта 2024 г.). ПМЦ 4436107 . ПМИД 25981462 .
{{cite journal}}: CS1 maint: DOI неактивен по состоянию на март 2024 г. ( ссылка ) - ^ Спитцер М.Х., Нолан Г.П. (май 2016 г.). «Массовая цитометрия: отдельные клетки, множество функций» . Клетка . 165 (4): 780–91. дои : 10.1016/j.cell.2016.04.019 . ПМК 4860251 . ПМИД 27153492 .
- ^ Гонг Х., Холкомб И., Оой А., Ван Х., Майонис Д., Унгер М.А. и др. (январь 2016 г.). «Простой метод получения конъюгированных с олигонуклеотидами антител и его применение для мультиплексного обнаружения белков в одиночных клетках» . Биоконъюгатная химия . 27 (1): 217–25. doi : 10.1021/acs.bioconjchem.5b00613 . ПМИД 26689321 .
- ^ Смитс А.Х., Линдебум Р.Г., Перино М., ван Херинген С.Дж., Венстра Г.Дж., Вермюлен М. (сентябрь 2014 г.). «Глобальная абсолютная количественная оценка показывает жесткую регуляцию экспрессии белка в отдельных яйцах Xenopus» . Исследования нуклеиновых кислот . 42 (15): 9880–9891. дои : 10.1093/nar/gku661 . ПМК 4150773 . ПМИД 25056316 .
- ^ Перейти обратно: а б с Ломбард-Банек С., Редди С., Муди С.А., Немес П. (август 2016 г.). «Количественное определение белков в одиночных эмбриональных клетках с нейронной судьбой без меток в эмбрионах лягушки (Xenopus laevis) на стадии дробления с использованием капиллярного электрофореза, электрораспылительной ионизации, масс-спектрометрии высокого разрешения (CE-ESI-HRMS)» . Молекулярная и клеточная протеомика . 15 (8): 2756–2768. дои : 10.1074/mcp.M115.057760 . ПМЦ 4974349 . ПМИД 27317400 .
- ^ Перейти обратно: а б с Сунь Л., Дубиак К.М., Пеучен Э.Х., Чжан З., Чжу Г., Хубер П.В. и др. (июль 2016 г.). «Одноклеточная протеомика с использованием бластомеров лягушки (Xenopus laevis), выделенных из эмбрионов ранних стадий, которые образуют геометрическую прогрессию по содержанию белка» . Аналитическая химия . 88 (13): 6653–6657. дои : 10.1021/acs.analchem.6b01921 . ПМК 4940028 . ПМИД 27314579 .
- ^ Перейти обратно: а б с Вирант-Клун И., Лейхт С., Хьюз К., Крийгсвельд Дж. (август 2016 г.). «Идентификация белков, специфичных для созревания, с помощью одноклеточной протеомики ооцитов человека» . Молекулярная и клеточная протеомика . 15 (8): 2616–2627. дои : 10.1074/mcp.M115.056887 . ПМЦ 4974340 . ПМИД 27215607 .
- ^ Перейти обратно: а б с Будник Б, Леви Э, Славов Н (15 марта 2017 г.). «Масс-спектрометрия отдельных клеток млекопитающих позволяет количественно оценить гетерогенность протеома во время дифференцировки клеток». биоRxiv 10.1101/102681 .
- ^ Будник Б., Леви Э., Харманж Г., Славов Н. (октябрь 2018 г.). «SCoPE-MS: масс-спектрометрия отдельных клеток млекопитающих количественно определяет гетерогенность протеома во время дифференцировки клеток» . Геномная биология . 19 (1): 161. дои : 10.1186/s13059-018-1547-5 . ПМК 6196420 . ПМИД 30343672 .
- ^ Шпехт Х, Эммотт Э, Коллер Т, Славов Н (09 июля 2019 г.). «Высокопроизводительная протеомика одиночных клеток позволяет количественно оценить появление гетерогенности макрофагов» . биоRxiv . дои : 10.1101/665307 .
- ^ Шпехт Х., Эммотт Э., Петельски А.А., Хаффман Р.Г., Перлман Д.Х., Серра М. и др. (январь 2021 г.). «Одноклеточный протеомный и транскриптомный анализ гетерогенности макрофагов с использованием SCoPE2» . Геномная биология . 22 (1): 50. дои : 10.1186/s13059-021-02267-5 . ПМЦ 7839219 . ПМИД 33504367 .
- ^ Шпехт Х., Харманж Г., Перлман Д.Х., Эммотт Э., Низиолек З., Будник Б. и др. (25 августа 2018 г.). «Автоматическая пробоподготовка для высокопроизводительной протеомики одиночных клеток» . bioRxiv : 399774. doi : 10.1101/399774 .
- ^ Хаффман Р.Г., Чен А., Шпехт Х., Славов Н. (июнь 2019 г.). «DO-MS: оптимизация методов масс-спектрометрии на основе данных» . Журнал исследований протеома . 18 (6): 2493–2500. doi : 10.1021/acs.jproteome.9b00039 . ПМЦ 6737531 . ПМИД 31081635 .
- ^ Чен А.Т., Франкс А., Славов Н. (июль 2019 г.). Кокс Дж. (ред.). «DART-ID увеличивает покрытие протеома одной клетки» . PLOS Вычислительная биология . 15 (7): e1007082. Бибкод : 2019PLSCB..15E7082C . дои : 10.1371/journal.pcbi.1007082 . ПМЦ 6625733 . ПМИД 31260443 .
- ^ Хаффман Р.Г., Ледюк А., Вихманн С., ди Джоя М., Борриелло Ф., Шпехт Х. и др. (18 марта 2022 г.). «Приоритетная протеомика одиночных клеток выявляет молекулярную и функциональную поляризацию первичных макрофагов». bioRxiv : 2022.03.16.484655. дои : 10.1101/2022.03.16.484655 . S2CID 247599981 .
- ^ Ледюк А., Хаффман Р.Г., Кантлон Дж., Хан С., Славов Н. (декабрь 2022 г.). «Изучение функциональной ковариации белков в отдельных клетках с использованием nPOP» . Геномная биология . 23 (1): 261. doi : 10.1186/s13059-022-02817-5 . ПМЦ 9756690 . ПМИД 36527135 .
- ^ «Система мультипликативного масштабирования протеомики одноклеточных». Природная биотехнология . 41 (1): 23–24. Июль 2022 г. doi : 10.1038/s41587-022-01411-1 . ПМИД 35851377 . S2CID 250642572 .
- ^ Деркс Дж., Ледюк А., Валлманн Г., Хаффман Р.Г., Уиллеттс М., Хан С. и др. (июль 2022 г.). «Увеличение производительности чувствительной протеомики с помощью plexDIA» . Природная биотехнология . 41 (1): 50–59. дои : 10.1038/s41587-022-01389-w . ПМЦ 9839897 . ПМИД 35835881 .
- ^ Деркс Дж., Славов Н. (05.11.2022). «Стратегии увеличения глубины и производительности анализа белков с помощью plexDIA». bioRxiv : 2022.11.05.515287. дои : 10.1101/2022.11.05.515287 . S2CID 253399025 .
- ^ Смитс А.Х., Линдебум Р.Г., Перино М., ван Херинген С.Дж., Винстра Г.Дж., Вермюлен М. (сентябрь 2014 г.). «Глобальная абсолютная количественная оценка показывает жесткую регуляцию экспрессии белка в отдельных яйцах Xenopus» . Исследования нуклеиновых кислот . 42 (15): 9880–91. дои : 10.1093/nar/gku661 . ПМК 4150773 . ПМИД 25056316 .
- ^ «SCoPE-MS — Наконец-то мы можем заняться протеомикой отдельных клеток!!!» . Новости в области протеомных исследований . 09.03.2017 . Проверено 28 июня 2017 г.
- ^ Уотсон Дж.Т., Спаркман ОД (октябрь 2007 г.). Введение в масс-спектрометрию: приборы, приложения и стратегии интерпретации данных (4-е изд.). Джон Уайли и сыновья, 2007. с. 8. ISBN 978-0-470-51634-8 .
- ^ Бергман Х.М., Ланекофф I (2017). «Профилирование и количественная оценка эндогенных молекул в отдельных клетках с использованием nano-DESI MS» . Аналитик . 142 (19): 3639–3647. Бибкод : 2017Ана...142.3639B . дои : 10.1039/C7AN00885F . ISSN 0003-2654 . ПМИД 28835951 .
- ^ Тейлор М.Дж., Лию А., Вертес А., Андертон Ч.Р. (сентябрь 2021 г.). «Анализ одиночных клеток в окружающей среде и визуализация нативных тканей с использованием масс-спектрометрии с ионизацией и ионизацией лазерной абляции и повышенным пространственным разрешением». Журнал Американского общества масс-спектрометрии . 32 (9): 2490–2494. дои : 10.1021/jasms.1c00149 . ОСТИ 1824325 . ПМИД 34374553 . S2CID 236968123 .
- ^ Ланекофф И., Шарма В.В., Маркес С. (июнь 2022 г.). «Метаболомика одноклеточных: где мы и куда идем?» . Современное мнение в области биотехнологии . 75 : 102693. doi : 10.1016/j.copbio.2022.102693 . ПМИД 35151979 . S2CID 246773056 .
- ^ Парик В., Тиан Х., Виноград Н., Бенкович С.Дж. (апрель 2020 г.). «Метаболомика и масс-спектрометрическая визуализация показывают направленный синтез пуринов de novo в клетках» . Наука . 368 (6488): 283–290. Бибкод : 2020Sci...368..283P . дои : 10.1126/science.aaz6465 . ПМЦ 7494208 . ПМИД 32299949 .
- ^ Се В., Гао Д., Цзинь Ф., Цзян Ю., Лю Х. (июль 2015 г.). «Исследование фосфолипидов в одиночных клетках с использованием интегрированного микрофлюидного устройства в сочетании с масс-спектрометрией лазерной десорбции / ионизации с матричной поддержкой». Аналитическая химия . 87 (14): 7052–7059. дои : 10.1021/acs.analchem.5b00010 . ПМИД 26110742 .
- ^ Бурсо П., Гейер Б., Суердик В., Бьен Т., Солтвиш Дж., Драйзеверд К. и др. (октябрь 2023 г.). «Визуализация метаболитов и микробов с высоким пространственным разрешением с использованием масс-спектрометрической визуализации MALDI и флуоресцентной маркировки in situ». Протоколы природы . 18 (10): 3050–3079. дои : 10.1038/s41596-023-00864-1 . ПМИД 37674095 . S2CID 261580460 .
- ^ Раппез Л., Стадлер М., Триана С., Гатунгу Р.М., Овчинникова К., Фапале П. и др. (июль 2021 г.). «SpaceM раскрывает метаболические состояния отдельных клеток» . Природные методы . 18 (7): 799–805. дои : 10.1038/s41592-021-01198-0 . ПМЦ 7611214 . ПМИД 34226721 .
- ^ Перейти обратно: а б с Зеноби Р. (декабрь 2013 г.). «Метаболомика одноклеточных: аналитические и биологические перспективы». Наука . 342 (6163): 1243259. doi : 10.1126/science.1243259 . ПМИД 24311695 . S2CID 21381091 .
- ^ Чжан Л., Форман Д.П., Грант П.А., Шреста Б., Муди С.А., Вильерс Ф. и др. (октябрь 2014 г.). «Метаболический анализ отдельных растительных клеток in situ методом капиллярного микроотбора и масс-спектрометрии с ионизацией электрораспылением и разделением по подвижности ионов» . Аналитик . 139 (20): 5079–85. Бибкод : 2014Ана...139.5079Z . дои : 10.1039/C4AN01018C . ПМИД 25109271 .
- ^ Дункан К.Д., Фирестам Дж., Ланекофф И. (январь 2019 г.). «Достижения в области метаболомики отдельных клеток на основе масс-спектрометрии» . Аналитик . 144 (3): 782–793. Бибкод : 2019Ана...144..782D . дои : 10.1039/C8AN01581C . ПМИД 30426983 .
- ^ Хагверди Л., Бюттнер М., Вольф Ф.А., Бюттнер Ф., Тайс Ф.Дж. (октябрь 2016 г.). «Псевдовремя диффузии надежно реконструирует ветвление линии» (PDF) . Природные методы . 13 (10): 845–8. дои : 10.1038/nmeth.3971 . ПМИД 27571553 . S2CID 3594049 .
- ^ Сетти М. и др. Wishbone идентифицирует раздвоенные траектории развития на основе данных об отдельных клетках. Нат. Биотехнология. 34, 637–645 (2016).
- ^ Перейти обратно: а б Шибингер Г., Шу Дж., Табака М., Клири Б., Субраманиан В., Соломон А. и др. (февраль 2019 г.). «Анализ оптимального транспорта экспрессии генов в отдельных клетках определяет траектории развития при перепрограммировании» . Клетка . 176 (4): 928–943.e22. дои : 10.1016/j.cell.2019.01.006 . ПМК 6402800 . ПМИД 30712874 .
- ^ Перейти обратно: а б Чен Х., Альберганте Л., Сюй Ю.И., Ларо К.А., Ло Боско Г., Гуань Дж. и др. (апрель 2019 г.). «Реконструкция одноклеточных траекторий, исследование и картирование данных омики с помощью STREAM» . Природные коммуникации . 10 (1): 1903. Бибкод : 2019NatCo..10.1903C . дои : 10.1038/s41467-019-09670-4 . ПМК 6478907 . ПМИД 31015418 .
- ^ Пандей К., Зафар Х. (август 2022 г.). «Вывод о переходах состояний клеток и пластичности судеб клеток из одиночных клеток с помощью MARGARET» . Исследования нуклеиновых кислот . 50 (15): е86. дои : 10.1093/nar/gkac412 . ПМЦ 9410915 . ПМИД 35639499 .
- ^ Лаборатория Пинелло. Исследование и картографирование реконструкции траектории одной ячейки
Дальнейшее чтение
[ редактировать ]- Лим SB, Лим CT, Лим WT (октябрь 2019 г.). «Одноклеточный анализ циркулирующих опухолевых клеток: почему гетерогенность имеет значение» . Раки . 11 (10): 1595. doi : 10.3390/cancers11101595 . ПМК 6826423 . ПМИД 31635038 .
- Чжоу ВМ, Ян ЙЮ, Го QR, Цзи Х, Ван Х, Сюй ТТ и др. (октябрь 2021 г.). «Приложения микрофлюидики для высокопроизводительного секвенирования отдельных клеток» . Журнал нанобиотехнологий . 19 (1): 312. дои : 10.1186/s12951-021-01045-6 . ПМЦ 8507141 . ПМИД 34635104 .
- Дин Л., Радфар П., Резаи М., Варкиани М.Э. (июль 2021 г.). «Простой в использовании метод изоляции и извлечения одиночных клеток с использованием микрофлюидной статической матрицы капель». Микрохимика Акта . 188 (8): 242. doi : 10.1007/s00604-021-04897-9 . ПМИД 34226955 . S2CID 235738076 .
- Луо С., Лю Х., Се Ф., Арманд Э.Дж., Силетти К., Баккен Т.Э. и др. (март 2022 г.). «Одноядерная мультиомика определяет разнообразие регуляторного генома корковых клеток человека» . Клеточная геномика . 2 (3): 100107. doi : 10.1016/j.xgen.2022.100107 . ПМЦ 9004682 . ПМИД 35419551 .
- Декамп Л., Ле Рой Д., Деман А.Л. (февраль 2022 г.). «Микрофлюидные технологии для выделения ЦОК: обзор 10 лет интенсивных усилий в области жидкой биопсии» . Международный журнал молекулярных наук . 23 (4): 1981. doi : 10.3390/ijms23041981 . ПМЦ 8875744 . ПМИД 35216097 .