Макромен
| Макро | |||
|---|---|---|---|
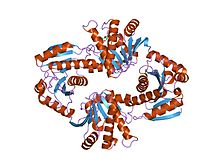 Кристаллическая структура макро-доменного варианта гистонов человека Macroh2a1.1 | |||
| Идентификаторы | |||
| Символ | Макро | ||
| Pfam | PF01661 | ||
| PFAM клан | CL0223 | ||
| InterPro | IPR002589 | ||
| Краткое содержание | 1vhu / scope / supfam | ||
| CDD | CD02749 | ||
| |||
В молекулярной биологии макромен (часто также написанный макродомен) или домен A1PP представляют собой модуль около 180 аминокислот , которые могут связывать ADP-рибозу , NAD метаболит или родственные лиганды . Связывание с ADP-рибозой может быть либо ковалентным , либо нековалентным : [ 1 ] В некоторых случаях это считается нековалентно связывающим, [ 2 ] В то время как в других случаях (таких как апретаксин ) он, по-видимому, связывается как нековалентно через мотив цинкового пальца , так и ковалентно через отдельную область белка . [ 3 ]
Функция
[ редактировать ]Первоначально был описан домен в связи с 1- фосфата ADP-рибозы (APP-1 активностью -P) (A1PP) белка YBR022W дрожжей YBR022W и называемого A1PP. [ 4 ] Тем не менее, домен был переименован в макрос, поскольку он является С-концевым доменом млекопитающих ядра сердечного макро-H2A . [ 5 ] [ 6 ] Макроментные белки могут быть обнаружены у эукариот , в (в основном патогенных) бактериях , у археи и в вирусах ssRNA , таких как коронавирусы , рубца и вирусы гепатита Е. У позвоночных домен происходит в EG гистоновых макрох2A, прогнозируемых поли-адп-рибозных полимераз (PARPS) и B-агрессивной лимфомы (BAL).
ADP-ribosylation of proteins is an important post-translational modification that occurs in a variety of biological processes, including DNA repair, regulation of transcription, chromatin biology, maintenance of genomic stability, telomere dynamics,[7] cell differentiation and proliferation,[8] necrosis and apoptosis,[9] and long-term memory formation.[10] The Macro domain recognises the ADP-ribose nucleotide and in some cases poly-ADP-ribose, and is thus a high-affinity ADP-ribose-binding module found in a number of otherwise unrelated proteins.[11] ADP-ribosylation of DNA is relatively uncommon and has only been described for a small number of toxins that include pierisin,[12] scabin[13] and DarT.[14][15] The Macro domain from the antitoxin DarG of the toxin-antitoxin system DarTG, both binds and removes the ADP-ribose modification added to DNA by the toxin DarT.[14][15] The Macro domain from human, macroH2A1.1, binds an NAD metabolite O-acetyl-ADP-ribose.[16]
| Class | Subclass | Species | Activity |
|---|---|---|---|
| MacroH2A-like | e | ADP-ribose binding | |
| MacroD-type | ‘classic’ | a, b, e, v | ADP-ribosyl bond hydrolysis |
| Zn-dependent | b, e | ADP-ribosyl bond hydrolysis | |
| GDAP2-like | e | ADP-ribose binding | |
| ALC1-like | b, e | ADP-ribose binding or ADP-ribosyl bond hydrolysis | |
| PARG-like | PARG_cat | e | ADP-ribosyl bond hydrolysis |
| mPARG (DUF2263) | b, e, v | ADP-ribosyl bond hydrolysis | |
| Macro2-type | e, v | ADP-ribosyl bond hydrolysis | |
| SUD-M-like | v | RNA binding | |
| DUF2362 | e | unknown | |
| a, Archaea; b, Bacteria; e, Eukarya; v, Virus | |||
Structure
[edit]The 3D structure of the Macro domain describes a mixed alpha/beta fold of a mixed beta sheet sandwiched between four helices with the ligand-binding pocket lies within the fold.[11] Several Macro domain-only domains are shorter than the structure of AF1521 and lack either the first strand or the C-terminal helix 5. Well conserved residues form a hydrophobic cleft and cluster around the AF1521-ADP-ribose binding site.[6][11][16][17]
See also
[edit]References
[edit]- ^ Hassa PO, Haenni SS, Elser M, Hottiger MO (September 2006). "Nuclear ADP-ribosylation reactions in mammalian cells: where are we today and where are we going?". Microbiol. Mol. Biol. Rev. 70 (3): 789–829. doi:10.1128/MMBR.00040-05. PMC 1594587. PMID 16959969.
- ^ Neuvonen M, Ahola T (January 2009). "Differential activities of cellular and viral macro domain proteins in binding of ADP-ribose metabolites". J. Mol. Biol. 385 (1): 212–25. doi:10.1016/j.jmb.2008.10.045. PMC 7094737. PMID 18983849.
- ^ Ahel I, Ahel D, Matsusaka T, Clark AJ, Pines J, Boulton SJ, West SC (January 2008). "Poly(ADP-ribose)-binding zinc finger motifs in DNA repair/checkpoint proteins". Nature. 451 (7174): 81–5. Bibcode:2008Natur.451...81A. doi:10.1038/nature06420. PMID 18172500. S2CID 4417693.
- ^ Martzen MR, McCraith SM, Spinelli SL, Torres FM, Fields S, Grayhack EJ, Phizicky EM (November 1999). "A biochemical genomics approach for identifying genes by the activity of their products". Science. 286 (5442): 1153–5. doi:10.1126/science.286.5442.1153. PMID 10550052.
- ^ Aravind L (May 2001). "The WWE domain: a common interaction module in protein ubiquitination and ADP ribosylation". Trends Biochem. Sci. 26 (5): 273–5. doi:10.1016/s0968-0004(01)01787-x. PMID 11343911.
- ^ Jump up to: a b Allen MD, Buckle AM, Cordell SC, Löwe J, Bycroft M (July 2003). "The crystal structure of AF1521 a protein from Archaeoglobus fulgidus with homology to the non-histone domain of macroH2A". J. Mol. Biol. 330 (3): 503–11. doi:10.1016/S0022-2836(03)00473-X. PMID 12842467.
- ^ Tennen RI, Chua KF (January 2011). "Chromatin regulation and genome maintenance by mammalian SIRT6". Trends in Biochemical Sciences. 36 (1): 39–46. doi:10.1016/j.tibs.2010.07.009. PMC 2991557. PMID 20729089.
- ^ Ji Y, Tulin AV (October 2010). "The roles of PARP1 in gene control and cell differentiation". Current Opinion in Genetics & Development. 20 (5): 512–8. doi:10.1016/j.gde.2010.06.001. PMC 2942995. PMID 20591646.
- ^ Han W, Li X, Fu X (2011). "The macro domain protein family: Structure, functions, and their potential therapeutic implications". Mutation Research. 727 (3): 86–103. doi:10.1016/j.mrrev.2011.03.001. PMC 7110529. PMID 21421074.
- ^ Schreiber V, Dantzer F, Ame JC, de Murcia G (July 2006). "Poly(ADP-ribose): novel functions for an old molecule". Nature Reviews Molecular Cell Biology. 7 (7): 517–28. doi:10.1038/nrm1963. PMID 16829982. S2CID 22030625.
- ^ Jump up to: a b c Karras GI, Kustatscher G, Buhecha HR, Allen MD, Pugieux C, Sait F, Bycroft M, Ladurner AG (June 2005). "The macro domain is an ADP-ribose binding module". EMBO J. 24 (11): 1911–20. doi:10.1038/sj.emboj.7600664. PMC 1142602. PMID 15902274.
- ^ Takamura-Enya, Takeji; Watanabe, Masahiko; Totsuka, Yukari; Kanazawa, Takashi; Matsushima-Hibiya, Yuko; Koyama, Kotaro; Sugimura, Takashi; Wakabayashi, Keiji (2001-10-23). "Mono(ADP-ribosyl)ation of 2′-deoxyguanosine residue in DNA by an apoptosis-inducing protein, pierisin-1, from cabbage butterfly". Proceedings of the National Academy of Sciences. 98 (22): 12414–12419. Bibcode:2001PNAS...9812414T. doi:10.1073/pnas.221444598. ISSN 0027-8424. PMC 60068. PMID 11592983.
- ^ Lyons, Bronwyn; Ravulapalli, Ravikiran; Lanoue, Jason; Lugo, Miguel R.; Dutta, Debajyoti; Carlin, Stephanie; Merrill, A. Rod (2016-05-20). "Scabin, a Novel DNA-acting ADP-ribosyltransferase from Streptomyces scabies". The Journal of Biological Chemistry. 291 (21): 11198–11215. doi:10.1074/jbc.M115.707653. ISSN 1083-351X. PMC 4900268. PMID 27002155.
- ^ Jump up to: a b Jankevicius, Gytis; Ariza, Antonio; Ahel, Marijan; Ahel, Ivan (2016). "The Toxin-Antitoxin System DarTG Catalyzes Reversible ADP-Ribosylation of DNA". Molecular Cell. 64 (6): 1109–1116. doi:10.1016/j.molcel.2016.11.014. PMC 5179494. PMID 27939941.
- ^ Jump up to: a b Schuller, Marion; Butler, Rachel E.; Ariza, Antonio; Tromans-Coia, Callum; Jankevicius, Gytis; Claridge, Tim D. W.; Kendall, Sharon L.; Goh, Shan; Stewart, Graham R.; Ahel, Ivan (2021-08-18). "Molecular basis for DarT ADP-ribosylation of a DNA base". Nature. 596 (7873): 597–602. doi:10.1038/s41586-021-03825-4. hdl:2299/25013. ISSN 1476-4687. PMID 34408320. S2CID 237214909.
- ^ Jump up to: a b Kustatscher G, Hothorn M, Pugieux C, Scheffzek K, Ladurner AG (July 2005). "Splicing regulates NAD metabolite binding to histone macroH2A". Nat. Struct. Mol. Biol. 12 (7): 624–5. doi:10.1038/nsmb956. PMID 15965484. S2CID 29456363.
- ^ Egloff MP, Malet H, Putics A, Heinonen M, Dutartre H, Frangeul A, Gruez A, Campanacci V, Cambillau C, Ziebuhr J, Ahola T, Canard B (September 2006). "Structural and functional basis for ADP-ribose and poly(ADP-ribose) binding by viral macro domains". J. Virol. 80 (17): 8493–502. doi:10.1128/JVI.00713-06. PMC 1563857. PMID 16912299.