Химерная РНК
Химерная РНК , иногда называемая слитым транскриптом , состоит из экзонов двух или более разных генов, которые потенциально могут кодировать новые белки. [ 1 ] Эти мРНК отличаются от мРНК, полученных обычным сплайсингом , поскольку они производятся двумя или более генными локусами.
Обзор производства РНК
[ редактировать ]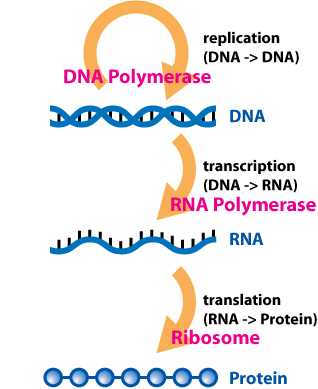
В 1956 году Фрэнсис Крик предложил то, что сейчас известно как « центральная догма » биологии: [ 3 ]
ДНК кодирует генетическую информацию, необходимую организму для осуществления его жизненного цикла. По сути, ДНК служит «жестким диском», на котором хранятся генетические данные. ДНК реплицируется и служит собственной матрицей для репликации. ДНК образует структуру двойной спирали и состоит из сахарофосфатного остова и азотистых оснований; это можно рассматривать как лестничную структуру, где стороны лестницы состоят из дезоксирибозы сахара и фосфата , а ступеньки лестницы состоят из парных азотистых оснований . [ 4 ] В молекуле ДНК четыре основания: аденин (А), цитозин (С), тимин (Т) и гуанин (G). Нуклеотиды являются структурным компонентом ДНК и РНК и состоят из молекулы сахара и молекулы фосфорной кислоты. Двойная спираль ДНК состоит из двух антипараллельных нитей, ориентированных в противоположных направлениях. ДНК состоит из пар оснований, в которых аденин соединяется с тимином, а гуанин соединяется с цитозином. В то время как ДНК служит матрицей для производства рибонуклеиновой кислоты (РНК), РНК обычно отвечает за производство белка. Процесс образования РНК из ДНК называется транскрипцией. РНК использует аналогичный набор оснований, за исключением того, что тимин заменен урацилом . Группа ферментов, называемых РНК-полимеразами (выделенными биохимиками Джерардом Гурвицем и Сэмюэлем Б. Вайсом), функционирует в присутствии ДНК. Эти ферменты производят РНК, используя в качестве матрицы сегменты хромосомной ДНК. В отличие от репликации, при которой создается полная копия ДНК, транскрипция копирует только тот ген, который должен экспрессироваться в виде белка. [ 5 ]
Первоначально считалось, что РНК служит структурной матрицей для синтеза белка , по существу упорядочивая аминокислоты с помощью ряда полостей, специально сформированных так, чтобы подходили только определенные аминокислоты. Крика эта гипотеза не удовлетворила, учитывая, что четыре основания РНК гидрофильны и что многие аминокислоты предпочитают взаимодействовать с гидрофобными группами. Кроме того, некоторые аминокислоты очень схожи по структуре, и Крик считал, что точное различение невозможно, учитывая сходство. Затем Крик предположил, что перед включением в белки аминокислоты сначала прикрепляются к адаптерным молекулам, имеющим уникальные особенности поверхности, которые могут связываться со специфическими основаниями на матрицах РНК. [ 5 ] Эти адаптерные молекулы называются транспортными РНК (тРНК).
Благодаря серии экспериментов с участием E. coli и фага Т4 в 1960 г. [ 5 ] было показано, что информационная РНК (мРНК) переносит информацию от ДНК к рибосомальным участкам синтеза белка. Предшественники тРНК-аминокислот помещаются в положение рибосом , где они могут считывать информацию, предоставленную матрицами мРНК, для синтеза белка.
Сплайсинг РНК
[ редактировать ]Создание белка состоит из двух основных этапов: транскрипции ДНК в РНК и трансляции РНК в белок. После того, как ДНК транскрибируется в РНК, молекула известна как пре-мессенджерная РНК (мРНК) и состоит из экзонов и интронов , которые можно разделить и перестроить множеством различных способов. Исторически экзоны считались кодирующей последовательностью, а интроны считались «мусорной» ДНК. Хотя было доказано, что это неверно, действительно, экзоны часто сливаются. В зависимости от потребностей клетки регуляторные механизмы выбирают, к каким экзонам, а иногда и интронам, присоединиться. Этот процесс удаления фрагментов транскрипта пре-мРНК и объединения их с другими фрагментами называется сплайсингом. Геном человека кодирует около 25 000 генов, но белков производится значительно больше. Это достигается за счет сплайсинга РНК. Экзоны этих 25 000 генов можно соединять разными способами, создавая бесчисленные комбинации транскриптов РНК и, в конечном итоге, бесчисленное количество белков. Обычно экзоны одного и того же транскрипта пре-мРНК сплайсируются вместе. Однако иногда продукты генов или транскрипты пре-мРНК соединяются вместе, так что экзоны из разных транскриптов смешиваются вместе в продукт слияния, известный как химерная РНК. Химерная РНК часто включает экзоны высокоэкспрессируемых генов. [ 1 ] но сам химерный транскрипт обычно экспрессируется на низких уровнях.
Эту химерную РНК затем можно транслировать в слитый белок. Слитые белки очень тканеспецифичны. [ 1 ] и они часто связаны с такими видами рака, как колоректальный рак, рак простаты, [ 6 ] и мезотелиомы. [ 7 ] Они в значительной степени используют сигнальные пептиды и трансмембранные белки , которые могут изменять локализацию белков, что, возможно, способствует фенотипу заболевания.
Открытие химерной РНК
[ редактировать ]В одном из первых исследований по изучению генерации химерной РНК изучалось слияние первых трех экзонов гена, известного как JAZF1, с последними 15 экзонами гена, известного как JJAZ1. [ 8 ] Этот точный транскрипт и полученный в результате белок были обнаружены именно в ткани эндометрия. Хотя эти транскрипты часто обнаруживаются при раке эндометрия, они экспрессируются и в нормальных тканях. Первоначально считалось, что это результат слияния хромосом, но одна группа исследовала, было ли это правдой. Используя Саузерн-блоттинг и флуоресцентную гибридизацию in situ (FISH) на геноме, исследователи не обнаружили никаких доказательств перестройки ДНК. Они решили продолжить исследование, объединив клетки эндометрия человека с фибробластами резус , и обнаружили химерные продукты, содержащие последовательности обоих видов. Эти данные позволяют предположить, что химерная РНК генерируется путем сращивания частей генов, а не путем хромосомных перестроек. Они также провели масс-спектрометрию транслированного белка, чтобы убедиться, что химерная РНК транслируется в белок.
В последнее время достижения в области секвенирования нового поколения значительно снизили стоимость секвенирования, что позволило RNAseq проводить больше проектов . Эти проекты RNAseq способны обнаруживать новые транскрипты РНК вместо традиционных микрочипов , в которых можно обнаружить только известные транскрипты. Глубокое секвенирование позволяет обнаруживать транскрипты даже на очень низких уровнях. Это позволило исследователям обнаружить гораздо больше химерных РНК и слитых белков и облегчило понимание их роли в здоровье и болезнях.
Химерные белковые продукты
[ редактировать ]Многочисленные предполагаемые химерные транскрипты были идентифицированы среди меток экспрессируемых последовательностей высокопроизводительного с использованием технологии секвенирования РНК . У людей химерные транскрипты могут генерироваться несколькими способами, такими как транс-сплайсинг пре-мРНК, сток транскрипции РНК, другие ошибки транскрипции РНК, а также они могут быть результатом слияния генов после межхромосомных транслокаций или перестроек. Среди немногих соответствующих белковых продуктов, которые были охарактеризованы до сих пор, большинство возникает в результате хромосомных транслокаций и связано с раком. Например, слияние генов при хроническом миелогенном лейкозе (ХМЛ) приводит к образованию транскрипта мРНК, который включает 5'-конец гена белка кластерной области точки разрыва (BCR) и 3'-конец гомолога 1 вирусного онкогена мышиного лейкоза Абельсона (ABL). ген. Трансляция этого транскрипта приводит к образованию химерного белка BCR-ABL, который обладает повышенной тирозинкиназной активностью. Химерные транскрипты характеризуют специфические клеточные фенотипы и предположительно функционируют не только в раковых, но и в нормальных клетках. Одним из примеров химеры в нормальных клетках человека является транс-сплайсинг 5'-экзонов Ген JAZF1 на хромосоме 7p15 и 3'-экзоны JJAZ1 ( SUZ12 ) на хромосоме 17q1. Эта химерная РНК транслируется в клетках стромы эндометрия и кодирует антиапоптотический белок. Яркими примерами химерных генов при раке являются слитые гены BCR-ABL, FUS - ERG , MLL -AF6 и MOZ-CBP, экспрессируемые при остром миелоидном лейкозе (ОМЛ), а также химера TMPRSS2-ETS, связанная со сверхэкспрессией онкогена в предстательной железе. рак. [ 1 ]
Характеристики химерных белков
[ редактировать ]Френкель-Моргенштерн и др. определили две основные особенности химерных белков. Они сообщили, что химеры используют сигнальные пептиды и трансмембранные домены для изменения клеточной локализации связанных с ними активностей. Во-вторых, химеры включают родительские гены, экспрессирующиеся на высоком уровне. [ 1 ] Обзор всех функциональных доменов белков, кодируемых химерными транскриптами, показал, что химеры содержат полные белковые домены значительно чаще, чем в случайных наборах данных. [ 9 ]
Базы данных химерных транскриптов
[ редактировать ]Было создано несколько баз данных для включения химерных транскриптов из разных ресурсов с использованием различных вычислительных процедур:
- ХиТаРС [ 10 ] [ 11 ] [ 12 ]
- ХимерБД 2.0 [ 13 ]
- Гибридная база данных [ 14 ]
- TICdb [ 15 ]
- дбкрид [ 16 ]
Вычислительные инструменты для обнаружения химерной РНК
[ редактировать ]Недавние достижения в области высокопроизводительного секвенирования транскриптома проложили путь к новым вычислительным методам обнаружения слияний. Ниже приведены вычислительные инструменты, доступные для обнаружения транскриптов слияния на основе данных RNA-Seq:
- Fusim — это программный инструмент для моделирования транскриптов слияния для всестороннего сравнения методов обнаружения слияния. [ 17 ]
- CRAC объединяет геномные местоположения и локальное покрытие, что позволяет прогнозировать соединения сплайсинга или слитые РНК непосредственно на основе анализа считывания секвенирования РНК. [ 18 ]
- TopHat-Fusion может обнаруживать продукты слияния, происходящие из известных генов, неизвестных генов и неаннотированных вариантов сплайсинга известных генов. [ 19 ]
- FusionAnalyser — это инструмент, предназначенный для выявления перестроек слияния драйверов при раке человека посредством анализа данных парного высокопроизводительного секвенирования транскриптома . [ 20 ]
- ChimeraScan предлагает обнаружение химерной транскрипции между двумя независимыми транскриптами в данных высокопроизводительного секвенирования транскриптома, предоставляя такие функции, как способность обрабатывать длинные (>75 п.н.) парные чтения, обработку неоднозначно картированных прочтений и обнаружение прочтений, охватывающих слияние. . [ 21 ]
- FusionHunter идентифицирует транскрипты слияния на основе транскрипционного анализа считываний парных концов РНК-seq. [ 22 ]
- SplitSeek позволяет de novo прогнозировать соединения сплайсинга в данных короткого считывания РНК-секвенирования, что подходит для обнаружения новых событий сплайсинга и химерных транскриптов. [ 23 ]
- Trans-AB ySS представляет собой конвейер сборки и анализа транскриптома de novo с коротким считыванием, который помогает идентифицировать известные, новые и альтернативные структуры в экспрессируемых транскриптах, таких как химерные транскрипты. [ 24 ]
- FusionSeq идентифицирует транскрипты слияния в результате секвенирования РНК с парными концами. Он включает в себя фильтры для удаления ложных слияний кандидатов с артефактами, такими как несовпадение или случайное спаривание фрагментов транскрипта. [ 25 ]
Необходимо проявлять некоторую осторожность при интерпретации событий транс-сплайсинга, обнаруженных в экспериментах по высокопроизводительному секвенированию, поскольку ферменты обратной транскриптазы, повсеместно используемые для определения последовательностей РНК, способны вводить очевидные события транс-сплайсинга, которые не присутствовали в исходной РНК. [ 26 ] [ 27 ] Однако наличие некоторых химерных РНК было подтверждено другими методами. [ 28 ]
Химерная РНК у низших эукариот
[ редактировать ]Различные низшие эукариоты, включая нематоды и трипаносомы, редко встречаются у высших эукариот, но широко используют транс-сплайсинг для создания химерных РНК. [ 29 ] [ 30 ] В этих организмах реакции сплайсинга между белком, кодирующим РНК, и универсальной последовательностью приводят к присоединению лидера сплайсинга к 5'-концу РНК, образуя функциональную информационную РНК . Эта система позволяет использовать опероны — наборы генов, кодирующих белки, с общей функцией, которые одновременно транскрибируются в одну РНК, а затем соединяются в отдельные информационные РНК, каждая из которых кодирует один белок.
Ссылки
[ редактировать ]- ^ Перейти обратно: а б с д и Френкель-Моргенштерн, М.; Лакруа, В.; Эзкурдия, И.; Левин Ю.; Габашвили А.; Прилуски Дж.; дель Посо, А.; Тресс, М.; Джонсон, Р.; Гиго, Р.; Валенсия, А. (15 мая 2012 г.). «Химеры обретают форму: потенциальные функции белков, кодируемых химерными транскриптами РНК» . Геномные исследования . 22 (7): 1231–1242. дои : 10.1101/гр.130062.111 . ПМК 3396365 . ПМИД 22588898 .
- ^ Хорспул, Дэниел (28 ноября 2008 г.). «Центральная догма молекулярной биохимии ферментов» . Проверено 22 июля 2013 г.
- ^ КРИК, ФРЭНСИС (август 1970 г.). «Центральная догма молекулярной биологии». Природа . 227 (5258): 561–563. Бибкод : 1970Natur.227..561C . дои : 10.1038/227561a0 . ПМИД 4913914 . S2CID 4164029 .
- ^ Гир, RC «Введение в ресурсы молекулярной биологии» . Проверено 22 июля 2013 г.
- ^ Перейти обратно: а б с Джеймс Д. Уотсон; и др. (2007). Молекулярная биология гена (6-е изд.). Сан-Франциско, Калифорния: Бенджамин Каммингс. ISBN 9780805395921 .
- ^ Томлинс, ЮАР; Мехра, Р; Родос, ДР; Смит, ЛР; Роулстон, Д; Хельгесон, Бельгия; Цао, X; Вэй, Джей Ти; Рубин, Массачусетс; Шах, РБ; Чиннаян, AM (1 апреля 2006 г.). «Слияние генов TMPRSS2:ETV4 определяет третий молекулярный подтип рака простаты» . Исследования рака . 66 (7): 3396–400. дои : 10.1158/0008-5472.CAN-06-0168 . ПМИД 16585160 .
- ^ Пангопулос, Иоаннис; Торсен, Джим; Горунова, Людмила; Миччи, Франческа; Хаугом, Лисбет; Дэвидсон, Бен; Хайм, Сверре (1 августа 2013 г.). «Секвенирование РНК идентифицирует слияние генов EWSR1 и YY1 при мезотелиоме с t(14;22)(q32;q12)» Гены, хромосомы и рак . 52 (8): 733–740. дои : 10.1002/gcc.22068 . ПМИД 23630070 . S2CID 28377909 .
- ^ Кунц, Дж.И.; Соренг, Алабама; Нуччи, М.; Куо, ФК; Пауэлс, П.; ван ден Берге, Х.; Цин, ПД; Флетчер, Дж.А.; Склар, Дж. (22 мая 2001 г.). «Частое слияние генов JAZF1 и JJAZ1 в стромальных опухолях эндометрия» . Труды Национальной академии наук . 98 (11): 6348–6353. Бибкод : 2001PNAS...98.6348K . дои : 10.1073/pnas.101132598 . ПМК 33471 . ПМИД 11371647 .
- ^ Френкель-Моргенштерн, М.; Валенсия, А. (11 июня 2012 г.). «Новые комбинации доменов в белках, кодируемых химерными транскриптами» . Биоинформатика . 28 (12): i67–i74. doi : 10.1093/биоинформатика/bts216 . ПМЦ 3371848 . ПМИД 22689780 .
- ^ Гороховский А.; Тагор, С.; Паланде, В.; Малка, А.; Равив-Шай, Д.; Френкель-Моргенштерн, М. (4 января 2017 г.). «ChiTaRS-3.1 - расширенные химерные транскрипты и база данных РНК-секвенирования, соответствующие белок-белковым взаимодействиям» . Исследования нуклеиновых кислот . 45 (Д1): Д790–Д795. дои : 10.1093/nar/gkw1127 . ПМК 5210585 . ПМИД 27899596 .
- ^ Френкель-Моргенштерн, М.; Гороховский А.; Вученович, Д.; Маэстре, Л.; Валенсия, А. (28 января 2015 г.). «ChiTaRS 2.1 — улучшенная база данных химерных транскриптов и данных секвенирования РНК с новыми смысловыми и антисмысловыми химерными РНК-транскриптами» . Исследования нуклеиновых кислот . 43 (Д1): Д68–Д75. дои : 10.1093/nar/gku1199 . ПМЦ 4383979 . ПМИД 25414346 .
- ^ Френкель-Моргенштерн, М.; Гороховский А.; Лакруа, В.; Роджерс, М.; Ибанез, К.; Буллоса, К.; Андрес Леон, Э.; Бен-Гур, А.; Валенсия, А. (9 ноября 2012 г.). «ChiTaRS: база данных химерных транскриптов человека, мыши и плодовых мух, а также данные секвенирования РНК» . Исследования нуклеиновых кислот . 41 (Д1): Д142–Д151. дои : 10.1093/nar/gks1041 . ПМК 3531201 . ПМИД 23143107 .
- ^ Ким, П.; Юн, С.; Ким, Н.; Ли, С.; Ко, М.; Ли, Х.; Канг, Х.; Ким, Дж.; Ли, С. (11 ноября 2009 г.). «ChimerDB 2.0 — обновленная база знаний по слитым генам» . Исследования нуклеиновых кислот . 38 (База данных): D81–D85. дои : 10.1093/nar/gkp982 . ПМЦ 2808913 . ПМИД 19906715 .
- ^ Ким, Дэ Су; Ха, Джэ Вон; Ким, Хеуи-Су (1 января 2007 г.). «HYBRIDdb: база данных гибридных генов в геноме человека» . БМК Геномика . 8 (1): 128. дои : 10.1186/1471-2164-8-128 . ПМК 1890557 . ПМИД 17519042 .
- ^ Ново, Ф.Дж.; де Мендибиль, ИО; Визманос, Дж.Л. (26 января 2007 г.). «TICdb: коллекция картируемых генами контрольных точек транслокации при раке» . БМК Геномика . 8:33 . дои : 10.1186/1471-2164-8-33 . ПМЦ 1794234 . ПМИД 17257420 .
- ^ Конг, Ф.; Чжу, Дж.; Ву, Дж.; Пэн, Дж.; Ван, Ю.; Ван, К.; Фу, С.; Юань, Л.-Л.; Ли, Т. (4 ноября 2010 г.). «dbCRID: база данных хромосомных перестроек при заболеваниях человека» . Исследования нуклеиновых кислот . 39 (База данных): D895–D900. дои : 10.1093/nar/gkq1038 . ПМК 3013658 . ПМИД 21051346 .
- ^ Бруно, Эндрю; Джеффри Мечниковски; Маочунь Цинь; Цзяньминь Ван; Сун Лю (январь 2013 г.). «FUSIM: программный инструмент для моделирования транскриптов слияния» . БМК Биоинформатика . 14 (13): 13. дои : 10.1186/1471-2105-14-13 . ПМК 3637076 . ПМИД 23323884 .
- ^ Филипп, Николя; Салсон, Микаэль; Коммес, Тереза; Соперники, Эрик (1 января 2013 г.). «CRAC: комплексный подход к анализу считываний RNA-seq» . Геномная биология . 14 (3): 30 рандов. дои : 10.1186/gb-2013-14-3-r30 . ПМК 4053775 . ПМИД 23537109 .
- ^ Ким, Дэхван; Зальцберг, Стивен Л. (1 января 2011 г.). «TopHat-Fusion: алгоритм обнаружения новых транскриптов слияния» . Геномная биология . 12 (8): С72. дои : 10.1186/gb-2011-12-8-r72 . ПМК 3245612 . ПМИД 21835007 .
- ^ Пьяцца, Р.; Пирола, А.; Спинелли, Р.; Валлетта, С.; Редаэлли, С.; Магистрони, В.; Гамбакорти-Пассерини, К. (8 мая 2012 г.). «FusionAnalyser: новый графический, управляемый событиями инструмент для обнаружения перестановок слияния» . Исследования нуклеиновых кислот . 40 (16): е123. дои : 10.1093/nar/gks394 . ПМЦ 3439881 . ПМИД 22570408 .
- ^ Айер, МК; Чиннайян, AM; Махер, Калифорния (11 августа 2011 г.). «ChimeraScan: инструмент для идентификации химерной транскрипции в данных секвенирования» . Биоинформатика . 27 (20): 2903–2904. doi : 10.1093/биоинформатика/btr467 . ПМК 3187648 . ПМИД 21840877 .
- ^ Ли, Ю.; Чиен, Дж.; Смит, Д.И.; Ма, Дж. (5 мая 2011 г.). «FusionHunter: идентификация слитых транскриптов при раке с использованием секвенирования парной РНК» . Биоинформатика . 27 (12): 1708–1710. doi : 10.1093/биоинформатика/btr265 . ПМИД 21546395 .
- ^ Амер, Адам; Веттербом, Анна; Феук, Ларс; Голдстоун, Вольф (1 января 2010 г.). «Глобальное и объективное обнаружение соединений сплайсинга на основе данных RNA-seq» . Геномная биология . 11 (3):R3 дои : 10.1186/gb-2010-11-3-r34 . ПМЦ 2864574 . ПМИД 20236510 .
- ^ Робертсон, Гордон; Шейн, Жаклин; Чиу, Ридман; Корбетт, Ричард; Филд, Мэтью; Джекман, Шон Д.; Мангалл, Карен; Ли, Сэм; Окада, Хисанага Марк; Цянь, Дженни Кью; Гриффит, Малачи; Раймонд, Энтони; Тиссен, Нина; Сезард, Тимоти; Баттерфилд, Ярон С; Ньюсом, Ричард; Чан, Саймон К; Она, Ронг; Вархол, Ричард; Камох, Балджит; Прабху, Анна-Лиза; Тэм, Анджела; Чжао, Юн Цзюнь; Мур, Ричард А; Херст, Мартин; Марра, Марк А; Джонс, Стивен Дж.М.; Худлесс, Памела А; Бироль, Инанк (10 октября 2010 г.). «Сборка de novo и анализ данных РНК-секвенирования». Природные методы 7 (11): 909–912. дои : 10.1038/nmeth.1517 . ПМИД 20935650 . S2CID 1034682 .
- ^ Сбонер, Андреа; Хабеггер, Лукас; Пфлюгер, Дороти; Терри, Стефан; Чен, Дэвид З.; Розовский, Джоэл С; Тевари, Ашутош К; Китабаяси, Наоки; Мосс, Бенджамин Дж; Чи, Марк С; Демикелис, Франческа; Рубин, Марк А; Герштейн, Марк Б. (1 января 2010 г.). «FusionSeq: модульная система для поиска слияний генов путем анализа данных секвенирования РНК с парными концами» . Геномная биология . 11 (10): Р104. дои : 10.1186/gb-2010-11-10-r104 . ПМК 3218660 . ПМИД 20964841 .
- ^ Хаусли, Дж; Толлерви, Д. (18 августа 2010 г.). «Очевидный неканонический транс-сплайсинг генерируется обратной транскриптазой in vitro» . ПЛОС ОДИН . 5 (8): e12271. Бибкод : 2010PLoSO...512271H . дои : 10.1371/journal.pone.0012271 . ПМЦ 2923612 . ПМИД 20805885 .
- ^ Макманус, CJ; Дафф, Миссури; Эйппер-Мэйнс, Дж; Грейвли, Британская Колумбия (20 июля 2010 г.). «Глобальный анализ транс-сплайсинга у дрозофилы» . Труды Национальной академии наук Соединенных Штатов Америки . 107 (29): 12975–9. Бибкод : 2010PNAS..10712975M . дои : 10.1073/pnas.1007586107 . ПМЦ 2919919 . ПМИД 20615941 .
- ^ Джебали, С; Лагард, Дж; Капранов П ; Лакруа, В; Борель, С; Мадж, Дж. М.; Ховальд, К.; Фуассак, С; Укла, К; Храст, Дж; Ребекка, П; Мартин, Д; Мюррей, РР; Ян, Х; Гамсари, Л; Лин, С; Белл, я; Дюмэ, Э; Дренков, Дж.; Тресс, ML; Гелпи, JL; Ороско, М; Валенсия, А; ван Беркум, Нидерланды; Лажуа, BR ; Видаль, М; Стаматояннопулос, Дж .; Батут, П; Добин, А; Харроу, Дж.; Хаббард, Т; Декер, Дж; Франкиш, А; Салехи-Аштиани, К; Реймонд, А; Антонаракис, SE; Гиго, Р; Гингерас, ТР (2012). «Доказательства существования сетей транскриптов, состоящих из химерных РНК в клетках человека» . ПЛОС ОДИН 7 (1): e28213. Бибкод : 2012PLoSO... 728213D дои : 10.1371/journal.pone.0028213 . ПМК 3251577 . ПМИД 22238572 .
- ^ Блюменталь, Т. (25 июня 2005 г.). «Транс-сплайсинг и опероны» . Червячная книга : 1–9. дои : 10.1895/wormbook.1.5.1 . ПМИД 18050426 .
- ^ Михаэли, С. (апрель 2011 г.). «Транс-сплайсинг трипаносом: механизм и его влияние на транскриптом паразита». Будущая микробиология . 6 (4): 459–74. дои : 10.2217/fmb.11.20 . ПМИД 21526946 .