Полногеномный нокаут CRISPR-Cas9
Полногеномные скрининги нокаута CRISPR-Cas9 направлены на выяснение взаимосвязи между генотипом и фенотипом путем устранения экспрессии генов в масштабе всего генома и изучения возникающих в результате фенотипических изменений. В этом подходе используется система редактирования генов CRISPR-Cas9 в сочетании с библиотеками одиночных направляющих РНК (sgRNA) , которые предназначены для воздействия на каждый ген в геноме. За последние годы полногеномный скрининг CRISPR стал мощным инструментом для проведения крупномасштабных скринингов потери функции с низким уровнем шума, высокой эффективностью нокаута и минимальными нецелевыми эффектами.

История
[ редактировать ]Ранние исследования Caenorhabditis elegans [ 1 ] и дрозофила меланогастер [ 2 ] [ 3 ] видел крупномасштабные систематические скрининги потери функции (LOF), проводимые посредством насыщающего мутагенеза , демонстрирующие потенциал этого подхода для характеристики генетических путей и идентификации генов с уникальными и важными функциями. Метод насыщающего мутагенеза позже был применен к другим организмам, например к рыбкам данио. [ 4 ] [ 5 ] и мыши. [ 6 ] [ 7 ]
Целенаправленные подходы к нокдауну генов появились в 1980-х годах с помощью таких методов, как гомологичная рекомбинация , [ 8 ] [ 9 ] трансрасщепляющие рибозимы , [ 10 ] [ 11 ] и антисмысловые технологии . [ 12 ] [ 13 ]
К 2000 году технология РНК-интерференции (РНКи) стала быстрым, простым и недорогим методом целенаправленного нокдауна генов и регулярно использовалась для изучения функции генов in vivo у C. elegans . [ 14 ] [ 15 ] [ 16 ] [ 17 ] Действительно, всего через несколько лет после его открытия Файром и др . (1998), [ 18 ] почти все из ~19 000 генов C. elegans были проанализированы с использованием нокдауна на основе RNAi . [ 19 ]
Производство библиотек РНКи облегчило применение этой технологии в масштабе всего генома, а методы на основе РНКи стали преобладающим подходом для полногеномных нокдаун-скринов. [ нужна ссылка ]
Тем не менее, подходы к полногеномному нокдаун-скринингу на основе RNAi имеют свои ограничения. Во-первых, высокие отклонения от цели вызывают проблемы с ложноположительными наблюдениями. [ 20 ] [ 21 ] Кроме того, поскольку РНКи снижает экспрессию генов на посттранскрипционном уровне путем воздействия на РНК, скрининг на основе РНКи приводит лишь к частичному и кратковременному подавлению генов. Хотя в определенных ситуациях частичное нокдаун может быть желательным, требовалась технология с улучшенной эффективностью нацеливания и меньшим количеством побочных эффектов. [ нужна ссылка ]
С момента первоначальной идентификации как прокариотической адаптивной иммунной системы, [ 22 ] бактериальная система типа II, кластеризованная с регулярными интервалами коротких палиндромных повторов (CRISPR) / Cas9, стала простым и эффективным инструментом для генерации целевых LOF-мутаций. [ 23 ] Он был успешно применен для редактирования геномов человека и начал вытеснять РНКи в качестве доминирующего инструмента в исследованиях млекопитающих. [ 24 ] В контексте полногеномных скринингов недавние исследования показали, что скрининги CRISPR/Cas9 способны обеспечить высокоэффективное и полное истощение белка и преодолеть проблемы нецелевого использования, наблюдаемые при скринингах RNAi. [ 25 ] [ 26 ] Подводя итог, можно сказать, что недавнее появление CRISPR-Cas9 значительно расширило наши возможности по проведению крупномасштабных LOF-сканирований. Универсальность и программируемость Cas9 в сочетании с низким уровнем шума, высокой эффективностью нокаутирования и минимальными нецелевыми эффектами сделали CRISPR платформой выбора для многих исследователей, занимающихся нацеливанием и редактированием генов. [ 24 ] [ 27 ]
Методы
[ редактировать ]CRISPR/Cas9 Потеря функции
[ редактировать ]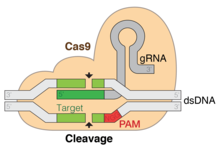
Система кластеризованных коротких палиндромных повторов с регулярными интервалами (CRISPR) / Cas9 представляет собой технологию редактирования генов, которая может вводить двухцепочечные разрывы (DSB) в целевом геномном локусе. Используя одну направляющую РНК (sgRNA) , эндонуклеазу Cas9 можно доставить к определенной последовательности ДНК, где она расщепляет нуклеотидную цепь. [ 28 ] Специфичность sgRNA определяется последовательностью длиной 20 нт, гомологичной интересующему локусу генома, а связывание с Cas9 опосредуется константной каркасной областью sgRNA. За желаемым сайтом-мишенью должен сразу же (от 5' до 3') следовать консервативный мотив из 3 нуклеотидов, примыкающий к протоспейсеру (PAM). [ 29 ] [ 30 ] Чтобы восстановить DSB, клетка может использовать негомологическое соединение концов, подверженное ошибкам , или гомологичную рекомбинацию . Путем создания подходящих sgRNA в геном можно вводить запланированные вставки или делеции. В контексте полногеномных скринингов LOF цель состоит в том, чтобы вызвать разрушение и нокаут генов. [ нужна ссылка ]
библиотеки sgRNA
[ редактировать ]Создание библиотеки
[ редактировать ]Чтобы выполнить нокаут CRISPR в масштабе всего генома, необходимо создать коллекции sgRNA, известные как библиотеки sgRNA или библиотеки нокаута CRISPR. Первым шагом в создании библиотеки sgRNA является идентификация интересующих геномных областей на основе известных правил нацеливания sgRNA. [ 31 ] Например, sgRNA наиболее эффективны при воздействии на кодирующие области генов, а не на 5'- и 3'- UTR . Консервативные экзоны представляют собой привлекательные мишени, поэтому следует учитывать положение относительно места начала транскрипции. [ 31 ] Во-вторых, идентифицируются и выбираются все возможные сайты PAM. [ 31 ] Следует анализировать попадающую и нецелевую активность, а также содержание GC и избегать растяжений гомополимера. [ 31 ] Наиболее часто используемая эндонуклеаза Cas9, полученная из Streptococcus pyogenes , распознает последовательность PAM NGG. [ 32 ]
Более того, определенные нуклеотиды, по-видимому, предпочтительнее в определенных местах. Гуанин имеет явное преимущество перед цитозином в положении 20 рядом с мотивом PAM, а в положении 16 цитозин предпочтительнее гуанина. [ 33 ] Для вариабельного нуклеотида в мотиве NGG PAM было показано, что предпочтительным является цитозин и нежелательным является тимин. [ 33 ] С учетом таких критериев библиотека sgRNA создается с помощью вычислений на основе выбранных сайтов PAM. [ 31 ] [ 33 ] [ 34 ]
Для ограничения ложноположительного обнаружения необходимо создать несколько sgRNA (не менее 4–6) против каждого отдельного гена, а также следует включить sgRNA отрицательного контроля без известных мишеней. [ 31 ] [ 33 ] Затем sgRNA создаются путем синтеза in situ , амплифицируются с помощью ПЦР и клонируются в векторную систему доставки.
Существующие библиотеки
[ редактировать ]Разработка новой библиотеки sgRNA — трудоемкий и трудоемкий процесс. На практике исследователи могут выбрать существующую библиотеку в зависимости от цели эксперимента и интересующих клеточных линий. По состоянию на февраль 2020 года наиболее широко используемыми ресурсами для полногеномного скрининга CRISPR были две библиотеки геномного CRISPR Knock-Out (GeCKO), созданные лабораторией Чжана. [ 35 ] Эти лентивирусные библиотеки, доступные через Addgene, нацелены соответственно на экзоны человека и мыши, и обе доступны в виде одновекторной системы (где sgRNA и Cas9 присутствуют в одной и той же плазмиде) или в виде двухвекторной системы (где sgRNA и Cas9 присутствуют в одной и той же плазмиде). присутствуют на отдельных плазмидах). Каждая библиотека поставляется в виде двух полубиблиотек, что позволяет исследователям проводить скрининг с использованием 3 или 6 sgRNA/ген. [ 36 ]
Помимо GeCKO, был создан ряд других библиотек CRISPR, которые доступны через Addgene. В лабораториях Сабатини и Лендера в настоящее время имеется 7 отдельных библиотек человека и мышей, включая целевые подбиблиотеки для отдельных подпулов, таких как киназы и рибосомальные гены (Addgene # 51043–51048). Кроме того, улучшение специфичности sgRNA привело к созданию библиотек «второго поколения», таких как библиотеки Бри (Addgene #73632) и Брунелло (Addgene #73178), созданные лабораториями Doench и Root, а также нокаутная библиотека Торонто (TKO). (Addgene #1000000069), созданный лабораторией Моффата. [ 36 ]
Лентивирусные векторы
[ редактировать ]
Направленный нокаут гена с использованием CRISPR/Cas9 требует использования системы доставки для введения sgRNA и Cas9 в клетку. Хотя для CRISPR потенциально доступен ряд различных систем доставки, [ 37 ] [ 38 ] полногеномный скрининг потери функции преимущественно проводится с использованием лентивирусных векторов третьего поколения. [ 35 ] [ 39 ] [ 40 ] Эти лентивирусные векторы способны эффективно трансдуцировать широкий спектр типов клеток и стабильно интегрироваться в геном делящихся и неделящихся клеток. [ 41 ] [ 42 ] Лентивирусные частицы третьего поколения получают путем совместной трансфекции клеток эмбриональной почки человека 293T (HEK) с:
- две упаковочные плазмиды, одна кодирует Rev , а другая Gag и Pol ;
- сменную плазмиду оболочки , которая кодирует гликопротеин оболочки другого вируса (чаще всего G-белок вируса везикулярного стоматита (VSV-G));
- одну или две (в зависимости от применяемой библиотеки) плазмиды-переносчики, кодирующие Cas9 и sgRNA, а также маркеры селекции. [ 35 ] [ 43 ] [ 44 ]
Супернатант, содержащий лентивирусные частицы, собирают, концентрируют и впоследствии используют для инфицирования клеток-мишеней. [ 45 ] Точный протокол производства лентивирусов будет варьироваться в зависимости от цели исследования и прикладной библиотеки. [ 35 ] [ 43 ] [ 44 ] Например, если используется двухвекторная система, клетки последовательно трансдуцируются Cas9 и sgRNA в двухэтапной процедуре. [ 35 ] [ 44 ] Несмотря на более сложную структуру, это имеет преимущество более высокого титра вируса библиотеки sgRNA. [ 35 ]
Фенотипический отбор
[ редактировать ]В целом существует два разных формата полногеномных скринингов CRISPR: массив и пул. В массивном скрининге каждая лунка содержит специфическую и известную sgRNA, нацеленную на определенный ген. [ 46 ] Поскольку sgRNA, ответственная за каждый фенотип, известна на основе местоположения лунки, фенотипы можно идентифицировать и анализировать без необходимости генетического секвенирования. Этот формат позволяет измерять более специфические клеточные фенотипы, возможно, с помощью флуоресценции или люминесценции, а также позволяет исследователям использовать больше типов библиотек и методов доставки. [ 46 ] Однако для крупномасштабных скринингов LOF массивные форматы считаются малоэффективными и дорогостоящими с точки зрения финансовых и материальных ресурсов, поскольку популяции клеток необходимо изолировать и культивировать индивидуально. [ 46 ]
При объединенном скрининге клетки, выращенные в одном сосуде, трансдуцируются вирусными векторами, в совокупности содержащими всю библиотеку sgRNA. Чтобы гарантировать, что количество клеток, инфицированных более чем одной sgRNA-содержащей частицей, ограничено, используется низкая множественность заражения (MOI) (обычно 0,3-0,6). [ 46 ] [ 47 ] Имеющиеся данные свидетельствуют о том, что каждая sgRNA должна быть представлена как минимум в 200 клетках. [ 48 ] [ 23 ] Трансдуцированные клетки будут отобраны, после чего будет проведен положительный или отрицательный отбор по интересующему фенотипу, а для идентификации интегрированных sgRNA потребуется генетическое секвенирование. [ 46 ]
Секвенирование и анализ попаданий нового поколения
[ редактировать ]После фенотипической селекции из выбранных клонов экстрагируют геномную ДНК вместе с контрольной популяцией клеток. [ 23 ] [ 46 ] [ 49 ] В наиболее распространенных протоколах полногеномного нокаута «библиотека секвенирования следующего поколения (NGS)» создается с помощью двухэтапной полимеразной цепной реакции (ПЦР). [ 23 ] [ 46 ] На первом этапе амплифицируется область sgRNA с использованием праймеров, специфичных для последовательности интеграции лентивируса, а на втором этапе добавляются последовательности Illumina i5 и i7. [ 23 ] NGS продуктов ПЦР позволяет идентифицировать извлеченные sgRNA, а этап количественного определения можно использовать для определения относительного содержания каждой sgRNA. [ 23 ]
Последним шагом скрининга является компьютерная оценка значительно обогащенных или обедненных sgRNA, отслеживание их соответствующих генов и, в свою очередь, определение того, какие гены и пути могут быть ответственны за наблюдаемый фенотип. В настоящее время для этой цели доступно несколько алгоритмов, наиболее популярным из которых является метод полногеномного анализа CRISPR/Cas9 (MAGeCK). [ 50 ] Разработанный специально для экранов отключения CRISPR/Cas9 в 2014 году, MAGeCK продемонстрировал лучшую производительность по сравнению с альтернативными алгоритмами того времени. [ 50 ] и с тех пор продемонстрировал надежные результаты и высокую чувствительность в различных экспериментальных условиях. [ 51 ] С 2015 года алгоритм MAGeCK был расширен за счет включения измерений контроля качества и учета ранее упускаемой из виду эффективности нокаута sgRNA. [ 51 ] Также был интегрирован веб-инструмент визуализации (VISPR), позволяющий пользователям в интерактивном режиме изучать результаты, анализ и контроль качества. [ 51 ]
Приложения
[ редактировать ]Клеточные сигнальные механизмы
[ редактировать ]За последние годы полногеномный экран CRISPR стал мощным инструментом для изучения сложных сетей клеточной передачи сигналов. [ 52 ] Передача клеточных сигналов необходима для ряда фундаментальных биологических процессов, включая рост клеток, пролиферацию, дифференцировку и апоптоз .
Одним из практических примеров является идентификация генов, необходимых для передачи пролиферативных сигналов в раковых клетках. Клетки трансдуцируются с помощью библиотеки sgRNA CRISPR и изучаются на предмет роста с течением времени. Сравнивая содержание sgRNA в выбранных клетках с контролем, можно определить, какие sgRNA истощаются и, в свою очередь, какие гены могут быть ответственны за дефект пролиферации. Такие скрининги использовались для выявления генов, важных для рака, при остром миелолейкозе. [ 53 ] и нейробластома , [ 54 ] и описать опухолеспецифические различия между линиями раковых клеток. [ 55 ]
Выявление синтетических летальных партнеров
[ редактировать ]Таргетная терапия рака предназначена для воздействия на конкретные гены, белки или среды, способствующие росту или выживанию опухолевых клеток. Однако после периода длительного лечения этими методами у опухолевых клеток может развиться резистентность. Хотя механизмы, лежащие в основе устойчивости рака к лекарственным средствам, плохо изучены, потенциальные причины включают: изменение мишени, деградацию лекарств, ускользание от апоптоза и эпигенетические изменения. [ 56 ] Резистентность широко известна и представляет собой серьезную проблему в лечении рака. [ нужна ссылка ]
Чтобы решить эту проблему, синтетического летального можно идентифицировать партнера. Полногеномные скрининги LOF с использованием CRISPR-Cas9 можно использовать для выявления синтетических летальных партнеров. [ 57 ] Для этого клеточную линию дикого типа и линию опухолевых клеток, содержащую мутацию, вызывающую устойчивость, трансдуцируют с помощью библиотеки sgRNA CRISPR. Две клеточные линии культивируются, и любые недостаточно представленные или мертвые клетки анализируются для выявления потенциальных синтетических летальных генов-партнеров. Недавнее исследование Hinze et al. (2019) [ 58 ] использовали этот метод для выявления синтетического летального взаимодействия между химиотерапевтическим препаратом аспарагиназой и двумя генами сигнального пути Wnt NKD2 и LGR6.
Факторы зависимости от хозяина при вирусной инфекции
[ редактировать ]Из-за своих небольших геномов и ограниченного количества кодируемых белков вирусы используют белки-хозяева для проникновения, репликации и передачи. Идентификация таких белков-хозяев, также называемых факторами зависимости от хозяина (HDF), особенно важна для определения терапевтических целей. В последние годы многие группы успешно использовали полногеномный CRISPR/Cas9 в качестве стратегии скрининга HDF при вирусных инфекциях. [ 59 ]
Один из примеров предоставлен Марсо и др. (2017), [ 60 ] целью которого было проанализировать факторы-хозяева, связанные с инфекцией денге и гепатита С (ВГС) (два вируса семейства Flaviviridae ). Было обнаружено, что ELAVL1 , РНК-связывающий белок, кодируемый геном ELAVL1, является критическим рецептором для проникновения вируса гепатита С, и между двумя флавивирусами было продемонстрировано значительное расхождение в факторах зависимости от хозяина. [ 60 ]
Дальнейшие применения
[ редактировать ]Дополнительные сообщения о применении полногеномных скринингов CRISPR включают изучение: митохондриального метаболизма, [ 61 ] устойчивость к бактериальным токсинам, [ 62 ] генетические факторы метастазирования, [ 63 ] лекарственная устойчивость рака, [ 64 ] Гибель клеток, вызванная вирусом Западного Нила, [ 65 ] и генные сети иммунных клеток. [ 66 ] [ 67 ]
Ограничения
[ редактировать ]В этом разделе конкретно будут рассмотрены полногеномные скрининги CRISPR. Обзор ограничений CRISPR см. в Lino et al. (2018) [ 38 ]
Библиотека sgRNA
[ редактировать ]Полногеномные скрининги CRISPR в конечном итоге будут ограничены свойствами выбранной библиотеки sgRNA. Каждая библиотека будет содержать свой набор sgRNA, и средний охват каждого гена может варьироваться. Доступные в настоящее время библиотеки имеют тенденцию отдавать предпочтение sgRNA, нацеленным на ранние (5') экзоны, кодирующие белок, а не к тем, которые нацелены на более функциональные домены белка. [ 58 ] Эта проблема была подчеркнута Hinze et al. (2019), [ 58 ] которые отметили, что гены, связанные с чувствительностью к аспарагиназе, не удалось выявить в полногеномном скрининге устойчивых к аспарагиназе лейкозных клеток.
Если подходящая библиотека недоступна, создание и амплификация новой библиотеки sgRNA — длительный процесс, который может занять многие месяцы. Потенциальные проблемы включают: (i) эффективный дизайн sgRNA; (ii) обеспечение полного охвата sgRNA по всему геному; (iii) дизайн основной цепи лентивирусного вектора; (iv) производство достаточного количества высококачественного лентивируса; (v) преодоление низкой эффективности трансформации; (vi) правильное масштабирование бактериальной культуры. [ 68 ]
Поддержание клеточного покрытия sgRNA
[ редактировать ]Одним из самых серьезных препятствий для полногеномного скрининга CRISPR является обеспечение адекватного охвата библиотеки sgRNA всей популяции клеток. [ 23 ] Имеющиеся на данный момент данные свидетельствуют о том, что каждая sgRNA должна быть представлена и поддерживаться как минимум в 200-300 клетках. [ 23 ] [ 48 ]
Учитывая, что стандартный протокол использует множественность заражения ~0,3 и эффективность трансдукции 30-40% [ 44 ] [ 23 ] количество клеток, необходимое для создания и поддержания соответствующего покрытия, становится очень большим. Например, самой популярной библиотекой sgRNA человека является библиотека GeCKO v2, созданная лабораторией Чжана; [ 30 ] он содержит 123 411 sgRNA. Исследования с использованием этой библиотеки обычно преобразуют более 1x10 8 клетки [ 58 ] [ 59 ] [ 69 ]
Поскольку CRISPR продолжает демонстрировать низкий уровень шума и минимальные нецелевые эффекты, альтернативной стратегией является уменьшение количества sgRNA на ген для первичного скрининга. Для отбора совпадений используются менее строгие пороговые значения, а дополнительные sgRNA позже используются в более специфичном вторичном скрининге. Этот подход продемонстрирован Doench et al . (2016), [ 33 ] которые обнаружили, что> 92% генов, восстановленных с использованием стандартного протокола, также были восстановлены с использованием меньшего количества sgRNA на ген. Они предполагают, что эта стратегия может быть полезна в исследованиях, где расширение масштабов исследования является непомерно дорогостоящим. [ нужна ссылка ]
Лентивирусные ограничения
[ редактировать ]Лентивирусные векторы имеют определенные общие ограничения. Во-первых, невозможно контролировать, где вирусный геном интегрируется в геном хозяина, и это может повлиять на важные функции клетки. Вануччи и др. [ 70 ] предоставить отличный обзор вирусных векторов, а также их общих преимуществ и недостатков. В конкретном контексте полногеномных скринингов CRISPR производство и трансдукция лентивирусных частиц является относительно трудоемким и трудоемким процессом, занимающим в общей сложности около двух недель. [ 44 ] Кроме того, поскольку ДНК интегрируется в геном хозяина, доставка лентивируса приводит к долгосрочной экспрессии Cas9, что потенциально приводит к нецелевым эффектам. [ нужна ссылка ]
Массивные и объединенные экраны
[ редактировать ]В массивном скрининге каждая лунка содержит специфическую и известную sgRNA, нацеленную на определенный ген. Таким образом, матричные скрининги позволяют получить детальное профилирование одной клетки, но ограничены высокими затратами и трудозатратами, необходимыми для выделения и культивирования большого количества отдельных популяций клеток. [ 46 ] Обычные объединенные скрининги CRISPR относительно просты и экономически эффективны, но ограничиваются исследованием всей популяции клеток. Это означает, что редкие фенотипы может быть труднее идентифицировать, и только грубые фенотипы могут быть выбраны, например, для выживания клеток, пролиферации или экспрессии репортерного гена. [ нужна ссылка ]
Культурные СМИ
[ редактировать ]Выбор культуральной среды может повлиять на физиологическую значимость результатов экспериментов с культурами клеток из-за различий в составе и концентрации питательных веществ. [ 71 ] Недавно была продемонстрирована систематическая погрешность в сгенерированных наборах данных при CRISPR и RNAi скрининге подавления генов (особенно метаболических генов). [ 72 ] и для метаболического профиля линий раковых клеток . [ 71 ] Например, более сильная зависимость от ASNS (аспарагинсинтетазы) была обнаружена в клеточных линиях, культивируемых в DMEM , в которой отсутствует аспарагин, по сравнению с клеточными линиями, культивируемыми в RPMI или F12 (содержащими аспарагин). [ 72 ] Избежать такой систематической ошибки можно, используя единую среду для всех проверяемых клеточных линий и, в идеале, используя питательную среду , которая лучше отражает физиологические уровни питательных веществ. В последнее время такие типы носителей, как Plasmax [ 73 ] и плазмоподобная среда человека (HPLM), [ 74 ] были разработаны.
Будущие направления
[ редактировать ]CRISPR + секвенирование одноклеточной РНК
[ редактировать ]Новые технологии направлены на объединение объединенных скринингов CRISPR с детальным разрешением массивно-параллельного секвенирования одноклеточной РНК (RNA-seq) . Исследования с использованием «CRISP-seq», [ 75 ] «КРОП-сек», [ 76 ] и «PERTURB-seq» [ 77 ] продемонстрировали богатые геномные данные, точно определяя сигнатуры экспрессии генов для отдельных нокаутов генов в сложном пуле клеток. Эти методы имеют дополнительное преимущество, заключающееся в создании профилей транскрипции клеток, индуцированных sgRNA. [ 78 ]
Ссылки
[ редактировать ]- ^ Бреннер С. (май 1974 г.). «Генетика Caenorhabditis elegans» . Генетика . 77 (1): 71–94. дои : 10.1093/генетика/77.1.71 . ПМЦ 1213120 . ПМИД 4366476 .
- ^ Ганс М., Аудит С., Массон М. (декабрь 1975 г.). «Выделение и характеристика сцепленных с полом мутантов с женской стерильностью у Drosophila melanogaster» . Генетика . 81 (4): 683–704. дои : 10.1093/генетика/81.4.683 . ПМЦ 1213428 . ПМИД 814037 .
- ^ Нюсляйн-Фольхард С., Вишаус Е (октябрь 1980 г.). «Мутации, влияющие на количество и полярность сегментов у дрозофилы». Природа . 287 (5785): 795–801. Бибкод : 1980Natur.287..795N . дои : 10.1038/287795a0 . ПМИД 6776413 . S2CID 4337658 .
- ^ Хаффтер П., Гранато М., Брэнд М., Маллинз М.К., Хаммершмидт М., Кейн Д.А. и др. (декабрь 1996 г.). «Идентификация генов с уникальными и важными функциями в развитии рыбок данио Danio rerio». Разработка . 123 : 1–36. дои : 10.1242/dev.123.1.1 . ПМИД 9007226 .
- ^ Дривер В., Солника-Крезель Л., Шир А.Ф., Нойхаусс С.К., Малицкий Дж., Стемпл Д.Л. и др. (декабрь 1996 г.). «Генетический скрининг мутаций, влияющих на эмбриогенез рыбок данио» (PDF) . Разработка . 123 : 37–46. дои : 10.1242/dev.123.1.37 . ПМИД 9007227 .
- ^ Нолан П.М., Питерс Дж., Стривенс М., Роджерс Д., Хаган Дж., Сперр Н. и др. (август 2000 г.). «Систематическая программа полногеномного фенотипического мутагенеза для изучения функций генов у мышей». Природная генетика . 25 (4): 440–3. дои : 10.1038/78140 . ПМИД 10932191 . S2CID 9028853 .
- ^ Касарскис А., Манова К., Андерсон К.В. (июнь 1998 г.). «Скрининг на основе фенотипа эмбриональных летальных мутаций у мышей» . Труды Национальной академии наук Соединенных Штатов Америки . 95 (13): 7485–90. Бибкод : 1998PNAS...95.7485K . дои : 10.1073/pnas.95.13.7485 . ПМК 22659 . ПМИД 9636176 .
- ^ Госслер А., Дучман Т., Корн Р., Серфлинг Э., Кемлер Р. (декабрь 1986 г.). «Трансгенез с помощью линий эмбриональных стволовых клеток, полученных из бластоцист» . Труды Национальной академии наук Соединенных Штатов Америки . 83 (23): 9065–9. Бибкод : 1986PNAS...83.9065G . дои : 10.1073/pnas.83.23.9065 . ПМК 387075 . ПМИД 3024164 .
- ^ Капечки М.Р. (июнь 1989 г.). «Изменение генома путем гомологичной рекомбинации». Наука . 244 (4910): 1288–92. Бибкод : 1989Sci...244.1288C . дои : 10.1126/science.2660260 . ПМИД 2660260 .
- ^ Зауг А.Я., Гроссханс CA, Чехия TR (декабрь 1988 г.). «Последовательность-специфическая эндорибонуклеазная активность рибозима Tetrahymena: усиленное расщепление определенных олигонуклеотидных субстратов, которые образуют несовпадающие комплексы рибозим-субстрат». Биохимия . 27 (25): 8924–31. дои : 10.1021/bi00425a008 . ПМИД 3069131 .
- ^ Кэмерон Ф.Х., Дженнингс, Пенсильвания (декабрь 1989 г.). «Специфическое подавление генов с помощью сконструированных рибозимов в клетках обезьян» . Труды Национальной академии наук Соединенных Штатов Америки . 86 (23): 9139–43. Бибкод : 1989PNAS...86.9139C . дои : 10.1073/pnas.86.23.9139 . ПМК 298449 . ПМИД 2556702 .
- ^ Экер-младший, Дэвис Р.В. (август 1986 г.). «Ингибирование экспрессии генов в растительных клетках путем экспрессии антисмысловой РНК» . Труды Национальной академии наук Соединенных Штатов Америки . 83 (15): 5372–6. Бибкод : 1986PNAS...83.5372E . дои : 10.1073/pnas.83.15.5372 . ПМК 386288 . ПМИД 16593734 .
- ^ Тульме Ж.-Дж., Элен С. (декабрь 1988 г.). «Антимессенджерные олигодезоксирибонуклеотиды: альтернатива антисмысловой РНК для искусственной регуляции экспрессии генов - обзор». Джин . 72 (1–2): 51–8. дои : 10.1016/0378-1119(88)90127-8 . ПМИД 2468575 .
- ^ Чейз Д., Серафинас С., Эшкрофт Н., Косински М., Лонго Д., Феррис Д.К., Голден А. (январь 2000 г.). «Поло-подобная киназа PLK-1 необходима для разрушения ядерной оболочки и завершения мейоза у Caenorhabditis elegans». Бытие . 26 (1): 26–41. doi : 10.1002/(sici)1526-968x(200001)26:1<26::aid-gene6>3.0.co;2-o . ПМИД 10660671 . S2CID 21234791 .
- ^ Кавано Т., Фудзита М., Сакамото Х. (июль 2000 г.). «Уникальные и избыточные функции белков SR, консервативного семейства факторов сплайсинга, в развитии Caenorhabditis elegans» . Механизмы развития . 95 (1–2): 67–76. дои : 10.1016/s0925-4773(00)00339-7 . ПМИД 10906451 . S2CID 17256368 .
- ^ Пауэрс Дж., Боссинджер О., Роуз Д., Строум С., Сакстон В. (октябрь 1998 г.). «Кинезин нематод, необходимый для продвижения борозды дробления» . Современная биология . 8 (20): 1133–6. дои : 10.1016/s0960-9822(98)70470-1 . ПМК 3209536 . ПМИД 9778533 .
- ^ Сюй Ю., Сунь З.В., Ли Х, Рубен М., Тэтчелл К., Бишоп Д.К. и др. (август 2000 г.). «Митотическое фосфорилирование гистона H3 регулируется киназой Ipl1/aurora и фосфатазой Glc7/PP1 у почкующихся дрожжей и нематод» . Клетка . 102 (3): 279–91. дои : 10.1016/s0092-8674(00)00034-9 . ПМИД 10975519 . S2CID 16057773 .
- ^ Файр А, Сюй С, Монтгомери МК, Костас С.А., Драйвер С.Е., Мелло CC (февраль 1998 г.). «Мощное и специфическое генетическое вмешательство двухцепочечной РНК в Caenorhabditis elegans». Природа . 391 (6669): 806–11. Бибкод : 1998Natur.391..806F . дои : 10.1038/35888 . ПМИД 9486653 . S2CID 4355692 .
- ^ Агравал Н., Дасарадхи П.В., Мохмед А., Малхотра П., Бхатнагар Р.К., Мукерджи С.К. (декабрь 2003 г.). «РНК-интерференция: биология, механизм и применение» . Обзоры микробиологии и молекулярной биологии . 67 (4): 657–85. doi : 10.1128/mmbr.67.4.657-685.2003 . ПМК 309050 . ПМИД 14665679 .
- ^ Сигойо Ф.Д., Лайман С., Хакинс Дж.Ф., Адамсон Б., Чунг Э., Кваттроки Б., Кинг Р.В. (февраль 2012 г.). «Метод биоинформатики выявляет заметные нецелевые транскрипты при скрининге РНКи» . Природные методы . 9 (4): 363–6. дои : 10.1038/nmeth.1898 . ПМЦ 3482495 . ПМИД 22343343 .
- ^ Эчеверри С.Дж., Бичи П.А., Баум Б., Бутрос М., Буххольц Ф., Чанда С.К. и др. (октябрь 2006 г.). «Минимизация риска сообщения о ложноположительных результатах при крупномасштабном скрининге РНКи». Природные методы . 3 (10): 777–9. дои : 10.1038/nmeth1006-777 . hdl : 1874/21016 . ПМИД 16990807 . S2CID 1737581 .
- ^ Баррангу Р., Фремо С., Дево Х., Ричардс М., Бояваль П., Муано С. и др. (март 2007 г.). «CRISPR обеспечивает приобретенную устойчивость прокариот к вирусам». Наука . 315 (5819): 1709–12. Бибкод : 2007Sci...315.1709B . дои : 10.1126/science.1138140 . hdl : 20.500.11794/38902 . ПМИД 17379808 . S2CID 3888761 .
- ^ Jump up to: а б с д и ж г час я Яу Э.Х., Рана ТМ (2018). «Секвенирование следующего поколения полногеномных экранов CRISPR». Секвенирование нового поколения . Методы молекулярной биологии. Том. 1712. стр. 203–216. дои : 10.1007/978-1-4939-7514-3_13 . ISBN 978-1-4939-7512-9 . ПМК 6089254 . ПМИД 29224076 .
- ^ Jump up to: а б Бетчер М., Макманус М.Т. (май 2015 г.). «Выбор правильного инструмента для работы: RNAi, TALEN или CRISPR» . Молекулярная клетка . 58 (4): 575–85. doi : 10.1016/j.molcel.2015.04.028 . ПМЦ 4441801 . ПМИД 26000843 .
- ^ Гилберт Л.А., Хорлбек М.А., Адамсон Б., Виллалта Дж.Е., Чен Ю., Уайтхед Э.Х. и др. (октябрь 2014 г.). «CRISPR-опосредованный контроль репрессии и активации генов в масштабе генома» . Клетка . 159 (3): 647–61. дои : 10.1016/j.cell.2014.09.029 . ПМЦ 4253859 . ПМИД 25307932 .
- ^ Конг Л., Ран Ф.А., Кокс Д., Лин С., Барретто Р., Хабиб Н. и др. (февраль 2013 г.). «Мультиплексная геномная инженерия с использованием систем CRISPR/Cas» . Наука . 339 (6121): 819–23. Бибкод : 2013Sci...339..819C . дои : 10.1126/science.1231143 . ПМЦ 3795411 . ПМИД 23287718 .
- ^ Эверс Б., Ястржебски К., Хейманс Дж.П., Грернрум В., Бейерсберген Р.Л., Бернардс Р. (июнь 2016 г.). «Скрининг нокаута CRISPR превосходит shRNA и CRISPRi в идентификации важных генов». Природная биотехнология . 34 (6): 631–3. дои : 10.1038/nbt.3536 . ПМИД 27111720 . S2CID 22384060 .
- ^ Джинек М., Чилински К., Фонфара И., Хауэр М., Дудна Дж.А., Шарпантье Э. (август 2012 г.). «Программируемая ДНК-эндонуклеаза, управляемая двойной РНК, в адаптивном бактериальном иммунитете» . Наука . 337 (6096): 816–21. Бибкод : 2012Sci...337..816J . дои : 10.1126/science.1225829 . ПМК 6286148 . ПМИД 22745249 .
- ^ Ву X, Криз Эй Джей, Sharp PA (июнь 2014 г.). «Целевая специфичность системы CRISPR-Cas9» . Количественная биология . 2 (2): 59–70. дои : 10.1007/s40484-014-0030-x . ПМЦ 4338555 . ПМИД 25722925 .
- ^ Jump up to: а б Чжан Ф, Вэнь Ю, Го X (сентябрь 2014 г.). «CRISPR/Cas9 для редактирования генома: прогресс, последствия и проблемы» . Молекулярная генетика человека . 23 (Р1): Р40-6. дои : 10.1093/hmg/ddu125 . ПМИД 24651067 .
- ^ Jump up to: а б с д и ж Йонг Дж., Конерманн С., Гутенберг Дж.С., Абудайе О.О., Платт Р.Дж., Бригам М.Д. и др. (апрель 2017 г.). «Скрининг нокаута CRISPR-Cas9 и активации транскрипции в масштабе генома» . Протоколы природы . 12 (4): 828–863. дои : 10.1038/нпрот.2017.016 . ПМК 5526071 . ПМИД 28333914 .
- ^ Эндо М., Миками М., Эндо А., Кая Х., Ито Т., Нисимасу Х. и др. (январь 2019 г.). «Редактирование генома растений с помощью разработанного CRISPR-Cas9, распознающего NG PAM». Природные растения . 5 (1): 14–17. дои : 10.1038/s41477-018-0321-8 . ПМИД 30531939 . S2CID 54462288 .
- ^ Jump up to: а б с д и Дёнч Дж.Г., Фузи Н., Саллендер М., Хегде М., Ваймберг Э.В., Донован К.Ф. и др. (февраль 2016 г.). «Оптимизированный дизайн sgRNA для максимизации активности и минимизации нецелевых эффектов CRISPR-Cas9» . Природная биотехнология . 34 (2): 184–191. дои : 10.1038/nbt.3437 . ПМЦ 4744125 . ПМИД 26780180 .
- ^ Канчельери С., Канвер МС, Бомбьери Н., Джуньо Р., Пинелло Л. (ноябрь 2019 г.). «CRISPRitz: быстрая, высокопроизводительная и учитывающая варианты in silico идентификация нецелевых сайтов для редактирования генома CRISPR» . Биоинформатика . 36 (7): 2001–2008. doi : 10.1093/биоинформатика/btz867 . ПМК 7141852 . ПМИД 31764961 .
- ^ Jump up to: а б с д и ж Санджана Н.Е., Шалем О., Чжан Ф. (август 2014 г.). «Улучшенные векторы и полногеномные библиотеки для скрининга CRISPR» . Природные методы . 11 (8): 783–784. дои : 10.1038/nmeth.3047 . ПМЦ 4486245 . ПМИД 25075903 .
- ^ Jump up to: а б «Объединенные библиотеки» . Аддген .
- ^ Сюй С.Л., Руан М.З., Махаджан В.Б., Цанг Ш.Х. (январь 2019 г.). «Системы вирусной доставки для CRISPR» . Вирусы . 11 (1): 28. дои : 10.3390/v11010028 . ПМК 6356701 . ПМИД 30621179 .
- ^ Jump up to: а б Лино К.А., Харпер Дж.К., Карни Дж.П., Тимлин Дж.А. (ноябрь 2018 г.). «Реализация CRISPR: обзор проблем и подходов» . Доставка лекарств . 25 (1): 1234–1257. дои : 10.1080/10717544.2018.1474964 . ПМК 6058482 . ПМИД 29801422 .
- ^ Ван Т., Вэй Джей Джей, Сабатини Д.М., Ландер Э.С. (январь 2014 г.). «Генетические скрининги клеток человека с использованием системы CRISPR-Cas9» . Наука . 343 (6166): 80–4. Бибкод : 2014Sci...343...80W . дои : 10.1126/science.1246981 . ПМК 3972032 . ПМИД 24336569 .
- ^ Шалем О., Санджана Н.Е., Хартениан Э., Ши Х., Скотт Д.А., Миккельсон Т. и др. (январь 2014 г.). «Геномный скрининг нокаута CRISPR-Cas9 в клетках человека» . Наука . 343 (6166): 84–87. Бибкод : 2014Sci...343...84S . дои : 10.1126/science.1247005 . ПМК 4089965 . ПМИД 24336571 .
- ^ Яниз-Галенде Э., Хаджар Р.Дж. (январь 2014 г.). «Стволовые клетки и генная терапия для регенерации сердца». Сердечная регенерация и восстановление . Издательство Вудхед. стр. 347–379. дои : 10.1533/9780857096708.4.347 . ISBN 9780857096586 .
- ^ Пауэлс К., Гейсберс Р., Тулен Дж., Шамбах А., Уиллард-Галло К., Верхойст С. и др. (декабрь 2009 г.). «Современные лентивирусные векторы для исследовательского использования: оценка риска и рекомендации по биобезопасности». Современная генная терапия . 9 (6): 459–74. дои : 10.2174/156652309790031120 . ПМИД 20021330 .
- ^ Jump up to: а б Далл Т., Зуфери Р., Келли М., Мандель Р.Дж., Нгуен М., Троно Д., Налдини Л. (ноябрь 1998 г.). «Лентивирусный вектор третьего поколения с системой условной упаковки» . Журнал вирусологии . 72 (11): 8463–71. doi : 10.1128/JVI.72.11.8463-8471.1998 . ПМЦ 110254 . ПМИД 9765382 .
- ^ Jump up to: а б с д и «Руководство пользователя системы Lenti-X CRISPR/Cas9» (PDF) . Такара Био США.
- ^ Тискорния Г, Сингер О, Верма И.М. (2006). «Производство и очистка лентивирусных векторов». Протоколы природы . 1 (1): 241–5. дои : 10.1038/nprot.2006.37 . ПМИД 17406239 . S2CID 37763028 .
- ^ Jump up to: а б с д и ж г час Агротис А., Кеттелер Р. (2015). «Новая эра функциональной геномики с использованием CRISPR/Cas9 при скрининге массивных библиотек» . Границы генетики . 6 : 300. дои : 10.3389/fgene.2015.00300 . ПМЦ 4585242 . ПМИД 26442115 .
- ^ Юнг А.Т., Чой Ю.Х., Ли А.Х., Хейл С., Понстингл Х., Пикард Д. и др. (октябрь 2019 г.). «Сальмонеллезная инфекция» . мБио . 10 (5). дои : 10.1128/mBio.02169-19 . ПМК 6786873 . ПМИД 31594818 .
- ^ Jump up to: а б Харт Т., Чандрашекхар М., Ареггер М., Стейнхарт З., Браун К.Р., МакЛауд Г. и др. (декабрь 2015 г.). «Экраны CRISPR высокого разрешения выявляют гены фитнеса и генотипическую предрасположенность к раку» . Клетка . 163 (6): 1515–26. дои : 10.1016/j.cell.2015.11.015 . ПМИД 26627737 .
- ^ Слесарев А., Вишванатан Л., Тан Й., Боргшульте Т., Ахтиен К., Разафски Д. и др. (март 2019 г.). «CRISPR/CAS9 нацелен на ЗАХВАТ геномных областей млекопитающих для характеристики с помощью NGS» . Научные отчеты . 9 (1): 3587. Бибкод : 2019НатСР...9.3587С . дои : 10.1038/s41598-019-39667-4 . ПМК 6401131 . ПМИД 30837529 .
- ^ Jump up to: а б Ли В, Сюй Х, Сяо Т, Конг Л, Лав Ми, Чжан Ф и др. (2014). «MAGeCK обеспечивает надежную идентификацию основных генов с помощью скрининга нокаута CRISPR/Cas9 в масштабе генома» . Геномная биология . 15 (12): 554. дои : 10.1186/s13059-014-0554-4 . ПМК 4290824 . ПМИД 25476604 .
- ^ Jump up to: а б с Ли В., Кёстер Дж., Сюй Х., Чен Ч., Сяо Т., Лю Дж. С. и др. (декабрь 2015 г.). «Контроль качества, моделирование и визуализация CRISPR-экранов с помощью MAGeCK-VISPR» . Геномная биология . 16 : 281. дои : 10.1186/s13059-015-0843-6 . ПМЦ 4699372 . ПМИД 26673418 .
- ^ Шарма С., Петсалаки Э. (март 2018 г.). «Применение подходов полногеномного скрининга на основе CRISPR-Cas9 для изучения механизмов клеточной сигнализации» . Международный журнал молекулярных наук . 19 (4): 933. doi : 10.3390/ijms19040933 . ПМК 5979383 . ПМИД 29561791 .
- ^ Целепис К., Койке-Юса Х., Де Брекелер Е., Ли Й., Мецакопян Е., Дови О.М. и др. (октябрь 2016 г.). «Скрининг исключения CRISPR определяет генетические уязвимости и терапевтические цели при остром миелоидном лейкозе» . Отчеты по ячейкам . 17 (4): 1193–1205. дои : 10.1016/j.celrep.2016.09.079 . ПМК 5081405 . ПМИД 27760321 .
- ^ Чен Л., Алекс Г., Дхария Н.В., Росс Л., Инигуес А.Б., Конвей А.С. и др. (январь 2018 г.). «Скрининг CRISPR-Cas9 выявил зависимость нейробластомы, усиленной MYCN, от EZH2» . Журнал клинических исследований . 128 (1): 446–462. дои : 10.1172/JCI90793 . ПМК 5749506 . ПМИД 29202477 .
- ^ Ван Т., Бирсой К., Хьюз Н.В., Крупчак К.М., Пост Ю., Вэй Дж.Дж. и др. (ноябрь 2015 г.). «Идентификация и характеристика важнейших генов в геноме человека» . Наука . 350 (6264): 1096–101. Бибкод : 2015Sci...350.1096W . doi : 10.1126/science.aac7041 . ПМЦ 4662922 . ПМИД 26472758 .
- ^ Лири М., Хирбот С., Лапинска К., Саркар С. (декабрь 2018 г.). «Сенсибилизация раковых клеток, устойчивых к лекарствам: вопрос комбинированной терапии» . Раки . 10 (12): 483. doi : 10.3390/cancers10120483 . ПМК 6315347 . ПМИД 30518036 .
- ^ Ван С., Ван Г., Фэн Икс, Шепард П., Чжан Дж., Тан М. и др. (апрель 2019 г.). «Полногеномные тесты CRISPR выявляют синтетическую летальность дефицита RNASEH2 и ингибирования ATR» . Онкоген . 38 (14): 2451–2463. дои : 10.1038/s41388-018-0606-4 . ПМК 6450769 . ПМИД 30532030 .
- ^ Jump up to: а б с д Хинце Л., Пфиррманн М., Карим С., Дегар Дж., МакГакин Дж., Винджамур Д. и др. (апрель 2019 г.). «Синтетическая летальность активации пути Wnt и аспарагиназы при острых лейкозах, устойчивых к лекарствам» . Раковая клетка . 35 (4): 664–676.e7. doi : 10.1016/j.ccell.2019.03.004 . ПМК 6541931 . ПМИД 30991026 .
- ^ Jump up to: а б Ли Б., Клохиси С.М., Чиа Б.С., Ван Б., Цуй А., Эйзенхауре Т. и др. (январь 2020 г.). «Полногеномный CRISPR-скрин выявляет факторы зависимости от хозяина для заражения вирусом гриппа А» . Природные коммуникации . 11 (1): 164. Бибкод : 2020NatCo..11..164L . дои : 10.1038/s41467-019-13965-x . ПМЦ 6952391 . ПМИД 31919360 .
- ^ Jump up to: а б Марсо С.Д., Пушник А.С., Майзуб К., Оой Ю.С., Брюэр С.М., Фукс Г. и др. (июль 2016 г.). «Генетическое исследование факторов-хозяев Flaviviridae с помощью полногеномного скрининга CRISPR» . Природа . 535 (7610): 159–63. Бибкод : 2016Natur.535..159M . дои : 10.1038/nature18631 . ПМЦ 4964798 . ПМИД 27383987 .
- ^ Бирсой К., Ван Т., Чен В.В., Фрейнкман Э., Абу-Ремайлех М., Сабатини Д.М. (июль 2015 г.). «Основная роль митохондриальной цепи транспорта электронов в пролиферации клеток заключается в обеспечении синтеза аспартата» . Клетка . 162 (3): 540–51. дои : 10.1016/j.cell.2015.07.016 . ПМЦ 4522279 . ПМИД 26232224 .
- ^ Койке-Юса Х., Ли Й., Тан Э.П., Веласко-Эррера М., Юса К. (март 2014 г.). «Полногеномный рецессивный генетический скрининг в клетках млекопитающих с использованием лентивирусной библиотеки CRISPR-гид РНК». Природная биотехнология . 32 (3): 267–73. дои : 10.1038/nbt.2800 . ПМИД 24535568 . S2CID 23071292 .
- ^ Чен С., Санджана Н.Э., Чжэн К., Шалем О., Ли К., Ши Икс и др. (март 2015 г.). «Полногеномный CRISPR-скрин на мышиной модели роста опухоли и метастазирования» . Клетка . 160 (6): 1246–60. дои : 10.1016/j.cell.2015.02.038 . ПМК 4380877 . ПМИД 25748654 .
- ^ Ши Дж., Ван Э., Милаццо Дж. П., Ван З., Кинни Дж. Б., Вакоч Ч. Р. (июнь 2015 г.). «Обнаружение мишеней для лечения рака с помощью скрининга белковых доменов CRISPR-Cas9» . Природная биотехнология . 33 (6): 661–7. дои : 10.1038/nbt.3235 . ПМЦ 4529991 . ПМИД 25961408 .
- ^ Ма Х., Данг Ю., Ву Ю., Цзя Г., Анайя Э., Чжан Дж. и др. (июль 2015 г.). «Скрининг на основе CRISPR идентифицирует гены, необходимые для гибели клеток, вызванной вирусом Западного Нила» . Отчеты по ячейкам . 12 (4): 673–83. дои : 10.1016/j.celrep.2015.06.049 . ПМК 4559080 . ПМИД 26190106 .
- ^ Парнас О., Йованович М., Эйзенхауре Т.М., Хербст Р.Х., Диксит А., Йе С.Дж. и др. (июль 2015 г.). «Полногеномный CRISPR-скрин в первичных иммунных клетках для анализа регуляторных сетей» . Клетка . 162 (3): 675–86. дои : 10.1016/j.cell.2015.06.059 . ПМЦ 4522370 . ПМИД 26189680 .
- ^ Шмид-Бургк Дж.Л., Чаухан Д., Шмидт Т., Эберт Т.С., Рейнхардт Дж., Эндл Э., Хорнунг В. (январь 2016 г.). «Полногеномный экран CRISPR (кластеризованные регулярные короткие палиндромные повторы) идентифицирует NEK7 как важный компонент активации воспаления NLRP3» . Журнал биологической химии . 291 (1): 103–9. дои : 10.1074/jbc.C115.700492 . ПМЦ 4697147 . ПМИД 26553871 .
- ^ Куинн Т. «Преимущества и проблемы объединенных библиотек» . Серия вебинаров . Такара Био США.
- ^ Фан З., Вэн С., Ли Х., Тао Р., Май В., Лю Икс и др. (март 2019 г.). «Анализ одноклеточной гетерогенности и скрининг CRISPR позволяют выявить ключевые гены заболеваний, специфичных для β-клеток» . Отчеты по ячейкам . 26 (11): 3132–3144.e7. дои : 10.1016/j.celrep.2019.02.043 . ПМК 6573026 . PMID 30865899 .
- ^ Ваннуччи Л., Лай М., Кьюппези Ф., Чеккерини-Нелли Л., Пистелло М. (январь 2013 г.). «Вирусные векторы: взгляд назад и вперед на технологию переноса генов». Новая микробиология . 36 (1): 1–22. ПМИД 23435812 .
- ^ Jump up to: а б Лагзиел С., Готлиб Э., Шломи Т. (2020). «Заботьтесь о своих СМИ» . Природный метаболизм . 2 (12): 1369–1372. дои : 10.1038/s42255-020-00299-y . ПМИД 33046912 . S2CID 222319735 .
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка ) - ^ Jump up to: а б Лагзиел С., Ли В.Д., Шломи Т. (2019). «Вывод о зависимости рака от метаболических генов на основе крупномасштабного генетического скрининга» . БМК Биол . 17 (1): 37. дои : 10.1186/s12915-019-0654-4 . ПМК 6489231 . ПМИД 31039782 .
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка ) - ^ Ванде Вурде Дж., Акерманн Т., Пфетцер Н., Самптон Д., Маккей Г., Кална Г.; и др. (2019). «Улучшение метаболической точности моделей рака с помощью физиологической среды для культивирования клеток» . Научный адв . 5 (1): eaau7314. Бибкод : 2019SciA....5.7314V . дои : 10.1126/sciadv.aau7314 . ПМК 6314821 . ПМИД 30613774 .
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка ) - ^ Кантор-младший, Абу-Ремайлех М, Канарек Н, Фрейнкман Э, Гао X, Луисен А; и др. (2017). «Физиологическая среда изменяет клеточный метаболизм и обнаруживает мочевую кислоту как эндогенный ингибитор UMP-синтазы» . Клетка . 169 (2): 258–272.e17. дои : 10.1016/j.cell.2017.03.023 . ПМЦ 5421364 . ПМИД 28388410 .
{{cite journal}}: CS1 maint: несколько имен: список авторов ( ссылка ) - ^ Джайтин Д.А., Вайнер А., Йофе И., Лара-Астиасо Д., Керен-Шауль Х., Дэвид Э. и др. (декабрь 2016 г.). «Рассечение иммунных цепей путем объединения скринингов, полученных с помощью CRISPR, с секвенированием одноклеточной РНК» . Клетка . 167 (7): 1883–1896.e15. дои : 10.1016/j.cell.2016.11.039 . ПМИД 27984734 .
- ^ Датлингер П., Рендейро А.Ф., Шмидл С., Краусгрубер Т., Тракслер П., Клугхаммер Дж. и др. (март 2017 г.). «Объединенный скрининг CRISPR с считыванием транскриптома одной клетки» . Природные методы . 14 (3): 297–301. дои : 10.1038/nmeth.4177 . ПМЦ 5334791 . ПМИД 28099430 .
- ^ Диксит А., Парнас О., Ли Б., Чен Дж., Фулко С.П., Джерби-Арнон Л. и др. (декабрь 2016 г.). «Perturb-Seq: анализ молекулярных цепей с помощью масштабируемого профилирования одноклеточной РНК объединенных генетических экранов» . Клетка . 167 (7): 1853–1866.e17. дои : 10.1016/j.cell.2016.11.038 . ПМК 5181115 . ПМИД 27984732 .
- ^ Альбертс, Брюс (2014). Молекулярная биология клетки (6-е изд.). Гирляндная наука.