Цифровая полимеразная цепная реакция
Судя по всему, основной автор этой статьи тесно связан с ее предметом. ( Август 2019 г. ) |
Цифровая полимеразная цепная реакция ( цифровая ПЦР , цифровая ПЦР , дПЦР или деПЦР ) представляет собой биотехнологическое усовершенствование традиционных методов полимеразной цепной реакции , которое можно использовать для прямого количественного определения и клональной амплификации цепей нуклеиновых кислот, включая ДНК , кДНК или РНК . Ключевое различие между дПЦР и кПЦР заключается в методе измерения количества нуклеиновых кислот, причем первый метод является более точным, чем ПЦР, но также более подвержен ошибкам в руках неопытных пользователей. [1] ПЦР проводит одну реакцию на один образец. dPCR также выполняет одну реакцию внутри образца, однако образец разделен на большое количество частей, и реакция проводится в каждой части отдельно. Такое разделение обеспечивает более надежный сбор и чувствительное измерение количества нуклеиновой кислоты. Было продемонстрировано, что этот метод полезен для изучения вариаций последовательностей генов, таких как варианты числа копий и точковые мутации.
Принципы
[ редактировать ]
Метод полимеразной цепной реакции используется для количественного определения нуклеиновых кислот путем амплификации молекулы нуклеиновой кислоты ферментом ДНК-полимеразой . [2] Обычная ПЦР основана на теории, согласно которой амплификация является экспоненциальной. Следовательно, нуклеиновые кислоты можно определить количественно путем сравнения количества циклов амплификации и количества конечного продукта ПЦР с таковыми в эталонном образце. [3] Однако многие факторы усложняют этот расчет, создавая неопределенности и неточности. К этим факторам относятся следующие: начальные циклы амплификации могут не быть экспоненциальными; ПЦР-амплификация в конечном итоге выходит на плато после неопределенного количества циклов; и низкие начальные концентрации молекул нуклеиновой кислоты-мишени могут не амплифицироваться до обнаруживаемых уровней. Однако наиболее существенным ограничением ПЦР является то, что эффективность ПЦР-амплификации в интересующем образце может отличаться от эффективности эталонных образцов.

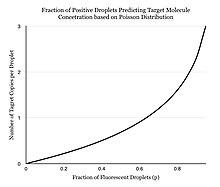
Вместо выполнения одной реакции на лунку, дПЦР предполагает разделение раствора ПЦР на десятки тысяч капель размером нанолитра, в каждой из которых происходит отдельная реакция ПЦР. [4] [5] Раствор для ПЦР готовится аналогично анализу TaqMan , который состоит из матричной ДНК (или РНК), зондов-гасителей флуоресценции, праймеров и мастер-микса ПЦР , который содержит ДНК-полимеразу , dNTP , MgCl 2 и реакционные буферы в оптимальных концентрациях. . Для разделения образцов можно использовать несколько различных методов, включая планшеты с микролунками, капилляры, масляную эмульсию и массивы миниатюрных камер с поверхностями, связывающими нуклеиновые кислоты. [6] Раствор для ПЦР разделен на более мелкие блоки, каждый из которых содержит необходимые компоненты для амплификации. Затем разделенные единицы подвергают термоциклированию, чтобы каждая единица могла независимо подвергаться ПЦР-амплификации. После нескольких циклов ПЦР-амплификации образцы проверяются на флуоресценцию с двоичным показанием «0» или «1». Регистрируют долю флуоресцирующих капель. [5] Разделение образца позволяет оценить количество различных молекул, предполагая, что популяция молекул соответствует распределению Пуассона , что учитывает возможность существования нескольких целевых молекул в одной капле. Используя закон малых чисел Пуассона, можно точно аппроксимировать распределение целевой молекулы в образце, что позволяет количественно оценить целевую цепь в продукте ПЦР. [7] Эта модель просто предсказывает, что по мере увеличения количества образцов, содержащих хотя бы одну целевую молекулу, увеличивается вероятность того, что образцы будут содержать более одной целевой молекулы. [8] В обычной ПЦР количество циклов ПЦР-амплификации пропорционально начальному числу копий. В отличие от мнения многих людей о том, что дПЦР обеспечивает абсолютную количественную оценку, цифровая ПЦР использует статистическую мощность для обеспечения относительной количественной оценки. Например, если Образец А при анализе в 1 миллионе делений дает одну положительную реакцию, это не означает, что Образец А имеет одну исходную молекулу. [ нужна ссылка ]
Преимущества dPCR включают повышенную точность за счет массового разделения образцов, что обеспечивает надежные измерения в желаемой последовательности ДНК благодаря воспроизводимости. [5] Частота ошибок выше при обнаружении различий в небольших кратных изменениях с помощью базовой ПЦР, тогда как частота ошибок меньше при использовании дПЦР из-за различий в меньших кратных изменениях, которые можно обнаружить в последовательности ДНК. Сама методика позволяет сократить использование большего объема необходимого реагента, что неизбежно снизит стоимость эксперимента. Кроме того, dPCR является высококоличественным, поскольку он не зависит от относительной флуоресценции раствора для определения количества амплифицированной целевой ДНК.
Сравнение dPCR и ПЦР в реальном времени (qPCR)
[ редактировать ]dPCR измеряет фактическое количество молекул (целевой ДНК), поскольку каждая молекула находится в одной капле, что делает его дискретным «цифровым» измерением. Он обеспечивает абсолютную количественную оценку, поскольку dPCR измеряет положительную фракцию образцов, то есть количество капель, флуоресцирующих вследствие правильной амплификации. Эта положительная фракция точно указывает на исходное количество матричной нуклеиновой кислоты. Точно так же в qPCR используется флуоресценция; однако он измеряет интенсивность флуоресценции в определенное время (обычно после каждого цикла амплификации), чтобы определить относительное количество целевой молекулы (ДНК), но не может указать точное количество без построения стандартной кривой с использованием различных количеств определенного стандарта. Он дает порог на цикл (CT), а разница в CT используется для расчета количества исходной нуклеиновой кислоты. По сути, qPCR представляет собой аналоговое измерение, которое может быть не столь точным из-за экстраполяции, необходимой для получения измерения. [6] [9]
dPCR измеряет количество ДНК после завершения амплификации, а затем определяет долю повторов. Это типично для измерения конечной точки, поскольку требует наблюдения за данными после завершения эксперимента. Напротив, кПЦР записывает относительную флуоресценцию ДНК в определенных точках процесса амплификации, что требует остановки экспериментального процесса. Этот аспект qPCR «в реальном времени» теоретически может повлиять на результаты из-за остановки эксперимента. [ нужна ссылка ] Однако на практике большинство термоциклеров кПЦР очень быстро считывают флуоресценцию каждого образца в конце этапа отжига/удлинения, прежде чем переходить к следующему этапу плавления, а это означает, что эта гипотетическая проблема на самом деле не актуальна и не применима для подавляющего большинства исследователей. dPCR измеряет амплификацию путем измерения продуктов конечного цикла ПЦР и, следовательно, менее восприимчив к артефактам, возникающим из-за снижения эффективности амплификации из-за присутствия ингибиторов ПЦР или несоответствия матрицы праймеров. [10] [11]
Цифровая ПЦР в реальном времени (rdPCR) сочетает в себе методологии цифровой ПЦР (dPCR) и количественной ПЦР (qPCR), объединяя точность dPCR с возможностями анализа в реальном времени qPCR. Целью такой интеграции является обеспечение повышенной чувствительности, специфичности и возможности абсолютного количественного определения последовательностей нуклеиновых кислот, что способствует количественной оценке генетического материала в научных и клинических исследованиях. [12] [13]
qPCR не может различить различия в экспрессии генов или вариации числа копий, меньшие, чем в два раза. С другой стороны, dPCR имеет более высокую точность и, как было показано, обнаруживает различия менее 30% в экспрессии генов, различает вариации числа копий, которые отличаются только на 1 копию, и идентифицирует аллели, которые встречаются с частотой менее 0,1%. [14] [5]
Приложения
[ редактировать ]Цифровая ПЦР имеет множество применений в фундаментальных исследованиях , клинической диагностике и тестировании окружающей среды. Его использование включает обнаружение патогенов и здоровья пищеварительной системы ; анализ [15] [16] жидкая биопсия для мониторинга рака , мониторинга отторжения трансплантата и неинвазивного пренатального тестирования на серьезные генетические аномалии ; [17] [18] [19] [20] [21] [22] [23] [24] изменения количества копий , анализ [25] [26] [27] анализ экспрессии одного гена, [28] обнаружение редкой последовательности, [24] [29] [30] профилирование экспрессии генов и анализ отдельных клеток ; [31] [32] [30] [33] [34] [35] [36] обнаружение примесей ДНК при биопереработке, [37] проверка правок генов и обнаружение специфических изменений метилирования ДНК как биомаркеров рака . [38] [39] [40] dPCR также часто используется в качестве ортогонального метода для подтверждения редких мутаций, обнаруженных с помощью секвенирования следующего поколения (NGS), и для проверки библиотек NGS . [41] [42] [43]
Абсолютная количественная оценка
[ редактировать ]dPCR обеспечивает абсолютную и воспроизводимую количественную оценку целевых нуклеиновых кислот с разрешением одной молекулы. [30] [44] [45] [46] Однако, в отличие от аналоговой количественной ПЦР (кПЦР), абсолютное количественное определение с помощью дПЦР не требует стандартной кривой . [44] dPCR также имеет большую толерантность к веществам-ингибиторам и ПЦР-анализам, которые неэффективно амплифицируются по сравнению с qPCR. [47] [11]
dPCR может, например, количественно определить наличие специфических последовательностей, контаминирующих генетически модифицированные организмы в пищевых продуктах, [48] вирусная нагрузка в крови, [49] МКПК , [50] [51] образцы сыворотки, [52] ткани ворсин хориона, [50] [51] биомаркеры нейродегенеративных заболеваний в спинномозговой жидкости, [53] и фекальное загрязнение питьевой воды. [54]
Копировать вариант номера
[ редактировать ]Изменение состояния числа копий по отношению к референтному локусу с одной копией называется « вариацией числа копий » (CNV), если оно появляется в клетках зародышевой линии, или изменением числа копий (CNA), если оно появляется в соматических клетках. [55] CNV или CNA могут возникать в результате делеции или амплификации локуса в зависимости от количества копий эталонного локуса, присутствующих в клетке, и вместе они вносят основной вклад в изменчивость генома человека . [56] [57] [58] Они были связаны с раком; [59] [60] [61] неврологический, [62] психиатрический, [63] [64] и аутоиммунные заболевания; [65] и побочные реакции на лекарства. [66] Однако эти аллельные вариации трудно измерить с высокой точностью, используя другие методы, такие как qPCR, что затрудняет связь фенотипов и заболеваний с измененным статусом CNV. [67] [68]
Большое количество «оцифрованных» измерений конечных точек, ставших возможными благодаря разделению образцов, позволяет dPCR разрешать небольшие различия в количестве копий с большей точностью и точностью по сравнению с другими методами, такими как микрочипы на основе SNP. [69] или кПЦР. [70] [71] qPCR ограничена в своей способности точно количественно определять амплификацию генов при некоторых заболеваниях, включая болезнь Крона, инфекцию ВИЧ-1 и ожирение. [72] [68] [71]
dPCR был разработан для измерения концентрации целевой нуклеиновой кислоты в копиях на единицу объема образца. При работе в разбавленных реакциях, когда менее ~10% перегородок содержат желаемую мишень (так называемое «ограничивающее разведение»), число копий можно оценить путем сравнения количества флуоресцентных капель, возникающих из целевого CNV, с количеством флуоресцентных капель. капли, возникающие из инвариантного однокопийного референсного локуса. [25] Фактически, как при этих более низких концентрациях цели, так и при более высоких, когда несколько копий одной и той же цели могут совместно локализоваться в одном разделе, статистика Пуассона используется для коррекции этих множественных заполнений, чтобы дать более точное значение для каждой концентрации цели. [73] [6]
Цифровая ПЦР использовалась для выявления как зародышевых, так и соматических различий в количестве копий генов между людьми. [74] и изучить связь между усилением HER2 (ERBB2) и прогрессированием рака молочной железы . [75] [76] [77] [27]
Редкая мутация и обнаружение редких аллелей
[ редактировать ]Разделение в цифровой ПЦР повышает чувствительность и позволяет обнаруживать редкие события, особенно однонуклеотидные варианты (SNV), путем изоляции или значительного уменьшения сигнала целевого биомаркера от потенциально конкурирующего фона. [9] [6] Эти события можно разделить на два класса: обнаружение редких мутаций и обнаружение редких последовательностей.
Обнаружение редких мутаций
[ редактировать ]Обнаружение редких мутаций происходит, когда биомаркер существует на фоне очень распространенного аналога, который отличается только одним нуклеотидным вариантом (SNV). Было показано, что цифровая ПЦР способна обнаруживать мутантную ДНК в присутствии 200 000-кратного избытка фона дикого типа , что в 2000 раз более чувствительно, чем достижимое с помощью обычной кПЦР. [9]
Обнаружение редкой последовательности
[ редактировать ]Цифровая ПЦР может обнаружить редкие последовательности, такие как ДНК ВИЧ, у пациентов с ВИЧ. [24] и ДНК фекальных бактерий в океанских и других пробах воды для оценки качества воды. [78] dPCR может обнаружить последовательности, встречающиеся всего в 1 на каждые 1 250 000 клеток. [24]
Жидкая биопсия
[ редактировать ]Способность дПЦР выявлять редкие мутации может оказаться особенно полезной в клинике благодаря использованию жидкой биопсии , как правило, неинвазивной стратегии обнаружения и мониторинга заболеваний с помощью жидкостей организма. [17] [79] Исследователи использовали жидкую биопсию для мониторинга опухолевой нагрузки, реакции на лечение и прогрессирования заболевания у онкологических больных путем измерения редких мутаций в циркулирующей опухолевой ДНК (цДНК) в различных биологических жидкостях пациентов, включая кровь , мочу и спинномозговую жидкость . [17] [80] [81] Раннее выявление ктДНК (как при молекулярном рецидиве ) может привести к более раннему назначению иммунотерапии или таргетной терапии, специфичной для сигнатуры мутации пациента, потенциально повышая шансы на эффективность лечения, а не дожидаясь клинического рецидива перед изменением лечения. Срок выполнения жидкостной биопсии может составлять несколько дней, по сравнению с двумя-четырьмя неделями или дольше для тестов на тканях. [82] [83] Это сокращение времени получения результатов было использовано врачами для ускорения лечения, адаптированного к биопсии . данным [82]
В 2016 году проспективное исследование с использованием dPCR в Институте рака Дана-Фарбер подтвердило клиническую пользу жидкой биопсии как инструмента прогностической диагностики для пациентов с немелкоклеточным раком легких . [84] Применение тестов жидкой биопсии также изучалось у пациентов с заболеваниями молочной железы , [85] колоректальный , [86] [87] гинекологический , [88] и мочевого пузыря рак [80] [89] для мониторинга как нагрузки заболевания, так и реакции опухоли на лечение.
Экспрессия генов и количественная оценка РНК
[ редактировать ]Исследования экспрессии генов и количественного определения РНК выиграли от повышения точности и абсолютного количественного определения dPCR. [90] Количественное определение РНК можно выполнить с помощью RT-PCR , где РНК подвергается обратной транскрипции в кДНК в самой реакции разделения, а количество молекул РНК, происходящих из каждого транскрипта (или аллельного транскрипта), количественно определяется с помощью dPCR. [31]
Часто можно добиться большей чувствительности и точности, используя дПЦР вместо кПЦР для количественного определения молекул РНК, отчасти потому, что для количественного определения не требуется использование стандартной кривой. [91] dPCR также более устойчив к ингибиторам ПЦР для количественного определения РНК, чем qPCR. [47] [16] [90]
dPCR может обнаруживать и количественно определять больше отдельных видов мишеней на каждый канал обнаружения, чем qPCR, благодаря способности различать цели на основе их дифференциальной амплитуды флуоресценции или за счет использования отличительных цветовых комбинаций для их обнаружения. [92] [90] В качестве примера можно привести двухканальную систему dPCR для обнаружения в одной лунке экспрессии четырех различных вариантов сплайсинга обратной транскриптазы теломеразы человека , белка, который более активен в большинстве опухолевых клеток, чем в здоровых клетках. [93]
Альтернативное использование разделения
[ редактировать ]Используя возможности динамического разделения, используемые в dPCR, улучшенное секвенирование NGS может быть достигнуто путем разделения сложных реакций ПЦР перед амплификацией, чтобы обеспечить более равномерную амплификацию по множеству различных ампликонов для анализа NGS . [94] [95] Кроме того, было показано, что улучшенная специфичность сложных реакций ПЦР-амплификации в каплях значительно сокращает количество итераций, необходимых для выбора аптамеров с высоким сродством в методе SELEX . [96] Разделение также может обеспечить более надежные измерения активности теломеразы из клеточных лизатов. [97] [98] Возможности динамического разделения dPCR также можно использовать для разделения тысяч ядер или целых клеток на отдельные капли, чтобы облегчить подготовку библиотеки для анализа одной клетки на доступный для транспозазы хроматин с использованием секвенирования (scATAC-seq). [99]
Капельная цифровая ПЦР
[ редактировать ]Цифровая ПЦР с каплями (ddPCR) — это метод dPCR, при котором реакционная проба объемом 20 микролитров, включая праймеры для анализа и либо зонды Такмана, либо интеркалирующий краситель, разделяется на ~ 20 000 капель масла размером примерно в 20 000 нанолитров с помощью метода водно-масляной эмульсии , подвергаемой термоциклированию для конечная точка в 96-луночном планшете для ПЦР и амплитуда флуоресценции, считываемая для всех капель в каждой лунке для образца в капельном проточном цитометре. [100]
Цифровая ПЦР на основе чипа
[ редактировать ]Цифровая ПЦР на основе чипа (дПЦР) также представляет собой метод дПЦР, при котором реакционная смесь (также при использовании в кПЦР) делится на чипе на от ~10 000 до ~45 000 частей, затем амплифицируется с помощью термоциклической машины для конечной точки ПЦР и считывайте с помощью мощного устройства считывания с камеры с флуоресцентным фильтром (HEX, FAM, Cy5, Cy5.5 и Texas Red) для всех разделов каждого чипа. [101]
История
[ редактировать ]dPCR возникла из подхода, впервые опубликованного в 1988 году корпорацией Cetus, когда исследователи показали, что единственная копия гена β-глобина может быть обнаружена и амплифицирована с помощью ПЦР. [102] [103] Это было достигнуто путем разбавления образцов ДНК из нормальной клеточной линии человека ДНК из мутантной линии, имеющей гомозиготную делецию гена β-глобина, до тех пор, пока она не перестанет присутствовать в реакции. В 1989 году Питер Симмондс, Эй.Дж. Браун и др. впервые использовал эту концепцию для количественной оценки молекулы. [104] Алекс Морли и Памела Сайкс официально утвердили этот метод как количественный метод в 1992 году. [45]
В 1999 году Берт Фогельштейн и Кеннет Кинцлер ввели термин «цифровая ПЦР» и показали, что этот метод можно использовать для обнаружения редких раковых мутаций. [105] Однако dPCR было трудно выполнить; это было трудоемко, требовало много тренировок, чтобы сделать это правильно, и его было трудно выполнять в больших количествах. [105] В 2003 году Кинцлер и Фогельштейн продолжили совершенствовать dPCR и создали улучшенный метод, который они назвали технологией BEAMing , что означает «бусины, эмульсия, амплификация и магнетизм». В новом протоколе использовалась эмульсия для разделения реакций амплификации в одной пробирке. Это изменение позволило ученым масштабировать метод до тысяч реакций за один проход. [106] [107] [108]
Компании, разрабатывающие коммерческие системы дПЦР, интегрировали такие технологии, как автоматическое разделение образцов, цифровой подсчет мишеней нуклеиновых кислот и увеличение количества капель, которые могут помочь процессу стать более эффективным. [109] [110] [111] В последние годы ученые разработали и коммерциализировали диагностику на основе dPCR для нескольких заболеваний, включая немелкоклеточный рак легких и синдром Дауна . [112] [113] Первая система дПЦР для клинического использования получила маркировку CE в 2017 году и одобрена Управлением по контролю за продуктами и лекарствами США в 2019 году для диагностики хронического миелолейкоза . [114]
Ссылки
[ редактировать ]- ^ Перкель Дж. (май 2015 г.). «Руководство нашими экспериментами по ПЦР» . БиоТехники . 58 (5): 217–221. дои : 10.2144/000114283 . ПМИД 25967899 .
- ^ «Полимеразная цепная реакция (ПЦР)» . Национальный центр биотехнологической информации, Национальная медицинская библиотека США.
- ^ Хигучи, Рассел; Фоклер, Карита; Доллинджер, Гэвин; Уотсон, Роберт (сентябрь 1993 г.). «Кинетический ПЦР-анализ: мониторинг реакций амплификации ДНК в реальном времени» . Био/Технологии . 11 (9): 1026–1030. дои : 10.1038/nbt0993-1026 . ISSN 1546-1696 . ПМИД 7764001 . S2CID 5714001 .
- ^ Дьювер Д.Л., Клайн М.К., Ромсос Э.Л., Томан Б. (май 2018 г.). «Оценка капельной цифровой ПЦР для количественного определения геномной ДНК человека: преобразование копий на нанолитр в нанограммы ядерной ДНК на микролитр» . Аналитическая и биоаналитическая химия . 410 (12): 2879–2887. дои : 10.1007/s00216-018-0982-1 . ПМЦ 5996397 . ПМИД 29556737 .
- ^ Jump up to: а б с д Бейкер М. (2012). «Цифровая ПЦР набирает обороты» . Природные методы . 9 (6): 541–544. дои : 10.1038/nmeth.2027 . S2CID 46347563 .
- ^ Jump up to: а б с д Цюань П.Л., Саузаде М., Брузес Э. (апрель 2018 г.). «дПЦР: обзор технологий» . Датчики . 18 (4): 1271. Бибкод : 2018Senso..18.1271Q . дои : 10.3390/s18041271 . ПМЦ 5948698 . ПМИД 29677144 .
- ^ Предигер Э. «Цифровая ПЦР (дПЦР) — что это такое и зачем ее использовать?» . Интегрированные ДНК-технологии .
- ^ Батлер Д.М., Паколд М.Э., Джордан П.С., Ричман Д.Д., Смит Д.М. (декабрь 2009 г.). «Эффективность амплификации и секвенирования одного генома повышается за счет количественного анализа и использования инструментов биоинформатики» . Журнал вирусологических методов . 162 (1–2): 280–283. doi : 10.1016/j.jviromet.2009.08.002 . ПМК 2761514 . ПМИД 19698751 .
- ^ Jump up to: а б с Пекин Д., Схири Ю., Барет Дж.К., Ле Корр Д., Мазутис Л., Салем С.Б. и др. (июль 2011 г.). «Количественное и чувствительное обнаружение редких мутаций с использованием капельной микрофлюидики». Лаборатория на чипе . 11 (13): 2156–2166. дои : 10.1039/c1lc20128j . ПМИД 21594292 .
- ^ Швец Д., Тихопад А., Новосадова В., Пфаффл М.В., Кубиста М. (март 2015 г.). «Насколько хороша оценка эффективности ПЦР: рекомендации по точной и надежной оценке эффективности кПЦР» . Биомолекулярное обнаружение и количественный анализ . 3 :9–16. дои : 10.1016/j.bdq.2015.01.005 . ПМЦ 4822216 . ПМИД 27077029 .
- ^ Jump up to: а б Дингл Т.С., Седлак Р.Х., Кук Л., Джером К.Р. (ноябрь 2013 г.). «Толерантность капельно-цифровой ПЦР по сравнению с количественной ПЦР в реальном времени к ингибирующим веществам» . Клиническая химия . 59 (11): 1670–1672. дои : 10.1373/clinchem.2013.211045 . ПМК 4247175 . ПМИД 24003063 .
- ^ Павшич, Ерней; Жел, Яна; Милавец, Мойча (01 января 2016 г.). «Оценка ПЦР в реальном времени и различных платформ цифровой ПЦР для количественного определения ДНК» . Аналитическая и биоаналитическая химия . 408 (1): 107–121. дои : 10.1007/s00216-015-9107-2 . ISSN 1618-2650 . ПМЦ 4706846 . ПМИД 26521179 .
- ^ Сюй, Цзячэнь; Дуонг, Кира; Ян, Чжэньлинь; Каджи, Кавано; Оу, Цзяцзя; Руководитель Стивен Р.; Крайнен, Гогче; Ордуханян, Филипп; Ханна, Лорен; Ханна, Ава; Ван, Ян; Ван, Чжицзе; Ван, Цзе (декабрь 2021 г.). «Цифровая полимеразная цепная реакция (ПЦР) в реальном времени как новая технология повышает предел обнаружения для анализа редких аллелей» . Трансляционное исследование рака легких . 10 (12): 4336–4352. doi : 10.21037/tlcr-21-728 . ISSN 2226-4477 . ПМЦ 8743530 . ПМИД 35070745 .
- ^ Салипанте С.Дж., Джером К.Р. (январь 2020 г.). «Цифровая ПЦР — новая технология с широким применением в микробиологии» . Клиническая химия . 66 (1): 117–123. дои : 10.1373/clinchem.2019.304048 . ПМИД 31704712 .
- ^ Витте А.К., Фистер С., Местер П., Шодер Д., Россманит П. (ноябрь 2016 г.). «Оценка эффективности количественного обнаружения локуса Listeria monocytogenes prfA с помощью капельной цифровой ПЦР» . Аналитическая и биоаналитическая химия . 408 (27): 7583–7593. дои : 10.1007/s00216-016-9861-9 . ПМК 5061835 . ПМИД 27558101 .
- ^ Jump up to: а б Стаубер Дж. , Шейх Н., Ордиз М.И., Тарр П.И., Манари М.Дж. (май 2016 г.). «Капельная цифровая ПЦР надежно и воспроизводимо количественно определяет воспалительные транскрипты хозяина в фекалиях» . Клеточная иммунология . 303 : 43–49. дои : 10.1016/j.cellimm.2016.03.007 . ПМЦ 4863679 . ПМИД 27063479 .
- ^ Jump up to: а б с Скибо С (23 февраля 2018 г.). «Догнало ли профилирование опухоли рак?» . Проверено 23 июля 2019 г.
- ^ Хирш Ф (27 июля 2018 г.). «В рекомендациях освещаются «лучшие практики» жидкой биопсии при лечении немелкоклеточного рака легких» . Проверено 23 июля 2019 г.
- ^ Джонсон М. (12 января 2018 г.). «Bio-Rad продолжает продвигать технологию цифровой ПЦР и тесты для жидкой биопсии на коммерческий клинический рынок» . Проверено 23 июля 2019 г.
- ^ Окснард Г.Р., Павелец С.П., Куанг Й., Мах С.Л., О'Коннелл А., Мессинео М.М. и др. (март 2014 г.). «Неинвазивное обнаружение ответа и устойчивости при раке легких с мутацией EGFR с использованием количественного генотипирования нового поколения бесклеточной ДНК плазмы» . Клинические исследования рака . 20 (6): 1698–1705. дои : 10.1158/1078-0432.CCR-13-2482 . ПМЦ 3959249 . ПМИД 24429876 .
- ^ Шютц Э., Фишер А., Бек Дж., Харден М., Кох М., Вюнш Т. и др. (апрель 2017 г.). «Бесклеточная ДНК, полученная из трансплантата, неинвазивное раннее отторжение и маркер повреждения трансплантата при трансплантации печени: проспективное обсервационное многоцентровое когортное исследование» . ПЛОС Медицина . 14 (4): e1002286. дои : 10.1371/journal.pmed.1002286 . ПМК 5404754 . ПМИД 28441386 .
- ^ Ли С.Ю., Хван С.Ю. (2015). «Применение технологии цифровой полимеразной цепной реакции для неинвазивного пренатального тестирования» . Журнал генетической медицины . 12 (2): 72–78. дои : 10.5734/JGM.2015.12.2.72 . ISSN 2383-8442 .
- ^ Гу В., Ко В., Блюменфельд Ю.Дж., Эль-Сайед Ю.Ю., Хаджинс Л., Хинц С.Р., Quake SR (июль 2014 г.). «Неинвазивная пренатальная диагностика у плода с риском развития метилмалоновой ацидемии» . Генетика в медицине . 16 (7): 564–567. дои : 10.1038/gim.2013.194 . ПМК 4079742 . ПМИД 24406457 .
- ^ Jump up to: а б с д Штамм MC, Lada SM, Luong T, Rought SE, Gianella S, Terry VH и др. (2013). «Высокоточное измерение ДНК ВИЧ с помощью цифровой капельной ПЦР» . ПЛОС ОДИН . 8 (4): e55943. Бибкод : 2013PLoSO...855943S . дои : 10.1371/journal.pone.0055943 . ПМК 3616050 . ПМИД 23573183 .
- ^ Jump up to: а б Белл А.Д., Ашер К.Л., МакКэрролл С.А. (2018). «Анализ изменения количества копий с помощью капельной цифровой ПЦР». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. Клифтон, Нью-Джерси, стр. 143–160. дои : 10.1007/978-1-4939-7778-9_9 . ISBN 978-1-4939-7776-5 . ПМИД 29717442 .
{{cite book}}: CS1 maint: отсутствует местоположение издателя ( ссылка ) - ^ Шода К., Итикава Д., Фудзита Ю., Масуда К., Хирамото Х., Хамада Дж. и др. (январь 2017 г.). «Мониторинг статуса количества копий HER2 в циркулирующей опухолевой ДНК с помощью капельной цифровой ПЦР у пациентов с раком желудка» . Рак желудка . 20 (1): 126–135. дои : 10.1007/s10120-016-0599-z . ПМИД 26874951 .
- ^ Jump up to: а б Гевенслебен Х., Гарсиа-Мурильяс И., Гразер М.К., Скьявон Г., Осин П., Партон М. и др. (июнь 2013 г.). «Неинвазивное обнаружение амплификации HER2 с помощью цифровой ПЦР ДНК плазмы» . Клинические исследования рака . 19 (12): 3276–3284. дои : 10.1158/1078-0432.CCR-12-3768 . ПМК 6485473 . ПМИД 23637122 .
- ^ Маццони Э., Фронтини Ф., Ротондо Х.К., Занотта Н., Фиораванти А., Минелли Ф. и др. (апрель 2019 г.). «Антитела, реагирующие на мимотопы большого Т-антигена обезьяньего вируса 40, вирусного онкопротеина, в сыворотке детей». Журнал клеточной физиологии . 234 (4): 3170–3179. дои : 10.1002/jcp.27490 . HDL : 11392/2397717 . ПМИД 30362540 . S2CID 53106591 .
- ^ Утияма И., Накашима М., Ватанабэ С., Миядзима М., Тагури М., Миятаке С. и др. (март 2016 г.). «Сверхчувствительная капельная цифровая ПЦР для обнаружения малораспространенной соматической мутации GNAQ при синдроме Стерджа-Вебера» . Научные отчеты . 6 (1): 22985. Бибкод : 2016NatSR...622985U . дои : 10.1038/srep22985 . ПМЦ 4783707 . ПМИД 26957145 .
- ^ Jump up to: а б с Марусина К. (1 октября 2017 г.). «Позиционирование цифровой ПЦР для более четкого представления генома» . Проверено 23 июля 2019 г.
- ^ Jump up to: а б Камитаки Н., Ашер К.Л., МакКэрролл С.А. (2018). «Использование капельной цифровой ПЦР для анализа экспрессии аллель-специфической РНК». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 401–422. дои : 10.1007/978-1-4939-7778-9_23 . ISBN 978-1-4939-7776-5 . ПМИД 29717456 .
- ^ Миллиер М.Дж., Штамп Л.К., Гессен, Пенсильвания (декабрь 2017 г.). «Цифровая ПЦР для экспрессии генов: влияние деградации тканевой РНК» . Научные отчеты . 7 (1): 17235. Бибкод : 2017НатСР...717235М . дои : 10.1038/s41598-017-17619-0 . ПМЦ 5722939 . ПМИД 29222437 .
- ^ «Высокочувствительное выявление гепатита В с помощью ddPCR» . 12 апреля 2018 г. Проверено 23 июля 2019 г.
- ^ Чан М, Чон С.В., Бэ Н.Х., Сон Ю, Ли ТиДжей, Ли МК, Ли С.Дж., Ли К.Г. (2017). «Система цифровой ПЦР на основе капель для определения одноклеточного уровня патогенов пищевого происхождения». Журнал БиоЧип . 11 (4): 329–337. дои : 10.1007/s13206-017-1410-x . ISSN 2092-7843 . S2CID 89829687 .
- ^ Игараси Ю., Утияма Т., Минэгиши Т., Такахаши С., Ватанабэ Н., Каваи Т. и др. (сентябрь 2017 г.). «Отслеживание векторов на основе отдельных клеток у пациентов с ADA-SCID, получающих генную терапию стволовыми клетками» . Молекулярная терапия. Методы и клинические разработки . 6 :8–16. дои : 10.1016/j.omtm.2017.05.005 . ПМЦ 5466583 . ПМИД 28626778 .
- ^ Албайрак С., Хорди К.А., Зехнер С., Лин Дж., Бичсел К.А., Хаммаш М., Тай С. (март 2016 г.). «Цифровая количественная оценка белков и мРНК в одиночных клетках млекопитающих» . Молекулярная клетка . 61 (6): 914–924. doi : 10.1016/j.molcel.2016.02.030 . ПМИД 26990994 .
- ^ Хуссейн М., Фантуццо Р., Меркорелли С., Каллен С. (май 2016 г.). «Метод прямой капельной цифровой ПЦР для количественного определения остаточной ДНК в белковых препаратах, вырабатываемых в дрожжевых клетках». Журнал фармацевтического и биомедицинского анализа . 123 : 128–131. дои : 10.1016/j.jpba.2016.01.050 . ПМИД 26896631 .
- ^ Мияока И., Чан А.Х., судья Л.М., Ю Дж., Хуан М., Нгуен Т.Д. и др. (март 2014 г.). «Выделение IPS-клеток человека с одноосновным отредактированным геномом без отбора антибиотиков» . Природные методы . 11 (3): 291–293. дои : 10.1038/nmeth.2840 . ПМК 4063274 . ПМИД 24509632 .
- ^ Нельсон С.Э., Хаким С.Х., Оустераут Д.Г., Такоре П.И., Мореб Э.А., Кастельянос Ривера Р.М. и др. (январь 2016 г.). «Редактирование генома in vivo улучшает мышечную функцию на мышиной модели мышечной дистрофии Дюшенна» . Наука . 351 (6271): 403–407. Бибкод : 2016Sci...351..403N . дои : 10.1126/science.aad5143 . ПМЦ 4883596 . ПМИД 26721684 .
- ^ Мияока Ю., Берман Дж.Р., Купер С.Б., Майерл С.Дж., Чан А.Х., Чжан Б. и др. (март 2016 г.). «Систематическая количественная оценка HDR и NHEJ выявляет влияние локуса, нуклеазы и типа клеток на редактирование генома» . Научные отчеты . 6 : 23549. Бибкод : 2016NatSR...623549M . дои : 10.1038/srep23549 . ПМЦ 4814844 . ПМИД 27030102 .
- ^ Guttery DS, Page K, Hills A, Woodley L, Marchese SD, Rghebi B и др. (июль 2015 г.). «Неинвазивное обнаружение активирующих мутаций рецептора эстрогена 1 (ESR1) при метастатическом раке молочной железы, положительном по рецептору эстрогена» . Клиническая химия . 61 (7): 974–982. дои : 10.1373/clinchem.2015.238717 . ПМИД 25979954 .
- ^ Робин Дж.Д., Ладлоу А.Т., ЛаРейнджер Р., Райт В.Е., Шей Дж.В. (апрель 2016 г.). «Сравнение методов количественного определения ДНК для секвенирования следующего поколения» . Научные отчеты . 6 (1): 24067. Бибкод : 2016NatSR...624067R . дои : 10.1038/srep24067 . ПМЦ 4822169 . ПМИД 27048884 .
- ^ Эгрейн Л., Гу Ю., Квейл М.А. (июнь 2016 г.). «Количественная оценка эффективности протокола подготовки библиотеки секвенирования следующего поколения с использованием капельной цифровой ПЦР - систематическое сравнение наборов для подготовки библиотеки ДНК для секвенирования Illumina» . БМК Геномика . 17 (1): 458. doi : 10.1186/s12864-016-2757-4 . ПМЦ 4906846 . ПМИД 27297323 .
- ^ Jump up to: а б Брунетто Г.С., Массуд Р., Лейбович Э.К., Карузо Б., Джонсон К., Охайон Дж. и др. (август 2014 г.). «Цифровая капельная ПЦР (ddPCR) для точного количественного определения провирусной нагрузки Т-лимфотропного вируса 1 человека в периферической крови и спинномозговой жидкости пациентов с HAM/TSP и идентификации вирусных мутаций» . Журнал нейровирусологии . 20 (4): 341–351. дои : 10.1007/s13365-014-0249-3 . ПМК 4085507 . ПМИД 24781526 .
- ^ Jump up to: а б Сайкс П.Дж., Нео С.Х., Бриско М.Дж., Хьюз Э., Кондон Дж., Морли А.А. (сентябрь 1992 г.). «Количественное определение мишеней для ПЦР с использованием предельного разведения». БиоТехники . 13 (3): 444–449. ПМИД 1389177 .
- ^ Фогельштейн Б., Кинцлер К.В. (август 1999 г.). «Цифровая ПЦР» . Труды Национальной академии наук Соединенных Штатов Америки . 96 (16): 9236–9241. Бибкод : 1999PNAS...96.9236V . дои : 10.1073/pnas.96.16.9236 . ЧВК 17763 . ПМИД 10430926 .
- ^ Jump up to: а б Рачки Н., Дрео Т., Гутьеррес-Агирре И., Блеец А., Равникар М. (2014). «Цифровая капельная ПЦР с обратной транскриптазой демонстрирует высокую устойчивость к ингибиторам ПЦР из образцов растений, почвы и воды» . Растительные методы . 10 (1): 42. дои : 10.1186/s13007-014-0042-6 . ПМК 4307183 . ПМИД 25628753 .
- ^ Добник Д., Спилсберг Б., Богожалец Кошир А., Штебих Д., Мориссет Д., Хольст-Йенсен А., Жел Дж. (2018). «Протоколы мультиплексной капельной цифровой ПЦР для количественной оценки событий ГМ-кукурузы». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 69–98. дои : 10.1007/978-1-4939-7778-9_5 . ISBN 978-1-4939-7776-5 . ПМИД 29717438 .
- ^ Веллуччи А., Лейбович ЕС, Якобсон С. (2018). «Использование капельной цифровой ПЦР для обнаружения коинфекции вирусами герпеса человека 6A и 6B (HHV-6A и HHV-6B) в клинических образцах». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 99–109. дои : 10.1007/978-1-4939-7778-9_6 . ISBN 978-1-4939-7776-5 . ПМИД 29717439 .
- ^ Jump up to: а б Тальяпьетра А., Ротондо Х.К., Бонони И., Маццони Е., Маганьоли Ф., Гонсалес Л.О. и др. (март 2020 г.). «Капельно-цифровая ПЦР для обнаружения последовательностей полиомавируса клеток Меркеля в ворсинках хориона у женщин, пострадавших от самопроизвольного аборта» . Журнал клеточной физиологии . 235 (3): 1888–1894. дои : 10.1002/jcp.29213 . hdl : 11392/2409453 . ПМИД 31549405 .
- ^ Jump up to: а б Тальяпьетра А., Ротондо Х.К., Бонони И., Маццони Э., Маганьоли Ф., Маритати М. и др. (март 2019 г.). «Следы полиомавирусов BK и JC в образцах женщин, перенесших самопроизвольный аборт». Репродукция человека . 34 (3): 433–440. дои : 10.1002/jcp.27490 . HDL : 11392/2397717 . ПМИД 30590693 . S2CID 53106591 .
- ^ Маццони Э., Ротондо Х.К., Маррачино Л., Селватичи Р., Бонони И., Торреджиани Э. и др. (2017). «Обнаружение ДНК полиомавируса клеток Меркеля в образцах сыворотки здоровых доноров крови» . Границы онкологии . 7 : 294. doi : 10.3389/fonc.2017.00294 . ПМЦ 5712532 . ПМИД 29238698 .
- ^ Подлесный П., Труллас Р. (2018). «Биомаркеры в спинномозговой жидкости: анализ бесклеточной циркулирующей митохондриальной ДНК с помощью цифровой ПЦР». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 111–126. дои : 10.1007/978-1-4939-7778-9_7 . ISBN 978-1-4939-7776-5 . ПМИД 29717440 .
- ^ Цао Ю, Райт М.Р., Гриффит Дж.Ф. (2018). «Исследование общего и антропогенного фекального загрязнения воды». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 127–140. дои : 10.1007/978-1-4939-7778-9_8 . ISBN 978-1-4939-7776-5 . ПМИД 29717441 .
- ^ Ли В., Ли А., Грегерсен П.К. (январь 2009 г.). «Обнаружение области изменения количества копий и изменения количества копий с помощью кумулятивных графиков» . БМК Биоинформатика . 10 (С1): С67. arXiv : 0909.3129 . Бибкод : 2009arXiv0909.3129L . дои : 10.1186/1471-2105-10-S1-S67 . ПМЦ 2648736 . ПМИД 19208171 .
- ^ Корен А., Рукосакер Р.Э., Камитаки Н., Карлич Р., Гош С., Полак П. и др. (ноябрь 2014 г.). «Генетические вариации времени репликации ДНК человека» . Клетка . 159 (5): 1015–1026. дои : 10.1016/j.cell.2014.10.025 . ПМЦ 4359889 . ПМИД 25416942 .
- ^ Сандерс С. (16 июля 2008 г.). «CNV против SNP: понимание структурных изменений при заболеваниях человека» . Проверено 24 июля 2019 г.
- ^ Маршалл С.Р., Хориган Д.П., Мерико Д., Тирувахиндрапурам Б., Ву В., Грир Д.С. и др. (январь 2017 г.). «Вклад вариантов числа копий в шизофрению по результатам полногеномного исследования 41 321 субъекта» . Природная генетика . 49 (1): 27–35. дои : 10.1038/ng.3725 . ПМЦ 5737772 . ПМИД 27869829 .
- ^ Шлиен А., Малкин Д. (июнь 2009 г.). «Вариации числа копий и рак» . Геномная медицина . 1 (6): 62. дои : 10,1186/гм62 . ПМК 2703871 . ПМИД 19566914 .
- ^ Лауэр С., Грешам Д. (декабрь 2019 г.). «Развивающийся взгляд на варианты количества копий». Современная генетика . 65 (6): 1287–1295. дои : 10.1007/s00294-019-00980-0 . ПМИД 31076843 . S2CID 149444714 .
- ^ «Обнаружено, что изменение номера копии связано со смертностью от рака» . 5 сентября 2018 г. Проверено 24 июля 2019 г.
- ^ Гу В, Лупски-младший (2008). «ХНВ и заболевания нервной системы – что нового?» . Цитогенетические и геномные исследования . 123 (1–4): 54–64. дои : 10.1159/000184692 . ПМК 2920183 . ПМИД 19287139 .
- ^ Тапар А., Купер М. (август 2013 г.). «Вариация номера экземпляра: что это такое и что это говорит нам о детских психических расстройствах?» . Журнал Американской академии детской и подростковой психиатрии . 52 (8): 772–774. дои : 10.1016/j.jaac.2013.05.013 . ПМК 3919207 . ПМИД 23880486 .
- ^ Секар А., Биалас А.Р., де Ривера Х., Дэвис А., Хаммонд Т.Р., Камитаки Н. и др. (февраль 2016 г.). «Риск шизофрении из-за сложных вариаций компонента 4 комплемента» . Природа . 530 (7589): 177–183. Бибкод : 2016Natur.530..177. . дои : 10.1038/nature16549 . ПМЦ 4752392 . ПМИД 26814963 .
- ^ Йим Ш., Юнг Ш., Чунг Б., Чунг Ю.Дж. (май 2015 г.). «Клинические последствия изменений числа копий при аутоиммунных заболеваниях» . Корейский журнал внутренней медицины . 30 (3): 294–304. дои : 10.3904/kjim.2015.30.3.294 . ПМЦ 4438283 . ПМИД 25995659 .
- ^ Хе Ю, Хоскинс Дж. М., Маклеод Х. Л. (май 2011 г.). «Варианты числа копий в фармакогенетических генах» . Тенденции молекулярной медицины . 17 (5): 244–251. doi : 10.1016/j.molmed.2011.01.007 . ПМК 3092840 . ПМИД 21388883 .
- ^ Гонсалес Э., Кулкарни Х., Боливар Х., Мангано А., Санчес Р., Катано Г. и др. (март 2005 г.). «Влияние сегментных дупликаций, содержащих ген CCL3L1, на восприимчивость к ВИЧ-1/СПИДу». Наука . 307 (5714): 1434–1440. Бибкод : 2005Sci...307.1434G . дои : 10.1126/science.1101160 . ПМИД 15637236 . S2CID 8815153 .
- ^ Jump up to: а б Лю С., Яо Л., Дин Д., Чжу Х. (декабрь 2010 г.). «Изменение числа копий CCL3L1 и восприимчивость к инфекции ВИЧ-1: метаанализ» . ПЛОС ОДИН . 5 (12): e15778. Бибкод : 2010PLoSO...515778L . дои : 10.1371/journal.pone.0015778 . ПМК 3012711 . ПМИД 21209899 .
- ^ Дубе С., Цинь Дж., Рамакришнан Р. (август 2008 г.). «Математический анализ изменения числа копий в образце ДНК с использованием цифровой ПЦР на нанофлюидном устройстве» . ПЛОС ОДИН . 3 (8): e2876. Бибкод : 2008PLoSO...3.2876D . дои : 10.1371/journal.pone.0002876 . ПМК 2483940 . ПМИД 18682853 .
- ^ Хьюзман CB, Лу XJ, Лю К.Ю., Чжу Ю., Таул Р.М., Хейнс С., Пох К.Ф. (сентябрь 2017 г.). «Обнаружение клинически значимых изменений числа копий при прогрессировании рака полости рта с использованием мультиплексной цифровой капельной ПЦР» . Научные отчеты . 7 (1): 11855. Бибкод : 2017NatSR...711855H . дои : 10.1038/s41598-017-11201-4 . ПМК 5605662 . ПМИД 28928368 .
- ^ Jump up to: а б Usher CL, Handsaker RE, Esko T, Tuke MA, Weedon MN, Hastie AR и др. (август 2015 г.). «Структурные формы локуса амилазы человека и их связь с SNP, гаплотипами и ожирением» . Природная генетика . 47 (8): 921–925. дои : 10.1038/ng.3340 . ПМЦ 4712930 . ПМИД 26098870 .
- ^ Олдхаус М.К., Абу Бакар С., Прескотт Н.Дж., Палла Р., Су К., Мэнсфилд Дж.К. и др. (декабрь 2010 г.). «Методы измерения и точность изменения количества копий: неспособность воспроизвести связь количества копий бета-дефенсина с болезнью Крона» . Молекулярная генетика человека . 19 (24): 4930–4938. дои : 10.1093/hmg/ddq411 . ПМЦ 2989891 . ПМИД 20858604 .
- ^ Пиньейру Л., Эмсли КР (2018). «Основные концепции и проверка измерений цифровой ПЦР». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 11–24. дои : 10.1007/978-1-4939-7778-9_2 . ISBN 978-1-4939-7776-5 . ПМИД 29717435 .
- ^ Handsaker RE, Ван Дорен В., Берман Дж.Р., Дженовезе Дж., Кашин С., Беттгер Л.М., МакКэрролл С.А. (март 2015 г.). «Большие многоаллельные вариации числа копий у людей» . Природная генетика . 47 (3): 296–303. дои : 10.1038/ng.3200 . ПМК 4405206 . ПМИД 25621458 .
- ^ Гарсия-Мурильяс I, Тернер, Северная Каролина (2018). «Оценка амплификации HER2 в плазме вкДНК». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 161–172. дои : 10.1007/978-1-4939-7778-9_10 . ISBN 978-1-4939-7776-5 . ПМИД 29717443 .
- ^ Кристген М., ван Луттихуизен Дж.Л., Раап М., Браубах П., Шмидт Л., Джонигк Д. и др. (декабрь 2016 г.). «Точная оценка количества копий ERBB2 при раке молочной железы с помощью анализа матрицы молекулярных инверсионных зондов» . Онкотаргет . 7 (50): 82733–82740. дои : 10.18632/oncotarget.12421 . ПМК 5347728 . ПМИД 27716627 .
- ^ Борли А., Мерсер Т., Морган М., Даттон П., Барретт-Ли П., Брунелли М., Джасани Б. (апрель 2014 г.). «Влияние количества копий HER2 при IHC2+/FISH-амплифицированном раке молочной железы на результат адъювантного лечения трастузумабом в крупной онкологической сети Великобритании» . Британский журнал рака . 110 (8): 2139–2143. дои : 10.1038/bjc.2014.147 . ПМЦ 3992505 . ПМИД 24691421 .
- ^ Цао Ю, Райт М.Р., Гриффит Дж.Ф. (март 2015 г.). «Капельная цифровая ПЦР для одновременной количественной оценки общих и антропогенных показателей кала для оценки качества воды». Исследования воды . 70 : 337–349. Бибкод : 2015WatRe..70..337C . дои : 10.1016/j.watres.2014.12.008 . ПМИД 25543243 .
- ^ Европейское общество медицинской онкологии (17 ноября 2017 г.). «Исследование анализирует мутации в спинномозговой жидкости при раке легких с метастазами в головной мозг» . Проверено 24 июля 2019 г.
- ^ Jump up to: а б Петроне Дж (8 июня 2017 г.). «Норвежская команда планирует к концу года дебютировать с цифровым ПЦР-тестом на рак мочевого пузыря» . Проверено 24 июля 2019 г.
- ^ Хиемке-Джива Л.С., Миннема М.К., Радерсма-ван Лун Дж.Х., Джива Н.М., де Бур М., Легит Р.Дж. и др. (апрель 2018 г.). «Использование капельной цифровой ПЦР в жидкостных биопсиях: высокочувствительный метод обнаружения MYD88 p.(L265P) в спинномозговой жидкости» . Гематологическая онкология . 36 (2): 429–435. дои : 10.1002/hon.2489 . ПМИД 29210102 . S2CID 4968214 .
- ^ Jump up to: а б Пакстон А. (октябрь 2017 г.). «Возрожденные надежды, новые проблемы с жидкой биопсией» . Проверено 24 июля 2019 г.
- ^ Бхадра К., Меллерт Х., Пестано Дж. (5 июня 2017 г.). «Внедрение тестов жидкой биопсии при НМРЛ» . Проверено 24 июля 2019 г.
- ^ Захер А.Г., Павелец С., Дальберг С.Е., Олден Р.С., О'Коннелл А., Фини Н. и др. (август 2016 г.). «Проспективная валидация быстрого генотипирования плазмы для обнаружения мутаций EGFR и KRAS при распространенном раке легких» . JAMA Онкология . 2 (8): 1014–1022. дои : 10.1001/jamaoncol.2016.0173 . ПМЦ 4982795 . ПМИД 27055085 .
- ^ Олссон Э., Винтер С., Джордж А., Чен Ю., Хоулин Дж., Тан М.Х. и др. (август 2015 г.). «Серийный мониторинг циркулирующей опухолевой ДНК у больных первичным раком молочной железы для выявления скрытого метастатического заболевания» . ЭМБО Молекулярная медицина . 7 (8): 1034–1047. дои : 10.15252/emmm.201404913 . ПМЦ 4551342 . ПМИД 25987569 .
- ^ Карпинетти П., Доннард Э., Беттони Ф., Асприно П., Кояма Ф., Розански А. и др. (ноябрь 2015 г.). «Использование персонализированных биомаркеров и жидкой биопсии для мониторинга ответа на лечение и рецидива заболевания при местно-распространенном раке прямой кишки после неоадъювантной химиолучевой терапии» . Онкотаргет . 6 (35): 38360–38371. дои : 10.18632/oncotarget.5256 . ПМК 4742005 . ПМИД 26451609 .
- ^ Рейнерт Т., Шолер Л.В., Томсен Р., Тобиасен Х., Ванг С., Нордентофт И. и др. (апрель 2016 г.). «Анализ циркулирующей опухолевой ДНК для мониторинга тяжести заболевания после операции по поводу колоректального рака» . Гут . 65 (4): 625–634. дои : 10.1136/gutjnl-2014-308859 . ПМИД 25654990 .
- ^ Перейра Э., Камачо-Ванегас О., Ананд С., Себра Р., Каталина Камачо С., Гарнар-Ворцель Л. и др. (2015). «Персонализированные биомаркеры циркулирующей опухолевой ДНК динамически прогнозируют реакцию на лечение и выживаемость при гинекологическом раке» . ПЛОС ОДИН . 10 (12): e0145754. Бибкод : 2015PLoSO..1045754P . дои : 10.1371/journal.pone.0145754 . ПМК 4696808 . ПМИД 26717006 .
- ^ Дамке К.М., Стивен К.Е., Ларсен Л.К., Поулсен А.Л., Абдул-Ал А., Даль С., Гульдберг П. (декабрь 2016 г.). «Проспективная слепая оценка анализа ДНК мочи для выявления уротелиальной карциномы мочевого пузыря у пациентов с макрогематурией». Европейская урология . 70 (6): 916–919. дои : 10.1016/j.eururo.2016.06.035 . ПМИД 27417036 .
- ^ Jump up to: а б с Линднер Л., Кайру П., Жако С., Бирлинг М.К., Эро Ю., Павлович Дж. (июль 2021 г.). «Надежные и надежные протоколы капельной цифровой ПЦР (ddPCR) и RT-ddPCR для исследований на мышах». Методы . 191 : 95–106. дои : 10.1016/j.ymeth.2020.07.004 . ПМИД 32721466 . S2CID 220851187 .
- ^ Тейлор С.С., Карбонно Дж., Шелтон Д.Н., Бойвен Дж. (ноябрь 2015 г.). «Оптимизация капельной цифровой ПЦР из экстрактов РНК и ДНК с прямым сравнением с RT-qPCR: клинические последствия для количественной оценки субпопуляций, устойчивых к озельтамивиру» . Журнал вирусологических методов . 224 : 58–66. дои : 10.1016/j.jviromet.2015.08.014 . ПМИД 26315318 .
- ^ Кит А.С., Хаггетт Дж.Ф., Цонев С. (декабрь 2016 г.). «Основы мультиплексирования при цифровой ПЦР» . Биомолекулярное обнаружение и количественный анализ . 10 :15–23. дои : 10.1016/j.bdq.2016.05.002 . ПМК 5154634 . ПМИД 27990345 .
- ^ Сунь Б, Тао Л, Чжэн ЮЛ (июнь 2014 г.). «Одновременная количественная оценка альтернативно сплайсированных транскриптов в реакции цифровой ПЦР с одной каплей» . БиоТехники . 56 (6): 319–325. дои : 10.2144/000114179 . ПМИД 24924392 .
- ^ Валенсия Калифорния, Роденайзер Д., Бхайд С., Чин Э., Литтлджон М.Р., Кеонг Л.М. и др. (2012). «Оценка платформ целевого обогащения с использованием массового параллельного секвенирования для обнаружения мутаций при врожденной мышечной дистрофии» . Журнал молекулярной диагностики . 14 (3): 233–246. дои : 10.1016/j.jmoldx.2012.01.009 . ПМЦ 3349841 . ПМИД 22426012 .
- ^ Филипп Дж., Дерурхи М., Дюран Э., Вайян Э., Дешаум А., Рабиривело И. и др. (2015). «Какой метод обогащения NGS лучше всего подходит для молекулярной диагностики моногенного диабета и ожирения?» . ПЛОС ОДИН . 10 (11): e0143373. Бибкод : 2015PLoSO..1043373P . дои : 10.1371/journal.pone.0143373 . ПМЦ 4657897 . ПМИД 26599467 .
- ^ Уэлле Э., Фоли Дж. Х., Конвей Э. М., Хейнс К. (август 2015 г.). «Hi-Fi SELEX: высококачественная платформа для обнаружения терапевтических аптамеров на основе цифровой ПЦР». Биотехнология и биоинженерия . 112 (8): 1506–1522. дои : 10.1002/бит.25581 . ПМИД 25727321 . S2CID 39450798 .
- ^ Ладлоу А.Т., Шелтон Д., Райт М.Е., Шей Дж.В. (2018). «DdTRAP: метод чувствительного и точного количественного определения теломеразной активности». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 513–529. дои : 10.1007/978-1-4939-7778-9_29 . ISBN 978-1-4939-7776-5 . ПМК 6046637 . ПМИД 29717462 .
- ^ Сайед М.Е., Слашер А.Л., Ладлоу А.Т. (май 2019 г.). «Droplet Digital TRAP (ddTRAP): адаптация протокола амплификации повторов теломер к капельной цифровой полимеразной цепной реакции». Журнал визуализированных экспериментов (147). дои : 10.3791/59550 . ПМИД 31107456 . S2CID 155519448 .
- ^ Штейн Р.А. (1 июля 2019 г.). «Секвенирование одной клетки через несколько омиков» . Проверено 1 августа 2019 г.
- ^ Вуд-Бауэнс СМ, Джи Х.П. (2018). «Измерение количества копий DDPCR с использованием одноцветного мультиплексирования и генотипирование вариантов одного нуклеотида». Цифровая ПЦР . Методы молекулярной биологии. Том. 1768. стр. 323–333. дои : 10.1007/978-1-4939-7778-9_18 . ISBN 978-1-4939-7776-5 . ПМИД 29717451 .
- ^ Лоу, Х., Чан, С.Дж., Су, GH. и др. Цифровая система ПЦР Clarity™: новая платформа для абсолютного количественного определения нуклеиновых кислот. Анальная биоанальная химия 409, 1869–1875 (2017). https://doi.org/10.1007/s00216-016-0131-7
- ^ Сайки Р.К., Гельфанд Д.Х., Стоффель С., Шарф С.Дж., Хигучи Р., Хорн Г.Т. и др. (январь 1988 г.). «Праймер-направленная ферментативная амплификация ДНК с помощью термостабильной ДНК-полимеразы». Наука . 239 (4839): 487–491. Бибкод : 1988Sci...239..487S . дои : 10.1126/science.239.4839.487 . ПМИД 2448875 .
- ^ Морли А.А. (сентябрь 2014 г.). «Цифровая ПЦР: Краткая история» . Биомолекулярное обнаружение и количественный анализ . 1 (1): 1–2. дои : 10.1016/j.bdq.2014.06.001 . ПМК 5129430 . ПМИД 27920991 .
- ^ Рутсарт С., Босман К., Трипстин В., Нейхейс М., Вандекеркхове Л. (январь 2018 г.). «Цифровая ПЦР как инструмент измерения персистенции ВИЧ» . Ретровирусология . 15 (1): 16. дои : 10.1186/s12977-018-0399-0 . ПМЦ 5789538 . ПМИД 29378600 .
- ^ Jump up to: а б Перкель Дж. (11 апреля 2014 г.). «Цифровая ПЦР-революция» . Проверено 22 июля 2019 г.
- ^ Пол Г., Ши I (январь 2004 г.). «Принцип и применение цифровой ПЦР». Экспертный обзор молекулярной диагностики . 4 (1): 41–47. дои : 10.1586/14737159.4.1.41 . ПМИД 14711348 . S2CID 28271641 .
- ^ Дрессман Д., Ян Х., Траверсо Г., Кинцлер К.В., Фогельштейн Б. (июль 2003 г.). «Преобразование отдельных молекул ДНК во флуоресцентные магнитные частицы для обнаружения и подсчета генетических вариаций» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (15): 8817–8822. Бибкод : 2003PNAS..100.8817D . дои : 10.1073/pnas.1133470100 . ПМК 166396 . ПМИД 12857956 .
- ^ Диль Ф., Ли М., Хе Ю., Кинцлер К.В., Фогельштейн Б., Дрессман Д. (июль 2006 г.). «BEAMing: ПЦР одиночных молекул на микрочастицах в эмульсиях вода в масле». Природные методы . 3 (7): 551–559. дои : 10.1038/nmeth898 . ПМИД 16791214 . S2CID 7059151 .
- ^ Буткус Б (8 июля 2010 г.). «Пространство цифровой ПЦР накаляется, поскольку поставщики инструментов для медико-биологических наук начинают подавать заявки» . Проверено 22 июля 2019 г.
- ^ Рамакришнан Р., Цинь Дж., Джонс Р.К., Уивер Л.С. (2013). «Интегрированные жидкостные схемы (IFC) для цифровой ПЦР». Микрофлюидная диагностика . Методы молекулярной биологии. Том. 949. стр. 423–431. дои : 10.1007/978-1-62703-134-9_27 . ISBN 978-1-62703-133-2 . ПМИД 23329458 .
- ^ Буткус Б (29 марта 2012 г.). «RainDance запускает цифровую платформу ПЦР; заявляет о чувствительности и превосходстве эксплуатационных затрат» . Проверено 22 июля 2019 г.
- ^ « Анализ крови «жидкой биопсии» обнаруживает генетические мутации при распространенной форме рака легких» . 7 апреля 2016 г. Проверено 22 июля 2019 г.
- ^ «Корейская компания BioCore первой начала коммерциализировать НИПТ на основе цифровой ПЦР» . 2 марта 2018 г. Проверено 22 июля 2019 г.
- ^ «Bio-Rad получает первую маркировку CE по результатам клинического теста ddPCR» . 5 декабря 2017 г. Проверено 22 июля 2019 г.