Циркулирующая опухолевая ДНК
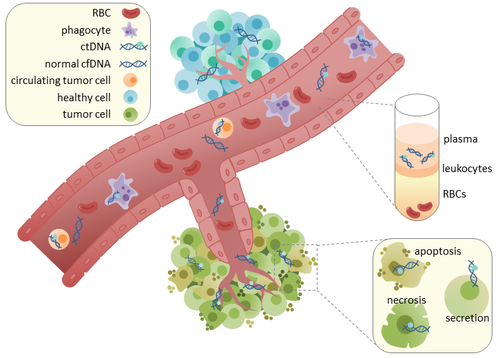
Циркулирующая опухолевая ДНК (цДНК) представляет собой опухолевого фрагментированную ДНК происхождения, находящуюся в кровотоке и не связанную с клетками. ctDNA не следует путать с внеклеточной ДНК ( cfDNA ), более широким термином, который описывает ДНК, которая свободно циркулирует в кровотоке, но не обязательно имеет опухолевое происхождение. Поскольку ктДНК может отражать весь геном опухоли , она приобрела популярность благодаря своей потенциальной клинической полезности; « Жидкостная биопсия » в виде забора крови может проводиться в различные моменты времени для мониторинга прогрессирования опухоли на протяжении всего курса лечения. [1] [2]
Недавние исследования заложили основу для определения экспрессии генов на основе вкДНК (и ктДНК), при этом EPIC-seq стал заметным достижением. [3] Этот метод существенно поднял планку неинвазивного определения уровней экспрессии отдельных генов, тем самым увеличив применимость анализа для характеристики заболеваний, гистологической классификации и мониторинга эффективности лечения. [3] [4] [5]
ктДНК происходит непосредственно из опухоли или из циркулирующих опухолевых клеток (ЦОК), [6] который описывает жизнеспособные, неповрежденные опухолевые клетки, которые выделяются из первичных опухолей и попадают в кровоток или лимфатическую систему. Точный механизм высвобождения цДНК неясен. Биологические процессы, которые, как предполагается, участвуют в высвобождении ктДНК, включают апоптоз и некроз умирающих клеток или активное высвобождение из жизнеспособных опухолевых клеток. [7] [8] [9] [10] [11] Исследования на людях (здоровых и онкологических больных) [12] и ксенотрансплантированные мыши [13] показывают, что размер фрагментированной вкДНК преимущественно составляет 166 п.о., что соответствует длине ДНК, обернутой вокруг нуклеосомы плюс линкер. Фрагментация такой длины может указывать на апоптотическую фрагментацию ДНК , что позволяет предположить, что апоптоз может быть основным методом высвобождения цДНК. Фрагментация вкДНК изменяется в плазме онкологических больных. [14] [15] В здоровых тканях инфильтрирующие фагоциты ответственны за очистку апоптотического или некротического клеточного мусора, включая вкДНК. [16] ctDNA у здоровых пациентов присутствует только на низких уровнях, но более высокие уровни ctDNA у онкологических больных могут быть обнаружены с увеличением размера опухоли. [17] Возможно, это происходит из-за неэффективной инфильтрации иммунных клеток в участки опухоли, что снижает эффективный клиренс цДНК из кровотока. [16] Сравнение мутаций в цДНК и ДНК, выделенных из первичных опухолей одних и тех же пациентов, выявило наличие идентичных генетических изменений, связанных с раком. [18] [19] Это привело к возможности использования ктДНК для более раннего выявления рака и последующего лечения. [20]
Методы
[ редактировать ]Преаналитические соображения
[ редактировать ]Когда кровь собирается в пробирки с ЭДТА и хранится, лейкоциты начинают лизировать и высвобождать геномную ДНК дикого типа в образец в количествах, обычно во много раз превышающих количество цДНК, в котором присутствует цДНК. [21] Это затрудняет обнаружение мутаций или других биомаркеров цтДНК. [22] Использование коммерчески доступных пробирок для стабилизации клеток может предотвратить или задержать лизис лейкоцитов, тем самым уменьшая эффект разбавления цДНК. [23] Шервуд и др. продемонстрировали превосходное обнаружение мутаций KRAS в соответствующих образцах, собранных как в пробирки EDTA K3, так и в пробирки Streck BCT. [23] Преимущества пробирок для стабилизации клеток могут быть реализованы в ситуации, когда кровь не может быть немедленно переработана в плазму.
Другие процедуры также могут уменьшить количество «загрязняющей» ДНК дикого типа и сделать обнаружение цДНК более осуществимым: [23]
- Никогда не замораживайте образец крови перед извлечением плазмы для анализа цтДНК.
- Обработайте образец до плазмы в течение 2–4 часов (если он собран в пробирку с ЭДТА).
- Никогда не используйте гепаринизированные пробирки, гепарин ингибирует ПЦР, имитируя спиральную структуру ДНК.
- Выполните этап двойного центрифугирования (центрифугируйте кровь для извлечения плазмы, затем повторите с плазмой, чтобы удалить мусор на дне пробирки), чтобы удалить больше клеточного мусора перед экстракцией ДНК.
- Плазма лучше, чем сыворотка, для восстановления цДНК [24]
Экстракция цДНК
[ редактировать ]Основная привлекательность анализа ктДНК заключается в том, что она извлекается неинвазивным способом путем сбора крови. Для получения вкДНК или ктДНК обычно требуется сбор примерно 3 мл крови в пробирки, покрытые ЭДТА . Использование ЭДТА важно для снижения свертываемости крови. Фракции плазмы и сыворотки крови можно разделить на этапе центрифугирования. ctDNA или cfDNA могут быть впоследствии экстрагированы из этих фракций. Хотя сыворотка имеет тенденцию иметь более высокие уровни вкДНК, это в первую очередь связано с ДНК лимфоцитов. [25] Высокие уровни загрязнения вкДНК неоптимальны, поскольку это может снизить чувствительность обнаружения ктДНК. Поэтому в большинстве исследований для выделения ктДНК используют плазму. Затем плазму снова обрабатывают центрифугированием для удаления остаточных неповрежденных клеток крови. Супернатант используют для экстракции ДНК, которую можно проводить с использованием коммерчески доступных наборов. [ нужна ссылка ]
Анализ цДНК
[ редактировать ]Анализ ктДНК после экстракции требует использования различных методов амплификации и секвенирования. Эти методы можно разделить на две основные группы в зависимости от того, является ли цель опроса всех генов при нецелевом подходе или цель состоит в мониторинге конкретных генов и мутаций при таргетном подходе. [ нужна ссылка ]
Нецелевые подходы
[ редактировать ]Для обнаружения новых мутаций в опухолевой ДНК при мониторинге бремени заболевания или отслеживании лекарственной устойчивости могут потребоваться подходы к секвенированию всего генома или всего экзома. [26] Нецелевые подходы также полезны в исследованиях по наблюдению гетерогенности опухолей или открытию новых мишеней для лекарств. Однако, хотя в некоторых приложениях могут потребоваться нецелевые методы, они более дороги и имеют более низкое разрешение. Это затрудняет выявление редких мутаций или ситуаций, когда присутствуют низкие уровни ctDNA (например, минимальная остаточная болезнь). Кроме того, могут возникнуть проблемы с различием ДНК опухолевых клеток и ДНК нормальных клеток при использовании полногеномного подхода. [ нужна ссылка ]
При секвенировании всего генома или экзома обычно используются технологии высокопроизводительного секвенирования ДНК . Ограничение секвенирования только всем экзомом вместо этого может снизить затраты и увеличить скорость, но за счет потери информации о мутациях в некодирующих регуляторных областях ДНК. [27] Хотя простой просмотр полиморфизма ДНК посредством секвенирования не позволяет дифференцировать ДНК от опухолевых или нормальных клеток, эту проблему можно решить путем сравнения с контрольным образцом нормальной ДНК (например, ДНК, полученной с помощью буккального мазка ). секвенирование экзома полезно для первоначального обнаружения мутаций. Это дает информацию для использования более чувствительных целевых методов, которые затем можно использовать в целях мониторинга заболеваний.
Полногеномное секвенирование позволяет восстановить структурные свойства вкДНК, размер фрагментов и закономерности их фрагментации. Эти уникальные закономерности могут быть важным источником информации для улучшения обнаружения ктДНК или локализации ткани происхождения этих фрагментов. [28] Выбор размера коротких фрагментов (<150 пар оснований) с помощью методов in vitro или in silico может улучшить восстановление мутаций и аберраций числа копий. [15]
Цифровое кариотипирование
[ редактировать ]Первоначально этот метод был разработан лабораторией Берта Фогельштейна , Луиса Диаса и Виктора Велкулеску в Университете Джонса Хопкинса . [29] В отличие от обычного кариотипирования, при котором краситель используется для окрашивания хромосомных полос с целью визуализации хромосом, цифровое кариотипирование использует последовательности ДНК локусов по всему геному для расчета вариаций числа копий . [29] Вариации числа копий распространены при раке и описывают ситуации, когда потеря гетерозиготности гена может привести к снижению функции из-за более низкой экспрессии или дупликации гена, что приводит к сверхэкспрессии.
Персонализированный анализ перестроенных концов
[ редактировать ]После секвенирования всего генома с использованием высокопроизводительного метода секвенирования, такого как Illumina HiSeq, к данным применяется персонализированный анализ перестроенных концов (PARE) для анализа хромосомных перестроек и транслокаций. Этот метод изначально был разработан для анализа ДНК солидных опухолей, но был модифицирован для приложений ctDNA. [29]
Метилирование ДНК и гидроксиметилирование
[ редактировать ]Правильная эпигенетическая маркировка необходима для нормальной экспрессии генов и функционирования клеток, а аберрантные изменения в эпигенетических паттернах являются отличительным признаком рака. [30] Нормальный эпигенетический статус поддерживается в клетке, по крайней мере частично, за счет метилирования ДНК . [31] Измерение закономерностей аберрантного метилирования в ctDNA возможно благодаря стабильному метилированию участков ДНК, называемых « островками CpG ». Метилирование ктДНК можно обнаружить путем обработки бисульфитом . Обработка бисульфитом химически превращает неметилированные цитозины в урацил, оставляя метилированные цитозины неизмененными. Впоследствии ДНК секвенируют, и можно выявить любые изменения в характере метилирования ДНК. Гидроксиметилирование ДНК представляет собой аналогичный признак, который, как было показано, является прогностическим маркером здоровых и болезненных состояний вкДНК, включая рак. [32] [33] )
Целевые подходы
[ редактировать ]При целенаправленном подходе секвенирование ктДНК может быть направлено на создание генетической панели, созданной на основе мутационных «горячих точек» интересующего рака. Это особенно важно для информирования о лечении в ситуациях, когда мутации выявляются в мишенях, поддающихся лечению. [27] Персонализированный целевой анализ ктДНК для каждого пациента также возможен путем сочетания жидкой биопсии со стандартной биопсией первичной ткани. Секвенирование всего генома или всего экзома биопсии первичной опухоли позволяет обнаружить генетические мутации, специфичные для опухоли пациента, и может использоваться для последующего целевого секвенирования цДНК пациента. Наивысшая чувствительность обнаружения ктДНК достигается за счет целевого секвенирования специфических однонуклеотидных полиморфизмов (SNP). Часто мутирующие гены, такие как онкогены, которые обычно имеют мутации «горячих точек», являются хорошими кандидатами для подходов целевого секвенирования. И наоборот, большинство генов-супрессоров опухолей имеют широкий спектр возможных мутаций потери функции по всему гену и как таковые не подходят для целевого секвенирования. [ нужна ссылка ]
Преимущество таргетных подходов заключается в амплификации ктДНК посредством полимеразной цепной реакции (ПЦР) или цифровой ПЦР . Это особенно важно при анализе ктДНК не только потому, что в кровотоке циркулирует относительно низкий уровень ДНК, но и потому, что ктДНК составляет небольшую долю от общего количества экстрагированной внеклеточной ДНК. [27] Следовательно, амплификация интересующих областей может значительно повысить чувствительность обнаружения цтДНК. Однако амплификация посредством ПЦР может привести к ошибкам, учитывая присущую ДНК-полимеразам частоту ошибок. Ошибки, допущенные во время секвенирования, также могут снизить чувствительность обнаружения мутаций цтДНК. [ нужна ссылка ]
Капельная цифровая полимеразная цепная реакция
[ редактировать ]Капельная цифровая ПЦР (ddPCR) основана на цифровой полимеразной цепной реакции , первоначально названной группой Берта Фогельштейна из Университета Джонса Хопкинса . Капельная цифровая ПЦР использует генератор капель для разделения отдельных фрагментов ДНК на капли с использованием масляно-водной эмульсии. Затем в каждой капле происходят отдельные полимеразные цепные реакции с использованием выбранных праймеров против участков цтДНК и продолжаются до конечной точки. Наличие интересующих последовательностей измеряется с помощью флуоресцентных зондов, которые связываются с амплифицированной областью. ddPCR позволяет проводить высококоличественную оценку частот аллелей и мутантов в ctDNA, но ограничена количеством флуоресцентных зондов, которые можно использовать в одном анализе (до 5). [34] Чувствительность анализа может варьироваться в зависимости от количества анализируемой ДНК и составляет около 1 на 10 000. [34] Специфичность следует повысить за счет использования либо модифицированных зондов, связывающихся с малой бороздкой (MGB), либо альтернативы, такой как заблокированные нуклеиновые кислоты (LNA).
Шарики, эмульгирование, амплификация и магнетизм
[ редактировать ]Бисер, эмульгирование, амплификация и магнетизм (BEAMing) — это метод, основанный на капельной цифровой ПЦР для выявления мутаций в цДНК с помощью проточной цитометрии. [35] После того, как ктДНК извлечена из крови, проводится ПЦР с использованием праймеров, предназначенных для воздействия на интересующие области. Эти праймеры также содержат специфические последовательности ДНК или метки. Амплифицированную ДНК смешивают с покрытыми стрептавидином магнитными шариками и эмульгируют в капли. Биотинилированные праймеры, предназначенные для связывания с метками, используются для амплификации ДНК. Биотинилирование позволяет амплифицированной ДНК связываться с магнитными шариками, покрытыми стрептавидином. После завершения ПЦР связанные с ДНК шарики разделяются с помощью магнита. Затем ДНК на шариках денатурируют и позволяют гибридизоваться с флуоресцентными олигонуклеотидами, специфичными для каждой матрицы ДНК. Полученные комплексы шарик-ДНК затем анализируют с помощью проточной цитометрии. Этот метод способен фиксировать частоты аллелей и мутаций благодаря взаимодействию с ddPCR. Однако, в отличие от ddPCR, можно исследовать большее количество последовательностей ДНК благодаря гибкости использования флуоресцентно связанных зондов. Еще одним преимуществом этой системы является то, что выделенную ДНК также можно использовать для последующего секвенирования. [36] Чувствительность 1,6 из 10. 4 до 4,3 из 10 5 . [34]
Персонализированное профилирование рака с помощью глубокого секвенирования
[ редактировать ]Персонализированное профилирование рака путем глубокого секвенирования (CAPP-Seq) было первоначально описано группами Эша Ализаде и Максимилиана Дина в Стэнфордском университете . В этом методе используются селекторные зонды биотинилированных олигонуклеотидов для нацеливания на последовательности ДНК, необходимые для обнаружения цДНК. [37] Общедоступные базы данных о раке были использованы для создания библиотеки зондов против рецидивирующих мутаций рака путем расчета их индекса рецидивирования. Протокол был оптимизирован для низких уровней ДНК, наблюдаемых в коллекции ctDNA. Затем выделенная ДНК подвергается глубокому секвенированию для повышения чувствительности. Этот метод позволяет исследовать сотни участков ДНК. Сообщается, что чувствительность обнаружения ктДНК CAPP-Seq составляет 2,5 молекулы на 1 000 000. [38]
Tagged глубокое секвенирование ампликонов
[ редактировать ]Глубокое секвенирование меченых ампликонов (TAM-Seq) позволяет целенаправленно секвенировать целые гены для обнаружения мутаций в ctDNA. [39] Сначала выполняется общий этап амплификации с использованием праймеров, которые охватывают весь интересующий ген участками длиной 150–200 п.н. Затем с помощью системы микрофлюидики к каждому ампликону прикрепляют адаптеры с уникальным идентификатором для дальнейшей амплификации ДНК в параллельных одиночных реакциях. Было показано, что этот метод успешно выявляет мутации, рассеянные в гене-супрессоре опухоли TP53, у больных раком яичников на поздних стадиях. Чувствительность этого метода составляет 1 к 50.
Безопасное секвенирование
[ редактировать ]Безопасное секвенирование (Safe-Seq) было первоначально описано Бертом Фогельштейном и его группой в Университете Джонса Хопкинса . Safe-Seq снижает частоту ошибок при массовом параллельном секвенировании, чтобы повысить чувствительность к редким мутантам. [40] Это достигается путем добавления последовательности уникального идентификатора (UID) к каждой матрице ДНК. Затем ДНК амплифицируют с использованием добавленных UID и секвенируют. Все молекулы ДНК с одинаковым UID (семейство UID) должны иметь одну и ту же заявленную последовательность ДНК, поскольку они были амплифицированы из одной молекулы. Однако мутации могут быть внесены посредством амплификации, или на этапах секвенирования и анализа могут быть выявлены неправильные назначения оснований. Наличие UID позволяет отделить эти методологические ошибки от истинных мутаций цтДНК. Мутация считается «супермутантной», если 95% секвенированных прочтений совпадают. Чувствительность этого подхода составляет 9 на 1 миллион. [34]
Дуплексное секвенирование
[ редактировать ]Дуплексное секвенирование — это улучшение по сравнению с отдельными UID, добавленными в методе Safe-Seq. [41] При дуплексном секвенировании рандомизированная двухцепочечная ДНК действует как уникальные метки и прикрепляется к инвариантному спейсеру. Метки прикрепляются к обоим концам фрагмента ДНК (α- и β-теги), в результате чего образуются две уникальные матрицы для ПЦР — одна цепь с α-тегом на 5'-конце и β-тегом на 3'-конце, а другая цепь. с меткой β на 5'-конце и меткой α на 3'-конце. Эти фрагменты ДНК затем амплифицируются с помощью праймеров против инвариантных последовательностей меток. Амплифицированную ДНК секвенируют и анализируют. ДНК с дуплексными адаптерами сравниваются, и мутации принимаются только в том случае, если между обеими цепями существует консенсус. Этот метод учитывает как ошибки секвенирования, так и ошибки ранней стадии ПЦР-амплификации. Чувствительность подхода к обнаружению мутантов составляет 1 из 10^7.
CAPP-Seq со встроенным цифровым подавлением ошибок
[ редактировать ]Интегрированное цифровое подавление ошибок (iDES) улучшает анализ ctDNA CAPP-Seq, уменьшая количество ошибок и, следовательно, повышая чувствительность обнаружения. [38] Как сообщалось в 2016 году, iDES сочетает в себе CAPP-Seq с технологией дуплексного секвенирования штрих-кодов и вычислительным алгоритмом, который удаляет стереотипные ошибки, связанные с этапом гибридизации CAPP-Seq. Этот метод также включает в себя, где это возможно, дуплексное секвенирование и включает методы более эффективного восстановления дуплекса из бесклеточной ДНК. Чувствительность этой улучшенной версии CAPP-Seq составляет 4 на 100 000 копий.
Полногеномное секвенирование
[ редактировать ]Исследования полногеномного секвенирования были выполнены на ctDNA, присутствующей у различных пациентов с резистентным к лечению раком простаты (подавляющее большинство, а в некоторых случаях и метастатическим ), раком мочевого пузыря и контрольными пациентами, у которых эта ДНК не была представлена, включая соматические мутации и структурные изменения. перестройки в их геномах.
Этот новый и многообещающий метод предоставил информацию о резистентности к лечению андрогенных рецепторов ингибиторами передачи сигналов , внутриопухолевой гетерогенности (благодаря филогенетической эволюции и молекулярной хронологии), хромосомной нестабильности , вкладе ктДНК в метастазирование через глобальные транскриптомные паттерны (с учетом нуклеосом, присутствующих в транскрипции). стартовые сайты (TSS) и сайты связывания AR (ARB). Таким образом, можно наблюдать геномную и транскриптомную эволюцию ктДНК, происходящую у живых пациентов, у которых развивается резистентность к лечению, поэтому секвенирование ктДНК является элементарным для выявления клинически значимого. различия в фенотипе рака и увидеть, как терапия влияет на пациентов. Кроме того, относительная однородность изменений генов-драйверов среди метастазов подтверждает, что геномные и функциональные изменения при раке простаты являются общими для ctDNA и ткани.
Это делает ктДНК мощным новым инструментом для обнаружения генетических мутаций на уровне генома у пациентов, страдающих метастатическим раком, для наблюдения за клинической значимостью клонального состава этих опухолей и понимания лучшего контроля рака. Эта субклональная реконструкция на основе цДНК благодаря полногеномному секвенированию представляет собой уникальный набор задач и возможностей для научных исследований в области онкологии. Более того, серийное исследование ктДНК показывает отбор, обусловленный лечением.увеличение количества андрогенных рецепторов, поскольку оно увеличивает размерность данных.
Необходима дальнейшая работа, чтобы понять, как расположение и размер метастазов в зависимости от опухолевой нагрузки влияют на циркулирующую опухолевую ДНК и на выбор новых методов для выбора других поражений, которые отражают клинически доминантное заболевание. [42]
Соображения
[ редактировать ]«Нормальное» и обнаружение ДНК опухоли
[ редактировать ]Одна из проблем при использовании ктДНК в качестве биомаркера рака заключается в том, можно ли отличить ккДНК от нормальных клеток с помощью вкДНК. вкДНК высвобождается доброкачественными клетками во время нормального клеточного обновления, а также во время таких процедур, как хирургическое вмешательство , лучевая терапия или химиотерапия . Считается, что лейкоциты вносят основной вклад в вкДНК в сыворотке. [27]
Исследовать
[ редактировать ]ctDNA в скрининге рака
[ редактировать ]Клиническая полезность ктДНК для выявления первичного заболевания частично ограничена чувствительностью современных технологий для обнаружения небольших опухолей с низким уровнем присутствия ктДНК и априорно неизвестных соматических мутаций. [17] [34]
ctDNA в мониторинге рака
[ редактировать ]Признаки заболевания традиционными методами визуализации, такими как КТ , ПЭТ или МРТ, могут отсутствовать после резекции опухоли. Таким образом, анализ ктДНК представляет собой потенциальную возможность обнаружить минимальную остаточную болезнь (МОБ) и, следовательно, возможность рецидива опухоли в тех случаях, когда объемные опухоли отсутствуют с помощью традиционных методов визуализации. [17] Сравнение обнаружения MRD с помощью компьютерной томографии по сравнению с ктДНК ранее проводилось у людей с раком толстой кишки II стадии; В этом исследовании исследователи смогли обнаружить ктДНК у людей, у которых не было обнаружено признаков клинического злокачественного новообразования с помощью компьютерной томографии, что позволяет предположить, что обнаружение ктДНК имеет большую чувствительность для оценки MRD. [27] Однако авторы признают, что анализ ктДНК не лишен ограничений; образцы плазмы, собранные после операции, позволили предсказать рецидив только через 36 месяцев в 48% случаев. [27] Впоследствии были разработаны методы анализа ctDNA как для колоректального рака, так и для диагностики колоректального рака. [43] и меланома . [44]
цДНК как прогностический биомаркер
[ редактировать ]Вопрос о том, можно ли использовать измерение количества или качества ктДНК для определения исходов у людей, больных раком, был предметом исследования. По состоянию на 2015 год это было очень неопределенно. [45] Хотя некоторые исследования показали тенденцию к более высоким уровням ктДНК у людей с метастатическим раком высокой стадии, нагрузка ктДНК не всегда коррелирует с традиционной стадией рака. [34] По состоянию на 2013 год казалось маловероятным, что ктДНК будет иметь клиническое значение в качестве единственного предсказателя прогноза. [46]
Исследования рака
[ редактировать ]Возникновение лекарственно-устойчивых опухолей из-за внутри- и межопухолевой гетерогенности является проблемой эффективности лечения. Минорный генетический клон внутри опухоли может размножиться после лечения, если он несет мутацию, устойчивую к лекарствам. Первоначальная биопсия может пропустить эти клоны из-за низкой частоты или пространственного разделения клеток внутри опухоли. Например, поскольку при биопсии берется только небольшая часть опухоли, клоны, находящиеся в другом месте, могут остаться незамеченными. Это может ввести в заблуждение исследования, направленные на изучение роли гетерогенности опухоли в прогрессировании и рецидиве рака. Использование ктДНК в исследованиях может облегчить эти проблемы, поскольку может предоставить более репрезентативный «скриншот» генетического разнообразия рака как на первичных, так и на метастатических участках. Например, было показано, что ктДНК полезна при изучении клональной эволюции рака пациента до и после схем лечения. [47] Раннее выявление рака по-прежнему является сложной задачей, но недавний прогресс в анализе эпигенетических особенностей вкДНК или раскрытии паттерна фрагментации повышает чувствительность жидкой биопсии. [28] Кроме того, анализ ктДНК является новым инструментом для понимания клонального состава метастатических опухолей, обнаружения различных мутаций в геномном масштабе, изучения субклонального разнообразия, которое влияет на прогноз заболевания, поскольку могут быть обнаружены различные резистентные фенотипы и появление новых механизмов геномного изменения. и транскриптомная устойчивость к лечению. [42]
Проблемы реализации
[ редактировать ]Внедрение ктДНК в клиническую практику во многом затруднено отсутствием стандартизированных методов обработки и анализа ктДНК. Прежде чем анализ ктДНК сможет стать рутинным клиническим анализом, необходимо установить стандартизацию методов сбора образцов (включая время сбора), последующей обработки (экстракция и амплификация ДНК), количественной оценки и проверки. Кроме того, может потребоваться создание панели «стандартных» опухолеассоциированных биомаркеров, учитывая разрешение современных методов секвенирования и обнаружения ctDNA. Секвенирование опухолеспецифичных аберраций из образцов плазмы также может помочь исключить примеси вкДНК из анализа; повышенные уровни вкДНК в нормальных клетках могут быть связаны с причинами, не связанными с раком. [27] Эти методы секвенирования также могут определять клональную эволюцию рака, гетерогенность опухоли и механизмы лекарственной устойчивости, связанные с раком. [42]
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Ван Дж., Мэсси С., Гарсиа-Корбачо Дж., Мульер Ф., Брентон Дж., Калдас К., Пейси С., Бэрд Р., Розенфельд Н. (апрель 2017 г.). «Жидкие биопсии достигают зрелости: на пути к внедрению циркулирующей опухолевой ДНК» . Обзоры природы Рак . 17 (4): 223–238. дои : 10.1038/nrc.2017.7 . ПМИД 28233803 . S2CID 4561229 .
- ^ Нонака, Т; Вонг, DTW (13 июня 2022 г.). «Диагностика по слюне» . Ежегодный обзор аналитической химии . 15 (1): 107–121. Бибкод : 2022ARAC...15..107N . doi : 10.1146/annurev-anchem-061020-123959 . ПМЦ 9348814 . ПМИД 35696523 .
- ^ Jump up to: а б Исфахани, Мохаммад Шахрох; Гамильтон, Эмили Г.; Мехрмохамади, Махья; и др. (апрель 2022 г.). «Вывод об экспрессии генов на основе профилей фрагментации внеклеточной ДНК» . Природная биотехнология . 40 (4): 585–597. дои : 10.1038/s41587-022-01222-4 . ПМЦ 9337986 . ПМИД 35361996 .
- ^ Муттер, Юрик А; Шахрох Исфахани, Мохаммед; Шрерс-Мартин, Йозеф; и др. (28 ноября 2023 г.). «Предполагаемая экспрессия генов с помощью профилирования бесклеточной ДНК позволяет неинвазивную классификацию лимфомы» . Кровь . 142 (Приложение 1): 245–245. дои : 10.1182/blood-2023-186853 .
- ^ Алиг, Стефан К.; Шахрох Исфахани, Мохаммед; Гарофало, Андреа; и др. (25 января 2024 г.). «Отличные подтипы лимфомы Ходжкина, определенные с помощью неинвазивного геномного профилирования» . Природа . 625 (7996): 778–787. дои : 10.1038/s41586-023-06903-x . ПМИД 38081297 .
- ^ Акча Х., Демирай А., Ярен А., Бир Ф., Коселер А., Ивакава Р., Багчи Г., Ёкота Дж. (март 2013 г.). «Применение сывороточной ДНК и пиросеквенирования для обнаружения мутаций EGFR при немелкоклеточном раке легких». Генетика рака . 206 (3): 73–80. doi : 10.1016/j.cancergen.2013.01.005 . ПМИД 23491080 .
- ^ Шварценбах Х., Хун Д.С., Пантел К. (июнь 2011 г.). «Бесклеточные нуклеиновые кислоты как биомаркеры у онкологических больных». Обзоры природы. Рак . 11 (6): 426–37. дои : 10.1038/nrc3066 . ПМИД 21562580 . S2CID 6061607 .
- ^ Строун М., Анкер П. (июль 1972 г.). «Нуклеиновые кислоты спонтанно высвобождаются ушами живой лягушки» . Биохимический журнал . 128 (3): 100П–101П. дои : 10.1042/bj1280100pb . ПМЦ 1173871 . ПМИД 4634816 .
- ^ Строун М., Лиоти Дж., Ледерри С., Олсон-Сэнд А., Анкер П. (ноябрь 2001 г.). «О возможном происхождении и механизме апоптоза циркулирующей ДНК и активного высвобождения ДНК». Клиника Химика Акта; Международный журнал клинической химии . 313 (1–2): 139–42. дои : 10.1016/S0009-8981(01)00665-9 . ПМИД 11694251 .
- ^ Анкер П., Строун М., Морис П.А. (сентябрь 1975 г.). «Спонтанное высвобождение ДНК лимфоцитами крови человека, как показано в системе in vitro» . Исследования рака . 35 (9): 2375–82. ПМИД 1149042 .
- ^ Роджерс Дж.К., Болдт Д., Корнфельд С., Скиннер А., Валерий Ч.Р. (июль 1972 г.). «Выделение дезоксирибонуклеиновой кислоты лимфоцитами, стимулированными фитогемагглютинином или антигеном» . Труды Национальной академии наук Соединенных Штатов Америки . 69 (7): 1685–9. Бибкод : 1972PNAS...69.1685R . дои : 10.1073/pnas.69.7.1685 . ПМК 426778 . ПМИД 4505646 .
- ^ Хейцер Э., Ауэр М., Хоффманн Э.М., Пихлер М., Гаш С., Ульц П., Лакс С., Вальдиспюль-Гейгль Дж., Мауэрманн О., Мохан С., Пристауз Г., Лакнер С., Хёфлер Г., Эйснер Ф., Петру Е., Силл Х., Самонигг Х., Пантель К., Ритдорф С., Бауэрнхофер Т., Гейгль Дж.Б., Спайхер М.Р. (июль 2013 г.). «Установление опухолеспецифических изменений числа копий ДНК плазмы больных раком» . Международный журнал рака . 133 (2): 346–56. дои : 10.1002/ijc.28030 . ПМК 3708119 . ПМИД 23319339 .
- ^ Тьерри А.Р., Мульер Ф., Гонгора С., Олье Дж., Роберт Б., Ичу М., Дель Рио М., Молина Ф. (октябрь 2010 г.). «Происхождение и количественная оценка циркулирующей ДНК у мышей с ксенотрансплантатами колоректального рака человека» . Исследования нуклеиновых кислот . 38 (18): 6159–75. дои : 10.1093/nar/gkq421 . ПМЦ 2952865 . ПМИД 20494973 .
- ^ Мульер Ф, Робер Б, Арнау Пейротт Э, Дель Рио М, Ичу М, Молина Ф, Гонгора С, Тьерри А.Р. (2011). «Высокая фрагментация характеризует циркулирующую ДНК опухолевого происхождения» . ПЛОС ОДИН . 6 (9): e23418. Бибкод : 2011PLoSO...623418M . дои : 10.1371/journal.pone.0023418 . ПМК 3167805 . ПМИД 21909401 .
- ^ Jump up to: а б Мульер Ф., Чандрананда Д., Пискорз А.М., Мур Э.К., Моррис Дж., Алборн Л.Б. и др. (ноябрь 2018 г.). «Улучшенное обнаружение циркулирующей опухолевой ДНК с помощью анализа размера фрагментов» . Научный перевод Мед . 10 (466). doi : 10.1126/scitranslmed.aat4921 . ПМК 6483061 . ПМИД 30404863 .
- ^ Jump up to: а б Писецкий Д.С., Фэрхерст А.М. (июнь 2007 г.). «Происхождение внеклеточной ДНК при удалении мертвых и умирающих клеток». Аутоиммунитет . 40 (4): 281–4. дои : 10.1080/08916930701358826 . ПМИД 17516210 . S2CID 11499768 .
- ^ Jump up to: а б с Аванзини С., Курц Д.М., Чабон Дж.Дж., Модинг Э.Дж., Хори С.С., Гамбхир С.С., Ализаде А.А., Дин М., Райтер Дж.Г. (декабрь 2020 г.). «Математическая модель выделения цтДНК предсказывает размер обнаруженной опухоли» . Достижения науки . 6 (50): eabc4308. Бибкод : 2020SciA....6.4308A . дои : 10.1126/sciadv.abc4308 . ПМЦ 7732186 . ПМИД 33310847 . S2CID 228096858 .
- ^ Васюхин В., Анкер П., Морис П., Лиоти Дж., Ледерри С., Строун М. (апрель 1994 г.). «Точечные мутации гена N-ras в ДНК плазмы крови больных миелодиспластическим синдромом или острым миелогенным лейкозом». Британский журнал гематологии . 86 (4): 774–779. дои : 10.1111/j.1365-2141.1994.tb04828.x . ПМИД 7918071 . S2CID 26365875 .
- ^ Васюхин В., Строун М., Морис П., Лиоти Дж., Ледерри С., Анкер П. (май 1994 г.). Точечные K-ras мутации в ДНК плазмы крови пациентов с колоректальными опухолями в проблемах современной медицины . Том. 5. С. 141–150.
- ^ Ён Э (июль 2014 г.). «Биомаркеры рака: написано кровью» . Природа . 511 (7511): 524–526. Бибкод : 2014Natur.511..524Y . дои : 10.1038/511524a . ПМИД 25079538 . S2CID 4445938 .
- ^ Сюэ Икс, Тир, доктор медицинских наук, Холен И, Чжу Ю.М., Уолл П.Дж. (июнь 2009 г.). «Оптимизация выхода и полезности циркулирующей бесклеточной ДНК из плазмы и сыворотки» (PDF) . Клиника Химика Акта; Международный журнал клинической химии . 404 (2): 100–4. doi : 10.1016/j.cca.2009.02.018 . ПМИД 19281804 .
- ^ Нортон С.Е., Лехнер Дж.М., Уильямс Т., Фернандо М.Р. (октябрь 2013 г.). «Стабилизирующий реагент предотвращает загрязнение внеклеточной ДНК клеточной ДНК в плазме во время хранения и транспортировки образцов крови, что определяется цифровой ПЦР» . Клиническая биохимия . 46 (15): 1561–5. doi : 10.1016/j.clinbiochem.2013.06.002 . ПМИД 23769817 .
- ^ Jump up to: а б с Шервуд Дж.Л., Коркоран С., Браун Х., Шарп А.Д., Мусилова М., Кольманн А. (2016). «Оптимизированные преаналитические методы улучшают обнаружение мутаций KRAS в циркулирующей опухолевой ДНК (цДНК) у пациентов с немелкоклеточным раком легких (НМРЛ)» . ПЛОС ОДИН . 11 (2): e0150197. Бибкод : 2016PLoSO..1150197S . дои : 10.1371/journal.pone.0150197 . ПМЦ 4769175 . ПМИД 26918901 .
- ^ Валле А, Марк М, Бизье А, Кури CE, Лакруа Х, Беннуна Ж, Дуйяр ЖЮ, Дени МГ (ноябрь 2013 г.). «Плазма является лучшим источником циркулирующей бесклеточной ДНК опухолевого происхождения, чем сыворотка, для обнаружения изменений EGFR у пациентов с опухолями легких». Рак легких . 82 (2): 373–4. дои : 10.1016/j.lungcan.2013.08.014 . ПМИД 24007628 .
- ^ Ли Т.Х., Монтальво Л., Хребтоу В., член парламента Буша (февраль 2001 г.). «Количественный анализ геномной ДНК в образцах плазмы и сыворотки: в сыворотке обнаружены более высокие концентрации геномной ДНК, чем в плазме». Переливание . 41 (2): 276–82. дои : 10.1046/j.1537-2995.2001.41020276.x . ПМИД 11239235 . S2CID 45714834 .
- ^ Цинь З, Любимов В.А., Чжоу С, Тонг Ю, Лян Дж (апрель 2016 г.). «Бесклеточная циркулирующая опухолевая ДНК при раке» . Китайский журнал рака . 35:36 . дои : 10.1186/s40880-016-0092-4 . ПМЦ 4823888 . ПМИД 27056366 .
- ^ Jump up to: а б с д и ж г Хейцер Э., Ульц П., Гейгл Дж.Б. (январь 2015 г.). «Циркулирующая опухолевая ДНК как жидкая биопсия рака» . Клиническая химия . 61 (1): 112–23. дои : 10.1373/clinchem.2014.222679 . ПМИД 25388429 .
- ^ Jump up to: а б ван дер Поль И., Мульер Ф. (октябрь 2019 г.). «На пути к раннему выявлению рака путем расшифровки эпигенетических и экологических отпечатков бесклеточной ДНК» . Раковая клетка . 36 (4): 350–368. doi : 10.1016/j.ccell.2019.09.003 . ПМИД 31614115 .
- ^ Jump up to: а б с Лири Р.Дж., Саусен М., Кинд И., Пападопулос Н., Карптен Дж.Д., Крейг Д., О'Шонесси Дж., Кинцлер К.В., Пармиджани Г., Фогельштейн Б., Диас Л.А., Велкулеску В.Е. (ноябрь 2012 г.). «Выявление хромосомных изменений в кровообращении онкологических больных с помощью полногеномного секвенирования» . Наука трансляционной медицины . 4 (162): 162ра154. doi : 10.1126/scitranslmed.3004742 . ПМЦ 3641759 . ПМИД 23197571 .
- ^ Преображенский Б.С. (1966). «[Современные перспективы и метод систематического лечения неврита улитки и хронической лабиринтопатии]». Вестник Оториноларингологии . 28 (1): 3–11. ПМИД 5988180 .
- ^ Бомонт Дж., Доббинс С., Латта Д., Макмиллин В.П. (май 1990 г.). «Мекитазин в лечении сенной лихорадки». Британский журнал клинической практики . 44 (5): 183–8. ПМИД 1975200 .
- ^ Ли В, Чжан Икс, Лу Икс, Ю Л, Сун Ю, Ло Цзы и др. (октябрь 2017 г.). «Сигнатуры 5-гидроксиметилцитозина в циркулирующей внеклеточной ДНК как диагностические биомаркеры рака человека» . Клеточные исследования . 27 (10): 1243–1257. дои : 10.1038/cr.2017.121 . ПМЦ 5630683 . ПМИД 28925386 .
- ^ Сонг CX, Инь С, Ма Л, Уиллер А, Чен Ю, Чжан Ю, Лю Б, Сюн Дж, Чжан В, Ху Дж, Чжоу З, Донг Б, Тянь З, Джеффри СС, Чуа М.С., Со С, Ли В , Вэй Ю, Дяо Дж, Се Д, Quake SR (октябрь 2017 г.). «Сигнатуры 5-гидроксиметилцитозина во внеклеточной ДНК предоставляют информацию о типах и стадиях опухолей» . Клеточные исследования . 27 (10): 1231–1242. дои : 10.1038/cr.2017.106 . ПМК 5630676 . ПМИД 28820176 .
- ^ Jump up to: а б с д и ж Батлер Т.М., Спеллман П.Т., Грей Дж. (февраль 2017 г.). «ДНК циркулирующих опухолей как инструмент раннего обнаружения и диагностики». Текущее мнение в области генетики и развития . 42 : 14–21. дои : 10.1016/j.gde.2016.12.003 . ПМИД 28126649 .
- ^ Дрессман Д., Ян Х., Траверсо Г., Кинцлер К.В., Фогельштейн Б. (июль 2003 г.). «Преобразование отдельных молекул ДНК во флуоресцентные магнитные частицы для обнаружения и подсчета генетических вариаций» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (15): 8817–22. Бибкод : 2003PNAS..100.8817D . дои : 10.1073/pnas.1133470100 . ПМК 166396 . ПМИД 12857956 .
- ^ Диль Ф., Ли М., Хе Ю., Кинцлер К.В., Фогельштейн Б., Дрессман Д. (июль 2006 г.). «BEAMing: ПЦР одиночных молекул на микрочастицах в эмульсиях вода в масле». Природные методы . 3 (7): 551–9. дои : 10.1038/nmeth898 . ПМИД 16791214 . S2CID 7059151 .
- ^ Ньюман А.М., Брэтман С.В., То Дж., Винн Дж.Ф., Эклов Н.К., Модлин Л.А., Лю КЛ, Нил Дж.В., Уэйкли Х.А., Мерритт Р.Э., Шрагер Дж.Б., Лу Б.В., Ализаде А.А., Дин М. (май 2014 г.). «Сверхчувствительный метод количественного определения циркулирующей опухолевой ДНК с широким охватом пациентов» . Природная медицина . 20 (5): 548–54. дои : 10.1038/нм.3519 . ПМК 4016134 . ПМИД 24705333 .
- ^ Jump up to: а б Ньюман А.М., Лавджой А.Ф., Класс Д.М., Курц Д.М., Чабон Дж.Дж., Шерер Ф. и др. (май 2016 г.). «Интегрированное цифровое подавление ошибок для улучшения обнаружения циркулирующей опухолевой ДНК» . Природная биотехнология . 34 (5): 547–555. дои : 10.1038/nbt.3520 . ПМЦ 4907374 . ПМИД 27018799 .
- ^ Форшью Т., Муртаза М., Паркинсон С., Гейл Д., Цуй Д.В., Капер Ф., Доусон С.Дж., Пискорз А.М., Хименес-Линан М., Бентли Д., Хэдфилд Дж., Мэй А.П., Калдас С., Брентон Дж.Д., Розенфельд Н. (май 2012 г.) . «Неинвазивная идентификация и мониторинг раковых мутаций путем целенаправленного глубокого секвенирования ДНК плазмы». Наука трансляционной медицины . 4 (136): 136ра68. doi : 10.1126/scitranslmed.3003726 . ПМИД 22649089 . S2CID 34723244 .
- ^ Кинде И., Ву Дж., Пападопулос Н., Кинцлер К.В., Фогельштейн Б. (июнь 2011 г.). «Обнаружение и количественная оценка редких мутаций с помощью массового параллельного секвенирования» . Труды Национальной академии наук Соединенных Штатов Америки . 108 (23): 9530–5. Бибкод : 2011PNAS..108.9530K . дои : 10.1073/pnas.1105422108 . ПМК 3111315 . ПМИД 21586637 .
- ^ Кеннеди С.Р., Шмитт М.В., Фокс Э.Дж., Корн Б.Ф., Солк Дж.Дж., Ан Э.Х., Приндл М.Дж., Куонг К.Дж., Шен Дж.К., Рискес Р.А., Леб Л.А. (ноябрь 2014 г.). «Обнаружение ультранизкочастотных мутаций методом дуплексного секвенирования» . Протоколы природы . 9 (11): 2586–606. дои : 10.1038/nprot.2014.170 . ПМЦ 4271547 . ПМИД 25299156 .
- ^ Jump up to: а б с Гербертс, Кэмерон; Аннала, Матти; Сипола, Джунатан; Нг, Сара В.С.; Чен, Синьи Э.; Нурминен, Ансси; Корхонен, Ольга В.; Мунзур, Аслы Д.; Бежа, Кевин; Шенлау, Елена; Берналес, Сесили К.; Ритч, Эли; Бэкон, Джек Фольксваген; Лак, Натан А.; Никтер, Матти (август 2022 г.). «Глубокая полногеномная ctDNA хронология резистентного к лечению рака простаты» . Природа . 608 (7921): 199–208. Бибкод : 2022Natur.608..199H . дои : 10.1038/s41586-022-04975-9 . ISSN 1476-4687 . ПМИД 35859180 . S2CID 250730778 .
- ^ Цзоу, Дунхуэй; Дэй, Роберт; Кокадис, Джуди А; Паракал, Сара; Митчелл, Уилсон; Блэк, Майкл А; Лоуренс, Бен; Фицджеральд, Сандра; Печать, Кристин; Джексон, Кристофер; Гилфорд, Парри (13 ноября 2020 г.). «Циркулирующая опухолевая ДНК является чувствительным маркером для рутинного мониторинга ответа на лечение при распространенном колоректальном раке» . Канцерогенез . 41 (11): 1507–1517. дои : 10.1093/carcin/bgaa102 . ISSN 0143-3334 . ПМИД 32955091 .
- ^ Фицджеральд, Сандра; Бленкирон, Шери; Стивенс, Розали; Мэти, Джон А.; Сомерс-Эдгар, Тиффани; Рольф, Гилл; Мартин, Ричард; Джексон, Кристофер; Экклс, Майкл; Робб, Тэмсин; Роджер, Юан; Лоуренс, Бен; Гилфорд, Парри; Лэшэм, Аннетт; Печать, Кристин Г. (2023). «Динамическая мутационная сложность ctDNA у пациентов с меланомой, получающих иммунотерапию» . Молекулярная диагностика и терапия . 27 (4): 537–550. дои : 10.1007/s40291-023-00651-4 . ISSN 1177-1062 . ПМЦ 10131510 . ПМИД 37099071 .
- ^ Раписувон С., Витч Э.Э., Веллштейн А. (2016). «Циркулирующие биомаркеры для мониторинга прогрессирования рака и лечения» . Журнал вычислительной и структурной биотехнологии . 14 : 211–22. дои : 10.1016/j.csbj.2016.05.004 . ПМЦ 4913179 . ПМИД 27358717 .
- ^ Кроули Э., Ди Николантонио Ф., Лупакис Ф., Барделли А. (август 2013 г.). «Жидкостная биопсия: мониторинг генетики рака в крови» . Обзоры природы. Клиническая онкология . 10 (8): 472–84. дои : 10.1038/nrclinonc.2013.110 . ПМИД 23836314 . S2CID 25537784 .
- ^ Муртаза М, Доусон С.Дж., Погребняк К., Руэда ОМ, Провенцано Е, Грант Дж., Чин С.Ф., Цуй Д.В., Марас Ф, Гейл Д., Али ХР, Шах П., Контенте-Куомо Т., Фарахани Х., Шуманский К., Кингсбери З., Хамфри С., Бентли Д., Шах С.П., Уоллис М., Розенфельд Н., Калдас К. (ноябрь 2015 г.). «Мультифокальная клональная эволюция, характеризующаяся использованием циркулирующей опухолевой ДНК в случае метастатического рака молочной железы» . Природные коммуникации . 6 (1): 8760. Бибкод : 2015NatCo...6.8760M . дои : 10.1038/ncomms9760 . ПМЦ 4659935 . ПМИД 26530965 .
Дальнейшее чтение
[ редактировать ]- Применение метилирования циркулирующей опухолевой ДНК в диагностике рака легких. Май 2019 г.
- Циркулирующая опухолевая ДНК: новое поколение биомаркеров рака. Февраль 2014 г.
- «Жидкая биопсия» ктДНК может произвести революцию в лечении рака, ноябрь 2014 г.
- Карачалиу Н., Майо-де-Лас-Касас К., Молина-Вила М.А., Роселл Р. (март 2015 г.). «Жидкая биопсия в реальном времени становится реальностью в лечении рака» . Анналы трансляционной медицины . 3 (3): 36. doi : 10.3978/j.issn.2305-5839.2015.01.16 . ПМЦ 4356857 . ПМИД 25815297 .
- Марусина, Катя (8 февраля 2018 г.). «Выявление циркулирующей опухолевой ДНК» . КлиническиеOMIC . Проверено 5 марта 2018 г.
- Дю-Буа, Асанте (2019). «Жидкостная биопсия при раке яичников с использованием циркулирующей опухолевой ДНК и клеток: готовы к прайм-тайму?» . Письма о раке . 468 : 59–71. дои : 10.1016/j.canlet.2019.10.014 . ПМИД 31610267 .
- NucPosDB: база данных о расположении нуклеосом in vivo и нуклеосомике внеклеточной ДНК.