Прогнозирование межбелкового взаимодействия
Прогнозирование межбелкового взаимодействия — это область, объединяющая биоинформатику и структурную биологию в попытке идентифицировать и каталогизировать физические взаимодействия между парами или группами белков. Понимание белок-белковых взаимодействий важно для исследования внутриклеточных сигнальных путей, моделирования структур белковых комплексов и для понимания различных биохимических процессов.
Экспериментально физические взаимодействия между парами белков можно выявить с помощью различных методов, включая дрожжевые двухгибридные системы, анализы комплементации белковых фрагментов (PCA), аффинную очистку/ масс-спектрометрию , белковые микрочипы , флуоресцентный резонансный перенос энергии (FRET), и микромасштабный термофорез (MST). Попытки экспериментально определить интерактом многих видов продолжаются. Экспериментально установленные взаимодействия обычно служат основой для вычислительных методов прогнозирования взаимодействий, например, с использованием гомологичных белковых последовательностей у разных видов. Однако существуют также методы, которые прогнозируют взаимодействия заново , без предварительного знания существующих взаимодействий.
Методы
[ редактировать ]Белки, которые взаимодействуют, с большей вероятностью будут эволюционировать совместно, [ 1 ] [ 2 ] [ 3 ] [ 4 ] следовательно, можно делать выводы о взаимодействиях между парами белков на основе их филогенетических расстояний. В некоторых случаях также наблюдалось, что пары взаимодействующих белков имеют слитые ортологи в других организмах. Кроме того, структурно решен ряд связанных белковых комплексов, и их можно использовать для идентификации остатков, которые опосредуют взаимодействие, чтобы аналогичные мотивы могли быть расположены в других организмах.
Филогенетическое профилирование
[ редактировать ]
филогенетического профиля Метод основан на гипотезе о том, что если два или более белка одновременно присутствуют или отсутствуют в нескольких геномах, то они, вероятно, функционально связаны. [ 5 ] Рисунок A иллюстрирует гипотетическую ситуацию, в которой белки A и B идентифицируются как функционально связанные из-за их идентичных филогенетических профилей в 5 различных геномах. Объединенный институт генома предоставляет интегрированную базу данных микробных геномов и микробиомов ( JGI IMG ), в которой есть инструмент филогенетического профилирования отдельных генов и генных кассет.
Прогнозирование совместно эволюционировавших пар белков на основе схожих филогенетических деревьев
[ редактировать ]Было замечено, что филогенетические деревья лигандов и рецепторов часто оказывались более похожими, чем случайно. [ 4 ] Вероятно, это связано с тем, что они столкнулись с одинаковым давлением отбора и одновременно эволюционировали. Этот метод [ 6 ] использует филогенетические деревья пар белков, чтобы определить, существуют ли взаимодействия. Для этого находят гомологи интересующих белков (с использованием инструмента поиска последовательностей, такого как BLAST ), и выполняют выравнивание нескольких последовательностей (с помощью инструментов выравнивания, таких как Clustal ), чтобы построить матрицы расстояний для каждого из представляющих интерес белков. [ 4 ] Матрицы расстояний затем следует использовать для построения филогенетических деревьев. Однако сравнение между филогенетическими деревьями затруднено, и современные методы позволяют обойти это, просто сравнивая матрицы расстояний. [ 4 ] . Матрицы расстояний белков используются для расчета коэффициента корреляции, большее значение которого соответствует коэволюции. Преимущество сравнения матриц расстояний вместо филогенетических деревьев состоит в том, что результаты не зависят от использованного метода построения дерева. Обратной стороной является то, что разностные матрицы не являются идеальным представлением филогенетических деревьев, и использование такого сокращения может привести к неточностям. [ 4 ] Еще один фактор, заслуживающий внимания, заключается в том, что между филогенетическими деревьями любого белка, даже не взаимодействующего между собой, существует фоновое сходство. Если это не учитывать, это может привести к высокому уровню ложноположительных результатов. По этой причине некоторые методы создают фоновое дерево с использованием последовательностей 16S рРНК, которые они используют в качестве канонического дерева жизни. Матрица расстояний, построенная на основе этого древа жизни, затем вычитается из матриц расстояний интересующих белков. [ 7 ] Однако, поскольку матрицы расстояний РНК и матрицы расстояний ДНК имеют разный масштаб, предположительно из-за того, что РНК и ДНК имеют разные скорости мутаций, матрицу РНК необходимо масштабировать, прежде чем ее можно будет вычесть из матриц ДНК. [ 7 ] Используя белки молекулярных часов, можно рассчитать коэффициент масштабирования расстояния между белками и РНК. [ 7 ] Этот коэффициент используется для масштабирования матрицы РНК.

Метод Розеттского камня (слияния генов)
[ редактировать ]Метод Розеттского камня или слияния доменов основан на гипотезе о том, что взаимодействующие белки иногда сливаются в один белок. [ 3 ] Например, два или более отдельных белка в геноме могут быть идентифицированы как слитые в один белок в другом геноме. Отдельные белки, вероятно, будут взаимодействовать и, таким образом, вероятно, функционально связаны. Примером этого является фермент сукцинил-коА-трансфераза человека , который у человека встречается в виде одного белка, а - в виде двух отдельных белков: ацетат-коА-трансферазы альфа и ацетат-коА-трансферазы бета у Escherichia coli . [ 3 ] Чтобы идентифицировать эти последовательности, алгоритм сходства последовательностей, такой как тот, который используется BLAST необходим . Например, если бы у нас были аминокислотные последовательности белков A и B и аминокислотные последовательности всех белков в определенном геноме, мы могли бы проверить каждый белок в этом геноме на наличие непересекающихся областей сходства последовательностей с белками A и B. . На рисунке B показано выравнивание последовательности BLAST сукцинил-коА-трансферазы с ее двумя отдельными гомологами в E. coli Эти две субъединицы имеют неперекрывающиеся области сходства последовательностей с человеческим белком, обозначенные розовыми областями, при этом альфа-субъединица похожа на первую половину белка, а бета-субъединица похожа на вторую половину. Одним из ограничений этого метода является то, что не все взаимодействующие белки могут быть обнаружены слитыми в другом геноме и, следовательно, не могут быть идентифицированы этим методом. С другой стороны, слияние двух белков не требует их физического взаимодействия. Например, домены SH2 и SH3 в Известно, что белок src взаимодействует. Однако многие белки обладают гомологами этих доменов, и не все они взаимодействуют. [ 3 ]
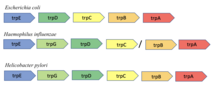
Сохраненное генное окружение
[ редактировать ]Метод консервативного соседства основан на гипотезе о том, что если гены, кодирующие два белка, являются соседями по хромосоме во многих геномах, то они, вероятно, функционально связаны. Метод основан на наблюдении Bork et al. сохранения пар генов в девяти бактериальных и архейных геномах. Этот метод наиболее эффективен для прокариот с оперонами, поскольку организация генов в опероне обычно связана с функцией. [ 8 ] Например, гены trpA и trpB в Escherichia coli кодируют две субъединицы фермента триптофансинтазы, которые, как известно, взаимодействуют и катализируют одну реакцию. Было показано, что соседство этих двух генов сохраняется в девяти различных бактериальных и архейных геномах. [ 8 ]
Методы классификации
[ редактировать ]Методы классификации используют данные для обучения программы (классификатора) различению положительных примеров взаимодействующих пар белок/домен от отрицательных примеров невзаимодействующих пар. Популярными классификаторами являются машины случайных лесных решений (RFD) и машины опорных векторов. RFD дает результаты, основанные на доменном составе взаимодействующих и невзаимодействующих пар белков. Когда RFD получает пару белков для классификации, сначала создает представление пары белков в векторе. [ 9 ] Вектор содержит все типы доменов, используемые для обучения RFD, и для каждого типа домена вектор также содержит значение 0, 1 или 2. Если пара белков не содержит определенного домена, то значение для этого домена равно 0. Если один из белков пары содержит домен, то значение равно 1. Если оба белка содержат домен, то значение равно 2. [ 9 ] Используя данные обучения, RFD создает лес решений, состоящий из множества деревьев решений. Каждое дерево решений оценивает несколько доменов и на основе наличия или отсутствия взаимодействий в этих доменах принимает решение о том, взаимодействует ли пара белков. Векторное представление пары белков оценивается каждым деревом, чтобы определить, являются ли они взаимодействующей парой или невзаимодействующей парой. Лес суммирует все данные, поступающие от деревьев, и принимает окончательное решение. [ 9 ] Сила этого метода в том, что он не предполагает, что домены взаимодействуют независимо друг от друга. Это позволяет использовать для предсказания несколько доменов в белках. [ 9 ] Это большой шаг вперед по сравнению с предыдущими методами, которые могли прогнозировать только на основе одной пары доменов. Ограничением этого метода является то, что для получения результатов он полагается на набор обучающих данных. Таким образом, использование различных наборов обучающих данных может повлиять на результаты. Недостатком большинства методов является отсутствие отрицательных данных, например, отсутствие взаимодействия белков, которое можно преодолеть с помощью отрицательной выборки на основе топологии. [ 10 ]
Вывод о взаимодействиях из гомологичных структур
[ редактировать ]Эта группа методов [ 11 ] [ 9 ] [ 12 ] [ 13 ] [ 14 ] использует известные структуры белковых комплексов для прогнозирования и структурного моделирования взаимодействий между последовательностями запрашиваемых белков. Процесс прогнозирования обычно начинается с использования метода на основе последовательностей (например, Interolog ) для поиска структур белковых комплексов, которые гомологичны запрашиваемым последовательностям. Эти известные сложные структуры затем используются в качестве шаблонов для структурного моделирования взаимодействия между последовательностями запросов. Преимущество этого метода состоит в том, что он не только позволяет сделать вывод о взаимодействиях белков, но и предлагает модели структурного взаимодействия белков, что может дать некоторое представление о механизме этого взаимодействия на атомном уровне. С другой стороны, способность этих методов делать прогнозы ограничена ограниченным числом известных структур белковых комплексов.
Методы ассоциации
[ редактировать ]Методы ассоциации ищут характерные последовательности или мотивы, которые могут помочь различать взаимодействующие и невзаимодействующие пары. Классификатор обучается путем поиска пар последовательность-сигнатура, где один белок содержит одну сигнатуру последовательности, а его взаимодействующий партнер содержит другую сигнатуру последовательности. [ 15 ] Они специально ищут сигнатуры последовательностей, которые встречаются вместе чаще, чем случайно. При этом используется показатель логарифмических шансов, который рассчитывается как log2(Pij/PiPj), где Pij — наблюдаемая частота доменов i и j, встречающихся в одной паре белков; Pi и Pj — фоновые частоты доменов i и j в данных. Взаимодействия с прогнозируемыми доменами — это взаимодействия с положительными значениями логарифмических шансов, а также имеющие несколько вхождений в базу данных. [ 15 ] Недостатком этого метода является то, что он рассматривает каждую пару взаимодействующих доменов отдельно и предполагает, что они взаимодействуют независимо друг от друга.
Выявление структурных закономерностей
[ редактировать ]Этот метод [ 16 ] [ 17 ] создает библиотеку известных интерфейсов белок-белок из PDB , где интерфейсы определяются как пары полипептидных фрагментов, которые находятся ниже порога, немного превышающего радиус Ван-дер-Ваальса задействованных атомов. Затем последовательности в библиотеке группируются на основе структурного выравнивания, а избыточные последовательности удаляются. Остатки, которые имеют высокий (обычно >50%) уровень частоты для данной позиции, считаются горячими точками. [ 18 ] Эта библиотека затем используется для идентификации потенциальных взаимодействий между парами целей при условии, что они имеют известную структуру (т.е. присутствуют в PDB ).
Моделирование байесовской сети
[ редактировать ]Байесовские методы [ 19 ] интегрируйте данные из самых разных источников, включая как экспериментальные результаты, так и предыдущие вычислительные прогнозы, и используйте эти функции для оценки вероятности того, что конкретное потенциальное взаимодействие белков является истинно положительным результатом. Эти методы полезны, поскольку экспериментальные процедуры, особенно эксперименты с двумя гибридами дрожжей, чрезвычайно зашумлены и дают множество ложноположительных результатов, в то время как ранее упомянутые вычислительные методы могут предоставить только косвенные доказательства того, что определенная пара белков может взаимодействовать. [ 20 ]
Анализ исключения пар доменов
[ редактировать ]Анализ исключения пар доменов [ 21 ] обнаруживает специфические взаимодействия доменов, которые трудно обнаружить с помощью байесовских методов. Байесовские методы хороши для обнаружения неспецифических беспорядочных взаимодействий и не очень хороши для обнаружения редких специфических взаимодействий. Метод анализа исключения пар доменов рассчитывает E-оценку, которая измеряет взаимодействие двух доменов. Он рассчитывается как log (вероятность взаимодействия двух белков при условии, что домены взаимодействуют/вероятность взаимодействия двух белков при условии, что домены не взаимодействуют). Вероятности, необходимые в формуле, рассчитываются с использованием процедуры максимизации ожидания, которая представляет собой метод оценки параметров в статистических моделях. Высокие баллы E указывают на то, что два домена могут взаимодействовать, тогда как низкие баллы указывают на то, что другие домены, образующие пару белков, с большей вероятностью будут нести ответственность за взаимодействие. Недостатком этого метода является то, что он не учитывает ложноположительные и ложноотрицательные результаты в экспериментальных данных.
Проблема контролируемого обучения
[ редактировать ]Проблему прогнозирования PPI можно сформулировать как задачу обучения с учителем. В этой парадигме известные белковые взаимодействия контролируют оценку функции, которая может предсказать, существует или нет взаимодействие между двумя белками, учитывая данные о белках (например, уровни экспрессии каждого гена в различных экспериментальных условиях, информацию о местоположении, филогенетический профиль и т. д.). .).
Связь с методами стыковки
[ редактировать ]Область предсказания белок-белковых взаимодействий тесно связана с областью белок-белкового докинга , которая пытается использовать геометрические и стерические соображения для объединения двух белков известной структуры в связанный комплекс. Это полезный способ исследования в тех случаях, когда оба белка в паре имеют известные структуры и известно (или, по крайней мере, есть сильное подозрение), что они взаимодействуют, но поскольку многие белки не имеют экспериментально определенных структур, методы прогнозирования взаимодействия на основе последовательностей являются полезными. организма особенно полезен в сочетании с экспериментальными исследованиями интерактома .
См. также
[ редактировать ]- Интерактом
- Белково-белковое взаимодействие
- Прогнозирование функции белка
- Прогнозирование структуры белка
- Программное обеспечение для прогнозирования структуры белка
- Генное предсказание
- Макромолекулярный стыковка
- Предиктор сайта взаимодействия белок-ДНК
- Двухгибридный скрининг
- ФастКонтакт
Ссылки
[ редактировать ]- ^ Перейти обратно: а б Дандекар Т., Снел Б., Хайнен М. и Борк П. (1998) «Сохранение порядка генов: отпечаток пальцев белков, которые физически взаимодействуют». Тенденции биохимии. наук. (23), 324-328
- ^ Энрайт А.Дж., Илиопулос И., Кирипид Н.К. и Узунис К.А. (1999) «Карты взаимодействия белков для полных геномов, основанные на событиях слияния генов». Природа (402), 86-90
- ^ Перейти обратно: а б с д Маркотт Э.М., Пеллегрини М., Нг Х.Л., Райс Д.В., Йейтс Т.О., Айзенберг Д. (1999) «Обнаружение функции белка и белок-белковых взаимодействий по последовательностям генома». Наука (285), 751-753
- ^ Перейти обратно: а б с д и Пасос, Ф.; Валенсия, А. (2001). «Сходство филогенетических деревьев как показатель белок-белкового взаимодействия» . Белковая инженерия . 9 (14): 609–614. дои : 10.1093/белок/14.9.609 . ПМИД 11707606 .
- ^ Перейти обратно: а б Раман, Картик (15 февраля 2010 г.). «Построение и анализ сетей белок-белкового взаимодействия» . Автоматизированное экспериментирование . 2 (1): 2. дои : 10.1186/1759-4499-2-2 . ISSN 1759-4499 . ПМЦ 2834675 . ПМИД 20334628 .
- ^ Тан Ш., Чжан З., Нг С.К. (2004) «СОВЕТ: автоматическое обнаружение и проверка взаимодействия посредством совместной эволюции». Нуклеиновые кислоты Рез. , 32 (проблема с веб-сервером): W69-72.
- ^ Перейти обратно: а б с Пасос, Ф; Ранея, Дж.А.; Хуан, Д; Штернберг, MJ (2005). «Оценка коэволюции белков в контексте древа жизни помогает прогнозировать интерактом». Дж Мол Биол . 352 (4): 1002–1015. дои : 10.1016/j.jmb.2005.07.005 . ПМИД 16139301 .
- ^ Перейти обратно: а б Дандекар, Т. (1 сентября 1998 г.). «Сохранение порядка генов: отпечаток белков, которые физически взаимодействуют». Тенденции биохимических наук . 23 (9): 324–328. дои : 10.1016/S0968-0004(98)01274-2 . ISSN 0968-0004 . ПМИД 9787636 .
- ^ Перейти обратно: а б с д и Чен, XW; Лю, М (2005). «Прогнозирование белок-белковых взаимодействий с использованием структуры леса случайных решений» . Биоинформатика . 21 (24): 4394–4400. doi : 10.1093/биоинформатика/bti721 . ПМИД 16234318 .
- ^ Чаттерджи, Аян; Раванди, Бабак; Филип, Наоми Х.; Абдельмесих, Марио; Моури, Уильям Р.; Риккьюто, Пьеро; Лян, Юпу; Дин, Вэй; Мобарек, Хуан К. (29 апреля 2024 г.), Отрицательная выборка на основе топологии повышает обобщаемость прогнозирования белок-белковых взаимодействий , doi : 10.1101/2024.04.27.591478 , получено 4 мая 2024 г.
- ^ Элой, П.; Рассел, РБ (2003). «InterPreTS: прогнозирование взаимодействия белков через третичную структуру» . Биоинформатика . 19 (1): 161–162. дои : 10.1093/биоинформатика/19.1.161 . ПМИД 12499311 .
- ^ Фукухара, Наоши и Такеши Кавабата. (2008) «HOMCOS: сервер для прогнозирования взаимодействующих пар белков и взаимодействующих сайтов путем моделирования гомологии сложных структур» Nucleic Acids Research , 36 (S2): 185-.
- ^ Киттихотират В., М. Геркин, Р.Э. Бамгарнер и Р. Самудрала (2009) «Protinfo PPC: веб-сервер для прогнозирования белковых комплексов на атомном уровне» Nucleic Acids Research , 37 (выпуск веб-сервера): 519-25.
- ^ Шумейкер, бакалавр искусств; Чжан, Д; Тангуду, РР; Тьяги, М; Фонг, Дж. Х.; Марчлер-Бауэр, А; Брайант, Ш.; Мадей, Т; Панченко А.Р. (январь 2010 г.). «Сервер предполагаемого биомолекулярного взаимодействия - веб-сервер для анализа и прогнозирования партнеров по взаимодействию белков и сайтов связывания» . Нуклеиновые кислоты Рез . 38 (Проблема с базой данных): D518–24. дои : 10.1093/нар/gkp842 . ПМЦ 2808861 . ПМИД 19843613 .
- ^ Перейти обратно: а б Спринзак, Э; Маргалит, Х (2001). «Коррелированные последовательности-сигнатуры как маркеры белок-белкового взаимодействия». Дж Мол Биол . 311 (4): 681–692. дои : 10.1006/jmbi.2001.4920 . ПМИД 11518523 .
- ^ Айтуна, А.С.; Кескин, О.; Гурсой, А. (2005). «Прогнозирование белок-белковых взаимодействий путем объединения сохранения структуры и последовательности в интерфейсах белков» . Биоинформатика . 21 (12): 2850–2855. doi : 10.1093/биоинформатика/bti443 . ПМИД 15855251 .
- ^ Огмен, У.; Кескин, О.; Айтуна, А.С.; Нусинов Р.; Гурсой, А. (2005). «ПРИЗМА: белковые взаимодействия посредством структурного соответствия» . Нуклеиновые кислоты Рез . 33 (проблема с веб-сервером): W331–336. дои : 10.1093/nar/gki585 . ПМЦ 1160261 . ПМИД 15991339 .
- ^ Кескин, О.; Ма, Б.; Нусинов, Р. (2004). «Горячие области в белок-белковых взаимодействиях: организация и вклад структурно консервативных остатков горячих точек». Дж. Мол. Биол . 345 (5): 1281–1294. дои : 10.1016/j.jmb.2004.10.077 . ПМИД 15644221 .
- ^ Янсен, Р; Ю, Х; Гринбаум, Д; Клюгер, Ю; Кроган, Нью-Джерси; Чунг, С; Эмили, А; Снайдер, М; Гринблатт, Дж. Ф.; Герштейн, М. (2003). «Подход байесовских сетей для прогнозирования белок-белковых взаимодействий на основе геномных данных». Наука . 302 (5644): 449–53. Бибкод : 2003Sci...302..449J . CiteSeerX 10.1.1.217.8151 . дои : 10.1126/science.1087361 . ПМИД 14564010 . S2CID 5293611 .
- ^ Чжан, королевский адвокат; Петри, Д; Дэн, Л; Цян, Л; Ши, Ю; Четверг, Калифорния; Бисикирская, Б; Лефевр, К; Акчили, Д; Хантер, Т; Маниатис, Т; Калифано, А; Хониг, Б. (2012). «Структурное предсказание белок-белковых взаимодействий в масштабе всего генома» . Природа . 490 (7421): 556–60. Бибкод : 2012Natur.490..556Z . дои : 10.1038/nature11503 . ПМЦ 3482288 . ПМИД 23023127 .
- ^ Шумейкер, бакалавр искусств; Панченко А.Р. (2007). «Расшифровка белок-белковых взаимодействий. Часть II. Вычислительные методы прогнозирования партнеров по взаимодействию белков и доменов» . ПЛОС Компьютерная Биол . 3 (4): е43. Бибкод : 2007PLSCB...3...43S . дои : 10.1371/journal.pcbi.0030043 . ПМЦ 1857810 . ПМИД 17465672 .