Незаменимый ген
Эссенциальные гены — это незаменимые гены , необходимые организмам для роста и воспроизводства потомства в определенной среде. [1] Однако необходимость существования во многом зависит от обстоятельств, в которых живет организм. Например, ген, необходимый для переваривания крахмала , необходим только в том случае, если крахмал является единственным источником энергии. В последнее время предпринимаются систематические попытки выявить те гены, которые абсолютно необходимы для поддержания жизни при условии наличия всех питательных веществ. [2] Такие эксперименты привели к выводу, что абсолютно необходимое количество генов для бактерий составляет порядка 250–300. Основные гены одноклеточных организмов кодируют белки, выполняющие три основные функции, включая обработку генетической информации, клеточную оболочку и производство энергии. [1] Эти функции генов используются для поддержания центрального метаболизма , репликации ДНК , перевода генов в белки , поддержания базовой клеточной структуры и опосредования процессов транспорта в клетку и из нее. По сравнению с одноклеточными организмами, многоклеточные имеют больше важных генов, связанных с общением и развитием. Большинство важнейших генов вирусов связаны с обработкой и сохранением генетической информации. В отличие от большинства одноклеточных организмов, вирусам не хватает многих важных генов метаболизма. [1] что заставляет их захватывать метаболизм хозяина. Большинство генов не являются существенными, но обеспечивают селективные преимущества и повышенную приспособленность . Следовательно, подавляющее большинство генов не являются существенными, и многие из них могут быть удалены без последствий, по крайней мере, в большинстве случаев.
Бактерии: полногеномные исследования
[ редактировать ]Для идентификации важных генов на уровне всего генома использовались две основные стратегии: направленная делеция генов и случайный мутагенез с использованием транспозонов . В первом случае аннотированные отдельные гены (или ORF полностью удаляются из генома ) систематически . При транспозон-опосредованном мутагенезе транспозоны случайным образом встраиваются в максимально возможное количество позиций генома с целью нарушить функцию целевых генов (см. рисунок ниже). Инсерционные мутанты, которые все еще способны выживать или расти, предполагают, что транспозон встроен в ген, который не является необходимым для выживания. Местоположение вставок транспозонов можно определить путем гибридизации с микрочипами. [3] или посредством секвенирования транспозонов . С развитием CRISPR эссенциальность генов также была определена посредством ингибирования экспрессии генов посредством интерференции CRISPR . Краткое описание таких экранов показано в таблице. [2] [4]
| Организм | Мутагенез | Метод | Зачитать | ORF | Не-эсс. | Существенный | % Эсс. | Примечания | Ссылка. |
|---|---|---|---|---|---|---|---|---|---|
| Микоплазма гениталиум/пневмония | случайный | Население | Секвенирование | 482 | 130 | 265–350 | 55–73% | --- | [5] |
| Микоплазма гениталиум | случайный | Клоны | Секвенирование | 482 | 100 | 382 | 79% | б, в | [6] |
| Золотистый стафилококк WCUH29 | случайный | Клоны | Секвенирование | 2,600 | н/д | 168 | н/д | б, в | [7] |
| Золотистый стафилококк RN4220 | случайный | Клоны | Секвенирование | 2,892 | н/д | 658 | 23% | --- | [8] |
| Haemophilus influenzae Rd | случайный | Население | Футпринт-ПЦР | 1,657 | 602 | 670 | 40% | --- | [9] |
| Стрептококк пневмонии Rx-1 | Целевой | Клоны | Формирование колонии | 2,043 | 234 | 113 | н/д | с | [10] |
| Стрептококк пневмонии D39 | Целевой | Клоны | Формирование колонии | 2,043 | 560 | 133 | н/д | с | [11] |
| Стрептококк пиогенес 5448 | случайный | Транспозон | Tn-seq | 1,865 | ? | 227 | 12% | --- | [12] |
| Стрептококк пиогенный NZ131 | случайный | Транспозон | Tn-seq | 1,700 | ? | 241 | 14% | --- | [12] |
| Стрептококк крови SK36 | Целевой | Клоны | Формирование колонии | 2,270 | 2,052 | 218 | 10% | а, дж | [1] [13] |
| Микобактерия туберкулеза H37Rv | случайный | Население | Микрочип | 3,989 | 2,567 | 614 | 15% | --- | [14] |
| Микобактерия туберкулеза | случайный | Транспозон | ? | 3,989 | ? | 401 | 10% | --- | [15] |
| Микобактерия туберкулеза H37Rv | случайный | Транспозон | OF-секвенирование | 3,989 | ? | 774 | 19% | --- | [16] [17] |
| Микобактерия туберкулеза H37Rv | случайный | Транспозон | OF-секвенирование | 3,989 | 3,364 | 625 | 16% | привет | [18] |
| Микобактерия туберкулеза | --- | вычислительный | вычислительный | 3,989 | ? | 283 | 7% | --- | [19] |
| Микобактерия туберкулеза H37Rv | Целевой | CRISPRi | OF-секвенирование | 4,052 | 33,15 | 737 | 18% | --- | [20] |
| Бацилла субтилис 168 | Целевой | Клоны | Формирование колонии | 4,105 | 3,830 | 261 | 7% | а, г, г | [21] [22] |
| Эшерихия коли К-12 MG1655 | случайный | Население | Футпринт-ПЦР | 4,308 | 3,126 | 620 | 14% | --- | [23] |
| Эшерихия коли К-12 MG1655 | Целевой | Клоны | Формирование колонии | 4,308 | 2,001 | н/д | н/д | а, е | [24] |
| Эшерихия коли К-12 BW25113 | Целевой | Клоны | Формирование колонии | 4,390 | 3,985 | 303 | 7% | а | [25] |
| Pseudomonas aeruginosa PAO1 | случайный | Клоны | Секвенирование | 5,570 | 4,783 | 678 | 12% | а | [26] |
| Porphyromonas gingivalis | случайный | Транспозон | Секвенирование | 1,990 | 1,527 | 463 | 23% | --- | [27] |
| Синегнойная палочка PA14 | случайный | Клоны | Секвенирование | 5,688 | 4,469 | 335 | 6% | а, е | [28] |
| Сальмонелла тифимуриум | случайный | Клоны | Секвенирование | 4,425 | н/д | 257 | ~11% | б, в | [29] |
| Хеликобактер пилори G27 | случайный | Население | Микрочип | 1,576 | 1,178 | 344 | 22% | --- | [30] |
| Коринебактерия глютамикум | случайный | Население | ? | 3,002 | 2,352 | 650 | 22% | --- | [31] |
| Франциселла Новицида | случайный | Транспозон | ? | 1,719 | 1,327 | 392 | 23% | --- | [32] |
| Микоплазма легочная UAB CTIP | случайный | Транспозон | ? | 782 | 472 | 310 | 40% | --- | [35] |
| Холерный вибрион N16961 | случайный | Транспозон | ? | 3,890 | ? | 779 | 20% | --- | [36] |
| Сальмонелла Тифи | случайный | Транспозон | ? | 4,646 | ? | 353 | 8% | --- | [37] |
| Золотистый стафилококк | случайный | Транспозон | ? | ~2,600 | ? | 351 | 14% | --- | [38] |
| Каулобактер крецентус | случайный | Транспозон | Tn-Seq | 3,876 | 3,240 | 480 | 12.2% | --- | [39] |
| Нейссерийный менингит | случайный | Транспозон | ? | 2,158 | ? | 585 | 27% | --- | [40] |
| Десульфовибрио аляскенсис | случайный | Транспозон | Секвенирование | 3,258 | 2,871 | 387 | 12% | --- | [41] |
Таблица 1. Основные гены бактерий . Мутагенез : целевые мутанты представляют собой делеции генов; случайные мутанты представляют собой вставки транспозонов . Методы : Клоны указывают на делеции одного гена, популяция указывает на мутагенез всей популяции, например, с использованием транспозонов. К важнейшим генам, полученным при популяционном скрининге, относятся гены, необходимые для приспособленности (см. текст). ORF : количество всех открытых рамок считывания в этом геноме. Примечания : (a) имеется коллекция мутантов; (b) метод прямого скрининга существенности (например, с помощью антисмысловой РНК), который не дает информации о несущественных генах. (c) Доступен только частичный набор данных. (d) Включает прогнозируемую существенность генов и сбор данных из опубликованных исследований существенности отдельных генов. (д) Проект в стадии реализации. ( f ) Выведено путем сравнения двух наборов данных об существенности генов, полученных независимо у штаммов P. aeruginosa PA14 и PAO1. (g) Первоначальный результат о 271 существенном гене был исправлен до 261, причем 31 ген, который считался важным, на самом деле оказался несущественным, тогда как с тех пор было описано 20 новых существенных генов. [22] (h) Подсчет генов с важными доменами и тех, которые приводят к дефектам роста при нарушении, как существенных, и тех, которые приводят к преимуществу роста при разрушении, как несущественных. (i) Включалась полностью насыщенная мутантная библиотека из 14 повторов с 84,3% возможных сайтов вставки по крайней мере с одной вставкой транспозона. (j) Каждый существенный ген был независимо подтвержден не менее пяти раз.

На основе полногеномных экспериментальных исследований и анализа системной биологии Kong et al. разработали важную базу данных генов. (2019) для прогнозирования > 4000 видов бактерий. [42]
Эукариоты
[ редактировать ]У Saccharomyces cerevisiae (почкующиеся дрожжи) 15-20% всех генов являются существенными. У Schizosaccharomyces pombe (делящихся дрожжей) было сконструировано 4836 гетерозиготных делеций, охватывающих 98,4% из 4914 открытых рамок считывания, кодирующих белок. 1260 из этих делеций оказались существенными. [43]
Подобные скрининги сложнее провести на других многоклеточных организмах, в том числе на млекопитающих (как модель для человека), по техническим причинам, и их результаты менее однозначны. были разработаны различные методы Однако для нематодного червя C. elegans . [44] фруктовая мушка, [45] и рыбка данио [46] (см. таблицу). Недавнее исследование 900 генов мышей пришло к выводу, что 42% из них являются незаменимыми, хотя выбранные гены не были репрезентативными. [47]
Эксперименты по нокауту генов на людях невозможны или, по крайней мере, неэтичны. Однако естественные мутации привели к выявлению мутаций, которые приводят к ранней эмбриональной или поздней смерти. [48] Обратите внимание, что многие гены у человека не являются абсолютно необходимыми для выживания, но при мутации могут вызывать тяжелые заболевания. Такие мутации занесены в базу данных Online Mendelian Inheritance in Man (OMIM). В ходе компьютерного анализа генетических вариаций и мутаций у 2472 человеческих ортологов известных важных генов мышей Georgi et al. обнаружили сильный, очищающий отбор и сравнительно низкие уровни вариаций последовательностей, что указывает на то, что эти человеческие гены также важны. [49]
Хотя доказать, что тот или иной ген необходим для человека, может оказаться затруднительно, можно продемонстрировать, что ген не является необходимым или даже не вызывает заболевания. Например, секвенирование геномов 2636 граждан Исландии и генотипирование 101584 дополнительных субъектов выявили 8041 человека, у которых был полностью нокаут один ген (т.е. эти люди были гомозиготны по нефункциональному гену). [50] Из 8041 человека с полным нокаутом 6885 были оценены как гомозиготы , а 1249 - как компаунд -гетерозиготы (т.е. у них были нокаутированы оба аллеля гена, но эти два аллеля имели разные мутации). У этих людей в общей сложности 1171 из 19 135 генов человека ( RefSeq ) (6,1%) были полностью нокаутированы. Был сделан вывод, что эти 1171 ген несущественны для человека — по крайней мере, о связанных с ними заболеваниях не сообщалось. [50] Аналогичным образом, последовательности экзома 3222 взрослых британско-пакистанского происхождения с высоким родительским родством выявили 1111 редких вариантов гомозиготных генотипов с прогнозируемой потерей функции гена (LOF = нокаут) в 781 гене. [51] В этом исследовании обнаружено в среднем 140 прогнозируемых генотипов LOF (на каждого субъекта), включая 16 редких ( частота минорных аллелей <1%) гетерозигот, 0,34 редких гомозигот, 83,2 частых гетерозигот и 40,6 частых гомозигот. Почти все редкие гомозиготные генотипы LOF были обнаружены в пределах автозиготных сегментов (94,9%). [51] Несмотря на то, что у большинства из этих людей не было очевидных проблем со здоровьем, связанных с их дефектными генами, вполне возможно, что незначительные проблемы со здоровьем могут быть обнаружены при более детальном обследовании.
Краткое изложение скрининга существенности показано в таблице ниже (в основном на основе базы данных основных генов). [52]
| Организм | Метод | Основные гены | Ссылка. |
| Арабидопсис Талиана | Вставка Т-ДНК | 777 | [53] |
| Caenorhabditis elegans (червь) | РНК-интерференция | 294 | [44] |
| Данио рерио (рыбка данио) | Инсерционный мутагенез | 288 | [46] |
| Drosophila melanogaster (дрозофила) | Инсерционный мутагенез P-элемента | 339 | [45] |
| Homo sapiens (человек) | Поиск литературы | 118 | [48] |
| Homo sapiens (человек) | Экран на базе CRISPR/Cas9 | 1,878 | [54] |
| Homo sapiens (человек) | Экран-ловушка гаплоидных генов | ~2,000 | [55] |
| Homo sapiens (человек) | мышиные ортологи | 2,472 | [56] |
| Mus musculus (мышь) | Поиск литературы | 2114 | [57] |
| Saccharomyces cerevisiae (дрожжи) | Делеции одного гена | 878 | [58] |
| Saccharomyces cerevisiae (дрожжи) | Делеции одного гена | 1,105 | [59] |
| Schizosaccharomyces pombe (дрожжи) | Делеции одного гена | 1,260 | [43] |
Вирусы
[ редактировать ]Вирусы лишены многих генов, необходимых для метаболизма. [1] заставляя их захватывать метаболизм хозяина. Скрининг основных генов был проведен у нескольких вирусов. Например, было обнаружено, что цитомегаловирус человека (ЦМВ) имеет 41 эссенциальную, 88 несущественных и 27 усиливающих ORF (всего 150 ORF). Большинство существенных и дополняющих генов расположены в центральной области, а несущественные гены обычно группируются вблизи концов вирусного генома. [60]
Чарк и Добсон (2015) составили комплексный обзор основных генов вируса осповакцины и назначили роли каждой из 223 ORF штамма Western Reserve (WR) и 207 ORF штамма Copenhagen, оценивая их роль в репликации в клеточной культуре. Согласно их определению, ген считается незаменимым (т.е. играет роль в культуре клеток), если его делеция приводит к снижению титра вируса более чем в 10 раз как на одноступенчатой, так и на многоступенчатой кривой роста. Все гены, участвующие в производстве обернутых вирионов, формировании актинового хвоста и внеклеточном высвобождении вирионов, также считались важными. Гены, влияющие на размер бляшек, но не на репликацию, были определены как несущественные. Согласно этому определению, для репликации вируса коровьей оспы в культуре клеток необходимы 93 гена, тогда как 108 и 94 ORF из WR и Копенгагена соответственно не являются необходимыми. [61] Вирусы коровьей оспы с делециями на обоих концах генома вели себя ожидаемо, демонстрируя лишь легкие дефекты или дефекты диапазона хозяев. Напротив, объединение делеций на обоих концах генома штамма WR VACV вызвало разрушительный дефект роста во всех протестированных клеточных линиях. Это демонстрирует, что делеции одного гена недостаточно для оценки важности генов и что для вируса осповакцины необходимо больше генов, чем первоначально предполагалось. [61]
Один из бактериофагов, прошедших скрининг на наличие важных генов, включает микобактериофаг Джайлса. По крайней мере, 35 из 78 предсказанных генов Джайлза (45%) не являются необходимыми для литического роста. Было обнаружено, что 20 генов являются важными. [62] Основная проблема с фаговыми генами заключается в том, что большинство их генов остаются функционально неизвестными, поэтому их роль трудно оценить. Скрининг фага Salmonella enterica SPN3US выявил 13 основных генов, хотя остается неясным, сколько генов действительно было протестировано. [63]
Количественный анализ эссенциальности генов
[ редактировать ]Теоретически существенные гены являются качественными. [1] Однако в зависимости от окружающей среды некоторые мутанты существенных генов могут проявлять частичные функции, которые можно количественно определить в некоторых исследованиях. Например, делеция определенного гена может снизить скорость роста (или уровень фертильности или других признаков) до 90% от дикого типа. Если существуют изоферменты или альтернативные пути для основных генов, их можно полностью удалить. [1] Используя CRISPR-интерференцию , экспрессию основных генов можно модулировать или «настраивать», что приводит к количественным (или непрерывным) взаимосвязям между уровнем экспрессии генов и величиной стоимости приспособленности, проявляемой данным мутантом. [20]
Синтетическая летальность
[ редактировать ]Два гена являются синтетическими летальными, если ни один из них не является существенным, но когда оба мутированы, двойной мутант является летальным. По оценкам некоторых исследований, количество синтетических летальных генов может составлять порядка 45% всех генов. [64] [65]
Условно эссенциальные гены
[ редактировать ]
Многие гены необходимы только при определенных обстоятельствах. Например, если в клетку поступает аминокислота лизин, любой ген, необходимый для выработки лизина, не является необходимым. Однако когда лизин не поступает, гены, кодирующие ферменты биосинтеза лизина, становятся необходимыми, поскольку без лизина невозможен синтез белка. [4]
Streptococcus pneumoniae , по-видимому, требует 147 генов для роста и выживания в слюне . [66] больше, чем 113-133, которые были обнаружены в предыдущих исследованиях.
Удаление гена может привести к смерти или блокировке клеточного деления . Хотя последний случай может означать «выживание» в течение некоторого времени, без клеточного деления клетка в конечном итоге все равно может умереть. Аналогичным образом, вместо блокировки клеточного деления клетка может иметь замедленный рост или метаболизм в диапазоне от почти необнаружимого до почти нормального. Таким образом, существует градиент от «существенного» до совершенно несущественного, опять же в зависимости от состояния. Некоторые авторы, таким образом, проводят различие между генами, « необходимыми для выживания » и « необходимыми для приспособленности ». [4]
Роль генетического фона . Подобно условиям окружающей среды, генетический фон может определять существенность гена: ген может быть существенным для одного человека, но не для другого, учитывая его или ее генетическую основу. Дупликация генов является одним из возможных объяснений (см. ниже).
Метаболическая зависимость . Гены, участвующие в определенных путях биосинтеза, таких как синтез аминокислот , могут стать несущественными, если одна или несколько аминокислот поставляются из культуральной среды. [1] или другим организмом. [67] Это основная причина, по которой многие паразиты (например, Cryptosporidium hominis ) [68] или эндосимбионтные бактерии потеряли многие гены (например, хламидии ). Такие гены могут быть существенными, но присутствовать только в организме хозяина. Например, Chlamydia trachomatis не может синтезировать пуриновые и пиримидиновые нуклеотиды de novo , поэтому эти бактерии зависят от генов биосинтеза нуклеотидов хозяина. [69]
Другой вид метаболической зависимости, не связанный с межвидовыми взаимодействиями, можно обнаружить, когда бактерии выращиваются в определенных питательных условиях . Например, более 100 генов становятся незаменимыми, когда Escherichia coli выращивается на среде с ограниченным количеством питательных веществ. В частности, изоцитратдегидрогеназа (icd) и цитратсинтаза (gltA) представляют собой два фермента, которые являются частью цикла трикарбоновых кислот (TCA) . Оба гена необходимы в минимальной среде M9 (которая обеспечивает только самые основные питательные вещества). Однако когда в среду добавляют 2-оксоглутарат или глутамат , эти гены перестают быть необходимыми. [70]
Дупликация генов и альтернативные пути метаболизма
[ редактировать ]Многие гены дублируются в геноме, и многие организмы имеют разные метаболические пути (альтернативный метаболический путь). [1] ) для синтеза тех же продуктов. Такие дупликации ( паралоги ) и альтернативные метаболические пути часто делают важные гены несущественными, поскольку дубликат может заменить исходную копию. Например, ген, кодирующий фермент аспартокиназу, важен для E. coli . Напротив, геном Bacillus subtilis содержит три копии этого гена, ни одна из которых сама по себе не является существенной. Однако тройная делеция всех трех генов смертельна. В таких случаях существенность гена или группы паралогов часто можно предсказать на основе существенности одного важного гена у другого вида. У дрожжей лишь немногие важные гены дублируются в геноме: 8,5% несущественных генов, но только 1% незаменимых генов имеют гомолог в дрожжевом геноме. [59]
У червя C. elegans несущественные гены чрезмерно представлены среди дубликатов, возможно, потому, что дупликация существенных генов вызывает сверхэкспрессию этих генов. Вудс и др. обнаружили, что несущественные гены чаще успешно дублируются (фиксируются) и теряются по сравнению с незаменимыми генами. Напротив, важные гены дублируются реже, но при успешной дупликации сохраняются в течение более длительных периодов времени. [71]
Сохранение
[ редактировать ]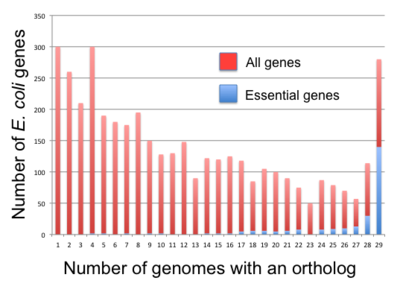
У бактерий незаменимые гены более консервативны, чем несущественные. [73] но корреляция не очень сильная. Например, только 34% основных генов B. subtilis имеют надежные ортологи у всех Bacillota , а 61% основных генов E. coli имеют надежные ортологи у всех гамма-протеобактерий . [72] Фанг и др. (2005) определили персистентные гены как гены, присутствующие более чем в 85% геномов клады. [72] Они обнаружили 475 и 611 таких генов для B. subtilis и E. coli соответственно. Кроме того, они классифицировали гены на пять классов в зависимости от их персистенции и существенности: персистентные гены, эссенциальные гены, персистентные несущественные (PNE) гены (276 у B. subtilis , 409 у E. coli ), эссенциальные непостоянные (ENP) гены (73 у B. subtilis , 33 в E. coli ) и непостоянные несущественные (NPNE) гены (3558 в B. subtilis , 3525 в E. coli ). Фанг и др. обнаружили 257 персистентных генов, которые существуют как у B. subtilis (для Bacillota), так и для E. coli (для гамма-протеобактерий). Среди них 144 (соответственно 139) были ранее идентифицированы как незаменимые у B. subtilis (соответственно E. coli ) и 25 (соответственно 18) из 257 генов отсутствуют в 475 B. subtilis (соответственно 611 E. coli). персистирующих генах гены. Все остальные члены пула являются генами PNE. [72]
У эукариот 83% однозначных ортологов между Schizosaccharomyces pombe и Saccharomyces cerevisiae сохранили эссенциальность, то есть они несущественны у обоих видов или незаменимы у обоих видов. Остальные 17% генов несущественны у одного вида и незаменимы у другого. [74] Это весьма примечательно, учитывая, что S. pombe отделен от S. cerevisiae примерно 400 миллионами лет эволюции. [75]
В целом высококонсервативные и, следовательно, более старые гены (т.е. гены более раннего филогенетического происхождения) с большей вероятностью будут иметь важное значение, чем более молодые гены, даже если они были дублированы. [76]
Изучать
[ редактировать ]Экспериментальное изучение незаменимых генов ограничено тем фактом, что инактивация незаменимого гена по определению летальна для организма. Следовательно, их нельзя просто удалить или мутировать для анализа полученных фенотипов (распространенный метод в генетике ).
Однако существуют обстоятельства, при которых важными генами можно манипулировать. В диплоидных организмах может быть необходима только одна функциональная копия некоторых важных генов ( гаплодостаточность ), при этом гетерозигота демонстрирует поучительный фенотип. Некоторые важные гены могут переносить вредные, но не полностью смертельные мутации, поскольку они не отменяют полностью функцию гена.
Компьютерный анализ может выявить многие свойства белков без их экспериментального анализа, например, путем изучения гомологичных белков , функций, структуры и т. д. (см. также ниже «Прогнозирование основных генов »). Продукты основных генов также можно изучать при экспрессии в других организмах или при очистке и изучении in vitro .
Условно незаменимые гены легче изучать. Были идентифицированы термочувствительные варианты основных генов, которые кодируют продукты, которые теряют функцию при высоких температурах и поэтому проявляют фенотип только при повышенной температуре. [77]
Воспроизводимость
[ редактировать ]Если скрининг основных генов повторяется в независимых лабораториях, в результате часто получаются разные списки генов. Например, скрининг E. coli выявил от ~300 до ~600 основных генов (см. Таблицу 1 ). Такие различия становятся еще более выраженными при использовании разных бактериальных штаммов (см. рисунок 2 ). Распространенное объяснение состоит в том, что условия эксперимента различны или что природа мутации может быть различной (например, полная делеция гена или мутант транспозона). [4] В частности, скрининг транспозонов трудно воспроизвести, поскольку транспозон может вставляться во многие позиции гена. Вставки к 3'-концу существенного гена могут не иметь летального фенотипа (или вообще не иметь фенотипа) и, следовательно, не могут быть распознаны как таковые. Это может привести к ошибочным аннотациям (здесь: ложноотрицательным). [78]
Сравнение CRISPR /cas9 и RNAi скринингов . Скрининг для идентификации основных генов в клеточной линии хронического миелогенного лейкоза человека K562 с помощью этих двух методов показал лишь ограниченное перекрытие. При уровне ложноположительных результатов 10% при скрининге Cas9 было идентифицировано ~ 4500 генов по сравнению с ~ 3100 при скрининге shRNA , при этом в обоих случаях было идентифицировано только ~ 1200 генов. [79]
Различные важные гены у разных организмов
[ редактировать ]Разные организмы могут иметь разные важные гены. Например, Bacillus subtilis имеет 271 незаменимый ген. [21] Около половины (150) ортологичных генов E. coli также являются существенными. Еще 67 генов, которые необходимы для E. coli, не являются существенными для B. subtilis , в то время как 86 существенных генов E. coli не имеют B. subtilis . ортолога [25] У Mycoplasmagentium по крайней мере 18 генов являются существенными, но не являются незаменимыми у M. bovis. [80] Многие из этих различных важных генов вызваны паралогами или альтернативными метаболическими путями. [1]
Такие различные важные гены в бактериях могут быть использованы для разработки таргетной антибактериальной терапии против определенных специфических патогенов для снижения устойчивости к антибиотикам в эпоху микробиома. [81] Stone и соавт. (2015) использовали разницу в основных генах бактерий для разработки селективных препаратов против орального возбудителя Porphyromonas gingivalis , а не полезных бактерий Streptococcus sanguis . [82]
Прогноз
[ редактировать ]Основные гены можно предсказать с помощью вычислений. Однако большинство методов в той или иной степени используют экспериментальные данные («обучающие наборы»). Чен и др. [83] определил четыре критерия выбора обучающих наборов для таких прогнозов: (1) основные гены в выбранном обучающем наборе должны быть надежными; (2) условия роста, в которых определяются основные гены, должны быть согласованными в наборах обучения и прогнозирования; (3) виды, используемые в качестве обучающей выборки, должны быть тесно связаны с целевым организмом; и (4) организмы, используемые в качестве наборов для обучения и прогнозирования, должны демонстрировать схожие фенотипы или образ жизни. Они также обнаружили, что размер обучающего набора должен составлять не менее 10% от общего числа генов, чтобы давать точные прогнозы. Некоторые подходы к прогнозированию основных генов:
Сравнительная геномика . Вскоре после того, как стали доступны первые геномы ( Hamophilus influenzae и Mycoplasmagentium ), Mushegian et al. [84] попытались предсказать количество необходимых генов на основе общих генов у этих двух видов. Было высказано предположение, что на длительном эволюционном расстоянии, разделяющем две бактерии, должны сохраняться только важные гены. В ходе этого исследования было выявлено около 250 кандидатов на незаменимые гены. [84] По мере того, как становилось доступно все больше геномов, число предсказанных основных генов продолжало сокращаться, поскольку все больше геномов имели все меньше и меньше общих генов. В результате был сделан вывод, что универсальное консервативное ядро состоит менее чем из 40 генов. [85] [86] Однако этот набор консервативных генов не идентичен набору основных генов, поскольку разные виды полагаются на разные существенные гены.
Аналогичный подход использовался для вывода основных генов из пангенома видов бруцелл . 42 полных генома бруцелл и в общей сложности 132 143 гена, кодирующих белки, были использованы для предсказания 1252 потенциально важных генов, полученных из основного генома путем сравнения с базой данных основных генов прокариот. [87]
Сетевой анализ . первые сети взаимодействия белков дрожжей , После того, как были опубликованы [88] было обнаружено, что белки с высокой степенью связи (например, посредством белок-белковых взаимодействий ) с большей вероятностью будут иметь важное значение. [89] Однако белки с высокой степенью связи могут быть экспериментальными артефактами, а высокая связь может скорее представлять собой плейотропию, а не эссенциальность. [90] Тем не менее, сетевые методы были улучшены за счет добавления других критериев и, следовательно, имеют некоторую ценность для прогнозирования важных генов. [91]
Машинное обучение . Хуа и др. использовали машинное обучение для прогнозирования основных генов у 25 видов бактерий . [92]
Индекс Херста . Лю и др. (2015) [93] использовал показатель Херста , характерный параметр для описания дальней корреляции в ДНК для предсказания важных генов. В 31 из 33 бактериальных геномов уровни значимости показателей Херста существенных генов были достоверно выше, чем для соответствующего полного набора генов, тогда как уровни значимости показателей Херста несущественных генов остались неизменными или незначительно увеличились.
Минимальные геномы . Считалось также, что существенные гены можно вывести из минимальных геномов , которые предположительно содержат только существенные гены. Проблема здесь в том, что самые маленькие геномы принадлежат паразитическим (или симбионтным) видам, которые могут выжить с уменьшенным набором генов, поскольку получают много питательных веществ от своих хозяев. Например, одним из самых маленьких геномов является геном Hodgkinia cicadicola , симбионта цикад, содержащий всего 144 КБ ДНК, кодирующей только 188 генов. [94] Как и другие симбионты, ходжкиния получает многие питательные вещества от хозяина, поэтому ее гены не обязательно должны быть важными.
Метаболическое моделирование . Незаменимые гены также можно предсказать в полностью секвенированных геномах путем метаболической реконструкции , то есть путем реконструкции полного метаболизма на основе содержания генов и затем идентификации тех генов и путей, которые оказались важными у других видов. Однако этот метод может быть скомпрометирован белками с неизвестной функцией. Кроме того, у многих организмов есть резервные или альтернативные пути, которые необходимо учитывать (см. рисунок 1). Метаболическое моделирование также использовалось Basler (2015) для разработки метода прогнозирования основных метаболических генов. [95] Анализ баланса потоков , метод метаболического моделирования, недавно был использован для прогнозирования основных генов в метаболизме светлоклеточной почечно-клеточной карциномы. [96]
Гены неизвестной функции . Удивительно, но значительное количество важных генов не имеет известной функции. Например, среди 385 основных кандидатов в M. Genitalium ни одна функция не может быть приписана 95 генам. [6] хотя к 2011 году это число сократилось до 75. [86] Большинство неизвестных функционально важных генов обладают потенциальными биологическими функциями, связанными с одной из трех фундаментальных функций. [1]
ЗУПЛС . Сонг и др. представили новый метод прогнозирования важных генов, который использует только Z-кривую и другие функции, основанные на последовательностях. [97] Такие особенности можно легко рассчитать на основе последовательностей ДНК/аминокислот. Однако надежность этого метода остается немного неясной.
Основные серверы предсказания генов . Го и др. (2015) разработали три онлайн-сервиса для прогнозирования основных генов в бактериальных геномах. Эти свободно доступные инструменты применимы для отдельных последовательностей генов без аннотированных функций, отдельных генов с определенными названиями и полных геномов бактериальных штаммов. [98] Конг и др. (2019) разработали базу данных ePath , которую можно использовать для поиска > 4000 видов бактерий для прогнозирования важных генов. [42]
Основные белковые домены
[ редактировать ]Хотя большинство незаменимых генов кодируют белки, многие незаменимые белки состоят из одного домена. Этот факт был использован для идентификации основных белковых доменов. Гудакр и др. идентифицировали сотни существенных доменов неизвестной функции (eDUF). [99] Лу и др. [100] представили аналогичный подход и определили 3450 доменов, которые необходимы по крайней мере для одного вида микробов.
См. также
[ редактировать ]- Незаменимая аминокислота
- Незаменимые белки в белковых комплексах
- Ген
- Геном
- Минимальный геном
- Мутация
Ссылки
[ редактировать ]- ^ Jump up to: а б с д и ж г час я дж к Сюй, Пин; Гэ, Сючунь; Чен, Лей; Ван, Сяоцзин; Доу, Юэтан; Сюй, Джерри З.; Патель, Дженишкумар Р.; Стоун, Виктория; Трин, Мой; Эванс, Карра; Котенок, Тодд (декабрь 2011 г.). «Полногеномная идентификация основных генов Streptococcus sanguinis» . Научные отчеты . 1 (1): 125. Бибкод : 2011NatSR...1E.125X . дои : 10.1038/srep00125 . ISSN 2045-2322 . ПМК 3216606 . ПМИД 22355642 .
- ^ Jump up to: а б Чжан Р., Линь Ю (январь 2009 г.). «DEG 5.0, база данных основных генов как прокариот, так и эукариотов» . Исследования нуклеиновых кислот . 37 (Проблема с базой данных): D455-8. дои : 10.1093/нар/gkn858 . ПМК 2686491 . ПМИД 18974178 .
- ^ Сассетти CM, Бойд Д.Х., Рубин Э.Дж. (2003). «Гены, необходимые для роста микобактерий, определенные с помощью мутагенеза высокой плотности» . Мол Микробиол . 48 (1): 77–84. дои : 10.1046/j.1365-2958.2003.03425.x . ПМИД 12657046 .
- ^ Jump up to: а б с д Гердес С., Эдвардс Р., Кубал М., Фонстейн М., Стивенс Р., Остерман А. (октябрь 2006 г.). «Основные гены на метаболических картах». Современное мнение в области биотехнологии . 17 (5): 448–56. дои : 10.1016/j.copbio.2006.08.006 . ПМИД 16978855 .
- ^ Хатчисон К.А., Петерсон С.Н., Гилл С.Р., Клайн Р.Т., Уайт О., Фрейзер К.М., Смит Х.О., Вентер Дж.К. (декабрь 1999 г.). «Глобальный мутагенез транспозонов и минимальный геном микоплазмы». Наука . 286 (5447): 2165–9. дои : 10.1126/science.286.5447.2165 . ПМИД 10591650 . S2CID 235447 .
- ^ Jump up to: а б Гласс Дж.И., Асад-Гарсия Н., Альперович Н., Юзеф С., Льюис М.Р., Маруф М., Хатчисон К.А., Смит Х.О., Вентер Дж.К. (январь 2006 г.). «Основные гены минимальной бактерии» . Труды Национальной академии наук Соединенных Штатов Америки . 103 (2): 425–30. Бибкод : 2006PNAS..103..425G . дои : 10.1073/pnas.0510013103 . ПМЦ 1324956 . ПМИД 16407165 .
- ^ Цзи Й, Чжан Б., Ван С.Ф., Уоррен П., Вуднатт Дж., Бернхэм М.К., Розенберг М. (сентябрь 2001 г.). «Идентификация критических стафилококковых генов с использованием условных фенотипов, генерируемых антисмысловой РНК». Наука . 293 (5538): 2266–9. Бибкод : 2001Sci...293.2266J . дои : 10.1126/science.1063566 . ПМИД 11567142 . S2CID 24126939 .
- ^ Форсайт Р.А., Хазельбек Р.Дж., Олсен К.Л., Ямамото Р.Т., Сюй Х, Трэвик Дж.Д., Уолл Д., Ван Л., Браун-Драйвер В., Фролих Дж.М., С.К.Г., Кинг П., Маккарти М., Мэлоун С., Мизинер Б., Роббинс Д., Тан З, Чжу Зи ЗЮ, Карр Г, Моска Д.А., Замудио С, Фоулкс Дж.Г., Зискинд Дж.В. (март 2002 г.). «Полногеномная стратегия идентификации основных генов золотистого стафилококка» . Молекулярная микробиология . 43 (6): 1387–400. дои : 10.1046/j.1365-2958.2002.02832.x . ПМИД 11952893 .
- ^ Акерли Б.Дж., Рубин Э.Дж., Новик В.Л., Амайя К., Джадсон Н., Мекаланос Дж.Дж. (январь 2002 г.). «Анализ в масштабе генома для идентификации генов, необходимых для роста или выживания Haemophilus influenzae» . Труды Национальной академии наук Соединенных Штатов Америки . 99 (2): 966–71. Бибкод : 2002PNAS...99..966A . дои : 10.1073/pnas.012602299 . ПМК 117414 . ПМИД 11805338 .
- ^ Танасси Дж.А., Хартман-Нойман С.Л., Догерти Т.Дж., Догерти Б.А., Пуччи М.Дж. (июль 2002 г.). «Идентификация 113 консервативных основных генов с использованием высокопроизводительной системы разрушения генов у Streptococcus pneumoniae» . Исследования нуклеиновых кислот . 30 (14): 3152–62. дои : 10.1093/nar/gkf418 . ПМЦ 135739 . ПМИД 12136097 .
- ^ Сон JH, Ко KS, Ли JY, Пэк JY, О WS, Юн HS, Чон JY, Чун Дж (июнь 2005 г.). «Идентификация основных генов Streptococcus pneumoniae методом аллельно-заместительного мутагенеза» . Молекулы и клетки . 19 (3): 365–74. дои : 10.1016/S1016-8478(23)13181-5 . ПМИД 15995353 .
- ^ Jump up to: а б Ле Бретон И., Белью А.Т., Вальдес К.М., Ислам Э., Карри П., Теттлин Х., Ширтлифф М.Е., Эль-Сайед Н.М., Макивер К.С. (май 2015 г.). «Основные гены в основном геноме человеческого патогена Streptococcus pyogenes» . Научные отчеты . 5 : 9838. Бибкод : 2015NatSR...5E9838L . дои : 10.1038/srep09838 . ПМК 4440532 . ПМИД 25996237 .
- ^ Чен Л, Ге Икс, Сюй П (2015). «Идентификация основных генов Streptococcus sanguinis с использованием полногеномной делеционной мутации». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 15–23. дои : 10.1007/978-1-4939-2398-4_2 . ISBN 978-1-4939-2397-7 . ПМЦ 4819415 . ПМИД 25636610 .
- ^ Сассетти CM, Бойд Д.Х., Рубин Э.Дж. (октябрь 2001 г.). «Комплексная идентификация условно-эссенциальных генов микобактерий» . Труды Национальной академии наук Соединенных Штатов Америки . 98 (22): 12712–7. Бибкод : 2001PNAS...9812712S . дои : 10.1073/pnas.231275498 . ПМК 60119 . ПМИД 11606763 .
- ^ Ламиххан Г., Фрейндлих Дж. С., Экинс С., Викрамаратне Н., Нолан С. Т., Бишай В. Р. (февраль 2011 г.). «Незаменимые метаболиты микобактерий туберкулеза и их мимики» . мБио . 2 (1): e00301-10. дои : 10.1128/mBio.00301-10 . ПМК 3031304 . ПМИД 21285434 .
- ^ Jump up to: а б Гриффин Дж. Э., Гавронски Дж. Д., Деджесус М. А., Йоргер Т. Р., Акерли Б. Дж., Сассетти CM (сентябрь 2011 г.). «Фенотипическое профилирование с высоким разрешением определяет гены, необходимые для роста микобактерий и катаболизма холестерина» . ПЛОС Патогены . 7 (9): e1002251. дои : 10.1371/journal.ppat.1002251 . ПМК 3182942 . ПМИД 21980284 .
- ^ Лонг Дж.Э., ДеДжесус М., Уорд Д., Бейкер Р.Э., Йоргер Т., Сассетти СМ (2015). «Идентификация основных генов микобактерий туберкулеза с помощью глобального фенотипического профилирования». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 79–95. дои : 10.1007/978-1-4939-2398-4_6 . ISBN 978-1-4939-2397-7 . ПМИД 25636614 .
- ^ ДеДжесус М.А., Геррик Э.Р., Сюй В., Парк С.В., Лонг Дж.Э., Бутте CC, Рубин Э.Дж., Шнаппингер Д., Эрт С., Форчун С.М., Сассетти СМ, Йоргер Т.Р. (январь 2017 г.). «Комплексный анализ эссенциальности генома микобактерии туберкулеза посредством мутагенеза насыщающих транспозонов» . мБио . 8 (1): e02133–16. дои : 10.1128/mBio.02133-16 . ПМК 5241402 . ПМИД 28096490 .
- ^ Гош С., Балони П., Мукерджи С., Ананд П., Чандра Н. (декабрь 2013 г.). «Многоуровневый многомасштабный подход к изучению основных генов микобактерии туберкулеза» . Системная биология BMC . 7 : 132. дои : 10.1186/1752-0509-7-132 . ПМЦ 4234997 . ПМИД 24308365 .
- ^ Jump up to: а б Бош, Б, ДеДжесус, М.А., Поултон, Северная Каролина, Чжан, В, Энгельхарт, К.А., Завери, А, Лавалетт, С, Рюкер, Н, Трухильо, К, Уоллах, Дж.Б., Ли, С, Эрт, С, Чейт, Б.Т., Шнаппингер, С., Рок, Дж.М. (июль 2021 г.). «Настройка экспрессии генов в масштабе всего генома выявляет разнообразные уязвимости M. Tuberculosis» . Клетка . 184 (17): 4579–4592.e24. дои : 10.1016/j.cell.2021.06.033 . ПМЦ 8382161 . ПМИД 34297925 .
- ^ Jump up to: а б Кобаяши К., Эрлих С.Д., Альбертини А., Амати Г., Андерсен К.К., Арно М. и др. (апрель 2003 г.). «Основные гены Bacillus subtilis» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (8): 4678–83. Бибкод : 2003PNAS..100.4678K . дои : 10.1073/pnas.0730515100 . ПМК 153615 . ПМИД 12682299 .
- ^ Jump up to: а б Коммишау FM, Питак Н., Штюльке Дж (июнь 2013 г.). «Основные гены Bacillus subtilis: переоценка через десять лет» (PDF) . Молекулярные биосистемы . 9 (6): 1068–75. дои : 10.1039/c3mb25595f . ПМИД 23420519 . S2CID 23769853 .
- ^ Гердес С.Ю., Шолле, доктор медицинских наук, Кэмпбелл Дж.В., Балажи Г., Равас Э., Догерти, доктор медицинских наук и др. (октябрь 2003 г.). «Экспериментальное определение и анализ на системном уровне основных генов Escherichia coli MG1655» . Журнал бактериологии . 185 (19): 5673–84. дои : 10.1128/JB.185.19.5673-5684.2003 . ЧВК 193955 . ПМИД 13129938 .
- ^ Кан Ю., Дерфи Т., Гласнер Дж. Д., Цю Ю., Фриш Д., Винтерберг К. М. и др. (август 2004 г.). «Систематический мутагенез генома Escherichia coli» . Журнал бактериологии . 186 (15): 4921–30. дои : 10.1128/JB.186.15.4921-4930.2004 . ПМК 451658 . ПМИД 15262929 .
- ^ Jump up to: а б Баба Т., Ара Т., Хасегава М., Такай Ю., Окумура Ю., Баба М. и др. (2006). «Создание мутантов Escherichia coli K-12 с нокаутом одного гена в рамке: коллекция Кейо» . Молекулярная системная биология . 2 : 2006.0008. дои : 10.1038/msb4100050 . ПМК 1681482 . ПМИД 16738554 .
- ^ Джейкобс М.А., Алвуд А., Тайписуттикул И., Спенсер Д., Хауген Э., Эрнст С. и др. (ноябрь 2003 г.). «Комплексная библиотека транспозонных мутантов Pseudomonas aeruginosa» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (24): 14339–44. Бибкод : 2003PNAS..10014339J . дои : 10.1073/pnas.2036282100 . ПМЦ 283593 . ПМИД 14617778 .
- ^ Хатчерсон Дж.А., Гогенени Х., Йодер-Хаймс Д., Хендриксон Э.Л., Хакетт М., Уайтли М. и др. (август 2016 г.). «Сравнение существенно важных генов Porphyromonas gingivalis, выявленных в двух библиотеках секвенирования транспозонов» . Молекулярная оральная микробиология . 31 (4): 354–64. дои : 10.1111/omi.12135 . ПМЦ 4788587 . ПМИД 26358096 .
- ^ Либерати Н.Т., Урбах Дж.М., Мията С., Ли Д.Г., Дренкард Э., Ву Г. и др. (февраль 2006 г.). «Упорядоченная, неизбыточная библиотека мутантов вставки транспозона штамма Pseudomonas aeruginosa PA14» . Труды Национальной академии наук Соединенных Штатов Америки . 103 (8): 2833–8. Бибкод : 2006PNAS..103.2833L . дои : 10.1073/pnas.0511100103 . ПМЦ 1413827 . ПМИД 16477005 .
- ^ Кнут К., Нисалла Х., Хьюк С.Дж., Фукс Т.М. (март 2004 г.). «Крупномасштабная идентификация основных генов сальмонеллы путем улавливания летальных вставок» . Молекулярная микробиология . 51 (6): 1729–44. дои : 10.1046/j.1365-2958.2003.03944.x . ПМИД 15009898 . S2CID 45267476 .
- ^ Салама Н.Р., Шеперд Б., Фалькоу С. (декабрь 2004 г.). «Глобальный мутагенез транспозонов и анализ основных генов Helicobacter pylori» . Журнал бактериологии . 186 (23): 7926–35. дои : 10.1128/JB.186.23.7926-7935.2004 . ПМК 529078 . ПМИД 15547264 .
- ^ Сузуки Н., Инуи М., Юкава Х. (2011). «Высокопроизводительный транспозонный мутагенез Corynebacteriumlutamicum». Штаммовая инженерия . Методы молекулярной биологии. Том. 765. стр. 409–17. дои : 10.1007/978-1-61779-197-0_24 . ISBN 978-1-61779-196-3 . ПМИД 21815106 .
- ^ Галлахер Л.А., Рэймидж Э., Джейкобс М.А., Каул Р., Бриттнахер М., Маноил С. (январь 2007 г.). «Полная библиотека транспозонных мутантов Francisco novicida, суррогата биологического оружия» . Труды Национальной академии наук Соединенных Штатов Америки . 104 (3): 1009–14. Бибкод : 2007PNAS..104.1009G . дои : 10.1073/pnas.0606713104 . ПМЦ 1783355 . ПМИД 17215359 .
- ^ Шталь М., Стинци А. (июнь 2011 г.). «Идентификация основных генов в геноме C. jejuni выявляет области гипервариабельной пластичности». Функциональная и интегративная геномика . 11 (2): 241–57. дои : 10.1007/s10142-011-0214-7 . ПМИД 21344305 . S2CID 24054117 .
- ^ Шталь М., Стинци А. (2015). «Отслеживание транспозонов на микрочипах для картирования условно незаменимых генов у Campylobacter jejuni». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 1–14. дои : 10.1007/978-1-4939-2398-4_1 . ISBN 978-1-4939-2397-7 . ПМИД 25636609 .
- ^ Френч КТ, Лао П., Лорейн А.Е., Мэтьюз Б.Т., Ю Х., Дибвиг К. (июль 2008 г.). «Крупномасштабный транспозонный мутагенез Mycoplasma pulmonis» . Молекулярная микробиология . 69 (1): 67–76. дои : 10.1111/j.1365-2958.2008.06262.x . ПМЦ 2453687 . ПМИД 18452587 .
- ^ Кэмерон Д.Э., Урбах Дж.М., Мекаланос Дж.Дж. (июнь 2008 г.). «Определенная библиотека мутантов транспозонов и ее использование для идентификации генов подвижности у холерного вибриона» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (25): 8736–41. Бибкод : 2008PNAS..105.8736C . дои : 10.1073/pnas.0803281105 . ПМЦ 2438431 . ПМИД 18574146 .
- ^ Лэнгридж Г.К., Фан М.Д., Тернер Д.Д., Перкинс Т.Т., Партс Л., Хаасе Дж. и др. (декабрь 2009 г.). «Одновременный анализ каждого гена Salmonella Typhi с использованием миллиона мутантов транспозонов» . Геномные исследования . 19 (12): 2308–16. дои : 10.1101/гр.097097.109 . ПМК 2792183 . ПМИД 19826075 .
- ^ Чаудхури Р.Р., Аллен А.Г., Оуэн П.Дж., Шалом Дж., Стоун К., Харрисон М. и др. (июль 2009 г.). «Комплексная идентификация основных генов Staphylococcus aureus с использованием транспозонов-опосредованной дифференциальной гибридизации (TMDH)» . БМК Геномика . 10 : 291. дои : 10.1186/1471-2164-10-291 . ПМК 2721850 . ПМИД 19570206 .
- ^ Кристен Б., Абелюк Э., Коллиер Дж.М., Калогераки В.С., Пассарелли Б., Коллер Дж.А., Феро М.Дж., МакАдамс Х.Х., Шапиро Л. (август 2011 г.). «Основной геном бактерии» . Молекулярная системная биология . 7 : 528. дои : 10.1038/msb.2011.58 . ПМК 3202797 . ПМИД 21878915 .
- ^ Мендум Т.А., Ньюкомб Дж., Маннан А.А., Киржек А.М., Макфадден Дж. (декабрь 2011 г.). «Опрос данных глобального мутагенеза с помощью масштабной модели генома Neisseria meningitidis для оценки пригодности генов in vitro и в сыворотке» . Геномная биология . 12 (12): 127 р. дои : 10.1186/gb-2011-12-12-r127 . ПМЦ 3334622 . ПМИД 22208880 .
- ^ Куэль Дж.В., Прайс М.Н., Рэй Дж., Ветмор К.М., Эскивель З., Казаков А.Е. и др. (май 2014 г.). «Функциональная геномика с обширной библиотекой мутантов транспозонов сульфатредуцирующей бактерии Desulfovibrio alaskensis G20» . мБио . 5 (3): e01041-14. дои : 10.1128/mBio.01041-14 . ПМК 4045070 . ПМИД 24865553 .
- ^ Jump up to: а б Конг, Сянчжэнь; Чжу, Бин; Стоун, Виктория Н.; Гэ, Сючунь; Эль-Рами, Фади Э.; Дунхай, Хуанфу; Сюй, Пин (декабрь 2019 г.). «ePath: онлайн-база данных для всесторонней аннотации основных генов прокариот» . Научные отчеты . 9 (1): 12949. Бибкод : 2019НатСР...912949К . дои : 10.1038/s41598-019-49098-w . ISSN 2045-2322 . ПМК 6737131 . ПМИД 31506471 .
- ^ Jump up to: а б Ким Д.У., Хейлс Дж., Ким Д., Вуд В., Пак Х.О., Вон М. и др. (июнь 2010 г.). «Анализ полногеномного набора делеций генов у делящихся дрожжей Schizosaccharomyces pombe» . Природная биотехнология . 28 (6): 617–623. дои : 10.1038/nbt.1628 . ПМЦ 3962850 . ПМИД 20473289 .
- ^ Jump up to: а б Камат Р.С., Фрейзер А.Г., Донг Й., Пулин Г., Дурбин Р., Готта М. и др. (январь 2003 г.). «Систематический функциональный анализ генома Caenorhabditis elegans с использованием РНКи». Природа . 421 (6920): 231–7. Бибкод : 2003Natur.421..231K . дои : 10.1038/nature01278 . hdl : 10261/63159 . ПМИД 12529635 . S2CID 15745225 .
- ^ Jump up to: а б Спрэдлинг AC, Стерн Д., Битон А., Рем Э.Дж., Лаверти Т., Мозден Н. и др. (сентябрь 1999 г.). «Проект Беркли по разрушению генов дрозофилы: вставки отдельных P-элементов мутируют 25% жизненно важных генов дрозофилы» . Генетика . 153 (1): 135–77. дои : 10.1093/генетика/153.1.135 . ПМК 1460730 . ПМИД 10471706 .
- ^ Jump up to: а б Амстердам А., Ниссен Р.М., Сан З., Суинделл Э.К., Фаррингтон С., Хопкинс Н. (август 2004 г.). «Идентификация 315 генов, необходимых для раннего развития рыбок данио» . Труды Национальной академии наук Соединенных Штатов Америки . 101 (35): 12792–7. Бибкод : 2004PNAS..10112792A . дои : 10.1073/pnas.0403929101 . ПМК 516474 . ПМИД 15256591 .
- ^ Уайт Дж.К., Гердин А.К., Карп Н.А., Райдер Э., Бульян М., Басселл Дж.Н. и др. (июль 2013 г.). «Полногеномная генерация и систематическое фенотипирование нокаутных мышей открывают новую роль многих генов» . Клетка . 154 (2): 452–64. дои : 10.1016/j.cell.2013.06.022 . ПМЦ 3717207 . ПМИД 23870131 .
- ^ Jump up to: а б Ляо БАЙ, Чжан Дж (май 2008 г.). «Нулевые мутации у ортологов человека и мыши часто приводят к различным фенотипам» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (19): 6987–92. Бибкод : 2008PNAS..105.6987L . дои : 10.1073/pnas.0800387105 . ПМЦ 2383943 . ПМИД 18458337 .
- ^ Георгий Б., Войт Б.Ф., Бьюкан М. (май 2013 г.). Флинт Дж. (ред.). «От мыши к человеку: эволюционный геномный анализ человеческих ортологов основных генов» . ПЛОС Генетика . 9 (5): e1003484. дои : 10.1371/journal.pgen.1003484 . ПМЦ 3649967 . ПМИД 23675308 .
- ^ Jump up to: а б Сулем П., Хельгасон Х., Оддсон А., Стефанссон Х., Гудьонссон С.А., Цинк Ф. и др. (май 2015 г.). «Идентификация большого набора редких полных нокаутов человека». Природная генетика . 47 (5): 448–52. дои : 10.1038/ng.3243 . ПМИД 25807282 . S2CID 205349719 .
- ^ Jump up to: а б Нарасимхан В.М., Хант К.А., Мейсон Д., Бейкер К.Л., Карчевски К.Дж., Барнс М.Р. и др. (апрель 2016 г.). «Влияние редких нокаутов генов на здоровье и популяцию у взрослых людей, имеющих родственных родителей» . Наука . 352 (6284): 474–7. Бибкод : 2016Sci...352..474N . дои : 10.1126/science.aac8624 . ПМЦ 4985238 . ПМИД 26940866 .
- ^ Луо Х, Линь Ю, Гао Ф, Чжан ЧТ, Чжан Р (январь 2014 г.). «DEG 10, обновление базы данных основных генов, включающее как гены, кодирующие белки, так и некодирующие геномные элементы» . Исследования нуклеиновых кислот . 42 (Проблема с базой данных): D574-80. дои : 10.1093/nar/gkt1131 . ПМЦ 3965060 . ПМИД 24243843 .
- ^ Цафрир И., Пена-Муралла Р., Дикерман А., Берг М., Роджерс Р., Хатченс С. и др. (июль 2004 г.). «Идентификация генов, необходимых для развития эмбрионов арабидопсиса» . Физиология растений . 135 (3): 1206–20. дои : 10.1104/стр.104.045179 . ПМК 519041 . ПМИД 15266054 .
- ^ Ван Т., Бирсой К., Хьюз Н.В., Крупчак К.М., Пост Ю., Вэй Дж.Дж. и др. (ноябрь 2015 г.). «Идентификация и характеристика важнейших генов в геноме человека» . Наука . 350 (6264): 1096–101. Бибкод : 2015Sci...350.1096W . doi : 10.1126/science.aac7041 . ПМЦ 4662922 . ПМИД 26472758 .
- ^ Бломен В.А., Маек П., Джае Л.Т., Бигензан Дж.В., Ньювенхейс Дж., Старинг Дж. и др. (ноябрь 2015 г.). «Эссенциальность гена и синтетическая летальность в гаплоидных клетках человека». Наука . 350 (6264): 1092–6. Бибкод : 2015Sci...350.1092B . doi : 10.1126/science.aac7557 . ПМИД 26472760 . S2CID 26529733 .
- ^ Георгий Б., Войт Б.Ф., Бьюкан М. (май 2013 г.). «От мыши к человеку: эволюционный геномный анализ человеческих ортологов основных генов» . ПЛОС Генетика . 9 (5): e1003484. дои : 10.1371/journal.pgen.1003484 . ПМЦ 3649967 . ПМИД 23675308 .
- ^ Ляо БАЙ, Чжан Дж (август 2007 г.). «Дубликаты генов мыши так же важны, как и одиночные гены». Тенденции в генетике . 23 (8): 378–81. дои : 10.1016/j.tig.2007.05.006 . ПМИД 17559966 .
- ^ Мьюс Х.В., Фришман Д., Гюльденер У., Маннхаупт Г., Майер К., Мокрейс М. и др. (январь 2002 г.). «MIPS: база данных геномов и белковых последовательностей» . Исследования нуклеиновых кислот . 30 (1): 31–4. дои : 10.1093/нар/30.1.31 . ПМК 99165 . ПМИД 11752246 .
- ^ Jump up to: а б Гиавер Г., Чу А.М., Ни Л., Коннелли С., Райлз Л., Веронно С. и др. (июль 2002 г.). «Функциональное профилирование генома Saccharomyces cerevisiae». Природа . 418 (6896): 387–91. Бибкод : 2002Natur.418..387G . дои : 10.1038/nature00935 . ПМИД 12140549 . S2CID 4400400 .
- ^ Ю Д, Сильва MC, Шенк Т (октябрь 2003 г.). «Функциональная карта цитомегаловируса человека AD169, определенная с помощью глобального мутационного анализа» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (21): 12396–401. Бибкод : 2003PNAS..10012396Y . дои : 10.1073/pnas.1635160100 . ПМК 218769 . ПМИД 14519856 .
- ^ Jump up to: а б Добсон Б.М., Чарк, округ Колумбия (ноябрь 2015 г.). «Избыточность усложняет определение основных генов вируса коровьей оспы» . Журнал общей вирусологии . 96 (11): 3326–37. дои : 10.1099/jgv.0.000266 . ПМЦ 5972330 . ПМИД 26290187 .
- ^ Дедрик Р.М., Маринелли Л.Дж., Ньютон Г.Л., Польяно К., Польяно Дж., Хэтфулл Г.Ф. (май 2013 г.). «Функциональные требования для роста бактериофагов: существенность и экспрессия генов в микобактериофаге Джайлза» . Молекулярная микробиология . 88 (3): 577–89. дои : 10.1111/mmi.12210 . ПМЦ 3641587 . ПМИД 23560716 .
- ^ Томас Х.А., Бенитес Кинтана А.Д., Бош М.А., Колл Де Пенья А., Агилера Э., Кулибали А. и др. (ноябрь 2016 г.). «Идентификация основных генов в фаге сальмонеллы SPN3US открывает новое понимание структуры и сборки головки гигантского фага» . Журнал вирусологии . 90 (22): 10284–10298. дои : 10.1128/JVI.01492-16 . ПМК 5105663 . ПМИД 27605673 .
- ^ Пал С., Папп Б., Лерчер М.Дж., Чермели П., Оливер С.Г., Херст Л.Д. (март 2006 г.). «Шанс и необходимость в эволюции минимальных метаболических сетей». Природа . 440 (7084): 667–70. Бибкод : 2006Natur.440..667P . дои : 10.1038/nature04568 . ПМИД 16572170 . S2CID 4424895 .
- ^ Мори Х., Баба Т., Ёкояма К., Такеучи Р., Номура В., Макиси К., Оцука Ю., Дозе Х., Ваннер Б.Л. (2015). «Идентификация основных генов и синтетических летальных комбинаций генов в Escherichia coli K-12». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 45–65. дои : 10.1007/978-1-4939-2398-4_4 . ISBN 978-1-4939-2397-7 . ПМИД 25636612 .
- ^ Верхаген Л.М., де Йонге М.И., Бургхаут П., Шраа К., Спаньоло Л., Менненс С., Элевелд М.Дж., ван дер Гааст-де Йонг К.Э., Зомер А., Херманс П.В., Бутсма Х.Дж. (2014). «Полногеномная идентификация генов, необходимых для выживания Streptococcus pneumoniae в слюне человека» . ПЛОС ОДИН . 9 (2): e89541. Бибкод : 2014PLoSO...989541V . дои : 10.1371/journal.pone.0089541 . ПМЦ 3934895 . ПМИД 24586856 .
- ^ Д'Суза Дж., Кост К. (ноябрь 2016 г.). «Экспериментальная эволюция метаболической зависимости у бактерий» . ПЛОС Генетика . 12 (11): e1006364. дои : 10.1371/journal.pgen.1006364 . ПМК 5096674 . ПМИД 27814362 .
- ^ Сюй, Пин; Видмер, Джованни; Ван, Инпин; Одзаки, Луис С.; Алвес, Жоао М.; Серрано, Мирна Г.; Пуйу, Даниэла; Манке, Патрисио; Акиёси, Донна; Макки, Аарон Дж.; Пирсон, Уильям Р. (октябрь 2004 г.). «Геном Cryptosporidium hominis» . Природа . 431 (7012): 1107–1112. Бибкод : 2004Natur.431.1107X . дои : 10.1038/nature02977 . ISSN 0028-0836 . ПМИД 15510150 . S2CID 4394344 .
- ^ Типплс Дж., МакКларти Дж. (июнь 1993 г.). «Облигатная внутриклеточная бактерия Chlamydia trachomatis ауксотрофна по трем из четырех рибонуклеозидтрифосфатов». Молекулярная микробиология . 8 (6): 1105–14. дои : 10.1111/j.1365-2958.1993.tb01655.x . ПМИД 8361355 . S2CID 46389854 .
- ^ Коте, Жан-Филипп; Френч, Шон; Герке, Себастьян С.; Макнейр, Крейг Р.; Мангат, Чанд С.; Бхарат, Амрита; Браун, Эрик Д. (22 ноября 2016 г.). «Сеть общегеномного взаимодействия генов питательного стресса в Escherichia coli» . мБио . 7 (6). дои : 10.1128/mBio.01714-16 . ISSN 2150-7511 . ПМК 5120140 . ПМИД 27879333 .
- ^ Вудс С., Коглан А., Риверс Д., Варнеке Т., Джеффрис С.Дж., Квон Т. и др. (май 2013 г.). Штернберг П.В. (ред.). «Смещения дублирования и удержания существенных и несущественных генов, выявленные с помощью систематического нокдаун-анализа» . ПЛОС Генетика . 9 (5): e1003330. дои : 10.1371/journal.pgen.1003330 . ПМЦ 3649981 . ПМИД 23675306 .
- ^ Jump up to: а б с д Фанг Дж., Роча Э., Данчин А. (ноябрь 2005 г.). «Насколько важны несущественные гены?». Молекулярная биология и эволюция . 22 (11): 2147–56. дои : 10.1093/molbev/msi211 . ПМИД 16014871 .
- ^ Джордан И.К., Рогозин И.Б., Вольф Ю.И., Кунин Е.В. (июнь 2002 г.). «Незаменимые гены более консервативны в эволюционном отношении, чем несущественные гены у бактерий» . Геномные исследования . 12 (6): 962–8. дои : 10.1101/гр.87702 . ПМЦ 1383730 . ПМИД 12045149 .
- ^ Райан Си Джей, Кроган, Нью-Джерси, Каннингем П., Кэгни Дж. (2013). «Все или ничего: белковые комплексы меняют сущность между отдаленно родственными эукариотами» . Геномная биология и эволюция . 5 (6): 1049–59. дои : 10.1093/gbe/evt074 . ПМЦ 3698920 . ПМИД 23661563 .
- ^ Сипички М (2000). «Где на древе жизни сидят делящиеся дрожжи?» . Геномная биология . 1 (2): ОБЗОРЫ1011. doi : 10.1186/gb-2000-1-2-reviews1011 . ПМЦ 138848 . ПМИД 11178233 .
- ^ Чен В.Х., Трачана К., Лерчер М.Дж., Борк П. (июль 2012 г.). «Молодые гены менее важны, чем старые гены, а дубликаты менее важны, чем одиночные гены того же возраста» . Молекулярная биология и эволюция . 29 (7): 1703–6. дои : 10.1093/molbev/mss014 . ПМЦ 3375470 . ПМИД 22319151 .
- ^ Кофоед М., Милбери К.Л., Чанг Дж.Х., Синха С., Бен-Аройя С., Гиавер Г. и др. (июль 2015 г.). «Обновленная коллекция термочувствительных аллелей незаменимых генов дрожжей со штрих-кодом» . Г3 . 5 (9): 1879–87. дои : 10.1534/g3.115.019174 . ПМЦ 4555224 . ПМИД 26175450 .
- ^ Дэн Дж, Су С, Лин X, Хассетт DJ, Лу LJ (2013). Ким П.М. (ред.). «Статистическая основа для улучшения геномных аннотаций основных генов прокариот» . ПЛОС ОДИН . 8 (3): e58178. Бибкод : 2013PLoSO...858178D . дои : 10.1371/journal.pone.0058178 . ПМЦ 3592911 . ПМИД 23520492 .
- ^ Моргенс Д.В., Динс Р.М., Ли А., Бассик М.С. (июнь 2016 г.). «Систематическое сравнение скринингов CRISPR/Cas9 и RNAi на предмет основных генов» . Природная биотехнология . 34 (6): 634–6. дои : 10.1038/nbt.3567 . ПМК 4900911 . ПМИД 27159373 .
- ^ Шарма С., Маркхэм П.Ф., Браунинг Г.Ф. (2014). «Гены, обнаруженные в других микоплазмах, не нужны для Mycoplasma bovis» . ПЛОС ОДИН . 9 (6): е97100. Бибкод : 2014PLoSO...997100S . дои : 10.1371/journal.pone.0097100 . ПМК 4045577 . ПМИД 24897538 .
- ^ Стоун В.Н., Сюй П (декабрь 2017 г.). «Таргетная противомикробная терапия в эпоху микробиома» . Молекулярная оральная микробиология . 32 (6): 446–454. дои : 10.1111/omi.12190 . ПМЦ 5697594 . ПМИД 28609586 .
- ^ Стоун В.Н., Парих Х.И., Эль-рами Ф., Ге Х, Чен В., Чжан Ю и др. (06.11.2015). Мерритт Дж. (ред.). «Идентификация низкомолекулярных ингибиторов мезо-2,6-диаминопимелатдегидрогеназы Porphyromonas gingivalis» . ПЛОС ОДИН . 10 (11): e0141126. Бибкод : 2015PLoSO..1041126S . дои : 10.1371/journal.pone.0141126 . ПМЦ 4636305 . ПМИД 26544875 .
- ^ Ченг Дж, Сюй З, Ву В, Чжао Л, Ли Х, Лю Ю, Тао С (2014). «Выбор обучающего набора для предсказания важнейших генов» . ПЛОС ОДИН . 9 (1): е86805. Бибкод : 2014PLoSO...986805C . дои : 10.1371/journal.pone.0086805 . ПМЦ 3899339 . ПМИД 24466248 .
- ^ Jump up to: а б Мушегян А.Р., Кунин Е.В. (сентябрь 1996 г.). «Минимальный набор генов клеточной жизни, полученный путем сравнения полных бактериальных геномов» . Труды Национальной академии наук Соединенных Штатов Америки . 93 (19): 10268–73. Бибкод : 1996PNAS...9310268M . дои : 10.1073/pnas.93.19.10268 . ПМК 38373 . ПМИД 8816789 .
- ^ Шарлебуа Р.Л., Дулиттл В.Ф. (декабрь 2004 г.). «Вычисление повсеместности прокариотических генов: спасение ядра от вымирания» . Геномные исследования . 14 (12): 2469–77. дои : 10.1101/гр.3024704 . ПМК 534671 . ПМИД 15574825 .
- ^ Jump up to: а б Юхас М., Эберл Л., Гласс Дж.И. (октябрь 2011 г.). «Суть жизни: важные гены минимальных геномов». Тенденции в клеточной биологии . 21 (10): 562–8. дои : 10.1016/j.tcb.2011.07.005 . ПМИД 21889892 .
- ^ Ян X, Ли Ю, Занг Дж, Ли Ю, Би П, Лу Ю, Ву Ц (апрель 2016 г.). «Анализ пангенома для идентификации основных и основных генов Brucella spp». Молекулярная генетика и геномика . 291 (2): 905–12. дои : 10.1007/s00438-015-1154-z . ПМИД 26724943 . S2CID 14565579 .
- ^ Швиковски Б., Уетц П., Филдс С. (декабрь 2000 г.). «Сеть белок-белковых взаимодействий у дрожжей». Природная биотехнология . 18 (12): 1257–61. дои : 10.1038/82360 . ПМИД 11101803 . S2CID 3009359 .
- ^ Чон Х., Мейсон С.П., Барабаси А.Л., Олваи З.Н. (май 2001 г.). «Летальность и центральность в белковых сетях». Природа . 411 (6833): 41–2. arXiv : cond-mat/0105306 . Бибкод : 2001Natur.411...41J . дои : 10.1038/35075138 . ПМИД 11333967 . S2CID 258942 .
- ^ Ю Х., Браун П., Йилдирим М.А., Лемменс И., Венкатесан К., Сахали Дж. и др. (октябрь 2008 г.). «Высококачественная карта взаимодействия бинарных белков сети интерактома дрожжей» . Наука . 322 (5898): 104–10. Бибкод : 2008Sci...322..104Y . дои : 10.1126/science.1158684 . ПМЦ 2746753 . ПМИД 18719252 .
- ^ Ли Х, Ли В, Цзэн М, Чжэн Р, Ли М (февраль 2019 г.). «Сетевые методы прогнозирования основных генов или белков: обзор». Брифинги по биоинформатике . 21 (2): 566–583. дои : 10.1093/нагрудник/bbz017 . ПМИД 30776072 .
- ^ Хуа Х.Л., Чжан Ф.З., Лабена А.А., Донг С., Цзинь Ю.Т., Го Ф.Б. (01.01.2016). «Подход к прогнозированию основных генов с использованием множественного картирования гомологии и алгоритмов машинного обучения» . БиоМед Исследования Интернэшнл . 2016 : 7639397. doi : 10.1155/2016/7639397 . ПМК 5021884 . ПМИД 27660763 .
- ^ Лю X, Ван Б, Сюй Л (2015). «Статистический анализ показателей Херста существенных/несущественных генов в 33 бактериальных геномах» . ПЛОС ОДИН . 10 (6): e0129716. Бибкод : 2015PLoSO..1029716L . дои : 10.1371/journal.pone.0129716 . ПМЦ 4466317 . ПМИД 26067107 .
- ^ Маккатчеон Дж. П., Макдональд Б. Р., Моран Н. А. (июль 2009 г.). Матич I (ред.). «Происхождение альтернативного генетического кода в чрезвычайно маленьком и богатом GC геноме бактериального симбионта» . ПЛОС Генетика . 5 (7): e1000565. дои : 10.1371/journal.pgen.1000565 . ПМК 2704378 . ПМИД 19609354 .
- ^ Баслер Г (2015). «Вычислительное прогнозирование основных метаболических генов с использованием подходов, основанных на ограничениях». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 183–204. дои : 10.1007/978-1-4939-2398-4_12 . ISBN 978-1-4939-2397-7 . ПМИД 25636620 .
- ^ Гатто Ф., Мисс Х., Шульце А., Нильсен Дж. (июнь 2015 г.). «Анализ баланса потоков предсказывает важные гены в метаболизме светлоклеточной почечно-клеточной карциномы» . Научные отчеты . 5 : 10738. Бибкод : 2015NatSR...510738G . дои : 10.1038/srep10738 . ПМЦ 4603759 . ПМИД 26040780 .
- ^ Сон К, Тонг Т, Ву Ф (апрель 2014 г.). «Прогнозирование основных генов в геномах прокариот с использованием линейного метода: ZUPLS» . Интегративная биология . 6 (4): 460–9. дои : 10.1039/c3ib40241j . ПМИД 24603751 .
- ^ Го ФБ, Е ЮН, Нин ЛВ, Вэй В (2015). «Три вычислительных инструмента для прогнозирования основных генов бактерий». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 205–17. дои : 10.1007/978-1-4939-2398-4_13 . ISBN 978-1-4939-2397-7 . ПМИД 25636621 .
- ^ Гудакр Н.Ф., Герлофф Д.Л., Утц П. (декабрь 2013 г.). «Белковые домены неизвестной функции необходимы бактериям» . мБио . 5 (1): e00744-13. дои : 10.1128/mBio.00744-13 . ПМК 3884060 . ПМИД 24381303 .
- ^ Лу Й, Лу Й, Дэн Дж, Лу Х, Лу LJ (2015). «Обнаружение основных доменов в основных генах». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 235–45. дои : 10.1007/978-1-4939-2398-4_15 . ISBN 978-1-4939-2397-7 . ПМИД 25636623 .
Дальнейшее чтение
[ редактировать ]- Гао Ф., Луо Х., Чжан Ч.Т., Чжан Р. (2015). «Анализ эссенциальности генов на основе DEG 10, обновленной базы данных основных генов». Генная эссенциальность . Методы молекулярной биологии. Том. 1279. стр. 219–33. дои : 10.1007/978-1-4939-2398-4_14 . ISBN 978-1-4939-2397-7 . ПМИД 25636622 .
- Лонг Дж.Л., изд. (2015). Эссенциальность гена — методы и протоколы Springer . Методы молекулярной биологии. Том. 1279. Хумана Пресс. п. 248. дои : 10.1007/978-1-4939-2398-4 . ISBN 978-1-4939-2397-7 . S2CID 27547825 .
- Чжан Р., изд. (2022). Основные гены и геномы . Методы молекулярной биологии. Том. 2377. Хумана Пресс. п. 434. дои : 10.1007/978-1-0716-1720-5 . ISBN 978-1-0716-1719-9 . S2CID 240006552 .