Информатика биоизображений
Информатика биоизображений — это раздел биоинформатики и вычислительной биологии . [1] Основное внимание уделяется использованию вычислительных методов для анализа биоизображений, особенно клеточных и молекулярных изображений, в больших масштабах и с высокой пропускной способностью. Цель состоит в том, чтобы получить полезные знания из сложных и разнородных изображений и связанных с ними метаданных .
Автоматизированные микроскопы способны собирать большое количество изображений с минимальным вмешательством. Это привело к взрывному росту объема данных, который абсолютно требует автоматической обработки. Кроме того, что удивительно, для некоторых из этих задач есть свидетельства того, что автоматизированные системы могут работать лучше, чем люди. [2] [3] Кроме того, автоматизированные системы непредвзяты, в отличие от человеческого анализа, на оценку которого может (даже неосознанно) влиять желаемый результат.
Все большее внимание уделяется разработке новых методов обработки изображений , компьютерного зрения , интеллектуального анализа данных , баз данных и методов визуализации для извлечения, сравнения, поиска и управления биологическими знаниями в этих проблемах, требующих большого объема данных. [4] [5]
Модальность данных [ править ]
Используется несколько систем и платформ сбора данных, для оптимального использования которых требуются разные методы.
Люминесцентная микроскопия [ править ]
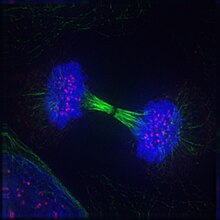
Флуоресцентная микроскопия позволяет напрямую визуализировать молекулы на субклеточном уровне как в живых, так и в фиксированных клетках. Интересующие молекулы помечены либо зеленым флуоресцентным белком (GFP), другим флуоресцентным белком, либо флуоресцентно меченным антителом . Регулярно используются несколько типов микроскопов: широкопольный, конфокальный или двухфотонный . Большинство систем микроскопии также поддерживают сбор временных рядов (фильмов).
Обычно фильтры используются таким образом, чтобы каждый краситель отображался отдельно (например, синий фильтр используется для изображения Hoechst , а затем быстро переключается на зеленый фильтр для изображения GFP). Для потребления изображения часто отображаются в искусственных цветах , показывая каждый канал в другом цвете, но они могут даже не быть связаны с исходными используемыми длинами волн. В некоторых случаях исходное изображение могло быть получено даже в невидимых длинах волн (обычно инфракрасное).
Выбор на этапе получения изображения будет влиять на анализ и часто требует специальной обработки. Конфокальные стеки потребуют 3D-обработки, а широкопольные псевдостеки часто выигрывают от цифровой деконволюции для удаления расфокусированного света.
Появление автоматизированных микроскопов, которые могут автоматически получать множество изображений, является одной из причин, по которой анализ невозможно проводить на глаз (в противном случае аннотации быстро стали бы узким местом в исследованиях). Использование автоматических микроскопов означает, что некоторые изображения могут быть не в фокусе (автоматические системы определения фокуса иногда могут давать неверные результаты), содержать небольшое количество клеток или быть заполнены мусором. Следовательно, созданные изображения будет труднее анализировать, чем изображения, полученные оператором, поскольку он выбрал бы другие места для изображения и правильной фокусировки. С другой стороны, оператор может внести неосознанную предвзятость в свой выбор, выбирая только те клетки, фенотип которых наиболее похож на ожидаемый до эксперимента.
Гистология [ править ]

Гистология — это применение микроскопии, при котором срезы тканей окрашиваются и наблюдаются под микроскопом (обычно световой микроскоп, но также используется электронная микроскопия).
При использовании светового микроскопа, в отличие от флуоресцентной визуализации, изображения обычно получают с помощью стандартных систем цветных камер. Это частично отражает историю этой области, где люди часто интерпретировали изображения, а также тот факт, что образец можно осветить белым светом и собрать весь свет без необходимости возбуждать флуорофоры. Когда используется более одного красителя, необходимым шагом предварительной обработки является разделение каналов и восстановление оценки интенсивности чистого красителя.
Было показано, что субклеточное расположение окрашенных белков можно определить по гистологическим изображениям.
Если целью является медицинская диагностика, то приложения гистологии часто попадают в область цифровой патологии или автоматического анализа изображений тканей , которые являются родственными областями информатики биоизображений. Часто применимы одни и те же вычислительные методы, но цели ориентированы скорее на медицину, чем на исследования.
Важные проблемы [ править ]
местоположения субклеточного Анализ

Анализ субклеточного местоположения был одной из первых задач в этой области. В контролируемом режиме задача состоит в том, чтобы изучить классификатор, который сможет распознавать изображения основных клеточных органелл на основе изображений.
Используемые методы основаны на машинном обучении и построении дискриминационного классификатора на основе числовых признаков, вычисленных на основе изображения. Характеристики представляют собой либо общие характеристики компьютерного зрения , такие как особенности текстуры Харалика , либо функции, специально разработанные для улавливания биологических факторов (например, типичным примером является совместная локализация с ядерным маркером).
Для основной задачи идентификации органелл можно получить очень высокие значения точности, в том числе лучше, чем ? результаты. [2] Эти методы полезны в фундаментальных исследованиях клеточной биологии, но также применяются для открытия белков, расположение которых меняется в раковых клетках. [6]
Однако классификация по органеллам представляет собой ограниченную форму проблемы, поскольку многие белки локализуются в нескольких местах одновременно (смешанные паттерны), и многие паттерны можно различить, даже если они не являются разными мембраносвязанными компонентами. В этой области существует ряд нерешенных проблем, и исследования продолжаются.
Показ высокого содержания [ править ]

Высокопроизводительный скрининг с использованием технологии автоматизированной визуализации (иногда называемый скринингом высокого содержания ) стал стандартным методом как для открытия лекарств, так и для фундаментальных биологических исследований. Используя многолуночные планшеты, робототехнику и автоматизированную микроскопию, один и тот же анализ можно очень быстро применить к большой библиотеке возможных реагентов (обычно малых молекул или РНКи ), получая тысячи изображений за короткий промежуток времени. Из-за большого объема генерируемых данных автоматический анализ изображений является необходимостью. [7]
Когда доступны положительные и отрицательные элементы управления, к проблеме можно подойти как к проблеме классификации и можно применить те же методы вычисления и классификации признаков, которые используются для анализа субклеточного местоположения.
Сегментация [ править ]
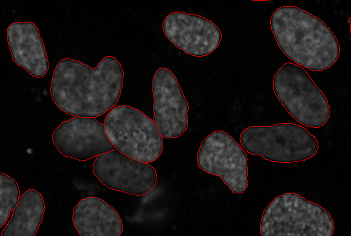
Сегментация клеток является важной подзадачой во многих из перечисленных ниже областей (и иногда полезна сама по себе, если цель состоит только в том, чтобы получить количество клеток в анализе жизнеспособности ). Цель состоит в том, чтобы определить границы ячеек в многоклеточном изображении. Это позволяет обрабатывать каждую ячейку индивидуально для измерения параметров. В 3D-данных сегментация должна выполняться в 3D-пространстве.
Поскольку визуализация ядерного маркера является общей для многих изображений, широко используемым протоколом является сегментация ядер. Это может быть полезно само по себе, если необходимы ядерные измерения, или может послужить водоразделом , который расширит сегментацию на все изображение.
На изображениях клеток описаны все основные методы сегментации: от простого определения порога до методов установки уровней. Поскольку существует множество модальностей изображения и разные типы клеток, каждый из которых подразумевает разные компромиссы, не существует единого принятого решения этой проблемы.
Сегментация изображений клеток как важная процедура часто используется для изучения экспрессии генов, взаимоотношений колокализации и т. д. отдельных клеток. В таких случаях анализа отдельных клеток часто необходимо однозначно определить идентичность клеток при их сегментации. Такая задача распознавания часто является нетривиальной в вычислительном отношении. Для модельных организмов, таких как C. elegans, которые имеют четко определенные клеточные линии, можно явно распознать идентичность клеток с помощью анализа изображений, комбинируя методы сегментации изображений и распознавания образов. [9] Одновременная сегментация и распознавание клеток [10] также было предложено как более точное решение этой проблемы, когда доступен «атлас» или другая предварительная информация о ячейках. Поскольку экспрессию генов с разрешением в одной клетке можно получить с использованием этих типов подходов, основанных на визуализации, можно комбинировать эти методы с другими методами количественного определения экспрессии генов в одной клетке, такими как RNAseq.
Отслеживание [ править ]
Отслеживание — еще одна традиционная проблема обработки изображений, возникающая в информатике биоизображений. Проблема состоит в том, чтобы связать объекты, которые появляются в последующих кадрах фильма. Как и в случае с сегментацией, задача может быть поставлена как в двух-, так и в трехмерной форме. [11]
В случае флуоресцентной визуализации отслеживание часто необходимо выполнять на изображениях с очень низкой контрастностью. Поскольку для достижения высокого контраста требуется больше света, который повреждает образец и разрушает краситель , освещение поддерживается на минимальном уровне. Часто бывает полезно подумать о бюджете фотонов: количество фотонов, которые можно использовать для визуализации до того, как образец будет поврежден, настолько велико, что данным больше нельзя доверять. Следовательно, если необходимо получить высококонтрастные изображения, то можно использовать лишь несколько кадров; в то время как в длинных фильмах каждый кадр будет иметь очень низкую контрастность.
Регистрация [ править ]
Когда рассматриваются образцы данных изображений разной природы, например, соответствующие различным методам маркировки, разным людям, образцам в разные моменты времени и т. д., изображения часто необходимо регистрировать для лучшего сравнения. Одним из примеров является то, что при сборе данных о динамике во времени часто приходится регистрировать изображения в последующих кадрах , чтобы можно было скорректировать незначительные сдвиги в положении камеры. Другим примером является то, что когда собрано множество изображений модельного животного (например, мозга C. elegans , мозга дрозофилы или мозга мыши ), часто возникает существенная необходимость зарегистрировать эти изображения, чтобы сравнить их образцы (например, те, которые соответствуют одинаковым или разным изображениям). популяция нейронов, имеющих общую или различную экспрессию генов и т. д.).
Пакеты программного обеспечения для регистрации медицинских изображений были первыми попытками использования приложений для регистрации микроскопических изображений. Однако из-за гораздо большего размера файла изображения и гораздо большего количества образцов в экспериментах во многих случаях необходимо разработать новое программное обеспечение для регистрации 3D-изображений. BrainAligner [12] это программное обеспечение, которое использовалось для автоматизации процесса трехмерной деформируемой и нелинейной регистрации с использованием стратегии надежного сопоставления ориентиров. В первую очередь он использовался для создания более 50 000 стандартизированных трехмерных изображений мозга плодовых мух на ферме Джанелия в HHMI, а также в других приложениях, включая стрекоз и мышей.
Важные места [ править ]
Консорциум ученых из университетов и научно-исследовательских институтов организовал ежегодные встречи по информатике биоизображений. [13] с 2005 года. На конференции ISMB есть трек «Биоизображения и визуализация данных» с 2010 года. Журнал «Биоинформатика» также представил трек «Информатика биоизображений» в 2012 году. В журнале OpenAccess BMC Bioinformatics есть раздел, посвященный анализу биоизображений, визуализации и связанным с ними приложениям. Другие журналы по вычислительной биологии и биоинформатике также регулярно публикуют работы по информатике биоизображений. С 2017 года программа Европейского Союза по затратам под названием NEUBIAS (сеть европейских аналитиков биоизображений) организует ежегодные конференции, а также школы подготовки аналитиков биоизображений и таггатоны.
Программное обеспечение [ править ]
Существует несколько пакетов, которые делают методы информатики биоизображений доступными через графический интерфейс пользователя, например ImageJ , FIJI , CellProfiler , chunkflow или Icy . Платформы визуализации и анализа, такие как Vaa3D, появились в последние годы и использовались как в крупномасштабных проектах, особенно в нейробиологии, так и в настольных приложениях.

Другие исследователи разрабатывают свои собственные методы, обычно основанные на языках программирования с хорошей поддержкой компьютерного зрения, таких как Python , C++ или MATLAB . Библиотека Mahotas для Python — один из популярных примеров. Хотя существуют примеры разработанных исследователями методов на языках программирования с меньшей поддержкой компьютерного зрения, чем R (например, trackdem [14] ).
См. также [ править ]
- Фокус-стекинг Техника объединения нескольких изображений с разным фокусным расстоянием в одно.
- Высококонтентный показ
- цифровая патология
- Медицинская визуализация
Внешние ссылки [ править ]
- Vaa3D: Высокопроизводительная многомерная визуализация и анализ изображений.
- Биоформаты Механизм ввода-вывода файлов изображений, поддерживающий десятки форматов.
Ссылки [ править ]
- ^ Пэн, Х; Бейтман А; Валенсия А; Рен Джей Ди (2012). «Информатика биоизображений: новая категория в биоинформатике» . Биоинформатика . 28 (8): 1057. doi : 10.1093/bioinformatics/bts111 . ПМЦ 3324521 . ПМИД 22399678 .
- ^ Jump up to: Перейти обратно: а б Мерфи, Роберт; Веллисте, М.; Поррека, Г. (2003). «Надежные числовые характеристики для описания и классификации моделей субклеточного расположения на изображениях флуоресцентного микроскопа». Журнал обработки сигналов СБИС . 35 (3): 311–321. CiteSeerX 10.1.1.186.9521 . doi : 10.1023/b:vlsi.0000003028.71666.44 . S2CID 8134907 .
- ^ Натткемпер, Тим; Торстен Твеллманн; Хельге Риттер; Вальтер Шуберт (2003). «Человек против машины: оценка флуоресцентных микрофотографий». Компьютеры в биологии и медицине . 33 (1): 31–43. CiteSeerX 10.1.1.324.4664 . дои : 10.1016/S0010-4825(02)00060-4 . ПМИД 12485628 .
- ^ Пэн Х (сентябрь 2008 г.). «Информатика биоизображений: новое направление инженерной биологии» . Биоинформатика . 24 (17): 1827–36. doi : 10.1093/биоинформатика/btn346 . ПМК 2519164 . ПМИД 18603566 .
- ^ «В поисках количественной микроскопии» . Природные методы . 9 (7): 627. 2012. doi : 10.1038/nmeth.2102 . ПМИД 22930824 .
- ^ Слава, Эстель; Джастин Ньюберг; Роберт Ф. Мерфи (2008). «Автоматическое сравнение моделей субклеточного расположения белков между изображениями нормальных и раковых тканей». Биомедицинская визуализация: от нано к макросу, 2008. ISBI 2008. 5-й международный симпозиум IEEE по .
- ^ Шариф, Абид; Джошуа Кангас; Луис Педро Коэльо; Шеннон Куинн; Роберт Ф. Мерфи (2010). «Автоматизированный анализ изображений для проверки и анализа большого количества контента» . Журнал биомолекулярного скрининга . 15 (7): 726–734. дои : 10.1177/1087057110370894 . ПМИД 20488979 .
- ^ Коэльо, Луис Педро; Абид Шариф; Роберт Ф. Мерфи (2009). «Ядерная сегментация в изображениях клеток микроскопа: сегментированный вручную набор данных и сравнение алгоритмов». Биомедицинская визуализация: от нано к макросу, 2009. ISBI'09. Международный симпозиум IEEE. ИИЭЭ . дои : 10.1109/ISBI.2009.5193098 . ПМК 2901896 .
- ^ Лонг, Фухуэй; Пэн, Х.; Лю, X.; Ким, С.; Майерс, EW (сентябрь 2009 г.). «Цифровой 3D-атлас C. elegans и его применение для анализа одиночных клеток» . Природные методы . 6 (9): 667–672. дои : 10.1038/nmeth.1366 . ПМК 2882208 . ПМИД 19684595 .
- ^ Цюй, Лей; Лонг, Ф.; Лю, X.; Ким, С.; Майерс, EW; Пэн, Х. (2011). «Одновременное распознавание и сегментация клеток: применение у C. elegans» . Биоинформатика . 27 (20): 2895–2902. doi : 10.1093/биоинформатика/btr480 . ПМК 3187651 . ПМИД 21849395 .
- ^ Дюфур, Александр; Василий Шинин; Шахрагим Таджбахш; Нэнси Гильен-Агион; Джей Си. Оливо-Марин; Кристоф Циммер (2005). «Сегментация и отслеживание флуоресцентных клеток в динамической трехмерной микроскопии со связанными активными поверхностями» (PDF) . Обработка изображений, транзакции IEEE на 14, вып. 9 . стр. 1396–1410. дои : 10.1109/TIP.2005.852790 . Архивировано из оригинала (PDF) 2 марта 2014 г. .
- ^ Пэн, Ханьчуань; Чунг, П.; Лонг, Ф.; Цюй, Л.; Дженетт, А.; Семена, А.; Майерс, EW; Симпсон, Дж. Х. (2011). «BrainAligner: 3D-атласы регистрации мозга дрозофилы» . Природные методы . 8 (6): 493–498. дои : 10.1038/nmeth.1602 . ПМК 3104101 . ПМИД 21532582 .
- ^ «Ежегодное собрание по информатике биоизображений» .
- ^ Брюйнинг, Маржолейн; Виссер, Марко Д.; Халлманн, Каспар А.; Йонгеянс, Элке; Голдинг, Ник (2018). «trackdem: автоматическое отслеживание частиц для получения количества населения и распределения размеров по видео в r» . Методы экологии и эволюции . 9 (4): 965–973. дои : 10.1111/2041-210X.12975 . hdl : 2066/184075 . ISSN 2041-210X .