Метабаркодирование
| Часть серии о |
| ДНК-штрихкодирование |
|---|
 |
| По таксонам |
| Другой |
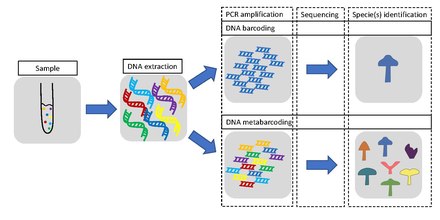
Метабаркодирование это штрих-кодирование ДНК — / РНК (или eDNA / eRNA ) способом, позволяющим одновременно идентифицировать множество таксонов в одном образце. Основное различие между штрих-кодированием и метабаркодированием заключается в том, что метабаркодирование не фокусируется на одном конкретном организме, а вместо этого направлено на определение видового состава в образце.
Штрих-код состоит из короткой вариабельной области гена (например, см. различные маркеры/штрих-коды ), которая полезна для таксономического назначения, окруженной высококонсервативными областями гена, которые можно использовать для разработки праймеров . [1] Идея общего штрих-кодирования возникла в 2003 году у исследователей из Университета Гвельфа . [2]
Процедура метабаркодирования, как и обычное штрихкодирование, последовательно проходит через этапы выделения ДНК , ПЦР-амплификации , секвенирования и анализа данных . Используются разные гены в зависимости от того, ставится ли цель штрих-кодирования одного вида или метабаркодирования нескольких видов. В последнем случае используется более универсальный ген. Метабаркодирование не использует ДНК/РНК одного вида в качестве отправной точки, а ДНК/РНК нескольких разных организмов, полученных из одного образца окружающей среды или общего образца.
Экологическая ДНК
[ редактировать ]Экологическая ДНК или эДНК описывает генетический материал, присутствующий в образцах окружающей среды, таких как отложения, вода и воздух, включая целые клетки, внеклеточную ДНК и, возможно, целые организмы. [3] [4] еДНК может быть получена из образцов окружающей среды и сохранена , извлечена , амплифицирована , секвенирована и классифицирована на основе ее последовательности. [5] На основе этой информации возможно обнаружение и классификация видов. ЭДНК может происходить из кожи, слизистых, слюны, спермы, секретов, яиц, фекалий, мочи, крови, корней, листьев, фруктов, пыльцы и гниющих тел более крупных организмов, тогда как микроорганизмы могут быть получены целиком. [6] [7] [4] Производство эДНК зависит от биомассы , возраста и пищевой активности организма, а также физиологии, истории жизни и использования пространства. [4] [8] [9] [10]
К 2019 году методы исследования электронной ДНК были расширены, и теперь можно оценивать целые сообщества по одному образцу. Этот процесс включает в себя метабаркодирование, которое можно точно определить как использование общих или универсальных полимеразной цепной реакции (ПЦР) праймеров на смешанных образцах ДНК любого происхождения с последующим высокопроизводительным секвенированием следующего поколения (NGS) для определения видового состава ДНК. образец. Этот метод уже много лет распространен в микробиологии, но по состоянию на 2020 год он только начинает применяться в оценке макроорганизмов. [11] [12] [13] Применение метабаркодирования eDNA в масштабах всей экосистемы может не только описывать сообщества и биоразнообразие, но также обнаруживать взаимодействия и функциональную экологию в больших пространственных масштабах, хотя это может быть ограничено ложными показаниями из-за загрязнения или других ошибок. [7] [14] [12] [9] В целом метабаркодирование eDNA увеличивает скорость, точность и идентификацию по сравнению с традиционным штрих-кодированием и снижает стоимость, но требует стандартизации и унификации, интеграции таксономии и молекулярных методов для полного экологического исследования. [11] [15] [16] [17] [9] [10]



с метабаркодированием ДНК окружающей среды [10]
Метабаркодирование eDNA применяется для мониторинга разнообразия во всех средах обитания и таксономических группах, реконструкции древних экосистем, взаимодействия растений и опылителей, анализа рациона, обнаружения инвазивных видов, реагирования на загрязнение и мониторинга качества воздуха. Метабаркодирование eDNA — это уникальный метод, который все еще находится в разработке и, вероятно, будет продолжать меняться в течение некоторого времени по мере развития технологий и стандартизации процедур. Однако по мере того, как метабаркодирование будет оптимизировано и его использование станет более распространенным, оно, вероятно, станет важным инструментом экологического мониторинга и глобальных исследований по сохранению природы. [10]
ДНК сообщества
[ редактировать ]С момента появления высокопроизводительного секвенирования ( HTS ) [19] Использование метабаркодирования в качестве инструмента обнаружения биоразнообразия вызвало огромный интерес. [12] [20] Однако до сих пор не ясно, какой исходный материал используется для проведения анализа метабаркодирования (например, ДНК окружающей среды или ДНК сообщества ). Без ясности между этими двумя исходными материалами различия в отборе проб, а также различия в лабораторных процедурах могут повлиять на последующие процессы биоинформатики, используемые для обработки данных, и усложнить интерпретацию пространственных и временных моделей биоразнообразия. Здесь мы стремимся четко различать преобладающие используемые исходные материалы и их влияние на последующий анализ и интерпретацию метабаркодирования ДНК окружающей среды животных и растений по сравнению с метабаркодированием ДНК сообщества. [13]
При метабаркодировании ДНК животных и растений целевые группы чаще всего собираются большими партиями (например, из почвы, ловушки или сети), а особи отделяются от других остатков образцов и объединяются вместе перед массовым извлечением ДНК. [12] Напротив, эДНК макроорганизма изолируется непосредственно из материала окружающей среды (например, почвы или воды) без предварительного отделения отдельных организмов или растительного материала от образца и неявно предполагает, что весь организм не присутствует в образце. Конечно, образцы ДНК сообщества могут содержать ДНК из частей тканей, клеток и органелл других организмов (например, содержимого кишечника, кожной внутриклеточной или внеклеточной ДНК). Аналогично, образцы эДНК макроорганизмов могут непреднамеренно захватывать целые микроскопические нецелевые организмы (например, простейших, бактерии). Таким образом, на практике это различие может, по крайней мере частично, исчезнуть. [13]
Еще одно важное различие между ДНК сообщества и эДНК макроорганизма заключается в том, что последовательности, полученные в результате метабаркодирования ДНК сообщества, могут быть таксономически проверены, если образцы не уничтожаются в процессе экстракции. Здесь последовательности можно затем генерировать из ваучерных образцов с использованием секвенирования по Сэнгеру. Поскольку в образцах для метабаркодирования эДНК отсутствуют целые организмы, такие сравнения in situ провести невозможно. Таким образом, таксономическое сходство может быть установлено только путем прямого сравнения полученных последовательностей (или с помощью биоинформатически сгенерированных операционных таксономических единиц (MOTU)) с последовательностями, которые таксономически аннотированы, например, в базе данных нуклеотидов GenBank NCBI. [21] СМЕЛЫЙ , [22] или к самостоятельно созданным справочным базам данных из ДНК, секвенированной по Сэнгеру. [23] [24] [25] (Молекулярная операционная таксономическая единица (MOTU) — это группа, идентифицируемая с помощью кластерных алгоритмов и заранее определенного процентного сходства последовательностей, например, 97%)). [26] [13] Затем, чтобы хотя бы частично подтвердить полученный список таксонов, проводятся сравнения с традиционными физическими, акустическими или визуальными методами исследования, проводимыми в то же время, или с историческими записями исследований определенного местоположения (см. Таблицу 1). [13]
Таким образом, разница в исходном материале между ДНК сообщества и эДНК имеет различные последствия для интерпретации масштаба выводов во времени и пространстве об обнаруженном биоразнообразии. Из ДНК сообщества ясно, что отдельные виды были обнаружены в это время и в этом месте, но что касается эДНК, то организм, продуцирующий ДНК, может находиться выше места отбора пробы. [27] или ДНК могла быть перенесена с фекалиями более мобильных хищных видов (например, птиц, откладывающих эДНК рыб, [28] или ранее присутствовал, но больше не активен в сообществе, и обнаружение происходит по ДНК, которая была выделена годами или десятилетиями ранее. [29] Последнее означает, что при выводе о присутствии вида в сообществе на основе эДНК необходимо тщательно учитывать масштаб выводов как в пространстве, так и во времени. [13]
Этапы метабаркодирования
[ редактировать ]
Существует шесть стадий или этапов штрих-кодирования и метабаркодирования ДНК. Штрих-кодирование ДНК животных (и, в частности, летучих мышей ) используется в качестве примера на диаграмме справа и в обсуждении непосредственно ниже.
Сначала выбираются подходящие области штрих-кодирования ДНК, чтобы ответить на какой-то конкретный исследовательский вопрос. Наиболее часто используемая область штрих-кода ДНК для животных представляет собой сегмент около 600 пар оснований длиной митохондриального гена цитохромоксидазы I (CO1). [2] Этот локус обеспечивает большую изменчивость последовательностей между видами, но относительно небольшую вариацию внутри вида. [31] Другими часто используемыми областями штрих-кода, используемыми для идентификации видов животных, являются области рибосомальной ДНК (рДНК), такие как 16S , 18S и 12S , и митохондриальные области, такие как цитохром B. [32] [33] [34] [35] Эти маркеры имеют преимущества и недостатки и используются для разных целей. [36] [37] Более длинные области штрих-кода (длиной не менее 600 пар оснований) часто необходимы для точного разграничения видов, особенно для дифференциации близких родственников. Идентификация производителя останков организма, таких как фекалии, волосы и слюна, может использоваться в качестве косвенной меры для проверки отсутствия/присутствия вида в экосистеме. ДНК в этих останках обычно имеет низкое качество и количество, поэтому в этих случаях используются более короткие штрих-коды длиной около 100 пар оснований. Точно так же остатки ДНК в навозе также часто разлагаются, поэтому для идентификации потребляемой добычи необходимы короткие штрих-коды. [30]
Во-вторых, необходимо создать справочную базу данных всех штрих-кодов ДНК, которые могут встретиться в исследовании. В идеале эти штрих-коды должны быть созданы на основе сертифицированных образцов, хранящихся в общедоступном месте, например, в музее естествознания или другом научно-исследовательском институте. [30] Создание таких справочных баз данных в настоящее время ведется во всем мире. Партнерские организации сотрудничают в международных проектах, таких как Международный проект по штрих-коду жизни (iBOL) и Консорциум по штрих-коду жизни (CBOL), стремясь создать эталонный штрих-код ДНК, который станет основой для идентификации мирового биома на основе ДНК. Хорошо известными репозиториями штрих-кодов являются NCBI GenBank и Barcode of Life Data System (BOLD). [30]
В-третьих, клетки, содержащие интересующую ДНК, необходимо вскрыть, чтобы обнажить ее ДНК. Этот этап — ДНК экстракция и очистка — должен выполняться из исследуемого субстрата. Для этого существует несколько процедур. [38] Необходимо выбрать специальные методы для выделения ДНК из субстратов с частично деградированной ДНК, например образцов ископаемых и образцов, содержащих ингибиторы, таких как кровь, фекалии и почва. Экстракции, при которых выход или качество ДНК, как ожидается, будут низкими, должны проводиться в древнем центре ДНК в соответствии с установленными протоколами, чтобы избежать загрязнения современной ДНК. [39] [40] Эксперименты всегда следует проводить в двух экземплярах. [41] и с включенным положительным контролем. [30]
В-четвертых, ампликоны должны быть созданы из ДНК, выделенной либо из одного образца, либо из сложных смесей с праймерами кодов ДНК, выбранных на этапе 1. Чтобы отслеживать их происхождение, на основе штрих - необходимо добавляется в случае метабаркодирования. Эти метки понадобятся в дальнейшем при анализе для отслеживания операций чтения из массива данных до их источника. [30]

В-пятых, необходимо выбрать соответствующие методы секвенирования ДНК . Классический метод обрыва цепи Сэнгера основан на селективном включении ингибиторов ДНК-полимеразы, удлиняющих цепь, во время репликации ДНК . Эти четыре основания разделяются по размеру с помощью электрофореза , а затем идентифицируются с помощью лазерного обнаружения. Метод Сэнгера ограничен и может производить одно считывание одновременно и поэтому подходит для создания штрих-кодов ДНК из субстратов, содержащих только один вид. [30] Новые технологии, такие как секвенирование нанопор, привели к снижению стоимости секвенирования ДНК с примерно 30 000 долларов США за мегабайт в 2002 году до примерно 0,60 долларов США в 2016 году. [43] [44] Современные технологии секвенирования нового поколения (NGS) могут обрабатывать тысячи и миллионы считываний параллельно и, следовательно, подходят для массовой идентификации смеси различных видов, присутствующих в субстрате, что называется метабаркодированием. [30]
Наконец, биоинформатический анализ необходимо провести для сопоставления штрих-кодов ДНК, полученных с номерами индексов штрих-кодов (BIN) в справочных библиотеках. [45] Каждый BIN или кластер BIN может быть идентифицирован на уровне вида, если он демонстрирует высокое (>97%) соответствие штрих-кодам ДНК, связанным с видом, присутствующим в справочной библиотеке, или когда таксономическая идентификация на уровне вида все еще отсутствует , таксономическая единица (ОТЕ), которая относится к группе видов (т.е. роду, семейству или более высокому таксономическому рангу). [30] (См. бинирование (метагеномика) ). Результаты биоинформатического конвейера должны быть сокращены, например, путем фильтрации ненадежных одиночных результатов, лишних дубликатов, низкокачественных чтений и/или химерных чтений . Обычно это делается путем выполнения последовательного поиска BLAST в сочетании со сценариями автоматической фильтрации и обрезки. [46] Стандартизированные пороговые значения необходимы для различения разных видов или для правильной и неправильной идентификации. [30]
Рабочий процесс метабаркодирования
[ редактировать ]Несмотря на очевидную мощь этого подхода, на метабаркодирование eDNA влияют проблемы точности и аккуратности, возникающие в ходе рабочего процесса в полевых условиях, в лаборатории и на клавиатуре. [47] Как показано на диаграмме справа, в соответствии с первоначальным дизайном исследования (гипотеза/вопрос, целевая таксономическая группа и т. д.) текущий рабочий процесс с eDNA состоит из трех компонентов: полевых, лабораторных и биоинформатических. [13] Полевой компонент состоит из сбора проб (например, воды, отложений, воздуха), которые консервируются или замораживаются перед экстракцией ДНК. Лабораторный компонент состоит из четырех основных этапов: (i) ДНК концентрируется (если не проводится в полевых условиях) и очищается, (ii) ПЦР используется для амплификации целевого гена или области, (iii) создаются уникальные нуклеотидные последовательности, называемые «индексами» ( также называемые «штрих-кодами»), встраиваются с помощью ПЦР или лигируются (связываются) с различными продуктами ПЦР, создавая «библиотеку», посредством которой несколько образцов могут быть объединены вместе, и (iv) объединенные библиотеки затем секвенируются на высокопроизводительном компьютере. машина . Последним шагом после лабораторной обработки образцов является вычислительная обработка выходных файлов секвенатора с использованием надежного биоинформационного конвейера. [13]

исследования метабаркодирования ДНК окружающей среды [13]

OTU и концепция вида
[ редактировать ]Этот раздел пуст. Вы можете помочь, добавив к нему . ( март 2021 г. ) |
Метод и визуализация
[ редактировать ]
б) Модели бета-разнообразия , проиллюстрированные с помощью анализа основных координат, выполненного в QIIME , [48] где каждая точка представляет образец, а цвета различают разные классы образцов. Чем ближе две точки выборки в трехмерном пространстве, тем более похожи их сообщества.
в) Филогенетическая визуализация данных об окружающей среде GraPhalAn с круговыми тепловыми картами и полосами численности, используемыми для передачи количественных характеристик таксона. [49]
d) Edge PCA, метрика разнообразия на основе дерева, которая определяет конкретные линии (зеленые/оранжевые ветви), которые в наибольшей степени способствуют изменениям сообщества, наблюдаемым в образцах, распределенных по разным осям PCA. [50] [51]
Этот метод требует, чтобы каждая собранная ДНК была заархивирована с соответствующим «типовым образцом» (по одному для каждого таксона) в дополнение к обычным данным сбора. Эти типы хранятся в конкретных учреждениях (музеях, молекулярных лабораториях, университетах, зоологических садах, ботанических садах, гербариях и т. д.) по одному на каждую страну, а в некоторых случаях одному и тому же учреждению поручается содержать типы более чем одной страны. , в тех случаях, когда у некоторых стран нет для этого технологий или финансовых ресурсов.
Таким образом, создание типовых образцов генетических кодов представляет собой методологию, параллельную той, которая применяется в традиционной систематике.
На первом этапе была определена область ДНК, которая будет использоваться для создания штрих-кода. Он должен был быть коротким и обеспечивать высокий процент уникальных последовательностей. У животных, водорослей и грибов часть митохондриального гена, который кодирует субъединицу 1 фермента цитохромоксидазы CO1, обеспечивает высокий процент (95%), область длиной около 648 пар оснований. [52]
В случае растений использование CO1 не было эффективным, поскольку они имеют низкий уровень изменчивости в этом регионе, в дополнение к трудностям, которые возникают из-за частых эффектов полиплоидии , интрогрессии и гибридизации, поэтому геном хлоропластов кажется более подходящий. [53] [54]
Приложения
[ редактировать ]Сети опылителей
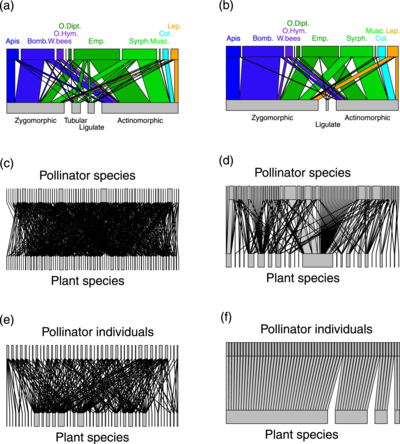
[ редактировать ]а, б – группы растений-опылителей
в,г – виды растений-опылителей
(д, е) отдельные виды растений-опылителей
( Empis leptempis pandellei )
Толщина линии подчеркивает пропорцию взаимодействий
На диаграмме справа показано сравнение сетей опыления, основанных на метабаркодировании ДНК, с более традиционными сетями, основанными на прямых наблюдениях за посещением растений насекомыми. Обнаруживая многочисленные дополнительные скрытые взаимодействия, данные метабаркодирования в значительной степени меняют свойства сетей опыления по сравнению с опросами посещений. Молекулярные данные показывают, что опылители гораздо более универсальны , чем можно было ожидать по результатам опросов. Однако виды опылителей состояли из относительно специализированных особей и образовывали функциональные группы, узкоспециализированные по цветочным морфам . [55]
В результате продолжающихся глобальных изменений во всем мире резкое и параллельное сокращение численности опылителей и видов растений, опыляемых животными. наблюдается [56] Понимание реакции сетей опыления на это сокращение крайне необходимо для диагностики рисков, которым могут подвергнуться экосистемы, а также для разработки и оценки эффективности природоохранных действий. [57] Ранние исследования опыления животными касались упрощенных систем, то есть специфических парных взаимодействий, или включали небольшие подмножества растительно-животных сообществ. Однако воздействие возмущений происходит через очень сложные сети взаимодействия. [58] и в настоящее время эти сложные системы находятся в центре внимания исследований. Оценка истинных сетей (определяемых экологическим процессом) на основе полевых исследований, которые подвержены эффектам выборки, по-прежнему представляет собой проблему. [59] [55]
Недавние исследования явно выиграли от сетевых концепций и инструментов для изучения моделей взаимодействия в крупных сообществах видов. [60] Они показали, что сети растений-опылителей были высокоструктурированы и значительно отклонялись от случайных ассоциаций. [61] Обычно сети имеют (1) низкую степень связности (реализованную долю всех потенциальных связей в сообществе), что предполагает низкую степень обобщения; (2) высокая вложенность (более специализированные организмы с большей вероятностью будут взаимодействовать с подмножествами видов, с которыми взаимодействуют более универсальные организмы); более специализированные виды взаимодействуют только с соответствующими подмножествами тех видов, которые взаимодействуют с более универсальными видами; [62] (3) совокупное распределение связности (количество связей на вид, с), которое следует степенному или усеченному степенному закону. [63] характеризуется небольшим количеством суперуниверсалов с большим количеством связей, чем ожидалось по случайности, и большим количеством специалистов; (4) модульная организация. Модуль — это группа видов растений и опылителей, которая демонстрирует высокий уровень связности внутри модуля и плохо связана с видами других групп. [64] [55]
Низкий уровень связности и высокая доля специалистов в сетях опыления контрастируют с мнением, что в сетях нормой является обобщение, а не специализация. [65] [66] Действительно, большинство видов растений посещают разнообразные опылители, которые используют цветочные ресурсы самых разных видов растений. [67] [68] Основная причина, объясняющая это кажущееся противоречие, — неполная выборка взаимодействий. [69] Действительно, большинство свойств сети очень чувствительны к интенсивности выборки и размеру сети. [61] Сетевые исследования в основном фитоцентричны, т.е. основаны на наблюдениях за посещением цветов опылителями. Тем не менее, этот подход, ориентированный на растения, страдает от присущих ему ограничений, которые могут препятствовать пониманию механизмов, способствующих объединению сообществ и моделям биоразнообразия. Во-первых, прямых наблюдений за посещением опылителями определенных таксонов, таких как орхидеи, часто недостаточно. [70] а редкие взаимодействия вообще очень трудно обнаружить в полевых условиях. [57] [58] [59] [60] Сообщества опылителей и растений обычно состоят из небольшого числа распространенных видов и множества редких видов, которые плохо регистрируются при посещениях. [71] [72] Эти редкие виды выглядят как специалисты, хотя на самом деле они могут быть типичными универсалами. Из-за положительной связи между частотой взаимодействия (f) и связностью (s), недостаточная выборка взаимодействий может привести к переоценке степени специализации сетей. [73] Во-вторых, сетевой анализ в основном проводился на видовом уровне. Сети очень редко масштабировались до функциональных групп или уменьшались до индивидуальных сетей. [74] и большинство из них были сосредоточены только на одном или двух видах. Поведение особей или колоний обычно игнорируется, хотя оно может влиять на структуру видовых сетей. [74] Таким образом, виды, считающиеся универсальными в видовых сетях, могут включать в себя загадочно специализированных особей или колонии. В-третьих, посетители цветов далеко не всегда являются эффективными опылителями, поскольку они могут не откладывать пыльцу одного вида и/или много пыльцы гетероспецифичного вида. [75] [76] Подходы, ориентированные на животных, основанные на исследовании нагрузки пыльцы на посетителей и рыльцев растений, могут быть более эффективными для выявления взаимодействий растений и опылителей. [75] [76] [55]
Распутывание пищевых сетей
[ редактировать ]
(А) Трофическая сеть:
членистоногих и позвоночных хищников – стрелки обозначают поток биомассы между хищниками и жертвами.
(Б) Внутригильдейные взаимодействия: * Членистоногие хищники * Паразитоиды членистоногих: * Насекомоядные позвоночные: [77]
Метабаркодирование открывает новые возможности для расшифровки трофических связей между хищниками и их добычей в пищевых сетях. [78] [79] По сравнению с традиционными, трудоемкими методами, такими как микроскопический или серологический анализ , развитие метабаркодирования ДНК позволяет идентифицировать виды добычи без предварительного знания ареала добычи хищника. [80] Кроме того, метабаркодирование также можно использовать для характеристики большого количества видов в одной реакции ПЦР и для одновременного анализа нескольких сотен образцов. [6] Такой подход все чаще используется для изучения функционального разнообразия и структуры пищевых сетей в агроэкосистемах. [79] [81] [82] [83] [84] Как и другие молекулярные подходы, метабаркодирование дает только качественные результаты о наличии/отсутствии видов-жертв в кишечнике или образцах фекалий. [85] Однако это знание о идентичности добычи, потребляемой хищниками одного и того же вида в данной среде, дает «прагматичный и полезный суррогат действительно количественной информации». [86] [77]
В экологии пищевой сети вопрос «кто кого ест» является фундаментальным вопросом для лучшего понимания сложных трофических взаимодействий, существующих между вредителями и их естественными врагами в данной экосистеме. [36] [87] Анализ питания членистоногих и позвоночных хищников позволяет идентифицировать ключевых хищников, участвующих в естественной борьбе с членистоногими вредителями, и дает представление о широте их рациона ( универсальный или специализированный ) и внутригильдейском хищничестве . [77]
На диаграмме справа обобщены результаты исследования 2020 года, в котором метабаркодирование использовалось для того, чтобы распутать функциональное разнообразие и структуру пищевой сети, связанной с парой полей проса в Сенегале. После отнесения идентифицированных OTU к видам 27 таксонов-жертв членистоногих были идентифицированы у девяти хищников-членистоногих. Среднее количество таксонов-жертв, обнаруженных на образец, было самым высоким у жужелиц , муравьев и пауков и самым низким у остальных хищников, включая антокоридовых клопов, пентатомидных клопов и уховерток. Среди хищных членистоногих высокое разнообразие жертв членистоногих отмечено у пауков, жужелиц, муравьев и клопов-антокорид. Напротив, разнообразие видов добычи, выявленных у уховерток и пентатомидных клопов, было относительно низким. Lepidoptera , Hemiptera , Diptera и Coleoptera были наиболее распространенными таксонами насекомых-жертв, обнаруженными у хищных членистоногих. [77]
Сохранение функционального биоразнообразия и связанных с ним экосистемных услуг , особенно путем борьбы с вредителями с помощью их естественных врагов, открывает новые возможности для решения проблем устойчивой интенсификации систем производства продуктов питания. [88] [89] [90] Хищничество в отношении вредителей сельскохозяйственных культур со стороны хищников-универсалов, включая членистоногих и позвоночных, является основным компонентом естественной борьбы с вредителями . [91] Особенно важной чертой большинства хищников-универсалов является то, что они могут колонизировать посевы в начале сезона, сначала питаясь альтернативной добычей. [92] [93] Однако широта «универсальной» диеты влечет за собой некоторые недостатки в борьбе с вредителями, такие как хищничество внутри гильдии. [91] [94] [95] Таким образом, для лучшего распутывания пищевых цепочек (например, конкуренции за эксплуатацию и очевидной конкуренции) и, в конечном итоге, для определения ключевых факторов естественной борьбы с вредителями в агроэкосистемах необходима точная диагностика широты рациона хищников-универсалов, включая хищничество жертв, не являющихся вредителями. Однако важность хищников-универсалов в пищевой сети, как правило, трудно оценить из-за эфемерного характера индивидуальных взаимодействий хищник-жертва. [94] [96] Единственное убедительное свидетельство хищничества является прямым наблюдением за потреблением добычи, выявлением остатков добычи в кишках хищников. [94] и анализы срыгиваний [97] или фекалии. [98] [77]
Морская биобезопасность
[ редактировать ]
Распространение неместных видов (ННГ) представляет собой значительный и растущий риск для экосистем. [100] В морских системах ННГ, которые выживают при транспортировке и адаптируются к новым местам, могут оказывать существенное неблагоприятное воздействие на местное биоразнообразие, включая вытеснение местных видов, а также сдвиги в биологических сообществах и связанных с ними пищевых цепях. [101] [102] После того как ННГ возникли, искоренить их чрезвычайно сложно и дорого. [103] [104] дальнейшее региональное распространение может происходить за счет естественного расселения или антропогенных путей переноса. [104] [105] [106] Хотя обрастание корпусов судов и балластные воды судов хорошо известны как важные антропогенные пути международного распространения ННГ, [100] [107] [108] [109] Сравнительно мало известно о том, что региональные транзитные суда могут способствовать вторичному распространению морских вредителей посредством перемещения льяльных вод. [99]
Недавние исследования показали, что вода и связанный с ней мусор, попавшие в трюмные помещения небольших судов (менее 20 м), могут выступать в качестве вектора распространения ННГ в региональных масштабах. [110] [111] [112] [113] [114] [115] Трюмная вода определяется как любая вода, которая остается на судне (кроме балласта) и которая не закачивается на борт намеренно. Он может накапливаться на палубе судна или под ней (например, под панелями пола) в результате различных механизмов, включая воздействие волн, утечек, через кормовые сальники гребного винта, а также при загрузке такого оборудования, как оборудование для дайвинга, рыбалки, аквакультуры или научного оборудования. . [116] Таким образом, льяльные воды могут содержать морскую воду, а также живые организмы на различных стадиях жизни, клеточный мусор и загрязняющие вещества (например, масло, грязь, моющие средства и т. д.), все из которых обычно сбрасываются с помощью автоматических трюмных насосов или самоосушаются. с использованием клапанов-утконосов. Трюмная вода, откачиваемая из небольших судов (вручную или автоматически), обычно не очищается перед сбросом в море, в отличие от более крупных судов, которые требуют разделения нефти и воды с использованием систем фильтрации, центрифугирования или абсорбции углерода. [116] [117] Если в результате этого процесса размножения жизнеспособны, сброс льяльных вод может привести к распространению НИС. [99]
В 2017 году Флетчер и др. использовали комбинацию лабораторных и полевых экспериментов для изучения разнообразия, численности и выживаемости биологического материала, содержащегося в пробах льяльных вод, взятых с небольших прибрежных судов. [115] Их лабораторный эксперимент показал, что асцидий колонии или фрагменты , а также личинки мшанок могут выжить при прохождении через нефильтрованную насосную систему, практически не повредившись. Они также провели первую морфомолекулярную оценку (с использованием метабаркодирования eDNA) риска биобезопасности, создаваемого сбросами льяльных вод с 30 малых судов (парусников и моторных лодок) различного происхождения и времени плавания. С помощью метабаркодирования eDNA они охарактеризовали примерно в три раза больше таксонов, чем с помощью традиционных микроскопических методов, включая обнаружение пяти видов, признанных неместными для исследуемого региона. [99]
Чтобы помочь понять риски, связанные с различными векторами интродукции ННГ, традиционные оценки биоразнообразия с помощью микроскопа все чаще дополняются метабаркодированием эДНК. [118] [119] [120] [121] Это позволяет идентифицировать широкий спектр разнообразных таксономических комплексов на многих стадиях жизни. Это также позволяет обнаруживать NIS, которые могли быть не замечены традиционными методами. Несмотря на большой потенциал инструментов метабаркодирования eDNA для широкомасштабного таксономического скрининга, [122] [123] ключевой задачей для eDNA в контексте экологического мониторинга морских вредителей, и особенно при мониторинге закрытых сред, таких как некоторые трюмные помещения или балластные цистерны, является дифференциация мертвых и жизнеспособных организмов. [124] Внеклеточная ДНК может сохраняться в темной/холодной среде в течение длительных периодов времени (от месяцев до лет, [125] [126] таким образом, многие из организмов, обнаруженных с помощью метабаркодирования эДНК, могли быть нежизнеспособными в месте сбора образцов в течение нескольких дней или недель. Напротив, рибонуклеиновая кислота (РНК) быстро разрушается после гибели клеток, что, вероятно, обеспечивает более точное представление о жизнеспособных сообществах. [127] Недавние исследования метабаркодирования изучили использование совместно экстрагированных молекул эДНК и эРНК для мониторинга образцов донных отложений вокруг морских рыбных ферм и мест бурения нефтяных скважин. [128] [129] [130] [131] [132] и коллективно обнаружили несколько более сильные корреляции между биологическими и физико-химическими переменными вдоль градиентов воздействия при использовании eRNA. С точки зрения морской биобезопасности обнаружение живых ННГ может представлять собой более серьезную и непосредственную угрозу, чем обнаружение ННГ, основанное исключительно на сигнале ДНК. Таким образом, РНК окружающей среды может стать полезным методом идентификации живых организмов в образцах. [99]
Разнообразный
[ редактировать ]Создание библиотеки генетических штрих-кодов изначально было ориентировано на рыбу. [133] и птицы, [134] [135] [136] за которыми следовали бабочки и другие беспозвоночные. [137] У птиц образец ДНК обычно берут из грудной клетки.
Исследователи уже разработали специальные каталоги для крупных групп животных, таких как пчелы, птицы, млекопитающие или рыбы. Другое использование - анализ полного зооценоза данной географической области, например, проект «Штрих-код полярной жизни», целью которого является сбор генетических особенностей всех организмов, обитающих в полярных регионах; оба полюса Земли. С этой формой связана кодировка всей ихтиофауны гидрографического бассейна, например того, который начал развиваться в реке Рио-Сан-Франциско, на северо-востоке Бразилии . [138] [139]
Потенциал использования штрих-кодов очень широк, поскольку открытие многочисленных загадочных видов (это уже дало многочисленные положительные результаты), [140] использование при идентификации видов на любом этапе их жизни, надежная идентификация в случаях незаконного оборота охраняемых видов и т.д. [141] [142]
Он также использовался в качестве неинвазивного инструмента для определения рациона диких животных, таких как вомбаты. [143] и особо находящиеся под угрозой исчезновения виды, такие как северный волосатый вомбат ( Lasiorhinus krefftii ). [144]
Потенциал и недостатки
[ редактировать ]
Потенциалы
[ редактировать ]Штрих-кодирование ДНК было предложено как способ различения видов, который может использовать даже неспециалист. [145]
Недостатки
[ редактировать ]В целом недостатки штрих-кодирования ДНК справедливы и для метабаркодирования. Одним из особых недостатков исследований метабаркодирования является отсутствие единого мнения относительно оптимального плана эксперимента и критериев биоинформатики, которые будут применяться при метабаркодировании эДНК. [146] Однако в настоящее время предпринимаются совместные попытки, такие как сеть COST DNAqua-Net Европейского сотрудничества в области науки и технологий , [147] двигаться вперед путем обмена опытом и знаниями для установления передовых стандартов биомониторинга. [148]
Так называемый штрих-код — это участок митохондриальной ДНК внутри гена цитохром-с-оксидазы . База данных Barcode of Life Data Systems (BOLD) содержит последовательности штрих-кодов ДНК более чем 190 000 видов. [149] [150] Однако такие ученые, как Роб ДеСалле, выразили обеспокоенность тем, что классическую таксономию и штрих-кодирование ДНК, которые они считают неправильным, необходимо согласовать, поскольку они по-разному разграничивают виды. [151] Генетическая интрогрессия, опосредованная эндосимбионтами и другими векторами, может еще больше сделать штрих-коды неэффективными для идентификации видов. [152]
Статус видов штрих-кодов
[ редактировать ]В микробиологии гены могут свободно перемещаться даже между отдаленно родственными бактериями, возможно, распространяясь на весь бактериальный домен. Как правило, микробиологи предполагают, что виды бактерий или архей с последовательностями генов 16S рибосомальной РНК, более похожими друг на друга более чем на 97%, необходимо проверять с помощью гибридизации ДНК-ДНК, чтобы решить, принадлежат ли они к одному и тому же виду или нет. [153] Это понятие было сужено в 2006 году до сходства 98,7%. [154]
Гибридизация ДНК-ДНК устарела, и ее результаты иногда приводили к вводящим в заблуждение выводам о видах, как, например, в случае с крупным поморником и большим поморником . [155] [156] Современные подходы сравнивают сходство последовательностей с использованием вычислительных методов. [157]
См. также
[ редактировать ]- Система данных «Штрих-код жизни » (жирный шрифт)
- Консорциум штрих-кода жизни (CBOL)
- Международное сотрудничество по базам данных нуклеотидных последовательностей (INSDC)
- Молекулярный маркер
- Таксономическое препятствие
Ссылки
[ редактировать ]- ^ Пьер, Таберле (2 февраля 2018 г.). Экологическая ДНК: для исследования и мониторинга биоразнообразия . Бонен, Орели, 1979-. Оксфорд. ISBN 9780191079993 . OCLC 1021883023 .
{{cite book}}: CS1 maint: отсутствует местоположение издателя ( ссылка ) - ^ Перейти обратно: а б Хеберт, Пол Д.Н.; Цивинская, Алина; Болл, Шелли Л.; Деваард, Джереми Р. (2003). «Биологическая идентификация посредством штрих-кодов ДНК» . Труды Лондонского королевского общества. Серия Б: Биологические науки . 270 (1512): 313–321. дои : 10.1098/рспб.2002.2218 . ПМЦ 1691236 . ПМИД 12614582 .
- ^ Фичетола, Джентиле Франческо; Мио, Клод; Помпанон, Франсуа; Таберле, Пьер (2008). «Обнаружение видов с использованием ДНК окружающей среды из проб воды» . Письма по биологии . 4 (4): 423–425. дои : 10.1098/rsbl.2008.0118 . ПМК 2610135 . ПМИД 18400683 .
- ^ Перейти обратно: а б с Барнс, Мэтью А.; Тернер, Кэмерон Р. (2016). «Экология ДНК окружающей среды и значение для сохранения генетики» . Сохраняющая генетика . 17 : 1–17. дои : 10.1007/s10592-015-0775-4 . hdl : 2346/87600 . S2CID 14914544 .
- ^ Дайнер, Кристи; Вальзер, Жан-Клод; Мехлер, Эльвира; Альтерматт, Флориан (2015). «Выбор методов улавливания и извлечения влияет на выявление пресноводного биоразнообразия по ДНК окружающей среды» . Биологическая консервация . 183 : 53–63. дои : 10.1016/j.biocon.2014.11.018 .
- ^ Перейти обратно: а б Таберле, Пьер; Куассак, Эрик; Помпанон, Франсуа; Брохманн, Кристиан; Виллерслев, Эске (2012). «На пути к оценке биоразнообразия следующего поколения с использованием метабаркодирования ДНК». Молекулярная экология . 21 (8): 2045–2050. дои : 10.1111/j.1365-294X.2012.05470.x . ПМИД 22486824 . S2CID 41437334 .
- ^ Перейти обратно: а б Боманн, Кристина; Эванс, Элис; Гилберт, М. Томас П.; Карвальо, Гэри Р.; Крир, Саймон; Кнапп, Майкл; Ю, Дуглас В.; Де Брюин, Марк (2014). «Экологическая ДНК для биологии дикой природы и мониторинга биоразнообразия». Тенденции в экологии и эволюции . 29 (6): 358–367. дои : 10.1016/j.tree.2014.04.003 . ПМИД 24821515 .
- ^ Голдберг, Карен С.; Тернер, Кэмерон Р.; Дайнер, Кристи; Климус, Кэти Э.; Томсен, Филип Фрэнсис; Мерфи, Мелани А.; Спир, Стивен Ф.; Макки, Анна; Ойлер-Макканс, Сара Дж.; Корнман, Роберт Скотт; Ларами, Мэтью Б.; Махон, Эндрю Р.; Лэнс, Ричард Ф.; Пиллиод, Дэвид С.; Стриклер, Кэтрин М.; Уэйтс, Лизетт П.; Фремьер, Александр К.; Такахара, Терухико; Гердер, Джелгер Э.; Таберле, Пьер (2016). «Важные соображения по применению методов ДНК окружающей среды для обнаружения водных видов» . Методы экологии и эволюции . 7 (11): 1299–1307. дои : 10.1111/2041-210X.12595 . hdl : 20.500.11850/502281 .
- ^ Перейти обратно: а б с Геринг, Дэниел; Борха, Ангел; Джонс, Дж.Иван; Пон, Дидье; Боэтс, Питер; Буше, Агнес; Брюс, Кэт; Дракаре, Стина; Хэнфлинг, Бернд; Калерт, Мария; Лиз, Флориан; Мейснер, Кристиан; Мерген, Патрисия; Рейджол, Йорик; Сегурадо, Педро; Фоглер, Альфрид; Келли, Мартин (2018). «Варианты внедрения идентификации на основе ДНК в оценку экологического статуса в соответствии с Европейской водной рамочной директивой» . Исследования воды . 138 : 192–205. Бибкод : 2018WatRe.138..192H . дои : 10.1016/j.watres.2018.03.003 . ПМИД 29602086 . S2CID 5008250 .
- ^ Перейти обратно: а б с д и Руперт, Криста М.; Клайн, Ричард Дж.; Рахман, доктор Сайдур (2019). «Прошлые, настоящие и будущие перспективы метабаркодирования ДНК окружающей среды (EDNA): систематический обзор методов, мониторинга и применения глобальной eDNA» . Глобальная экология и охрана природы . 17 : e00547. дои : 10.1016/j.gecco.2019.e00547 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0. - ^ Перейти обратно: а б Куассак, Эрик; Риаз, Тиайба; Пуйландр, Николя (2012). «Биоинформатические задачи метабаркодирования ДНК растений и животных» . Молекулярная экология . 21 (8): 1834–1847. дои : 10.1111/j.1365-294X.2012.05550.x . ПМИД 22486822 . S2CID 24398174 .
- ^ Перейти обратно: а б с д и ж г Крир, Саймон; Дайнер, Кристи; Фрей, Серита ; Поразинска, Дорота; Таберле, Пьер; Томас, В. Келли; Поттер, Кейтлин; Бик, Холли М. (2016). «Полевое руководство эколога по последовательной идентификации биоразнообразия» . Методы экологии и эволюции . 7 (9): 1008–1018. дои : 10.1111/2041-210X.12574 . hdl : 20.500.11850/125378 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0. - ^ Перейти обратно: а б с д и ж г час я Дайнер, Кристи; Бик, Холли М.; Мехлер, Эльвира; Сеймур, Мэтью; Лакурсьер-Руссель, Анаис; Альтерматт, Флориан; Крир, Саймон; Биста, Илиана; Лодж, Дэвид М.; Вере, Наташа; Пфрендер, Майкл Э.; Бернатчес, Луи (2017). «Метабаркодирование ДНК окружающей среды: изменение методов исследования сообществ животных и растений» . Молекулярная экология . 26 (21): 5872–5895. дои : 10.1111/mec.14350 . hdl : 20.500.11850/455284 . ПМИД 28921802 . S2CID 8001074 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0. - ^ Фичетола, Джентиле Франческо; Таберле, Пьер; Куассак, Эрик (2016). «Как ограничить ложные срабатывания ДНК окружающей среды и метабаркодирования?» . Ресурсы молекулярной экологии . 16 (3): 604–607. дои : 10.1111/1755-0998.12508 . ПМИД 27062589 . S2CID 785279 .
- ^ Ю, Дуглас В.; Цзи, Иньцю; Эмерсон, Брент К.; Ван, Сяоян; Е, Чэнси; Ян, Чунянь; Дин, Чжаоли (2012). «Суп биоразнообразия: метабаркодирование членистоногих для быстрой оценки биоразнообразия и биомониторинга» . Методы экологии и эволюции . 3 (4): 613–623. дои : 10.1111/j.2041-210X.2012.00198.x . hdl : 10261/193380 .
- ^ Кристеску, Мелания Э. (2014). «От штрих-кодирования отдельных людей к метабаркодированию биологических сообществ: на пути к интегративному подходу к изучению глобального биоразнообразия». Тенденции в экологии и эволюции . 29 (10): 566–571. дои : 10.1016/j.tree.2014.08.001 . ПМИД 25175416 . S2CID 18352572 .
- ^ Гибсон, Джоэл Ф.; Шокралла, Шади; Карри, Колин; Бэрд, Дональд Дж.; Монк, Венди А.; Кинг, Ян; Хаджибабаи, Мехрдад (2015). «Крупномасштабный биомониторинг удаленных и находящихся под угрозой исчезновения экосистем посредством высокопроизводительного секвенирования» . ПЛОС ОДИН . 10 (10): e0138432. Бибкод : 2015PLoSO..1038432G . дои : 10.1371/journal.pone.0138432 . ПМЦ 4619546 . ПМИД 26488407 .
- ^ Чуа, Физилия Ю.С.; Бурла, Сара Дж.; Фергюсон, Кэмерон; Корлевич, Петра; Чжао, Лея; Экрем, Торбьёрн; Мейер, Рудольф; Лавничак, Мара К.Н. (10 марта 2023 г.). «Будущее мониторинга насекомых на основе ДНК». Тенденции в генетике . 39 (7): 531–544. дои : 10.1016/j.tig.2023.02.012 . ПМИД 36907721 . S2CID 257470926 .
- ^ Маргулис, Марсель; и др. (2005). «Секвенирование генома в микропроизводных пиколитровых реакторах высокой плотности» . Природа . 437 (7057): 376–380. Бибкод : 2005Natur.437..376M . дои : 10.1038/nature03959 . ПМЦ 1464427 . ПМИД 16056220 .
- ^ Хаджибабаи, Мехрдад; Шокралла, Шади; Чжоу, Синь; Певец, Грегори AC; Бэрд, Дональд Дж. (2011). «Экологическое штрих-кодирование: подход к секвенированию нового поколения для приложений биомониторинга с использованием речного бентоса» . ПЛОС ОДИН . 6 (4): e17497. Бибкод : 2011PLoSO...617497H . дои : 10.1371/journal.pone.0017497 . ПМК 3076369 . ПМИД 21533287 .
- ^ Бенсон, Деннис А.; Кавано, Марк; Кларк, Карен; Карш-Мизрачи, Илен; Липман, Дэвид Дж.; Остелл, Джеймс; Сэйерс, Эрик В. (2012). «Ген Банк » . Исследования нуклеиновых кислот . 41 (Проблема с базой данных): D36–D42. дои : 10.1093/nar/gks1195 . ПМК 3531190 . ПМИД 23193287 .
- ^ Прюсс, Э.; Кваст, К.; Книттель, К.; Фукс, Б.М.; Людвиг, В.; Пеплиес, Дж.; Глокнер, Ф.О. (2007). «SILVA: комплексный онлайн-ресурс для проверенных и согласованных по качеству данных о последовательностях рибосомальных РНК, совместимых с ARB» . Исследования нуклеиновых кислот . 35 (21): 7188–7196. дои : 10.1093/нар/gkm864 . ПМЦ 2175337 . ПМИД 17947321 .
- ^ о'Доннелл, Джеймс Л.; Келли, Райан П.; Лоуэлл, Натали С.; Порт, Джесси А. (2016). «Индексированные праймеры для ПЦР вызывают смещение, специфичное для матрицы, в крупномасштабных исследованиях секвенирования ДНК» . ПЛОС ОДИН . 11 (3): e0148698. Бибкод : 2016PLoSO..1148698O . дои : 10.1371/journal.pone.0148698 . ПМЦ 4780811 . ПМИД 26950069 .
- ^ Сёнстебё, Дж. Х.; Джелли, Л.; Бристинг, АК; Эльф, Р.; Эдвардс, М.; Хейл, Дж.; Виллерслев, Э.; Куассак, Э.; Риу, Д.; Саннье, Дж.; Таберлет, П.; Брохманн, К. (2010). «Использование секвенирования нового поколения для молекулярной реконструкции прошлой арктической растительности и климата». Ресурсы молекулярной экологии . 10 (6): 1009–1018. дои : 10.1111/j.1755-0998.2010.02855.x . ПМИД 21565110 . S2CID 23029234 .
- ^ Виллерслев, Эске; и др. (2014). «Пятьдесят тысяч лет арктической растительности и мегафауны» (PDF) . Природа . 506 (7486): 47–51. Бибкод : 2014Natur.506...47W . дои : 10.1038/nature12921 . ПМИД 24499916 . S2CID 4461741 .
- ^ Блакстер, Марк; Манн, Дженна; Чепмен, Том; Томас, Фрэн; Уиттон, Клэр; Флойд, Робин; Абебе, Эйуалем (2005). «Определение оперативных таксономических единиц с использованием данных штрих-кода ДНК» . Философские труды Королевского общества B: Биологические науки . 360 (1462): 1935–1943. дои : 10.1098/rstb.2005.1725 . ПМК 1609233 . ПМИД 16214751 .
- ^ Дайнер, Кристи; Альтерматт, Флориан (2014). «Расстояние транспортировки ДНК беспозвоночных из окружающей среды в естественной реке» . ПЛОС ОДИН . 9 (2): е88786. Бибкод : 2014PLoSO...988786D . дои : 10.1371/journal.pone.0088786 . ПМК 3921251 . ПМИД 24523940 .
- ^ Меркес, Кристофер М.; МакКалла, С. Грейс; Дженсен, Натан Р.; Гайковски, Марк П.; Амберг, Джон Дж. (2014). «Стойкость ДНК в тушах, слизи и птичьих фекалиях может повлиять на интерпретацию данных ДНК из окружающей среды» . ПЛОС ОДИН . 9 (11): e113346. Бибкод : 2014PLoSO...9k3346M . дои : 10.1371/journal.pone.0113346 . ПМЦ 4234652 . ПМИД 25402206 .
- ^ Йоккоз, штат Нью-Йорк; Братен, Калифорния; Джелли, Л.; Хейл, Дж.; Эдвардс, Мэн; Гослар, Т.; фон Стедингк, Х.; Бристинг, АК; Куассак, Э.; Помпанон, Ф.; Сёнстебё, Дж. Х.; Микель, К.; Валентини, А.; Де Белло, Ф.; Чаве, Дж.; Туиллер, В.; Винкер, П.; Круо, К.; Гавори, Ф.; Расмуссен, М.; Гилберт, MTP; Орландо, Л.; Брохманн, К.; Виллерслев, Э.; Таберлет, П. (2012). «ДНК из почвенных зеркал таксономического разнообразия и форм роста растений». Молекулярная экология . 21 (15): 3647–3655. дои : 10.1111/j.1365-294X.2012.05545.x . ПМИД 22507540 .
- ^ Перейти обратно: а б с д и ж г час я дж Хаарсма, Анне-Йифке; Сипель, Хенк; Гравендил, Барбара (2016). «Дополнительная ценность метабаркодирования в сочетании с микроскопией для эволюционных исследований млекопитающих» . Зоологика Скрипта . 45 : 37–49. дои : 10.1111/zsc.12214 . hdl : 2066/161842 . S2CID 89048681 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0. - ^ Кресс, В. Джон; Гарсия-Робледо, Карлос; Уриарте, Мария; Эриксон, Дэвид Л. (2015). «Штрих-коды ДНК для экологии, эволюции и сохранения». Тенденции в экологии и эволюции . 30 (1): 25–35. дои : 10.1016/j.tree.2014.10.008 . ПМИД 25468359 .
- ^ Галан, Максим; Пажес, Мари; Коссон, Жан-Франсуа (2012). «Секвенирование нового поколения для штрих-кодирования грызунов: идентификация видов по свежим, деградированным образцам и образцам из окружающей среды» . ПЛОС ОДИН . 7 (11): е48374. Бибкод : 2012PLoSO...748374G . дои : 10.1371/journal.pone.0048374 . ПМЦ 3492341 . ПМИД 23144869 .
- ^ Иванова Наталья Владимировна; Клэр, Элизабет Л.; Борисенко, Алексей В. (2012). «Штрих-кодирование ДНК у млекопитающих». ДНК-штрих-коды . Методы молекулярной биологии. Том. 858. стр. 153–182. дои : 10.1007/978-1-61779-591-6_8 . ISBN 978-1-61779-590-9 . ПМИД 22684956 .
- ^ Клэр, Элизабет Л. (2014). «Молекулярное обнаружение трофических взаимодействий: новые тенденции, явные преимущества, важные соображения и способы сохранения» . Эволюционные приложения . 7 (9): 1144–1157. дои : 10.1111/eva.12225 . ПМК 4231602 . ПМИД 25553074 .
- ^ Орландо, Людовик; Ханни, Кэтрин; Дуади, Кристоф Дж. (2007). «Филогенетические отношения мамонта и слона: Mammut Americanum, пропавшая внешняя группа» . Эволюционная биоинформатика . 3 : 45–51. дои : 10.1177/117693430700300019 . ПМЦ 2674638 . ПМИД 19430604 .
- ^ Перейти обратно: а б Помпанон, Франсуа; Дигл, Брюс Э.; Симондсон, Уильям О.К.; Браун, Дэвид С.; Джарман, Саймон Н.; Таберле, Пьер (2012). «Кто что ест: оценка диеты с использованием секвенирования следующего поколения» . Молекулярная экология . 21 (8): 1931–1950. дои : 10.1111/j.1365-294X.2011.05403.x . ПМИД 22171763 . S2CID 10013333 .
- ^ Дигл, Брюс Э.; Джарман, Саймон Н.; Куассак, Эрик; Помпанон, Франсуа; Таберле, Пьер (2014). «Метабаркодирование ДНК и маркер субъединицы I цитохром с-оксидазы: не идеальное совпадение» . Письма по биологии . 10 (9). дои : 10.1098/rsbl.2014.0562 . ПМК 4190964 . ПМИД 25209199 .
- ^ Даливал, Анандика (2013). «Выделение и очистка ДНК». Материалы и методы . 3 . дои : 10.13070/mm.en.3.191 .
- ^ Купер, А.; Пойнар, Х.Н. (2000). «Древняя ДНК: делай это правильно или не делай вообще». Наука . 289 (5482): 1139б–1139. дои : 10.1126/science.289.5482.1139b . ПМИД 10970224 . S2CID 11030200 .
- ^ Виллерслев, Эске; Купер, Алан (2005). «Обзорная статья. Древняя ДНК» . Труды Королевского общества B: Биологические науки . 272 (1558): 3–16. дои : 10.1098/rspb.2004.2813 . ПМЦ 1634942 . ПМИД 15875564 .
- ^ Фичетола, Джентиле Ф.; Пансу, Йохан; Бонен, Орели; Куассак, Эрик; Жиге-Ковекс, Шарлин; Де Барба, Марта; Джелли, Людовик; Лопес, Карла М.; Бойер, Фредерик; Помпанон, Франсуа; Райе, Жиль; Таберле, Пьер (2015). «Уровни репликации, ложное присутствие и оценка присутствия/отсутствия на основе данных метабаркодирования eDNA». Ресурсы молекулярной экологии . 15 (3): 543–556. дои : 10.1111/1755-0998.12338 . ПМИД 25327646 . S2CID 24432585 .
- ^ Ян, Аймин; Чжан, Вэй; Ван, Цзяхао; Ян, Кэ; Хан, Ян; Чжан, Лимин (2020). «Обзор применения алгоритмов машинного обучения для анализа последовательностей ДНК» . Границы биоинженерии и биотехнологии . 8 : 1032. дои : 10.3389/fbioe.2020.01032 . ПМЦ 7498545 . ПМИД 33015010 .
- ^ Шендюр, Джей; Эйден, Эрез Либерман (2012). «Расширяющиеся возможности секвенирования ДНК» . Природная биотехнология . 30 (11): 1084–1094. дои : 10.1038/nbt.2421 . ПМК 4149750 . ПМИД 23138308 .
- ^ Панда, Даршан; Молла, Кутубуддин Али; Байг, Мирза Джайнул; Суэйн, Алака; Бехера, Диптирекха; Даш, Манасвини (2018). «ДНК как устройство хранения цифровой информации: надежда или обман?» , 3 Биотехнологии . 8 (5): 239. дои : 10.1007/s13205-018-1246-7 . ПМЦ 5935598 . ПМИД 29744271 .
- ^ Ратнасингем, С. (2013). «Реестр всех видов животных на основе ДНК: система индексного номера штрих-кода (BIN)» . ПЛОС ОДИН . 8 (7): e66213. Бибкод : 2013PLoSO...866213R . дои : 10.1371/journal.pone.0066213 . ПМК 3704603 . ПМИД 23861743 .
- ^ Нельсон, Карен Э. (3 января 2015 г.). Энциклопедия метагеномики: гены, геномы и метагеномы. Основы, методы, базы данных и инструменты . Спрингер. ISBN 9781489974778 .
- ^ Абаренков, Кесси; Хенрик Нильссон, Р.; Ларссон, Карл-Хенрик; Александр, Ян Дж.; Эберхардт, Урсула; Эрланд, Сюзанна; Хайленд, Клаус; Хьёллер, Расмус; Ларссон, Эллен; Пеннанен, Тайна; Сен, Робин; Тейлор, Энди Ф.С.; Тедерсоо, Лехо; Урсинг, Бьёрн М.; Вролстад, Труде; Лииматайнен, Каре; Пейнтнер, Урсула; Кылялг, Урмас (2010). «База данных UNITE для молекулярной идентификации грибов – последние обновления и перспективы на будущее» . Новый фитолог . 186 (2): 281–285. дои : 10.1111/j.1469-8137.2009.03160.x . ПМИД 20409185 .
- ^ Капорасо, Дж. Грегори; Кучински, Джастин; Стомбо, Джесси; Биттингер, Кайл; Бушман, Фредерик Д.; Костелло, Элизабет К.; Фирер, Ной; Пенья, Антонио Гонсалес; Гудрич, Джулия К.; Гордон, Джеффри И.; Хаттли, Гэвин А.; Келли, Скотт Т.; Найтс, Дэн; Кениг, Джереми Э.; Лей, Рут Э.; Лозупоне, Кэтрин А.; Макдональд, Дэниел; Мюгге, Брайан Д.; Пиррунг, Мэг; Ридер, Йенс; Севински, Джоэл Р.; Тернбо, Питер Дж.; Уолтерс, Уильям А.; Видманн, Джереми; Яцуненко Таня; Заневельд, Джесси; Найт, Роб (2010). «QIIME позволяет анализировать данные общественного секвенирования с высокой пропускной способностью» . Природные методы . 7 (5): 335–336. дои : 10.1038/nmeth.f.303 . ПМК 3156573 . ПМИД 20383131 .
- ^ Асникар, Франческо; Вайнгарт, Джордж; Тикл, Тимоти Л.; Хаттенхауэр, Кертис; Сегата, Никола (2015). «Компактное графическое представление филогенетических данных и метаданных с помощью GraPhl An » . ПерДж . 3 : е1029. дои : 10.7717/peerj.1029 . ПМЦ 4476132 . ПМИД 26157614 .
- ^ Мацен Ив, Фредерик А.; Эванс, Стивен Н. (2013). «Главные краевые компоненты и сквош-кластеризация: использование специальной структуры данных филогенетического размещения для сравнения образцов» . ПЛОС ОДИН . 8 (3): e56859. Бибкод : 2013PLoSO...856859M . дои : 10.1371/journal.pone.0056859 . ПМЦ 3594297 . ПМИД 23505415 .
- ^ Дарлинг, Аарон Э.; Жоспен, Гийом; Лоу, Эрик; Матсен, Фредерик А.; Бик, Холли М.; Эйзен, Джонатан А. (2014). «Phylo Sift : Филогенетический анализ геномов и метагеномов» . ПерДж . 2 : е243. дои : 10.7717/peerj.243 . ПМЦ 3897386 . ПМИД 24482762 .
- ^ Стокл, М.Ю. и Хеберт, П.Д. (2008). Штрих-код жизни. Исследования и наука, (387), 42-47.
- ^ Ньюмастер С.Г. и др. (2007). Тестирование областей штрих-кода растений-кандидатов Myristicaceae. Заметки по молекулярной экологии. 1-11.
- ^ Хаэн-Молина, Р., Кожапе-Кастельс, Дж., Фернандес-Паласиос, О., де Пас, Дж. П., Феблес, Р., Брамвелл, Д., ... и Халик, К. А. Молекулярная филогения макаронезийских Matthioleae по данным региона ИТС.
- ^ Перейти обратно: а б с д и Порнон, Эндрю; Андало, Кристоф; Буррус, Моник; Эскаравадж, Натали (2017). «Данные метабаркодирования ДНК раскрывают невидимые сети опыления» . Научные отчеты . 7 (1): 16828. Бибкод : 2017НатСР...716828П . дои : 10.1038/s41598-017-16785-5 . ПМК 5715002 . ПМИД 29203872 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0. - ^ Бисмейер, Дж. К.; Робертс, СП; Ример, М.; Олемюллер, Р.; Эдвардс, М.; Питерс, Т.; Шафферс, AP; Поттс, СГ; Клейкерс, Р.; Томас, CD; Сеттеле, Дж.; Кунин, МЫ (2006). «Параллельное сокращение количества опылителей и насекомоопыляемых растений в Великобритании и Нидерландах». Наука . 313 (5785): 351–354. Бибкод : 2006Sci...313..351B . дои : 10.1126/science.1127863 . ПМИД 16857940 . S2CID 16273738 .
- ^ Перейти обратно: а б Хегланд, Штейн Джоар; Данн, Дженнифер; Нильсен, Андерс; Меммотт, Джейн (2010). «Как экономически эффективно контролировать экологические сообщества: пример сетей растений и опылителей». Биологическая консервация . 143 (9): 2092–2101. doi : 10.1016/j.biocon.2010.05.018 .
- ^ Перейти обратно: а б Джордано, Педро (2016). «В погоне за экологическими взаимодействиями» . ПЛОС Биология . 14 (9): e1002559. дои : 10.1371/journal.pbio.1002559 . ПМК 5025190 . ПМИД 27631692 .
- ^ Перейти обратно: а б Васкес, Диего П.; Чакофф, Наташа П.; Каньоло, Лучано (2009). «Оценка множества факторов, определяющих структуру мутуалистических сетей растений и животных». Экология . 90 (8): 2039–2046. дои : 10.1890/08-1837.1 . HDL : 11336/20750 . ПМИД 19739366 .
- ^ Перейти обратно: а б Бартомеус, Игнаси (2013). «Понимание правил связи в сетях растения-опылители с помощью иерархических моделей, включающих обнаруживаемость опылителей и свойства растений» . ПЛОС ОДИН . 8 (7): е69200. Бибкод : 2013PLoSO...869200B . дои : 10.1371/journal.pone.0069200 . ПМЦ 3698228 . ПМИД 23840909 .
- ^ Перейти обратно: а б Блютген, Нико; Мензель, Флориан; Ховестадт, Томас; Фиала, Бриджит; Блютген, Нильс (2007). «Специализация, ограничения и конфликт интересов во мутуалистических сетях» . Современная биология . 17 (4): 341–346. дои : 10.1016/j.cub.2006.12.039 . PMID 17275300 . S2CID 17241040 .
- ^ Баскомпт, Дж.; Джордано, П.; Мелиан, CJ; Олесен, Дж. М. (2003). «Вложенная сборка мутуалистических сетей растений и животных» . Труды Национальной академии наук . 100 (16): 9383–9387. Бибкод : 2003PNAS..100.9383B . дои : 10.1073/pnas.1633576100 . ПМК 170927 . ПМИД 12881488 .
- ^ Джордано, Педро; Баскомпт, Хорди; Олесен, Йенс М. (2002). «Инвариантные свойства в коэволюционных сетях взаимодействий растений и животных». Экологические письма . 6 : 69–81. дои : 10.1046/j.1461-0248.2003.00403.x . hdl : 10261/41710 .
- ^ Олесен, Дж. М.; Баскомпт, Дж.; Дюпон, ЮЛ; Джордано, П. (2007). «Модульность сетей опыления» . Труды Национальной академии наук . 104 (50): 19891–19896. Бибкод : 2007PNAS..10419891O . дои : 10.1073/pnas.0706375104 . ПМК 2148393 . ПМИД 18056808 .
- ^ Оллертон, Джефф (1996). «Согласование экологических процессов с филогенетическими закономерностями: очевидный парадокс систем растения-опылители». Журнал экологии . 84 (5): 767–769. дои : 10.2307/2261338 . JSTOR 2261338 .
- ^ Джонсон, Стивен Д.; Штайнер, Ким Э. (2000). «Обобщение против специализации в системах опыления растений». Тенденции в экологии и эволюции . 15 (4): 140–143. дои : 10.1016/S0169-5347(99)01811-X . ПМИД 10717682 .
- ^ Васер, Николас М.; Читтка, Ларс; Прайс, Мэри В.; Уильямс, Нил М.; Оллертон, Джефф (1996). «Обобщение в системах опыления и почему это важно». Экология . 77 (4): 1043–1060. дои : 10.2307/2265575 . JSTOR 2265575 .
- ^ Фенстер, Чарльз Б.; Армбрустер, В. Скотт; Уилсон, Пол; Дудаш, Мишель Р.; Томсон, Джеймс Д. (2004). «Синдромы опыления и специализация цветов». Ежегодный обзор экологии, эволюции и систематики . 35 : 375–403. doi : 10.1146/annurev.ecolsys.34.011802.132347 .
- ^ Босх, Джорди; Мартин Гонсалес, Ана М.; Родриго, Ансельм; Наварро, Дэвид (2009). «Сети растений-опылителей: добавление точки зрения опылителя». Экологические письма . 12 (5): 409–419. дои : 10.1111/j.1461-0248.2009.01296.x . ПМИД 19379135 .
- ^ Видмер, А.; Коццолино, С.; Пеллегрино, Дж.; Солива, М.; Дафни, А. (2000). «Молекулярный анализ орхидейных поллинариев и остатков поллинариев, обнаруженных на насекомых». Молекулярная экология . 9 (11): 1911–1914. дои : 10.1046/j.1365-294x.2000.01103.x . ПМИД 11091327 . S2CID 13242534 .
- ^ Петаниду, Т. и Поттс, С.Г. (2006). «Взаимное использование ресурсов в средиземноморских сообществах растений и опылителей: насколько специализированы сети опыления?» В: Васер Н.М., Оллертон Дж. (ред.) Взаимодействие растений и опылителей, от специализации к обобщению , University of Chicago Press, страницы 221–244.
- ^ Гомес, Хосе М.; Босх, Джорди; Перфектти, Франциско; Фернандес, Хуанде; Абдельазиз, Мохамед (2007). «Разнообразие опылителей влияет на размножение и пополнение растений: компромиссы при обобщении». Экология . 153 (3): 597–605. Бибкод : 2007Oecol.153..597G . дои : 10.1007/s00442-007-0758-3 . ПМИД 17576602 . S2CID 4219676 .
- ^ Васкес, Диего П.; Айзен, Марсело А. (2003). «Анализ нулевой модели специализации во взаимодействиях растений и опылителей». Экология . 84 (9): 2493–2501. дои : 10.1890/02-0587 . S2CID 54219364 .
- ^ Перейти обратно: а б Дюпон, Йоко Л.; Тройельсгаард, Кристиан; Хаген, Мелани; Хенриксен, Мари В.; Олесен, Йенс М.; Педерсен, Нанна МЭ; Кисслинг, В. Дэниел (2014). «Пространственная структура индивидуальной сети растений-опылителей» (PDF) . Ойкос . 123 (11): 1301–1310. дои : 10.1111/oik.01426 . S2CID 31139363 .
- ^ Перейти обратно: а б Кинг, Кэролайн; Баллантайн, Гэвин; Уиллмер, Пэт Г. (2013). «Почему посещение цветов является плохим показателем опыления: измерение осаждения пыльцы за один визит с последствиями для сетей опыления и сохранения» . Методы экологии и эволюции . 4 (9): 811–818. дои : 10.1111/2041-210X.12074 . hdl : 10023/5299 .
- ^ Перейти обратно: а б Лопесарайса-Микель, Марта Э.; Хейс, Ричард Б.; Уолли, Мартин Р.; Меммотт, Джейн (2007). «Воздействие чужеродного растения на сеть местных растений и опылителей: экспериментальный подход». Экологические письма . 10 (7): 539–550. дои : 10.1111/j.1461-0248.2007.01055.x . ПМИД 17542933 .
- ^ Перейти обратно: а б с д и ж Соу, Ахмаду; Харан, Жюльен; Бенуа, Лора; Галан, Максим; Брево, Тьерри (2020). «Метабаркодирование ДНК как инструмент распутывания пищевых сетей в агроэкосистемах» . Насекомые . 11 (5): 294. doi : 10.3390/insects11050294 . ПМК 7290477 . ПМИД 32403224 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0. - ^ Галан, Максим; Понс, Жан-Батист; Турнейр, Орианна; Пьер, Эрик; Лейхтманн, Максим; Понтье, Доминик; Шарбоннель, Натали (2018). «Метабаркодирование для параллельной идентификации нескольких сотен хищников и их жертв: применение к анализу рациона видов летучих мышей» . Ресурсы молекулярной экологии . 18 (3): 474–489. дои : 10.1111/1755-0998.12749 . ПМИД 29288544 . S2CID 1004450 .
- ^ Перейти обратно: а б Паула, Дебора П.; Линард, Бенджамин; Крэмптон-Платт, Алекс; Шриватсан, Амрита; Тиммерманс, Мартин Дж.Т.Н.; Суджи, Эдисон Р.; Пирес, Кармен СС; Соуза, Лукас М.; Андоу, Дэвид А.; Фоглер, Альфрид П. (2016). «Выявление трофических взаимодействий у членистоногих-хищников посредством секвенирования ДНК содержимого кишечника» . ПЛОС ОДИН . 11 (9): e0161841. Бибкод : 2016PLoSO..1161841P . дои : 10.1371/journal.pone.0161841 . ПМК 5021305 . ПМИД 27622637 .
- ^ Прингл, Роберт М.; Хатчинсон, Мэтью К. (2 ноября 2020 г.). «Разрешение структуры пищевой сети» . Ежегодный обзор экологии, эволюции и систематики . 51 (1): 55–80. doi : 10.1146/annurev-ecolsys-110218-024908 . ISSN 1543-592X . S2CID 225457667 .
- ^ Фирлей, Аннабель; Дойон, Жозе; Харвуд, Джеймс Д.; Бродер, Жак (2013). «Многоподходное исследование для определения взаимодействия между жужелицами и соевой тлей» . Экологическая энтомология . 42 (1): 89–96. дои : 10.1603/EN11303 . ПМИД 23339789 . S2CID 7903342 .
- ^ Карцинель, Тайлер Р.; Чен, Патрисия А.; Ковердейл, Тайлер С.; Эриксон, Дэвид Л.; Кресс, В. Джон; Кузьмина Мария Львовна; Рубинштейн, Дэниел И.; Ван, Вэй; Прингл, Роберт М. (2015). «Метабаркодирование ДНК проливает свет на разделение пищевых ниш крупных травоядных африканских животных» . Труды Национальной академии наук . 112 (26): 8019–8024. Бибкод : 2015PNAS..112.8019K . дои : 10.1073/pnas.1503283112 . ПМЦ 4491742 . ПМИД 26034267 .
- ^ Лопес, СМ; Де Барба, М.; Бойер, Ф.; Мерсье, К.; Да Силва Фильо, П.Дж. С.; Хайдтманн, LM; Гальяно, Д.; Кубяк, Б.Б.; Лангоне, П.; Гарсиас, FM; Джелли, Л.; Куассак, Э.; Де Фрейтас, TR O.; Таберлет, П. (2015). «Анализ диеты с метабаркодированием ДНК для видов с парапатрическим и симпатрическим распределением: тематическое исследование на подземных грызунах» . Наследственность . 114 (5): 525–536. дои : 10.1038/hdy.2014.109 . ПМЦ 4815513 . ПМИД 25649502 . S2CID 5077455 .
- ^ Молло, Грегори; Дайк, Пьер-Франсуа; Лефевр, Пьер; Лескурре, Франсуаза; Мартин, Жан-Франсуа; Пири, Сильвен; Утка, Эльза; Тиксье, Филипп (2014). «Покровное выращивание меняет рацион членистоногих на банановой плантации: подход метабаркодирования» . ПЛОС ОДИН . 9 (4): е93740. Бибкод : 2014PLoSO...993740M . дои : 10.1371/journal.pone.0093740 . ПМЦ 3973587 . ПМИД 24695585 .
- ^ Гринстоун, Мэтью Х.; Пэйтон, Марк Э.; Вебер, Дональд К.; Симмонс, Элвин М. (2014). «Период полураспада обнаруживаемости в исследованиях хищник-жертва членистоногих: что это такое, зачем он нам нужен, как его измерить и как его использовать». Молекулярная экология . 23 (15): 3799–3813. дои : 10.1111/mec.12552 . ПМИД 24303920 . S2CID 41032815 .
- ^ Симондсон, Уильям О.К.; Харвуд, Джеймс Д. (2014). «Специальный выпуск по молекулярному обнаружению трофических взаимодействий: распутывание запутанного банка» (PDF) . Молекулярная экология . 23 (15): 3601–3604. дои : 10.1111/mec.12831 . ПМИД 25051891 . S2CID 26082315 .
- ^ Валентини, Алиса; Помпанон, Франсуа; Таберле, Пьер (2009). «Штрих-код ДНК для экологов». Тенденции в экологии и эволюции . 24 (2): 110–117. дои : 10.1016/j.tree.2008.09.011 . ПМИД 19100655 .
- ^ Бегг, Грэм С.; Кук, Саманта М.; Дай, Ричард; Ферранте, Марко; Франк, Пьер; Лавин, Клэр; Лёвей, Габор Л.; Особняк-Ваки, Агата; Пелл, Джудит К.; Пети, Сандрин; Кесада, Нора; Риччи, Бенуа; Раттен, Стивен Д.; Берч, А. Николас Э. (2017). «Функциональный обзор консервационного биологического контроля». Защита урожая . 97 : 145–158. дои : 10.1016/j.cropro.2016.11.008 .
- ^ Гурр, генеральный менеджер; Ван Эмден, ХФ; Раттен, SD (1998). «Манипулирование средой обитания и эффективность естественных врагов». Природоохранный биологический контроль . стр. 155–183. дои : 10.1016/B978-012078147-8/50055-4 . ISBN 9780120781478 .
- ^ Викхейс, Крис АГ; Лу, Яньхуэй; Моралес, Хельда; Васкес, Луис Л.; Легаспи, Хесуса К.; Элиопулос, Панайотис А.; Эрнандес, Луис М. (2013). «Текущее состояние и потенциал сохранения биологического контроля в сельском хозяйстве в развивающемся мире». Биологический контроль . 65 : 152–167. doi : 10.1016/j.biocontrol.2012.11.010 .
- ^ Перейти обратно: а б Саймондсон, WOC; Сандерленд, КД; Гринстоун, Миннесота (2002). «Могут ли хищники-универсалы быть эффективными агентами биоконтроля?». Ежегодный обзор энтомологии . 47 : 561–594. дои : 10.1146/annurev.ento.47.091201.145240 . ПМИД 11729085 .
- ^ Харвуд, Джей Ди и Обрицкий, Джей Джей (2005). «Роль альтернативной добычи в поддержании популяции хищников». В: М.С. Ходдл (ред.), Труды второго международного симпозиума по биологической борьбе с членистоногими , том 2, страницы 453–462.
- ^ Харвуд, Джеймс Д.; Десне, Николя; Ю, ХО Юнг С.; Роули, Дэниел Л.; Гринстоун, Мэтью Х.; Обрицкий, Джон Дж.; о'Нил, Роберт Дж. (2007). «Отслеживание роли альтернативной добычи в хищничестве соевой тли Orius insidiosus: молекулярный подход». Молекулярная экология . 16 (20): 4390–4400. дои : 10.1111/j.1365-294X.2007.03482.x . ПМИД 17784913 . S2CID 21211301 .
- ^ Перейти обратно: а б с Ферлонг, Майкл Дж. (2015). «Знай своих врагов: интеграция молекулярных и экологических методов для оценки воздействия членистоногих-хищников на вредителей сельскохозяйственных культур». Наука о насекомых . 22 (1): 6–19. дои : 10.1111/1744-7917.12157 . ПМИД 25081301 . S2CID 27851198 .
- ^ Йонссон, Маттиас; Раттен, Стив Д.; Лэндис, Дуг А.; Гурр, Джефф М. (2008). «Последние достижения в сохранении биологического контроля членистоногих со стороны членистоногих». Биологический контроль . 45 (2): 172–175. doi : 10.1016/j.biocontrol.2008.01.006 .
- ^ Помпанон, Франсуа; Дигл, Брюс Э.; Симондсон, Уильям О.К.; Браун, Дэвид С.; Джарман, Саймон Н.; Таберле, Пьер (2012). «Кто что ест: оценка диеты с использованием секвенирования следующего поколения» . Молекулярная экология . 21 (8): 1931–1950. дои : 10.1111/j.1365-294X.2011.05403.x . ПМИД 22171763 . S2CID 10013333 .
- ^ Вальднер, Томас; Трауготт, Майкл (2012). «Анализ срыгиваний на основе ДНК: неинвазивный подход к изучению рациона потребителей беспозвоночных». Ресурсы молекулярной экологии . 12 (4): 669–675. дои : 10.1111/j.1755-0998.2012.03135.x . ПМИД 22443278 . S2CID 21254959 .
- ^ Боманн, Кристина; Монадджем, Ара; Лемкуль Ноер, Кристина; Расмуссен, Мортен; Зил, Мэтт РК; Клэр, Элизабет; Джонс, Гарет; Виллерслев, Эске; Гилберт, М. Томас П. (2011). «Анализ молекулярной диеты двух африканских летучих мышей со свободным хвостом (Molossidae) с использованием высокопроизводительного секвенирования» . ПЛОС ОДИН . 6 (6): e21441. Бибкод : 2011PLoSO...621441B . дои : 10.1371/journal.pone.0021441 . ПМК 3120876 . ПМИД 21731749 .
- ^ Перейти обратно: а б с д и Почон, Ксавьер; Зайко, Анастасия; Флетчер, Лорен М.; Ларош, Оливье; Вуд, Сюзанна А. (2017). «Разыскивается живым или мертвым? Использование метабаркодирования ДНК и РНК окружающей среды для различения живых ансамблей для целей биобезопасности» . ПЛОС ОДИН . 12 (11): e0187636. Бибкод : 2017PLoSO..1287636P . дои : 10.1371/journal.pone.0187636 . ПМК 5667844 . ПМИД 29095959 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0. - ^ Перейти обратно: а б Молнар, Дженнифер Л.; Гамбоа, Ребекка Л.; Ревенга, Кармен; Сполдинг, Марк Д. (2008). «Оценка глобальной угрозы инвазивных видов морскому биоразнообразию». Границы в экологии и окружающей среде . 6 (9): 485–492. дои : 10.1890/070064 .
- ^ Оленин, Сергей; Минчин, Дэн; Даунис, Дариус (2007). «Оценка биозагрязнения водных экосистем». Бюллетень о загрязнении морской среды . 55 (7–9): 379–394. Бибкод : 2007MarPB..55..379O . дои : 10.1016/j.marpolbul.2007.01.010 . ПМИД 17335857 .
- ^ Кацаневакис, Стелиос; Валлентинус, Ингер; Зенетос, Аргиро; Леппякоски, Эркки; Чинар, Мелих Эртан; Озтюрк, Байрам; Грабовский, Михал; Голани, Дэниел; Кардосо, Ана Кристина (2014). «Воздействие инвазивных чужеродных морских видов на экосистемные услуги и биоразнообразие: общеевропейский обзор» . Водные нашествия . 9 (4): 391–423. дои : 10.3391/ai.2014.9.4.01 .
- ^ Пиментел, Дэвид; Сунига, Родольфо; Моррисон, Дуг (2005). «Обновленная информация об экологических и экономических издержках, связанных с чужеродными инвазивными видами в Соединенных Штатах». Экологическая экономика . 52 (3): 273–288. дои : 10.1016/j.ecolecon.2004.10.002 .
- ^ Перейти обратно: а б Харви, Чад Т.; Куреши, Самир А.; МакИсаак, Хью Дж. (2009). «Обнаружение колонизирующих водных неместных видов» . Разнообразие и распространение . 15 (3): 429–437. дои : 10.1111/j.1472-4642.2008.00550.x . S2CID 53602450 .
- ^ Леппякоски, Эркки; Голлаш, Стефан; Оленин, Сергей (29 июня 2013 г.). Инвазивные водные виды Европы. Распространение, воздействие и управление . Спрингер. ISBN 9789401599566 .
- ^ Халм, Филип Э. (2009). «Торговля, транспорт и проблемы: управление путями проникновения инвазивных видов в эпоху глобализации» . Журнал прикладной экологии . 46 : 10–18. дои : 10.1111/j.1365-2664.2008.01600.x .
- ^ Инглис Г., Флоерл О., Ахьонг С., Кокс С., Анвин М., Пондер-Саттон А. и др. (2010) «Риски биобезопасности, связанные с биообрастанием на международных судах, прибывающих в Новую Зеландию: краткое изложение закономерностей и предсказателей загрязнения». Биобезопасность Новой Зеландии , Технический отчет №: 2008.
- ^ Руис, Грегори М.; Роулингс, Тоня К.; Доббс, Фред К.; Дрейк, Лиза А.; Маллади, Тимоти; Хук, Анварул; Колвелл, Рита Р. (2000). «Глобальное распространение микроорганизмов кораблями». Природа . 408 (6808): 49–50. Бибкод : 2000Natur.408...49R . дои : 10.1038/35040695 . ПМИД 11081499 . S2CID 205010602 .
- ^ Голлаш, С. (2002). «Значение обрастания корпусов кораблей как вектора интродукции видов в Северное море». Биологическое обрастание . 18 (2): 105–121. дои : 10.1080/08927010290011361 . S2CID 85620063 .
- ^ Минёр, Фредерик; Джонсон, Марк П.; Мэггс, Кристин А. (2008). «Интродукция макроводорослей в результате обрастания корпуса на прогулочных судах: водоросли и моряки». Экологический менеджмент . 42 (4): 667–676. Бибкод : 2008EnMan..42..667M . дои : 10.1007/s00267-008-9185-4 . ПМИД 18704562 . S2CID 25680517 .
- ^ Джонсон, Лэдд Э.; Риккарди, Энтони; Карлтон, Джеймс Т. (2001). «Распространение водных инвазивных видов по суше: оценка риска временного прогулочного катания на лодках». Экологические приложения . 11 (6): 1789. doi : 10.1890/1051-0761(2001)011[1789:ODOAIS]2.0.CO;2 . ISSN 1051-0761 .
- ^ Дарбисон, Эмили (2009). «Привычки морского плавания на лодках и потенциал распространения инвазивных видов в заливе Св. Лаврентия» . Водные нашествия . 4 : 87–94. дои : 10.3391/ai.2009.4.1.9 .
- ^ Акоста, Эрнандо; Форрест, Барри М. (2009). «Распространение морских неместных видов посредством прогулочного катания на лодках: концептуальная модель оценки риска, основанная на анализе дерева неисправностей». Экологическое моделирование . 220 (13–14): 1586–1598. doi : 10.1016/j.ecolmodel.2009.03.026 .
- ^ МакМахон, Роберт (2011). «Структура популяции мидий Quagga (Dreissena rostriformis bugensis) во время раннего вторжения в озера Мид и Мохаве, январь-март 2007 г.» . Водные нашествия . 6 (2): 131–140. дои : 10.3391/ai.2011.6.2.02 .
- ^ Перейти обратно: а б Флетчер, Лорен М.; Зайко, Анастасия; Атала, Хавьер; Рихтер, Ингрид; Дюфур, Селин М.; Почон, Ксавьер; Вуд, Сусана А.; Хопкинс, Грант А. (2017). «Трюмные воды как вектор распространения морских вредителей: морфологическое, метабаркодирование и экспериментальная оценка». Биологические инвазии . 19 (10): 2851–2867. дои : 10.1007/s10530-017-1489-y . S2CID 25513146 .
- ^ Перейти обратно: а б Синнер, Джим, Барри Форрест, Марк Ньютон, Грэм Инглис, Крис Вудс, Дон Морриси, Орели Кастинель и Роуэн Стрикленд (2013) «Управление распространением вредных морских организмов внутри страны» Часть B: Законодательная база и анализ вариантов. Каутронский институт .
- ^ Инглис Г., Моррисси Д., Вудс С., Синнер Дж., Ньютон М. (2013) «Управление внутренним распространением вредных морских организмов», Часть A: Оперативные инструменты управления. Министерство первичной промышленности Новой Зеландии , Отчет № 2013/xx.
- ^ Конте, Тьерри; Сандиониги, Анна; Виар, Фредерик; Казираги, Маурицио (2015). «ДНК (мета)баркодирование биологических вторжений: мощный инструмент для выяснения процессов вторжения и помощи в управлении инопланетянами». Биологические инвазии . 17 (3): 905–922. дои : 10.1007/s10530-015-0854-y . S2CID 12799342 .
- ^ Зайко, Анастасия; Шимански, Кейт; Почон, Ксавьер; Хопкинс, Грант А.; Голдстиен, Шарин; Флёрл, Оливер; Вуд, Сюзанна А. (2016). «Метабаркодирование улучшает обнаружение эукариот из ранних сообществ биообрастания: значение для мониторинга вредителей и управления их маршрутами». Биологическое обрастание . 32 (6): 671–684. дои : 10.1080/08927014.2016.1186165 . ПМИД 27212415 . S2CID 46842367 .
- ^ Браун, Эмили А.; Чейн, Фредерик Джей-Джей; Жан, Айбин; МакИсаак, Хью Дж.; Кристеску, Мелания Э. (2016). «Раннее обнаружение водных захватчиков с помощью метабаркодирования выявило большое количество неместных видов в канадских портах» . Разнообразие и распространение . 22 (10): 1045–1059. дои : 10.1111/ddi.12465 .
- ^ Зайко, Анастасия; Мартинес, Хосе Л.; Шмидт-Петерсен, Юлия; Рибичич, Дени; Самуиловиене, Аурелия; Гарсиа-Васкес, Ева (2015). «Метабаркодирование для контроля балластных вод – выгодное решение или трудная задача?» . Бюллетень о загрязнении морской среды . 92 (1–2): 25–34. Бибкод : 2015MarPB..92...25Z . doi : 10.1016/j.marpolbul.2015.01.008 . ПМИД 25627196 .
- ^ Шокралла, Шади; Сполл, Дженнифер Л.; Гибсон, Джоэл Ф.; Хаджибабаи, Мехрдад (2012). «Технологии секвенирования нового поколения для исследования ДНК окружающей среды» . Молекулярная экология . 21 (8): 1794–1805. дои : 10.1111/j.1365-294X.2012.05538.x . ПМИД 22486820 . S2CID 5944083 .
- ^ Цзи, Иньцю; Эштон, Луиза; Педли, Скотт М.; Эдвардс, Дэвид П.; Тан, Юн; Накамура, Акихиро; Китчинг, Роджер; Долман, Пол М.; Вудкок, Пол; Эдвардс, Фелисити А.; Ларсен, Тронд Х.; Сюй, Уэйн В.; Бенедикт, Сьюзан; Хамер, Кейт С.; Уилков, Дэвид С.; Брюс, Кэтрин; Ван, Сяоян; Леви, Таал; Лотт, Мартин; Эмерсон, Брент К.; Ю, Дуглас В. (2013). «Надежный, проверяемый и эффективный мониторинг биоразнообразия с помощью метабаркодирования» . Экологические письма . 16 (10): 1245–1257. дои : 10.1111/ele.12162 . hdl : 10072/56911 . ПМИД 23910579 .
- ^ Дарлинг, Джон А.; Фредерик, Раймонд М. (2018). «Инструменты на основе нуклеиновых кислот для наблюдения, мониторинга и исследований балластных вод» . Журнал морских исследований . 133 : 43–52. Бибкод : 2018JSR...133...43D . дои : 10.1016/j.seares.2017.02.005 . ПМК 6104837 . ПМИД 30147432 .
- ^ Коринальдези, К.; Беолчини, Ф.; Делл'Анно, А. (2008). «Скорость повреждения и деградации внеклеточной ДНК в морских отложениях: значение для сохранения последовательностей генов». Молекулярная экология . 17 (17): 3939–3951. дои : 10.1111/j.1365-294X.2008.03880.x . ПМИД 18643876 . S2CID 22062643 .
- ^ Делл'Анно, А.; Дановаро, Р. (2005). «Внеклеточная ДНК играет ключевую роль в функционировании глубоководной экосистемы». Наука . 309 (5744): 2179. doi : 10.1126/science.1117475 . ПМИД 16195451 . S2CID 39216262 .
- ^ Менгони, Алессио; Татти, Энрико; Декорози, Франческа; Вити, Карло; Баззикалупо, Марко; Джованнетти, Лусиана (2005). «Сравнение подходов T-RFLP с 16S рРНК и 16S рДНК к изучению бактериальных сообществ в почвенных микрокосмах, обработанных хроматом в качестве возмущающего агента». Микробная экология . 50 (3): 375–384. дои : 10.1007/s00248-004-0222-4 . ПМИД 16254761 . S2CID 23943691 .
- ^ Павловский, Ян; Эслинг, Филипп; Лейзерович, Франк; Седхаген, Томас; Уайлдинг, Томас А. (2014). «Экологический мониторинг с помощью метабаркодирования секвенирования протистов нового поколения: оценка воздействия рыбоводства на сообщества донных фораминифер». Ресурсы молекулярной экологии . 14 (6): 1129–1140. дои : 10.1111/1755-0998.12261 . ПМИД 24734911 . S2CID 2303206 .
- ^ Виско, Джоана Аморим; Апотелоз-Перре-Жантиль, Лор; Кордонье, Ариэль; Эслинг, Филипп; Пилле, Лоик; Павловский, Ян (2015). «Экологический мониторинг: определение индекса диатомовых водорослей на основе данных секвенирования следующего поколения». Экологические науки и технологии . 49 (13): 7597–7605. Бибкод : 2015EnST...49.7597V . дои : 10.1021/es506158m . ПМИД 26052741 .
- ^ Доул, Эдди; Почон, Ксавьер; Кили, Найджел; Вуд, Сюзанна А. (2015). «Оценка последствий обогащения морского дна разведением лосося с использованием разнообразия бактериальных сообществ и высокопроизводительного секвенирования» . ФЭМС Микробиология Экология . 91 (8): fiv089. дои : 10.1093/femsec/fiv089 . ПМИД 26207046 .
- ^ Почон, X.; Вуд, ЮАР; Кили, Северная Каролина; Лейзерович, Ф.; Эслинг, П.; Дрю, Дж.; Павловский, Дж. (2015). «Точная оценка влияния разведения лосося на обогащение донных отложений с использованием метабаркодирования фораминифер». Бюллетень о загрязнении морской среды . 100 (1): 370–382. Бибкод : 2015МартПБ.100..370П . doi : 10.1016/j.marpolbul.2015.08.022 . ПМИД 26337228 .
- ^ Ларош, Оливье; Вуд, Сюзанна А.; Трамбле, Луи А.; Эллис, Джоан И.; Лейзерович, Франк; Павловский, Ян; Лир, Гэвин; Атала, Хавьер; Почон, Ксавье (2016). «Первая оценка метабаркодирования фораминифер для мониторинга воздействия на окружающую среду морской буровой площадки» . Морские экологические исследования . 120 : 225–235. Бибкод : 2016MarER.120..225L . дои : 10.1016/j.marenvres.2016.08.009 . hdl : 10754/622291 . ПМИД 27595900 .
- ^ Казарес Каррильо, DE Описание личинки Eucinostomus jonesii (Pisces, Gerreidae) морфологическими и генетическими методами.
- ^ Керр, К.К., Лийтмаер, Д.А., Барьер, А.С., Хеберт, П.Д. и Тубаро, П.Л. (2009). Исследование закономерностей эволюции неотропических птиц с помощью штрих-кодов ДНК. ПЛОС ОДИН, 4(2), е4379.
- ^ Лийтмаер, Д.А., Керр, К.К., Барьер, А.С., Хеберт, П.Д. и Тубаро, П.Л. (2011). Библиотеки штрих-кодов ДНК дают представление о континентальных моделях диверсификации птиц. ПЛОС ОДИН, 6(7), е20744.
- ^ Лийтмаер, Д.А., Керр, К.К., Стокль, М.Ю., и Тубаро, П.Л. (2012). ДНК-штрихкодирование птиц: от полевого сбора до анализа данных. В «Штрих-кодах ДНК» (стр. 127–152). Хумана Пресс.
- ^ Гарсиа Моралес, AE (2013). Штрих-кодирование и филогеографический анализ коловраток (Monogononta, Ploima) из юго-восточной Мексики.
- ^ де Карвальо, Даниэль Кардозу; Сесилия Гонтихо Леаль; Пауло душ Сантос Помпеу; Хосе Вандерваль Мело Джуниор и Дениз А.А. де Оливейра (2011). Применение метода генетической идентификации — штрих-кода ДНК — у рыб из бассейна реки Сан-Франциско. Бюллетень Бразильского общества ихтиологии № 104. Кафедра морфологии, Институт биологических наук, Ботукату, Сан-Паулу, Бразилия.
- ^ де Карвальо, Даниэль Кардозу; Пауло душ Сантос Помпеу; Сесилия Гонтихо Леаль; Дениз А.А. де Оливейра и Ханнер, Р. (2011). Дивергенция глубоководных пресноводных рыб Бразилии: пример бассейна реки Сан-Франциско. Митохондриальная ДНК , 2011.
- ^ Эрнандес, Эсмеральда Сальгадо (2008). Генетический штрих-код («штрих-код ДНК») как инструмент идентификации видов. Эррериана. Научно-пропагандистский журнал. Том 4 (1).
- ^ Хеберт П. и Г. Райан (2005). Перспективы штрих-кодирования ДНК для таксономии. Систематическая биология. 54(5):852-859.
- ^ Холлингсворт, П. (2007). Штрих-кодирование ДНК: потенциальные пользователи. Геномика, общество и политика. 3(2):44-47.
- ^ Олд, Джули М.; Валлин, Блэр Л.; Торли, Роуэн К.; Кейси, Фиона; Стэннард, Хейли Дж. (2023). «Анализ метабаркодирования ДНК диеты вомбата с голым носом ( Vombatus ursinus )» . Экология и эволюция . 14 (5): e11432. дои : 10.1002/ece3.11432 . ПМЦ 11103767 . ПМИД 38770127 .
- ^ Кейси, Фиона; Олд, Джули М.; Стэннард, Хейли Дж. (2023). «Оценка рациона находящегося под угрозой исчезновения северного волосатого вомбата ( Lasiorhinus krefftii ) с использованием метабаркодирования ДНК» . Экология и эволюция . 13 (9): e10469. дои : 10.1002/ece3.10469 . ПМЦ 10485309 . ПМИД 37693933 .
- ^ «Что такое штрих-кодирование ДНК?» . Штрих-код жизни. Архивировано из оригинала 1 июля 2017 года . Проверено 11 октября 2017 г.
- ^ Эванс, Даррен М.; Китсон, Джеймс Дж. Н.; Лант, Дэвид Х.; Стро, Найджел А.; Покок, Майкл Джо (2016). «Объединение метабаркодирования ДНК и анализа экологических сетей для понимания и создания устойчивых наземных экосистем» (PDF) . Функциональная экология . 30 (12): 1904–1916. дои : 10.1111/1365-2435.12659 . ISSN 1365-2435 .
- ^ DNAqua-Net Европейское сотрудничество в области науки и технологий. Доступ: 9 октября 2022 г.
- ^ Павловский, Ян; Келли-Куинн, Мэри; Альтерматт, Флориан; Апотелоз-Перре-Жантиль, Лор; Бежа, Педро; Богджеро, Анджела; Борха, Ангел; Буше, Аньес; Кордье, Тристан (2018). «Будущее биотических индексов в экогеномную эпоху: интеграция метабаркодирования (е) ДНК в биологическую оценку водных экосистем» . Наука об общей окружающей среде . 637–638: 1295–1310. Бибкод : 2018ScTEn.637.1295P . doi : 10.1016/j.scitotenv.2018.05.002 . hdl : 20.500.12327/138 . ПМИД 29801222 .
- ^ Ратнасингем, Судживан; Хеберт, Пол Д.Н. (2007). «ЖИРНЫЙ: Система данных «Штрих-код жизни» (http://www.barcodinglife.org)» . Заметки по молекулярной экологии . 7 (3): 355–364. дои : 10.1111/j.1471-8286.2007.01678.x . ПМК 1890991 . ПМИД 18784790 .
- ^ Стокль, Марк (ноябрь – декабрь 2013 г.). «Штрих-кодирование ДНК готово к прорыву» . ДжинВотч . 26 (5). Архивировано из оригинала 3 апреля 2014 года . Проверено 14 февраля 2021 г.
- ^ ДеСалле, Р.; Иган, Миннесота; Сиддалл, М. (2005). «Несвятая троица: таксономия, разграничение видов и штрих-кодирование ДНК» . Философские труды Королевского общества B: Биологические науки . 360 (1462): 1905–1916. дои : 10.1098/rstb.2005.1722 . ПМК 1609226 . ПМИД 16214748 .
- ^ Уитворт, ТЛ; Доусон, РД; Магалон, Х.; Бодри, Э. (2007). «Штрих-кодирование ДНК не может надежно идентифицировать виды рода мух Protocalliphora (Diptera: Calliphoridae)» . Труды Королевского общества B: Биологические науки . 274 (1619): 1731–1739. дои : 10.1098/rspb.2007.0062 . ПМЦ 2493573 . ПМИД 17472911 .
- ^ Стакебрандт, Э.; Гебель, Б.М. (1994). «Таксономическое примечание: место для реассоциации ДНК-ДНК и анализа последовательности 16S рРНК в современном определении вида в бактериологии» . Межд. Дж. Сист. Бактериол . 44 (4): 846–849. дои : 10.1099/00207713-44-4-846 .
- ^ Стакебрандт, Э.; Эберс, Дж. (2006). «Пересмотр таксономических параметров: потускневшие золотые стандарты» (PDF) . Микробиология сегодня . 33 (4): 152–155. Архивировано (PDF) из оригинала 25 марта 2018 г.
- ^ Ньютон, Ян (2003). Видообразование и биогеография птиц . Академическая пресса. п. 69. ИСБН 978-0-08-092499-1 . Архивировано из оригинала 7 февраля 2018 года.
- ^ Андерссон, Мальте (1999). Гибридизация и филогения поморников . Том. 266 стр. 1579–1585. дои : 10.1098/rspb.1999.0818 . ISBN 9780080924991 . ПМК 1690163 . Архивировано из оригинала 7 февраля 2018 года.
{{cite book}}:|journal=игнорируется ( помогите ) - ^ Кесвани, Дж.; Уитмен, ВБ (2001). «Связь сходства последовательности 16S рРНК с гибридизацией ДНК у прокариот» . Международный журнал систематической и эволюционной микробиологии . 51 (2): 667–78. дои : 10.1099/00207713-51-2-667 . ПМИД 11321113 .
Дальнейшие ссылки
[ редактировать ]- ^ Сантоферрара, Лусиана; Бурки, Фабьен; Филкер, Сабина; Логарес, Рамиро; Данторн, Мика; Макманус, Джордж Б. (2020). «Перспективы десяти лет исследований протистов с помощью высокопроизводительного метабаркодирования». Журнал эукариотической микробиологии . 67 (5): 612–622. дои : 10.1111/jeu.12813 . hdl : 10261/223228 . ПМИД 32498124 . S2CID 219331807 .