Личность по происхождению
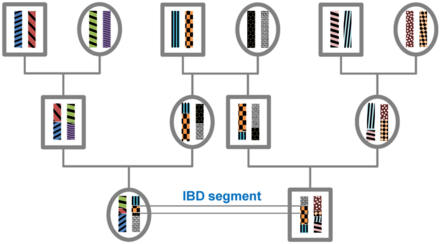
Сегмент ДНК идентичен по состоянию (IBS) у двух или более индивидуумов, если они имеют идентичные нуклеотидные последовательности в этом сегменте. Сегмент СРК идентичен по происхождению ( ВЗК ) у двух или более индивидуумов, если они унаследовали его от общего предка без рекомбинации , то есть сегмент имеет одинаковое наследственное происхождение у этих индивидуумов. Сегменты ДНК, которые являются ВЗК, по определению являются СРК, но сегменты, которые не являются ВЗК, все равно могут быть СРК из-за одних и тех же мутаций у разных людей или рекомбинаций, которые не изменяют сегмент. [ нужна ссылка ]

Теория [ править ]
Все люди в конечной популяции являются родственниками, если их проследить достаточно долго, и, следовательно, будут иметь общие сегменты генома ВЗК . Во время мейоза сегменты ВЗК распадаются путем рекомбинации. Следовательно, ожидаемая длина сегмента IBD зависит от количества поколений с момента появления самого последнего общего предка в локусе сегмента. Длина сегментов IBD, возникших в результате n поколений общего предка в прошлом (следовательно, с участием 2 n мейоза), экспоненциально распределяется со средним значением 1/(2 n ) Моргана (M). [1] Ожидаемое количество сегментов IBD уменьшается с увеличением количества поколений с момента появления общего предка в этом локусе. Для определенного сегмента ДНК вероятность ВЗК уменьшается как 2 −2−2n так как в каждом мейозе вероятность передачи этого сегмента равна 1/2. [2]
Приложения [ править ]
Идентифицированные сегменты ВЗК можно использовать для самых разных целей. Как отмечалось выше, степень (продолжительность и количество) распространения ВЗК зависит от семейных отношений между тестируемыми людьми. Таким образом, одним из применений обнаружения сегментов IBD является количественная оценка родства. [3] [4] [5] [6] Измерение родства может быть использовано в судебной генетике . [7] но также может увеличить информацию при генетических связей . картировании [3] [8] и помочь уменьшить предвзятость за счет недокументированных связей в стандартных исследованиях ассоциаций . [6] [9] Еще одним применением IBD является вменение генотипа и гаплотипа вывод фазы . [10] [11] [12] Длинные общие сегменты ВЗК, разбитые короткими участками, могут указывать на ошибки фазировки. [5] [13] : ДА
Картирование ВЗК [ править ]
картирование ВЗК [3] аналогичен анализу сцепления, но может быть выполнен без известной родословной на группе неродственных людей. Картирование ВЗК можно рассматривать как новую форму ассоциативного анализа, которая увеличивает возможности картирования генов или геномных областей, содержащих несколько вариантов восприимчивости к редким заболеваниям. [6] [14]
Используя смоделированные данные, Браунинг и Томпсон показали, что картирование ВЗК имеет более высокую эффективность, чем тестирование ассоциаций, когда несколько редких вариантов гена способствуют восприимчивости к заболеванию. [14] области всего генома С помощью картирования IBD были обнаружены значимые в изолированных популяциях, а также в беспородных популяциях, хотя стандартные ассоциативные тесты не дали результатов. [11] [15] Хаувен и др. использовали совместное использование ВЗК для определения хромосомного местоположения гена, ответственного за доброкачественный рецидивирующий внутрипеченочный холестаз в изолированной рыбацкой популяции. [16] Кенни и др. также использовали изолированную популяцию для точного картирования сигнала, обнаруженного в ходе полногеномного исследования ассоциаций в плазме (GWAS) уровней растительных стеринов (PPS), суррогатного показателя всасывания холестерина из кишечника. [17] Франкс и др. смог идентифицировать потенциальный локус восприимчивости к шизофрении и биполярному расстройству с помощью данных генотипа образцов «случай-контроль». [18] Лин и др. обнаружили значимый сигнал связи по всему геному в наборе данных пациентов с рассеянным склерозом . [19] Летузе и др. использовали картирование IBD для поиска мутаций-основателей в образцах рака . [20]
в генетике ВЗК популяционной
Обнаружение естественного отбора в геноме человека также возможно с помощью обнаруженных сегментов IBD. Отбор обычно имеет тенденцию к увеличению количества сегментов ВЗК среди людей в популяции. Сканируя регионы с избыточным распространением ВЗК, можно выявить регионы человеческого генома, которые совсем недавно подверглись сильному отбору. [21] [22]
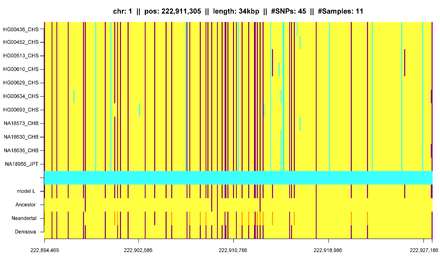
Кроме того, сегменты ВЗК могут быть полезны для измерения и выявления других влияний на структуру населения. [6] [23] [24] [25] [26] Гусев и др. показали, что сегменты IBD могут использоваться с дополнительным моделированием для оценки демографической истории, включая узкие места и примеси . [24] Используя аналогичные модели, Паламара и др. и Карми и др. реконструировал демографическую историю евреев -ашкенази и кенийских масаи . [25] [26] [27] Ботиге и др. исследовали различия в африканском происхождении среди европейского населения. [28] Ральф и Куп использовали обнаружение ВЗК для количественной оценки общего происхождения различных европейских популяций. [29] и Гравель и др. аналогичным образом пытались сделать выводы из генетической истории популяций Америки. [30] Рингбауэр и др. использовали географическую структуру сегментов IBD для оценки распространения в Восточной Европе в течение последних столетий. [31] Используя данные «1000 геномов», Хохрейтер обнаружил различия в распространении ВЗК между африканскими, азиатскими и европейскими популяциями, а также сегменты ВЗК, которые являются общими с древними геномами, такими как неандертальцы или денисовцы . [13]
Методы и программное обеспечение [ править ]
Программы для выявления сегментов ВЗК у неродственных лиц:
- БЫСТРО : сверхбыстрая идентификация путем обнаружения происхождения в когортах масштаба биобанка с использованием позиционного преобразования Берроуза – Уиллера [32]
- Parente : идентифицирует сегменты IBD между парами людей в нефазированных данных генотипа. [33]
- BEAGLE/fastIBD : находит сегменты ВЗК между парами людей в полногеномных SNP . данных [34]
- BEAGLE/RefinedIBD : находит сегменты IBD в парах людей, используя метод хеширования, и оценивает их значимость с помощью отношения правдоподобия. [35]
- IBDseq : обнаруживает парные сегменты IBD в данных секвенирования. [36]
- ЗАродышевая линия : обнаруживает сегменты ВЗК в линейном времени у пар людей. [5]
- DASH : основан на парных сегментах IBD для определения кластеров людей, которые, вероятно, будут иметь один и тот же гаплотип. [15]
- PLINK : набор инструментов для анализа ассоциаций всего генома и популяционного анализа сцепления, включая метод парного обнаружения сегментов ВЗК. [6]
- Связать : оценивает вероятность ВЗК между парами людей в определенном локусе с использованием SNP. [3]
- MCMC_IBDfinder : основан на цепи Маркова Монте-Карло (MCMC) для поиска сегментов воспалительного заболевания кишечника у нескольких людей. [37]
- IBD-Groupon : обнаруживает групповые сегменты IBD на основе парных отношений IBD. [38]
- HapFABIA : идентифицирует очень короткие сегменты ВЗК, характеризующиеся редкими вариантами в больших данных секвенирования одновременно у нескольких людей. [13]
См. также [ править ]
- Сопоставление ассоциаций
- Генетическая ассоциация
- Генетическая связь
- Полногеномное исследование ассоциаций
- Идентичность по типу
- Неравновесие связей
- Популяционная генетика
Ссылки [ править ]
- ^ Браунинг, SR (2008). «Оценка парной идентичности по происхождению на основе плотных данных генетических маркеров в популяционной выборке гаплотипов» . Генетика . 178 (4): 2123–2132. doi : 10.1534/genetics.107.084624 . ПМК 2323802 . ПМИД 18430938 .
- ^ Томпсон, Э.А. (2008). «Процесс ВЗК по четырем хромосомам» . Теоретическая популяционная биология . 73 (3): 369–373. дои : 10.1016/j.tpb.2007.11.011 . ПМК 2518088 . ПМИД 18282591 .
- ↑ Перейти обратно: Перейти обратно: а б с д Альбрехцен, А.; Санд Корнелиуссен, Т.; Мольтке, И.; Ван Оверсим Хансен, Т.; Нильсен, ФК; Нильсен, Р. (2009). «Картирование родства и участки родства для общегеномных данных при наличии неравновесия по сцеплению». Генетическая эпидемиология . 33 (3): 266–274. дои : 10.1002/gepi.20378 . ПМИД 19025785 . S2CID 12029712 .
- ^ Браунинг, SR; Браунинг, Б.Л. (2010). «Обнаружение личности с высоким разрешением по происхождению у неродственных лиц» . Американский журнал генетики человека . 86 (4): 526–539. дои : 10.1016/j.ajhg.2010.02.021 . ПМЦ 2850444 . ПМИД 20303063 .
- ↑ Перейти обратно: Перейти обратно: а б с Гусев А.; Лоу, Дж. К.; Стоффель, М.; Дейли, MJ; Альтшулер Д.; Бреслоу, Дж. Л.; Фридман, Дж. М.; Пе'Эр, И. (2008). «Вся популяция, полногеномное картирование скрытого родства» . Геномные исследования . 19 (2): 318–326. дои : 10.1101/гр.081398.108 . ПМЦ 2652213 . ПМИД 18971310 .
- ↑ Перейти обратно: Перейти обратно: а б с д и Перселл, С.; Нил, Б.; Тодд-Браун, К.; Томас, Л.; Феррейра, Маргарита; Бендер, Д.; Маллер, Дж.; Склар, П.; Де Баккер, PIW; Дейли, MJ; Шам, ПК (2007). «PLINK: Набор инструментов для полногеномного анализа связей и популяционного анализа связей» . Американский журнал генетики человека . 81 (3): 559–575. дои : 10.1086/519795 . ПМК 1950838 . ПМИД 17701901 .
- ^ Ян В. Эветт; Брюс С. Вейр (январь 1998 г.). Интерпретация доказательств ДНК: статистическая генетика для судебно-медицинских экспертов . Синауэр Ассошиэйтс, Инкорпорейтед. ISBN 978-0-87893-155-2 .
- ^ Лейтенеггер, А.; Прум, Б.; Генин, Э.; Верный, Ц.; Леменк, А.; Клержедарпу, Ф.; Томпсон, Э. (2003). «Оценка коэффициента инбридинга посредством использования геномных данных» . Американский журнал генетики человека . 73 (3): 516–523. дои : 10.1086/378207 . ПМК 1180677 . ПМИД 12900793 .
- ^ Войт, Б.Ф.; Причард, Дж. К. (2005). «Смешение из-за загадочной связи в исследованиях ассоциаций случаев-контроля» . ПЛОС Генетика . 1 (3): е32. дои : 10.1371/journal.pgen.0010032 . ПМК 1200427 . ПМИД 16151517 .
- ^ Конг, А.; Массон, Г.; Фригг, МЛ; Гилфасон, А.; Зусманович П.; Торлейфссон, Г.; Оласон, ИП; Ингасон, А.; Стейнберг, С.; Рафнар, Т.; Сулем, П.; Муи, М.; Йонссон, Ф.; Торстейнсдоттир, У.; Гудбьартссон, DF; Стефанссон, Х.; Стефанссон, К. (2008). «Обнаружение совместного использования по происхождению, фазировке на большие расстояния и вменению гаплотипов» . Природная генетика . 40 (9): 1068–1075. дои : 10.1038/ng.216 . ПМК 4540081 . ПМИД 19165921 .
- ↑ Перейти обратно: Перейти обратно: а б Гусев А.; Шах, MJ; Кенни, Э.Э.; Рамачандран, А.; Лоу, Дж. К.; Салит, Дж.; Ли, CC; Левандовский, ЕС; Уивер, Теннесси; Доан, королевский адвокат; Пекхэм, HE; Маклафлин, Сан-Франциско; Лайонс, MR; Шет, В.Н.; Стоффель, М.; Де Ла Вега, FM; Фридман, Дж. М.; Бреслоу, Дж. Л.; Пеэр, И. (2011). «Низкопроходное полногеномное секвенирование и вариантный вывод с использованием идентичности по происхождению в изолированной человеческой популяции» . Генетика . 190 (2): 679–689. дои : 10.1534/genetics.111.134874 . ПМЦ 3276614 . ПМИД 22135348 .
- ^ Браунинг, БЛ; Браунинг, SR (2009). «Единый подход к вменению генотипа и выводу фазы гаплотипа для больших наборов данных о тройках и неродственных индивидуумах» . Американский журнал генетики человека . 84 (2): 210–223. дои : 10.1016/j.ajhg.2009.01.005 . ПМК 2668004 . ПМИД 19200528 .
- ↑ Перейти обратно: Перейти обратно: а б с Хохрайтер, С. (2013). «HapFABIA: Идентификация очень коротких сегментов идентичности по происхождению, характеризующихся редкими вариантами в больших данных секвенирования» . Исследования нуклеиновых кислот . 41 (22): е202. дои : 10.1093/нар/gkt1013 . ПМЦ 3905877 . ПМИД 24174545 .
- ↑ Перейти обратно: Перейти обратно: а б Браунинг, SR; Томпсон, Э.А. (2012). «Обнаружение редких вариантов ассоциаций путем сопоставления идентичности по происхождению в исследованиях случай-контроль» . Генетика . 190 (4): 1521–1531. дои : 10.1534/genetics.111.136937 . ПМЦ 3316661 . ПМИД 22267498 .
- ↑ Перейти обратно: Перейти обратно: а б Гусев А.; Кенни, Э.Э.; Лоу, Дж. К.; Салит, Дж.; Саксена, Р.; Катиресан, С.; Альтшулер, Д.М.; Фридман, Дж. М.; Бреслоу, Дж. Л.; Пеэр, И. (2011). «DASH: метод картирования гаплотипов, идентичных по происхождению, обнаруживает связь с недавними вариациями» . Американский журнал генетики человека . 88 (6): 706–717. дои : 10.1016/j.ajhg.2011.04.023 . ПМЦ 3113343 . ПМИД 21620352 .
- ^ Хауэн, RHJ; Бахарлоо, С.; Бланкеншип, К.; Раймакерс, П.; Джуйн, Дж.; Сандкуйл, Луизиана; Фреймер, НБ (1994). «Скрининг генома путем поиска общих сегментов: картирование гена доброкачественного рецидивирующего внутрипеченочного холестаза» . Природная генетика . 8 (4): 380–386. дои : 10.1038/ng1294-380 . hdl : 1765/55192 . ПМИД 7894490 . S2CID 8131209 .
- ^ Кенни, Э.Э.; Гусев А.; Ригель, К.; Лутьоханн, Д.; Лоу, Дж. К.; Салит, Дж.; Маллер, Дж.Б.; Стоффель, М.; Дейли, MJ; Альтшулер, Д.М.; Фридман, Дж. М.; Бреслоу, Дж. Л.; Пе'Эр, И.; Сехайек, Э. (2009). «Систематический анализ гаплотипов позволяет выявить сложный локус стерола плазменного растения на микронезийском острове Косраэ» . Труды Национальной академии наук . 106 (33): 13886–13891. Бибкод : 2009PNAS..10613886K . дои : 10.1073/pnas.0907336106 . ПМК 2728990 . ПМИД 19667188 .
- ^ Франкс, К.; Тоцци, Ф.; Фармер, А.; Винсент, JB; Руеску, Д.; Сент-Клер, Д.; Маглия, П. (2008). «Популяционный анализ связи когорт шизофрении и биполярного расстройства случай-контроль идентифицирует потенциальный локус восприимчивости на 19q13» . Молекулярная психиатрия . 15 (3): 319–325. дои : 10.1038/mp.2008.100 . hdl : 11858/00-001M-0000-0012-C935-9 . ПМИД 18794890 .
- ^ Лин, Р.; Чарльзворт, Дж.; Станкович Дж.; Перро, В.М.; Браун, Массачусетс; Анзгене, Б.В.; Тейлор, Б.В. (2013). Толанд, Аманда Юарт (ред.). «Картирование идентичности по происхождению для обнаружения редких вариантов, вызывающих предрасположенность к рассеянному склерозу» . ПЛОС ОДИН . 8 (3): e56379. Бибкод : 2013PLoSO...856379L . дои : 10.1371/journal.pone.0056379 . ПМЦ 3589405 . ПМИД 23472070 .
- ^ Летузе, Э.; Соу, А.; Петель, Ф.; Розати, Р.; Фигейредо, Британская Колумбия; Бурничон, Н.; Хименес-Рокепло, AP; Лалли, Э.; Де Рейньес, Алабама (2012). Майлунд, Томас (ред.). «Идентификация мутаций-основателей при раке по происхождению с использованием данных SNP опухолей высокого разрешения» . ПЛОС ОДИН . 7 (5): e35897. Бибкод : 2012PLoSO...735897L . дои : 10.1371/journal.pone.0035897 . ПМЦ 3342326 . ПМИД 22567117 .
- ^ Альбрехцен, А.; Мольтке, И.; Нильсен, Р. (2010). «Естественный отбор и распределение идентичности по происхождению в геноме человека» . Генетика . 186 (1): 295–308. дои : 10.1534/genetics.110.113977 . ПМК 2940294 . ПМИД 20592267 .
- ^ Хан, Л.; Эбни, М. (2011). «Оценка идентичности по происхождению с использованием плотных данных генотипа по всему геному» . Генетическая эпидемиология . 35 (6): 557–567. дои : 10.1002/gepi.20606 . ПМЦ 3587128 . ПМИД 21769932 .
- ^ Кокерхэм, CC; Вейр, Б.С. (1983). «Разница фактического инбридинга». Теоретическая популяционная биология . 23 (1): 85–109. дои : 10.1016/0040-5809(83)90006-0 . ПМИД 6857551 .
- ↑ Перейти обратно: Перейти обратно: а б Гусев А.; Паламара, ПФ; Апонте, Г.; Чжуан, З.; Дарваси, А.; Грегерсен, П.; Пеэр, И. (2011). «Архитектура дальнодействующих гаплотипов, общих внутри и между популяциями» . Молекулярная биология и эволюция . 29 (2): 473–486. дои : 10.1093/molbev/msr133 . ПМК 3350316 . ПМИД 21984068 .
- ↑ Перейти обратно: Перейти обратно: а б Паламара, ПФ; Ленц, Т.; Дарваси, А.; Пеэр, И. (2012). «Распределение идентичности по длине по происхождению раскрывает мелкомасштабную демографическую историю» . Американский журнал генетики человека . 91 (5): 809–822. дои : 10.1016/j.ajhg.2012.08.030 . ПМЦ 3487132 . ПМИД 23103233 .
- ↑ Перейти обратно: Перейти обратно: а б Паламара, ПФ; Пеэр, И. (2013). «Вывод об исторических темпах миграции посредством обмена гаплотипами» . Биоинформатика . 29 (13): i180–i188. doi : 10.1093/биоинформатика/btt239 . ПМЦ 3694674 . ПМИД 23812983 .
- ^ Карми, С.; Паламара, ПФ; Вачич, В.; Ленц, Т.; Дарваси, А.; Пеэр, И. (2013). «Разница в разделении идентичности по происхождению в модели Райта-Фишера» . Генетика . 193 (3): 911–928. arXiv : 1206.4745 . дои : 10.1534/genetics.112.147215 . ПМК 3584006 . ПМИД 23267057 .
- ^ Ботиг, LR; Хенн, Б.М.; Гравий, С.; Мэйплс, Британская Колумбия; Жиньу, Чехия; Корона, Э.; Ацмон, Г.; Бернс, Э.; Острер, Х.; Флорес, К.; Бертранпети, Ж.; Комас, Д.; Бустаманте, компакт-диск (2013). «Поток генов из Северной Африки способствует дифференцированному генетическому разнообразию человека в южной Европе» . Труды Национальной академии наук . 110 (29): 11791–11796. Бибкод : 2013PNAS..11011791B . дои : 10.1073/pnas.1306223110 . ПМК 3718088 . ПМИД 23733930 .
- ^ Ральф, П.; Куп, Г. (2013). Тайлер-Смит, Крис (ред.). «География недавнего генетического происхождения в Европе» . ПЛОС Биология . 11 (5): e1001555. дои : 10.1371/journal.pbio.1001555 . ПМЦ 3646727 . ПМИД 23667324 .
- ^ Гравий, С.; Захария Ф.; Морено-Эстрада, А.; Бирнс, Дж. К.; Муццио, М.; Родригес-Флорес, JL; Кенни, Э.Э.; Жиньу, Чехия; Мэйплс, Британская Колумбия; Гибле, В.; Дутиль, Дж.; Виа, М.; Сандовал, К.; Бедойя, Г.; 1000 геномов, ТЗ; Олексик А.; Руис-Линарес, Е.Г.; Берчард, Дж. К.; Мартинес-Крусадо, компакт-диск; Бустаманте, компакт-диск (2013). Уильямс, Скотт М. (ред.). «Реконструкция миграций коренных американцев на основе данных всего генома и всего экзома» . ПЛОС Генетика . 9 (12): e1004023. arXiv : 1306.4021 . дои : 10.1371/journal.pgen.1004023 . ПМЦ 3873240 . ПМИД 24385924 .
{{cite journal}}: CS1 maint: числовые имена: список авторов ( ссылка ) - ^ Рингбауэр, Харальд; Куп, Грэм; Бартон, Николас Х. (01 марта 2017 г.). «Вывод о современной демографии на основе изоляции по расстоянию между длинными общими блоками последовательностей» . Генетика . 205 (3): 1335–1351. doi : 10.1534/genetics.116.196220 . ISSN 0016-6731 . ПМК 5340342 . ПМИД 28108588 .
- ^ Насери А., Лю X, Чжан С., Чжи Д. Сверхбыстрая идентификация путем обнаружения происхождения в когортах масштаба биобанка с использованием позиционного преобразования Берроуза – Уиллера BioRxiv 2017.
- ^ Родригес Дж. М., Бацоглу С., Берковичи С. Точный метод определения родства в больших наборах данных нефазированных генотипов с помощью встроенного теста отношения правдоподобия. РЕКОМБ 2013, ЛНБИ 7821:212-229.
- ^ Браунинг, БЛ; Браунинг, СР (2011). «Быстрый и мощный метод определения личности по происхождению» . Американский журнал генетики человека . 88 (2): 173–182. дои : 10.1016/j.ajhg.2011.01.010 . ПМК 3035716 . ПМИД 21310274 .
- ^ Браунинг, БЛ; Браунинг, SR (2013). «Повышение точности и эффективности определения личности по происхождению в демографических данных» . Генетика . 194 (2): 459–471. дои : 10.1534/genetics.113.150029 . ПМЦ 3664855 . ПМИД 23535385 .
- ^ Браунинг, БЛ; Браунинг, СР (2013). «Определение идентичности по происхождению и оценка частоты ошибок генотипа в данных последовательностей» . Американский журнал генетики человека . 93 (5): 840–851. дои : 10.1016/j.ajhg.2013.09.014 . ПМЦ 3824133 . ПМИД 24207118 .
- ^ Мольтке, И.; Альбрехцен, А.; Хансен, ТВО; Нильсен, ФК; Нильсен, Р. (2011). «Метод обнаружения областей воспалительного заболевания кишечника одновременно у нескольких людей - с применением к генетике заболеваний» . Геномные исследования . 21 (7): 1168–1180. дои : 10.1101/гр.115360.110 . ПМК 3129259 . ПМИД 21493780 .
- ^ Он, Д. (2013). «IBD-Groupon: эффективный метод обнаружения групповых областей идентичности по происхождению одновременно у нескольких людей на основе парных отношений IBD» . Биоинформатика . 29 (13): i162–i170. doi : 10.1093/биоинформатика/btt237 . ПМЦ 3694672 . ПМИД 23812980 .