Филогенетическое примирение

В филогенетике — это подход , примирение позволяющий связать истории двух или более совместно развивающихся биологических объектов. Общая идея согласования состоит в том, что филогенетическое дерево , представляющее эволюцию объекта (например, гомологичных генов или симбионтов ), может быть нарисовано внутри другого филогенетического дерева, представляющего охватывающий объект (соответственно, виды, хозяева), чтобы выявить их взаимозависимость и эволюционные события, которые ознаменовали их общую историю. Разработка подходов к примирению началась в 1980-х годах, главным образом для описания совместной эволюции гена и генома, а также хозяина и симбионта, который может быть мутуалистическим, комменсалистским или паразитическим. Его также использовали, например, для обнаружения горизонтального переноса генов или понимания динамики эволюции генома .
Филогенетическое согласование может объяснить разнообразие эволюционных траекторий того, что составляет историю жизни, переплетающихся друг с другом на всех масштабах, которые можно рассматривать, от молекул до популяций или культур. Недавним воплощением важности взаимодействия между уровнями организации является концепция холобионта , согласно которой макроорганизм рассматривается как сложное партнерство различных видов. Моделирование эволюции таких сложных сущностей является одним из сложных и интересных направлений современных исследований примирения.
Филогенетические деревья как вложенные структуры
[ редактировать ]
Филогенетические деревья переплетаются на всех уровнях организации, объединяя конфликты и зависимости внутри и между уровнями. Популяции макроорганизмов мигрируют между континентами, их микробы- симбионты переключаются между популяциями, гены их симбионтов передаются между видами микробов, происходит обмен доменами между генами.Этот список уровней организации не является репрезентативным или исчерпывающим, но дает представление об уровнях, на которых использовались методы выверки. В качестве общего метода примирение может учитывать множество других уровней. Например, он мог бы рассмотреть синтенную организацию генов, [1] история взаимодействия мобильных элементов и видов, [2] эволюция белкового комплекса у разных видов. [3] Масштаб рассматриваемых эволюционных событий может варьироваться от популяционных событий, таких как географическая диверсификация, до уровней нуклеотидов внутри генов, [4] включая, например, события на уровне хромосом внутри геномов, такие как дупликация всего генома. [5]
Филогении использовались для представления разнообразия жизни на многих уровнях организации : макроорганизмы, [6] их клетки на протяжении всего развития , [7] микроорганизмов через маркерные гены , [8] хромосомы , [9] белки , [10] белковые домены , [6] а также может быть полезным для понимания эволюции элементов человеческой культуры, таких как языки. [11] или сказки . [12] На каждом из этих уровней филогенетические деревья описывают разные истории конкретных событий диверсификации, которые могут или не могут быть общими для всех уровней. Тем не менее, поскольку они структурно вложены друг в друга (подобно матрешкам ) или функционально зависимы, эволюция на определенном уровне связана с эволюцией на других уровнях.
Филогенетическая сверка — это выявление связей между уровнями путем сравнения как минимум двух связанных деревьев. Первоначально разработанные для двух деревьев, недавно были построены сверки для более чем двух уровней (см. раздел « Явное моделирование трех и более уровней »). Таким образом, примирение обеспечивает эволюционные сценарии, которые раскрывают конфликт и сотрудничество между развивающимися сущностями. Эти связи могут быть неинтуитивными, например, гены, присутствующие в одном и том же геноме, могут демонстрировать некоррелированную эволюционную историю, в то время как некоторые гены, присутствующие в геноме симбионта, могут демонстрировать сильный сигнал коэволюции с филогенией хозяина. Следовательно, примирение может быть полезным инструментом для понимания ограничений и эволюционных стратегий, лежащих в основе совокупности, образующей холобионт .
Поскольку все уровни по существу имеют дело с одним и тем же объектом — филогенетическим деревом , одни и те же модели согласования — в частности модели, основанные на событиях дупликации - переноса - потери , которые являются центральными для этой статьи, — могут быть перенесены с небольшими изменениями в любую пару связанных уровней: [13] «внутренний», «нижний» или «ассоциированный» объект (например, ген, вид-симбионт, популяция) развивается внутри «верхнего» или «хозяина» (соответственно вида, хозяина или географической области).Верхние и нижние сущности частично связаны одной и той же историей, что приводит к сходству их филогенетических деревьев, но ассоциации могут со временем меняться, становиться более или менее строгими или переключаться на других партнеров.
История
[ редактировать ]Принцип филогенетического согласования был введен в 1979 году. [14] для объяснения различий между генами и филогениями на уровне видов. В скромных условиях для объяснения несоответствий между генным деревом и древом вида были привлечены два эволюционных события: дупликация генов и потеря генов. В нем также описывалась оценка генных деревьев, зная дерево видов и выровненную последовательность, используя количество дупликаций генов, потерь и замен нуклеотидов для эволюции выровненной последовательности - подход, который по-прежнему занимает центральное место сегодня с новыми моделями согласования и филогенетического вывода. . [15]
Термин «примирение» был использован Уэйном Мэддисоном в 1997 году. [16] как обратную концепцию «филогенетического разногласия», возникающего в результате эволюционных событий на уровне генов.
Затем примирение было разработано совместно для совместной эволюции хозяина и симбионта и географического разнообразия видов . В обоих случаях было важно смоделировать горизонтальное событие, которое подразумевало параллельные ветви дерева-хозяина: смену хозяина на хозяина и симбионта и расселение видов из одной области в другую в биогеографии . В отличие от генов и геномов, совместная эволюция хозяина и симбионта и объяснение видовой диверсификации географическим положением не всегда являются нулевой гипотезой . Визуальное изображение двух филогений в танглграмме может помочь оценить такую коэволюцию, хотя она не имеет очевидной статистической интерпретации. [17]
Символьные методы, такие как Brooks Parsimony Analysis , [18] были предложены для проверки коэволюции и реконструкции сценариев коэволюции. В этих методах одно из деревьев забывается, за исключением его листьев, которые затем используются в качестве персонажа, развивающегося на втором дереве.Первые модели примирения, явно учитывающие две топологии и использующие механистический подход, основанный на событиях, были предложены для хозяина, симбионта и биогеографии. [19] [20] Последовали дебаты, поскольку методы еще не были полностью надежными, но интегрировали полезную информацию в новую структуру. [21]
Затраты на каждое событие и метод динамического программирования, учитывающий все пары узлов-хозяев и симбионтов, были затем включены в подход «хозяин-симбионт», оба из которых до сих пор лежат в основе большинства современных методов согласования хозяина и симбионта, а также видов и генов. [22]
Примирение вернулось в рамки, в которых оно было введено, ген и вид. После того, как модели персонажей были рассмотрены для горизонтального переноса генов, [23] новая модель согласования, следующая и улучшающая подход динамического программирования, представленный для хозяина и симбионта, эффективно ввела горизонтальный перенос генов в согласование генов и видов поверх модели дупликации и потери. [24]
Таким образом, прогрессивное развитие филогенетического согласования стало возможным благодаря обменам между многочисленными исследовательскими сообществами, изучающими филогению на уровне хозяина и симбионта, гена и вида или биогеографии. Эта история и ее современное развитие рассматривались несколько раз, в основном с упором на определенные пары уровней, за некоторыми исключениями. [25] [13] Новые разработки начинают объединять различные структуры с новыми интегративными моделями.
Карманные суслики и их грызуны: классический пример
[ редактировать ]
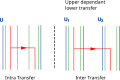
Карманные суслики ( Geomyidae ) и их жуки ( Trichodectidae ) образуют хорошо изученную систему коэволюции хозяина и симбионта. [26] Слева изображена филогения хозяина и симбионта, а также совпадение листьев их деревьев. Для хозяина О. обозначает Ортогеомис , Г. — Геомис и Т. — Томомис ; для симбионта Г. означает Geomydoecus и Т. означает Thomoydoecus .Согласование двух деревьев означает создание сценария с эволюционными событиями и сопоставлением исходных узлов, изображающих коэволюцию двух деревьев. События, рассматриваемые в этой системе, являются событиями модели DTL: дублирование, передача (или переключение хоста), потеря и совместное видообразование, нулевое событие коэволюции.В двух исследованиях были предложены два сценария: [27] [28] использование двух разных фреймворков, которые можно рассматривать как DTL предварительного динамического программирования. [29] примирение.В современных системах выверки DTL затраты присваиваются событиям. Затем было показано, что эти два сценария соответствуют максимально экономному согласованию с различными распределениями затрат. [22] В сценарии A для согласования двух деревьев используются 6 ковидеций, 2 дублирования, 3 потери и 2 переключения хостов, а в сценарии B используются 5 ковидеций, 3 дублирования, 3 потери и 2 переключения хостов. Стоимость сценария — это сумма стоимости его событий. Например, при стоимости 0 за совместное видообразование, 2 за дублирование, 1 за потерю и 3 за смену хоста, сценарий А имеет стоимость и сценарий Б , и поэтому, согласно принципу экономии, сценарий А будет считаться более вероятным (сценарий А остается более вероятным до тех пор, пока стоимость совместного видообразования меньше стоимости дублирования).
Разработка моделей филогенетического согласования
[ редактировать ]
Модели и методы, используемые сегодня в филогении, являются результатом нескольких десятилетий исследований, которые постепенно усложнялись, что обусловлено характером данных и стремлением к биологическому реализму, с одной стороны, и ограничениями и прогрессом математических и алгоритмических методов, с другой. .
Модели до согласования: символы на деревьях
[ редактировать ]Символьные методы можно использовать, когда для одного из уровней нет дерева, а есть только значения для символа на листьях филогенетического дерева для другого уровня. Модель определяет события изменения значения персонажа, их скорость, вероятность или стоимость. Например, характером может быть наличие хозяина на дереве-симбионте, [18] географический регион на видовом дереве, [31] количество генов на геномном дереве, [32] или нуклеотиды в последовательности. [4] Таким образом, такие методы направлены на реконструкцию наследственных персонажей во внутренних узлах дерева. [33]
Хотя эти методы дали результаты по эволюции генома, полезность второго дерева проявляется на очень простых примерах. Если симбионт недавно приобрел способность распространяться в группе видов и, таким образом, присутствует у большинства из них, методы определения признаков ошибочно покажут, что симбионт уже был у общего предка хозяев. Напротив, сравнение деревьев-симбионтов и деревьев-хозяев показало бы расхождения, свидетельствующие о горизонтальных переносах.
Истоки согласования: модель потери дублирования и отображение наименьшего общего предка
[ редактировать ]Дупликация и потеря были использованы в первую очередь для объяснения наличия нескольких копий гена в геноме или его отсутствия у определенных видов. [10] С помощью этих двух событий можно примирить любые два дерева, [14] т.е. нанести на карту узлы и ветви нижнего и верхнего деревьев или, что то же самое, дать список эволюционных событий, объясняющих несоответствия между верхним деревом и нижним деревом.Наиболее экономное согласование дублирования и потерь (DL) вычисляется с помощью сопоставления наименьшего общего предка (LCA): от листьев к корню каждый внутренний узел сопоставляется с наименьшим общим предком отображения двух его дочерних элементов.
Марковская модель примирения
[ редактировать ]Отображение LCA в модели DL следует принципу экономии : ни одно событие не должно вызываться, если в этом нет необходимости. Однако использование этого принципа дискутируется. [4] и общепризнано, что в молекулярной эволюции более точно будет соответствовать вероятностной модели случайного блуждания , которая не обязательно приводит к экономным сценариям. Марковская модель рождения и смерти — это такая модель, которая может генерировать нижнее дерево «внутри» фиксированного верхнего от корня до листьев. [34] Статистический вывод обеспечивает основу для поиска наиболее вероятных сценариев, и в этом случае согласование двух деревьев с максимальной вероятностью также является экономным. Кроме того, с помощью такой структуры можно отбирать сценарии или интегрировать несколько возможных сценариев для проверки различных гипотез, например, для исследования пространства нижних деревьев. Более того, вероятностные модели могут быть интегрированы в более крупные модели, поскольку вероятности просто умножаются при предположении независимости, например, сочетая эволюцию последовательностей и согласование DL. [35]
Представляем горизонтальную передачу
[ редактировать ]
Смена хозяина, то есть наследование симбионта от родственной линии, является решающим событием в эволюции паразитических или симбиотических отношений между видами. Этот горизонтальный перенос также моделирует миграционные события в биогеографии и стал интересен для согласования генных и видовых деревьев, когда выяснилось, что многие несоответствия нельзя просто объяснить дублированием и утратой и что горизонтальный перенос генов (HGT) был основным эволюционным процессом в эволюция микроорганизмов. Этот паттерн переключения или горизонтального переноса также может моделировать примесь или интрогрессию . [36] Учитывается в характерных методах, без информации о филогении симбионтов. [18] [37] Помимо модели DL, горизонтальный перенос обеспечивает новые и совершенно разные сценарии сверки.
Простой, но мощный подход к динамическому программированию
[ редактировать ]Метод согласования LCA дает уникальное решение, которое, как было показано, является оптимальным для проблемы минимизации взвешенного числа событий, независимо от относительных весов дублирования и потери. [38] Напротив, при дублировании, горизонтальном переносе и потере (DTL) может быть несколько одинаково экономных сверок. Например, последовательность дублирований и потерь может быть заменена одной передачей. Одна из первых идей по определению вычислительной проблемы и подходу к ее решению заключалась в том, чтобы в рамках системы хозяин/симбионт максимизировать количество совместных видообразований с помощью эвристического алгоритма. [27] Другое решение состоит в том, чтобы указать относительные затраты на события и найти сценарий, который минимизирует сумму затрат на его события. [22] В рамках вероятностных моделей эквивалентная задача состоит из присвоения ставок или вероятностей событиям и поиска сценариев максимального правдоподобия или выборочных сценариев в соответствии с их вероятностью. Все эти проблемы решаются с помощью подхода динамического программирования . Этот метод динамического программирования предполагает обход двух деревьев в обратном порядке .Исходя из листьев и затем поднимаясь вверх по двум деревьям, для каждой пары внутренних узлов (по одному на каждое дерево) вычисляется стоимость наиболее экономного согласования DTL. [22]
В рамках экономии затраты на согласование нижнего поддерева с корнем в с верхним поддеревом, корнем которого является инициализируется для листьев с их сопоставлением:
А затем индуктивно, обозначив дети дети затраты, связанные с видообразованием, дублированием, горизонтальным переносом и потерей соответственно (при часто фиксируется на 0),
Затраты и , поскольку они не зависят от , можно вычислить один раз для всех , тем самым достигая квадратичной сложности для вычисления для всех пар и . Стоимость потерь появляется только в связи с другими событиями, поскольку в целях экономии потеря всегда может быть связана с предыдущим событием в дереве.
Индукция, лежащая в основе использования динамического программирования, основана на постоянном движении по деревьям к корням. Однако некоторые комбинации событий, которые могут происходить последовательно, могут сделать эту индукцию нечеткой. Одна из таких комбинаций состоит из переноса, за которым немедленно следует утрата донорской линии (TL). Ограничение использования этого события TL устраняет индукцию. [39] При неограниченном использовании необходимо использовать или добавлять другие известные методы решения систем уравнений, такие как методы с фиксированной точкой , [40] или численное решение дифференциальных уравнений. [41] В 2016 году только две из семи наиболее часто используемых программ согласования экономии обрабатывали события TL. [42] хотя их рассмотрение может кардинально изменить результат примирения. [43]
В отличие от сопоставления LCA, сверка DTL обычно дает несколько сценариев с минимальными затратами, в некоторых случаях экспоненциальными. Сильная сторона подхода динамического программирования заключается в том, что он позволяет вычислить минимальную стоимость совместной эволюции входного верхнего и нижнего дерева за квадратичное время : [44] и получить наиболее экономичный сценарий путем возврата .Его также можно перенести в вероятностную структуру, чтобы вычислить вероятность коэволюции и получить наиболее вероятное согласование, заменив затраты ставками, минимумы суммами и суммы продуктами. [45] Более того, с помощью множественных возвратов этот подход подходит для перечисления всех экономных решений или выборки сценариев, оптимальных и неоптимальных, в зависимости от их вероятности.
Оценка стоимости и расценок мероприятия
[ редактировать ]
Динамическое программирование само по себе является лишь частичным решением и не решает некоторых проблем, возникающих при согласовании.Определение наиболее экономного согласования DTL требует присвоения затрат различным видам событий (D, T и L). Различные назначения затрат могут привести к различным сценариям сверки, поэтому существует необходимость в способе выбора этих затрат.Для этого существует множество подходов. КоРе-ПА [46] рекурсивно исследует пространство векторов затрат в поисках хорошего совпадения с частотами событий при сверке.ЭЛЬ [45] использует ту же идею в вероятностной структуре для оценки частоты событий по максимальному правдоподобию.Альтернативно, КОАЛА [47] представляет собой предварительный процесс, использующий приближенные байесовские вычисления с последовательным Монте-Карло : моделирование и статистическое отклонение или принятие параметров с последовательным уточнением.
В рамках принципа экономии также возможно разделить пространство возможных издержек событий на области издержек, которые приводят к одному и тому же оптимальному по Парето решению . [48] Оптимальные по Парето согласования таковы, что никакое другое согласование не имеет строго меньшей стоимости для одного типа событий (дублирование, перенос или потеря) и меньшей или равной для других.
При выборе затрат на мероприятие также можно полагаться на внешние факторы. Например, программа Angst [49] выбирает затраты, которые минимизируют различия в размере генома и количестве генов между родительскими и дочерними видами.
Проблема временной осуществимости
[ редактировать ]
Метод динамического программирования работает для датированных (внутренние узлы полностью упорядочены) или недатированных верхних деревьев. Однако в случае недатированных деревьев возникает проблема временной осуществимости. Действительно, горизонтальная передача подразумевает, что донор и получатель работают одновременно, что подразумевает ограничение по времени на дереве. Как следствие, две горизонтальные передачи могут быть несовместимы, поскольку они предполагают противоречивые временные ограничения.Подход динамического программирования не позволяет легко выявить такие несовместимости. Если верхнее дерево не датировано, найти временно осуществимое наиболее экономное согласование будет NP-трудно . [50] [51] [52] Это фиксированный параметр tractable , что означает, что существуют алгоритмы, работающие во времени, ограниченном экспонентой количества передач в выходных сценариях. [51] Некоторые решения подразумевают целочисленное линейное программирование. [53] или ветвящееся исследование. [13] Если верхнее дерево датировано, то проблемы несовместимости нет, поскольку горизонтальные передачи могут быть ограничены таким образом, чтобы они никогда не возвращались назад во времени. Затем поиск последовательного оптимального согласования решается за полиномиальное время. [51] или с ускорением в RASCAL, [54] [55] тестируя только часть сопоставлений узлов.
Большая часть программного обеспечения, использующего недатированные деревья, не ищет временную осуществимость, за исключением Джейн, [56] который исследует пространство общего количества заказов с помощью генетического алгоритма или, в постпроцессе, Notung, [57] и эвкалипт, [58] который ищет внутри множества оптимальных решений непротиворечивые во времени. Другие методы работают как дополнительные уровни сверки, корректируя сверки. [59] или возвращение подмножества возможных переводов, [60] который можно использовать для датировки видового дерева. [60] [61]
Расширение филогении: переходы из мертвых
[ редактировать ]
В филогенетике в целом важно иметь в виду, что существующие и предковые виды, представленные в любой филогении, представляют собой лишь редкую выборку видов, которые существуют в настоящее время или когда-либо существовали. Поэтому можно смело предположить, что все переносы, которые можно обнаружить филогенетическими методами, произошли от линий, строго говоря, отсутствующих в изучаемой филогении. [62] Учет вымершего или невыбранного биоразнообразия в филогенетических исследованиях может дать лучшее понимание этих процессов. [63] Первоначально методы согласования DTL не распознавали это явление и допускали передачу только между одновременными ветвями дерева, игнорируя, следовательно, наиболее правдоподобные решения. Однако методы, работающие с недатированными верхними деревьями, можно рассматривать как неявную обработку неизвестного разнообразия, позволяя осуществлять перенос «в будущее» с точки зрения одной филогении, то есть донор более древний, чем получатель. Переход в будущее может быть переведен в видообразование неизвестных видов, за которым следует переход от неизвестных видов.ЭЛЬ [62] в его датированной версии явно учитывается неизвестное разнообразие, добавляя Морана процесс видообразования/вымирания видов к датированной модели рождения/смерти эволюции генов. Переносы из мертвых также выполняются Tera и ecceTERA в бережливых условиях. [64] [42] показывающий, что учет этих переносов улучшает способность реконструировать генные деревья с использованием сверки и более явной модели. [65] и в вероятностной ситуации – в ALE без даты. [66]
Специфика биогеографии: древовидная структура «эволюции» территорий
[ редактировать ]
В биогеографии некоторые применения подходов согласования рассматривают в качестве верхнего дерева кладограмму площади с определенными наследственными узлами. Например, корнем может быть Пангея , а узлами — современные континенты . Иногда внутренние узлы представляют собой не наследственные области, а объединения областей их потомков, чтобы учесть возможность видов, эволюционирующих вдоль нижнего дерева, для заселения одной или нескольких областей. В этом случае эволюционными событиями являются миграция, когда один вид колонизирует новую территорию, аллопатрическое видообразование или викариантность, что эквивалентно совместному видообразованию при сравнении хозяина и симбионта.Несмотря на то, что такой подход не всегда дает дерево (при наличии объединений AB и BC листьев A, B, C у ребенка может быть несколько родителей), и эта структура не связана со временем (возможна у вида перейти от А к АВ путем миграции, а также от АВ к А путем вымирания), методы согласования — с событиями и динамическим программированием — могут вывести эволюционные сценарии между верхней географической структурой и нижним деревом видов. Дива [67] и Лагранж [68] [41] представляют собой две модели согласования, создающие такую древовидную структуру и затем применяющие согласование: первая с принципом экономии, вторая - с вероятностной структурой. Кроме того, BioGeoBEARS [69] это пакет биогеографических выводов, который переопределил модели DIVA и Лагранжа и допускает новые опции, такие как отдаленные зависимые переводы [70] и обсуждение выбора статистической модели. [69]
Графический вывод
[ редактировать ]Учитывая два дерева и множество эволюционных событий, связывающих их, просмотр согласованных деревьев является сложным, но необходимым вопросом, чтобы сделать исследования согласования более доступными. Некоторые программы сверки включают аннотации эволюционных событий на нижних деревьях. [57] в то время как другие, [56] [71] [58] [46] и конкретные пакеты в DL [72] или ДТЛ, [73] проследить нижнее дерево, встроенное в верхнее. Одной из трудностей в этом отношении является разнообразие выходных форматов для различных программ выверки данных. Общий стандарт, Recphyloxml, [74] был создан и одобрен частью сообщества, и доступен просмотрщик, способный отображать выверку в многоуровневых системах. [75]
Решение дополнительных практических вопросов
[ редактировать ]Применение согласования DTL к биологическим данным поднимает несколько проблем, связанных с уровнями неопределенности и достоверности входных и выходных данных. Что касается результатов, неопределенность ответа требует исследования всего пространства решений. Что касается входных данных, филогенетическое согласование должно учитывать неопределенности в разрешении или укоренении верхних или нижних деревьев или даже предлагать корни или разрешения в соответствии с их достоверностью.
Исследование пространства примирения
[ редактировать ]
Динамическое программирование позволяет выбирать согласования равномерно среди оптимальных. [76] или в зависимости от их вероятности. Также можно перечислить их за время, пропорциональное числу решений: [58] число, которое может быстро стать трудноразрешимым (даже только для оптимальных). Поиск и представление структуры среди множества возможных согласований был в центре недавних методологических разработок, особенно для методов, нацеленных на хозяина и симбионта. Несколько работ были сосредоточены на компактном представлении набора согласований из единой выборки оптимальных. [76] или путем построения графика, суммирующего оптимальные решения. [77] Этого можно достичь, присвоив значения поддержки конкретным событиям на основе всех оптимальных (или неоптимальных) согласований. [78] или с использованием согласованного дерева. [79] [59] В модели DL можно определить медианную сверку на основе общих событий и вычислить ее за полиномиальное время. [80] Императрица [71] может группировать подобные сверки посредством кластеризации, [81] при этом все попарные расстояния между согласованиями вычислимы за полиномиальное время (независимо от количества наиболее экономных согласований). [82] С той же целью Капибара [83] определяет классы эквивалентности среди согласований, эффективно вычисляет представителей для всех классов и выводит с линейной задержкой заданное количество согласований (сначала оптимальные, затем субоптимальные).Пространство наиболее экономного согласования может быть расширено или уменьшено при увеличении или уменьшении допустимого расстояния горизонтальной передачи. [58] что легко сделать с помощью динамического программирования.
Вывод филогенетических деревьев с согласованием
[ редактировать ]Сверка и неопределенность исходных данных
[ редактировать ]Согласование работает с двумя фиксированными деревьями, нижним и верхним, которые считаются правильными и укорененными. Однако эти деревья не являются данными из первых рук. Наиболее часто используемые данные для филогенетики состоят из выровненных нуклеотидных или белковых последовательностей. Извлечение ДНК, секвенирование, сборка и аннотирование геномов, распознавание гомологических отношений между генами и создание множественных выравниваний для филогенетической реконструкции — все это сложные процессы, ошибки в которых могут в конечном итоге повлиять на реконструированное дерево. [84] Любая топологическая ошибка или ошибка корня могут быть неправильно истолкованы и вызвать систематическую ошибку. Например, при сверке DL ошибки в нижнем дереве смещают сверку в сторону большего количества событий дублирования ближе к корню и большего количества потерь ближе к листьям. [85]
С другой стороны, примирение, как макроэволюционная модель, может работать как дополнительный уровень к микроэволюционной модели эволюции последовательности, разрешая политомии (узлы с более чем двумя дочерними элементами) или укореняя деревья, или переплетаться с ней посредством интегративных моделей. чтобы получить лучшую филогению.
Большинство работ в этом направлении сосредоточено на согласовании генов и видов, тем не менее, некоторые первые шаги были сделаны в отношении хозяина/симбионта, например, рассмотрение неукорененных деревьев симбионтов. [86] или иметь дело с политомиями в Джейн. [56]
Исследование пространства нижних деревьев с примирением
[ редактировать ]Для сверки можно легко использовать в качестве входных данных некорневые нижние деревья, что является часто используемой функцией, поскольку деревья, полученные на основе молекулярных данных, обычно не имеют корней. Можно проверить все возможные корни, или продуманный тройной обход некорневого дерева позволяет сделать это без дополнительных временных затрат. [39] В модели потерь при дублировании набор корней, минимизирующих затраты, находится близко друг к другу, образуя «плато». [87] свойство, которое не обобщается на DTL. [86] [79]

Согласование также может принимать на вход небинарные деревья , то есть с внутренними узлами, имеющими более двух дочерних элементов. Такие деревья можно получить, например, путем сокращения ветвей с низкой статистической поддержкой. Вывод двоичного дерева из недвоичного дерева в соответствии с оценками согласования решается в DL с помощью эффективных методов. [57] [88] [89] [90] [91] В DTL проблема NP-сложная . [92] Эвристика [93] и точные управляемые алгоритмы с фиксированными параметрами [92] [94] [95] являются возможными решениями.
Другой способ справиться с неопределенностью в нижних деревьях — взять в качестве входных данных выборку альтернативных нижних деревьев вместо одного. Например, в статье, давшей название примирению, [14] предлагалось рассмотреть все наиболее вероятные нижние деревья и выбрать из этих деревьев лучшее по их стоимости DL - принцип, также используемый TreeFix-DTL. [96] Выборка нижних деревьев может аналогичным образом отражать их правдоподобие в соответствии с выровненными последовательностями, полученными с помощью методов Монте-Карло байесовской цепи Маркова , реализованных, например, в Phylobayes. [97] АнгСТ, [49] НО [40] и т. д. ТЕРА [64] используйте «объединение», расширение динамического программирования DTL, которое позволяет эффективно перемещаться по набору альтернативных нижних деревьев вместо одного дерева.
Локальный поиск в пространстве нижних деревьев, руководствуясь совместной правдоподобностью, с одной стороны, из множественных выравниваний последовательностей, а с другой стороны, из сверки с верхним деревом, достигается в Phyldog с помощью модели DL. [98] и в GeneRax с DTL. [15] В модели DL с эволюцией последовательностей и расслабленными молекулярными часами пространство нижнего дерева можно исследовать с помощью MCMC. [99] МауглиННИ [100] может изменить входное дерево генов в узлах с плохой поддержкой, чтобы увеличить оценку DTL, в то время как TreeSolve разрешает мультифуркации, добавленные путем свертывания узлов с плохой поддержкой. [101]
Наконец, интегративные модели, сочетающие эволюцию последовательностей и согласование, могут вычислять совместную вероятность посредством динамического программирования (как для согласования, так и для эволюции последовательностей генов). [40] использовать цепь Маркова Монте-Карло, чтобы включить молекулярные часы для оценки длины ветвей в модели DL [34] или с расслабленными молекулярными часами, [99] и в модели DTL. [102] Эти модели применялись в рамках генов/видов, но еще не в контексте хозяина/симбионта или биогеографии.
Вывод верхних деревьев с использованием сверки
[ редактировать ]Выведение верхнего дерева из набора нижних деревьев — давний вопрос, связанный с проблемой супердерева . [103] Это особенно интересно в случае согласования генов и видов, когда многие (обычно тысячи) генных деревьев доступны из полных последовательностей генома . Методы супердерева пытаются собрать дерево видов на основе наборов деревьев, которые могут отличаться от современных наборов видов и топологии, но обычно без учета биологического процесса, объясняющего эти различия. Однако некоторые подходы к супердереву статистически совместимы для реконструкции дерева видов, если деревья генов моделируются в рамках модели DL. Это означает, что если количество входных нижних деревьев, сгенерированных из истинного верхнего дерева с помощью модели DL, возрастает до бесконечности, при условии отсутствия дополнительных ошибок, выходное верхнее дерево почти наверняка сходится к истинному. Это было показано на примере квартетной дистанции . [104] и с обобщенным расстоянием мультикопии Робинсона Фулдса , [105] [106] с лучшим временем выполнения , но при условии, что деревья генов не содержат двудольных частей, противоречащих дереву видов, что кажется редким в модели DL.

Согласование также можно использовать для вывода верхних деревьев. Это вычислительно сложная проблема: уже разрешение политомий в недвоичном верхнем дереве с двоичным нижним деревом (минимизация оценки согласования DL) является NP-трудной. [91] В частности, реконструкция дерева видов, дающая наилучшую стоимость DL для нескольких генных деревьев, является NP-трудной и 2-аппроксимируемой. [107] Это называется проблемой дублирования генов или, в более общем смысле, экономией генного дерева. Проблема рассматривалась как способ обнаружить паралогию и улучшить реконструкцию дерева видов. [108] [109] Это NP-сложно, с интересными результатами по сложности задачи. [91] [110] и поведение модели при различном размере входных данных, структуре и наличии ILS. [111] Существует несколько решений с ILP [112] или эвристика, [113] [114] и с возможностью глубокого слияния. [115]
ОДТЛ [45] принимает в качестве входных данных генные деревья и ищет дерево видов максимального правдоподобия в соответствии с моделью DTL с поиском с восхождением на холм . Этот подход создает дерево видов с внутренними узлами, упорядоченными во времени, обеспечивая совместимость по времени для сценариев перехода между нижними деревьями {раздел ссылки|Проблема временной осуществимости}.
Решая более общую проблему, Филдог [98] ищет дерево видов с максимальной вероятностью, генные деревья и параметры DL из нескольких сопоставлений семейств с помощью нескольких раундов локального поиска. Таким образом, он одновременно исследует как верхние, так и нижние деревья. MixTreEM [116] представляет более быстрое решение.
Ограничения двухуровневой модели DTL
[ редактировать ]Предел динамического программирования: независимая эволюция дочерних линий
[ редактировать ]
Структура динамического программирования, как и обычные модели рождения и смерти, работает на основе гипотезы независимой эволюции дочерних линий в нижнем дереве. Однако эта гипотеза не справедлива, если модель дополняется несколькими другими задокументированными эволюционными событиями, такими как горизонтальный перенос с заменой гомологичного гена в линии реципиента или конверсия гена . Горизонтальный перенос с заменой обычно моделируется перестановкой верхнего дерева, называемой обрезкой и пересадкой поддерева (SPR). Согласование в рамках SPR является NP-сложной задачей, даже в устаревших деревьях, и допускает обработку с фиксированными параметрами в отношении размера выходных данных. [117] [118]
Другой способ смоделировать и сделать вывод о замене горизонтальных переносов — это использовать лес максимального согласия , где ветви нижнего и верхнего деревьев обрезаются, чтобы получить два одинаковых (или статистически неотличимых друг от друга) дерева. [119] ) верхние и нижние леса. Задача NP-сложная, [120] но было предложено несколько приближений. [121] Замену трансферов можно рассматривать поверх модели DL. [122] В том же духе конверсию генов можно рассматривать как «замещающую дупликацию». В последнем случае полиномиальный алгоритм, не использующий динамическое программирование и являющийся расширением метода LCA, может найти все оптимальные решения, включая преобразования генов. [118]
Интеграция уровней популяции: неспособность дивергенции и неполная сортировка по происхождению
[ редактировать ]В рамках структуры хозяин/симбионт один вид симбионтов иногда связан с несколькими видами хозяев. Это означает, что, хотя у хозяина наблюдается видообразование или диверсификация, у симбионта популяции неразличимы.Это решается, например, с помощью дополнительных политомий в дереве симбионтов, что может привести к неразрешимым проблемам вывода, поскольку политомии необходимо разрешить.Это также моделируется дополнительным эволюционным событием «неспособность дивергенции» (Джейн, [56] Амокоала [123] ).Неспособность дивергенции может быть способом обеспечить «свободную» смену хозяев в популяции, поток симбионтов между близкородственными хозяевами.Следуя этому видению, в Eucalypt рассматривается возможность переключения хостов, разрешенная только для близких хостов. [58] Эту идею горизонтального потока между близкими популяциями можно также применить к структурам генов и видов, при этом определение вида основано на градиенте потока генов между популяциями. [124]

Неспособность дивергенции является одним из способов примирения популяционной динамики , структуры, в основном адаптированной к многовидовому уровню, где популяции должны быть хорошо дифференцированы. Существуют и другие популяционные явления, ограничивающие эту структуру, одним из которых является глубокое слияние родословных, приводящее к неполной сортировке родословных (ILS), которая не обрабатывается моделью DTL. [88] [125] Слияние нескольких видов — это классическая модель эволюции аллелей по древу видов с рождением аллелей.и сортировка аллелей по видам, которая учитывает размеры популяции и, естественно, включает ILS. [126] [127] [111] [128] [129] В контексте сверки было предпринято несколько попыток учесть ILS без сложной интеграции модели населения. Например, ILS можно рассматривать как возможный образец эволюции генного дерева. В этом случае детские линии не являются независимыми друг от друга, что приводит к трудностям. С помощью LCA можно обрабатывать только ILS, но согласование ILS + DL является NP-сложным, даже без переносов. [130]
Обозначения [88] обрабатывает ILS, сжимая короткие ветви дерева видов в политомии и позволяя ILS свободно диверсифицировать генные деревья в этих политомиях. ecceTERA [131] связывает максимальный размер связанных частей дерева видов, где может произойти ILS, предлагая управляемый алгоритм с фиксированным параметром для этого параметра.
ILS и DL можно рассматривать в сети верхнего уровня , а не в дереве. Это моделирует, в частности, интрогрессию, с возможностью оценки параметров модели. [132]
Были предложены более интегративные модели согласования, учитывающие ILS, включая как DL, так и многовидовое слияние, [133] с ДЛКоал. Это вероятностная модель с экономным переводом, [134] предлагая две последовательные эвристики типа LCA, обрабатываемые через промежуточное дерево локуса между геном и видом.Однако за пределами структуры согласования генов и видов ILS, по-видимому, без какой-либо конкретной причины никогда не учитывается ни у хозяина/симбионта, ни в биогеографии.
Кофилогения с более чем двумя уровнями
[ редактировать ]
Поразительным аспектом согласования является общая методология, работающая с различными уровнями организации: она используется для сравнения деревьев доменов и белков, деревьев генов и видов, деревьев-хозяев и деревьев-симбионтов, популяционных и географических деревьев. Однако теперь, когда ученые склонны считать, что многоуровневые модели биологического функционирования привносят новый и меняющий взгляд на организмы и их окружающую среду, [136] вопрос в том, как использовать примирение, чтобы приблизить филогенетику к эре холобионтов.
В основе идеи холобионта лежит коэволюция сущностей на разных уровнях эволюции: макроорганизмы, микроорганизмы и их гены имеют разную историю, связанную с общим функционированием в единой экосистеме. Биологические системы, такие как переплетение хозяина, симбионтов и их генов, предполагают функциональные и эволюционные зависимости между более чем двумя уровнями.
Примеры многоуровневых систем со сложными эволюционными взаимозависимостями
[ редактировать ]Гены коэволюционируют за пределами генома
[ редактировать ]Концепция холобионта [137] подчеркивает возможность генов из разных геномов сотрудничать и совместно развиваться. [138] [139] [140] Например, определенные гены в геноме симбионта могут выполнять функцию своего хозяина, например, производить жизненно важное соединение, отсутствующее в доступных источниках питания. Ярким примером являются насекомые, питающиеся кровью или соком, которые часто зависят от одного или нескольких бактериальных симбионтов, чтобы процветать на ресурсе, который богат сахаром, но лишен незаменимых аминокислот или витаминов. [141] Другой пример — ассоциация Fabaceae с азотфиксирующими бактериями . Соединение-бенефициар хозяина обычно производится набором генов, закодированных в геноме симбионта, которые в ходе эволюции могут передаваться другим симбионтам и/или в геном хозяина и из него. Методы согласования потенциально могут выявить эволюционные связи между частями геномов разных видов. Поиск коэволюционирующих генов за пределами геномов, в которых они закодированы, высветил бы основу ассоциации организмов в холобионте.
Маршруты горизонтального переноса генов зависят от нескольких уровней
[ редактировать ]Во внутриклеточных симбионтных системах насекомых были идентифицированы многочисленные случаи горизонтального переноса генов, будь то от хозяина к симбионту, от симбионта к хозяину или от симбионта к симбионту. [142]
передача генов эндосимбионта, участвующих в путях питания-бенефициара, насекомому-хозяину происходит преимущественно, если линии донора и реципиента имеют одного и того же хозяина. Было показано, что [143] [144] [145] То же самое происходит и с насекомыми, бактериальные симбионты которых обеспечивают защитный белок. [146] или в облигатных клубеньковых бактериальных симбионтах, связанных с растениями. [147] Было показано, что у человека-хозяина перенос генов происходит преимущественно среди симбионтов, находящихся в одних и тех же органах. [148]
Обзор горизонтального переноса генов в системах хозяин/симбионт [149] подчеркивает важность поддержки HGT многочисленными доказательствами. В частности, утверждается, что перенос следует считать более поддерживаемым, когда в нем участвуют симбионты, имеющие общую среду обитания, географическую зону или одного и того же хозяина. Однако следует иметь в виду, что большая часть разнообразия хозяев и симбионтов неизвестна и что переносы могли произойти у невыбранных близкородственных видов, хозяев или симбионтов.
Идея о том, что перенос генов у симбионтов ограничен хозяином, также может быть использована для изучения филогенетической истории хозяина. Например, на основе филогеографических исследований в настоящее время признано, что бактерия Helicobacter pylori была связана с человеческими популяциями с момента возникновения человеческого вида . [150] [151] Анализ геномов Helicobacter pylori в Европе позволяет предположить, что они возникли в результате рекомбинации африканского и азиатского Helicobacter pylori . Это явно подразумевает ранние контакты между соответствующими человеческими популяциями.
Аналогичным образом, анализ HGT у коронавирусов разных видов млекопитающих с использованием методов согласования выявил частые контакты между вирусными линиями, что можно интерпретировать как частую смену хозяев. [152]
Культурная эволюция
[ редактировать ]Эволюция элементов человеческой культуры , например языков и народных сказок , в сочетании с генетикой человеческой популяции изучалась с использованием концепций филогенетики. Хотя примирение никогда не использовалось в этой структуре, некоторые из этих исследований охватывают несколько уровней организации, каждый из которых представлен деревом или эволюцией персонажа, с акцентом на коэволюцию этих уровней.
Языковые деревья можно сравнить с деревьями населения, чтобы выявить вертикально передаваемые народные сказки с помощью модели персонажа на этом языковом дереве. [153] Варианты семьи, языков, генетического разнообразия, населения и географии каждой сказки можно сравнить по два, чтобы связать разнообразие сказок с языками, с одной стороны, и географией, с другой. [154] Как и в случае с генетикой, когда симбионты имеют одного хозяина, способствующего HGT, языковые барьеры могут препятствовать передаче народных сказок или языковых элементов. [155]
Исследование трехуровневых систем с использованием двухуровневой сверки
[ редактировать ]Многоуровневая сверка не так развита, как двухуровневая сверка. Один из способов подойти к изучению эволюционных зависимостей между более чем двумя уровнями организации — попытаться использовать доступные стандартные двухуровневые методы, чтобы дать первое представление о сложности биологической системы.
- Множественные линии генов могут претерпевать совместные события, такие как сегментная дупликация, перенос или потеря или даже дупликация всего генома.
- Примирение может помочь определить пути перемещения и гибридизации.
- Три уровня могут быть согласованы вместе последовательно: промежуточный в верхнем перед добавлением нижнего или попытка найти общий наиболее экономичный сценарий для двух согласований.
- При более чем двух уровнях примирение нижнего и среднего уровней можно сравнить с примирением нижнего и верхнего.
Мультигенные события: неявное рассмотрение промежуточного уровня
[ редактировать ]На уровне дерева генов/видов обычно приходится иметь дело со множеством различных деревьев генов. В этом случае неявно выдвигается гипотеза о том, что разные семейства генов развиваются независимо. Однако это не обязательно так. Например, дупликация, перенос и потеря могут происходить для сегментов генома, охватывающих произвольное количество смежных генов. Учитывать такие мультигенные события можно, используя промежуточный ориентир для нижних деревьев внутри верхнего. Например, можно вычислить совместную вероятность согласования нескольких генных деревьев с датированным деревом видов с дублированием, потерей и дублированием всего генома. [5] или в скромной обстановке, [156] [157] [158] [159] и одно из определений проблемы — NP-трудное. [160] Аналогичным образом, каркас DL может быть обогащен за счет дупликации и потери сегментов хромосом вместо одного гена. Однако примирение DL становится невозможным из-за этой новой возможности. [161]
Связь между двумя последовательными генами также можно смоделировать как развивающийся признак, подверженный приобретению, потере, возникновению, разрушению, дупликации и переносу. [1] Эволюция этой связи проявляется как дополнительный уровень к видам и генным деревьям, частично ограничиваемый согласованием генного/видового дерева, а частично развивающийся сам по себе в соответствии с организацией генома. Таким образом, он моделирует синтению или близость между генами. В другом масштабе он также может моделировать эволюцию двух доменов, принадлежащих белку.
Обнаружение «магистралей переноса», то есть преимущественного приобретения групп генов от конкретного донора, является еще одним примером независимости генных историй. [162] Аналогичным образом можно обнаружить перенос нескольких генов. [163] Это также привело к методологическим разработкам, таким как сверка с использованием филогенетических сетей , рассматриваемых как дерево, дополненное ребрами переноса, которые можно использовать для ограничения переносов в модели DTL. [164] Сети также можно использовать для моделирования интрогрессии и неполной сортировки линий . [165] [166] [36]
Обнаружение коэволюции на нескольких парах уровней
[ редактировать ]Центральным вопросом для понимания эволюции холобионта является знание того, какие уровни развиваются совместно друг с другом, например, между видами-хозяевами, генами-хозяевами, симбионтами и генами-симбионтами. К множественным взаимозависимостям между всеми уровнями эволюции можно подойти путем множественных попарных сравнений двух развивающихся сущностей.
Примирение хозяина и симбионта, с одной стороны, и географии и симбионта, с другой, также может помочь выявить закономерности диверсификации хозяина и симбионта, которые отражают либо коэволюцию, либо закономерности, которые можно объяснить общей географической диверсификацией. [167] [168] [169] [170] Аналогичным образом, в исследовании использовались методы согласования, чтобы дифференцировать влияние эволюции диеты и филогенетической инерции на состав микробиомов кишечника млекопитающих . Реконструируя диету предков и состав микробиома с учетом филогении млекопитающих, исследование показало, что оба эффекта вносят свой вклад, но в разных временных масштабах. [171]
Явное моделирование трех и более уровней.
[ редактировать ]
В модели многоуровневой системы хозяин/симбионт/гены горизонтальный перенос генов должен быть более вероятен между двумя симбионтами одного и того же хозяина. Это незаметно для двухуровневого генного дерева/дерева видов или согласования хозяина/симбионта: в некоторых случаях рассмотрение любой комбинации двух уровней может привести к упущению эволюционного сценария, который может быть наиболее вероятным только в том случае, если информация из трех деревья рассматриваются вместе.
Пытаясь преодолеть ограничения использования стандартных двухуровневых согласований с системами, включающими взаимозависимости на нескольких уровнях, в последнее десятилетие были предприняты методологические усилия по построению и использованию многоуровневых моделей. Для этого необходимо выявить хотя бы один «промежуточный» уровень между верхним и нижним.
- Два уровня могут быть согласованы с ограничением верхнего, например хозяин и симбионт с географией.
- Персонажи могут развиваться на основе согласованных филогений, например, синтения генов при согласовании генов и видов.
- Переносы могут быть верхнезависимыми, что более вероятно между двумя промежуточными сущностями, принадлежащими одному и тому же верхнему.
- С помощью трехуровневых моделей согласования промежуточное дерево можно вывести из нижнего и верхнего деревьев.
Предварительное согласование: символы на согласованных деревьях
[ редактировать ]Первым шагом на пути к интегрированным трехуровневым моделям является рассмотрение филогенетических деревьев на двух уровнях, а другой уровень представлен только символами на листьях одного из деревьев.
Например, согласование филогении хозяина и симбионта может быть основано на географических данных. [172] Наследственные географические местоположения видов-хозяев и симбионтов, полученные с помощью метода вывода о характере, могут затем использоваться для ограничения примирения хозяина и симбионта: предковые хозяева и симбионты могут быть связаны только в том случае, если они принадлежат к одному и тому же географическому местоположению.
В другом масштабе к эволюции на субгенном уровне можно подойти с помощью характерного метода. [173] Здесь части генов (например, последовательность, кодирующая белковые домены) согласовываются согласно модели DL с деревом видов, а гены, которым они принадлежат, упоминаются как признаки этих частей. Затем предковые гены реконструируются апостериорно посредством слияния и разделения частей гена.
Двухуровневые сверки, информированные третьим уровнем
[ редактировать ]Как указано в нескольких исследованиях, упомянутых в § Горизонтальная передача на нескольких уровнях , верхний уровень может служить основой для согласования между промежуточным и нижним уровнями, особенно для горизонтальных передач.
Трехуровневые модели могут учитывать эти допущения для проведения сверки между промежуточным деревом и нижними уровнями со знанием верхнего дерева. Модель может, например, повысить вероятность сценариев примирения, когда горизонтальный перенос генов происходит между существами, живущими в одной и той же среде обитания . Это было достигнуто впервые за счет согласования генов и видов DTL, вложенных в домен гена DTL, и согласования генов. [174] Различные затраты на интер- и интраперенос зависят от того, происходит ли перенос между генами одних и тех же геномов.
Обратите внимание, что эта модель явно рассматривает три уровня и три дерева, но еще не определяет реальную трехуровневую сверку с соответствующей вероятностью или оценкой. [174] Он основан на последовательной операции, при которой вторая сверка определяется результатом первой.
Проблема согласования в многоуровневых моделях
[ редактировать ]Следующий шаг — определить оценку сверки, состоящей из трех вложенных деревьев, и вычислить по этим трем деревьям трехуровневые сверки в соответствии с их оценкой. Это было достигнуто с помощью системы вид/ген/домен, в которой гены развиваются внутри дерева видов с помощью модели DL, а домены развиваются внутри системы ген/вид с помощью модели DTL, что запрещает перенос доменов между генами двух разных видов. [175] Вывод включает сценарии-кандидаты с общими оценками. Вычисление сценария минимального балла является NP-сложным, но динамическое программирование или целочисленное линейное программирование могут предложить эвристику. [175] [176] Варианты задачи с учетом нескольких доменов [177] доступны, как и среда моделирования. [178]
Вывод промежуточного дерева с использованием моделей трехуровневой нижней/промежуточной/верхней сверки
[ редактировать ]Точно так же, как двухуровневое согласование можно использовать для улучшения нижних или верхних филогений или для помощи в их построении из выровненных последовательностей, таким же образом можно использовать модели совместного согласования.
В этом смысле объединенная модель DL гена/вида, DL домена гена и модели эволюции последовательности генов в байесовской системе улучшает реконструкцию генных деревьев. [179]
Программное обеспечение
[ редактировать ]Для реализации различных моделей выверки было разработано множество программ. Следующая таблица не претендует на полноту, но представляет ряд программных инструментов, предназначенных для согласования деревьев для определения сценариев согласования или для соответствующего использования, такого как исправление или вывод деревьев или тестирование коэволюции. В разделе «Уровни интереса» подробно описаны уровни, для которых было реализовано программное обеспечение, хотя вполне возможно, например, использовать программное обеспечение, созданное для согласования видов и генов, для согласования хозяина и симбионтов. [180] Экономия или вероятность — это базовая модель, которая используется для сверки.
| Имя | Уровни интереса | Платформа | Командная строка или графический интерфейс пользователя (GUI) | Использование | Вероятность или экономия | Наличие программного обеспечения и лицензия на программное обеспечение |
|---|---|---|---|---|---|---|
| Дива [181] | География и виды | Unix , Mac , Win | Командная строка | Вывод о примирении | бережливость | GNU GPLv2 |
| Лагранж [182] | География и виды | Линукс , Мак, Вин | Пакет Python | Вывод о примирении | Вероятность | GNU GPLv2 |
| БиоГеоБЕАРС [183] | География и виды | пакет R | пакет R | Вывод о сверке, проверка статистической модели | Вероятность | GNU GPLv2, GNU GPLv3 |
| Джейн [184] | Хозяин и симбионты | Юникс, Мак, Победа | Графический интерфейс или командная строка | Вывод о согласовании, неопределенность дерева | бережливость | Собственный , регистрация для скачивания |
| императрица [185] | Хозяин и симбионты | Юникс, Мак, Победа | Графический интерфейс или командная строка | Выводы по согласованию, оценка затрат, исследование пространства решений | бережливость | GNU GPLv3 |
| Эвкалипт [186] | Хозяин и симбионты | Ява | Командная строка, графический вывод с включенной программой просмотра CophyTrees | Вывод о согласовании, исследование пространства решений | бережливость | CeCILL |
| Капибара [187] | Хозяин и симбионты | Пакет Linux, Mac, Win и Python | Графический интерфейс, пакет Python | Вывод о согласовании, исследование пространства решений | бережливость | код доступен на github |
| Лист [188] | Хозяин и симбионты | Линукс, Мак | Командная строка | оценка стоимости | бережливость | CeCILL |
| РЕЙНДЖЕР-ДТЛ [189] | Виды и гены | Линукс, Мак, Вин | Командная строка | Вывод сверки, неопределенность дерева, выборка решений, замена переносов | бережливость | GNU GPLv3 |
| Обозначения [190] | Виды и гены | Linux, Mac, Win <7 | графический интерфейс | Вывод о примирении, неопределенность дерева, ген, генный домен, видовая модель | бережливость | Собственный |
| Маугли [191] | Виды и гены, Хозяин и симбионт | Линукс, Мак | Командная строка, графический вывод с помощью совместимой программы просмотра Sylvx | Вывод сверки, неопределенность дерева, ILS, географические ограничения, ввод датированного дерева | бережливость | Собственный |
| Сильвкс [192] | Виды и гены, Хозяин и симбионт | Линукс, Мак, Вин | графический интерфейс | Просмотрщик, совместимый с Маугли, ecceTERA | - | GNU GPLv2 или новее |
| АНГСТ [193] | Виды и гены | Питон 2 | Командная строка | Выводы о сверке, оценка затрат, датированные входные данные дерева, неопределенность дерева | бережливость | программное обеспечение, доступное в исходном коде [194] |
| и т. д. [195] | Виды и гены | Linux, Mac, построенные из кода | Командная строка, совместимая со средством просмотра Sylvx, и вывод Recphyloxml. | Выводы о сверке, оценка стоимости, датированные, частично датированные или недатированные входные данные дерева видов, неопределенность дерева, исследование пространства сверки, сеть видов | бережливость | CeCILL |
| НО [196] | Виды и гены | Линукс, Мак | Командная строка | Выводы о сверке, оценка затрат, датированные или недатированные входные данные дерева пород, неопределенность дерева | Вероятность | GNU GPLv3 |
| Трирекс [197] | Виды и гены | Линукс, Мак, Вин | Графический интерфейс, интегрированный в Seaview | Коррекция дерева DL | Экономия, Вероятность | GNU Affero GPL версии 3.0 или новее |
| ДжинРакс [198] | Виды и гены | Линукс, Мак | Командная строка, графический вывод с помощью Recphyloxml и Thirdkind | Вывод генного дерева на основе сверки и выровненных последовательностей, вывод дерева видов | Вероятность | GNU представляет GPL v3.0 |
| Филдог [199] | Виды и гены | Linux, докер, виртуальная машина | Командная строка | Вывод генного и видового дерева на основе сверки и выровненных последовательностей | Вероятность | CeCILL |
| КоРе-ПА [200] | Хозяин и симбионт | Линукс, Мак, Вин | Графический интерфейс, командная строка, графический вывод SVG | Вывод о сверке, оценка затрат, датированное дерево, статистический тест | бережливость | - |
| CoRe-ILP [201] | Хозяин и симбионт | Линукс, Мак, Вин | Командная строка | Вывод о примирении, временная осуществимость, датированное дерево | бережливость | требуется CPLEX (академическую лицензию можно получить бесплатно) |
| Мошенник [55] | Хозяин и симбионт | Ява | Командная строка | Вывод о согласовании, датированное дерево | бережливость | - |
| MixTreEM [ мертвая ссылка ] | Виды и ген | Linux, Mac, Win (сборка из исходников) | Командная строка | Вывод генного и видового дерева на основе сверки и выровненных последовательностей | Вероятность | |
| Основной [ мертвая ссылка ] | Виды и ген | Линукс, Мак | Командная строка, графический вывод (ПрИМЕТВ) | Вывод по согласованию, вывод генного и видового дерева на основе согласования и выровненных последовательностей, анализ ортологии | Вероятность | |
| Древовидная карта [ мертвая ссылка ] | Хозяин и симбионт | Ява | Командная строка | Вывод о сверке, статистический тест | бережливость | |
| ДЖПРАЙМ [202] | Ген и виды | Java-библиотека | Командная строка | Вывод о сверке, вывод о генном и видовом дереве на основе сверки и выровненных последовательностей | Вероятность | Новый БСД |
| СЕАДОГ [203] | Виды, гены и домены | Линукс, Мак | Командная строка | Трехуровневый вывод сверки | бережливость | GNU GPLv3 |
| iGTP [204] | Виды и гены | Линукс, Мак, Вин | графический интерфейс | Коррекция генного дерева при DL или глубоком слиянии | бережливость | Исходный код по запросу |
| TreeSolve [205] | Виды и гены | Линукс, Вин | графический интерфейс | Коррекция генного дерева в DTL | бережливость | Исходный код по запросу |
| ДеревоФикс, [206] TreeFix-DTL [207] | Виды и гены | Линукс | Командная строка | Коррекция генного дерева в DL и DTL | бережливость | GNU GPLv3 |
| АРТра [208] | Виды и гены | Линукс, Мак | Командная строка | Вывод аддитивных и заменяющих переносов | бережливость | GNU GPL |
| ДЛКоул [209] | Виды и гены | Линукс, Мак, Вин | Командная строка | Вывод сверки с ILS | бережливость | GNU GPL |
| Третий вид [210] | Виды и гены | Линукс, Мак, Вин | Командная строка | Просмотрщик, совместимый с Recphyloxml | - | CeCILL |
Ссылки
[ редактировать ]![]() Эта статья была адаптирована из следующего источника под лицензией CC BY 4.0 ( 2022 г. ): Хьюго Мене; Винсент Добен; Эрик Таннье (3 ноября 2022 г.). «Филогенетическое примирение» . PLOS Вычислительная биология . 18 (11): e1010621. doi : 10.1371/JOURNAL.PCBI.1010621 . ISSN 1553-734X . Викиданные Q115175616 .
Эта статья была адаптирована из следующего источника под лицензией CC BY 4.0 ( 2022 г. ): Хьюго Мене; Винсент Добен; Эрик Таннье (3 ноября 2022 г.). «Филогенетическое примирение» . PLOS Вычислительная биология . 18 (11): e1010621. doi : 10.1371/JOURNAL.PCBI.1010621 . ISSN 1553-734X . Викиданные Q115175616 .
- ↑ Перейти обратно: Перейти обратно: а б Дюшемен, В.; Ансельметти, Ю.; Паттерсон, М.; Понти, Ю.; Берар, С.; Шов, К.; Скорнавакка, К.; Добин, В.; Танньер, Э. (2017). «DeCoSTAR: реконструкция наследственной организации генов или геномов с использованием согласованных филогений» . Геномная биология и эволюция . 9 (5): 1312–1319. дои : 10.1093/gbe/evx069 . ПМЦ 5441342 . ПМИД 28402423 .
- ^ Лорето, Эль; Карарето, CM; Кэпи, П. (2008). «Возвращаясь к горизонтальному переносу мобильных элементов у дрозофилы» . Наследственность . 100 (6): 545–554. дои : 10.1038/sj.hdy.6801094 . ПМИД 18431403 . S2CID 32424144 .
- ^ Дениз, Р.; Эбби, СС; Роча EPC (2019). «Диверсификация суперсемейства филаментов типа IV в машины для адгезии, секреции белка, поглощения ДНК и подвижности» . ПЛОС Биология . 17 (7): e3000390. дои : 10.1371/journal.pbio.3000390 . ПМЦ 6668835 . ПМИД 31323028 .
- ↑ Перейти обратно: Перейти обратно: а б с Фельзенштейн Дж. (2004) Выводы о филогениях. Издательство Оксфордского университета
- ↑ Перейти обратно: Перейти обратно: а б Звенепол, А.; Ван Де Пер, Ю. (2019). «Вывод о древних дупликациях всего генома и эволюция дупликации и уровня потерь генов». Молекулярная биология и эволюция . 36 (7): 1384–1404. дои : 10.1093/molbev/msz088 . hdl : 1854/LU-8612728 . ПМИД 31004147 .
- ↑ Перейти обратно: Перейти обратно: а б Баговский, КП; Брюинз, В.; Те Велтуис, Эй Джей (2010). «Природа эволюции белковых доменов: формирование сети взаимодействия» . Современная геномика . 11 (5): 368–376. дои : 10.2174/138920210791616725 . ПМК 2945003 . ПМИД 21286315 .
- ^ Наир, НУ; Лин, Ю.; Манасовская А.; Антич, Дж.; Грнарова П.; Саху, AD; Бучер, П.; Море, Б.М. (2014). «Изучение дифференцировки клеток методом филогенетического анализа с использованием данных модификации гистонов» . БМК Биоинформатика . 15 (1): 269. дои : 10.1186/1471-2105-15-269 . ПМЦ 4138389 . ПМИД 25104072 .
- ^ Вёзе, ЧР; Кандлер, О.; Уилис, ML (1990). «На пути к естественной системе организмов: предложение по доменам архей, бактерий и эукариев» . Труды Национальной академии наук Соединенных Штатов Америки . 87 (12): 4576–4579. Бибкод : 1990PNAS...87.4576W . дои : 10.1073/pnas.87.12.4576 . ПМК 54159 . ПМИД 2112744 .
- ^ Добжанский Т.; Стертевант, А.Х. (1938). «Инверсии хромосом дрозофилы Pseudoobscura» . Генетика . 23 (1): 28–64. дои : 10.1093/генетика/23.1.28 . ПМК 1209001 . ПМИД 17246876 .
- ↑ Перейти обратно: Перейти обратно: а б Цукеркандль, Э.; Полинг, Л. (1965). «Молекулы как документы эволюционной истории». Журнал теоретической биологии . 8 (2): 357–366. Бибкод : 1965JThBi...8..357Z . дои : 10.1016/0022-5193(65)90083-4 . ПМИД 5876245 .
- ^ Грей, РД; Брайант, Д.; Гринхилл, SJ (2010). «О форме и ткани человеческой истории» . Философские труды Лондонского королевского общества. Серия Б, Биологические науки . 365 (1559): 3923–3933. дои : 10.1098/rstb.2010.0162 . ПМК 2981918 . ПМИД 21041216 .
- ^ Тегерани, Джей-Джей (2013). «Филогения Красной Шапочки» . ПЛОС ОДИН . 8 (11): e78871. Бибкод : 2013PLoSO...878871T . дои : 10.1371/journal.pone.0078871 . ПМЦ 3827309 . ПМИД 24236061 .
- ↑ Перейти обратно: Перейти обратно: а б с Визеке, Николас; Бернт, Матиас; Миддендорф, Мартин (30 июля 2013 г.). «Объединяющее экономное примирение деревьев». arXiv : 1307.7831 [ q-bio.QM ].
- ↑ Перейти обратно: Перейти обратно: а б с Гудман, Моррис; Челюсняк, Джон; Мур, Дж. Уильям; Ромеро-Эррера, А.Е.; Мацуда, Гэндзи (1979). «Вписывание генной линии в ее видовую линию: стратегия экономии, иллюстрированная кладограммами, построенными на основе последовательностей глобина». Систематическая зоология . 28 (2): 132–163. дои : 10.2307/2412519 . JSTOR 2412519 .
- ↑ Перейти обратно: Перейти обратно: а б Морель, Б.; Козлов А.М.; Стаматакис, А.; Сёллёси, Г.Дж. (2020). «GeneRax: инструмент для определения генеалогического древа генов на основе максимального правдоподобия с учетом древа видов при дублировании, переносе и потере генов» . Молекулярная биология и эволюция . 37 (9): 2763–2774. дои : 10.1093/molbev/msaa141 . ПМЦ 8312565 . ПМИД 32502238 .
- ^ Уэйн П. Мэддисон (1997) Генные деревья в деревьях видов, Систематическая биология, 46 (3) 523-536.
- ^ Де Вьен, DM (2019). «Танглограммы вводят в заблуждение при визуальной оценке конгруэнтности деревьев». Молекулярная биология и эволюция . 36 (1): 174–176. дои : 10.1093/molbev/msy196 . ПМИД 30351416 .
- ↑ Перейти обратно: Перейти обратно: а б с Брукс, Дэниел Р. (1981). «Паразитологический метод Хеннига: предлагаемое решение». Систематическая зоология . 30 (3): 229–249. дои : 10.2307/2413247 . JSTOR 2413247 .
- ^ Снит, PHA (1982). «Рецензируемая работа: Систематика и биогеография: кладистика и викариантность. II., Гарет Нельсон, Норман Платник». Систематическая зоология . 31 (2): 208–217. дои : 10.2307/2413040 . JSTOR 2413040 .
- ^ Ронквист, Фредрик; Найлин, Сорен (1990). «Процесс и закономерности эволюции видовых ассоциаций». Систематическая зоология . 39 (4): 323–344. дои : 10.2307/2992354 . JSTOR 2992354 .
- ^ Пейдж РДМ (1990). «Анализ компонентов: доблестный провал?». Кладистика . 6 (2): 119–136. дои : 10.1111/j.1096-0031.1990.tb00532.x . ПМИД 34933509 . S2CID 86307275 .
- ↑ Перейти обратно: Перейти обратно: а б с д Чарльстон, Массачусетс (1998). «Джунгли: новое решение проблемы согласования филогении хозяина и паразита». Математические биологические науки . 149 (2): 191–223. дои : 10.1016/s0025-5564(97)10012-8 . ПМИД 9621683 .
- ^ Хейн, Йотун (1993). «Эвристический метод восстановления истории последовательностей, подлежащих рекомбинации». Журнал молекулярной эволюции . 36 (4): 396. Бибкод : 1993JMolE..36..396H . дои : 10.1007/BF00182187 . S2CID 16346458 .
- ^ Халлетт, Монтана; Лагергрен, Дж. (2001). «Эффективные алгоритмы решения задач латерального переноса генов». Материалы пятой ежегодной международной конференции по вычислительной биологии - RECOMB '01 . стр. 149–156. дои : 10.1145/369133.369188 . ISBN 1581133537 . S2CID 5804061 .
- ^ Пейдж, RDM (1994). «Карты между деревьями и кладистический анализ исторических ассоциаций между генами, организмами и территориями». Систематическая биология . 43 : 58–77. дои : 10.1093/sysbio/43.1.58 .
- ^ Хафнер, М.С.; Надлер, С.А. (1988). «Филогенетические деревья поддерживают совместную эволюцию паразитов и их хозяев». Природа . 332 (6161): 258–259. Бибкод : 1988Natur.332..258H . дои : 10.1038/332258a0 . ПМИД 3347269 . S2CID 4236760 .
- ↑ Перейти обратно: Перейти обратно: а б Пейдж, Родерик Д.М. (июнь 1994 г.). «Параллельные филогении: реконструкция истории скоплений хозяина и паразита» . Кладистика . 10 (2): 155–173. дои : 10.1111/j.1096-0031.1994.tb00170.x .
- ^ Ронквист, Ф. (1995). «Реконструкция истории ассоциаций хозяин-паразит с использованием обобщенной экономности» . Кладистика . 11 (1): 73–89. дои : 10.1111/j.1096-0031.1995.tb00005.x . ПМИД 34920597 . S2CID 86298930 .
- ^ DTL означает «Дублирование, горизонтальная передача и потеря».
- ^ Эта таблица предназначена служить иллюстрацией к разделу о двухуровневой сверке Menet et al. (2022) Филогенетическое примирение. PLoS Comput Biol 18 (11): e1010621. doi:10.1371/journal.pcbi.1010621 и его можно прочитать вместе с ним. Добавление события горизонтального переноса добавляет новые, более экономичные решения по сравнению с предыдущей моделью DL (A). В этом новом событии затраты должны быть назначены событиям D, T и L, а разные затраты дают разные решения (B). Не все сценарии, включая трансферты, осуществимы во времени. Некоторые из них могут включать временные ограничения, несовместимые с верхним деревом (C). Передача может произойти от вида к одному из его потомков через вымершие сестринские линии (D). В биогеографии можно построить древовидную структуру для учета возможных миграций между различными географическими областями (E). В некоторых случаях экспоненциальное число сценариев может быть наиболее экономным, например, когда два эквивалентных шаблона имеют одинаковую стоимость (F). Нижнее дерево может быть некорневым (G), разветвленным (H) или представлено в качестве образца потенциальных деревьев (I), а сверка может использоваться для устранения этих неопределенностей и получения нижнего дерева с двоичным корнем. Показатель согласованности также можно использовать для построения верхнего дерева (J). Динамическое программирование ограничено тем фактом, что оно предполагает независимость между сестринскими линиями, что делает невозможным рассмотрение замены переноса или конверсии генов (K), а также отказ от дивергенции (L) и неполную сортировку линий (M), два уровня популяции. события.
- ^ Уайли, Э.О. (1988). «Анализ бережливости и биогеография викарианства». Систематическая зоология . 37 (3): 271–290. дои : 10.2307/2992373 . JSTOR 2992373 .
- ^ Чурос, М.; Миклош, И. (2009). «Упорядочение и большие предковые геномы архей, выявленные с помощью филогенетической модели рождения и смерти» . Молекулярная биология и эволюция . 26 (9): 2087–2095. дои : 10.1093/molbev/msp123 . ПМК 2726834 . ПМИД 19570746 .
- ^ Груссен, М.; Добин, В.; Гуи, М.; Танньер, Э. (2016). «Реконструкция предков: теория и практика». Энциклопедия эволюционной биологии (PDF) . стр. 70–77. дои : 10.1016/B978-0-12-800049-6.00166-9 . ISBN 9780128004265 .
- ↑ Перейти обратно: Перейти обратно: а б Арвестад, Л.; Берглунд, AC; Лагергрен, Дж.; Сеннблад, Б. (2003). «Сверка байесовского гена/дерева видов и анализ ортологии с использованием MCMC». Биоинформатика . 19 (Приложение 1): i7-15. doi : 10.1093/биоинформатика/btg1000 . ПМИД 12855432 .
- ^ Арвестад, Ларс; Берглунд, Анн-Шарлотта; Лагергрен, Йенс; Сеннблад, Бенгт (2004). «Реконструкция генного дерева и анализ ортологии на основе интегрированной модели дупликаций и эволюции последовательностей». Материалы восьмой ежегодной международной конференции по вычислительной молекулярной биологии - RECOMB '04 . стр. 326–335. дои : 10.1145/974614.974657 . ISBN 1581137559 . S2CID 12364792 .
- ↑ Перейти обратно: Перейти обратно: а б Ю, Ю.; Донг, Дж.; Лю, К.Дж.; Нахле, Л. (2014). «Вывод с максимальным правдоподобием сетчатой эволюционной истории» . Труды Национальной академии наук Соединенных Штатов Америки . 111 (46): 16448–16453. Бибкод : 2014PNAS..11116448Y . дои : 10.1073/pnas.1407950111 . ПМЦ 4246314 . ПМИД 25368173 .
- ^ Чурёш, М. (2010). «Подсчет: эволюционный анализ филогенетических профилей с экономией и правдоподобием» . Биоинформатика . 26 (15): 1910–1912. doi : 10.1093/биоинформатика/btq315 . ПМИД 20551134 .
- ^ Шов С. и Эль-Мабрук Н. (2009) Новые перспективы эволюции семейства генов: потери в примирении и связь с супердеревьями. РЕКОМБ 5541:46-58
- ↑ Перейти обратно: Перейти обратно: а б Дойон Дж., Скорнавакка С., Горбунов К., Сёллёси Г., Ранвез В. и др. (2010) Эффективный алгоритм для экономного согласования деревьев генов и видов с потерями, дублированием и переносами. РЕКОМБ-CG
- ↑ Перейти обратно: Перейти обратно: а б с Сёллёси, Г.Дж.; Росикевич, В.; Буссау, Б.; Танньер, Э.; Добин, В. (2013). «Эффективное исследование пространства согласованных генных деревьев» . Систематическая биология . 62 (6): 901–912. дои : 10.1093/sysbio/syt054 . ПМЦ 3797637 . ПМИД 23925510 .
- ↑ Перейти обратно: Перейти обратно: а б Ри, Р.Х.; Смит, С.А. (2008). «Вывод с максимальной вероятностью об эволюции географического ареала путем расселения, локального вымирания и кладогенеза» . Систематическая биология . 57 (1): 4–14. дои : 10.1080/10635150701883881 . ПМИД 18253896 . S2CID 14205291 .
- ↑ Перейти обратно: Перейти обратно: а б Жакокс, Э.; Шов, К.; Сёллёси, Г.Дж.; Понти, Ю.; Скорнавакка, К. (2016). «EcceTERA: Комплексное согласование генных и видовых деревьев с использованием экономии» . Биоинформатика . 32 (13): 2056–2058. doi : 10.1093/биоинформатика/btw105 . ПМИД 27153713 .
- ^ Дойон, JP; Ранвез, В.; Добин, В.; Берри, В. (2011). «Модели, алгоритмы и программы для согласования филогении» . Брифинги по биоинформатике . 12 (5): 392–400. дои : 10.1093/нагрудник/bbr045 . ПМИД 21949266 .
- ^ Бансал, Миссисипи; Альм, Э.Дж.; Келлис, М. (2012). «Эффективные алгоритмы решения проблемы согласования при дупликации генов, горизонтальном переносе и потере» . Биоинформатика . 28 (12): i283-91. doi : 10.1093/биоинформатика/bts225 . ПМК 3371857 . ПМИД 22689773 .
- ↑ Перейти обратно: Перейти обратно: а б с Сёллоси, Г.Дж.; Буссау, Б.; Эбби, СС; Танньер, Э.; Добин, В. (2012). «Филогенетическое моделирование латерального переноса генов реконструирует структуру и относительное время видообразования» . Труды Национальной академии наук Соединенных Штатов Америки . 109 (43): 17513–17518. Бибкод : 2012PNAS..10917513S . дои : 10.1073/pnas.1202997109 . ПМЦ 3491530 . ПМИД 23043116 .
- ↑ Перейти обратно: Перейти обратно: а б Меркл, Д.; Миддендорф, М.; Визеке, Н. (2010). «Подход к динамическому программированию, адаптивный к параметрам, для вывода кофилогений» . БМК Биоинформатика . 11 (Приложение 1): S60. дои : 10.1186/1471-2105-11-S1-S60 . ПМК 3009534 . ПМИД 20122236 .
- ^ Боде, К.; Донати, Б.; Синаймери, Б.; Крещенци, П.; Готье, К.; Матиас, К.; Саго, МФ (2015). «Реконструкция кофилогении с помощью приближенного байесовского расчета» . Систематическая биология . 64 (3): 416–431. дои : 10.1093/sysbio/syu129 . ПМЦ 4395844 . ПМИД 25540454 .
- ^ Либескинд-Хадас, Р.; Ву, ЮК; Бансал, Миссисипи; Келлис, М. (2014). «Согласование Парето-оптимального филогенетического дерева» . Биоинформатика . 30 (12): i87-95. doi : 10.1093/биоинформатика/btu289 . ПМК 4058917 . ПМИД 24932009 .
- ↑ Перейти обратно: Перейти обратно: а б Дэвид, Луизиана; Альм, Э.Дж. (2011). «Быстрые эволюционные инновации во время архейской генетической экспансии». Природа . 469 (7328): 93–96. Бибкод : 2011Natur.469...93D . дои : 10.1038/nature09649 . hdl : 1721.1/61263 . ПМИД 21170026 . S2CID 4420725 .
- ^ Халлетт, Монтана; Лагергрен, Дж. (2001). «Эффективные алгоритмы решения задач латерального переноса генов». Материалы пятой ежегодной международной конференции по вычислительной биологии . стр. 149–156. дои : 10.1145/369133.369188 . ISBN 1581133537 . S2CID 5804061 .
- ↑ Перейти обратно: Перейти обратно: а б с Тофиг, А.; Халлетт, М.; Лагергрен, Дж. (2011). «Одновременная идентификация дупликаций и латеральных переносов генов» . Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 8 (2): 517–535. дои : 10.1109/TCBB.2010.14 . ПМИД 21233529 . S2CID 16462428 .
- ^ Овадия, Ю.; Филдер, Д.; Коноу, К.; Либескинд-Хадас, Р. (2011). «Проблема реконструкции кофилогении NP-полна». Журнал вычислительной биологии . 18 (1): 59–65. дои : 10.1089/cmb.2009.0240 . ПМИД 20715926 .
- ^ Визеке, Н.; Хартманн, Т.; Бернт, М.; Миддендорф, М. (2015). «Кофилогенетическое согласование с ILP». Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 12 (6): 1227–1235. дои : 10.1109/TCBB.2015.2430336 . ПМИД 26671795 . S2CID 2291208 .
- ^ Дринкуотер, Бенджамин; Чарльстон, Майкл А. (2 января 2014 г.). «Улучшенный алгоритм сопоставления узлов для задачи реконструкции кофилогении» . Коэволюция . 2 (1): 1–17. дои : 10.1080/23256214.2014.906070 .
- ↑ Перейти обратно: Перейти обратно: а б Дринкуотер, Б.; Чарльстон, Массачусетс (2016). «RASCAL: рандомизированный подход к коэволюционному анализу» . Журнал вычислительной биологии . 23 (3): 218–227. дои : 10.1089/cmb.2015.0111 . ПМИД 26828619 .
- ↑ Перейти обратно: Перейти обратно: а б с д Коноу, К.; Филдер, Д.; Овадия, Ю.; Либескинд-Хадас, Р. (2010). «Джейн: Новый инструмент для решения проблемы реконструкции кофилогении» . Алгоритмы молекулярной биологии . 5:16 . дои : 10.1186/1748-7188-5-16 . ПМЦ 2830923 . ПМИД 20181081 .
- ↑ Перейти обратно: Перейти обратно: а б с Дюран, Д.; Халлдорссон, Б.В.; Верно, Б. (2006). «Гибридный микро-макроэволюционный подход к реконструкции генного дерева» . Журнал вычислительной биологии . 13 (2): 320–335. дои : 10.1089/cmb.2006.13.320 . ПМИД 16597243 .
- ↑ Перейти обратно: Перейти обратно: а б с д и Донати, Б.; Боде, К.; Синаймери, Б.; Крещенци, П.; Саго, МФ (2015). «ЕВКАЛИПТ: Эффективный счетчик согласования деревьев» . Алгоритмы молекулярной биологии . 10 (1): 3. дои : 10.1186/s13015-014-0031-3 . ПМК 4310143 . ПМИД 25648467 .
- ↑ Перейти обратно: Перейти обратно: а б Ма, В.; Смирнов Д.; Форман, Дж.; Швейкарт, А.; Слокам, К.; Шринивасан, С.; Либескинд-Хадас, Р. (2018). «DTL-RNB: Алгоритмы и инструменты суммирования пространства согласований DTL» . Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 15 (2): 411–421. дои : 10.1109/TCBB.2016.2537319 . ПМИД 26955051 . S2CID 4566350 .
- ↑ Перейти обратно: Перейти обратно: а б Шов, Седрик; Рафией, Акбар; Давин, Адриан А.; Скорнавакка, Селин; Вебер, Филипп; Буссау, Бастьен; Сёллёси, Гергеи Дж.; Добен, Винсент; Таннье, Эрик (14 апреля 2017 г.). «MaxTiC: Быстрое ранжирование филогенетического дерева по максимальной временной согласованности с боковым переносом генов». дои : 10.1101/127548 . S2CID 21910535 .
{{cite journal}}: Для цитирования журнала требуется|journal=( помощь ) - ^ Давин, А.А.; Танньер, Э.; Уильямс, штат Калифорния; Буссау, Б.; Добин, В.; Сёллёси, Г.Дж. (2018). «Перенос генов позволяет датировать древо жизни» . Экология и эволюция природы . 2 (5): 904–909. Бибкод : 2018NatEE...2..904D . дои : 10.1038/s41559-018-0525-3 . ПМЦ 5912509 . ПМИД 29610471 .
- ↑ Перейти обратно: Перейти обратно: а б Сёллоси, Г.Дж.; Танньер, Э.; Лартильо, Н.; Добин, В. (2013). «Боковой перенос генов от мертвых» . Систематическая биология . 62 (3): 386–397. дои : 10.1093/sysbio/syt003 . ПМЦ 3622898 . ПМИД 23355531 .
- ^ Давин, А.А.; Трику, Т.; Танньер, Э.; Де Вьен, DM; Сёллёси, Г.Дж. (2020). «Зомби: филогенетический симулятор деревьев, геномов и последовательностей, учитывающий мертвые линии» . Биоинформатика . 36 (4): 1286–1288. doi : 10.1093/биоинформатика/btz710 . ПМЦ 7031779 . PMID 31566657 .
- ^ Вайнер, Самсон; Бансал, Мукул С. (5 августа 2021 г.). «Улучшенное согласование дублирования, переноса и потерь с вымершими и невыбранными родословными» . Алгоритмы . 14 (8): 231. дои : 10.3390/a14080231 .
- ^ Сёллёси, Г.Дж.; Давин, А.А.; Танньер, Э.; Добин, В.; Буссау, Б. (2015). «Филогенетический анализ в масштабе генома обнаруживает обширный перенос генов среди грибов» . Философские труды Лондонского королевского общества. Серия Б, Биологические науки . 370 (1678). дои : 10.1098/rstb.2014.0335 . ПМЦ 4571573 . ПМИД 26323765 .
- ^ Ронквист Ф (1997)Филогенетические подходы в коэволюции и биогеографии. Зоологика Скрипта 26:313--322
- ^ Ри, Р.Х.; Мур, БР; Уэбб, Колорадо; Донохью, MJ (2005). «Система правдоподобия для вывода об эволюции географического ареала на филогенетических деревьях» . Эволюция; Международный журнал органической эволюции . 59 (11): 2299–2511. дои : 10.1111/j.0014-3820.2005.tb00940.x . ПМИД 16396171 . S2CID 23245573 .
- ↑ Перейти обратно: Перейти обратно: а б Мацке, Нью-Джерси (2014). «Выбор модели в исторической биогеографии показывает, что видообразование в результате события-основателя является решающим процессом в островных кладах» . Систематическая биология . 63 (6): 951–970. дои : 10.1093/sysbio/syu056 . ПМИД 25123369 .
- ^ Ван Дам, Мэтью Х.; Мацке, Николас Дж. (2016). «Оценка влияния связности и расстояния на биогеографические закономерности в юго-западных пустынях Северной Америки». Журнал биогеографии . 43 (8): 1514–1532. Бибкод : 2016JBiog..43.1514V . дои : 10.1111/jbi.12727 . S2CID 87276317 .
- ↑ Перейти обратно: Перейти обратно: а б Сантичайвекин, С.; Ян, К.; Лю, Дж.; Маухортер, Р.; Цзян, Дж.; Уэсли, Т.; Ву, ЮК; Либескинд-Хадас, Р. (2021). «EMPRess: инструмент систематического согласования кофилогении» . Биоинформатика . 37 (16): 2481–2482. doi : 10.1093/биоинформатика/btaa978 . ПМИД 33216126 .
- ^ Сеннблад, Б.; Шрейль, Э.; Берглунд Зоннхаммер, AC; Лагергрен, Дж.; Арвестад, Л. (2007). «Приметв: Зритель примирившихся деревьев» . БМК Биоинформатика . 8 : 148. дои : 10.1186/1471-2105-8-148 . ЧВК 1891116 . ПМИД 17484781 .
- ^ Шевене, Ф.; Дойон, JP; Скорнавакка, К.; Жакокс, Э.; Жуслен, Э.; Берри, В. (2016). «SylvX: средство просмотра согласований филогенетических деревьев» . Биоинформатика . 32 (4): 608–610. doi : 10.1093/биоинформатика/btv625 . ПМИД 26515823 .
- ^ Дюшемен, В.; Генс, Г.; Аригон Шифолло, AM; Рецензент Л.; Бансал, Миссисипи; Берри, В.; Буссау, Б.; Шевене, Ф.; Конт, Н.; Дэвин, А.А.; Дессимос, Дж.; Дилус, Д.; Хашич, Д.; Малло, Д.; Планель, Р.; Посада, Д.; Скорнавакка, К.; Соллози, Г.; Чжан, Л.; Добин, В. (2018). «RecPhyloXML: формат согласованных генных деревьев» . Биоинформатика . 34 (21): 3646–3652. doi : 10.1093/биоинформатика/bty389 . ПМК 6198865 . ПМИД 29762653 .
- ^ Пенель, С.; Мене, Х.; Трику, Т.; Добин, В.; Танньер, Э. (2022). «Третий род: отображение филогенетических встреч за пределами двухуровневого согласования». Биоинформатика . 38 (8): 2350–2352. doi : 10.1093/биоинформатика/btac062 . ПМИД 35139153 .
- ↑ Перейти обратно: Перейти обратно: а б Бансал, Миссисипи; Альм, Э.Дж.; Келлис, М. (2013). «Еще раз о сверке: обработка нескольких оптимумов при согласовании с дублированием, переносом и потерей» . Журнал вычислительной биологии . 20 (10): 738–754. дои : 10.1089/cmb.2013.0073 . ПМК 3791060 . ПМИД 24033262 .
- ^ Нгуен, TH; Ранвез, В.; Берри, В.; Скорнавакка, К. (2013). «Поддержка мер по оценке достоверности эволюционных событий, предсказанных методами согласования» . ПЛОС ОДИН . 8 (10): е73667. Бибкод : 2013PLoSO...873667N . дои : 10.1371/journal.pone.0073667 . ПМЦ 3790797 . ПМИД 24124449 .
- ↑ Перейти обратно: Перейти обратно: а б Кунду, С.; Бансал, М.С. (2018). «О влиянии неопределенного укоренения генного дерева на согласование потерь дублирования, переноса и потери» . БМК Биоинформатика . 19 (Приложение 9): 290. doi : 10.1186/s12859-018-2269-0 . ПМК 6101088 . ПМИД 30367593 .
- ^ Хубер К., Моултон В., Сагот М.Ф., Синаймери Б. (2018) Геометрические медианы в пространствах согласования филогенетических деревьев. Письмо об обработке информации. 136: 96–101
- ^ Маухортер, Р.; Либескинд-Хадас, Р. (2019). «Иерархическая группировка максимально экономных согласований» . БМК Биоинформатика . 20 (1): 612. doi : 10.1186/s12859-019-3223-5 . ПМК 6882150 . ПМИД 31775628 .
- ^ Сантичайвекин, С.; Маухортер, Р.; Либескинд-Хадас, Р. (2019). «Эффективный точный алгоритм для вычисления всех попарных расстояний между согласованиями в модели потерь при дублировании и передаче» . БМК Биоинформатика . 20 (Приложение 20): 636. doi : 10.1186/s12859-019-3203-9 . ПМЦ 6915856 . ПМИД 31842734 .
- ^ Ван, Ю.; Мэри, А.; Саго, МФ; Синаймери, Б. (2020). «Капибара: перечисление классов эквивалентности кофилогенных событийных согласований» . Биоинформатика . 36 (14): 4197–4199. doi : 10.1093/биоинформатика/btaa498 . ПМИД 32556075 .
- ^ Буссау, Б.; Добин, В. (2010). «Геномы как документы эволюционной истории». Тенденции в экологии и эволюции . 25 (4): 224–232. дои : 10.1016/j.tree.2009.09.007 . ПМИД 19880211 .
- ^ Хан, М.В. (2007). «Смещение в методах согласования филогенетических деревьев: последствия для эволюции генома позвоночных» . Геномная биология . 8 (7): Р141. дои : 10.1186/gb-2007-8-7-r141 . ПМК 2323230 . ПМИД 17634151 .
- ↑ Перейти обратно: Перейти обратно: а б Урбини, Л.; Синаймери, Б.; Матиас, К.; Саго, МФ (2018). «Исследование устойчивости метода экономного согласования в кофилогении хозяин-симбионт» (PDF) . Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 16 (3): 738–748. дои : 10.1109/TCBB.2018.2838667 . ПМИД 29993554 . S2CID 51613681 .
- ^ Гурецкий, П.; Эйленштейн, О.; Тюрин, Дж. (2013). «Сверка неукорененных деревьев: единый подход». Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 10 (2): 522–536. дои : 10.1109/TCBB.2013.22 . ПМИД 23929875 . S2CID 13416908 .
- ↑ Перейти обратно: Перейти обратно: а б с Штольцер, М.; Лай, Х.; Сюй, М.; Сатай, Д.; Верно, Б.; Дюран, Д. (2012). «Вывод о дублировании, потерях, переносах и неполной сортировке линий с использованием небинарных деревьев видов» . Биоинформатика . 28 (18): и409–и415. doi : 10.1093/биоинформатика/bts386 . ПМЦ 3436813 . ПМИД 22962460 .
- ^ Лафонд М., Нутахи Э. и Эль-Мабрук Н. (2016) Эффективное разрешение небинарного дерева генов с взвешенной стоимостью согласования. 27-й ежегодный симпозиум по комбинаторному сопоставлению с образцом (CPM 2016) 14:1--14:12
- ^ Конт, Н.; Морель, Б.; Хасич, Д.; Геген, Л.; Буссау, Б.; Добин, В.; Пенель, С.; Скорнавакка, К.; Гуи, М.; Стаматакис, А.; Танньер, Э.; Парсонс, ДП (2020). «Treerecs: интегрированный филогенетический инструмент, от последовательностей до согласований» . Биоинформатика . 36 (18): 4822–4824. doi : 10.1093/биоинформатика/btaa615 . ПМИД 33085745 .
- ↑ Перейти обратно: Перейти обратно: а б с Чжэн, Ю; У, Таоян; Чжан, Лусинь (2012). «Согласование деревьев генов и видов с помощью политомии». arXiv : 1201.3995 [ q-bio.PE ].
- ↑ Перейти обратно: Перейти обратно: а б Корди, М.; Бансал, М.С. (2017). «О сложности согласования потерь дублирования-переноса с небинарными генными деревьями». Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 14 (3): 587–599. дои : 10.1109/TCBB.2015.2511761 . ПМИД 28055898 . S2CID 4458502 .
- ^ Лай, Хан; Штольцер, Морин; Дюран, Дэнни (2017). «Быстрая эвристика для разрешения слабо поддерживаемых ветвей с использованием дублирования, переносов и потерь». Сравнительная геномика . Конспекты лекций по информатике. Том. 10562. стр. 298–320. дои : 10.1007/978-3-319-67979-2_16 . ISBN 978-3-319-67978-5 .
- ^ Жакокс, Э.; Веллер, М.; Танньер, Э.; Скорнавакка, К. (2017). «Разрешение и согласование небинарных генных деревьев с переносами, дупликациями и потерями» . Биоинформатика . 33 (7): 980–987. doi : 10.1093/биоинформатика/btw778 . ПМИД 28073758 .
- ^ Корди, М.; Бансал, М.С. (2019). «Точные алгоритмы согласования потерь дублирования-переноса с небинарными генными деревьями» . Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 16 (4): 1077–1090. дои : 10.1109/TCBB.2017.2710342 . ПМИД 28622673 . S2CID 54606569 .
- ^ Бансал, Миссисипи; Ву, ЮК; Альм, Э.Дж.; Келлис, М. (2015). «Улучшенная коррекция ошибок дерева генов при наличии горизонтального переноса генов» . Биоинформатика . 31 (8): 1211–1218. doi : 10.1093/биоинформатика/btu806 . ПМЦ 4393519 . ПМИД 25481006 .
- ^ Лартильо, Н.; Филипп, Х. (2004). «Модель байесовской смеси для неоднородностей между сайтами в процессе замены аминокислот» . Молекулярная биология и эволюция . 21 (6): 1095–1109. дои : 10.1093/molbev/msh112 . ПМИД 15014145 .
- ↑ Перейти обратно: Перейти обратно: а б Буссау, Б.; Сёллоси, Г.Дж.; Дюре, Л.; Гуи, М.; Танньер, Э.; Добин, В. (2013). «Геномная кооценка видов и генных деревьев» . Геномные исследования . 23 (2): 323–330. дои : 10.1101/гр.141978.112 . ПМК 3561873 . ПМИД 23132911 .
- ↑ Перейти обратно: Перейти обратно: а б Акерборг, О.; Сеннблад, Б.; Арвестад, Л.; Лагергрен, Дж. (2009). «Одновременная реконструкция байесовского генного дерева и анализ согласования» . Труды Национальной академии наук Соединенных Штатов Америки . 106 (14): 5714–5719. Бибкод : 2009PNAS..106.5714A . дои : 10.1073/pnas.0806251106 . ПМК 2667006 . ПМИД 19299507 .
- ^ Нгуен, TH; Ранвез, В.; Пуанте, С.; Шифолло, AM; Дойон, JP; Берри, В. (2013). «Примирение и локальная перестройка генного дерева могут принести взаимную выгоду» . Алгоритмы молекулярной биологии . 8 (1): 12. дои : 10.1186/1748-7188-8-12 . ПМЦ 3871789 . ПМИД 23566548 .
- ^ Корди, Мисах; Бансал, Мукул С. (2020). « Решение дерева : быстрое исправление ошибок микробных генных деревьев». Алгоритмы вычислительной биологии . Конспекты лекций по информатике. Том. 12099. стр. 125–139. дои : 10.1007/978-3-030-42266-0_10 . ISBN 978-3-030-42265-3 . S2CID 210171366 .
- ^ Сьёстранд, Дж.; Тофиг, А.; Добин, В.; Арвестад, Л.; Сеннблад, Б.; Лагергрен, Дж. (2014). «Байесовский метод анализа латерального переноса генов» . Систематическая биология . 63 (3): 409–420. дои : 10.1093/sysbio/syu007 . ПМИД 24562812 .
- ^ Варнау, Тэнди (2018). «Строительство супердерева: возможности и проблемы». arXiv : 1805.03530 [ q-bio.PE ].
- ^ Легрид, Б.; Моллой, ЕК; Варнов, Т.; Рох, С. (2021). «Статистическая оценка полиномиального времени деревьев видов при дупликации и потере генов». Журнал вычислительной биологии . 28 (5): 452–468. дои : 10.1089/cmb.2020.0424 . ПМИД 33325781 .
- ^ Моллой, ЕК; Варнов, Т. (2020). «FastMulRFS: Быстрая и точная оценка дерева видов с использованием общих моделей дупликации и потери генов» . Биоинформатика . 36 (Приложение_1): i57–i65. doi : 10.1093/биоинформатика/btaa444 . ПМЦ 7355287 . ПМИД 32657396 .
- ^ Чаудхари, Р.; Берли, Дж. Г.; Фернандес-Бака, Д. (2013). «Вывод деревьев видов из неконгруэнтных многокопийных генных деревьев с использованием расстояния Робинсона-Фоулдса» . Алгоритмы молекулярной биологии . 8 (1): 28. дои : 10.1186/1748-7188-8-28 . ПМЦ 3874668 . ПМИД 24180377 .
- ^ Ма Б, Ли М и Чжан Л (2000) От генных деревьев к деревьям видов. SIAM J. Comput., май, 729–752 24
- ^ Гиго, Родерик; Мучник, Илья; Смит, Темпл Ф. (1 октября 1996 г.). «Реконструкция древней молекулярной филогении» . Молекулярная филогенетика и эволюция . 6 (2): 189–213. Бибкод : 1996МОЛПЭ...6..189Г . дои : 10.1006/mpev.1996.0071 . ISSN 1055-7903 . ПМИД 8899723 .
- ^ Пейдж, Родерик DM (1 января 2000 г.). «Извлечение деревьев видов из сложных генных деревьев: согласованные деревья и филогения позвоночных» . Молекулярная филогенетика и эволюция . 14 (1): 89–106. Бибкод : 2000МолПЭ..14...89П . дои : 10.1006/mpev.1999.0676 . ISSN 1055-7903 . ПМИД 10631044 . Проверено 17 декабря 2022 г.
- ^ Бансал, Миссисипи; Шамир, Р. (2011). «Заметки о решимости проблемы дупликации генов с фиксированными параметрами». Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 8 (3): 848–850. дои : 10.1109/TCBB.2010.74 . ПМИД 20733245 . S2CID 7086924 .
- ↑ Перейти обратно: Перейти обратно: а б Мэддисон, WP; Ноулз, LL (2006). «Вывод о филогении, несмотря на неполную сортировку линий» . Систематическая биология . 55 (1): 21–30. дои : 10.1080/10635150500354928 . ПМИД 16507521 . S2CID 13453831 .
- ^ Чанг, туалет; Берли, Дж.Дж.; Фернандес-Бака, DF; Эйленштейн, О. (2011). «Решение ILP для проблемы дупликации генов» . БМК Биоинформатика . 12 (Приложение 1): S14. дои : 10.1186/1471-2105-12-S1-S14 . ПМК 3044268 . ПМИД 21342543 .
- ^ Пейдж, Р.Д. (1998). «GeneTree: сравнение филогении генов и видов с использованием согласованных деревьев» . Биоинформатика . 14 (9): 819–820. дои : 10.1093/биоинформатика/14.9.819 . ПМИД 9918954 .
- ^ Вехе, А.; Бансал, Миссисипи; Берли, Дж. Г.; Эйленштейн, О. (2008). «DupTree: программа для крупномасштабного филогенетического анализа с использованием экономии генного дерева» . Биоинформатика . 24 (13): 1540–1541. doi : 10.1093/биоинформатика/btn230 . ПМИД 18474508 .
- ^ Чаудхари, Р.; Бансал, Миссисипи; Вехе, А.; Фернандес-Бака, Д.; Эйленштейн, О. (2010). «IGTP: пакет программного обеспечения для крупномасштабного анализа генного дерева» . БМК Биоинформатика . 11 : 574. дои : 10.1186/1471-2105-11-574 . ПМК 3002902 . ПМИД 21092314 .
- ^ Улла, И.; Парвиайнен, П.; Лагергрен, Дж. (2015). «Вывод дерева видов с использованием модели смеси» . Молекулярная биология и эволюция . 32 (9): 2469–2482. дои : 10.1093/molbev/msv115 . ПМИД 25963975 .
- ^ Бордевич В. и Семпл С. (2005) О вычислительной сложности сокращения корневого поддерева и расстояния пересадки. Анналы Комбинаториса 8: 409–423.
- ↑ Перейти обратно: Перейти обратно: а б Хасич, Д.; Танньер, Э. (2019). «Согласование дерева видов генного дерева с преобразованием генов». Журнал математической биологии . 78 (6): 1981–2014. arXiv : 1703.08950 . дои : 10.1007/s00285-019-01331-w . ПМИД 30767052 . S2CID 8476555 .
- ^ Эбби, СС; Танньер, Э.; Гуи, М.; Добин, В. (2010). «Обнаружение латерального переноса генов путем статистической сверки филогенетических лесов» . БМК Биоинформатика . 11 : 324. дои : 10.1186/1471-2105-11-324 . ПМЦ 2905365 . ПМИД 20550700 .
- ^ Хейн Дж., Цзян Т., Ван Л., Чжан К. (1996) О сложности сравнения эволюционных деревьев. Дискретная прикладная математика 71:153--169
- ^ Родригес, Эстела М.; Саго, Мари-Франс; Вакабаяси, Ёсико (апрель 2007 г.). «Задача леса максимального согласия: алгоритмы аппроксимации и вычислительные эксперименты». Теоретическая информатика . 374 (1–3): 91–110. дои : 10.1016/j.tcs.2006.12.011 . S2CID 16479722 .
- ^ Корди М. (2019) Вывод об эволюции семейства микробных генов с использованием согласования дублирования, переноса и потерь: алгоритмы и сложность. Докторские диссертации. 2101.
- ^ Урбини Л. (2017) Модели и алгоритмы для изучения общей эволюционной истории хозяев и симбионтов. Докторская диссертация, Лионский университет
- ^ Марин, Дж.; Ачаз, Г.; Кромбах, А.; Ламберт, А. (2020). «Геномный взгляд на диверсификацию». Журнал эволюционной биологии . 33 (10): 1387–1404. дои : 10.1111/jeb.13677 . ПМИД 32654283 . S2CID 91543153 .
- ^ Сёллёси, Г.Дж.; Танньер, Э.; Добин, В.; Буссау, Б. (2015). «Связь генных деревьев с деревьями видов» . Систематическая биология . 64 (1): е42-62. дои : 10.1093/sysbio/syu048 . ПМЦ 4265139 . ПМИД 25070970 .
- ^ Раннала, Б.; Ян, З. (2003). «Байесовская оценка времени дивергенции видов и размеров предковой популяции с использованием последовательностей ДНК из нескольких локусов» . Генетика . 164 (4): 1645–1656. дои : 10.1093/генетика/164.4.1645 . ПМЦ 1462670 . ПМИД 12930768 .
- ^ Дегнан, Дж. Х.; Солтер, Луизиана (2005). «Распределение генного дерева в процессе слияния». Эволюция; Международный журнал органической эволюции . 59 (1): 24–37. дои : 10.1111/j.0014-3820.2005.tb00891.x . ПМИД 15792224 .
- ^ Лю, Л.; Перл, ДК (2007). «Деревья видов из генных деревьев: реконструкция байесовских апостериорных распределений филогении видов с использованием предполагаемых распределений генных деревьев» . Систематическая биология . 56 (3): 504–514. дои : 10.1080/10635150701429982 . ПМИД 17562474 .
- ^ Раннала Б., Эдвардс С., Личе А. и Ян З. (2020) Филогенетика в геномную эру, 3.3:1--3.3:21
- ^ Борк, Д.; Ченг, Р.; Ван, Дж.; Сун, Дж.; Либескинд-Хадас, Р. (2017). «О вычислительной сложности задачи согласования максимальной экономии в модели слияния дублирования-потерь» . Алгоритмы молекулярной биологии . 12 :6. дои : 10.1186/s13015-017-0098-8 . ПМК 5349084 . ПМИД 28316640 .
- ^ Чан, Ю.Б.; Ранвез, В.; Скорнавакка, К. (2017). «Вывод о неполной сортировке родословной, дублировании, переносе и потерях с помощью сверок» (PDF) . Журнал теоретической биологии . 432 : 1–13. Бибкод : 2017JThBi.432....1C . дои : 10.1016/j.jtbi.2017.08.008 . ПМИД 28801222 . S2CID 7854827 .
- ^ Дю П., Огилви Х.А. и Наклех Л. (2019) Объединение дупликации, потери и слияния генов в филогенетических сетях. Биоинформатические исследования и приложения. ISBRA 2019. Конспекты лекций по информатике, том 11490. Springer, Cham.
- ^ Расмуссен, доктор медицинских наук; Келлис, М. (2012). «Единое моделирование дупликации, потери и слияния генов с использованием дерева локусов» . Геномные исследования . 22 (4): 755–765. дои : 10.1101/гр.123901.111 . ПМК 3317157 . ПМИД 22271778 .
- ^ Ву, ЮК; Расмуссен, доктор медицинских наук; Бансал, Миссисипи; Келлис, М. (2014). «Наиболее экономное согласование при наличии дупликации, потери и глубокого слияния генов с использованием меченых сливающихся деревьев» . Геномные исследования . 24 (3): 475–486. дои : 10.1101/гр.161968.113 . ПМЦ 3941112 . ПМИД 24310000 .
- ^ Эта таблица предназначена служить иллюстрацией к разделу о трехуровневой сверке Menet et al. (2022) Филогенетическое примирение. PLoS Comput Biol 18 (11): e1010621. doi:10.1371/journal.pcbi.1010621 и его можно прочитать вместе с ним. Цвета соответствуют разным уровням (похожие, но разные цвета, если на одном уровне несколько деревьев). Легенда дает пример всех этих цветов и кодов формы. Множественные линии генов могут претерпевать совместные события, такие как дупликация всего генома (А) или сегментные события (Б), некоторые события могут быть более вероятными, чем другие, например, специфические горизонтальные переносы с путем переносов или гибридизация (С). Кофилогенетические паттерны можно сравнить, чтобы увидеть, например, не является ли общий паттерн хозяина и симбионта просто общим паттерном симбионта и географии (D). Персонажи могут развиваться в соответствии с согласованной филогенией, например, синтенией генов (E), или два уровня могут быть согласованы с ограничением верхнего уровня (F). Передачи могут быть верхнезависимыми, что более вероятно между двумя промежуточными сущностями, принадлежащими одному верхнему (G). Три уровня могут быть согласованы вместе, последовательно: промежуточный и верхний, прежде чем добавлять нижний, или пытаться найти совместный наиболее экономичный сценарий для двух согласований (H). Эти многоуровневые модели также можно использовать для реконструкции промежуточной филогении (I).
- ^ Тайс, КР; Дейли, Нью-Мексико; Классен, Дж.Л.; Брукер, Р.М.; Бейнс, Дж. Ф.; Бош, TC; Крайан, Дж. Ф.; Гилберт, Сан-Франциско; Спокойной ночи, СиДжей; Ллойд, Э.А.; Сапп, Дж.; Ванденкокорнхейсе, П.; Зильбер-Розенберг, И.; Розенберг, Э.; Борденштейн, СР (2016). «Правильное понимание концепции гологенома: эко-эволюционная основа хозяев и их микробиомов» . mSystems . 1 (2). дои : 10.1128/mSystems.00028-16 . ПМК 5069740 . ПМИД 27822520 .
- ^ Маргулис, Л.С.; Фестер, Р. (1991). Симбиоз как источник эволюционных инноваций: видообразование и морфогенез . Кембридж, Массачусетс: MIT Press. ISBN 9780262132695 .
- ^ Борденштейн, СР; Тайс, КР (2015). «Биология хозяина в свете микробиома: десять принципов холобионтов и гологеномов» . ПЛОС Биология . 13 (8): e1002226. дои : 10.1371/journal.pbio.1002226 . ПМЦ 4540581 . ПМИД 26284777 .
- ^ Розенберг, Э.; Корен, О.; Решеф, Л.; Эфрони, Р.; Зильбер-Розенберг, И. (2007). «Роль микроорганизмов в здоровье, болезнях и эволюции кораллов». Обзоры природы. Микробиология . 5 (5): 355–362. дои : 10.1038/nrmicro1635 . ПМИД 17384666 . S2CID 2967190 .
- ^ Зильбер-Розенберг, И.; Розенберг, Э. (2008). «Роль микроорганизмов в эволюции животных и растений: гологеномная теория эволюции» . Обзоры микробиологии FEMS . 32 (5): 723–735. дои : 10.1111/j.1574-6976.2008.00123.x . ПМИД 18549407 .
- ^ Моран, Северная Каролина; Маккатчеон, JP; Накабачи, А. (2008). «Геномика и эволюция наследственных бактериальных симбионтов». Ежегодный обзор генетики . 42 : 165–190. дои : 10.1146/annurev.genet.41.110306.130119 . ПМИД 18983256 .
- ^ Лопес-Мадригал, С.; Гил, Р. (2017). «И ты, Брут? Даже внутриклеточные мутуалистические симбионты не избегают горизонтального переноса генов» . Гены . 8 (10): 247. doi : 10.3390/genes8100247 . ПМК 5664097 . ПМИД 28961177 .
- ^ Пенц, Т.; Шмитц-Эссер, С.; Келли, SE; Касс, Б.Н.; Мюллер, А.; Войк, Т.; Малфатти, ЮАР; Хантер, Миссисипи; Хорн, М. (2012). «Сравнительная геномика предполагает независимое происхождение цитоплазматической несовместимости у Cardinium Hertigii» . ПЛОС Генетика . 8 (10): е1003012. дои : 10.1371/journal.pgen.1003012 . ПМЦ 3486910 . ПМИД 23133394 .
- ^ Никох, Н.; Хосокава, Т.; Морияма, М.; Осима, К.; Хаттори, М.; Фукацу, Т. (2014). «Эволюционное происхождение пищевого мутуализма насекомых и вольбахии» . Труды Национальной академии наук Соединенных Штатов Америки . 111 (28): 10257–10262. Бибкод : 2014PNAS..11110257N . дои : 10.1073/pnas.1409284111 . ПМК 4104916 . ПМИД 24982177 .
- ^ Мансано-Марин, А.; Кер д'Асье, А.; Кламенс, Алабама; Орвайн, К.; Круо, К.; Барбе, В.; Жуслен, Э. (2020). «Последовательный горизонтальный перенос генов биосинтеза витаминов позволяет создавать новые пищевые симбионты в дисимбиотических системах тлей» . Журнал ISME . 14 (1): 259–273. Бибкод : 2020ISMEJ..14..259M . дои : 10.1038/s41396-019-0533-6 . ПМК 6908640 . ПМИД 31624345 .
- ^ Накабачи, А.; Уеока, Р.; Осима, К.; Тета, Р.; Мангони, А.; Гурги, М.; Олдхэм, Нью-Джерси; Ван Эхтен-Декерт, Г.; Окамура, К.; Ямамото, К.; Иноуэ, Х.; Окума, М.; Хонго, Ю.; Миягишима, Ю.Ю.; Хаттори, М.; Пиль, Дж.; Фукацу, Т. (2013). «Защитный бактериомный симбионт с резко уменьшенным геномом» . Современная биология . 23 (15): 1478–1484. Бибкод : 2013CBio...23.1478N . дои : 10.1016/j.cub.2013.06.027 . ПМИД 23850282 . S2CID 1637956 .
- ^ Пинто-Карбо, М.; Зибер, С.; Дессейн, С.; Уикер, Т.; Верстраете, Б.; Гадеманн, К.; Эберл, Л.; Карлье, А. (2016). «Доказательства горизонтального переноса генов между симбионтами облигатных клубеньков» . Журнал ISME . 10 (9): 2092–2105. Бибкод : 2016ISMEJ..10.2092P . дои : 10.1038/ismej.2016.27 . ПМЦ 4989318 . ПМИД 26978165 .
- ^ Чон, Х.; Ариф, Б.; Каэтано-Аноллес, Ж.; Ким, К.М.; Насир, А. (2019). «Горизонтальный перенос генов в микроорганизмах, связанных с человеком, выведенный путем филогенетической реконструкции и согласования» . Научные отчеты . 9 (1): 5953. Бибкод : 2019NatSR...9.5953J . дои : 10.1038/s41598-019-42227-5 . ПМК 6459891 . ПМИД 30976019 .
- ^ Виджаявардена, Британская Колумбия; Минчелла, диджей; Девуди, Дж. А. (2013). «Хозяева, паразиты и горизонтальный перенос генов». Тенденции в паразитологии . 29 (7): 329–338. дои : 10.1016/j.pt.2013.05.001 . ПМИД 23759418 .
- ^ Мудли, Ю.; Линц, Б.; Бонд, РП; Ньювудт, М.; Судьял, Х.; Шлебуш, CM; Бернхофт, С.; Хейл, Дж.; Суербаум, С.; Мугиша, Л.; Ван дер Мерве, Юго-Запад; Ахтман, М. (2012). «Возраст ассоциации Helicobacter pylori и человека» . ПЛОС Патогены . 8 (5): e1002693. дои : 10.1371/journal.ppat.1002693 . ПМЦ 3349757 . ПМИД 22589724 .
- ^ Ахтман, М. (2016). «Сколько лет бактериальным патогенам?» . Слушания. Биологические науки . 283 (1836). дои : 10.1098/rspb.2016.0990 . ПМК 5013766 . ПМИД 27534956 .
- ^ Фу, Иран; Пистолоцци, Марко; Ян, Сяофэн; Линь, Чжанлинь (2020). «Комплексная классификация коронавирусов и предполагаемая передача между хостами». bioRxiv 10.1101/2020.08.11.232520 .
- ^ Да Силва, SG; Тегерани, Джей-Джей (2016). «Сравнительный филогенетический анализ раскрывает древние корни индоевропейских сказок» . Королевское общество открытой науки . 3 (1): 150645. Бибкод : 2016RSOS....350645D . дои : 10.1098/rsos.150645 . ПМЦ 4736946 . ПМИД 26909191 .
- ^ Росс, РМ; Гринхилл, SJ; Аткинсон, QD (2013). «Структура населения и культурная география сказки в Европе» . Слушания. Биологические науки . 280 (1756). дои : 10.1098/rspb.2012.3065 . ПМЦ 3574383 . ПМИД 23390109 .
- ^ Бортолини, Э.; Пагани, Л.; Крема, скорая помощь; Сарно, С.; Барбьери, К.; Боаттини, А.; Саццини, М.; Да Силва, SG; Мартини, Дж.; Метспалу, М.; Петтенер, Д.; Луизелли, Д.; Тегерани, Джей-Джей (2017). «Выявление закономерностей распространения сказок на основе геномных данных» . Труды Национальной академии наук Соединенных Штатов Америки . 114 (34): 9140–9145. Бибкод : 2017PNAS..114.9140B . дои : 10.1073/pnas.1614395114 . ПМК 5576778 . ПМИД 28784786 .
- ^ Гиго, Р.; Мучник И.; Смит, Т.Ф. (1996). «Реконструкция древней молекулярной филогении» . Молекулярная филогенетика и эволюция . 6 (2): 189–213. Бибкод : 1996МОЛПЭ...6..189Г . дои : 10.1006/mpev.1996.0071 . ПМИД 8899723 .
- ^ Пейдж, РД; Коттон, Дж.А. (2002). «Филогеномика позвоночных: согласованные деревья и дупликации генов». Тихоокеанский симпозиум по биокомпьютингу. Тихоокеанский симпозиум по биокомпьютингу : 536–547. дои : 10.1142/9789812799623_0050 . ISBN 978-981-02-4777-5 . ПМИД 11928506 . S2CID 5638166 .
- ^ Берли, Дж. Г.; Бансал, Миссисипи; Вехе, А.; Эйленштейн, О. (2009). «Обнаружение крупномасштабных событий дупликации генов с помощью согласованных деревьев: значение для выявления событий древней полиплоидии у растений». Журнал вычислительной биологии . 16 (8): 1071–1083. дои : 10.1089/cmb.2009.0139 . ПМИД 19689214 .
- ^ Бансал, Миссисипи; Эйленштейн, О. (2008). «Возвращение к проблеме множественной дупликации генов» . Биоинформатика . 24 (13): i132-8. doi : 10.1093/биоинформатика/btn150 . ПМЦ 2718628 . ПМИД 18586705 .
- ^ Майкл Р. Феллоуз, Майкл Т. Халлет и Ульрика Стеге. 1998. О проблеме множественной дупликации генов. В материалах 9-го Международного симпозиума по алгоритмам и вычислениям (ISAAC '98). Шпрингер-Верлаг, Берлин, Гейдельберг, 347–356.
- ^ Донди, Р.; Лафонд, М.; Скорнавакка, К. (2019). «Согласование нескольких деревьев генов посредством сегментных дупликаций и потерь» . Алгоритмы молекулярной биологии . 14 :7. дои : 10.1186/s13015-019-0139-6 . ПМК 6425616 . ПМИД 30930955 .
- ^ Бансал, Миссисипи; Банай, Г.; Гогартен, JP; Шамир, Р. (2011). «Обнаружение путей горизонтального переноса генов». Журнал вычислительной биологии . 18 (9): 1087–1114. дои : 10.1089/cmb.2011.0066 . hdl : 1721.1/69855 . ПМИД 21899418 .
- ^ Клуб, Л.; Госслен, С.; Фуллмер, М.; Граф, Дж.; Гогартен, JP; Бансал, Миссисипи (2021). «Систематическое обнаружение крупномасштабного мультигенного горизонтального переноса у прокариот» . Молекулярная биология и эволюция . 38 (6): 2639–2659. дои : 10.1093/molbev/msab043 . ПМЦ 8136488 . PMID 33565580 .
- ^ Ю, Ю.; Ристич, Н.; Нахле, Л. (2013). «Быстрые алгоритмы и эвристики филогеномики в условиях ILS и гибридизации» . БМК Биоинформатика . 14 (Дополнение 15): S6. дои : 10.1186/1471-2105-14-S15-S6 . ПМЦ 3852049 . ПМИД 24564257 .
- ^ Ю, Ю.; Барнетт, РМ; Нахле, Л. (2013). «Экономный вывод о гибридизации при неполной сортировке линий» . Систематическая биология . 62 (5): 738–751. дои : 10.1093/sysbio/syt037 . ПМЦ 3739885 . ПМИД 23736104 .
- ^ Нибердинг, КМ; Дюретт-Дессет, MC; Вандерпоортен, А.; Казанова, Дж. К.; Рибас, А.; Деффонтен, В.; Фелиу, К.; Моран, С.; Либуа, Р.; Мишо, младший (2008). «География и биогеография хозяина имеют значение для понимания филогеографии паразита». Молекулярная филогенетика и эволюция . 47 (2): 538–554. Бибкод : 2008МОЛПЭ..47..538Н . дои : 10.1016/j.ympev.2008.01.028 . hdl : 2268/9909 . ПМИД 18346916 .
- ^ Мартинес-Акино, А.; Чеккарелли, Ф.С.; Эгиарте, Луизиана; Васкес-Домингес, Э.; Де Леон, GP (2014). «Отражает ли историческая биогеография и история эволюции дигенейских видов Margotrema SPP. В центральной Мексике историю их пресноводных рыб-хозяев (Goodeinae)?» . ПЛОС ОДИН . 9 (7): е101700. Бибкод : 2014PLoSO...9j1700M . дои : 10.1371/journal.pone.0101700 . ПМК 4084993 . ПМИД 24999998 .
- ^ Векштейн, JD (2004). «Биогеография объясняет кофилогенетические закономерности тукановых вшей» . Систематическая биология . 53 (1): 154–164. дои : 10.1080/10635150490265085 . ПМИД 14965910 .
- ^ Фонтан, ЭД; Паули, Дж. Н.; Мендоса, JE; Карлсон, Дж.; Пири, МЗ (2017). «Кофилогенетика и биогеография показывают совместную эволюцию отношений между ленивцами и их водорослями-симбионтами» . Молекулярная филогенетика и эволюция . 110 : 73–80. Бибкод : 2017МолПЭ.110...73Ф . дои : 10.1016/j.ympev.2017.03.003 . ПМИД 28288943 .
- ^ Груссен, М.; Мазель, Ф.; Сандерс, Дж.Г.; Смилли, CS; Лавернь, С.; Туиллер, В.; Альм, Э.Дж. (2017). «Раскрытие процессов формирования микробиома кишечника млекопитающих в течение эволюционного времени» . Природные коммуникации . 8 : 14319. Бибкод : 2017NatCo...814319G . дои : 10.1038/ncomms14319 . ПМЦ 5331214 . ПМИД 28230052 .
- ^ Берри, В.; Шевене, Ф.; Дойон, JP; Жуслен, Э. (2018). «Метод согласования с учетом географии для исследования моделей диверсификации во взаимодействиях хозяина и паразита». Ресурсы молекулярной экологии . 18 (5): 1173–1184. дои : 10.1111/1755-0998.12897 . ПМИД 29697894 . S2CID 25758856 .
- ^ Ву, ЮК; Расмуссен, доктор медицинских наук; Келлис, М. (2012). «Эволюция на субгенном уровне: перестановки доменов в филогении дрозофилы» . Молекулярная биология и эволюция . 29 (2): 689–705. дои : 10.1093/molbev/msr222 . ПМЦ 3258039 . ПМИД 21900599 .
- ↑ Перейти обратно: Перейти обратно: а б Штольцер, М.; Зиверт, К.; Лай, Х.; Сюй, М.; Дюран, Д. (2015). «Вывод событий в многодоменных семьях с филогенетическим согласованием» . БМК Биоинформатика . 16 (Дополнение 14): S8. дои : 10.1186/1471-2105-16-S14-S8 . ПМК 4610023 . ПМИД 26451642 .
- ↑ Перейти обратно: Перейти обратно: а б Ли, Л.; Бансал, М.С. (2019). «Интегрированная структура согласования эволюции на уровне доменов, генов и видов» . Транзакции IEEE/ACM по вычислительной биологии и биоинформатике . 16 (1): 63–76. дои : 10.1109/TCBB.2018.2846253 . ПМИД 29994126 . S2CID 51614715 .
- ^ Ли, Лей; Бансал, Мукул С. (2018). «Решение целочисленного линейного программирования для проблемы согласования домена, гена и вида». Материалы Международной конференции ACM 2018 по биоинформатике, вычислительной биологии и медицинской информатике . стр. 386–397. дои : 10.1145/3233547.3233603 . ISBN 9781450357944 . S2CID 49426403 .
- ^ Ли, Лей; Бансал, Мукул С. (2019). «Одновременное согласование нескольких доменов, нескольких генов в рамках модели согласования доменов, генов и видов». Биоинформатические исследования и приложения . Конспекты лекций по информатике. Том. 11490. стр. 73–86. дои : 10.1007/978-3-030-20242-2_7 . ISBN 978-3-030-20241-5 . S2CID 85507475 .
- ^ Кунду, С.; Бансал, М.С. (2019). «SaGePhy: улучшенная система филогенетического моделирования эволюции генов и субгенов». Биоинформатика . 35 (18): 3496–3498. doi : 10.1093/биоинформатика/btz081 . ПМИД 30715213 .
- ^ Мухаммад, Сайед Аун; Сеннблад, Бенгт; Лагергрен, Йенс (2018). «Одновременная реконструкция эволюции генов и доменов с учетом древа видов» . дои : 10.1101/336453 . S2CID 90627553 .
{{cite journal}}: Для цитирования журнала требуется|journal=( помощь ) - ^ Байи-Беше, М.; Мартинс-Симойнс, П.; Сёллоси, Г.Дж.; Миалдеа, Г.; Саго, МФ; Шарла, С. (2017). «Как долго вольбахия остается на борту?» . Молекулярная биология и эволюция . 34 (5): 1183–1193. дои : 10.1093/molbev/msx073 . ПМИД 28201740 .
- ^ «Дива» . 26 апреля 2013 года . Проверено 21 ноября 2022 г.
- ^ «Лагранж» . Проверено 21 ноября 2022 г.
- ^ «БиоГеоМЕДВЕДИ» . Проверено 21 ноября 2022 г.
- ^ «Джейн» . Проверено 21 ноября 2022 г.
- ^ «ЭМПресс» . Проверено 21 ноября 2022 г.
- ^ «Эвкалипт» . 28 июля 2014 года . Проверено 21 ноября 2022 г.
- ^ «Капибара» . Проверено 21 ноября 2022 г.
- ^ «Коала» . 28 июля 2014 года . Проверено 21 ноября 2022 г.
- ^ «РЕЙНДЖЕР-ДТЛ» . 31 мая 2018 года . Проверено 21 ноября 2022 г.
- ^ «Нотунг» . Проверено 21 ноября 2022 г.
- ^ «Маугли» . Проверено 21 ноября 2022 г.
- ^ «Сильвкс» . Проверено 21 ноября 2022 г.
- ^ «АнГСТ» . Проверено 21 ноября 2022 г.
- ^ Код доступен на Github.
- ^ «экцеТЕРА» . Гитхаб . 21 сентября 2021 г. Проверено 21 ноября 2022 г.
- ^ «ЭЛЬ» . Гитхаб . 4 октября 2022 г. Проверено 21 ноября 2022 г.
- ^ «Treerecs – быстрое, универсальное и удобное для пользователя филогенетическое согласование» . Проверено 20 декабря 2022 г.
- ^ «ДжинРакс» . Гитхаб . 8 сентября 2022 г. Проверено 20 декабря 2022 г.
- ^ «PHYLDOG: совместная реконструкция видов и филогении генов» . pbil.univ-lyon1.fr (на французском языке) . Проверено 20 декабря 2022 г.
- ^ «Лейпцигский университет – Факультет математики и информатики – Группа роевого интеллекта и сложных систем» . pacosy.informatik.uni-leipzig.de . Проверено 20 декабря 2022 г.
- ^ «Лейпцигский университет – факультет математики и информатики – кафедра роевого интеллекта и сложных систем» . pacosy.informatik.uni-leipzig.de . Проверено 20 декабря 2022 г.
- ^ Арвестад, Ларс (6 сентября 2021 г.). «О JPrIME» . Гитхаб . Проверено 20 декабря 2022 г.
- ^ Орландо, Эчеваррия (31 мая 2018 г.). «SEADOG | Научно-исследовательская лаборатория вычислительной биологии» . compbio.engr.uconn.edu . Проверено 20 декабря 2022 г.
- ^ «iGTP Домой» . Лаборатория вычислительной биологии . Проверено 20 декабря 2022 г.
- ^ Мукул, Бансал (26 ноября 2018 г.). «TreeSolve | Научно-исследовательская лаборатория вычислительной биологии» . compbio.engr.uconn.edu .
- ^ «ТриФикс» . www.cs.hmc.edu . Проверено 20 декабря 2022 г.
- ^ «ТриФикс-ДТЛ» . www.cs.hmc.edu . Проверено 20 декабря 2022 г.
- ^ Мукул, Бансал (26 ноября 2018 г.). «ARTra: Аддитивный и заменяющий переносной вывод | Исследовательская лаборатория вычислительной биологии» . compbio.engr.uconn.edu . Проверено 20 декабря 2022 г.
- ^ «DLCoal: Моделирование дупликации, потерь и слияния генов» . compbio.mit.edu . Проверено 20 декабря 2022 г.
- ^ «Дом · simonpenel/третьего рода Wiki» . Гитхаб . Проверено 20 декабря 2022 г.
Внешние ссылки
[ редактировать ] СМИ, связанные с филогенетическим согласованием, на Викискладе?
СМИ, связанные с филогенетическим согласованием, на Викискладе?