N-ацетилглюкозамин-6-фосфатдеацетилаза
| N-ацетилглюкозамин-6-фосфатдеацетилаза в Mycobacterium smegmatis | |||
|---|---|---|---|
 | |||
| Идентификаторы | |||
| ЕС №. | 3.5.1.25 | ||
| CAS №. | 9027-50-3 | ||
| Базы данных | |||
| Intenz | Intenz View | ||
| Бренда | Бренда вход | ||
| Расширение | Вид Nicezyme | ||
| Кегг | Кегг вход | ||
| Метатический | Метаболический путь | ||
| Напрямую | профиль | ||
| PDB Структуры | RCSB PDB PDBE PDBSUM | ||
| Джин Онтология | Друг / Quickgo | ||
| |||
В фермете N-ацетилглюкозамин - N-ацетилглюкозамин-6-фосфатдеацетилаза ( EC 3.5.1.25 ), также известный как GLCNAC-6-фосфатдеацетилаза или NAGA, является ферментом , который катализирует деацетилирование 6 (Glcnac-6-6-ацетилгглакозамин-6 (Glcnac-6-ацетилглюкозамин-6 (Glcnac-6-ацетилгглакозамин-6 (Glcnac-6 P) глюкозамин-6-фосфату (GLCN-6-P):
- H 2 O + N-ацетил-D-глюкозамин 6-фосфат ацетат + D-глюкозамин 6-фосфат [ 1 ]
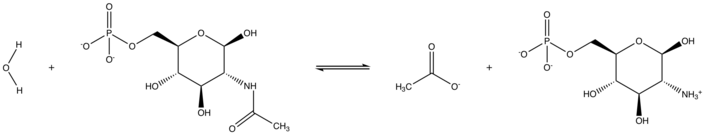
NagA Reaction
GLCNAC-6-фосфат деацетилаза кодируется геном NAGA. [ 2 ]
Этот фермент принадлежит к суперсемейству амидогидролазы . [ 3 ] Амидогидролазы являются типом гидролазы , которая действует на амидные связи. Все члены семейства амидогидролазы используют структуру Tim Barrel , а подавляющее большинство членов являются металлоферментами . [ 4 ] Семейство ферментов важно в метаболизме аминокислот и нуклеотидов, а также в биодеградации сельскохозяйственных и промышленных соединений. Нага участвует в метаболизме амино-сахара, особенно в биосинтезе амино-сахар-нуклеотидов. [ 5 ]
Структура
[ редактировать ]Нага - это гомодимерный фермент с двумя доменами в каждом димере структуры. [ 6 ] Каждый домен I содержит (β/α) 8 -структурную складку ствола, также известную как ствол TIM, и содержит активный сайт фермента. Каждый активный сайт состоит из каталитического сайта фермента и металлического связывающего сайта, которые участвуют в распознавании субстрата и металла, соответственно. Домен I также формирует димерный интерфейс с доменом I соседней субъединицы. [ 6 ] Меньший второй домен ферментов NAGA содержит β-болот, который потенциально действует для стабилизации фермента. [ 6 ] В то время как все члены суперсемейства амидогидролазы используют структурную складку TIM-Barrel, у Naga в Escherichia coli (Ecnaga) есть псевдо-Tim Barrel, который прилагает воронкоподобный каталитический сайт фермента. [ 7 ] Структура димера NAGA считается решающей для активности и термостабильности фермента. [ 8 ]

Метал-связывающий сайт
[ редактировать ]Ферменты амидогидролазы могут связывать один, два или три атома металла в активном сайте. Эти металлы могут включать Zn 2+ , Co 2+ , Fe 2+ , CD 2+ и другие. [ 1 ] Ecnaga содержит мононуклеарную металлическую связывание с Zn 2+ ион; [ 3 ] Кроме того, ECNAGA показывает фосфатный ион, связанный на металлическом сайте. [ 7 ] В отличие от Ecnaga, Naga of Mycobacterium smegmatis (MSNAGA) и Bacillus subtilis (BSNAGA) имеют бинуклеарные металлические сайты. MSNAGA имеет два иона с двухвалентными металлами, расположенные в каждом активном участке, которые необходимы для эффективного катализа и структурной стабильности. [ 6 ] В то время как большинство других видов бактерий используют Zn в качестве их металлического ко-фактора, Bsnaga использует железо в качестве преобладающего металла в металлическом сайте. [ 9 ]
КАТАЛИТИЧЕСКАЯ СВЕДЕНИЯ МЕСТ
[ редактировать ]Большинство остатков активного участка Ecnaga и Bsnaga сохраняются и имеют сходные структурные позиции. Примечательным различием между микобактериальными ферментами NAGA и ферментами NAGA из других видов бактерий является присутствие цистеина в положении 131. Другие виды бактерий имеют остаток лизина в этом положении. Этот цистеин расположен в гибкой петле, которая предотвращает связывание физиологического субстрата. [ 6 ]
Механизм
[ редактировать ]Каталитический механизм для ферментов NAGA, предложенных, использует нуклеофильную атаку с помощью координированной металлом молекула воды или гидроксид-иона. Механизм проходит через строго консервативный остаток аспарагиновой кислоты активной сайты (ASP-273), который первоначально действует в качестве основания для активации гидролитической молекулы воды, чтобы атаковать карбонильную группу субстрата. [ 3 ] Затем ASP-273 действует как кислота для протонирования группы покинув амина. Один предложенный механизм с использованием BSNAGA и его двух соавторов железа в сайте связывания металлов демонстрирует нуклеофильную атаку гидроксидом Fe-мостикой Fe, а затем стабилизация карбонильного кислорода одним из двух атомов Fe. [ 9 ]
Биологическая функция
[ редактировать ]
Нага расположена в цитоплазме клетки. N-ацетилглюкозамин (GLCNAC) входит в клетку как часть расщепления клеточной стенки. GLCNAC, моносахарид и производное глюкозы, является частью биополимера в бактериальной клеточной стенке. Этот биополимер образует многослойную структуру, называемую пептидогликаном (PG). GlcNac затем превращается в GLCNAC-6-P ферментом. [ 10 ] Этот субстрат затем деацетилизируется в ацетат и GLCN-6-P от NAGA. [ 11 ] NAGA важна для производства GLCN-6-P, который затем используется в двух основных путях: путь рециркуляции PG и путь гликолиза .
PG Recycling Pathway
[ редактировать ]В пути рециркуляции PG, как только GLCNAC-6-P метаболизируется с помощью NAGA, его продукт, GLCN-6-P, затем может быть преобразован в GLCN-1-P ферментом GLMM с последующим реакцией и реакцией с UTP с помощью GLMU для формирования udp-glcnac. [ 10 ] [ 11 ] UDP-GLCNAC является конечным продуктом этого пути, который затем используется для изготовления гликозаминогликанов , протеогликанов и гликолипидов , которые необходимы для пополнения PG для клеточной стенки. [ 12 ] Рециркуляция PG необходима для бактериальных клеток, чтобы обеспечить рост бактерий и предотвратить лизис клеток . [ 13 ]
Путь гликолиза
[ редактировать ]Вместо того, чтобы входить в путь рециркуляции PG, GLCN-6-P может быть преобразован в фруктозо-6-фосфат с помощью NAGB. Эта реакция обратима ферментом GLM, [ 10 ] [ 11 ] амидотрансфераза. [ 13 ] Производимый фруктозо-6-фосфат затем входит в путь гликолиза. Гликолиз катализирует выработку пирувата , что приводит к циклу лимонной кислоты и позволяет выработать аминокислоты. [14] GlcN-6-P and fructose-6-phosphate act as allosteric regulators of NagA, inhibiting further deacetylation of GlcNAc-6-P.[15]
Disease relevance
[edit]NagA is a potential drug target of Mycobacterium tuberculosis (Mtb). Eliminating NagA produces high levels of the allosteric activator GlcNAc-6-P,[2] which prevents the production of GlcN-6-P in order to proceed with the PG recycling pathway. NagA is, therefore, at a crucial metabolic chokepoint in Mtb,[16] representing the key enzymatic step in the generation of essential amino-sugar precursors. These precursors are required for Mtb cell wall biosynthesis and influence the PG recycling pathway. Additionally, the presence of cysteine in MSNagA's active site may represent a unique exploitative target in Mtb therapeutics.[6]
Structural studies
[edit]As of early 2019, 11 structures have been solved for this class of enzymes, with PDB accession codes 1O12, 1UN7, 1YMY, 1YRR, 2P50, 2P53, 6FV3, 6FV4, 3EGJ, 3IV8, and 2VHL.
Nomenclature
[edit]The systematic name of this enzyme class is N-acetyl-D-glucosamine-6-phosphate amidohydrolase. Other names in common use include acetylglucosamine phosphate deacetylase, acetylaminodeoxyglucosephosphate acetylhydrolase, and 2-acetamido-2-deoxy-D-glucose-6-phosphate amidohydrolase.[15]
References
[edit]- ^ Jump up to: a b "nagA - N-acetylglucosamine-6-phosphate deacetylase - Escherichia coli (strain K12) - nagA gene & protein". www.uniprot.org. Retrieved 2019-03-14.
- ^ Jump up to: a b Alvarez-Añorve LI, Bustos-Jaimes I, Calcagno ML, Plumbridge J (October 2009). "Allosteric regulation of glucosamine-6-phosphate deaminase (NagB) and growth of Escherichia coli on glucosamine". Journal of Bacteriology. 191 (20): 6401–7. doi:10.1128/JB.00633-09. PMC 2753035. PMID 19700525.
- ^ Jump up to: a b c Hall RS, Xiang DF, Xu C, Raushel FM (July 2007). "N-Acetyl-D-glucosamine-6-phosphate deacetylase: substrate activation via a single divalent metal ion". Biochemistry. 46 (27): 7942–52. doi:10.1021/bi700543x. PMC 2533526. PMID 17567047.
- ^ Liu A, Huo L (2014-08-15), John Wiley & Sons Ltd (ed.), "Amidohydrolase Superfamily", eLS, John Wiley & Sons, Ltd, doi:10.1002/9780470015902.a0020546.pub2, ISBN 9780470015902
- ^ Yadav V, Panilaitis B, Shi H, Numuta K, Lee K, Kaplan DL (2011-06-02). "N-acetylglucosamine 6-phosphate deacetylase (nagA) is required for N-acetyl glucosamine assimilation in Gluconacetobacter xylinus". PLOS ONE. 6 (6): e18099. Bibcode:2011PLoSO...618099Y. doi:10.1371/journal.pone.0018099. PMC 3107205. PMID 21655093.
- ^ Jump up to: a b c d e f Ahangar MS, Furze CM, Guy CS, Cooper C, Maskew KS, Graham B, Cameron AD, Fullam E (June 2018). "Mycobacterium tuberculosis N-acetylglucosamine-6-phosphate deacetylase (NagA)". The Journal of Biological Chemistry. 293 (25): 9770–9783. doi:10.1074/jbc.RA118.002597. PMC 6016474. PMID 29728457.
- ^ Jump up to: a b Ferreira FM, Mendoza-Hernandez G, Castañeda-Bueno M, Aparicio R, Fischer H, Calcagno ML, Oliva G (June 2006). "Structural analysis of N-acetylglucosamine-6-phosphate deacetylase apoenzyme from Escherichia coli". Journal of Molecular Biology. 359 (2): 308–21. doi:10.1016/j.jmb.2006.03.024. PMID 16630633.
- ^ Mine S, Kado Y, Watanabe M, Fukuda Y, Abe Y, Ueda T, Kawarabayasi Y, Inoue T, Ishikawa K (November 2014). "The structure of hyperthermophilic β-N-acetylglucosaminidase reveals a novel dimer architecture associated with the active site". The FEBS Journal. 281 (22): 5092–103. doi:10.1111/febs.13049. PMID 25227262. S2CID 21178562.
- ^ Jump up to: a b Vincent F, Yates D, Garman E, Davies GJ, Brannigan JA (January 2004). "The three-dimensional structure of the N-acetylglucosamine-6-phosphate deacetylase, NagA, from Bacillus subtilis: a member of the urease superfamily". The Journal of Biological Chemistry. 279 (4): 2809–16. doi:10.1074/jbc.M310165200. PMID 14557261.
- ^ Jump up to: a b c Park JT, Uehara T (June 2008). "How bacteria consume their own exoskeletons (turnover and recycling of cell wall peptidoglycan)". Microbiology and Molecular Biology Reviews. 72 (2): 211–27, table of contents. doi:10.1128/MMBR.00027-07. PMC 2415748. PMID 18535144.
- ^ Jump up to: a b c Plumbridge J (September 2009). "An alternative route for recycling of N-acetylglucosamine from peptidoglycan involves the N-acetylglucosamine phosphotransferase system in Escherichia coli". Journal of Bacteriology. 191 (18): 5641–7. doi:10.1128/JB.00448-09. PMC 2737974. PMID 19617367.
- ^ Milewski S, Gabriel I, Olchowy J (January 2006). "Enzymes of UDP-GlcNAc biosynthesis in yeast". Yeast. 23 (1): 1–14. doi:10.1002/yea.1337. PMID 16408321. S2CID 39940329.
- ^ Jump up to: a b Dhar S, Kumari H, Balasubramanian D, Mathee K (January 2018). "Cell-wall recycling and synthesis in Escherichia coli and Pseudomonas aeruginosa - their role in the development of resistance". Journal of Medical Microbiology. 67 (1): 1–21. doi:10.1099/jmm.0.000636. PMID 29185941.
- ^ Stryer L, Tymoczko JL, Berg JM (2002). "The Citric Acid Cycle". Biochemistry. 5th Edition.
- ^ Jump up to: a b White RJ, Pasternak CA (октябрь 1967 г.). «Очистка и свойства N-ацетилглюкозамин 6-фосфатдеацетилазы из Escherichia coli» . Биохимический журнал . 105 (1): 121–5. doi : 10.1042/bj1050121 . PMC 1198282 . PMID 4861885 .
- ^ «Молекулярные понимания фермента Naga могут помочь бороться с туберкулезом» . News-medical.net . 2018-07-12 . Получено 2019-03-11 .
Дальнейшее чтение
[ редактировать ]- Yamano N, Matsushita Y, Kamada Y, Fujishima S, Arita M (август 1996 г.). «Очистка и характеристика N-ацетилглюкозамин 6-фосфатной деацетилазы с активностью против N-ацетилглюкозамина из вибрио-холеры не O1» . Биоссака, биотехнология и биохимия . 60 (8): 1320–3. doi : 10.1271/bbb.60.1320 . PMID 8987551 .

