Ансамбль геномов
| Содержание | |
|---|---|
| Описание | Интегративный ресурс для данных в масштабе генома видов, не являющихся позвоночными. |
| Типы данных захвачен | Геномная база данных |
| Организмы | кастрюля |
| Контакт | |
| Исследовательский центр | Европейский институт биоинформатики |
| Первичное цитирование | Керси и др. (2012), [1] Хау и др. (2020) [2] |
| Дата выпуска | 2009 |
| Доступ | |
| Веб-сайт | https://ensemblgenomes.org/ |
| URL-адрес загрузки | ftp://ftp.ensemblgenomes.org/pub/current |
| веб-службы URL-адрес | https://rest.ensembl.org/ |
| Публичный к SQL доступ | [адрес электронной почты защищен] : 4157 |
| Разнообразный | |
| Лицензия | Апач 2.0 |
| Выпуск данных частота | 4 раза в год |
| Версия | Выпуск 52 (декабрь 2021 г.) |
Ensembl Genomes — это научный проект по предоставлению данных в масштабе генома видов, не являющихся позвоночными. [1] [2]
Проект осуществляется Европейским институтом биоинформатики и был запущен в 2009 году с использованием технологии Ensembl . [3] Основная цель базы данных Ensembl Genomes – дополнить основную базу данных Ensembl Genomes.базу данных путем создания пяти дополнительных веб-страниц, включающих данные о геномах бактерий , грибов , многоклеточных беспозвоночных , растений и простейших . [4] Для каждого из доменов Ensembl доступны инструменты для манипулирования, анализа и визуализации данных генома. Большая часть данных Ensembl Genomes хранится в реляционных базах данных MySQL, и доступ к ним можно получить через интерфейс Ensembl REST, Perl API, Biomart или онлайн. [5]
Ensembl Genomes — открытый проект, и большая часть кода, инструментов и данных доступна общественности. [6] Программное обеспечение Ensembl и Ensembl Genomes использует лицензию Apache 2.0. [7] лицензия.
Отображение геномных данных
[ редактировать ]
Ключевой особенностью Ensembl Genomes является графический интерфейс, который позволяет пользователям прокручивать геном и наблюдать относительное расположение таких функций, как концептуальные аннотации (например, гены , локусы SNP ), образцы последовательностей (например, повторы) и экспериментальные данные (например, последовательности). и внешние признаки последовательности, отображенные на геноме ). [1] Графические изображения доступны для различных уровней разрешения: от всего кариотипа до последовательности одного экзона . Информация о геноме распределена по четырем вкладкам: страница вида, вкладка «Местоположение», вкладка « Ген » и вкладка « Расшифровка », каждая из которых предоставляет информацию в более высоком разрешении.
При поиске определенного вида с помощью Ensembl Genomes происходит перенаправление на страницу вида. Часто предоставляется краткое описание вида, а также ссылки на дополнительную информацию и статистику о геноме , графический интерфейс и некоторые доступные инструменты.
Кариотип . доступен для некоторых видов в Ensembl Genomes [8] Если кариотип доступен, ссылка на него будет в разделе «Сборка генов» на странице вида. В качестве альтернативы, если пользователи находятся на вкладке «Местоположение», они также могут просмотреть кариотип, выбрав «Весь геном» в меню слева. Пользователи могут щелкнуть по местоположению кариотипа, чтобы увеличить масштаб одной конкретной хромосомы или участка генома. [8] Откроется вкладка «Местоположение».
На вкладке «Местоположение» пользователи могут просматривать гены , вариации, сохранение последовательностей и другие типы аннотаций по геному . [9] «Подробно о регионе» легко настраивается и масштабируется, и пользователи могут выбирать, что они хотят видеть, нажав кнопку «Настроить эту страницу» в нижней части меню слева. Добавляя и удаляя треки, пользователи смогут выбирать тип данных, которые они хотят включать в дисплеи. [9] На вкладке «Местоположение» можно легко добавлять или удалять данные из следующих категорий: « Последовательность и сборка », « Гены и транскрипты », « Выравнивание мРНК и белков », «Другие ДНК выравнивания », « зародышевой линии Вариации », « Сравнительная геномика » и др. [9] Пользователи также могут изменить параметры отображения, например ширину. [9] Дополнительная опция позволяет пользователям сбросить конфигурацию обратно к настройкам по умолчанию. [9]
Более конкретную информацию о выбранном гене можно найти на вкладке «Ген». Пользователи могут попасть на эту страницу, выполнив поиск нужного гена в строке поиска и щелкнув идентификатор гена или щелкнув один из генов, показанных на вкладке «Местоположение». Вкладка «Ген» содержит информацию, специфичную для гена, такую как структура гена, количество транскриптов , положение на хромосоме и информацию о гомологии в виде генных деревьев. [10] Доступ к этой информации можно получить через меню слева.
Вкладка «Расшифровка» также появится, когда пользователь решит просмотреть ген. Вкладка «Транскрипт» содержит большую часть той же информации, что и вкладка «Ген», однако она ориентирована только на один транскрипт. [10]
Инструменты
[ редактировать ]Добавление пользовательских треков в Ensembl Genomes
[ редактировать ]Ensembl Genomes позволяет сравнивать и визуализировать пользовательские данные при просмотре кариотипов и генов. Большинство представлений Ensembl Genomes включают кнопку «Добавить ваши данные» или «Управление вашими данными», которая позволяет пользователю загружать новые треки, содержащие чтения или последовательности, в Ensembl Genomes или изменять данные, которые были загружены ранее. [11] Загруженные данные можно визуализировать в виде областей или всего кариотипа. Загруженные данные можно локализовать с помощью координат хромосом или координат клона BAC. [12] Для загрузки файла данных на любую страницу Ensembl Genomes можно использовать следующие методы: [13]
- Файлы размером менее 5 МБ можно загружать непосредственно с любого компьютера или из веб-сайта (URL) на серверы Ensembl.
- Файлы большего размера можно загружать только из веб-сайтов (URL).
- Файлы BAM можно загружать только с использованием подхода на основе URL-адреса. Индексный файл (.bam.bai) должен находиться на том же веб-сервере.
- Источник распределенной системы аннотаций можно прикрепить из веб-сайтов.
Следующие типы файлов поддерживаются Ensembl Genomes: [14]

- КРОВАТЬ
- КроватьГраф
- Общий
- ГФФ/ГТФ
- ПСЛ
- ПАРИК
- БАМ
- Большая кровать
- БольшойПарик
- ВЦФ
Данные временно загружаются на серверы. Зарегистрированные пользователи могут войти в систему и сохранить свои данные для дальнейшего использования. Можно делиться и получать доступ к загруженным данным, используя назначенный URL-адрес. [15] Пользователям также разрешено удалять свои собственные треки из Ensembl Genomes.
БиоМарт
[ редактировать ]BioMart — это бесплатная поисковая система, встроенная в Ensembl и Ensembl Genomes (за исключением Ensembl Bacteria) с целью анализа и извлечения геномных данных из баз данных Ensembl в табличных форматах, таких как HTML, TSV, CSV или XLS. [16] В выпуске 45 (2019 г.) Ensembl Genomes на BioMarts доступны следующие данные:
- Ensembl Protists BioMart: включает 33 вида и вариации Phytophthora infestans и Phaeodactylum tricornutum. [17]
- Ensembl Fungi BioMart: включает 56 видов и вариаций Fusarium graminearum , Fusarium oxysporum , Schizosaccharomyces pombe , Puccinia graminis , Verticillium dahliae , Zymoseptoria tritici и Saccharomyces cerevisiae. [18]
- Ensembl Metazoa BioMart: включает 78 видов и вариаций Aedes aegypti , Anopheles gambiae и Ixodes scapularis. [19]
- Ансамбль растений: включает 67 видов и вариаций Arabidopsis thaliana , Brachypodium distachyon , Hordeum vulgare , Oryza glaberrima , Oryza glumipatula , Oryza sativa indica , Oryza sativa japonica , Solanum lycopersicum , Sorghum bicolor , Triticum aestivum , Vitis vinifera и Zea may. с [20]
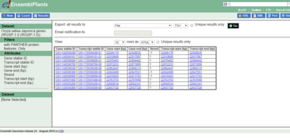
Целью BioMarts в Ensembl Genomes является предоставление пользователю возможности искать и загружать таблицы, содержащие все гены одного вида, гены в определенной области хромосомы или гены в одной области хромосомы, связанной с доменом InterPro. [21] BioMarts также включает в себя фильтры для уточнения извлекаемых данных, а атрибуты (идентификатор варианта, имя хромосомы, идентификатор ансамбля, местоположение и т. д.), которые появятся в конечном файле таблицы, могут быть выбраны пользователем.
Доступ к BioMart можно получить онлайн в каждом соответствующем домене Ensembl Genomes, либо исходный код можно установить в среде UNIX из git -репозитория BioMart. [22]
ВЗРЫВ
[ редактировать ]Предусмотрен интерфейс BLAST , позволяющий пользователям искать последовательности ДНК или белков в геномах Ensembl. Доступ к нему можно получить через заголовок, расположенный поверх всех страниц Ensembl Genome, под названием BLAST. Поиск BLAST можно настроить для поиска отдельных видов или коллекций видов (максимум 25). Существует таксономический браузер, позволяющий выбирать таксономически родственные виды. [23]
Последовательный поиск
[ редактировать ]Ensembl Genomes предоставляет второй инструмент поиска последовательностей, который использует алгоритм на основе Exonerate, предоставленный Европейским архивом нуклеотидов . [23] Доступ к этому инструменту можно получить через заголовок, расположенный в верхней части всех страниц Ensembl Genome, под названием Sequence Search. Затем пользователи могут выбрать, хотят ли они, чтобы Exonerate проводил поиск по всем видам в разделе Ensembl Genomes или по всем видам в Ensembl Genomes. Они также могут выбрать «Максимальное значение E», которое ограничит результаты, отображаемые только теми, у которых значения E ниже максимального. Наконец, пользователи могут выбрать альтернативный режим поиска, выбрав «Использовать объединенный запрос».
Предиктор эффекта варианта
[ редактировать ]Предиктор эффекта варианта — один из наиболее часто используемых инструментов в Ensembl и Ensembl Genomes. Это позволяет исследовать и анализировать, какое влияние варианты (SNP, CNV, индели или структурные вариации) оказывают на конкретный ген, последовательность, белок, транскрипт или фактор транскрипции. [24] Чтобы использовать VEP, пользователи должны ввести местоположение своих вариантов и вариации нуклеотидов, чтобы получить следующие результаты: [25]
- Гены и транскрипты, на которые влияет этот вариант
- Расположение вариантов
- Как вариант влияет на синтез белка (например, на образование стоп-кодона)
- Сравнение с другими базами данных для поиска равных известных вариантов.
Пользователи могут получить доступ к VEP двумя способами. Первая форма – онлайн. На этой странице пользователь генерирует входные данные, выбирая следующие параметры: [26]
- Виды, подлежащие сравнению. Базой данных по умолчанию для сравнения является Ensembl Transcripts, но для некоторых видов можно выбрать другие источники.
- Имя загруженных данных (это необязательно, но так будет проще идентифицировать данные, если было выполнено много заданий VEP)
- Выбор формата ввода данных. Если выбран неправильный формат файла, VEP при запуске выдаст ошибку.
- Поля для загрузки данных. Пользователи могут загружать данные со своих компьютеров, из URL-адреса или напрямую копируя их содержимое в текстовое поле.
Загрузка данных в VEP поддерживает нотации VCF, Pileup, HGVS и формат по умолчанию. [27] Формат по умолчанию — файл, разделенный пробелами, который содержит данные в столбцах. Первые пять столбцов указывают хромосому, начальное местоположение, конечное местоположение, аллель (пара аллелей, разделенных знаком «/», с эталонным аллелем первым) и цепь (+ для прямого или – для обратного). [28] Шестой столбец представляет собой идентификатор варианта и является необязательным. Если оставить это поле пустым, VEP присвоит идентификатор в выходном файле.
VEP также предоставляет пользователям дополнительные параметры идентификатора, дополнительные возможности для дополнения вывода и фильтрации. [29] Параметры фильтрации позволяют использовать такие функции, как удаление известных вариантов из результатов, возврат вариантов только в экзонах и ограничение результатов конкретными последствиями вариантов. [30]
Пользователи VEP также имеют возможность просматривать и управлять всеми заданиями, связанными с их сеансом, просматривая вкладку «Последние заявки». На этой вкладке пользователи могут просматривать статус своего поиска (успех, в очереди, выполняется или не удалось), а также сохранять, удалять или повторно отправлять задания. [31]
Второй вариант использования VEP — загрузка исходного кода для его использования в средах UNIX. [32] Все функции онлайн-версии и скриптовой версии одинаковы. VEP также можно использовать с онлайн-инстансами, такими как Galaxy.
По завершении задания VEP выходные данные представляют собой табличный файл, содержащий следующие столбцы: [33]
- Загруженный вариант – как хромосома_start_аллели.
- Местоположение — в стандартном формате координат (chr:start или chr:start-end).
- Аллель — вариант аллели, используемый для расчета последствий.
- Ген — стабильный идентификатор ансамбля затронутого гена.
- Функция — стабильный идентификатор функции Ensembl.
- Тип объекта — тип объекта. В настоящее время один из Transcript, RegulatoryFeature, MotifFeature.
- Последствие - тип последствия этого варианта.
- Положение в кДНК - относительное положение пары оснований в последовательности кДНК.
- Позиция в CDS - относительное положение пары оснований в кодирующей последовательности.
- Позиция в белке – относительное положение аминокислоты в белке.
- Аминокислотная замена - дается только в том случае, если вариация влияет на последовательность, кодирующую белок.
- Изменение кодона - альтернативные кодоны с вариантной основой в верхнем регистре.
- Совмещенный вариант – известный идентификатор существующего варианта.
- Дополнительно — этот столбец содержит дополнительную информацию в виде пар ключ=значение, разделенных знаком «;». Отображает дополнительные идентификаторы.

Другие распространенные форматы вывода для VEP включают форматы JSON и VDF. [34]
Программный доступ к данным
[ редактировать ]Интерфейс Ensembl Genomes [REST] обеспечивает доступ к данным с использованием вашего любимого языка программирования.
Вы также можете получить доступ к данным с помощью Perl API и Biomart.
Текущие виды
[ редактировать ]Ensembl Genomes не пытается включить все возможные геномы, а на сайте представлены те геномы, которые считаются важными с научной точки зрения. [35] На каждом участке присутствует следующее количество видов:
- Бактериальный отдел Ensembl теперь содержит все бактериальные геномы, которые были полностью секвенированы, аннотированы и отправлены в Международное сотрудничество по базам данных нуклеотидных последовательностей (Европейский архив нуклеотидов, GenBank и База данных ДНК Японии). [35] Текущий набор данных содержит 31 332 генома. [36]
- Ансамбль грибов содержит 1502 генома. [37]
- Ensembl Metazoa содержит 333 генома видов беспозвоночных . [38] Основной сайт Ensembl содержит 324 генома видов позвоночных . [39]
- Ансамбль растений содержит 153 генома. [40]
- Ансамбль протистов содержит 237 геномов. [41]
Сотрудничество
[ редактировать ]Ensembl Genomes постоянно расширяет данные аннотаций благодаря сотрудничеству с другими организациями, участвующими в проектах и исследованиях аннотаций геномов. Следующие организации являются сотрудниками Ensembl Genomes: [42]
- AllBio
- Ячмень
- Culicoides sonorensis
- Грамен
- ИНФРАКРАСНЫЙ
- Микром
- PomBase
- ФитоПат
- ТРАНСПЛАНТ
- Геномика Triticeae для устойчивого сельского хозяйства
- ВекторБаза
- Улучшение генома пшеничной ржавчины
- Червячная база
- WormBase ПараСайт
См. также
[ редактировать ]- Вместе
- Европейский институт биоинформатики
- ВЗРЫВ
- БЛАТ
- Червячная база
- ВекторБаза
- Список секвенированных геномов растений
- Список секвенированных бактериальных геномов
- Список секвенированных геномов протистов
- Список секвенированных геномов грибов
- Список секвенированных геномов архей
- Анализ последовательности
- Структурная вариация
Внешние ссылки
[ редактировать ]- Ансамбль геномов
- Документация по геномам ансамблей
- Вместе
- Домашняя страница ЭБИ
- Юридические уведомления Ensembl Genomes
- Домашняя страница Биомарта
Ссылки
[ редактировать ]- ^ Перейти обратно: а б с Керси, Пи Джей; Стейнс, DM; Лоусон, Д.; Кулеша, Е.; Дервент, П.; Хамфри, Джей Си; Хьюз, летнее время; Кинан, С.; Керхорну, А.; Косельни, Г.; Лэнгридж, Н.; Макдауэлл, доктор медицины; Меги, К.; Махешвари, У.; Нун, М.; Паулини, М.; Педро, Х.; Тонева И.; Уилсон, Д.; Йейтс, А.; Бирни, Э. (2011). «Ансамбль геномов: интегративный ресурс для данных в масштабе генома видов, не являющихся позвоночными» . Исследования нуклеиновых кислот . 40 (Проблема с базой данных): D91–D97. дои : 10.1093/nar/gkr895 . ПМК 3245118 . ПМИД 22067447 .
- ^ Перейти обратно: а б Хоу К.Л., Контрерас-Морейра Б., Де Силва Н., Маслен Г., Аканни В., Аллен Дж., Альварес-Харрета Дж., Барба М., Болсер Д.М., Камбел Л., Карбахо М., Чакиачвили М., Кристенсен М., Камминс С., Кьюзик А., Дэвис П., Фексова С., Галл А., Джордж Н., Гил Л., Гупта П., Хаммонд-Козак К.Е., Хаскелл Е., Хант С., Джайсвал П., Яначек С., Керси П.Дж., Лэнгридж Н., Махешвари Ю., Морел Т., Макдауэлл М.Д., Мур Б., Муффато М., Наамати Дж., Найтани С., Олсон А., Папатеодору И., Патрисио М., Паулини М., Педро Х., Перри Э., Прис Дж., Розелло М., Рассел М., Ситник В., Стейнс Д.М., Стайн Дж., Телло- Руис М.К., Треванион С.Дж., Урбан М., Вэй С., Уэр Д., Уильямс Дж., Йейтс А.Д., Фличек П. (январь 2020 г.). «Ensembl Genomes 2020 — возможность проведения геномных исследований беспозвоночных» . Исследования нуклеиновых кислот . 48(Д1) (Д1): Д689–Д695. дои : 10.1093/nar/gkz890 . ПМК 6943047 . ПМИД 31598706 .
- ^ Хаббард, TJP; Акен, БЛ ; Эйлинг, С.; Баллестер, Б.; Бил, К.; Брагин, Е.; Брент, С.; Чен, Ю.; Клэпхэм, П.; Кларк, Л.; Коутс, Г.; Фэрли, С.; Фицджеральд, С.; Фернандес-Банет, Дж.; Гордон, Л.; Граф, С.; Хайдер, С.; Хаммонд, М.; Холланд, Р.; Хау, К.; Дженкинсон, А.; Джонсон, Н.; Кахари, А.; Киф, Д.; Кинан, С.; Кинселла, Р.; Кокочински, Ф.; Кулеша, Е.; Лоусон, Д.; Лонгден, И. (2009). «Ансамбль 2009» . Исследования нуклеиновых кислот . 37 (Проблема с базой данных): D690–D697. дои : 10.1093/нар/gkn828 . ПМЦ 2686571 . ПМИД 19033362 .
- ^ «Об ансамблевых геномах» . Ансамбль геномов . Ансамбль . Проверено 2 сентября 2014 г.
- ^ «Ансамбль геномов MySQL» . ensemblgenomes.org . Ансамбль геномов . Проверено 11 сентября 2014 г.
- ^ Кинселла, Рода Дж.; Кяхари, Андреас; Сайед, Хайдер; Самора, Хорхе; Проктор, Гленн; Спудич, Джульетта; Алмейда-Кинг, Джефф; Стейнс, Дэниел; Дервент, Пол; Керурну, Арно; Керси, Пол; Фличек, Пол (2011). «Ensembl BioMarts: центр поиска данных в таксономическом пространстве» . База данных . 2011 (2011): 2. doi : 10.1093/database/bar030 . ПМК 3170168 . ПМИД 21785142 .
- ^ «Лицензия на программное обеспечение» . Ансамбль . Проверено 9 июня 2020 г.
- ^ Перейти обратно: а б «Целый геном» . Ансамбль геномов . Проверено 7 сентября 2014 г.
- ^ Перейти обратно: а б с д и «Часто задаваемые вопросы» . Ансамбль геномов . Проверено 7 сентября 2014 г.
- ^ Перейти обратно: а б Спудич, Г; Фернандес-Суарес, XM; Бирни, Э. (2007). «Просмотр генома с помощью Ensembl: практический обзор» . Брифинги по функциональной геномике и протеомике . 6 (3): 202–19. дои : 10.1093/bfgp/elm025 . ПМИД 17967807 .
- ^ «Загрузка ваших данных в Ensembl» . Ансамбль геномов . Ансамбль геномов . Проверено 9 сентября 2014 г.
- ^ «Координаты расположения данных в Ensembl Genomes» . Ансамбль геномов . Ансамбль геномов . Проверено 9 сентября 2014 г.
- ^ «Методы загрузки данных» . Ансамбль растений . Ансамбль геномов . Проверено 9 сентября 2014 г.
- ^ «Поддерживаемые файлы данных» . Ансамбль растений . Ансамбль геномов . Проверено 9 сентября 2014 г.
- ^ «Сохранение и обмен данными в Ensembl Genomes» . Ансамбль растений . Ансамбль геномов.
- ^ «Интеллектуальный анализ данных в Ensembl с помощью Data Mining в Ensembl с BioMart» (PDF) . Ансамбль : 2. 2014 . Проверено 11 сентября 2014 г.
- ^ «Ансамбль Протистов» . Ансамбль Протистов . Ансамбль геномов . Проверено 1 октября 2019 г.
- ^ «Ансамбль грибов» . Ансамбль грибов . Ансамбль геномов . Проверено 1 октября 2019 г.
- ^ «Ансамбль Метазоа» . Ансамбль Метазоа . Ансамбль геномов . Проверено 1 октября 2019 г.
- ^ «Ансамбль растений» . Ансамбль растений . Ансамбль геномов . Проверено 1 октября 2019 г.
- ^ «Интеллектуальный анализ данных в Ensembl с помощью Data Mining в Ensembl с BioMart» (PDF) . Ансамбль : 3. 2014 . Проверено 11 сентября 2014 г.
- ^ «Руководство пользователя BioMart 0.9.0» (PDF) . Май 2014. с. 5 . Проверено 11 сентября 2014 г.
- ^ Перейти обратно: а б «Часто задаваемые вопросы» . Ансамбль геномов. Архивировано из оригинала 10 сентября 2014 года . Проверено 11 сентября 2014 г.
- ^ «Вариантный предсказатель эффекта» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «Обзор результатов предсказания эффекта варианта» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «Ввод данных в ВЭП» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «Форматы файлов, поддерживаемые VEP» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «Файл VEP по умолчанию» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «Опции VEP и дополнительные возможности» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «ВЭП-фильтрация» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «ВЭП вакансии» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «Загрузка сценария VEP» . ensembl.org . Ансамбль . Проверено 11 сентября 2014 г.
- ^ «Выход ВЭП» . ensembl.org . Ансамбль геномов . Проверено 11 сентября 2014 г.
- ^ «Форматы вывода VEP» . ensembl.org . Ансамбль геномов . Проверено 11 сентября 2014 г.
- ^ Перейти обратно: а б Керси, Пи Джей; Аллен, Дж. Э.; Кристенсен, М; Дэвис, П; Фалин, ЖЖ; Грабмюллер, К; Хьюз, Д.С.; Хамфри, Дж; Керхорну, А; Хобова Дж.; Лэнгридж, Н.; Макдауэлл, доктор медицины; Махешвари, У; Маслен, Г; Нун, М; Онг, СК; Паулини, М; Педро, Х; Тонева, Я; Тули, Массачусетс; Уолтс, Б; Уильямс, Дж; Уилсон, Д; Юенс-Кларк, К.; Монако, МК; Штейн, Дж; Вэй, Х; Уэр, Д; Болсер, Д.М.; и др. (2014). «Ensembl Genomes 2013: Расширение доступа к полногеномным данным» . Исследования нуклеиновых кислот . 42 (Проблема с базой данных): D546–52. дои : 10.1093/нар/gkt979 . ПМЦ 3965094 . ПМИД 24163254 .
- ^ «Список видов» . Ансамбль геномов . Проверено 1 июля 2024 г.
- ^ «Список видов» . Ансамбль геномов . Проверено 1 июля 2024 г.
- ^ «Список видов» . Ансамбль геномов . Проверено 1 июля 2024 г.
- ^ «Список видов» . Ансамбль геномов . Проверено 1 июля 2024 г.
- ^ «Список видов» . Ансамбль геномов . Проверено 1 июля 2024 г.
- ^ «Список видов» . Ансамбль геномов . Проверено 1 июля 2024 г.
- ^ «Соавторы — ансамбли геномов» . Ансамбль геномов . Ансамбль геномов . Проверено 3 сентября 2014 г.
