Сплайсосома
Сплисосома в представляет собой большой комплекс рибонуклеопротеина (RNP), который можно найти в основном ядре эукариотических клеток . Сплисосома собирается из небольших ядерных РНК ( SNRNA ) и многочисленных белков. Маленькая ядерная РНК (SNRNA) молекулы связываются со специфическими белками с образованием небольшого ядерного рибонуклеопротеинового комплекса (SNRNP, произносится «Snurps»), который, в свою очередь, сочетается с другими SNRNP с образованием большого комплекса рибонуклеопротеина, называемой сплайсосомой. Сплисосома удаляет интроны из транскрибированной пре-мРНК, типа первичной транскрипта . Этот процесс обычно называют сплайсингом . [ 1 ] Аналогией является редактор фильма, который выборочно вырезает нерелевантный или неверный материал (эквивалентный интронам ) из первоначального фильма и отправляет очищенную версию режиссеру для окончательного сокращения. [ Цитация необходима ]
Однако иногда РНК внутри интрона действует как рибозим, сплайсинг без использования сплисосомы или ферментов белка.

История
[ редактировать ]В 1977 году работа Sharp and Roberts Labs показала, что гены высших организмов «разделены» или присутствуют в нескольких отдельных сегментах вдоль молекулы ДНК. [ 2 ] [ 3 ] Кодирующие области гена разделяются некодирующей ДНК , которая не участвует в экспрессии белка. Структура расщепления гена была обнаружена, когда аденовирусные мРНК были гибридизованы для фрагментов расщепления эндонуклеазы однократной вирусной ДНК. [ 2 ] Было отмечено, что мРНК гибридов мРНК-ДНК содержали 5 ' и 3' хвостов негидрогенных областей. Когда использовались более крупные фрагменты вирусных ДНК, раздвоенные структуры петлевой ДНК наблюдались при гибридизации до вирусных мРНК. Было понято, что зацикленные области, интроны , вырезаются из мРНК -предшественников в резком процессе, названном «сплайсинг». Впоследствии было обнаружено, что структура сплит -гена является общей для большинства эукариотических генов. Филипп Шарп и Ричард Дж. Робертс были удостоены Нобелевской премии по физиологии или медицине 1993 года за открытие интронов и процесса сплайсинга. [ Цитация необходима ]
Композиция
[ редактировать ]Каждая сплайсосома состоит из пяти небольших ядерных РНК (SNRNA) и ряда связанных белковых факторов. Когда эти небольшие РНК объединяются с белковыми факторами, они производят РНК-белковые комплексы, называемые SNRNP ( S Mall N Uclear R Ibo n Ucleo proteins , произносится «Snurps»). SNRNAS, которые составляют основную сплайсосому, называются U1 , U2 , U4 , U5 и U6 , так называемые, потому что они богаты уридина и участвуют в нескольких взаимодействиях РНК-РНК и РНК-белок. [ 1 ]
Сборка сплайсосомы происходит на каждой пре-мРНК (также известной как гетерогенная ядерная РНК, HN-RNA) в каждом экзоне: интрон соединение. Интроны до мРНК содержит специфические элементы последовательности, которые распознаются и используются во время сборки сплайсосомы. К ним относятся 5 -'конечный сайт сплайсинга, последовательность точек ветвления, полипиримидиновый тракт и 3 -дюймовый сайт сплайсинга. Сплисосома катализирует удаление интронов и перевязку фланкирующих экзонов. [ Цитация необходима ]
Интроны обычно имеют нуклеотидную последовательность Gu на месте 5 'конечного сплайсинга и AG на 3 -дюймовом участке сплайсинга. 3 'сайт сплайсинга может быть дополнительно определен с помощью переменной длины полипиримидинов, называемой полипиримидиновым трактом (PPT), который служит двойной функции рекрутинговых факторов на 3' сайт сплайсинга и, возможно, рекрутирующие коэффициенты в последовательность ветвей (BPS) Полем BPS содержит консервативный аденозин, необходимый для первого этапа сплайсинга. [ Цитация необходима ]
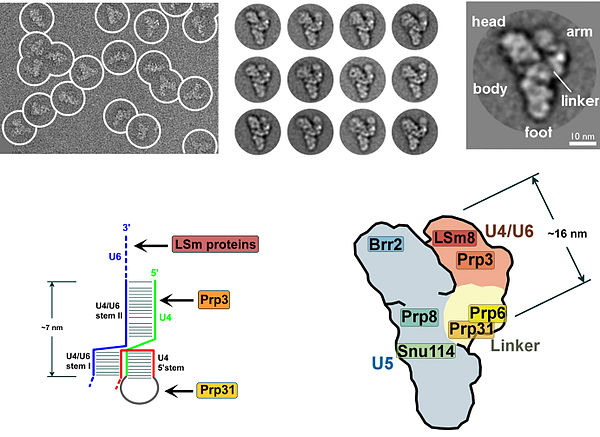
Многие белки демонстрируют цинк-связывающий мотив, который подчеркивает важность цинка в механизме сплайсинга. [ 4 ] [ 5 ] [ 6 ] Первая реконструкция молекулярного разрешения комплекса U4/U6.U5 Тройного малого ядерного рибонуклеопротеина (TRI-SNRNP) была зарегистрирована в 2016 году. [ 7 ]
Крио-Эм широко применяется Shi et al. чтобы выяснить почти/атомную структуру сплицеосомы у обоих дрожжей [ 9 ] и люди. [ 10 ] Молекулярная структура сплицеосомы в почти атомной разрешении демонстрирует компонент SPP42 U5 SNRNP образует центральный каркас и закрепляет каталитический центр у дрожжей. Атомная структура человеческой сплайсосомы иллюстрирует компонент II шага II, который использует расширенную структуру, готовую для выбора сайта 3'-сплит. Все пять металлов (назначенные как MG2+) в дрожжевом комплексе сохраняются в человеческом комплексе. [ Цитация необходима ]
Альтернативный сплайсинг
[ редактировать ]Альтернативный сплайсинг (повторная комбинация различных экзонов ) является основным источником генетического разнообразия у эукариот. Варианты сплайсинга использовались для учета относительно небольшого числа генов, кодирующих белок в геноме человека , в настоящее время оценивается в 20 000 человек. один конкретный Drosophila ген , DSCAM Предполагалось, что , альтернативно расплачивается на 38 000 различных мРНК , предполагая, что все его экзоны могут посыпать независимо друг от друга. [ 11 ]
Расположение сплайсинга
[ редактировать ]Первоначально было обнаружено, что факторы сплайсинга до мРНК сконцентрировались в ядерных телах, известных как ядерные крапки. [ 12 ] Первоначально было постулировано, что ядерные спекли являются либо местами, посвященными мРНК сплайсинга или хранилища факторов сплайсинга мРНК. В настоящее время понимается, что ядерные спекли помогают концентрировать факторы сплайсинга вблизи генов, которые физически расположены рядом с ними. Гены, расположенные дальше от краплей, все еще могут быть транскрибированы и расщеплены, но их скольжение менее эффективно по сравнению с тем, что ближе к спеклям. [ 13 ] Сплайсинг РНК является биохимической реакцией, и, как и все биохимические реакции, его скорость зависит от концентрации ферментов и субстратов. В этом случае ферменты являются сплайсосомами, а субстраты являются до-мРНК. Изменяя концентрацию сплицеосом и пре-мРНК на основе их близости к ядерным спеклям, клетки могут потенциально регулировать эффективность сплайсинга. [ 13 ]
Сборка
[ редактировать ]Модель для формирования активного сайта сплицеосом включает в себя упорядоченную, пошаговую сборку дискретных частиц SNRNP на подложке пре-мРНК. Первое распознавание пре-мРНК включает в себя связывание SNRNP U1 с 5-'конечным сайтом сплайсинга пре-мРНК и других не связанных с SNRNP факторов, чтобы сформировать комплекс обязательств или ранний (E) комплекс у млекопитающих. [ 14 ] [ 15 ] Комплекс обязательств является АТФ-независимым комплексом, который совершает пре-мРНК в путь сплайсинга. [ 16 ] U2 SNRNP набирается в область филиала посредством взаимодействия с комплексным компонентом E -комплекса U2AF (вспомогательный фактор U2 SNRNP) и, возможно, U1 SNRNP. В АТФ-зависимой реакции U2 SNRNP тесно связан с последовательности точек ветви Первая переэтерификация. [ 17 ]
Присутствие псевдоуридинового остатка в snRNA U2, почти противоположное сайту ветви, приводит к измененной конформации дуплекса РНК-РНК при связывании SNRNP U2. В частности, измененная структура дуплекса, индуцированная псевдоуридином, ставит 2 'OH выпивленного аденозина в благоприятное положение для первого этапа сплайсинга. [ 18 ] U4/U5/U6 TRI-SNRNP (см. Рисунок 1) рекрутируется в сборку сплайсосомы с образованием комплекса B, и после нескольких перестановки комплекс C активируется для катализа. [ 19 ] [ 20 ] Неясно, как TRI-SNRNP рекрутируется в комплекс A, но этот процесс может быть опосредован с помощью белковых взаимодействий и/или спаривания основания между SNRNA U2 и SNRNA U6. [ Цитация необходима ]
U5 SNRNP взаимодействует с последовательностями на 5 'и 3' сайтах сплайсинга через инвариантную петлю SnRNA U5 [ 21 ] и компоненты белка U5 взаимодействуют с областью 3 'сайта сплайсинга. [ 22 ]
После рекрутирования TRI-SNRNP несколько перестройков РНК-РНК предшествуют первой каталитической стадию, и дальнейшие перестройки происходят в каталитической активной сплайсосоме. Несколько взаимодействий РНК-РНК являются взаимоисключающими; Тем не менее, неизвестно, что запускает эти взаимодействия, ни порядок этих перестроек. Первая перестройка, вероятно, является смещением U1 SNRNP из 5 -дюймового сайта сплайсинга и образования взаимодействия SnRNA U6. Известно, что U1 SNRNP слабо связан с полностью сформированными сплайсосомами, [ 23 ] и U1 SNRNP ингибирует образование взаимодействия сплайсинга U6-5 'на модели субстратного олигонуклеотида, содержащего короткий 5' экзон и 5 'сайт сплайсинга. [ 24 ] Связывание U2 SNRNP с последовательности точек ветвей (BPS) является одним из примеров взаимодействия РНК-РНК, смещающего взаимодействие белка-РНК. При рекрутировании U2 SNRNP белок, связывающий ветвь, в комплексе обязательств смещен, поскольку сайт связывания SNRNA U2 и SF1 является взаимоисключающими событиями. [ Цитация необходима ]
Внутри SNRNA U2 существуют другие взаимоисключающие перестройки, которые происходят между конкурирующими конформациями. Например, в активной форме, петля STEM IIA предпочитается; В неактивной форме взаимно исключительное взаимодействие между петлей и нисходящей последовательности преобладает. [ 20 ] Неясно, как U4 смещена из SNRNA U6, хотя РНК была вовлечена в сборку сплайсосомы и может функционировать, чтобы расслабиться U4/U6 и способствовать образованию взаимодействия SnRNA U2/U6. Взаимодействие стволовых петлей U4/U6 I и II диссоциации, а область Freed STEM -петля II сгиба U6 на себя, образуя внутримолекулярную петлю STEM, и U4 больше не требуется в дальнейшей сборке сплисосомы. Область Freed STEM LOOP I в парных пар U6 с SNRNA U2, образующей спираль U2/U6 I. Однако структура спирали I взаимоисключает с 3 'половиной внутренней 5 -' области стволовой петли U2 SnRNA. [ Цитация необходима ]
Незначительная сплайсосома
[ редактировать ]У некоторых эукариот есть вторая сплайсосома, так называемая небольшая сплайсосома . [ 25 ] Группа менее распространенных SNRNAS, U11 , U12 , U4ATAC и U6ATAC вместе с U5 являются субъединицами незначительной сплайсосомы, которые разбивают редкий класс интронов до-мРНК, обозначенные типом U12. Незначительная сплайсосома расположена в ядре, как и его основной аналог, [ 26 ] Хотя в некоторых специализированных клетках есть исключения, включая Anucleate Lamestets [ 27 ] и дендроплазма ( дендритная цитоплазма) нейрональных клеток. [ 28 ]
Ссылки
[ редактировать ]- ^ Jump up to: а беременный Will Cl, Lührmann R (июль 2011 г.). «Структурная структура и функция» . Перспективы Cold Spring Harbor в биологии . 3 (7): A003707. doi : 10.1101/cshperspect.a003707 . PMC 3119917 . PMID 21441581 .
- ^ Jump up to: а беременный Berget SM, Moore C, Sharp PA (август 1977 г.). «Сплайсированные сегменты на 5 -дюймовом конце аденовируса 2 позднего мРНК» . Труды Национальной академии наук Соединенных Штатов Америки . 74 (8): 3171–5. Bibcode : 1977pnas ... 74.3171b . doi : 10.1073/pnas.74.8.3171 . PMC 431482 . PMID 269380 .
- ^ Чоу Л.Т., Робертс Дж. М., Льюис Дж.Б., брокер ТР (август 1977 г.). «Карта цитоплазматических транскриптов РНК из литического аденовируса 2 типа, определяемой электронной микроскопией РНК: гибриды ДНК». Клетка . 11 (4): 819–36. doi : 10.1016/0092-8674 (77) 90294-x . PMID 890740 . S2CID 37967144 .
- ^ Агафонов Д.Е., Декерт Дж., Вольф Э., Оденвалдер П., Бессонов С., Уилл К.Л. и др. (Июль 2011). «Полуколичественный протеомный анализ человеческой сплицеосомы с помощью нового двухмерного метода гель-электрофореза» . Молекулярная и клеточная биология . 31 (13): 2667–82. doi : 10.1128/mcb.05266-11 . PMC 3133382 . PMID 21536652 .
- ^ Кун А.Н., Ван Сантен М.А., Швиенхорст А., Урлауб Х., Люрманн Р. (январь 2009 г.). «Стал сплайсосомы сборки на отдельных стадиях с помощью малых молекул ингибиторов ацетилирования и деацетилирования белка» . РНК . 15 (1): 153–75. doi : 10.1261/rna.1332609 . PMC 2612777 . PMID 19029308 .
- ^ Патил В., Канзонери Дж.С., Саматов Т.Р., Люрманн Р., Ойелере А.К. (сентябрь 2012 г.). «Молекулярная архитектура цинка, хлежающих мелких молекул, которые ингибируют сборку сплайсосомы на ранней стадии» . РНК . 18 (9): 1605–11. doi : 10.1261/rna.034819.112 . PMC 3425776 . PMID 22832025 .
- ^ Кейт Дж. Х. (март 2016 г.). «Структура. Большой взрыв в структурной биологии сплайсосомы». Наука . 351 (6280): 1390–2. doi : 10.1126/science.aaf4465 . PMID 27013712 . S2CID 206648185 .
- ^ Häcker I, Sander B, Golas MM, Wolf E, Karagöz E, Kastner B, et al. (Ноябрь 2008 г.). «Локализация белков PRP8, BRR2, SNU114 и U4/U6 в дрожжевой Tri-SNRNP с помощью электронной микроскопии». Природа структурная и молекулярная биология . 15 (11): 1206–12. doi : 10.1038/nsmb.1506 . PMID 18953335 . S2CID 22982227 .
- ^ Yan C, Hang J, Wan R, Huang M, Wong CC, Shi Y (сентябрь 2015 г.). «Структура дрожжей сплайсосомы при разрешении 3,6-анго». Наука . 349 (6253): 1182–91. Bibcode : 2015sci ... 349.1182Y . doi : 10.1126/science.aac7629 . PMID 26292707 . S2CID 22194712 .
- ^ Zhang X, Yan C, Hang J, Finci Li, Lei J, Shi Y (май 2017). «Атомная структура человеческой сплайсосомы» . Клетка . 169 (5): 918–929.e14. doi : 10.1016/j.cell.2017.04.033 . PMID 28502770 .
- ^ Schmucker D, Clemens JC, Shu H, Worby CA, Xiao J, Muda M, et al. (Июнь 2000 г.). «Drosophila DSCAM - это рецептор Axon, демонстрирующий необычайное молекулярное разнообразие» . Клетка . 101 (6): 671–84. doi : 10.1016/s0092-8674 (00) 80878-8 . PMID 10892653 .
- ^ Spector DL, Lamond AI (февраль 2011 г.). "Ядерные спекли" . Обзор. Перспективы Cold Spring Harbor в биологии . 3 (2): A000646. doi : 10.1101/cshperspect.a000646 . PMC 3039535 . PMID 20926517 .
- ^ Jump up to: а беременный Bhat P, Chow A, Emert B, et al. (Май 2024). «Организация генома вокруг ядерных спеклз стимулирует эффективность сплайсинга мРНК» . Природа . 629 (5): 1165–1173. doi : 10.1038/s41586-024-07429-6 . PMC 11164319 . PMID 38720076 .
- ^ Jamison SF, Crow A, Garcia-Blanco MA (октябрь 1992 г.). «Путь сборки сплайсосомы в экстрактах млекопитающих» . Молекулярная и клеточная биология . 12 (10): 4279–87. doi : 10.1128/mcb.12.10.4279 . PMC 360351 . PMID 1383687 .
- ^ Seraphin B, Rosbash M (октябрь 1989 г.). «Идентификация функциональных комплексов snRNA-pre-мРНК U1, приверженных сборке сплайсосомы и сплайсинга». Клетка . 59 (2): 349–58. doi : 10.1016/0092-8674 (89) 90296-1 . PMID 2529976 . S2CID 18553973 .
- ^ Legrain P, Seraphin B, Rosbash M (сентябрь 1988 г.). «Ранняя приверженность дрожжей до-мРНК к пути сплайсосомы» . Молекулярная и клеточная биология . 8 (9): 3755–60. doi : 10.1128/mcb.8.9.3755 . PMC 365433 . PMID 3065622 .
- ^ Query CC, Moore MJ, Sharp PA (март 1994 г.). «Отбор нуклеофилов ветви в сплайсинге пре-мРНК: свидетельство выпиренной дуплексной модели» . Гены и развитие . 8 (5): 587–97. doi : 10.1101/gad.8.5.587 . PMID 7926752 .
- ^ Ньюби М.И., Гринбаум Н.Л. (декабрь 2002 г.). «Скульпирование мотива распознавания участка сплайсосомальной ветви консервативным псевдоуридином». Природа структурная биология . 9 (12): 958–65. doi : 10.1038/nsb873 . PMID 12426583 . S2CID 39628664 .
- ^ Burge CB, Tuschl T, Sharp PA (1999). «Предшественники сплайсинга в мРНК сплестисосомами». В Gesteland RF, Cech TR, Atkins JF (Eds.). Мир РНК . Cold Spring Harbor Lab. Нажимать. С. 525–60. ISBN 978-0-87969-380-0 .
- ^ Jump up to: а беременный Staley JP, Guthrie C (февраль 1998 г.). «Механические устройства сплайсосомы: двигатели, часы, пружины и вещи» . Клетка . 92 (3): 315–26. doi : 10.1016/s0092-8674 (00) 80925-3 . PMID 9476892 .
- ^ Newman AJ, Teigelkamp S, Beggs JD (ноябрь 1995 г.). «Взаимодействия снурны в 5 'и 3' сайтах сплайсинга, контролируемых с помощью фотоактивированного сшивания в дрожжевых сплайсосомах» . РНК . 1 (9): 968–80. PMC 1369345 . PMID 8548661 . Архивировано из оригинала 2005-02-23 . Получено 2008-03-07 .
- ^ Chiara MD, Palandjian L, Feld Kramer R, Reed R (август 1997 г.). «Доказательства того, что U5 SNRNP распознает 3 'сайт сплайсинга для каталитического этапа II у млекопитающих» . Embo Journal . 16 (15): 4746–59. doi : 10.1093/emboj/16.15.4746 . PMC 1170101 . PMID 9303319 .
- ^ Мур М.Дж., Шарп Па (сентябрь 1993 г.). «Доказательства двух активных сайтов в сплайсосоме, предоставленных стереохимией сплайсинга до мРНК». Природа . 365 (6444): 364–8. Bibcode : 1993natur.365..364M . doi : 10.1038/365364A0 . PMID 8397340 . S2CID 4361512 .
- ^ Konforti BB, Koziolkiewicz MJ, Konarska MM (декабрь 1993 г.). «Разрушение спаривания оснований между 5 -'сайтом сплайсинга и 5' конец SnRNA U1 требуется для сборки сплайсосомы». Клетка . 75 (5): 863–73. doi : 10.1016/0092-8674 (93) 90531-t . PMID 8252623 .
- ^ Патель А.А., Стейц Дж. (Декабрь 2003 г.). «Сплайсинг двойной: понимание второй сплайсосомы». Природные обзоры. Молекулярная клеточная биология . 4 (12): 960–70. doi : 10.1038/nrm1259 . PMID 14685174 . S2CID 21816910 .
- ^ Pessa HK, Will CL, Meng X, Schneider C, Watkins NJ, Perälä N, et al. (Июнь 2008 г.). «Незначительные компоненты сплайсосомы преимущественно локализованы в ядре» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (25): 8655–60. doi : 10.1073/pnas.0803646105 . PMC 2438382 . PMID 18559850 .
- ^ Денис М.М., Толли Н.Д., Бантинг М., Шверц Х., Цзян Х., Линдеманн С. и др. (Август 2005 г.). «Избавление от ядерных ограничений: зависящий от сигнального сплайсинга пре-мРНК в тромбоцитах с придурками» . Клетка . 122 (3): 379–91. doi : 10.1016/j.cell.2005.06.015 . PMC 4401993 . PMID 16096058 .
- ^ Glanzer J, Miyashiro Ky, Sul Jy, Barrett L, Belt B, Haydon P, et al. (Ноябрь 2005 г.). «Возможность сплайсинга РНК живых нейронов дендритов» . Труды Национальной академии наук Соединенных Штатов Америки . 102 (46): 16859–64. BIBCODE : 2005PNAS..10216859G . doi : 10.1073/pnas.0503783102 . PMC 1277967 . PMID 16275927 .
Дальнейшее чтение
[ редактировать ]- Мясник SE (2011). «Глава 8. Сплисосома и ее металлические ионы». В Sigel A, Sigel H, Sigel Rk (Eds.). Структурные и каталитические роли ионов металлов в РНК . Металлические ионы в науках о жизни. Тол. 9. RSC Publishing. С. 235–51. doi : 10.1039/9781849732512-00235 . ISBN 978-1-84973-094-5 Полем PMID 22010274 .
- Нильсен TW (декабрь 2003 г.). «Сплисосома: самая сложная макромолекулярная машина в ячейке?». Биологии . 25 (12): 1147–9. doi : 10.1002/bies.10394 . PMID 14635248 .
Внешние ссылки
[ редактировать ]- 3D макромолекулярные структуры сплицеосом из банка данных EM (EMDB)
- Сплицеосомы в Национальной библиотеке медицины США медицинские заголовки (Mesh)