Розетта@дома
 | |
 | |
| Разработчик(и) | лаборатория Бейкера, Вашингтонский университет; Розетта Коммонс |
|---|---|
| Первоначальный выпуск | 6 октября 2005 г |
| Стабильная версия | Розетта: 4.20 / 1 мая 2020 г Rosetta Mini: 3,78 / 3 октября 2017 г Rosetta для Android: 4.20 / 1 мая 2020 г |
| Статус разработки | Активный |
| Операционная система | Windows , macOS , Linux , Android |
| Платформа | БОИНК |
| Лицензия | Проприетарное бесплатное программное обеспечение для академического и некоммерческого использования. [1] |
| Средняя производительность | 225 041 гигафлопс [2] |
| Активные пользователи | 36,726 |
| Всего пользователей | 1,363,584 [3] |
| Активные хосты | 249,673 |
| Всего хостов | 529,112 |
| Веб-сайт | боинк |
Rosetta@home — это волонтерский компьютерный проект, исследующий прогнозирование структуры белков на платформе открытой инфраструктуры для сетевых вычислений Беркли (BOINC), управляемой лабораторией Бейкера. Rosetta@home стремится предсказать стыковку белков между белками и разработать новые белки с помощью около пятидесяти пяти тысяч активных компьютеров-добровольцев, обрабатывающих в среднем более 487 946 гигафлопс в среднем по состоянию на 19 сентября 2020 года. [4] Foldit , видеоигра Rosetta@home, стремится достичь этих целей с помощью краудсорсингового подхода. большая часть проекта ориентирована на фундаментальные исследования, направленные на повышение точности и надежности методов протеомики , Rosetta@home также проводит прикладные исследования малярии Хотя , болезни Альцгеймера и других патологий. [5]
Как и все проекты BOINC, Rosetta@home использует простаивающие вычислительные ресурсы компьютеров добровольцев для выполнения вычислений на отдельных рабочих единицах . Завершенные результаты отправляются на центральный сервер проекта , где они проверяются и вносятся в базы данных проекта . Проект является кроссплатформенным и работает на самых разных аппаратных конфигурациях. Пользователи могут наблюдать за ходом предсказания структуры индивидуального белка на заставке Rosetta@home.
Помимо исследований, связанных с болезнями, сеть Rosetta@home служит платформой для тестирования новых методов структурной биоинформатики . Такие методы затем используются в других приложениях на базе Rosetta, таких как RosettaDock или Human Proteome Folding Project и Microbiome Immunity Project , после того, как они достаточно разработаны и доказали свою стабильность на большом и разнообразном наборе компьютеров-добровольцев Rosetta@home. Двумя особенно важными тестами для новых методов, разработанных в Rosetta@home, являются эксперименты по критической оценке методов прогнозирования структуры белка (CASP) и критической оценке прогнозирования взаимодействий (CAPRI), проводимые раз в два года эксперименты, в которых оценивают современное состояние структуры белков. предсказание и предсказание межбелкового стыковки соответственно. Розетта неизменно входит в число лучших предсказателей стыковки и является одним из лучших доступных предсказателей третичной структуры . [6]
С притоком новых пользователей, желающих принять участие в борьбе с пандемией COVID-19 , вызванной SARS-CoV-2 , Rosetta@home увеличила свою вычислительную мощность до 1,7 петафлопс по состоянию на 28 марта 2020 года. [7] [8] 9 сентября 2020 года исследователи Rosetta@home опубликовали статью, описывающую 10 мощных противовирусных препаратов против SARS-CoV-2. Rosetta@home внесла свой вклад в это исследование, и эти кандидаты на противовирусные препараты приближаются к первой фазе клинических испытаний, которые могут начаться в начале 2022 года. [9] [10] [11] [12] По словам команды Rosetta@home, волонтеры Rosetta внесли свой вклад в разработку вакцины на основе наночастиц. [9] Эта вакцина была лицензирована и известна как IVX-411 компанией Icosavax, которая начала клинические испытания фазы I/II в июне 2021 года. [13] и GBP510 , который разрабатывается SK Bioscience и уже одобрен для клинического исследования фазы III в Южной Корее. [14] [15]
NL-201 , кандидат на лекарство от рака, который был впервые создан в Институте дизайна белков (IPD) и опубликован в статье в январе 2019 года. [16] в мае 2021 года при поддержке Neoleukin Therapeutics, дочерней компании IPD, началось клиническое исследование фазы 1 на людях. [17] Rosetta@home сыграла роль в разработке NL-201 и внесла свой вклад в эксперименты по «складыванию вперед», которые помогли проверить дизайн белков. [18]
Вычислительная платформа
[ редактировать ]Приложение Rosetta@home и волонтерская вычислительная платформа BOINC доступны для операционных систем Windows , Linux и macOS ; BOINC также работает на некоторых других платформах, например, на FreeBSD. [19] Для участия в Rosetta@home требуется центральный процессор (ЦП) с тактовой частотой не менее 500 МГц , 200 мегабайт свободного дискового пространства , 512 мегабайт физической памяти и подключение к Интернету. [20] По состоянию на 20 июля 2016 года актуальная версия приложения Rosetta Mini — 3.73. [21] Текущая рекомендуемая версия программы BOINC — 7.6.22. [19] Стандартный протокол передачи гипертекста (HTTP) ( порт 80) используется для связи между клиентом BOINC пользователя и серверами Rosetta@home в Вашингтонском университете; HTTPS (порт 443) используется при обмене паролями. Удаленное и локальное управление клиентом BOINC использует порт 31416 и порт 1043, которые, возможно, потребуется специально разблокировать, если они находятся за брандмауэром . [22] Рабочие единицы, содержащие данные об отдельных белках, передаются с серверов, расположенных в лаборатории Бейкера в Вашингтонском университете , на компьютеры добровольцев, которые затем рассчитывают прогноз структуры назначенного белка. Чтобы избежать дублирования предсказаний структуры данного белка, каждая рабочая единица инициализируется случайным начальным номером. белка Это дает каждому предсказанию уникальную траекторию движения по энергетическому ландшафту . [23] Прогнозы структуры белка, сделанные Rosetta@home, являются приближением глобального минимума в энергетическом ландшафте данного белка. Этот глобальный минимум представляет собой наиболее энергетически выгодную конформацию белка, т. е. его нативное состояние .
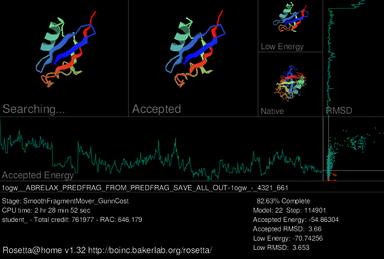
Основной особенностью графического пользовательского интерфейса (GUI) Rosetta@home является заставка , которая показывает прогресс текущего рабочего блока во время моделируемого процесса сворачивания белка . В левом верхнем углу текущей заставки показано, как целевой белок принимает различные формы (конформации) в поисках структуры с наименьшей энергией. Справа изображена структура последнего принятого документа. Вверху справа показана конформация текущей приманки с наименьшей энергией; ниже указана истинная, или нативная, структура белка, если она уже определена. В заставку включены три графика. график термодинамической свободной энергии Рядом с серединой отображается график среднеквадратического отклонения принятой модели, которая колеблется по мере изменения принятой модели. Справа показан принятой модели (RMSD), который показывает, насколько структурно схожа принятая модель с исходной моделью. Справа от принятого графика энергии и под графиком RMSD результаты этих двух функций используются для построения графика зависимости энергии от RMSD по мере постепенного уточнения модели. [24]
Как и все проекты BOINC, Rosetta@home работает в фоновом режиме на компьютере пользователя, используя мощность компьютера в режиме ожидания, либо во время входа в учетную запись в операционной системе хоста, либо до него . Программа освобождает ресурсы ЦП по мере того, как они необходимы другим приложениям, что не влияет на нормальное использование компьютера. Многие настройки программы можно указать через настройки учетной записи пользователя, в том числе: максимальный процент ресурсов ЦП, которые программа может использовать (для управления энергопотреблением или выработкой тепла на компьютере, работающем с постоянной производительностью), время суток, в течение которого программа может работать. и многие другие. [ нужна ссылка ]
Значение проекта
[ редактировать ]С распространением проектов секвенирования генома ученые могут сделать вывод об аминокислотной последовательности или первичной структуре многих белков, которые выполняют функции внутри клетки. Чтобы лучше понять функцию белка и помочь в разработке рациональных лекарств белка , ученым необходимо знать трехмерную третичную структуру .

Трехмерные структуры белков в настоящее время определяются экспериментально с помощью рентгеновской кристаллографии или спектроскопии ядерного магнитного резонанса (ЯМР). Этот процесс медленный (на то, чтобы понять, как впервые кристаллизовать белок, могут потребоваться недели или даже месяцы) и дорогостоящий (около 100 000 долларов США за белок). [25] К сожалению, скорость обнаружения новых последовательностей намного превышает скорость определения структуры – из более чем 7 400 000 белковых последовательностей, доступных в базе данных неизбыточных (nr) белков Национального центра биотехнологической информации (NCBI), менее 52 000 трехмерных структур белков были решены и помещены в Банк данных белков , главное хранилище структурной информации о белках. [26] Одна из основных целей Rosetta@home — прогнозировать структуры белков с той же точностью, что и существующие методы, но способом, требующим значительно меньше времени и денег. Rosetta@home также разрабатывает методы определения структуры и стыковки мембранных белков (например, рецепторов, связанных с G-белком (GPCR)), [27] которые исключительно трудно проанализировать с помощью традиционных методов, таких как рентгеновская кристаллография и ЯМР-спектроскопия, но которые, тем не менее, представляют собой большинство мишеней для современных лекарств. [28]
Прогресс в предсказании структуры белка оценивается в эксперименте «Критическая оценка методов предсказания структуры белка» (CASP), проводимом два раза в год, в котором исследователи со всего мира пытаются вывести структуру белка из его аминокислотной последовательности. Группы, набравшие высокие баллы в этом иногда конкурентном эксперименте, считаются де-факто знаменосцами современного уровня предсказания структуры белков. Rosetta, программа, на которой основана Rosetta@home, используется с CASP5 в 2002 году. В эксперименте CASP6 2004 года Rosetta вошла в историю, став первой, кто произвел разрешение, близкое к атомному уровню, ab initio предсказывая структуру белка в своей представленная модель для цели CASP T0281. [29] Ab initio моделирование считается особенно сложной категорией предсказания структуры белка, поскольку оно не использует информацию о структурной гомологии и должно полагаться на информацию о гомологии последовательностей и моделировании физических взаимодействий внутри белка. Rosetta@home используется в CASP с 2006 года, где он был одним из лучших средств прогнозирования во всех категориях прогнозирования структур в CASP7. [30] [31] [32] Эти высококачественные прогнозы стали возможны благодаря вычислительной мощности, предоставленной волонтерами Rosetta@home. [33] Увеличение вычислительной мощности позволяет Rosetta@home отбирать больше областей конформационного пространства (возможных форм, которые может принимать белок), которые, согласно парадоксу Левинталя , будут экспоненциально увеличиваться с длиной белка. [ нужна ссылка ]
Rosetta также используется для прогнозирования стыковки белков , что определяет структуру белков с множеством комплексов или четвертичную структуру . Этот тип взаимодействия белков влияет на многие клеточные функции, включая связывание антиген-антитело и фермент-ингибитор, а также клеточный импорт и экспорт. Определение этих взаимодействий имеет решающее значение для разработки лекарств . Rosetta используется в эксперименте по критической оценке прогнозирования взаимодействий (CAPRI), который оценивает состояние поля стыковки белков аналогично тому, как CASP измеряет прогресс в предсказании структуры белков. Вычислительная мощность, предоставленная волонтерами проекта Rosetta@home, была названа основным фактором производительности Rosetta на CAPRI 2007, где ее прогнозы стыковки были одними из самых точных и полных. [34]
В начале 2008 года Rosetta была использована для компьютерного проектирования белка с функцией, никогда ранее не наблюдавшейся в природе. [35] Частично это было вызвано отзывом громкой статьи 2004 года, в которой первоначально описывался вычислительный дизайн белка с улучшенной ферментативной активностью по сравнению с его естественной формой. [36] 2008 года Исследовательская статья группы Дэвида Бейкера, описывающая, как был создан белок, в которой упоминается Rosetta@home о предоставленных ею вычислительных ресурсах, представляет собой важное доказательство концепции этого метода проектирования белка. [35] Этот тип белковой конструкции может найти в будущем применение в разработке лекарств, зеленой химии и биоремедиации . [35]
Исследования, связанные с болезнями
[ редактировать ]Помимо фундаментальных исследований по прогнозированию структуры белка, стыковке и дизайну, Rosetta@home также используется в непосредственных исследованиях, связанных с болезнями. [37] Многочисленные мелкие исследовательские проекты описаны в журнале Rosetta@home Дэвида Бейкера. [38] По состоянию на февраль 2014 года на форуме обновляется информация о последних публикациях и краткое описание работы. [39] Ветка форума больше не используется с 2016 года, а новости об исследовании можно найти в разделе общих новостей проекта. [40]
болезнь Альцгеймера
[ редактировать ]Компонент пакета программного обеспечения Rosetta, RosettaDesign, использовался для точного прогнозирования того, какие области амилоидогенных белков с наибольшей вероятностью образуют амилоид -подобные фибриллы. [41] Взяв гексапептиды (фрагменты длиной шесть аминокислот) интересующего белка и выбрав наименьшую энергию, соответствующую структуре, аналогичной структуре известного гексапептида, образующего фибриллы, RosettaDesign смогла идентифицировать пептиды, которые в два раза чаще образуют фибриллы, чем случайные. белки. [42] Rosetta@home использовалась в том же исследовании для прогнозирования структур бета-амилоида , белка, образующего фибриллы, который, как предполагается, вызывает болезнь Альцгеймера . [43] Предварительные, но пока неопубликованные результаты были получены в отношении белков, разработанных Rosetta, которые могут предотвращать образование фибрилл, хотя неизвестно, могут ли они предотвратить заболевание. [44]
Сибирская язва
[ редактировать ]Другой компонент Rosetta, RosettaDock, [45] [46] [47] был использован в сочетании с экспериментальными методами для моделирования взаимодействия между тремя белками — летальным фактором (LF), фактором отека (EF) и защитным антигеном (PA), которые составляют токсин сибирской язвы . Компьютерная модель точно предсказала стыковку между LF и PA, помогая установить, какие домены соответствующих белков участвуют в комплексе LF-PA. Это открытие в конечном итоге было использовано в исследованиях, в результате которых были созданы улучшенные вакцины против сибирской язвы. [48] [49]
Вирус простого герпеса 1
[ редактировать ]RosettaDock использовался для моделирования стыковки между антителом ( иммуноглобулином G ) и поверхностным белком, экспрессируемым вирусом герпеса 1 (ВПГ-1), который служит для разрушения противовирусного антитела. Белковый комплекс, предсказанный RosettaDock, полностью соответствовал экспериментальным моделям, которые особенно трудно получить, что привело исследователей к выводу, что метод стыковки потенциально может решить некоторые проблемы, с которыми сталкивается рентгеновская кристаллография при моделировании интерфейсов белок-белок. [50]
ВИЧ
[ редактировать ]В рамках исследования, финансируемого за счет гранта в размере 19,4 миллиона долларов Фонда Билла и Мелинды Гейтс , [51] Rosetta@home использовалась при разработке множества возможных вакцин против вируса иммунодефицита человека ( ВИЧ ). [52] [53]
Малярия
[ редактировать ]В исследованиях, связанных с инициативой «Большие вызовы глобального здравоохранения» , [54] Rosetta использовалась для компьютерной разработки новых самонаводящихся эндонуклеазных белков, которые могли бы уничтожить Anopheles gambiae или иным образом сделать комара неспособным передавать малярию . [55] Возможность моделировать и изменять конкретно взаимодействия белок-ДНК, например, взаимодействия самонаводящихся эндонуклеаз, дает вычислительным методам проектирования белков, таким как Rosetta, важную роль в генной терапии (которая включает в себя возможные методы лечения рака). [37] [56]
COVID-19
[ редактировать ]В 2020 году пакет молекулярного моделирования Rosetta был использован для точного предсказания атомной структуры шипового белка SARS-CoV-2 за несколько недель до того, как его можно было измерить в лаборатории. [57] 26 июня 2020 года проект объявил, что ему удалось создать антивирусные белки, нейтрализующие вирионы SARS-CoV-2 в лаборатории, и что эти экспериментальные противовирусные препараты оптимизируются для испытаний на животных. [58]
Вслед за этим 9 сентября в журнале Science была опубликована статья, описывающая 10 ингибиторов минипротеина SARS-CoV-2. Два из этих ингибиторов, LCB1 и LCB3, в несколько раз более эффективны, чем лучшие моноклональные антитела, разрабатываемые против SARS-CoV. -2, как на молярной, так и на массовой основе. Кроме того, исследования показывают, что эти ингибиторы сохраняют свою активность при повышенных температурах, в 20 раз меньше антител и, следовательно, имеют в 20 раз больше потенциальных нейтрализующих участков, что повышает потенциальную эффективность препарата, вводимого местно. Ожидается, что небольшой размер и высокая стабильность ингибиторов сделают их пригодными для использования в виде геля, который можно наносить назально, или в виде порошка для введения непосредственно в дыхательную систему. В ближайшие месяцы исследователи будут работать над разработкой этих ингибиторов в качестве терапевтических и профилактических средств. [10] По прогнозам, по состоянию на июль 2021 года клинические испытания этих противовирусных препаратов начнутся в начале 2022 года, и они получили финансирование от Фонда Билла и Мелинды Гейтс на доклинические и ранние клинические испытания. [12] В испытаниях на животных эти противовирусные кандидаты оказались эффективны против вызывающих беспокойство вариантов, включая альфа, бета и гамма. [12] [59] [60]
Rosetta@home использовалась для скрининга более 2 миллионов белков, связывающих спайки SARS-CoV-2, которые были разработаны с помощью вычислений и, таким образом, внесли вклад в это исследование. [61] [62]
По словам команды Rosetta@home в Институте белкового дизайна, волонтеры Rosetta@home внесли свой вклад в разработку кандидатов на противовирусные препараты. [10] и к вакцине на основе белковых наночастиц. [63] Вакцина IVX-411 уже проходит первую фазу клинических испытаний, проводимых Icosavax. [13] в то время как та же вакцина, лицензированная другим производителем и под названием GBP510, была одобрена в Южной Корее для исследования фазы III, проводимого SK Bioscience . [15] [14] Противовирусные препараты-кандидаты также проходят клинические испытания фазы 1. [9]
Рак
[ редактировать ]Исследователи Rosetta@home разработали агонист рецептора IL-2 под названием Неолейкин-2/15, который не взаимодействует с альфа-субъединицей рецептора. Такие сигнальные молекулы иммунитета полезны при лечении рака. Хотя природный IL-2 страдает от токсичности из-за взаимодействия с альфа-субъединицей, разработанный белок гораздо безопаснее, по крайней мере, на животных моделях. [16] Rosetta@home участвовала в «экспериментах по складыванию вперед», которые помогли проверить проекты. [18]
В статье в журнале New Yorker за сентябрь 2020 года Дэвид Бейкер заявил, что клинические испытания Неолейкина-2/15 на людях начнутся «позже в этом году». Neoleukin-2/15 разрабатывается Neoleukin , дочерней компанией лаборатории Бейкера. [64] В декабре 2020 года компания Neoleukin объявила, что подаст заявку на новое исследуемое лекарство в Управление по контролю за продуктами и лекарствами , чтобы начать клиническое исследование фазы 1 NL-201, которое является дальнейшим развитием Neoleukin-2/15. Аналогичная заявка была подана в Австралии, и Неолейкин надеется привлечь 120 участников к клиническому исследованию фазы 1. [65] Клинические испытания фазы 1 на людях начались 5 мая 2021 года. [17]
Розеточное программное обеспечение
[ редактировать ]| Разработчик(и) | лаборатория Бейкера, Вашингтонский университет; Розетта Коммонс |
|---|---|
| Первоначальный выпуск | 1998 [66] |
| Стабильная версия | 3.13 [67] |
| Лицензия | доступен собственный исходный код [68] бесплатное программное обеспечение для академического использования; имеется коммерческая лицензия [69] |
| Веб-сайт | www |
Rosetta — это программное обеспечение, отвечающее за прогнозирование структуры в Rosetta@home. Помимо кластера BOINC, Rosetta может работать на одном локальном компьютере или на локальном суперкомпьютере. Подобно другим биоинформатическим программам, существуют общедоступные онлайн-серверы, предлагающие запуск Rosetta из веб-интерфейса. [70] Программное обеспечение свободно лицензируется академическому сообществу и доступно фармацевтическим компаниям за определенную плату. [71]
Первоначально представленный лабораторией Бейкера в Вашингтонском университете в 1998 году как ab initio подход к предсказанию структуры, Rosetta с тех пор разделился на несколько направлений разработки и отдельных услуг, предоставляя такие функции, как стыковка макромолекул и дизайн белков . [66] Многие из аспирантов и других исследователей, участвовавших в первоначальной разработке Rosetta, с тех пор перешли в другие университеты и исследовательские институты и впоследствии усовершенствовали различные части проекта Rosetta.
Платформа Rosetta получила свое название от Розеттского камня , поскольку она пытается расшифровать структурный «смысл» аминокислотных последовательностей белков. [72] Разработка кода Rosetta осуществляется Rosetta Commons. [71] Rosetta участвует в CASP и CAPRI .
Rosetta была переписана на C++ , чтобы упростить разработку по сравнению с исходной версией, написанной на Фортране . Эта новая версия является объектно-ориентированной и была выпущена для Rosetta@Home 8 февраля 2008 года. [21] [73]
Розетта Дизайн
[ редактировать ]
RosettaDesign, компьютерный подход к проектированию белков, основанный на Rosetta, начался в 2000 году с исследования по изменению пути сворачивания G. белка [74] В 2002 году RosettaDesign была использована для создания Top7 белка длиной 93 аминокислоты , α/β- , у которого общая складка никогда ранее не наблюдалась в природе. Эта новая конформация была предсказана Розеттой с точностью до 1,2 Å RMSD от структуры, определенной с помощью рентгеновской кристаллографии , что представляет собой необычайно точное предсказание структуры. [75] Rosetta и RosettaDesign получили широкое признание, будучи первыми, кто разработал и точно предсказал структуру нового белка такой длины, о чем свидетельствует статья 2002 года, описывающая двойной подход, что вызвало два положительных письма в журнале Science : [76] [77] и цитируется более чем в 240 других научных статьях. [78] Видимый продукт этого исследования, Top7 , был назван «Молекулой месяца» RCSB PDB в октябре 2006 года; [79] суперпозиция . соответствующих ядер (остатков 60–79) его предсказанной и рентгеновской кристаллических структур представлена в логотипе Rosetta@home [29]
Брайан Кульман, бывший научный сотрудник лаборатории Дэвида Бейкера , а ныне доцент Университета Северной Каролины в Чапел-Хилл , [80] предлагает RosettaDesign в качестве онлайн-сервиса. [81]
РозеттаДок
[ редактировать ]RosettaDock был добавлен в пакет программного обеспечения Rosetta во время первого эксперимента CAPRI лаборатории Бейкера в 2002 году в качестве алгоритма для прогнозирования стыковки белков между собой . [82] В этом эксперименте RosettaDock сделал прогноз высокой точности для стыковки между стрептококковым гноеродным экзотоксином А и β-цепью рецептора Т-клеток , а также прогноз средней точности для комплекса между свиньи альфа-амилазой и верблюда антителом . Хотя метод RosettaDock позволил сделать только два приемлемо точных прогноза из семи возможных, этого было достаточно, чтобы поставить его на седьмое место из девятнадцати методов прогнозирования в первой оценке CAPRI. [82]
Разработка RosettaDock разделилась на две ветви для последующих раундов CAPRI, поскольку Джеффри Грей, который заложил основу для RosettaDock во время работы в Вашингтонском университете , продолжил работу над методом на своей новой должности в Университете Джонса Хопкинса . Члены лаборатории Бейкера продолжили разработку RosettaDock в отсутствие Грея. Две версии немного различались в моделировании боковой цепи, выборе приманки и других областях. [47] [83] Несмотря на эти различия, методы Бейкера и Грея показали хорошие результаты во второй оценке CAPRI, заняв пятое и седьмое места соответственно из 30 групп предикторов. [84] Сервер RosettaDock Джеффри Грея доступен в качестве бесплатной службы прогнозирования стыковок для некоммерческого использования. [85]
В октябре 2006 года RosettaDock был интегрирован в Rosetta@home. В методе использовалась фаза быстрой и грубой модели стыковки с использованием только белкового остова . За этим последовала медленная фаза полноатомного уточнения, в которой ориентация двух взаимодействующих белков относительно друг друга и взаимодействия боковых цепей на границе белок-белок одновременно оптимизировались для поиска конформации с наименьшей энергией. [86] Значительно возросшая вычислительная мощность, обеспечиваемая сетью Rosetta@home, в сочетании с пересмотренным представлением складчатого дерева для обеспечения гибкости магистральной сети и моделирования петель , сделала RosettaDock шестой из 63 групп прогнозирования в третьей оценке CAPRI. [6] [34]
Робетта
[ редактировать ]Сервер Robetta (Rosetta Beta) — это автоматизированная служба прогнозирования структуры белков, предлагаемая лабораторией Бейкера для некоммерческого ab initio и сравнительного моделирования. [87] Он участвовал в качестве сервера автоматического прогнозирования в экспериментах CASP, проводимых два раза в год , начиная с CASP5 в 2002 году, показывая одни из лучших результатов в категории автоматизированного прогнозирования серверов. [88] С тех пор Робетта участвовала в соревнованиях CASP6 и 7, где показала результаты выше среднего среди групп автоматизированных серверов и людей-предсказателей. [32] [89] [90] Он также участвует в непрерывной оценке CAMEO3D . Задачи Robetta выполняются на серверах лаборатории Baker, компьютерах исследовательского кампуса Janelia и компьютерах участников Rosetta@home. [87]
При моделировании структуры белка CASP6 Робетта сначала ищет структурные гомологи, используя BLAST , PSI-BLAST и 3D-Jury , затем анализирует целевую последовательность на отдельные домены или независимо сворачивает единицы белков, сопоставляя последовательность со структурными семействами. в базе данных Pfam . Домены со структурными гомологами затем следуют протоколу «модели на основе шаблона» (т.е. моделирования гомологии ). Здесь собственная программа выравнивания лаборатории Бейкера, K*sync, создает группу гомологов последовательностей, и каждый из них моделируется методом Rosetta de novo для создания приманки (возможная структура). Окончательный прогноз структуры выбирается на основе модели с наименьшей энергией , определяемой энергетической функцией Розетты с низким разрешением. Для доменов, у которых не обнаружены структурные гомологи, применяется протокол de novo , в котором в качестве окончательного предсказания выбирается модель с наименьшей энергией из набора сгенерированных ложных целей. Эти предсказания доменов затем соединяются вместе для исследования междоменных взаимодействий на третичном уровне внутри белка. Наконец, вклады боковой цепи моделируются с использованием протокола для Монте-Карло . Конформационный поиск [91]
В CASP8 Робетта была дополнена для использования метода всеатомного уточнения Розетты с высоким разрешением. [92] отсутствие которого было названо основной причиной того, что Robetta была менее точной, чем сеть Rosetta@home в CASP7. [33] В CASP11 был добавлен способ прогнозирования карты контактов белков путем совместной эволюции остатков в родственных белках, называемый GREMLIN, что позволяет добиться большего de novo . успеха в складывании [93]
Другие серверы Rosetta
[ редактировать ]Rosetta доступна в виде онлайн-сервиса на ряде других общедоступных серверов. ROSIE предлагает множество функций: от прогнозирования и проектирования структуры РНК до стыковки лигандов и моделирования антител. [94]
Фолдит
[ редактировать ]9 мая 2008 года, после того как пользователи Rosetta@home предложили интерактивную версию волонтерской вычислительной программы, лаборатория Бейкера публично выпустила Foldit — онлайн-игру для предсказания структуры белков, основанную на платформе Rosetta. [95] По состоянию на 25 сентября 2008 г. [update]У Foldit было более 59 000 зарегистрированных пользователей. [96] Игра предоставляет пользователям набор элементов управления (например, встряхивать, покачивать, перестраивать), позволяющих манипулировать основной цепью аминокислот и боковыми цепями целевого белка, придавая им более энергетически выгодные конформации. Пользователи могут работать над решениями индивидуально в качестве солистов или коллективно в качестве разработчиков , накапливая баллы в любой категории по мере улучшения своих прогнозов структуры. [97]
Foldit может работать как графический интерфейс Rosetta в специальном «профессиональном режиме». [70]
РозаTTAFold
[ редактировать ]RoseTTAFold, вдохновленный AlphaFold , использует нейронную сеть для прогнозирования расстояния и ориентации между остатками. Эти прогнозы помогают программному обеспечению Rosetta создавать структуру. RoseTTAFold имеет открытый исходный код под лицензией MIT . [98]
Филиалы лабораторий, не принадлежащих компании Baker
[ редактировать ]Лаборатория Цзяньи Ян в Китае предлагает модифицированную версию Rosetta под названием tr-RosettaX2 (Rosetta с ограниченным преобразованием). [99] Он использует метод прогнозирования контактов на основе глубокого обучения, отличный от RoseTTAFold, для управления обычным алгоритмом складывания Rosetta. trRosetta предшествует RoseTTAFold. [100]
Сравнение с аналогичными волонтерскими компьютерными проектами
[ редактировать ]Существует несколько добровольных компьютерных проектов, области исследований которых аналогичны темам Rosetta@home, но отличаются исследовательским подходом:
Складной@дома
[ редактировать ]Из всех крупных добровольных компьютерных проектов, занимающихся исследованием белков, Folding@home — единственный, не использующий платформу BOINC . [101] [102] [103] И Rosetta@home, и Folding@home изучают заболевания, связанные с неправильным сворачиванием белков, такие как болезнь Альцгеймера , но Folding@home занимается гораздо более эксклюзивными исследованиями. [104] [105] всех атомов, молекулярной динамики Folding@home почти исключительно использует модели чтобы понять, как и почему белки сворачиваются (или потенциально неправильно сворачиваются и впоследствии агрегируются, вызывая заболевания). [106] [107] Другими словами, сила Folding@home заключается в моделировании процесса сворачивания белка, а сила Rosetta@home — в расчете конструкции белка и прогнозировании структуры и стыковки белка.
Некоторые результаты Rosetta@home используются в качестве основы для некоторых проектов Folding@home. Розетта предлагает наиболее вероятную структуру, но неясно, является ли именно эту форму молекула и жизнеспособна ли она. Folding@home затем можно использовать для проверки результатов Rosetta@home и предоставить дополнительную информацию на атомном уровне, а также подробную информацию о том, как молекула меняет форму. [107] [108]
Эти два проекта также существенно различаются по своей вычислительной мощности и разнообразию хостов. В среднем около 6650 терафлопс от хостовой базы центральных процессоров (ЦП), графических процессоров (ГП) и (ранее) PS3 , [109] У Folding@home почти в 108 раз больше вычислительной мощности, чем у Rosetta@home. [110]
Сетка мирового сообщества
[ редактировать ]И Фаза I, и Фаза II Проекта складывания протеома человека (HPF), подпроекта World Community Grid , использовали программу Rosetta для создания структурных и функциональных аннотаций различных геномов . [111] [112] Хотя теперь он использует его для создания баз данных для биологов, Ричард Бонно , главный научный сотрудник проекта «Складывание протеома человека», принимал активное участие в первоначальной разработке Rosetta в лаборатории Дэвида Бейкера, когда получил докторскую степень. [113] Более подробную информацию о взаимосвязи между HPF1, HPF2 и Rosetta@home можно найти на веб-сайте Ричарда Бонно. [114]
Предсказатель@дома
[ редактировать ]Как и Rosetta@home, Predictor@home специализируется на предсказании структуры белков. [115] В то время как Rosetta@home использует программу Rosetta для прогнозирования структуры, Predictor@home использовал методологию dTASSER. [116] В 2009 году Predictor@home закрылся.
Другие проекты волонтерских вычислений, связанные с белками, на BOINC включают QMC@home , Docking@home , POEM@home , SIMAP и TANPAKU . проект Rosetta@home RALPH@home, альфа- , который тестирует новые версии приложений, рабочие модули и обновления перед их переходом на Rosetta@home, также работает на BOINC. [117]
Добровольные взносы
[ редактировать ]Rosetta@home зависит от вычислительных мощностей, предоставленных отдельными участниками проекта для его исследований. По состоянию на 28 марта 2020 г. [update]Около 53 000 пользователей из 150 стран были активными участниками Rosetta@home, что в совокупности способствовало простою процессоров примерно 54 800 компьютеров, что привело к совокупной средней производительности более 1,7 петафлопс . [110] [118]

Пользователям предоставляются кредиты BOINC в качестве меры их вклада. Кредит, предоставленный для каждой рабочей единицы, представляет собой количество ложных целей, созданных для этой рабочей единицы, умноженное на средний заявленный балл за ложные копии, представленные всеми компьютерными хостами для этой рабочей единицы. Эта специальная система была разработана для устранения существенных различий между кредитами, предоставленными пользователям со стандартным клиентом BOINC и оптимизированным клиентом BOINC, а также различиями в кредитах между пользователями, использующими Rosetta@home в Windows и Linux . операционных системах [119] Сумма кредитов, предоставляемых за секунду работы ЦП, для Rosetta@home ниже, чем для большинства других проектов BOINC. [120] Rosetta@home занимает тринадцатое место из более чем 40 проектов BOINC по общему объему кредитов. [121]
Пользователи Rosetta@home, предсказывающие белковые структуры, представленные в эксперименте CASP, получили признание в научных публикациях относительно своих результатов. [33] Пользователи, которые предсказывают наименьшую энергетическую структуру для данного рабочего подразделения, отображаются на домашней странице Rosetta@home как Предсказатель дня вместе со всеми командами, членами которых они являются. [122] Пользователь дня выбирается случайным образом каждый день, чтобы он также был на главной странице, из числа пользователей, создавших профиль Rosetta@home. [123]
Ссылки
[ редактировать ]- ^ «Лицензионное соглашение Rosetta@home» . Boinc.bakerlab.org. Архивировано из оригинала 12 июня 2020 года . Проверено 12 июня 2020 г.
- ^ «Розетта@дома» . Архивировано из оригинала 2 апреля 2023 года . Проверено 18 июня 2023 г.
- ^ «Rosetta@Home – Подробная статистика | BOINCstats/BAM!» . Архивировано из оригинала 11 февраля 2019 года . Проверено 20 октября 2018 г.
- ^ «Розетта@дома» . Архивировано из оригинала 14 марта 2016 года . Проверено 24 марта 2020 г.
- ^ «Что такое Розетта@дома?» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 13 сентября 2008 года . Проверено 7 сентября 2008 г.
- ^ Jump up to: а б Ленсинк М.Ф., Мендес Р., Водак С.Дж. (декабрь 2007 г.). «Стыковка и оценка белковых комплексов: CAPRI, 3-е издание». Белки . 69 (4): 704–18. дои : 10.1002/прот.21804 . ПМИД 17918726 . S2CID 25383642 .
- ^ «Rosetta@home — Статус сервера «оценка терафлопс» » . Розетта@дома . 25 марта 2020 года. Архивировано из оригинала 3 января 2016 года . Проверено 25 марта 2020 г.
- ^ «Rosetta@home объединяет легион компьютеров против коронавируса» . HPCWire . 24 марта 2020 года. Архивировано из оригинала 9 апреля 2020 года . Проверено 25 марта 2020 г.
- ^ Jump up to: а б с «Проекты по борьбе с COVID-19 на нашей платформе переходят к клиническим испытаниям на людях! Наши замечательные онлайн-добровольцы сыграли свою роль в разработке новой многообещающей вакцины, а также потенциальных противовирусных методов лечения» . Твиттер . 25 июня 2021 года. Архивировано из оригинала 26 июня 2021 года . Проверено 26 июня 2021 г.
- ^ Jump up to: а б с Као Л., Горешник И., Ковентри Б., Кейс Дж.Б., Миллер Л., Козодой Л. и др. (октябрь 2020 г.). «Разработка de novo пикомолярных ингибиторов минипротеина SARS-CoV-2» . Наука . 370 (6515): 426–431. Бибкод : 2020Sci...370..426C . дои : 10.1126/science.abd9909 . ПМЦ 7857403 . ПМИД 32907861 .
- ^ «Информация о коронавирусе от Дэвида Бейкера. Спасибо всем за ваш вклад!» . Розетта@дома . 21 сентября 2020 года. Архивировано из оригинала 30 октября 2020 года . Проверено 23 сентября 2020 г.
- ^ Jump up to: а б с «Годовой отчет IPD за 2021 год» (PDF) . Институт белкового дизайна . 14 июля 2021 г. Архивировано (PDF) из оригинала 18 августа 2021 г. . Проверено 18 августа 2021 г.
- ^ Jump up to: а б «АНЗКТР – Регистрация» . anzctr.org.au . Архивировано из оригинала 30 октября 2021 года . Проверено 9 июля 2021 г.
- ^ Jump up to: а б «Южная Корея одобрила третью фазу испытаний вакцины против COVID-19 компании SK Bioscience» . Рейтер . 10 августа 2021 года. Архивировано из оригинала 18 августа 2021 года . Проверено 18 августа 2021 г.
- ^ Jump up to: а б Институт белкового дизайна (10 августа 2021 г.). «Архивная копия» . Твиттер . Архивировано из оригинала 18 августа 2021 года . Проверено 18 августа 2021 г.
{{cite web}}: CS1 maint: архивная копия в заголовке ( ссылка ) - ^ Jump up to: а б Сильва Д.А., Ю.С., Ульге Ю.И., Спенглер Дж.Б., Джуд К.М., Лабао-Алмейда С., Али Л.Р., Кихано-Рубио А., Рутербуш М., Люнг И., Биари Т., Кроули С.Дж., Маркос Э., Уокки К.Д., Вайцнер Б.Д., Пардо -Авила Ф., Кастельянос Дж., Картер Л., Стюарт Л., Ридделл С.Р., Пеппер М., Бернардес Дж.Дж., Дуган М., Гарсия К.К., Бейкер Д. (январь 2019 г.). «Разработка de novo мощных и селективных имитаторов IL-2 и IL-15» . Природа . 565 (7738): 186–191. Бибкод : 2019Natur.565..186S . дои : 10.1038/s41586-018-0830-7 . ПМК 6521699 . ПМИД 30626941 .
- ^ Jump up to: а б «Neoleukin Therapeutics объявляет о начале фазы 1 исследования NL-201 | Neoleukin Therapeutics, Inc» . www.neoleukin.com.investor.neoleukin.com . Архивировано из оригинала 24 июня 2021 года . Проверено 22 июня 2021 г.
- ^ Jump up to: а б «Еще одна публикация в журнале Nature, описывающая первые белки, созданные de novo и обладающие противораковой активностью» . Розетта@дома . 14 января 2020 года. Архивировано из оригинала 19 октября 2020 года . Проверено 19 сентября 2020 г.
- ^ Jump up to: а б «Загрузить клиентское программное обеспечение BOINC» . БОИНК . Калифорнийский университет. 2008. Архивировано из оригинала 7 декабря 2008 года . Проверено 1 декабря 2008 г.
- ^ «Rosetta@home: Рекомендуемые системные требования» . Розетта@дома . Университет Вашингтона. 2008. Архивировано из оригинала 25 сентября 2008 года . Проверено 7 октября 2008 г.
- ^ Jump up to: а б «Rosetta@home: Архив новостей» . Розетта@дома . Университет Вашингтона. 2016. Архивировано из оригинала 16 июня 2016 года . Проверено 20 июля 2016 г.
- ^ «Rosetta@home: Часто задаваемые вопросы (в разработке) (сообщение 10910)» . Форумы Rosetta@home . Университет Вашингтона. 2006. Архивировано из оригинала 26 февраля 2009 года . Проверено 7 октября 2008 г.
- ^ Ким Д.Э. (2005). «Rosetta@home: случайное начальное число (сообщение 3155)» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 26 февраля 2009 года . Проверено 7 октября 2008 г.
- ^ «Rosetta@home: Краткое руководство по Rosetta и ее графике» . Розетта@дома . Университет Вашингтона. 2007. Архивировано из оригинала 24 сентября 2008 года . Проверено 7 октября 2008 г.
- ^ Борн П.Е., Хельге В., ред. (2003). Структурная биоинформатика . Хобокен, Нью-Джерси: Вили-Лисс. ISBN 978-0-471-20199-1 . OCLC 50199108 .
- ^ «Ежегодный рост белковых структур» . Банк данных белков RCSB. 2021. Архивировано из оригинала 1 мая 2021 года . Проверено 1 мая 2021 г.
- ^ Бейкер Д. (2008). «Rosetta@home: журнал Rosetta@home Дэвида Бейкера (сообщение 55893)» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 23 июня 2008 года . Проверено 7 октября 2008 г.
- ^ Сантос, Рита; Урсу Олег; Голтон, Анна; Бенто, А. Патрисия; Донади, Рамеш С.; Болонья, Кристиан Г.; Карлссон, Аннели; Аль-Лазикани, Бисан; Херси, Энн; Опря, Тюдор И.; Оверингтон, Джон П. (2 декабря 2016 г.). «Полная карта целей молекулярных лекарств» . Nature Reviews Открытие лекарств . 16 (1): 19–34. дои : 10.1038/nrd.2016.230 . ПМК 6314433 . ПМИД 27910877 .
- ^ Jump up to: а б «Rosetta@home: Обзор исследования» . Розетта@дома . Университет Вашингтона. 2007. Архивировано из оригинала 25 сентября 2008 года . Проверено 7 октября 2008 г.
- ^ Копп Дж., Бордоли Л., Бэтти Дж.Н., Кифер Ф., Шведе Т. (2007). «Оценка прогнозов CASP7 для целей моделирования на основе шаблонов». Белки . 69 (Приложение 8): 38–56. дои : 10.1002/прот.21753 . ПМИД 17894352 . S2CID 31575350 .
- ^ Прочтите RJ, Chavali G (2007). «Оценка прогнозов CASP7 в категории высокоточного моделирования на основе шаблонов» . Белки . 69 (Приложение 8): 27–37. дои : 10.1002/прот.21662 . ПМИД 17894351 . S2CID 33172629 .
- ^ Jump up to: а б Джаух Р., Йео ХК, Колаткар П.Р., Кларк Н.Д. (2007). «Оценка прогнозов структуры CASP7 для целей без шаблонов» . Белки . 69 (Приложение 8): 57–67. дои : 10.1002/прот.21771 . ПМИД 17894330 . S2CID 38430899 .
- ^ Jump up to: а б с Дас Р., Цянь Б., Раман С. и др. (2007). «Прогнозирование структуры целей CASP7 с использованием обширного уточнения всех атомов с помощью Rosetta@home». Белки . 69 (Приложение 8): 118–28. дои : 10.1002/прот.21636 . ПМИД 17894356 . S2CID 17470364 .
- ^ Jump up to: а б Ван С., Шуелер-Фурман О., Андре И. и др. (декабрь 2007 г.). «RosettaDock в раундах CAPRI 6–12». Белки . 69 (4): 758–63. дои : 10.1002/prot.21684 . ПМИД 17671979 . S2CID 9028237 .
Большой объем стыковочных вычислений для предсказаний целей 27 и 28 был выполнен в Rosetta@Home (www.boinc.bakerlab.org), проекте распределенных вычислений, использующем программное обеспечение Rosetta на персональных компьютерах добровольцев из по всему миру с использованием технологии Berkeley Open Infrastructure for Network Computing (BOINC) (www.boinc.berkeley.edu).
- ^ Jump up to: а б с Цзян Л., Альтхофф Э.А., Клементе Ф.Р. и др. (март 2008 г.). «Вычислительный дизайн ретро-альдольных ферментов de novo» . Наука . 319 (5868): 1387–91. Бибкод : 2008Sci...319.1387J . дои : 10.1126/science.1152692 . ПМК 3431203 . ПМИД 18323453 .
- ^ Хайден ЕС (13 февраля 2008 г.). «Протеиновый приз можно получить после отзыва» . Природа . дои : 10.1038/news.2008.569 .
- ^ Jump up to: а б «Исследования, связанные с болезнями» . Розетта@дома . Университет Вашингтона. 2008. Архивировано из оригинала 23 сентября 2008 года . Проверено 8 октября 2008 г.
- ^ Бейкер Д. (2008). «Rosetta@home: журнал Дэвида Бейкера Rosetta@home» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 25 сентября 2008 года . Проверено 7 сентября 2008 г.
- ^ «Обновления исследований Rosetta@home» . Boinc.bakerlab.org. Архивировано из оригинала 19 апреля 2014 года . Проверено 18 апреля 2014 г.
- ^ «Архив новостей» . Розетта@дома . Архивировано из оригинала 10 мая 2019 года . Проверено 10 мая 2019 г.
- ^ Кульман Б., Бейкер Д. (сентябрь 2000 г.). «Нативные белковые последовательности близки к оптимальным по своей структуре» . Труды Национальной академии наук Соединенных Штатов Америки . 97 (19): 10383–88. Бибкод : 2000PNAS...9710383K . дои : 10.1073/pnas.97.19.10383 . ПМК 27033 . ПМИД 10984534 .
- ^ Томпсон М.Дж., Сиверс С.А., Караниколас Дж., Иванова М.И., Бейкер Д., Айзенберг Д. (март 2006 г.). «3D-профильный метод идентификации фибриллообразующих сегментов белков» . Труды Национальной академии наук Соединенных Штатов Америки . 103 (11): 4074–78. Бибкод : 2006PNAS..103.4074T . дои : 10.1073/pnas.0511295103 . ПМЦ 1449648 . ПМИД 16537487 .
- ^ Брэдли П. «Форум Rosetta@home: предсказание структуры амилоидных фибрилл» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 26 февраля 2009 года . Проверено 7 сентября 2008 г.
- ^ Бейкер Д. «Форум Rosetta@home: Публикации о работе R@H по борьбе с болезнью Альцгеймера? (сообщение 54681)» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 26 февраля 2009 года . Проверено 8 октября 2008 г.
- ^ Ван С., Шулер-Фурман О., Бейкер Д. (май 2005 г.). «Улучшенное моделирование боковой цепи для стыковки белков с белками» . Белковая наука . 14 (5): 1328–39. дои : 10.1110/ps.041222905 . ПМК 2253276 . ПМИД 15802647 .
- ^ Грей Дж.Дж., Мугон С., Ван С. и др. (август 2003 г.). «Стыковка белков с белками с одновременной оптимизацией смещения твердого тела и конформации боковой цепи». Журнал молекулярной биологии . 331 (1): 281–99. дои : 10.1016/S0022-2836(03)00670-3 . ПМИД 12875852 .
- ^ Jump up to: а б Шуелер-Фурман О., Ван С., Бейкер Д. (август 2005 г.). «Прогресс в стыковке белков между белками: прогнозы атомного разрешения в эксперименте CAPRI с использованием RosettaDock с улучшенной трактовкой гибкости боковой цепи». Белки . 60 (2): 187–94. дои : 10.1002/прот.20556 . ПМИД 15981249 . S2CID 17672050 .
- ^ Лейси Д.Б., Лин Х.К., Мельник Р.А. и др. (ноябрь 2005 г.). «Модель летального фактора токсина сибирской язвы, связанного с защитным антигеном» . Труды Национальной академии наук Соединенных Штатов Америки . 102 (45): 16409–14. Бибкод : 2005PNAS..10216409L . дои : 10.1073/pnas.0508259102 . ПМЦ 1283467 . ПМИД 16251269 .
- ^ Альбрехт М.Т., Ли Х., Уильямсон Э.Д. и др. (ноябрь 2007 г.). «Человеческие моноклональные антитела против летального фактора сибирской язвы и защитного антигена действуют независимо, защищая от инфекции Bacillus anthracis и усиливая эндогенный иммунитет к сибирской язве» . Инфекция и иммунитет . 75 (11): 5425–33. дои : 10.1128/IAI.00261-07 . ПМК 2168292 . ПМИД 17646360 .
- ^ Спраг Э.Р., Ван С., Бейкер Д., Бьоркман П.Дж. (июнь 2006 г.). «Кристаллическая структура Fc-рецептора HSV-1, связанного с Fc, раскрывает механизм биполярного мостикового соединения антител» . ПЛОС Биология . 4 (6): е148. дои : 10.1371/journal.pbio.0040148 . ПМЦ 1450327 . ПМИД 16646632 .
- ^ Полсон, Том (19 июля 2006 г.). «Фонд Гейтса выделил 287 миллионов долларов на исследования вакцины против ВИЧ» . Сиэтлский пост-разведчик . Архивировано из оригинала 5 сентября 2022 года . Проверено 7 сентября 2008 г.
- ^ Лю Ю и др. (2007). «Разработка каркасов IgG1 b12 и иммуногенов внешнего домена на основе env ВИЧ-1, способных вызывать и обнаруживать IgG1 b12-подобные антитела» (PDF) . Глобальное предприятие по производству вакцины против ВИЧ. Архивировано из оригинала (PDF) 25 февраля 2009 г. Проверено 28 сентября 2008 г.
- ^ Бейкер Д. «Архив журнала Дэвида Бейкера Rosetta@home (сообщение 40756)» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 26 февраля 2009 года . Проверено 7 сентября 2008 г.
- ^ «Наведение генов эндонуклеазы: новые инструменты для инженерии и контроля популяций комаров» . Большие проблемы глобального здравоохранения. Архивировано из оригинала 6 июля 2008 года . Проверено 7 сентября 2008 г.
- ^ Виндбихлер Н., Папатанос П.А., Каттеручча Ф., Рэнсон Х., Берт А., Крисанти А. (2007). «Нацеливание на гены, опосредованное эндонуклеазой Homeing, в клетках и эмбрионах Anopheles gambiae» . Исследования нуклеиновых кислот . 35 (17): 5922–33. дои : 10.1093/нар/gkm632 . ПМК 2034484 . ПМИД 17726053 .
- ^ Эшворт Дж., Хавранек Дж.Дж., Дуарте К.М. и др. (июнь 2006 г.). «Вычислительная реорганизация связывания ДНК эндонуклеаз и специфичности расщепления» . Природа . 441 (7093): 656–59. Бибкод : 2006Natur.441..656A . дои : 10.1038/nature04818 . ПМЦ 2999987 . ПМИД 16738662 .
- ^ «Роль Розетты в борьбе с коронавирусом – Институт дизайна белков» . 21 февраля 2020 года. Архивировано из оригинала 24 декабря 2021 года . Проверено 6 марта 2020 г.
- ^ «Обзор исследований по коронавирусу» . Rosetta@home Официальный Твиттер . 26 июня 2020 года. Архивировано из оригинала 26 июня 2020 года . Проверено 27 июня 2020 г.
- ^ Дело Дж.Б., Чен Р.Э., Цао Л., Ин Б., Винклер Э.С., Джонсон М. и др. (июль 2021 г.). «Ультрапотентные минипротеины, нацеленные на домен, связывающий рецептор SARS-CoV-2, защищают от инфекций и болезней» . Клетка-хозяин и микроб . 29 (7): 1151–1161.e5. дои : 10.1016/j.chom.2021.06.008 . ПМК 8221914 . ПМИД 34192518 .
- ^ Хант А.С., Кейс Дж.Б., Парк Ю.Дж., Цао Л., Ву К., Уоллс А.С. и др. (июль 2021 г.). «Мультивалентные белки защищают от вызывающих беспокойство вариантов SARS-CoV-2» . bioRxiv : 2021.07.07.451375. дои : 10.1101/2021.07.07.451375 . ПМК 8282097 . ПМИД 34268509 .
- ^ «Большие новости от @UWproteindesign: новый кандидат на лечение #COVID19! Все еще необходимы дополнительные лабораторные исследования. Спасибо всем волонтерам, которые помогли собрать данные для этого проекта!!» . Розетта@домашний Твиттер . 9 сентября 2020 года. Архивировано из оригинала 9 сентября 2020 года . Проверено 19 сентября 2020 г.
- ^ «Мини-связующие de novo нацелены на белок шипов SARS-CoV-2» . Бейкер Лаборатория . 9 сентября 2020 г. Архивировано из оригинала 30 сентября 2020 г. Проверено 19 сентября 2020 г.
- ^ Уоллс А.С., Фиала Б., Шефер А., Ренн С., Фам М.Н., Мерфи М. и др. (ноябрь 2020 г.). «Выявление мощных реакций нейтрализующих антител с помощью разработанных вакцин на основе белковых наночастиц против SARS-CoV-2» . Клетка . 183 (5): 1367–1382.e17. дои : 10.1016/j.cell.2020.10.043 . ПМЦ 7604136 . ПМИД 33160446 .
- ^ Хатсон М. (18 сентября 2020 г.). «Ученые продвигаются к одному из Святых Граалей технологий» . Житель Нью-Йорка . Архивировано из оригинала 18 сентября 2020 года . Проверено 19 сентября 2020 г.
- ^ «Neoleukin Therapeutics объявляет о подаче заявки на новое исследуемое лекарственное средство для кандидата на белковую иммунотерапию NL-201 De Novo для лечения рака» . Неолейкин Терапия . 10 декабря 2020 года. Архивировано из оригинала 10 декабря 2020 года . Проверено 10 декабря 2020 г.
- ^ Jump up to: а б Саймонс К.Т., Бонно Р., Ручински И., Бейкер Д. (1999). «Ab initio предсказание структуры белка целей CASP III с использованием Rosetta». Белки . Приложение 3 (S3): 171–76. doi : 10.1002/(SICI)1097-0134(1999)37:3+<171::AID-PROT21>3.0.CO;2-Z . ПМИД 10526365 . S2CID 5774447 .
- ^ «Примечания к выпуску» . Розетта Коммонс . Проверено 21 августа 2023 г.
- ^ "Начиная" . www.rosettacommons.org .
Для большинства целей мы не распространяем исполняемые/бинарные файлы, мы распространяем исходный код.
- ^ «Rosetta: пакет программного обеспечения для молекулярного моделирования» . UW CoMotion – Центр совместных инноваций. Архивировано из оригинала 12 июня 2020 года . Проверено 12 июня 2020 г.
- ^ Jump up to: а б «Способы использования» . РозеттаКоммонс .
- ^ Jump up to: а б «Розетта Коммонс» . RosettaCommons.org. 2008. Архивировано из оригинала 15 сентября 2008 года . Проверено 7 октября 2008 г.
- ^ «Интервью с Дэвидом Бейкером» . Команда Пикарда по распределенным вычислениям. 2006. Архивировано из оригинала 18 февраля 2009 года . Проверено 23 декабря 2008 г.
- ^ Ким Д.Э. (2008). «Rosetta@home: Проблемы с minirosetta версии 1.+ (Сообщение 51199)» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 26 февраля 2009 года . Проверено 7 сентября 2008 г.
- ^ Наули С., Кульман Б., Бейкер Д. (июль 2001 г.). «Компьютерная реконструкция пути сворачивания белка». Структурная биология природы . 8 (7): 602–05. дои : 10.1038/89638 . ПМИД 11427890 . S2CID 18542707 .
- ^ Кульман Б., Дантас Дж., Иретон Г.К., Варани Г., Стоддард Б.Л., Бейкер Д. (ноябрь 2003 г.). «Разработка новой складки глобулярного белка с точностью на атомном уровне». Наука . 302 (5649): 1364–68. Бибкод : 2003Sci...302.1364K . дои : 10.1126/science.1089427 . ПМИД 14631033 . S2CID 1939390 .
- ^ Джонс Д.Т. (ноябрь 2003 г.). «Структурная биология. Учимся говорить на языке белков». Наука . 302 (5649): 1347–48. дои : 10.1126/science.1092492 . ПМИД 14631028 . S2CID 83109899 .
- ^ фон Гротгусс М., Вирвич Л.С., Пас Дж., Рыхлевски Л. (июнь 2004 г.). «Точное предсказание белковых структур». Наука . 304 (5677): 1597–99, ответ автора 1597–99. дои : 10.1126/science.304.5677.1597b . ПМИД 15192202 . S2CID 29787060 .
- ^ «Статьи со ссылкой на: Кульман и др. (2003) «Дизайн новой складки глобулярного белка с точностью на атомном уровне» » . ISI Web of Science . Проверено 10 июля 2008 г.
- ^ «Молекула месяца, октябрь 2005 г.: Дизайнерские белки» . Банк данных белков RCSB. Архивировано из оригинала 28 сентября 2008 года . Проверено 7 сентября 2008 г.
- ^ «Домашняя страница лаборатории Кульмана» . Лаборатория Кульмана . Университет Северной Каролины. Архивировано из оригинала 10 октября 2008 года . Проверено 7 сентября 2008 г.
- ^ «Веб-сервер RosettaDesign» . Лаборатория Кульмана . Университет Северной Каролины. Архивировано из оригинала 13 сентября 2008 года . Проверено 7 сентября 2008 г.
- ^ Jump up to: а б Грей Дж.Дж., Мугон С.Е., Кортемме Т. и др. (июль 2003 г.). «Прогнозы стыковки белков между белками для эксперимента CAPRI». Белки . 52 (1): 118–22. CiteSeerX 10.1.1.80.9354 . дои : 10.1002/прот.10384 . ПМИД 12784377 . S2CID 1186127 .
- ^ Daily MD, Масика Д., Сивасубраманиан А., Сомаруту С., Грей Джей Джей (2005). «Этапы CAPRI 3–5 открывают многообещающие успехи и будущие задачи для RosettaDock» . Белки . 60 (2): 181–86. CiteSeerX 10.1.1.521.9981 . дои : 10.1002/прот.20555 . ПМИД 15981262 . S2CID 21137936 . Архивировано из оригинала 30 июня 2012 года.
- ^ Мендес Р., Лепла Р., Ленсинк М.Ф., Водак С.Дж. (2005). «Оценка прогнозов CAPRI в раундах 3–5 показывает прогресс в процедурах стыковки» . Белки . 60 (2): 150–69. дои : 10.1002/прот.20551 . ПМИД 15981261 . S2CID 24626361 . Архивировано из оригинала 30 июня 2012 года.
- ^ «Сервер RosettaDock» . Розетта Коммонс . Архивировано из оригинала 27 марта 2020 года . Проверено 28 марта 2020 г.
- ^ «Белково-белковая стыковка в Rosetta@home» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 23 июня 2008 года . Проверено 7 сентября 2008 г.
- ^ Jump up to: а б «Робетта» . robetta.bakerlab.org .
Вычислительные ресурсы предоставляются лабораторией Бейкера, исследовательским кампусом Джанелия HHMI, а также волонтерами проекта распределенных вычислений Rosetta@home. Вы можете помочь этому сервису, присоединившись к Rosetta@home.
- ^ Элой П., Старк А., Хэдли С., Рассел Р.Б. (2003). «Прогнозы без шаблонов: новые складки, вторичная структура и контакты в CASP5». Белки . 53 (Приложение 6): 436–56. дои : 10.1002/прот.10546 . ПМИД 14579333 . S2CID 22274928 .
- ^ Тресс М., Эзкурдия И., Гранья О., Лопес Г., Валенсия А. (2005). «Оценка прогнозов, представленных для категории сравнительного моделирования CASP6». Белки . 61 (Приложение 7): 27–45. дои : 10.1002/прот.20720 . ПМИД 16187345 . S2CID 24617067 .
- ^ Бэтти Дж.Н., Копп Дж., Бордоли Л., Рид Р.Дж., Кларк Н.Д., Шведе Т. (2007). «Автоматические прогнозы серверов в CASP7» . Белки . 69 (Приложение 8): 68–82. дои : 10.1002/прот.21761 . ПМИД 17894354 . S2CID 29879391 .
- ^ Чивиан Д., Ким Д.Э., Мальмстрем Л., Шонбрун Дж., Рол К.А., Бейкер Д. (2005). «Прогнозирование структур CASP6 с использованием автоматизированных протоколов Робетты». Белки . 61 (Приложение 7): 157–66. дои : 10.1002/прот.20733 . ПМИД 16187358 . S2CID 8122486 .
- ^ Бейкер Д. «Журнал Дэвида Бейкера Rosetta@home, сообщение 52902» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 23 июня 2008 года . Проверено 7 сентября 2008 г.
- ^ Овчинников С., Ким Д.Е., Ван Р.Ю., Лю Ю., ДиМайо Ф., Бейкер Д. (сентябрь 2016 г.). «Улучшено предсказание структуры de novo в CASP11 за счет включения информации о коэволюции в Rosetta» . Белки . 84 (Приложение 1): 67–75. дои : 10.1002/прот.24974 . ПМЦ 5490371 . ПМИД 26677056 .
- ^ «Серверы» . РозеттаКоммонс .
- ^ Бейкер Д. «Журнал Дэвида Бейкера Rosetta@home (сообщение 52963)» . Форумы Rosetta@home . Университет Вашингтона. Архивировано из оригинала 23 июня 2008 года . Проверено 16 сентября 2008 г.
- ^ «Форумы Foldit: Сколько пользователей у Foldit? И т. д. (сообщение 2)» . Университет Вашингтона. Архивировано из оригинала 22 сентября 2008 года . Проверено 27 сентября 2008 г.
- ^ «Foldit: Часто задаваемые вопросы» . сложить.it . Университет Вашингтона. Архивировано из оригинала 14 сентября 2008 года . Проверено 19 сентября 2008 г.
- ^ Пэк, Минкён; ДиМайо, Фрэнк; Анищенко Иван; Даупарас, Юстас; Овчинников Сергей; Ли, Гю Рие; Ван, Цзюэ; Конг, Цянь; Кинч, Лиза Н.; Шеффер, Р. Дастин; Миллан, Клаудия; Парк, Ханбом; Адамс, Карсон; Глассман, Калеб Р.; ДеДжованни, Энди; Перейра, Хосе Х.; Родригес, Андрия В.; ван Дейк, Альбердина А.; Эбрехт, Ана К.; Опперман, Дидерик Дж.; Сагмайстер, Тео; Бюльхеллер, Кристоф; Павлов-Келлер, Чай; Ратинасвами, Манодж К.; Далвади, Удит; Да, Кэлвин К.; Берк, Джон Э.; Гарсия, К. Кристофер; Гришин Ник В.; Адамс, Пол Д.; Прочтите, Рэнди Дж.; Бейкер, Дэвид (20 августа 2021 г.). «Точное предсказание белковых структур и взаимодействий с помощью трехдорожечной нейронной сети» . Наука . 373 (6557): 871–876. Бибкод : 2021Sci...373..871B . дои : 10.1126/science.abj8754 . ПМЦ 7612213 . ПМИД 34282049 .
- ^ Пэн, З; Ван, В; Хан, Р; Чжан, Ф; Ян, Дж. (декабрь 2022 г.). «Предсказание структуры белка в эпоху глубокого обучения». Современное мнение в области структурной биологии . 77 : 102495. doi : 10.1016/j.sbi.2022.102495 . ПМИД 36371845 . S2CID 253470306 .
- ^ Ду, Цзунян; Ван, Вэнькай; Вэй, Хун; Пэн, Чжэньлин, Иван; Бейкер, Ян, Цзяньи (декабрь 2021 г.) «Сервер trRosetta» . предсказание» . Протоколы природы . 16 (12): 5634–5651. : 10.1038 /s41596-021-00628-9 . PMID 34759384. . S2CID 243987300 doi
- ^ «Список проектов – BOINC» . Калифорнийский университет. Архивировано из оригинала 8 сентября 2008 года . Проверено 8 сентября 2008 г.
- ^ Панде Групп (2010). «Часто задаваемые вопросы о высокой производительности» . Стэнфордский университет . Архивировано из оригинала (FAQ) 19 августа 2012 года . Проверено 19 сентября 2011 г.
- ^ 7im (2 апреля 2010 г.). «Re: Ответы на вопрос: Причины не использования F@H» . Архивировано из оригинала 24 августа 2021 года . Проверено 19 сентября 2011 г.
{{cite web}}: CS1 maint: числовые имена: список авторов ( ссылка ) - ^ Виджай Панде (5 августа 2011 г.). «Обновлена страница результатов – опубликован новый ключевой результат в нашей работе по болезни Альцгеймера» . Архивировано из оригинала 15 сентября 2011 года . Проверено 19 сентября 2011 г.
- ^ Группа Панде. «Часто задаваемые вопросы по изучению болезней Folding@home» . Стэнфордский университет . Архивировано из оригинала (FAQ) 11 октября 2007 года . Проверено 12 сентября 2011 г.
- ^ Виджай Панде (26 сентября 2007 г.). «Как работает ФАГ: Молекулярная динамика» . Архивировано из оригинала 26 сентября 2011 года . Проверено 10 сентября 2011 г.
- ^ Jump up to: а б tjlane (9 июня 2011 г.). «Re: Курс приготовления зернистого протеина менее чем за 10 минут» . Архивировано из оригинала 11 ноября 2012 года . Проверено 19 сентября 2011 г.
- ^ jmn (29 июля 2011 г.). «Rosetta@home и Folding@home: дополнительные проекты» . Архивировано из оригинала 23 сентября 2011 года . Проверено 19 сентября 2011 г.
- ^ Группа Панде. «Статистика клиентов по ОС» . Стэнфордский университет. Архивировано из оригинала 13 мая 2013 года . Проверено 18 октября 2011 г.
- ^ Jump up to: а б «Rosetta@home: Кредитный обзор» . boincstats.com. Архивировано из оригинала 21 апреля 2020 года . Проверено 28 марта 2020 г.
- ^ Мальмстрем Л., Риффл М., Штраус К.Э. и др. (апрель 2007 г.). «Назначение суперсемейства протеома дрожжей посредством интеграции предсказания структуры с онтологией гена» . ПЛОС Биология . 5 (4): е76. дои : 10.1371/journal.pbio.0050076 . ПМЦ 1828141 . ПМИД 17373854 .
- ^ Бонно Р. (2006). «Сообщения на доске объявлений всемирного сообщества: переход HPF -> HPF2» . Лаборатория Бонно, Нью-Йоркский университет. Архивировано из оригинала 29 августа 2008 года . Проверено 7 сентября 2008 г.
- ^ «Список публикаций Ришара Бонно» . Лаборатория Бонно, Нью-Йоркский университет. Архивировано из оригинала 7 июля 2008 года . Проверено 7 сентября 2008 г.
- ^ Бонно Р. «Сообщения на доске объявлений в сети мирового сообщества» . Лаборатория Бонно, Нью-Йоркский университет. Архивировано из оригинала 4 июля 2008 года . Проверено 7 сентября 2008 г.
- ^ «Predictor@home: Разработка новых областей применения P@H» . Исследовательская группа Брукса . Проверено 7 сентября 2008 г. [ мертвая ссылка ]
- ^ Каррильо-Трипп М (2007). «дТАССЕР» . Исследовательский институт Скриппса. Архивировано из оригинала 6 июля 2007 года . Проверено 7 сентября 2008 г.
- ^ «РАЛЬФ@домашний сайт» . Форумы RALPH@home . Университет Вашингтона. Архивировано из оригинала 13 января 2016 года . Проверено 7 сентября 2008 г.
- ^ «Розетта@дома» . Архивировано из оригинала 3 января 2016 года . Проверено 19 марта 2020 г.
- ^ «Rosetta@home: объяснение новой кредитной системы» . Форумы Rosetta@home . Университет Вашингтона. 2006. Архивировано из оригинала 2 февраля 2009 года . Проверено 8 октября 2008 г.
- ^ «BOINCstats: Сравнение кредитоспособности проектов» . boincstats.com. 2008. Архивировано из оригинала 13 сентября 2008 года . Проверено 8 октября 2008 г.
- ^ «Кредит разделен на проекты» . boincstats.com. Архивировано из оригинала 20 февраля 2015 года . Проверено 19 февраля 2015 г.
- ^ «Rosetta@home: Архив предсказателя дня» . Розетта@дома . Университет Вашингтона. 2008. Архивировано из оригинала 24 сентября 2008 года . Проверено 8 октября 2008 г.
- ^ «Rosetta@home: сворачивание, дизайн и стыковка белков» . Розетта@дома . Университет Вашингтона. 2008. Архивировано из оригинала 28 февраля 2011 года . Проверено 8 октября 2008 г.
Внешние ссылки
[ редактировать ]- Официальный сайт
- Бейкер Лаборатория Бейкер Веб-сайт лаборатории
- Журнал Дэвида Бейкера Rosetta@home
- BOINC Включает обзор платформы и руководство по установке BOINC и прикреплению к Rosetta@home.
- BOINCstats – Rosetta@home Подробная статистика вкладов
- RALPH@home Веб-сайт проекта альфа-тестирования Rosetta@home
- Видео Rosetta@home на YouTube. Обзор Rosetta@home, предоставленный Дэвидом Бейкером и членами лаборатории.
- Академическое сотрудничество Rosetta Commons для разработки платформы Rosetta
- Канон Розетты , список знаковых документов в развитии Розетты.
- Веб-страница лаборатории Кульмана , дом RosettaDesign
Онлайн-сервисы Rosetta
- Rosetta Commons Список доступных серверов
- Robetta Сервер прогнозирования структуры белка
- ROSIE Стыковка, проектирование и т.п. многофункциональный серверный набор
- RosettaDesign Protein Сервер дизайна
- RosettaBackrub Гибкая магистральная сеть/сервер проектирования белков