Визуализация биологических данных
Визуализация биологических данных — раздел биоинформатики, занимающийся применением компьютерной графики , научной визуализации и визуализации информации в различных областях наук о жизни . Это включает в себя визуализацию последовательностей , геномов , выравниваний , филогений , макромолекулярных структур , системной биологии , микроскопии и магнитно-резонансной томографии данных . Программные инструменты, используемые для визуализации биологических данных, варьируются от простых автономных программ до сложных интегрированных систем.
Новая тенденция — стирание границ между визуализацией 3D-структур с атомным разрешением, визуализацией более крупных комплексов с помощью криоэлектронной микроскопии и визуализацией расположения белков и комплексов внутри целых клеток и тканей. [1] [2] Также возросла доступность и важность данных с временным разрешением из системной биологии , электронной микроскопии и визуализации клеток и тканей. [3] [4]
Выравнивание последовательности [ править ]

Визуализация выравнивания последовательностей играет решающую роль в биоинформатике и геномике , позволяя исследователям эффективно интерпретировать и анализировать сложные генетические данные. Визуализация выравнивания последовательностей позволяет идентифицировать сходства, различия, консервативные области и закономерности эволюции в последовательностях ДНК или белков , помогая понять генетические взаимоотношения, функциональные элементы и эволюционные процессы. Визуализация выравнивания последовательностей необходима по нескольким причинам:
Идентификация консервативной последовательности . Визуализация помогает исследователям идентифицировать консервативные области последовательностей, которые указывают на функциональную важность или эволюционные связи. [5]
Обнаружение мутаций и вариаций. Инструменты визуализации позволяют обнаруживать мутации , вставки, делеции и другие вариации в последовательностях, обеспечивая понимание генетического разнообразия и мутаций, вызывающих заболевания. [6]
Понимание эволюционных связей. Визуализируя выравнивание последовательностей, исследователи могут делать выводы об эволюционных связях, строить филогенетические деревья и изучать историю эволюции видов или генов. [7]
Прогнозирование функциональных элементов. Визуализация помогает прогнозировать функциональные элементы, такие как белковые домены, мотивы и регуляторные области внутри последовательностей, что облегчает исследования функциональной геномики. [8]

Сравнение геномов : сравнительная геномика опирается на выравнивания последовательностей визуализацию для сравнения геномов , идентификации ортологичных и паралогичных генов и изучения эволюции генома у разных видов. [9] Чтобы визуализировать выравнивание последовательностей и их особенности, исследователи часто полагаются на популярные программные инструменты биоинформатики, такие как Clustal Omega , MUSCLE , T-Coffee и MAFFT . Эти инструменты предоставляют интерактивные платформы для выравнивания последовательностей, выделения консервативных областей, отображения вариаций последовательностей и идентификации мотивов последовательностей. Кроме того, программное обеспечение для визуализации, такое как Jalview , BioEdit и Geneious, предлагает расширенные функции для визуализации и анализа выравнивания последовательностей, что упрощает исследователям интерпретацию и извлечение значимой информации из генетических данных.
Техники
Помимо программных инструментов, таких как Clustal Omega , MUSCLE , T-Coffee и MAFFT , существует несколько популярных методов визуализации выравнивания геномных последовательностей, которые играют решающую роль, помогая исследователям понять родовые отношения, функциональные элементы и эволюционные процессы. Среди популярных инструментов распространенные методы визуализации выравнивания последовательностей включают:

Логотип последовательности . Логотипы последовательностей представляют собой графическое изображение выравнивания последовательностей, которое отображает сохранение остатков в каждом положении, а также относительную частоту каждой аминокислоты или нуклеотида . Логотипы последовательностей обеспечивают компактную и информативную визуализацию консервативной последовательности и изменчивости. [10]
Множественное выравнивание последовательностей . Множественные средства просмотра выравнивания последовательностей, такие как Jalview и MEGA , предоставляют интерактивные платформы для визуализации и анализа множественного выравнивания последовательностей . Эти инструменты предлагают функции для выделения консервативных областей последовательностей, идентификации мотивов и изучения эволюционных связей внутри последовательностей. [11]

Инструменты выравнивания структуры белка: такие инструменты, как PyMOL и UCSF Chimera, позволяют визуализировать выравнивание последовательностей в контексте белковых структур. Накладывая выровненные последовательности на белковые структуры , исследователи могут анализировать пространственное расположение консервативных остатков и функциональных доменов. [12]
Визуализация филогенетического дерева. Инструменты визуализации филогенетического дерева, такие как FigTree и iTOL , позволяют исследователям визуализировать эволюционные отношения, выведенные на основе выравнивания последовательностей. Эти инструменты обеспечивают интерактивное отображение филогенетических деревьев, выделяя длину ветвей, значения поддержки узлов и эволюционные расстояния. [13]
Браузер генома. Браузеры генома, такие как UCSC Genome Browser и Ensembl, предоставляют комплексные платформы для визуализации выравнивания последовательностей во всех геномах. Исследователи могут изучать аннотации ДНК , регуляторные элементы и данные сравнительной геномики в контексте последовательностей генома. [14]

Приложения
Визуализация выравнивания геномных последовательностей используется в различных приложениях, играя решающую роль в различных областях геномики и биоинформатики , позволяя исследователям анализировать, интерпретировать и извлекать ценную информацию из генетических данных. Приложения визуализации выравнивания последовательностей разнообразны и охватывают широкий спектр областей исследований. Некоторые ключевые приложения включают в себя:
Сравнительная геномика . Визуализация выравнивания последовательностей необходима для исследований сравнительной геномики, где исследователи сравнивают генетические последовательности разных видов для выявления эволюционных связей, консервативных областей последовательностей и функциональных элементов. Инструменты визуализации помогают обнаруживать сходства и различия между геномами , помогая изучать эволюционные процессы. [15]
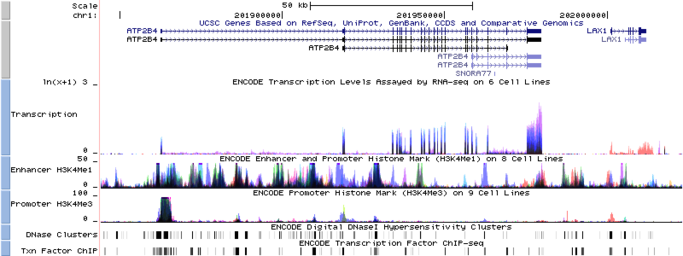
Вариантный анализ. В области генетики и медицины персонализированной визуализация выравнивания последовательностей используется для вариантного анализа для выявления однонуклеотидных полиморфизмов (SNP), вставок, делеций и других генетических вариаций . Инструменты визуализации помогают исследователям точно определить конкретные изменения в геномных последовательностях и оценить их потенциальное влияние на фенотипические признаки. [16]
Филогенетический анализ. Филогенетические исследования основаны на выравнивания последовательностей визуализации для построения филогенетических деревьев и анализа генетических связей между видами или популяциями . Инструменты визуализации позволяют исследователям визуализировать сходство последовательностей, рассчитывать эволюционные расстояния и делать выводы о филогенетических отношениях на основе выравнивания последовательностей. [17]
Функциональная геномика . В исследованиях функциональной геномики визуализация выравнивания последовательностей используется для изучения экспрессии генов , регуляторных элементов и белок-белковых взаимодействий . Визуализируя выравнивание последовательностей в контексте функциональных аннотаций и генных сетей, исследователи могут выяснить биологические функции и регуляторные механизмы генов. [18]
Структурная биоинформатика . Визуализация выравнивания последовательностей является неотъемлемой частью структурной биоинформатики , где исследователи анализируют белковые последовательности и структуры, чтобы понять их трехмерную организацию и функциональные свойства. Инструменты визуализации помогают выравнивать белковые последовательности, прогнозировать структурные мотивы и исследовать белок-белковые взаимодействия . [19]
Макромолекулярный [ править ]
Визуализация макромолекул имеет решающее значение для сложного понимания многогранных структур и функций, которые являются фундаментальными для биологических систем. Замечательный прогресс был достигнут в трехмерном изображении таких макромолекул, охватывающих углеводы, белки, нуклеиновые кислоты и их комплексы. Недавние достижения в методологиях визуализации ускорили квантовый скачок в нашей способности различать тонкости биологических данных. Эти сложные визуализации обеспечивают беспрецедентный уровень ясности и детализации, тем самым улучшая наше понимание механистических основ, управляющих поведением и взаимодействием биологических объектов.
Техники

Сегментация улучшает интерпретацию биологических изображений, а автоматизированные инструменты улучшают анализ данных. Это привело к росту популярности веб-визуализации для 3D-сегментации. Сегментация играет жизненно важную роль в расшифровке данных биологических изображений. Появление сложных технологий автоматизированной сегментации, а также их включение в общедоступные хранилища данных изображений значительно расширяет процесс интерпретации. [20]
Объемная визуализация выявляет внутренние макромолекулярные структуры без сегментации, обеспечивая неинвазивный вид внутри молекул.
Интеграция экспериментальных данных в визуализации, например наложение мутаций или данных связывания, дает более глубокое понимание. Это может отображаться в виде тепловых карт или градиентов молекулы, что жизненно важно для управления растущей сложностью биомолекулярных данных. [21]
Интерактивная 3D-визуализация предлагает практическое взаимодействие с макромолекулами, позволяя выполнять такие манипуляции, как вращение и масштабирование, что улучшает понимание.

Виртуальная реальность и дополненная реальность представляют собой иммерсивные методы взаимодействия с макромолекулами, обеспечивая трехмерную перспективу, с которой не могут сравниться экранные инструменты. Приложение AR также разработано, чтобы помочь учащимся визуализировать трехмерные макромолекулярные структуры и взаимодействовать с ними, устраняя ограничения традиционных двумерных изображений в передаче пространственных деталей и восприятии глубины. [22]
Анимация молекулярной активности иллюстрирует динамическое поведение биомолекул и служит мощным образовательным и исследовательским инструментом. Используя технологию игрового движка Unity3D, этот подход демократизирует создание интерактивных инструментов молекулярной визуализации, в результате чего создается удобная платформа, упрощающая представление сложных биологических данных. [23]
Высокопроизводительная компьютерная визуализация позволяет визуализировать в режиме реального времени большие и сложные наборы данных, что необходимо для расширенного макромолекулярного анализа. Программное обеспечение, использующее высокопроизводительные вычисления, динамически и эффективно анализирует взаимодействие лекарственного средства с рецептором посредством молекулярно-динамического моделирования, предлагая глубокую информацию и прогнозы об эффективности лекарств, а также облегчая визуализацию. [24]
Методы гибридной визуализации объединяют различные методы, чтобы обеспечить многогранное представление о молекулах, сочетая детальное положение атомов с целостным пониманием структуры и объема.
Визуализация в различных типах макромолекул

углеводов Визуализация
Визуализации углеводсвязывающего модуля (CBM) целлюлазы исследуют его взаимодействие с целлюлозой во время гидролиза с трех точек зрения: адсорбция CBM на целлюлозе, ее пространственное занятие и доступность поверхности целлюлозы для CBM.

белков Визуализация
Банк данных белков RCSB (RCSB PDB), поддерживаемый крупными научными агентствами США, стал ключевым ресурсом для структурных биологов во всем мире и действует как центр данных США в рамках партнерства Всемирного банка данных белков (wwPDB). В качестве назначенного хранителя архива RCSB PDB обеспечивает безопасность данных PDB и ежегодно обслуживает десятки тысяч депозиторов данных на всех населенных континентах, используя различные методы структурного определения. Веб-портал RCSB.org предоставляет неограниченный доступ к данным PDB миллионам людей по всему миру. В этой статье подробно описывается рост и эволюция архива с развитием экспериментальных методов, решающая роль стандартов данных и интеграции, а также внедрение новых инструментов и функций для трехмерного структурного анализа и визуализации за последний год. [25]
нуклеиновой кислоты Визуализация
Исследователи разработали быстрый, простой и точный метод обнаружения вируса инфекционного ринотрахеита крупного рогатого скота (IBRV) у крупного рогатого скота — вируса, известного тем, что он вызывает хронические инфекции и оказывает существенное экономическое воздействие. Этот метод объединяет амплификацию рекомбинантной полимеразы (RPA) с полосой визуализации вертикального потока (VF) для формирования анализа RPA-VF, который нацелен на ген тимидинкиназы, обеспечивая быстрое обнаружение, высокую специфичность и нулевую перекрестную реактивность с другими патогенами. [26]
Большие неполимерные молекулы
Визуализация наноразмерных материалов имеет решающее значение для понимания их структурно-функциональных взаимосвязей и обычно требует передовых микроскопических и аналитических методов, которые обеспечивают изображения с высоким разрешением и большим увеличением.

Наночастицы — это крошечные частицы размером от 1 до 100 нанометров. Благодаря небольшому размеру и высокому соотношению площади поверхности к объему они обладают уникальными химическими и физическими свойствами. Визуализация наночастиц обычно достигается с использованием методов высокого разрешения, таких как просвечивающая электронная микроскопия (ПЭМ), сканирующая электронная микроскопия (СЭМ), атомно-силовая микроскопия (АСМ) и динамическое рассеяние света (ДРС) для анализа распределения по размерам. [27] [28]

Нанокомпозиты — это материалы, которые включают наночастицы в матрицу другого материала, например полимеров, керамики или металлов. Эти композиты часто обладают улучшенными свойствами, такими как повышенная прочность или электропроводность. Визуализация распределения и взаимодействия наночастиц внутри матрицы может быть осуществлена с использованием таких методов, как TEM, SEM и рентгеновская дифракция (XRD).

Нанотрубки , в частности углеродные нанотрубки (УНТ), представляют собой цилиндрические структуры диаметром всего 1 нанометр. Они обладают замечательными механическими, электрическими и термическими свойствами и используются в различных приложениях: от материаловедения до нанотехнологий. Для визуализации нанотрубок обычно требуется ПЭМ, СЭМ или АСМ.

Нановолокна — это волокна диаметром в нанометровом масштабе. Они создаются с помощью таких процессов, как электропрядение, и находят применение в таких областях, как фильтрация, текстильная промышленность и биомедицина. Нановолокна можно визуализировать с помощью SEM, который дает подробные изображения их морфологии и распределения.
Раздел визуализации крупных неполимерных молекул демонстрирует всестороннее и четкое описание методов, используемых для изучения наноразмерных материалов. В нем подробно описывается применение передовых методов микроскопии, таких как TEM, SEM, AFM и XRD, а также их применимость к конкретным наноматериалам, таким как наночастицы мезопористого кремнезема, нанокомпозиты, углеродные нанотрубки и нановолокна. Каждый материал контекстуализируется в контексте его промышленного или биомедицинского применения, что подчеркивает важность этих методов визуализации для понимания свойств и поведения материала. Хотя раздел информативен и технически подробен, его можно улучшить, включив в него конкретные примеры результатов визуализации, обсудив ограничения существующих методов и, возможно, представив новые методы, чтобы обеспечить более всестороннее представление о данной области. В целом, описание эффективно предназначено для обучения и информирования о решающей роли визуализации в нанотехнологиях.
Визуализируйте взаимодействия между макромолекулами
Взаимодействия белок-углеводы визуализировались атомами водорода в пердейтерированном лектин-фукозном комплексе. [29] Вычислительная стыковка играет жизненно важную роль в структурной биологии, поскольку программное обеспечение обеспечивает удобную веб-платформу для моделирования различных макромолекулярных взаимодействий, таких как гибкие комплексы и мембранно-ассоциированные сборки. Это повышает доступность и обогащает опыт пользователей в сообществе структурной биологии. [30]
Инструменты
PyMOL, Chimera, ChimeraX, Jmol, VMD, Swiss-PdbViewer, Coot, Biovia Discovery Studio, LightDock и Schrodinger's Maestro — ключевые инструменты молекулярной визуализации, каждый из которых предлагает уникальные возможности, начиная от высококачественных 3D-изображений и интерактивного анализа и заканчивая поддержкой виртуальной реальности. и крупномасштабное моделирование, удовлетворяющее разнообразные потребности в молекулярном моделировании, публикациях и образовании как на платформах с открытым исходным кодом, так и на коммерческих платформах.
Системная биология

Системная биология — это раздел визуализации биологических данных, посвященный анализу и моделированию сложных биологических систем. Популярные вычислительные модели, используемые в системной биологии, включают исчисление процессов , такое как стохастическое π-исчисление , и реконструкцию и анализ на основе ограничений (COBRA), парадигму, которая учитывает физические, ферментативные и топологические ограничения, лежащие в основе фенотипа в метаболической сети . [32] [33]
Большая часть визуализации данных в системной биологии выполняется с использованием математически созданных моделей. Исследователи составят схему всех белков, генов или метаболических путей в данной биологической системе, а затем определят скорость реакций в этой системе, используя кинетику действия масс или кинетику ферментов . Эти значения используются в качестве параметров для построения дифференциальных уравнений , представляющих систему, которые затем можно использовать для определения поведения объектов внутри этой системы. Также существуют альтернативные решения математического моделирования; например, метод COBRA, такой как анализ баланса потоков, можно использовать для анализа потока метаболитов через определенную метаболическую сеть. [34]
Еще одним ключевым методом визуализации в системной биологии является масс-спектрометрия , которую можно использовать для визуализации пространственного распределения соединений, биомаркеров, метаболитов, пептидов и/или белков в организме. Это особенно полезно в метаболомике , разделе системной биологии, который использует масс-спектрометрию для измерения информации о распределении метаболитов, а затем использует измеренную интенсивность для построения изображения. [35]
Популярные программные инструменты, используемые в моделировании системной биологии, включают MassPy , Cytosim и PySB . Дополнительные примеры можно найти в списке программного обеспечения для моделирования системной биологии в Википедии .
Визуализация под микроскопом [ править ]
Помимо оптической и электронной микроскопии, при визуализации также используются другие методы, такие как сканирующий зонд, ультрафиолет, инфракрасный свет, цифровая голография, лазер и любительская микроскопия.

Новые подходыВ исследовании изучается использование двухфотонной микроскопии, метода, позволяющего получать изображения на глубине до 800 мкм за счет двухфотонного поглощения, для визуализации микророботических агентов под биологической тканью, демонстрируя ее преобразующий потенциал для применения в микроробототехнике как in vitro, так и in vivo. [36]
Исследователи использовали световую микроскопию яркого поля с высокоинтенсивным импульсным светодиодным освещением для получения детальных изображений живых клеток с разрешением 12 бит на канал, устраняя искажения данных, вызванные взаимодействием оптических путей и аномалиями датчиков, с помощью комплексного подхода спектроскопической калибровки, позволяющего визуализировать с минимальной потерей информации при глубине интенсивности 8 бит. [37]
Исследователи изучили инициативу сообщества, направленную на улучшение изображения данных световой микроскопии в научных публикациях путем соблюдения «Принципов FAIR Data», которые направлены на улучшение находимости, доступности, совместимости и воспроизводимости данных. Несмотря на постоянные проблемы, связанные с качеством данных и передачей данных, инициатива подчеркивает роль глобального научного сотрудничества в продвижении стандартов визуализации и использует исторические знания для направления и продвижения будущих достижений в области биологических изображений. [38]
Магнитно-резонансная томография [ править ]

Магнитно-резонансная томография (МРТ) — распространенная форма визуализации биологических данных, используемая для формирования изображений внутренних биологических процессов. Различные настройки радиочастотных импульсов и градиентов приводят к различному внешнему виду изображения; эти комбинации известны как последовательности МРТ . Особенно примечательным подразделом МРТ является магнитно-резонансная ангиография , которая представляет собой группу методов, используемых для визуализации артерий и вен. Возможности МРТ для визуализации дополнительно расширяются за счет диффузионной МРТ и функциональной МРТ , которые можно использовать для захвата нейронных путей и кровотока соответственно.
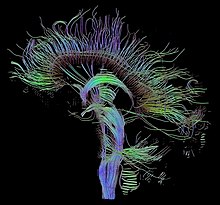
Диффузионная МРТ дополнительно опирается на визуализацию тензора диффузии (DTI), которая измеряет диффузию и направленность молекул воды, а также визуализацию базисного спектра диффузии (DBSI), которая извлекает несколько анизотропных и изотропных тензоров диффузии. [39] [40] Функциональная МРТ основана на контрасте, зависящем от уровня кислорода в крови (ЖИРНЫЙ), который измеряет долю насыщенного кислородом гемоглобина в определенных областях мозга; это позволяет ему измерять и моделировать активность мозга на основе кровотока. [41] Дополнительные методы МРТ включают импульсы насыщения (используются для уменьшения артефактов движения), градиентное эхо (например, динамическое усиление контраста), спиновое эхо и диффузионное взвешивание (метод генерации контраста сигнала, основанный на различиях в броуновском движении ). [42] [43] [44]
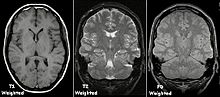
Чтобы создать наблюдаемое изображение с помощью МРТ, цель помещают в мощное магнитное поле, например, в аппарате МРТ. Это приводит к тому, что оси протонов водорода внутри мишени, которые обычно выстраиваются случайным образом в соответствии с равновесием, выстраиваются в одном направлении, создавая магнитный вектор, ориентированный вдоль оси магнита. Эта ориентация также позволяет измерить спин или частоту вращения протонов водорода. Затем выравнивание нарушается с помощью радиочастотных (РЧ) импульсов (РЧ является разновидностью неионизирующего электромагнитного излучения). [45] Когда магнитное поле удаляется, протоны водорода возвращаются в свои равновесные состояния в процессе, известном как релаксация , и при этом они излучают радиочастотную энергию. [46] Различные ткани расслабляются с разной скоростью, что позволяет ученым использовать определенные последовательности радиочастотных импульсов, чтобы подчеркнуть определенные ткани или аномалии.
Через некоторое время после радиочастотного импульса измеряются радиочастотные энергетические сигналы, излучаемые протонами, для получения информации о частоте из каждого места в плоскости изображения. Затем используется преобразование Фурье для преобразования этой частотной информации в уровни интенсивности, которые отображаются в сгенерированном изображении в виде оттенков серого.

В общем, измеряются два аспекта процесса релаксации: время, необходимое магнитному вектору для возвращения в состояние покоя (также известное как T 1 или спин-решеточная релаксация ), и время, необходимое для осевого спина протонов водорода. вернуться в состояние покоя (также известное как Т 2 или спин-спиновая релаксация ). [47] Чтобы создать Т 1- взвешенное изображение, МР-сигнал измеряется путем изменения промежутка времени между РЧ-импульсами (также известного как время повторения или TR). Для создания Т 2 -взвешенного изображения МР-сигнал измеряется путем изменения времени между подачей РЧ-импульса и получением РЧ-энергетических сигналов от протонов водорода (также известное как время до эха , или TE). Преобладающие интенсивности сигнала при взвешивании изображения T 1 — это жидкость (черный из-за низкой интенсивности), мышечный (серый из-за промежуточной интенсивности сигнала) и жир (белый из-за высокой интенсивности сигнала). Подавление жира применяется ко многим последовательностям, взвешенным по Т 1, для подавления яркости создаваемого им сигнала. Доминирующими интенсивностями сигнала взвешивания изображения Т 2 являются жидкость (белый), мышечная (серый) и жир (белый). Сигналы Т 2 также часто усиливаются или подавляются в зависимости от цели визуализации; Яркие примеры включают подавление жира, ослабление жидкости и взвешивание восприимчивости.
Также следует отметить взвешенные изображения по протонной плотности (PD), которые генерируются с использованием длинного TR и короткого TE. ПД полезен для дифференциации жидкости, гиалинового хряща и фиброзного хряща , что делает его идеальным для визуализации суставов. За пределами визуализации суставов он в значительной степени был заменен инверсионным восстановлением с ослаблением жидкости (FLAIR), последовательностью инверсионного восстановления , которая удаляет сигнал из спинномозговой жидкости. [48]
Томография [ править ]
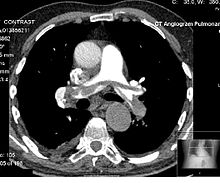

Компьютерная томография (КТ) и позитронно-эмиссионная томография (ПЭТ) аналогичны МРТ, но основаны на других методах визуализации (рентгеновское излучение и ионизирующее излучение соответственно). Вариант КТ, известный как контрастная КТ, также требует, чтобы субъект принимал контрастное вещество, называемое радиоконтрастом (обычно путем перорального приема, клизмы или инъекции). Положительные радиоконтрастные вещества, такие как сульфат бария, увеличивают ослабление рентгеновских лучей в организме, в результате чего содержащая их ткань становится более белой на рентгеновском изображении. Между тем, негативные агенты, такие как углекислый газ, позволяют рентгеновским лучам легко проходить сквозь них, в результате чего содержащие их ткани кажутся темнее. [49]
Как и магнитно-резонансная томография, при компьютерной томографии используются многочисленные методы для отображения и измерения данных, в том числе последовательная КТ (когда стол КТ перемещается от места к месту), спиральная КТ (когда вся рентгеновская трубка вращается вокруг объекта) и электронная КТ . лучевая томография (где с помощью отклоняющих катушек вращаются только пути электронов). ПЭТ-сканеры не имеют такого большого разнообразия аппаратного обеспечения и вместо этого используют разные радиоиндикаторы в зависимости от цели визуализации. Обратите внимание, что радиофармпрепараты отличаются от радиоконтрастов; первый полагается на радиоактивный распад, чтобы проследить свой путь, тогда как второй поглощается конкретной тканью и влияет на ослабление рентгеновского излучения в этой ткани. Поскольку эти методы не являются взаимоисключающими, ПЭТ и КТ можно выполнять одновременно с помощью сканеров ПЭТ-КТ, которые используются для большинства современных ПЭТ-сканирований. [50]
Любой или оба этих метода можно использовать в сочетании с проекцией максимальной интенсивности (MIP) для преобразования данных сканирования в трехмерное изображение. Этого может быть трудно добиться из-за артефактов, создаваемых дыханием и кровотоком, которые неподготовленному глазу могут показаться аномалиями; однако отличить эти артефакты от настоящей болезни можно, если им уделять пристальное внимание. [51] При правильном выполнении КТ и ПЭТ, выполненные с помощью MIP, отлично подходят для выявления небольших аномальных разрастаний тканей, особенно в легких. Сканирования, сделанные с этой целью с помощью MIP, как правило, имеют более высокую значимость, чем усредненные изображения, созданные с помощью традиционной КТ. [52]
MIP-визуализация также используется с магнитно-резонансной ангиографией, и исследования показали, что ее можно эффективно использовать с МРТ. [53] По крайней мере, одно исследование показало, что MIP-МРТ на самом деле значительно превосходит односрезовую МРТ при использовании нейронными сетями для классификации поражений на основе злокачественности. [54]
Выравнивание [ править ]
Выравнивание последовательностей — это способ упорядочения последовательностей белка, РНК или ДНК для выявления областей сходства, которые могут быть следствием функциональных, структурных или эволюционных взаимоотношений между последовательностями. Эта концепция изначально сравнивает только две такие последовательности в так называемом попарном выравнивании.Глобальные выравнивания, которые пытаются выровнять каждый остаток в каждой последовательности, наиболее полезны, когда последовательности в наборе запроса похожи и имеют примерно одинаковый размер. Локальные выравнивания более полезны для несходных последовательностей, которые предположительно содержат области сходства или сходные мотивы последовательностей в пределах их более широкого контекста последовательности. Множественное выравнивание последовательностей является расширением парного выравнивания, позволяющим включать более двух последовательностей одновременно. Множественные методы выравнивания пытаются выровнять все последовательности в каждом наборе запросов. Множественные выравнивания часто используются для идентификации консервативных областей последовательностей в группе последовательностей, предположительно связанных эволюционно.
Цели визуализации выравнивания:
- Помогите общему пониманию крупномасштабных выравниваний ДНК или белков. При анализе данных полезно каким-то образом визуализировать их, чтобы иметь возможность легко выявить четкие закономерности или взаимосвязи.
- Визуализируйте выравнивание рисунков и публикаций. Он суммирует множественное выравнивание последовательностей в удобной для восприятия форме.
- Вручную редактируйте и контролируйте автоматически созданные трассы. Несмотря на то, что существуют эффективные алгоритмы, ни один из них не идеален, а инструменты визуализации позволяют редактировать небольшие несоответствия.

Обычное множественное выравнивание последовательностей . Выровненные последовательности нуклеотидных или аминокислотных остатков обычно представляются в виде строк в матрице. Между остатками вставляются пробелы, чтобы одинаковые или похожие символы располагались в последовательных столбцах. Многие программы визуализации последовательностей также используют цвет для отображения информации о свойствах отдельных элементов последовательности; в последовательностях ДНК и РНК это соответствует присвоению каждому нуклеотиду своего цвета. При выравнивании белков цвет часто используется для обозначения свойств аминокислот, чтобы помочь судить о сохранении данной аминокислотной замены. Для нескольких последовательностей последняя строка в каждом столбце часто является консенсусной последовательностью, определяемой выравниванием; консенсусная последовательность также часто представляется в графическом формате с логотипом последовательности, в котором размер каждой буквы нуклеотида или аминокислоты соответствует степени ее консервативности.

Круговое множественное выравнивание последовательностей . Распространенным предположением о методах множественного выравнивания последовательностей является то, что крайние левая и правая позиции входных последовательностей имеют отношение к выравниванию. Однако позиция, в которой начинается или заканчивается последовательность, может быть совершенно произвольной. Например, при линеаризации круговой молекулярной структуры начало последовательности выбирается случайным образом. Это актуально, например, в процессе множественного выравнивания последовательностей митохондриальной ДНК, вироидных, вирусных или других геномов, имеющих кольцевую молекулярную структуру.

Спиральное выравнивание множественных последовательностей . Цвет используется для отображения информации о свойствах отдельных элементов последовательности. Также могут быть пробелы, благодаря которым последовательности лучше сочетаются друг с другом. Таким образом, топология спирального выравнивания последовательностей эквивалентна стандартной линейной матрице с тем преимуществом, что она практически суммирует очень длинные последовательности. Это означает, что каждая отдельная спираль представляет собой одну из выравниваемых последовательностей.

3D-визуализация . Обычное одномерное представление последовательности белка представляет собой список аминокислот, которые ее образуют. Однако трехмерное выравнивание показывает, как последовательности могут соответствовать друг другу. Средство просмотра группового выравнивания 1D-3D из банка данных белков RCSD поддерживает исследование множественного выравнивания последовательностей (MSA) на уровнях последовательностей и структур для экспериментальных структур PDB и моделей вычислительной структуры (CSM). Можно выбрать белки и/или области остатков из MSA, чтобы просмотреть их выровненные трехмерные структуры.
RCSB.org кластеризует белковые объекты (экспериментальные структуры PDB и CSM) по порогу идентичности последовательностей и присоединению UniProt. Для каждого кластера MSA рассчитывается с использованием Clustal Omega и отображается в средстве просмотра группового выравнивания 1D-3D с использованием определенных цветовых схем. Положения последовательности белка PDB представлены синим цветом, если остаток был определен экспериментально, и серым цветом, если нет. CSM окрашены в соответствии с их местными показателями pLDDT. [55]
Филогения [ править ]

Филогенетическое дерево — это диаграмма ветвления или дерево, показывающее эволюционные отношения между различными биологическими видами или другими объектами, основанные на сходстве и различиях в их физических или генетических характеристиках. Это визуальное представление, показывающее историю эволюции набора видов или таксонов в течение определенного времени.
На ветвях филогенетического дерева неявно происходят две вещи. Во-первых, это течение времени. Более глубокие узлы старше более мелких узлов, с которыми они связаны. Таким образом, более глубокие узлы указывают как на более отдаленные отношения между терминальными таксонами, которые они соединяют, так и на больший возраст самого недавнего общего предка этих таксонов. Во-вторых, это эволюционная модификация или накопление наследственных генетических и/или структурных изменений вдоль этих ветвей. Термин «длина ветки» обычно относится к количеству этих изменений. Если «длина ветвей» дерева измеряет эти изменения, мы также называем дерево филограммой. Обычное филогенетическое дерево . Обычно его называют дендрограммой . Это диаграмма с прямыми линиями, представляющими дерево. Он будет отображать столбец узлов, представляющих отдельные таксоны, а оставшиеся узлы представляют кластеры, к которым принадлежат данные, со стрелками, обозначающими расстояние: способ измерения того, насколько они различаются (несходство). Расстояние между сливающимися кластерами монотонно, увеличиваясь с уровнем слияния: высота каждого узла на графике пропорциональна величине межгруппового несходства между двумя его ветвями.

Кладограмма – это также диаграмма с прямыми линиями, представляющими дерево. Разница между кладограммой и эволюционным деревом состоит в том, что кладограмма не показывает, как предки связаны с потомками, а также не показывает, насколько они изменились. Это означает, что одной и той же кладограмме может соответствовать более одного эволюционного дерева.

Круглое филогенетическое дерево . Круглые деревья часто используются для иллюстрации взаимоотношений между членами основных групп современных организмов, и эти деревья могут иметь множество конечных таксонов. Это может показаться нелогичным, но та же информация, что и в обычном филогенетическом дереве, содержится и в круговом генетическом дереве. Топология структуры остается прежней, и она меняет лишь форму, чтобы лучше разместить большой объем информации на меньшем пространстве.

3D-визуализация . На филограмме эволюционное расстояние представлено на одной из осей, а гены — на другой. Чтобы можно было визуализировать паралоги, можно добавить третью ось. В стандартной (2D) схеме филогении не всегда легко отличить события дупликации генов (паралоги) от ветвления видообразования (виды), поскольку доступна только одна пространственная ось (гены), чтобы показать сочетание этих двух видов информации. Напротив, в 3DPE их легко различить, поскольку он проецирует их на две ортогональные оси: виды (X) и паралоги (Z). Например, эволюция многих паралогов визуально очевидна в 3DPE представлении (у трех видов эукариот справа), но эта закономерность менее ясна в 2D представлении. [56]
Программное обеспечение для визуализации [ править ]
| Имя | Описание | Тип данных | Автор(ы) | Год |
|---|---|---|---|---|
| Цитоскейп | Программная платформа с открытым исходным кодом для визуализации сложных биологических сетей. [57] | Системная биология | Команда Цитоскейп | июль 2002 г. |
| РисДерево | Средство просмотра деревьев Java, способное читать несколько форматов файлов деревьев, цветные ветви и создавать векторные изображения. | Филогенетическое дерево | Эндрю Рамбо | 6 ноября 2006 г. |
| Интерактивное Древо Жизни (ИТОЛ) | Строит деревья и аннотирует их различными типами данных. | Филогенетическое дерево | Чиккарелли Ф.Д. и др. [58] | 3 марта 2006 г. |
| Джмол | Бесплатный Java-апплет с открытым исходным кодом, способный загружать несколько молекул с независимым движением, поверхностями и молекулярными орбиталями, визуализацией полостей и симметрией кристаллов. [59] | Молекулярный | Тогда Гезельтер | 2001 |
| Обработка, анализ и визуализация медицинских изображений (MIPAV) | Количественный анализ и визуализация медицинских изображений для таких методов, как ПЭТ, МРТ, КТ или микроскопия. [60] | Медицинская визуализация | Центр информационных технологий Национального института здравоохранения | Неизвестный |
| Медуза | Программное обеспечение для построения и анализа ансамблей реконструкций метаболических сетей в масштабе генома. [61] | Системная биология | Грегори Л. Медлок, Томас Дж. Моутиньо, Джейсон А. Папин | 2001 |
| Молекулярно-эволюционно-генетический анализ (МЕГА) | Предоставляет несколько алгоритмов для построения филогенетических деревьев, включая UPGMA, максимальное правдоподобие, максимальную экономию и т. д. | Филогенетическое дерево | Масатоши Ней, Судхир Кумар, Коитиро Тамура, Глен Стечер, Дэниэл Петерсон, Николас Петерсон | 1993 |
| Молекулярная операционная среда (MOE) | Моделирует микро- и макромолекулы, белково-лигандные комплексы и кристаллические решетки. | Молекулярный | Группа химических вычислений | Неизвестный |
| ПиМОЛ | Приложение Python с открытым исходным кодом для моделирования биологических макромолекул | Молекулярный | Уоррен Делано | 2017 |
| Т-Кофе | Выполняет множественное выравнивание последовательностей, используя прогрессивный подход. | Последовательности | Седрик Нотредам | 15 октября 2020 г. |
- ИТОЛ древо жизни
- Визуализация экзотоксина А, созданная с помощью Jmol
- Сегмент ДНК, изображенный PyMOL
- Данные дрожжевой сети, визуализированные Cytoscape
Ссылки [ править ]
- ^ Лучич В., Фёрстер Ф., Баумайстер В. (2005). «Структурные исследования методом электронной томографии: от клеток к молекулам». Ежегодный обзор биохимии . 74 : 833–65. doi : 10.1146/annurev.biochem.73.011303.074112 . ПМИД 15952904 .
- ^ Стивен А.С., Баумейстер В. (сентябрь 2008 г.). «Будущее гибридное». Журнал структурной биологии . 163 (3): 186–95. дои : 10.1016/j.jsb.2008.06.002 . ПМИД 18602011 . S2CID 2432954 .
- ^ Платтнер Х, Хентшель Дж (2006). «Субсекундная клеточная динамика: электронная микроскопия с временным разрешением и функциональная корреляция» . Обзор клеточной биологии (представленная рукопись). Международный обзор цитологии. Том. 255. стр. 133–76. дои : 10.1016/S0074-7696(06)55003-X . ISBN 9780123735997 . ПМИД 17178466 .
- ^ Франк Дж., Шлихтинг I (сентябрь 2004 г.). «Визуализация макромолекулярных процессов и взаимодействий с временным разрешением». Журнал структурной биологии . 147 (3): 209–10. дои : 10.1016/j.jsb.2004.06.003 . ПМИД 15450290 .
- ^ Уотерхаус, А.М., Проктер, Дж.Б., Мартин, ДМА, Клэмп, М. и Бартон, Г.Дж. (2009). Jalview версии 2 — редактор множественного выравнивания последовательностей и инструментарий для анализа. Биоинформатика, 25(9), 1189-1191.
- ^ Като, К., и Стэндли, DM (2013). Программное обеспечение для множественного выравнивания последовательностей MAFFT, версия 7: улучшение производительности и удобства использования. Молекулярная биология и эволюция, 30(4), 772-780.
- ^ Кумар С., Стечер Г., Ли М., Князь К. и Тамура К. (2018). MEGA X: Молекулярно-эволюционно-генетический анализ на различных вычислительных платформах. Молекулярная биология и эволюция, 35 (6), 1547–1549.
- ^ Сиверс, Ф., Уилм, А., Дайнин, Д., Гибсон, Т.Дж., Карплюс, К., Ли, В., ... и Хиггинс, Д.Г. (2011). Быстрое и масштабируемое создание высококачественных выравниваний множественных последовательностей белков с использованием Clustal Omega. Молекулярная системная биология, 7(1), 539.
- ^ Альтшул, С.Ф., Гиш, В., Миллер, В., Майерс, Э.В., и Липман, DJ (1990). Базовый инструмент поиска локального выравнивания. Журнал молекулярной биологии, 215(3), 403-410.
- ^ Шнайдер, Т.Д., и Стивенс, Р.М. (1990). Логотипы последовательностей: новый способ отображения согласованных последовательностей. Исследования нуклеиновых кислот, 18(20), 6097-6100.
- ^ Уотерхаус, А.М., Проктер, Дж.Б., Мартин, ДМА, Клэмп, М. и Бартон, Г.Дж. (2009). Jalview версии 2 — редактор множественного выравнивания последовательностей и инструментарий для анализа. Биоинформатика, 25(9), 1189-1191.
- ^ Петтерсен, Э.Ф., Годдард, Т.Д., Хуанг, CC, Коуч, GS, Гринблатт, DM, Мэн, EC, и Феррин, TE (2004). UCSF Chimera — система визуализации для поисковых исследований и анализа. Журнал вычислительной химии, 25 (13), 1605–1612.
- ^ Рамбо, А. (2012). FigTree: инструмент для рисования древовидных фигур. Молекулярная эволюция, филогенетика и эпидемиология. Получено с http://tree.bio.ed.ac.uk/software/figtree/ .
- ^ Кент, В.Дж., Сагнет, К.В., Фьюри, Т.С., Роскин, К.М., Прингл, Т.Х., Залер, А.М., и Хаусслер, Д. (2002). Браузер генома человека в UCSC. Геномные исследования, 12(6), 996-1006.
- ^ Альтшул, С.Ф., Гиш, В., Миллер, В., Майерс, Э.В., и Липман, DJ (1990). Базовый инструмент поиска локального выравнивания. Журнал молекулярной биологии, 215(3), 403-410.
- ^ Макларен, В., Гил, Л., Хант, С.Э., Риат, Х.С., Ричи, Г.Р., Торманн, А., ... и Каннингем, Ф. (2016). Предиктор эффекта ансамблевого варианта. Геномная биология, 17 (1), 1-14.
- ^ Фельзенштейн, Дж. (1985). Доверительные пределы филогении: подход с использованием бутстрепа. Эволюция, 39(4), 783-791.
- ^ Хатри П., Сирота М. и Бьютт А.Дж. (2012). Десять лет анализа путей: текущие подходы и нерешенные проблемы. PLoS вычислительная биология, 8 (2), e1002375.
- ^ Уотерхаус, А.М., Проктер, Дж.Б., Мартин, ДМА, Клэмп, М. и Бартон, Г.Дж. (2009). Jalview версии 2 — редактор множественного выравнивания последовательностей и инструментарий для анализа. Биоинформатика, 25(9), 1189-1191.
- ^ Чарешнев А; Мидлик, А; Ионеску, CM (2023). «Объемы и сегментации Mol *: визуализация и интерпретация данных визуализации клеток наряду с данными о структуре макромолекул и биологическими аннотациями» . Исследования нуклеиновых кислот . 51(Ж1) (Ж1): Ж326–Ж330. дои : 10.1093/nar/gkad411 . ПМК 10320116 . ПМИД 37194693 .
- ^ Сенал, Д; Свободова, Р; Берка, К (2021). «Высокопроизводительная доставка и визуализация макромолекулярных данных для Интернета. Исправление» (PDF) . Acta Crystallographica Раздел D. 77 (1): 126-126. Бибкод : 2021AcCrD..77..126S . дои : 10.1107/S205979832001606X . ПМЦ 7787108 . ПМИД 33404533 .
- ^ Сун, Р.Дж.; Уилсон, AT; Ло, С.М. (2019). «BiochemAR: образовательный инструмент дополненной реальности для обучения структуре и функциям макромолекул» . Журнал химического образования . 97 (1): 147–153. doi : 10.1021/acs.jchemed.8b00691 .
- ^ Вернер, Э. (2022). «Стратегии создания молекулярной анимации» . Границы биоинформатики . 2 : 793914. дои : 10.3389/fbinf.2022.793914 . ПМЦ 9580893 . ПМИД 36304328 .
- ^ Пьерони, М; Мадедду, Ф; Ди Мартино, Дж (2023). «МД-лиганд-рецептор: высокопроизводительный вычислительный инструмент для характеристики связывающих взаимодействий лиганд-рецептор в траекториях молекулярной динамики» . . Международный журнал молекулярных наук . 24 (14): 11671. doi : 10.3390/ijms241411671 . ПМЦ 10380688 . ПМИД 37511429 .
- ^ Берли, СК; Бхикадия, К; Би, К (17 августа 2023 г.). «Банк данных белков RCSB: празднование 50-летия PDB с новыми инструментами для понимания и визуализации биологических макромолекул в 3D» . Белковая наука . 31 (1): 187–208. дои : 10.1128/spectrum.01859-23 . ПМЦ 10433874 . ПМИД 37382549 .
- ^ Гайдос, Л; Блейкли, член парламента; Кумар, А (2021). «Визуализация атомов водорода в пердейтерированном комплексе лектин-фукоза раскрывает ключевые детали белок-углеводных взаимодействий» (PDF) . Структура . 29 (9): 1003–1013.e4. doi : 10.1016/j.str.2021.03.003 . ПМИД 33765407 .
- ^ Ху, Кью; Ло, Ю (2021). «Наноносители на основе хитозана для инкапсуляции и доставки куркумина: обзор» . Международный журнал биологических макромолекул . 179 : 125–135. doi : 10.1016/j.ijbiomac.2021.02.216 . PMID 33667554 .
- ^ Ван, Т; Ху, Кью; Сюэ, Дж (2021). «Распределение и стабильность фолиевой кислоты и кофейной кислоты в полых частицах зеина, покрытых хитозаном» . Международный журнал биологических макромолекул . 183 : 2282–2292. doi : 10.1016/j.ijbiomac.2021.05.216 . ПМИД 34102238 .
- ^ Гайдос, Л; Блейкли, член парламента; Кумар, А (2021). «Визуализация атомов водорода в пердейтерированном комплексе лектин-фукоза раскрывает ключевые детали белок-углеводных взаимодействий». Структура . 29 (9): 1003–1013.e4. doi : 10.1016/j.str.2021.03.003 . ПМИД 33765407 .
- ^ Хименес-Гарсия, Б; Роэл-Турис, Дж; Баррадас-Баутиста, Д (2023). «Сервер LightDock: моделирование макромолекулярных взаимодействий с помощью искусственного интеллекта» . Исследования нуклеиновых кислот . 51(W1) (W1): W298–W304. дои : 10.1093/nar/gkad327 . ПМЦ 10320125 . ПМИД 37140054 .
- ^ Лабант, МэриЭнн (1 ноября 2013 г.). «Поднятие планки в доклинической визуализации» . Новости генной инженерии и биотехнологии . Мэри Энн Либерт, Inc. Проверено 24 апреля 2024 г.
- ^ Ананиаду, София (12 октября 2006 г.). «Интеллектуальный анализ текста и его потенциальные применения в системной биологии» . Тенденции в биотехнологии . 24 (12): 571–579. дои : 10.1016/j.tibtech.2006.10.002 . ПМИД 17045684 . Проверено 14 апреля 2024 г.
- ^ Ресендис-Антонио, Осбальдо (2013). «Моделирование на основе ограничений» . Энциклопедия системной биологии . Спрингер Природа. стр. 494–498. дои : 10.1007/978-1-4419-9863-7_1143 . ISBN 978-1-4419-9862-0 . Проверено 14 апреля 2024 г.
{{cite book}}:|website=игнорируется ( помогите ) - ^ Орт, Джеффри Д.; Тиле, Инес; Палссон, Бернхард О (март 2010 г.). «Что такое анализ баланса потоков?» . Природная биотехнология . 28 (3): 245–248. дои : 10.1038/nbt.1614 . ISSN 1087-0156 . ПМК 3108565 . ПМИД 20212490 .
- ^ «Визуальная метаболомика» . Шимадзу . Проверено 15 апреля 2024 г.
- ^ Уарото, Джей-Джей; Капуано, Л; Кая, М (2023). «Двухфотонная микроскопия для микроробототехники: визуализация микроагентов под фиксированной тканью» . ПЛОС ОДИН . 18 (8): e0289725. Бибкод : 2023PLoSO..1889725H . дои : 10.1371/journal.pone.0289725 . ПМЦ 10414647 . ПМИД 37561749 .
- ^ Платонова Г; Штыс, Д; Соучек, П (2021). «Спектроскопический подход к коррекции и визуализации биологических данных светлопольной светопропускающей микроскопии» . В Фотонике . 8 (8): 333. arXiv : 1903.06519 . Бибкод : 2021Фото...8..333P . doi : 10.3390/photonics8080333 .
- ^ Джамбор, Гонконг (2023 г.). «Общественный подход к повышению качества и интерпретируемости микроскопических изображений» . Журнал клеточной науки . 136 (24): jcs261837. дои : 10.1242/jcs.261837 . ПМИД 38095680 .
- ^ Вебер, Б. (2009). «Магнитно-резонансная томография в исследованиях эпилепсии: последние и предстоящие разработки» . Наука Директ . Энциклопедия фундаментальных исследований эпилепсии . Проверено 14 апреля 2024 г.
- ^ Вулискрофт, Линдси (апрель 2024 г.). «Визуализация базисного спектра диффузии и визуализация тензора диффузии предсказывают стойкое образование черных дыр при рассеянном склерозе» . Рассеянный склероз и связанные с ним заболевания . Эльзевир . Проверено 14 апреля 2024 г.
- ^ Винсент, К. (12 декабря 2008 г.). «Функциональная магнитно-резонансная томография, зависящая от уровня оксигенации крови: современное и потенциальное использование в акушерстве и гинекологии» . BJOG: Международный журнал акушерства и гинекологии . 116 (2): 240–246. дои : 10.1111/j.1471-0528.2008.01993.x . ПМК 2675013 . ПМИД 19076956 .
- ^ «Как работают импульсы насыщения?» . Вопросы и ответы по МРТ . ООО "Эльстер" . Проверено 14 апреля 2024 г.
- ^ Ча, Сунми (2013). «Динамические функциональные и физиологические методики» . Наука Директ . Изображение мозга . Проверено 14 апреля 2024 г.
- ^ Ча, Сунми (28 сентября 2016 г.). «Диффузионно-взвешенная визуализация: техника и применение» . Всемирный журнал радиологии . 8 (9). Издательская группа Байсидэн: 785–798. дои : 10.4329/wjr.v8.i9.785 . ПМК 5039674 . ПМИД 27721941 .
- ^ «Радиочастотные электромагнитные поля (ЭМП)» . Здоровье Канады . Правительство Канады. 8 декабря 2020 г. . Проверено 24 апреля 2024 г.
- ^ Престон, Дэвид К. (4 июля 2016 г.). «Магнитно-резонансная томография (МРТ) головного мозга и позвоночника: основы» . Университет Кейс Вестерн Резерв . Проверено 24 апреля 2024 г.
- ^ Бергер, Аби (5 января 2002 г.). «Магнитно-резонансная томография» . БМЖ . 324 (7328): 35. дои : 10.1136/bmj.324.7328.35 . ПМК 1121941 . ПМИД 11777806 .
- ^ Баба, Яхья (4 сентября 2022 г.). «МРТ-последовательности (обзор)» . Радиопедия . Радиопедия.орг . Проверено 24 апреля 2024 г.
- ^ Нетт, Брайан (4 декабря 2023 г.). «Контрастные вещества (рентгеноконтрастные вещества и йодсодержащие контрастные вещества)» . Как работает радиология . ООО «Как работает радиология» . Проверено 24 апреля 2024 г.
- ^ «ПЭТ/КТ» . RadiologyInfo.org . Радиологическое общество Северной Америки, Inc., 1 мая 2023 г. Проверено 14 апреля 2024 г.
- ^ Прокоп, М.; Шин, ХО; Шанц, А.; Шефер-Прокоп, CM (март 1997 г.). «Использование проекций максимальной интенсивности в КТ-ангиографии: базовый обзор». Рентгенография . 17 (2). Совет директоров Радиологического общества Северной Америки, Inc.: 433–451. doi : 10.1148/radiographics.17.2.9084083 . ПМИД 9084083 .
- ^ Озкан, Мехмет Бурак; Чеунер, Себастьян; Элиф, Озкан (декабрь 2016 г.). «Возврат диагностической точности методов срезов MIP для небольших легочных узелков у пациентов с детской онкологией: что еще есть в подходе детского радиолога?» . Египетский журнал радиологии и ядерной медицины . 47 (4). Египетское общество радиологии и ядерной медицины: 1629–1637. дои : 10.1016/j.ejrnm.2016.09.008 .
- ^ Адамсон, Юстус; Чжэн, Чанг; Ван, Чжихэн; Инь, Фан-Фан; Цай, Цзин (ноябрь 2010 г.). «Визуализация проекции максимальной интенсивности (MIP) с использованием МРТ с накоплением срезов». Мед. физ . 37 (11). Американский институт физики: 5914–5920. Бибкод : 2010MedPh..37.5914A . дои : 10.1118/1.3503850 . ПМИД 21158304 .
- ^ Антропова, Наталья; Абэ, Хироюки; Гигер, Мэриеллен Л. (5 января 2018 г.). «Использование клинических проекций максимальной интенсивности МРТ для улучшения классификации поражений молочной железы с помощью глубоких сверточных нейронных сетей» . Журнал медицинской визуализации (Беллингем, Вашингтон) . 5 (1). Общество фотооптических приборов: 014503. doi : 10.1117/1.JMI.5.1.014503 . ПМЦ 5798576 . ПМИД 29430478 .
- ^ Банк, Данные о белках RCSB. «Исследование нескольких выравниваний последовательностей в 1D и 3D» . www.rcsb.org . Проверено 26 апреля 2024 г.
- ^ Ким, Намшин; Ли, Кристофер (20 июня 2007 г.). «Трехмерный исследователь филогении: различение паралогов, латеральный перенос и нарушение предположения о «молекулярных часах» с помощью 3D-визуализации» . БМК Биоинформатика . 8 (1): 213. дои : 10.1186/1471-2105-8-213 . ISSN 1471-2105 . ПМК 1906840 . ПМИД 17584922 .
- ^ «Цитоскейп» . Цитоскейп . Консорциум Cytoscape . Проверено 16 апреля 2024 г.
- ^ «Интерактивное древо жизни ИТОЛ» . ИТОЛ . Исследования нуклеиновых кислот. 13 апреля 2024 г. . Проверено 16 апреля 2024 г.
- ^ Максуд, Муниза (11 октября 2020 г.). «Анализ и визуализация биологических данных» . Биокод . ООО "БиоКод" . Проверено 16 апреля 2024 г.
- ^ «О МИПАВ» . ИТОЛ . Центр информационных технологий. 13 апреля 2024 г. . Проверено 16 апреля 2024 г.
- ^ Медлок, GL; Моутинью, Ти Джей; Папин, Дж. А. (29 апреля 2020 г.). «Medusa: Программное обеспечение для построения и анализа ансамблей реконструкций метаболических сетей в масштабе генома» . PLOS Вычислительная биология . 16 (4). PLoS Компьютерная биология: e1007847. Бибкод : 2020PLSCB..16E7847M . дои : 10.1371/journal.pcbi.1007847 . ПМЦ 7213742 . ПМИД 32348298 .
Внешние ссылки [ править ]
Сопутствующие конференции [ править ]
- BioVis: Симпозиум по визуализации биологических данных
- Применение визуализации информации в биоинформатике
- CIBDV: вычислительный интеллект для визуализации биологических данных
- IVBI: Визуализация информации в симпозиуме по биомедицинской информатике
- VMLS: визуализация в медицине и науках о жизни
- ВИЗБИ: Семинар по визуализации биологических данных