ПЭТАза
| ПЭТАза | |||
|---|---|---|---|
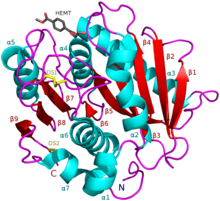 I. sakaiensis ПЭТаза ( A0A0K8P6T7 ) в комплексе с HEMT, аналогом ПЭТ ( PDB : 5XH3 ). | |||
| Идентификаторы | |||
| Номер ЕС. | 3.1.1.101 | ||
| Альт. имена | ПЭТ-гидролаза, поли(этилентерефталат)-гидролаза | ||
| Базы данных | |||
| ИнтЭнк | вид IntEnz | ||
| БРЕНДА | БРЕНДА запись | ||
| Экспаси | Просмотр NiceZyme | ||
| КЕГГ | КЕГГ запись | ||
| МетаЦик | метаболический путь | ||
| ПРЯМОЙ | профиль | ||
| PDB Структуры | RCSB PDB PDBe PDBsum | ||
| |||
ПЭТазы представляют собой эстераз класс ферментов , которые катализируют расщепление (посредством гидролиза ) полиэтилентерефталатного (ПЭТ) пластика до мономерного моно-2-гидроксиэтилтерефталата (МГЭТ). Идеализированная химическая реакция:
- (этилентерефталат) n + H 2 O → (этилентерефталат) n -1 + МГЭТ,
где n - количество мономеров в полимерной цепи, хотя следовые количества ПЭТ вместо этого распадаются до бис (2-гидроксиэтил) терефталата (БГЭТ). [1] ПЭТазы также могут расщеплять ПЭФ-пластик ( полиэтилен-2,5-фурандикарбоксилат ), который является заменителем ПЭТ биологического происхождения, до аналогичного MHEF . ПЭТазы не могут катализировать гидролиз алифатических полиэфиров, таких как полибутиленсукцинат или полимолочная кислота . [2]
В то время как разложение ПЭТ естественным (неферментативным) путем займет сотни лет, ПЭТазы могут разложить его за считанные дни. [3]
История
[ редактировать ]Первая ПЭТаза была обнаружена в 2016 году у Ideonella sakaiensis штамма 201-F6 бактерий , обнаруженных в образцах ила, собранных недалеко от японского предприятия по переработке ПЭТ-бутылок . [1] [4] Ранее были известны другие типы гидролаз, способные разлагать ПЭТ. [2] включая липазы, эстеразы и кутиназы. [5] Для сравнения, о существовании ферментов, расщепляющих полиэстер, было известно как минимум еще в 1975 году (в случае α -химотрипсина ). [6] и 1977 г. ( липаза ). [7]
ПЭТ-пластик получил широкое распространение в 1970-х годах, и было высказано предположение, что ПЭТазы в бактериях появились лишь недавно. [2] ПЭТАза, возможно, имела в прошлом ферментативную активность, связанную с разрушением воскового покрытия на растениях. [8]
Структура
[ редактировать ]По состоянию на апрель 2019 года было известно 17 трехмерных кристаллических структур ПЭТаз: 6QGC , 6ILX , 6ILW , 5YFE , 6EQD , 6EQE , 6EQF , 6EQG , 6EQH , 6ANE , 5XJH , 5YNS , 5XFY , 5XFZ , 5XG0 . , 5XH2 и 5XH3 .
ПЭТаза проявляет общие свойства как с липазами, так и с кутиназами в том, что она обладает α/β-гидролазной складкой; хотя щель активного центра, наблюдаемая у ПЭТазы, более открыта, чем у кутиназ. [2] , ПЭТАза Ideonella sakaiensis аналогична диенелактонгидролазе По данным Pfam . По данным ESTHER, он относится к семейству полиэстеразы-липазы-кутиназы.
Существует около 69 ферментов, подобных ПЭТАзе, включающих множество разнообразных организмов, и существует две классификации этих ферментов, включая тип I и тип II. Предполагается, что 57 ферментов относятся к категории I типа, тогда как остальные относятся к группе типа II, включая фермент ПЭТаза, обнаруженный у Ideonella sakaiensis . Во всех 69 ПЭТазоподобных ферментах в активном центре существуют одни и те же три остатка, что позволяет предположить, что каталитический механизм одинаков для всех форм ПЭТАзоподобных ферментов. [9]
- ПЭТазы Поверхность двойного мутанта ( R 103 G и S 131 A ) с HEMT (1-(2-гидроксиэтил) 4-метилтерефталатом), связанным с ее активным центром . HEMT является аналогом MHET и содержит дополнительный метанол этерифицированный . PDBID: 5XH3.
- Ленточная диаграмма ПЭТазы с тремя остатками Ser160, Asp206 и His237. Каталитическая триада представлена палочками голубого цвета. Активный сайт показан оранжевым цветом и представляет стимуляцию молекулой 2-HE(MHET) 4 . [9]
Мутации
[ редактировать ]Открытие ПЭТазы I. sakaiensis обеспечивает потенциальное решение проблемы накопления пластика в мире; однако встречающиеся в природе ферменты ограничены в своей способности к деградации из-за нестабильности, низкой активности и уровня экспрессии, что в конечном итоге приводит к необходимости улучшения, если они будут использоваться для крупномасштабного промышленного применения. [10] Большинство стратегий реализуют сайт-направленный мутагенез для создания улучшенной версии, известной как вариант или мутант фермента. Один вариант увеличил активность ПЭТазы на 22,4% за счет замены аргинина на аланин в аминокислотной цепи в 280-м положении. [11] Аналогично, двойной мутант был создан для сужения активного сайта и стал на 4,13% более активным, чем дикий тип. [12] Для сравнения, два других двойных мутанта создали дополнительные водородные связи, которые улучшили стабильность ПЭТазы. Другие успешные подходы к улучшению стабильности ПЭТазы включают добавление Ca 2+ или мг 2+ , дисульфидные связи и солевые мостики , а также гликозилирование . [10] Что касается термостабильности, у другого двойного мутанта наблюдалось увеличение по сравнению с диким типом. [11] Более того, соединительная петля β1-β2 фермента также может стать будущей мишенью для улучшения термостабильности благодаря ее гибкости и удалению от активного центра. [13]
Биологический путь
[ редактировать ]
У I. sakaiensis полученный MHET далее расщепляется под действием фермента MHETase до терефталевой кислоты и этиленгликоля . [1] Лабораторные эксперименты показали, что химерные белки, искусственно связывающие МГЕТазу и ПЭТазу, превосходят аналогичные смеси свободных ферментов. [15]
См. также
[ редактировать ]- Пластивор
- Galleria mellonella — гусеница, способная переваривать полиэтилен .
- Aspergillus Tubeingensis , грибок, способный переваривать полиуретан .
- Pestalotiopsis microspora — эндофитный вид грибов, способный расщеплять полиуретан.
- Кутиназа — фермент эстераза схожей геометрической формы.
Ссылки
[ редактировать ]- ^ Jump up to: а б с Ёсида С., Хирага К., Такехана Т., Танигучи И., Ямаджи Х., Маэда Ю. и др. (март 2016 г.). «Бактерия, которая разлагает и усваивает поли(этилентерефталат)». Наука . 351 (6278): 1196–9. Бибкод : 2016Sci...351.1196Y . дои : 10.1126/science.aad6359 . ПМИД 26965627 . S2CID 31146235 .
- «Открытие бактерии, которая разлагает и ассимилирует поли(этилентерефталат), может служить платформой для разложения и/или ферментации для биологической переработки отходов ПЭТ» (PDF) . Киотский технологический институт (пресс-релиз). 30 марта 2016 г.
- ^ Jump up to: а б с д Остин Х.П., Аллен М.Д., Донохо Б.С., Роррер Н.А., Кернс Ф.Л., Сильвейра Р.Л. и др. (май 2018 г.). «Характеристика и разработка ароматической полиэстеразы, разлагающей пластик» . Труды Национальной академии наук Соединенных Штатов Америки . 115 (19): Е4350–Е4357. Бибкод : 2018PNAS..115E4350A . дои : 10.1073/pnas.1718804115 . ПМЦ 5948967 . ПМИД 29666242 .
- ^ Докрилл, Питер. «Ученые случайно создали мутантный фермент, который поедает пластиковые отходы» . НаукаАлерт . Проверено 27 ноября 2018 г.
- ^ Танасупават С., Такехана Т., Ёсида С., Хирага К., Ода К. (август 2016 г.). «Ideonella sakaiensis sp. nov., выделенная из микробного консорциума, разлагающего поли(этилентерефталат)» . Международный журнал систематической и эволюционной микробиологии . 66 (8): 2813–8. дои : 10.1099/ijsem.0.001058 . ПМИД 27045688 .
- ^ Хань X, Лю В, Хуан Дж.В., Ма Дж., Чжэн Ю., Ко Т.П. и др. (декабрь 2017 г.). «Структурное понимание каталитического механизма ПЭТ-гидролазы» . Природные коммуникации . 8 (1): 2106. Бибкод : 2017NatCo...8.2106H . дои : 10.1038/s41467-017-02255-z . ПМЦ 5727383 . ПМИД 29235460 .
- ^ Табуси И., Ямада Х., Мацузаки Х., Фурукава Дж. (август 1975 г.). «Полиэфир легко гидролизуется химотрипсином». Журнал науки о полимерах: издание Polymer Letters . 13 (8): 447–450. Бибкод : 1975JPoSL..13..447T . дои : 10.1002/pol.1975.130130801 .
- ^ Токива Ю., Сузуки Т. (ноябрь 1977 г.). «Гидролиз полиэфиров липазами». Природа . 270 (5632): 76–8. Бибкод : 1977Natur.270...76T . дои : 10.1038/270076a0 . ПМИД 927523 . S2CID 4145159 .
- ^ «Лабораторный «несчастный случай» становится мутантным ферментом, пожирающим пластик» . Живая наука . Проверено 27 ноября 2018 г.
- ^ Jump up to: а б Джу С., Чо И.Дж., Со Х., Сон Х.Ф., Сагон Х.И., Шин Т.Дж. и др. (январь 2018 г.). «Структурное понимание молекулярного механизма разложения полиэтилентерефталата» . Природные коммуникации . 9 (1): 382. Бибкод : 2018NatCo...9..382J . дои : 10.1038/s41467-018-02881-1 . ПМК 5785972 . ПМИД 29374183 .
- ^ Jump up to: а б Ци, Синьхуа; Ян, Вэньлун; Цао, Жибэй; Дин, Минчжу; Юань, Инцзин (26 декабря 2021 г.). «Современные достижения в области биоразложения и биоконверсии полиэтилентерефталата» . Микроорганизмы . 10 (1): 39. doi : 10.3390/microorganisms10010039 . ISSN 2076-2607 . ПМЦ 8779501 . ПМИД 35056486 .
- ^ Jump up to: а б Урбанек, Анета К.; Косёровска, Катажина Е.; Мироньчук, Александра М. (30 ноября 2021 г.). «Современные знания о разложении полиэтилентерефталата генетически модифицированными микроорганизмами» . Границы биоинженерии и биотехнологии . 9 . дои : 10.3389/fbioe.2021.771133 . ISSN 2296-4185 . ПМЦ 8669999 . ПМИД 34917598 .
- ^ Остин, Гарри П.; Аллен, Марк Д.; Донохо, Брайон С.; Роррер, Николас А.; Кернс, Фиона Л.; Сильвейра, Родриго Л.; Поллард, Бенджамин К.; Доминик, Грэм; Думан, Рамона; Эль Омари, Камель; Михайлик, Виталий; Вагнер, Армин; Миченер, Уильям Э.; Аморе, Антонелла; Скаф, Мунир С. (08 мая 2018 г.). «Характеристика и разработка ароматической полиэстеразы, разлагающей пластик» . Труды Национальной академии наук . 115 (19): Е4350–Е4357. Бибкод : 2018PNAS..115E4350A . дои : 10.1073/pnas.1718804115 . ISSN 0027-8424 . ПМЦ 5948967 . ПМИД 29666242 .
- ^ да Коста, Клаубер Энрике Соуза; дос Сантос, Альберто М.; Алвес, Клаудио Наум; Марти, Серхио; Молинер, Висент; Сантана, Кауэ; Ламейра, Херонимо (октябрь 2021 г.). «Оценка конформационных изменений ПЭТазы, индуцированных связыванием поли(этилентерефталата)» . Белки: структура, функции и биоинформатика . 89 (10): 1340–1352. дои : 10.1002/прот.26155 . ISSN 0887-3585 . ПМИД 34075621 . S2CID 233620229 .
- ^ Эллисон Чан (2016). «Будущее бактерий, очищающих наши пластиковые отходы» (PDF) .
- ^ Нотт Б.С., Эриксон Э., Аллен М.Д., Гадо Дж.Э., Грэм Р., Кернс Ф.Л. и др. (октябрь 2020 г.). «Характеристика и разработка двухферментной системы для деполимеризации пластмасс» . Proc Natl Acad Sci США . 117 (41): 25476–25485. Бибкод : 2020PNAS..11725476K . дои : 10.1073/pnas.2006753117 . ПМЦ 7568301 . ПМИД 32989159 .

![Ленточная диаграмма ПЭТазы с тремя остатками Ser160, Asp206 и His237. Каталитическая триада представлена палочками голубого цвета. Активный сайт показан оранжевым цветом и представляет стимуляцию молекулой 2-HE(MHET)4.[9]](http://upload.wikimedia.org/wikipedia/commons/thumb/d/db/PETase_active_site.png/739px-PETase_active_site.png)