Функциональная геномика

Функциональная геномика — это область молекулярной биологии , которая пытается описать функции и взаимодействия генов (и белков ). Функциональная геномика использует обширные данные, полученные в результате геномных и транскриптомных проектов (таких как проекты секвенирования генома и секвенирования РНК ). Функциональная геномика фокусируется на динамических аспектах, таких как транскрипция генов , трансляция , регуляция экспрессии генов и межбелковых взаимодействиях , в отличие от статических аспектов геномной информации, таких как последовательность или структуры ДНК. Ключевой характеристикой исследований функциональной геномики является их полногеномный подход к этим вопросам, обычно включающий высокопроизводительные методы, а не более традиционный подход «ген-кандидат».
Определение и цели [ править ]
Чтобы понять функциональную геномику, важно сначала определить функцию. В своей статье [1] Граур и др. определить функцию двумя возможными способами. Это «избранное следствие» и «причинная роль». Функция «выбранный эффект» относится к функции, для которой выбирается признак (ДНК, РНК, белок и т. д.). Функция «причинная роль» относится к функции, для которой признак достаточен и необходим. Функциональная геномика обычно проверяет определение функции «причинной роли».
Цель функциональной геномики — понять функции генов или белков, в конечном итоге всех компонентов генома. Термин «функциональная геномика» часто используется для обозначения множества технических подходов к изучению генов и белков организма, включая «биохимические, клеточные и/или физиологические свойства каждого генного продукта». [2] в то время как некоторые авторы включают в свое определение изучение негенных элементов. [3] Функциональная геномика может также включать исследования естественных генетических изменений с течением времени (например, развитие организма) или пространства (например, областей его тела), а также функциональных нарушений, таких как мутации.
Перспективы функциональной геномики заключаются в создании и синтезе геномных и протеомных знаний для понимания динамических свойств организма. Потенциально это может дать более полную картину того, как геном определяет функции, по сравнению с исследованиями отдельных генов. Интеграция данных функциональной геномики часто является частью подходов системной биологии .
Методы и приложения [ править ]
Функциональная геномика включает в себя функциональные аспекты самого генома, такие как мутации и полиморфизм (например, анализ однонуклеотидного полиморфизма (SNP)), а также измерение молекулярной активности. Последние включают ряд «-омик » , таких как транскриптомика ( экспрессия генов ), протеомика ( производство белка ) и метаболомика . Функциональная геномика использует в основном мультиплексные методы для измерения содержания многих или всех генных продуктов, таких как мРНК или белки, в биологическом образце . Более сфокусированный подход функциональной геномики мог бы проверить функцию всех вариантов одного гена и количественно оценить эффекты мутантов, используя секвенирование для определения активности. Вместе эти методы измерения направлены на количественную оценку различных биологических процессов и улучшение нашего понимания функций и взаимодействий генов и белков.
На уровне ДНК [ править ]
взаимодействий генетических Картирование
Систематическое попарное удаление генов или ингибирование экспрессии генов можно использовать для идентификации генов со схожей функцией, даже если они не взаимодействуют физически. Эпистаз означает тот факт, что эффекты нокаута двух разных генов могут не суммироваться; то есть фенотип, возникающий в результате ингибирования двух генов, может отличаться от суммы эффектов одиночного нокаута.
ДНК Взаимодействие и белка
Белки, образующиеся в результате трансляции мРНК (информационная РНК, закодированная информация ДНК для синтеза белка), играют важную роль в регуляции экспрессии генов. Чтобы понять, как они регулируют экспрессию генов, необходимо идентифицировать последовательности ДНК, с которыми они взаимодействуют. Разработаны методы выявления участков ДНК-белковых взаимодействий. К ним относятся ChIP-секвенирование , секвенирование CUT&RUN и телефонные карты. [4]
ДНК доступности Анализы
Были разработаны анализы для выявления доступных областей генома. Эти области доступного хроматина являются кандидатами на регуляторные области. Эти анализы включают ATAC-seq , DNase-Seq и FAIRE-Seq .
На уровне РНК [ править ]
Микрочипы [ править ]

Микрочипы измеряют количество мРНК в образце, которое соответствует данному гену или последовательности ДНК-зонда. Последовательности зондов иммобилизуют на твердой поверхности и позволяют гибридизоваться с флуоресцентно меченной «мишенью» мРНК. Интенсивность флуоресценции пятна пропорциональна количеству целевой последовательности, гибридизовавшейся с этим пятном, и, следовательно, распространенности этой последовательности мРНК в образце. Микрочипы позволяют идентифицировать гены-кандидаты, участвующие в данном процессе, на основе различий между уровнями транскриптов для разных условий и общих паттернов экспрессии с генами с известной функцией.
МУДРЕЦ [ править ]
Серийный анализ экспрессии генов (SAGE) — это альтернативный метод анализа, основанный на секвенировании РНК, а не на гибридизации. SAGE опирается на секвенирование тегов из 10–17 пар оснований, которые уникальны для каждого гена. Эти метки производятся из мРНК поли-А и лигируются конец в конец перед секвенированием. SAGE дает объективное измерение количества транскриптов на клетку, поскольку оно не зависит от предварительного знания того, какие транскрипты изучать (как это делают микрочипы).
Секвенирование РНК [ править ]
Как отмечалось в 2016 году, в последние годы секвенирование РНК заменило технологию микрочипов и SAGE и стало наиболее эффективным способом изучения транскрипции и экспрессии генов. Обычно это делается с помощью секвенирования следующего поколения . [5]
Подмножество секвенированных РНК представляют собой малые РНК, класс некодирующих молекул РНК, которые являются ключевыми регуляторами транскрипционного и посттранскрипционного молчания генов или молчания РНК . Секвенирование нового поколения является золотым стандартом для обнаружения, профилирования и анализа экспрессии некодирующих РНК .
репортерные анализы (MPRA параллельные Массивно - )
Массивно-параллельные репортерные анализы — это технология проверки цис-регуляторной активности последовательностей ДНК. [6] [7] MPRA используют плазмиду с синтетическим цис-регуляторным элементом перед промотором, управляющим синтетическим геном, таким как зеленый флуоресцентный белок. Библиотеку цис-регуляторных элементов обычно тестируют с помощью MPRA, библиотека может содержать от сотен до тысяч цис-регуляторных элементов. Цис-регуляторную активность элементов анализируют с использованием нижестоящей репортерной активности. Активность всех членов библиотеки анализируется параллельно с использованием штрих-кодов для каждого цис-регуляторного элемента. Одним из ограничений MPRA является то, что активность анализируется на плазмиде и может не отражать все аспекты регуляции генов, наблюдаемые в геноме.
STARR-seq [ править ]
STARR-seq — это метод, аналогичный MPRA, для анализа энхансерной активности случайно расщепленных геномных фрагментов. В оригинальной публикации [8] случайно расщепленные фрагменты генома дрозофилы были помещены ниже минимального промотора. Кандидаты в энхансеры среди случайно расщепленных фрагментов будут транскрибировать себя с использованием минимального промотора. Используя секвенирование в качестве считывания и контроля входных количеств каждой последовательности, этим методом анализируют силу предполагаемых энхансеров.
Perturb-seq [ править ]

Perturb-seq сочетает нокдауны генов, опосредованные CRISPR, с экспрессией генов в одной клетке. Линейные модели используются для расчета влияния нокдауна одного гена на экспрессию нескольких генов.
На уровне белка [ править ]
Дрожжевая двугибридная система [ править ]
дрожжей Скрининг двухгибридных (Y2H) тестирует белок-приманку против многих потенциально взаимодействующих белков («жертва») для выявления физических межбелковых взаимодействий. Эта система основана на транскрипционном факторе, первоначально GAL4, [9] отдельные домены ДНК-связывания и активации транскрипции необходимы для того, чтобы белок вызывал транскрипцию репортерного гена. При скрининге Y2H белок-приманка сливается со связывающим доменом GAL4, а библиотека потенциальных белков-жертв (взаимодействующих) рекомбинантно экспрессируется в векторе с доменом активации. In vivo взаимодействие белков-приманок и жертв в дрожжевой клетке приводит к тому, что активационные и связывающие домены GAL4 сближаются достаточно близко друг к другу, что приводит к экспрессии репортерного гена . Также возможно систематически тестировать библиотеку белков-приманок с библиотекой белков-жертв, чтобы выявить все возможные взаимодействия в клетке.
MS и AP/MS [ править ]
Масс-спектрометрия (МС) позволяет идентифицировать белки и их относительные уровни, поэтому ее можно использовать для изучения экспрессии белков. При использовании в сочетании с аффинной очисткой масс -спектрометрию (AP/MS) можно использовать для изучения белковых комплексов, то есть того, какие белки взаимодействуют друг с другом в комплексах и в каких соотношениях. Чтобы очистить белковые комплексы, обычно белок-приманку помечают определенным белком или пептидом, который можно использовать для извлечения комплекса из сложной смеси. Очистку обычно проводят с использованием антитела или соединения, которое связывается с слитой частью. Затем белки расщепляются на короткие пептидные фрагменты, и для идентификации белков используется масс-спектрометрия на основе отношения массы к заряду этих фрагментов.
сканирование мутационное Глубокое
При глубоком мутационном сканировании сначала синтезируются все возможные аминокислотные замены в данном белке. [10] Активность каждого из этих вариантов белка анализируют параллельно с использованием штрих-кодов для каждого варианта. [11] Сравнивая активность с белком дикого типа, определяют эффект каждой мутации. Несмотря на то, что благодаря комбинаторике можно оценить каждое возможное изменение аминокислоты, две или более одновременные мутации трудно проверить. Эксперименты по глубокому мутационному сканированию также использовались для определения структуры белка и межбелковых взаимодействий. [12] Глубокое мутационное сканирование является примером мультиплексного анализа вариантного эффекта (MAVE), семейства методов, которые включают мутагенез кодируемого ДНК белка или регуляторного элемента с последующим мультиплексным анализом некоторых аспектов функции. MAVE позволяют создавать «вариантные карты эффектов», характеризующие аспекты функции каждого возможного отдельного нуклеотидного изменения в интересующем гене или функциональном элементе. [13]
Мутагенез и фенотипирование [ править ]
Важной функциональной особенностью генов является фенотип, обусловленный мутациями. Мутанты могут быть получены путем случайных мутаций или путем направленного мутагенеза, включая сайт-направленный мутагенез, удаление полных генов или другие методы.
Нокауты (делеции генов) [ править ]
Функцию генов можно исследовать, систематически «выбивая» гены один за другим. Это делается путем делеции или нарушения функции (например, путем инсерционного мутагенеза ), и полученные организмы проверяются на предмет фенотипов, которые дают ключ к пониманию функции нарушенного гена. Нокауты были произведены для целых геномов, то есть путем удаления всех генов в геноме. Для существенных генов это невозможно, поэтому используются другие методы, например, удаление гена во время экспрессии гена из плазмиды , использование индуцируемого промотора, так что уровень генного продукта может быть изменен по желанию (и, таким образом, «функциональное "удаление достигнуто).
Сайт-направленный мутагенез [ править ]
Сайт-направленный мутагенез используется для мутации определенных оснований (и, следовательно, аминокислот ). Это очень важно для исследования функции определенных аминокислот в белке, например, в активном центре фермента .
РНКи [ править ]
Методы РНК-интерференции (РНКи) могут использоваться для временного подавления или нокдауна экспрессии генов с использованием двухцепочечной РНК длиной ~20 пар оснований, обычно доставляемой путем трансфекции синтетических ~20-мерных молекул короткоинтерферирующей РНК (миРНК) или кодируемых вирусом коротких шпильковые РНК (shRNA). Скрининг РНКи, обычно выполняемый в анализах на основе клеточных культур или экспериментальных организмов (таких как C. elegans ), может использоваться для систематического разрушения почти каждого гена в геноме или подмножеств генов (субгеномов); возможные функции нарушенных генов могут быть определены на основе наблюдаемых фенотипов .
CRISPR-экраны [ править ]
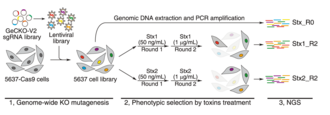
CRISPR-Cas9 использовался для мультиплексного удаления генов в клеточных линиях. Количественная оценка количества направляющих РНК для каждого гена до и после эксперимента может указывать на важные гены. Если направляющая РНК разрушает важный ген, это приведет к потере этой клетки и, следовательно, после скрининга произойдет истощение этой конкретной направляющей РНК. В недавнем эксперименте CRISPR-cas9 на клеточных линиях млекопитающих было обнаружено, что около 2000 генов являются важными для нескольких клеточных линий. [15] [16] Некоторые из этих генов были необходимы только в одной клеточной линии. Большинство генов входят в состав мультибелковых комплексов. Этот подход можно использовать для выявления синтетической летальности с использованием соответствующего генетического фона. CRISPRi и CRISPRa позволяют экранировать потерю и приобретение функции аналогичным образом. CRISPRi идентифицировал около 2100 основных генов в клеточной линии K562. [17] [18] Скрининг делеции CRISPR также использовался для выявления потенциальных регуляторных элементов гена. Например, была опубликована методика ScanDel, в которой применялся этот подход. Авторы удалили области за пределами интересующего гена (HPRT1, вовлеченного в менделевское расстройство) в попытке идентифицировать регуляторные элементы этого гена. [19] Гассперини и др. не выявили каких-либо дистальных регуляторных элементов для HPRT1 с использованием этого подхода, однако такие подходы могут быть распространены на другие представляющие интерес гены.
Функциональные аннотации для генов [ править ]
Аннотация генома [ править ]
Предполагаемые гены можно идентифицировать путем сканирования генома на предмет областей, которые, вероятно, кодируют белки, на основе таких характеристик, как длинные открытые рамки считывания , последовательности инициации транскрипции и полиаденилирования сайты . Последовательность, идентифицированная как предполагаемый ген, должна быть подтверждена дополнительными доказательствами, такими как сходство с последовательностями кДНК или EST из того же организма, сходство предсказанной последовательности белка с известными белками, связь с последовательностями промотора или свидетельство того, что мутация последовательности приводит к наблюдаемый фенотип.
камня Подход Розеттского
Подход Розеттского камня — это вычислительный метод прогнозирования функции белка de novo. В его основе лежит гипотеза о том, что некоторые белки, участвующие в том или ином физиологическом процессе, могут существовать в виде двух отдельных генов в одном организме и в виде одного гена в другом. Геномы сканируются на предмет последовательностей, которые независимы в одном организме и в одной открытой рамке считывания в другом. Если два гена слились, предполагается, что они имеют схожие биологические функции, которые делают такую совместную регуляцию выгодной.
Методы биоинформатики для функциональной геномики
Из-за большого количества данных, полученных с помощью этих методов, и желания найти биологически значимые закономерности, биоинформатика имеет решающее значение для анализа данных функциональной геномики. Примерами методов этого класса являются кластеризация данных или анализ главных компонентов для машинного обучения без учителя (обнаружение классов), а также искусственные нейронные сети или машины опорных векторов для машинного обучения с учителем (прогнозирование классов, классификация ). Анализ функционального обогащения используется для определения степени сверх- или недостаточной экспрессии (положительных или отрицательных регуляторов в случае скрининга РНКи) функциональных категорий по сравнению с фоновыми наборами. Анализ обогащения на основе онтологии генов предоставляется DAVID и анализом обогащения набора генов (GSEA). [20] анализ на основе путей от Ingenuity [21] и студия Pathway [22] и анализ на основе белковых комплексов с помощью COMPLEAT. [23]

Были разработаны новые вычислительные методы для понимания результатов эксперимента по глубокому мутационному сканированию. «phydms» сравнивает результат эксперимента по глубокому мутационному сканированию с филогенетическим деревом. [24] Это позволяет пользователю сделать вывод, применяет ли процесс отбора в природе аналогичные ограничения к белку, как показывают результаты глубокого мутационного сканирования. Это может позволить экспериментатору выбирать между различными экспериментальными условиями в зависимости от того, насколько хорошо они отражают природу. Глубокое мутационное сканирование также использовалось для вывода о межбелковых взаимодействиях. [25] Авторы использовали термодинамическую модель для прогнозирования эффектов мутаций в различных частях димера. Глубокая мутационная структура также может быть использована для определения структуры белка. Сильный положительный эпистаз между двумя мутациями при глубоком мутационном сканировании может указывать на то, что две части белка расположены близко друг к другу в трехмерном пространстве. Эту информацию затем можно использовать для определения структуры белка. Доказательство принципа этого подхода было продемонстрировано двумя группами, использующими белок GB1. [26] [27]
Результаты экспериментов MPRA потребовали применения подходов машинного обучения для интерпретации данных. Модель SVM k-меров с пробелами использовалась для вывода о кмерах, которые обогащены цис-регуляторными последовательностями с высокой активностью по сравнению с последовательностями с более низкой активностью. [28] Эти модели обеспечивают высокую предсказательную силу. Для интерпретации результатов этих многомерных экспериментов также использовались подходы глубокого обучения и случайного леса. [29] Эти модели начинают помогать лучше понимать функцию некодирующей ДНК в отношении регуляции генов.
Проекты консорциума [ править ]
Проект ENCODE [ править ]
Проект ENCODE . (Энциклопедия элементов ДНК) — это углубленный анализ генома человека, целью которого является выявление всех функциональных элементов геномной ДНК, как в кодирующих, так и в некодирующих областях Важные результаты включают в себя доказательства, полученные с помощью массивов геномных плиток, о том, что большинство нуклеотидов транскрибируются в виде кодирующих транскриптов, некодирующих РНК или случайных транскриптов, открытие дополнительных сайтов регуляции транскрипции, дальнейшее выяснение механизмов модификации хроматина.
Экспрессия генотипа-ткани» ( Проект « GTEx )

Проект GTEx — это проект генетики человека, направленный на понимание роли генетических вариаций в формировании вариаций транскриптома в разных тканях. В рамках проекта было собрано множество образцов тканей (более 50 различных тканей) от более чем 700 посмертных доноров. В результате было собрано более 11 000 образцов. GTEx помог понять совместное использование тканей и тканеспецифичность eQTL . [30] Геномный ресурс был разработан, чтобы «обогатить наше понимание того, как различия в последовательности нашей ДНК способствуют здоровью и болезням». [31]
Альянс Вариантов Атласа Эффектов
Альянс «Атлас вариантов эффектов» (AVE), [32] Основанный в 2020 году, это международный консорциум, цель которого каталогизировать влияние всех возможных генетических вариантов на функциональную геномику, связанную с заболеваниями, путем создания карт эффектов вариантов, которые раскрывают функцию каждого возможного отдельного нуклеотидного изменения в гене или регуляторном элементе. AVE частично финансируется Институтом Бротмана Бати при Вашингтонском университете и Национальным институтом исследований генома человека за счет гранта Центра передового опыта в области геномных наук (NHGRI RM1HG010461). [33]
См. также [ править ]
Ссылки [ править ]
- ^ Граур Д., Чжэн Ю., Прайс Н., Азеведо Р.Б., Зуфалл Р.А., Эльхайк Э. (20 февраля 2013 г.). «О бессмертии телевизоров: «функция» в человеческом геноме согласно неэволюционному евангелию ENCODE» . Геномная биология и эволюция . 5 (3): 578–90. дои : 10.1093/gbe/evt028 . ПМЦ 3622293 . ПМИД 23431001 .
- ^ Гибсон Г, Муза СВ. Букварь по геномной науке (3-е изд.). Сандерленд, Массачусетс: Sinauer Associates.
- ^ Певснер Дж (2009). Биоинформатика и функциональная геномика (2-е изд.). Хобокен, Нью-Джерси: Уайли-Блэквелл. ISBN 9780470085851 .
- ^ Ван Х, Мэйхью Д., Чен X, Джонстон М., Митра Р.Д. (май 2011 г.). «Визитные карточки позволяют мультиплексно идентифицировать геномные мишени ДНК-связывающих белков» . Геномные исследования . 21 (5): 748–55. дои : 10.1101/гр.114850.110 . ПМК 3083092 . ПМИД 21471402 .
- ^ Хрдличкова Р., Толуэ М., Тиан Б. (январь 2017 г.). «Методы RNA-Seq для анализа транскриптома» . Междисциплинарные обзоры Wiley: РНК . 8 (1): e1364. дои : 10.1002/wrna.1364 . ПМЦ 5717752 . ПМИД 27198714 .
- ^ Квасниески Дж.К., Фиоре С., Чаудхари Х.Г., Коэн Б.А. (октябрь 2014 г.). «Высокопроизводительное функциональное тестирование прогнозов сегментации ENCODE» . Геномные исследования . 24 (10): 1595–602. дои : 10.1101/гр.173518.114 . ПМК 4199366 . ПМИД 25035418 .
- ^ Патвардхан Р.П., Хиатт Дж.Б., Виттен Д.М., Ким М.Дж., Смит Р.П., Мэй Д. и др. (февраль 2012 г.). «Массовое параллельное функциональное рассечение энхансеров млекопитающих in vivo» . Природная биотехнология . 30 (3): 265–70. дои : 10.1038/nbt.2136 . ПМК 3402344 . ПМИД 22371081 .
- ^ Арнольд С.Д., Герлах Д., Стельцер С., Боринь Л.М., Рат М., Старк А. (март 2013 г.). «Карты количественной активности энхансеров по всему геному, определенные с помощью STARR-seq». Наука . 339 (6123): 1074–7. Бибкод : 2013Sci...339.1074A . дои : 10.1126/science.1232542 . ПМИД 23328393 . S2CID 54488955 .
- ^ Филдс С., Сонг О (июль 1989 г.). «Новая генетическая система для обнаружения белок-белковых взаимодействий». Природа . 340 (6230): 245–6. Бибкод : 1989Natur.340..245F . дои : 10.1038/340245a0 . ПМИД 2547163 . S2CID 4320733 .
- ^ Арайя С., Фаулер Д. (29 сентября 2011 г.). «Глубокое мутационное сканирование: оценка функции белка в огромных масштабах» . Тенденции в биотехнологии . 29 (9): 435–442. дои : 10.1016/j.tibtech.2011.04.003 . ПМК 3159719 . ПМИД 21561674 .
- ^ Пенн В.Д., Макки А.Г., Кунц С.П., Вудс Х., Нэш В., Грюнхаген Т.С. и др. (март 2020 г.). «Изучение ограничений биофизической последовательности в трансмембранных доменах родопсина путем глубокого мутационного сканирования» . Научный адв . 6 (10): eaay7505. Бибкод : 2020SciA....6.7505P . дои : 10.1126/sciadv.aay7505 . ПМК 7056298 . ПМИД 32181350 .
- ^ Роллинз Н., Брок К., Поелвейк Ф., Маркс Д. (2019). «Определение трехмерной структуры белка на основе сканирования глубоких мутаций» . Природная генетика . 51 (7): 1170–1176. дои : 10.1038/s41588-019-0432-9 . ПМК 7295002 . ПМИД 31209393 .
- ^ Фаулер Д.М., Адамс DJ, Глойн А.Л., Старита Л. (2023). «Атлас вариантов эффектов для понимания генома с разрешением нуклеотидов» . Геномная биология . 24 (1): 147. дои : 10.1186/s13059-023-02986-x . ПМЦ 10316620 . ПМИД 37394429 .
- ^ Тиан С., Мунируддин К., Чой М.Ю., Тао Л., Бхуян Р.Х., Оми Ю. и др. (27 ноября 2018 г.). «Полногеномный CRISPR-скрининг на шига-токсины и рицин выявляет белки Гольджи, имеющие решающее значение для гликозилирования» . ПЛОС Биология . 16 (11). е2006951. doi : 10.1371/journal.pbio.2006951 . ПМК 6258472 . ПМИД 30481169 .
- ^ Харт Т., Чандрашекхар М., Ареггер М., Стейнхарт З., Браун К.Р., МакЛауд Г. и др. (декабрь 2015 г.). «Экраны CRISPR высокого разрешения выявляют гены фитнеса и генотипическую предрасположенность к раку» . Клетка . 163 (6): 1515–26. дои : 10.1016/j.cell.2015.11.015 . ПМИД 26627737 .
- ^ Шалем О., Санджана Н.Е., Хартениан Э., Ши Х., Скотт Д.А., Миккельсон Т. и др. (январь 2014 г.). «Геномный скрининг нокаута CRISPR-Cas9 в клетках человека» . Наука . 343 (6166): 84–87. Бибкод : 2014Sci...343...84S . дои : 10.1126/science.1247005 . ПМК 4089965 . ПМИД 24336571 .
- ^ Гилберт Л.А., Хорлбек М.А., Адамсон Б., Виллалта Дж.Э., Чен Ю., Уайтхед Э.Х. и др. (октябрь 2014 г.). «CRISPR-опосредованный контроль репрессии и активации генов в масштабе генома» . Клетка . 159 (3): 647–61. дои : 10.1016/j.cell.2014.09.029 . ПМЦ 4253859 . ПМИД 25307932 .
- ^ Хорлбек М.А., Гилберт Л.А., Виллалта Дж.Э., Адамсон Б., Пак Р.А., Чен Ю. и др. (сентябрь 2016 г.). «Компактные и высокоактивные библиотеки нового поколения для репрессии и активации генов, опосредованных CRISPR» . электронная жизнь . 5 . doi : 10.7554/eLife.19760 . ПМК 5094855 . ПМИД 27661255 .
- ^ Гасперини М., Финдли Г.М., Маккенна А., Милбанк Дж.Х., Ли С., Чжан М.Д. и др. (август 2017 г.). «CRISPR/Cas9-опосредованное сканирование регуляторных элементов, необходимых для экспрессии HPRT1, посредством тысяч крупных запрограммированных геномных делеций» . Американский журнал генетики человека . 101 (2): 192–205. дои : 10.1016/j.ajhg.2017.06.010 . ПМЦ 5544381 . ПМИД 28712454 .
- ^ Субраманиан А., Тамайо П., Мута В.К., Мукерджи С., Эберт Б.Л., Джилетт М.А. и др. (октябрь 2005 г.). «Анализ обогащения генного набора: основанный на знаниях подход к интерпретации профилей экспрессии в масштабах всего генома» . Труды Национальной академии наук Соединенных Штатов Америки . 102 (43): 15545–50. Бибкод : 2005PNAS..10215545S . дои : 10.1073/pnas.0506580102 . ПМЦ 1239896 . ПМИД 16199517 .
- ^ «Системы изобретательности» . Архивировано из оригинала 25 января 1999 г. Проверено 31 декабря 2007 г.
- ^ «Ариадна Геномика: Студия Pathway» . Архивировано из оригинала 30 декабря 2007 г. Проверено 31 декабря 2007 г.
- ^ Винаягам А., Ху Ю., Кулкарни М., Розель С., Сопко Р., Мор С.Э. и др. (февраль 2013 г.). «Среда анализа на основе белковых комплексов для наборов данных с высокой пропускной способностью» . Научная сигнализация . 6 (264): сс5. дои : 10.1126/scisignal.2003629 . ПМЦ 3756668 . ПМИД 23443684 .
- ^ Хилтон С.К., Дауд М.Б., Блум Дж.Д. (2017). «phydms: программное обеспечение для филогенетического анализа, основанного на глубоком мутационном сканировании» . ПерДж . 5 : е3657. дои : 10.7717/peerj.3657 . ПМК 5541924 . ПМИД 28785526 .
- ^ Дисс Г., Ленер Б. (апрель 2018 г.). «Генетический ландшафт физического взаимодействия» . электронная жизнь . 7 . дои : 10.7554/eLife.32472 . ПМЦ 5896888 . ПМИД 29638215 .
- ^ Шмидель Дж. М., Ленер Б. (июль 2019 г.). «Определение белковых структур методами глубокого мутагенеза» . Природная генетика . 51 (7): 1177–1186. дои : 10.1038/s41588-019-0431-x . ПМК 7610650 . ПМИД 31209395 .
- ^ Роллинз Н.Дж., Брок К.П., Полвейк Ф.Дж., Стифлер М.А., Готье Н.П., Сандер С. и др. (июль 2019 г.). «Определение трехмерной структуры белка на основе сканирования глубоких мутаций» . Природная генетика . 51 (7): 1170–1176. дои : 10.1038/s41588-019-0432-9 . ПМК 7295002 . ПМИД 31209393 .
- ^ Ганди М., Ли Д., Мохаммад-Нури М., Бир М.А. (июль 2014 г.). «Улучшенное предсказание регуляторных последовательностей с использованием особенностей k-меров с пробелами» . PLOS Вычислительная биология . 10 (7): e1003711. Бибкод : 2014PLSCB..10E3711G . дои : 10.1371/journal.pcbi.1003711 . ПМК 4102394 . ПМИД 25033408 .
- ^ Ли Ю, Ши В, Вассерман В.В. (май 2018 г.). «Полногеномное предсказание цис-регуляторных регионов с использованием контролируемых методов глубокого обучения» . БМК Биоинформатика . 19 (1): 202. дои : 10.1186/s12859-018-2187-1 . ПМЦ 5984344 . ПМИД 29855387 .
- ^ Батл А, Браун К.Д., Энгельхардт Б.Е., Монтгомери С.Б. и др. (Консорциум GTEx) (октябрь 2017 г.). «Генетическое влияние на экспрессию генов в тканях человека» . Природа . 550 (7675): 204–213. Бибкод : 2017Natur.550..204A . дои : 10.1038/nature24277 . ПМК 5776756 . ПМИД 29022597 .
- ^ «GTEx создает эталонный набор данных для изучения генетических изменений и экспрессии генов» . Управление стратегической координации – Общий фонд . Национальные институты здравоохранения США. 8 февраля 2018 года . Проверено 13 января 2022 г.
- ^ «Атлас Альянса вариантов эффектов» . Реестр исследовательских организаций .
- ^ «Ученые запускают «геркулесовый» проект по созданию атласа вариантов генома человека | Институт Бротмана Батыя» . brotmanbaty.org . Проверено 5 февраля 2024 г.
Внешние ссылки [ править ]
- Программа Европейского научного фонда на передовых рубежах функциональной геномики
- MUGEN NoE — Интегрированная функциональная геномика в моделях мутантных мышей
- Прозрение природы: функциональная геномика
- КОДИРОВАТЬ