ДНК штрих -кодирование

| Часть серии на |
| ДНК штрих -кодирование |
|---|
 |
| Таксонами |
| Другой |
Шадкодирование ДНК является методом идентификации видов с использованием короткого участка ДНК из определенного гена или генов. Предпосылка штрих -кодирования ДНК заключается в том, что по сравнению с эталонной библиотекой таких секций ДНК (также называемых « последовательностями »), отдельная последовательность может быть использована для однозначного идентификации организма для видов, так же, как сканер супермаркета использует знакомые черные полосы Шалмный код UPC для определения элемента в его запасе против своей справочной базы данных. [ 1 ] Эти «штрих -коды» иногда используются в попытке выявить неизвестные виды или части организма, просто для каталогизации как можно большего количества таксонов или для сравнения с традиционной таксономией, чтобы определить границы видов. [ 2 ]
Различные области генов используются для идентификации различных организмов с использованием штрих -кодирования. Наиболее часто используемая область штрих -кода для животных и некоторых протистов является частью гена цитохрома Ссидазы I (COI или COX1 ), обнаруженного в митохондриальной ДНК . Другими генами, подходящими для штрих -кодирования ДНК, являются внутренняя транскрибированная прокладная (ITS) рРНК, часто используемая для грибов и Rubisco, используемых для растений. [ 3 ] [ 4 ] [ 5 ] Микроорганизмы обнаруживаются с использованием различных областей генов. ген 18S Например, ген 16S рРНК широко используется для идентификации прокариот, тогда как рРНК в основном используется для обнаружения микробных эукариот . Эти генные области выбираются из -за того, что они имеют менее внутривидовые (внутри видовых) изменений, чем межвидовые (между видами) вариациями, которые известны как «разрыв штрих -кодирования». [ 6 ]
Некоторые применения штрих -кодирования ДНК включают в себя: идентификацию листьев растений, даже когда цветы или фрукты недоступны; идентификация пыльцы, собранная на телах опыляющих животных; идентификация личинок насекомых, которые могут иметь меньше диагностических признаков, чем взрослые; или исследование диеты животного на основе содержания желудка, слюны или кала. [ 7 ] Когда штрих -кодирование используется для идентификации организмов из образца, содержащего ДНК из более чем одного организма, термин метабардодирование ДНК , используется [ 8 ] [ 9 ] Например, ДНК метабардодирование сообществ диатомовых сообществ в реках и ручьях, которая используется для оценки качества воды. [ 10 ]
Фон
[ редактировать ]Методы штрих -кодирования ДНК были разработаны на основе ранних работ секвенирования ДНК на микробных сообществах с использованием гена 5S рРНК . [ 11 ] В 2003 году конкретные методы и терминология современного штрих -кодирования ДНК были предложены в качестве стандартизированного метода для идентификации видов, а также потенциально выделяют неизвестные последовательности на более высокие таксоны, такие как заказы и Phyla, в статье Paul Dn Hebert et al. из Университета Гвельфа , Онтарио , Канада . [ 12 ] Хеберт и его коллеги продемонстрировали полезность гена цитохрома с оксидазы I (COI), впервые используемого Folmer et al. В 1994 году с использованием их опубликованных праймеров ДНК в качестве инструмента для филогенетических анализов на уровнях видов [ 12 ] в качестве подходящего дискриминационного инструмента между метазоанскими беспозвоночными. [ 13 ] «Фольмерная область» гена COI обычно используется для различия между таксонами на основе его моделей изменения на уровне ДНК. Относительная простота извлечения последовательности, а вариабельность, смешанная с сохранением между видами, являются одними из преимуществ COI. Называя профили «штрих -коды», Hebert et al. Предусмотрено разработку базы данных COI, которая может служить основой для «глобальной системы биоидентификации».
Методы
[ редактировать ]Выборка и сохранение
[ редактировать ]Штрих -кодирование может быть сделано из ткани из образца -мишени, из смеси организмов (объемный образец) или из ДНК, присутствующей в образцах окружающей среды (например, вода или почва). Методы выборки, сохранения или анализа различаются между этими различными типами образца.
Образцы ткани
Для штрих -кодирования образца ткани из образца -мишени, небольшого кусочка кожи, масштаба, ноги или антенны, вероятно, будет достаточно (в зависимости от размера образца). Чтобы избежать загрязнения, необходимо стерилизовать используемые инструменты между образцами. Рекомендуется собрать два образца из одного образца, один для архива и один для процесса штрих -кодирования. Сохранение выборки имеет решающее значение для преодоления проблемы деградации ДНК.
Объемные образцы
Образец - это тип экологической выборки, содержащей несколько организмов из таксономической группы исследуемой . Разница между объемными образцами (в том смысле, используемом здесь) и другими образцами окружающей среды состоит в том, что объемный образец обычно обеспечивает большое количество ДНК хорошего качества. Примеры объемных образцов включают образцы водных макробеспозвоночных, собранные с помощью Kick-Net или образцов насекомых, собранных с ловушкой недовольства. Отфильтрованные или фракционированные по размеру образцы воды, содержащие целые организмы, такие как одноклеточные эукариоты, также иногда определяются как объемные образцы. Такие образцы могут быть собраны с помощью тех же методов, которые использовались для получения традиционных образцов для идентификации на основе морфологии.
Эдна Образцы
Метод ДНК окружающей среды (EDNA) представляет собой неинвазивный подход для обнаружения и идентификации видов из клеточного мусора или внеклеточной ДНК, присутствующей в образцах окружающей среды (например, вода или почва) посредством штрих-кодирования или метабардодирования. Подход основан на том факте, что каждый живой организм оставляет ДНК в окружающей среде, и эта экологическая ДНК может быть обнаружена даже для организмов, которые находятся в очень низком численности. Таким образом, для отбора проб в полевых условиях наиболее важной частью является использование без ДНК материал и инструменты на каждом участке или образце, чтобы избежать загрязнения, если ДНК целевого организма (ов) может присутствовать в низких количествах. С другой стороны, образец Эдны всегда включает в себя ДНК цельных клеток, живых микроорганизмов, которые часто присутствуют в больших количествах. Следовательно, образцы микроорганизма, взятые в природной среде, также называются образцами EDNA, но загрязнение менее проблематично в этом контексте из -за большого количества целевых организмов. Метод EDNA применяется на большинстве типов образцов, таких как вода, осадок, почва, фекалии животных, содержание желудка или кровь от EG. [ 14 ]
Экстракция, амплификация и секвенирование ДНК
[ редактировать ]ДНК штрих -кодирование требует, чтобы ДНК в образце извлекали. Существуют несколько различных методов экстракции ДНК , и такие факторы, как стоимость, время, тип выборки и урожайность, влияют на выбор оптимального метода.
Когда ДНК из образцов организма или EDNA амплифицируется с использованием полимеразной цепной реакции (ПЦР), реакция может быть отрицательно влиять на молекулы ингибитора, содержащиеся в образце. [ 15 ] Удаление этих ингибиторов имеет решающее значение для обеспечения того, чтобы высококачественная ДНК была доступна для последующего анализа.
Усиление экстрагированной ДНК является обязательным шагом в штрих -кодировании ДНК. Как правило, только небольшой фрагмент общего материала ДНК секвенирует (как правило, 400–800 пар оснований ) [ 16 ] Чтобы получить штрих -код ДНК. Усиление материала EDNA обычно фокусируется на меньших размерах фрагментов (<200 пар оснований), так как EDNA с большей вероятностью будет фрагментирована, чем материал ДНК из других источников. Тем не менее, некоторые исследования утверждают, что нет никакой связи между размером ампликона и скоростью обнаружения EDNA. [ 17 ] [ 18 ]

Когда область маркера штрих -кода ДНК была амплифицирована, следующим шагом является последовательность маркера с использованием секвенирования ДНК . методов [ 19 ] Доступно много разных платформ секвенирования, и техническая разработка работает быстро.
Выбор маркера
[ редактировать ]
Маркеры, используемые для штрих -кодирования ДНК, называются штрих -кодами. Чтобы успешно охарактеризовать виды на основе штрих -кодов ДНК, выбор информативных областей ДНК имеет решающее значение. Хороший штрих-код ДНК должен иметь низкую внутриспецифическую и высокую межспецифическую изменчивость [ 12 ] и обладать консервативными фланговыми сайтами для разработки универсальных ПЦР праймеров для широкого таксономического применения. Цель состоит в том, чтобы спроектировать праймеры, которые будут обнаружить и различать большинство или все виды в исследуемой группе организмов (высокое таксономическое разрешение). Длина последовательности штрих -кода должна быть достаточно короткой, чтобы использоваться с источником отбора проб тока, экстракции ДНК , амплификации и секвенирования . методами [ 20 ]
В идеале одна последовательность генов будет использоваться для всех таксономических групп, от вирусов до растений и животных . Тем не менее, такая область гена еще не обнаружена, поэтому для разных групп организмов используются разные штрих -коды. [ Цитация необходима ] или в зависимости от вопроса об обучении.
Для животных наиболее широко используемым штрих -кодом является митохондриальный цитохром С оксидаза I ( COI ). [ 21 ] Другие митохондриальные гены, такие как CYTB , 12S или 16S также используются. Митохондриальные гены предпочтительнее ядерных генов из -за отсутствия интронов , их гаплоидного мода наследования и их ограниченной рекомбинации . [ 21 ] [ 22 ] Более того, каждая клетка имеет различные митохондрии (до нескольких тысяч), и каждая из них содержит несколько круговых молекул ДНК . Поэтому митохондрии могут предлагать обильный источник ДНК, даже когда ткань образца ограничена. [ Цитация необходима ]
Однако у растений митохондриальные гены не подходят для штрих -кодирования ДНК, потому что они демонстрируют низкую скорость мутаций . [ 23 ] Несколько генов -кандидатов были обнаружены в геноме хлоропласта , наиболее перспективным является ген Maturase K ( MATK ) само по себе или в связи с другими генами. внутренние маркеры , такие как рибосомные транскрибированные проставки (ее ДНК) вместе с MATK , RBCL , TRNH или другими генами. Для идентификации видов также использовались [ Цитация необходима ] Лучшая дискриминация между видами растений была достигнута при использовании двух или более штрих -кодов хлоропластов. [ 24 ]
Для бактерий небольшая субъединица гена рибосомной РНК ( 16S ) может использоваться для различных таксонов, так как она высоко консервативна. [ 25 ] Некоторые исследования показывают COI , [ 26 ] типа II Шаперонин ( CPN60 ) [ 27 ] или β -субъединица РНК -полимеразы ( RPOB ) [ 28 ] Также может служить бактериальными штрих -кодами ДНК.
Штриховые грибы более сложные, и может потребоваться более одного комбинации праймеров. [ 29 ] Маркер COI хорошо работает в определенных группах грибов, [ 30 ] но не одинаково хорошо в других. [ 31 ] Следовательно, используются дополнительные маркеры, такие как ее рДНК и большая субъединица ядерной рибосомной РНК (28S LSU RRNA). [ 32 ]
В группе протистов были предложены различные штрих -коды, такие как области D1 - D2 или D2 - D3 28S рДНК , субрегион V4 18S RRNA , ее рДНК и COI . Кроме того, некоторые специфические штрих-коды могут использоваться для фотосинтетических протистов, например, большой субъединицы гена рибралезы-1,5-бисфосфат-карбоксилаз-оксигеназы ( RBCL ) и гена хлоропластического 23S рРНК . [ Цитация необходима ]
Справочные библиотеки и биоинформатика
[ редактировать ]Справочные библиотеки используются для таксономической идентификации, также называемой аннотацией последовательностей, полученных из штрих -кодирования или метабардода. Эти базы данных содержат штрих -коды ДНК, назначенные ранее идентифицированным таксонам. Большинство справочных библиотек не охватывают все виды в группе организма, и новые записи постоянно создаются. В случае макро- и многих микроорганизмов (таких как водоросли) эти справочные библиотеки требуют подробной документации (местоположение и дату отбора проб, лицо, которое его собрало, изображение и т. Д.) и авторитетную таксономическую идентификацию образца ваучера, а также подача последовательностей в определенном формате. Однако такие стандарты выполняются только для небольшого числа видов. Процесс также требует хранения образцов ваучеров в музейных коллекциях, гербарии и других сотрудничающих учреждениях. Как таксономически комплексное покрытие, так и качество контента важны для точности идентификации. [ 33 ] В микробном мире нет информации о ДНК для большинства названий видов, и многие последовательности ДНК не могут быть назначены каким -либо биномиалам Linnaean . [ 34 ] Существуют несколько эталонных баз данных в зависимости от группы организма и используемого генетического маркера. Существуют меньшие, национальные базы данных (например, Finbol) и крупные консорциумы, такие как Международный проект штрих -кода жизни (IBOL). [ 35 ]
Запущен в 2007 году, штрих -код системы Life Data System (BOLD) [ 36 ] является одной из самых больших баз данных, содержащих около 780 000 бункеров (числа индексов штрих -кода). Это свободно доступный репозиторий для записей образца и последовательности для исследований штрих -кода, а также рабочая сумка, помогающая управлению, обеспечению качества и анализу данных штрих -кода. База данных в основном содержит записи бин для животных на основе генетического маркера COI. Для идентификации растений жирный жирный цвет принимает последовательности от Matk и RBCL .
База данных UNITE [ 37 ] был запущен в 2003 году и является эталонной базой данных для молекулярной идентификации грибковых (и с 2018 года всех эукариотических) видов с ядерной рибосомной внутренней транскрибированной областью генетического маркера (ITS). Эта база данных основана на концепции гипотез видов: вы выбираете %, в котором вы хотите работать, а последовательности сортируются по сравнению с последовательностями, полученными из образцов ваучеров, идентифицированных экспертами.
Diat.Barcode [ Постоянная мертвая ссылка ]
Diat.Barcode [ 38 ] База данных была впервые опубликована под названием R-Syst :: Diatom [ 39 ] В 2016 году начиная с данных из двух источников: Коллекция культуры Thonon (TCC) на гидробиологической станции Французского Национального института сельскохозяйственных исследований (INRA) и из базы данных NCBI (Национальный центр биотехнологической информации). Diat.Barcode предоставляет данные для двух генетических маркеров: RBC L (рибулоза-1,5-бисфосфат карбоксилаза/оксигеназа) и 18S (18S рибосомная РНК). База данных также включает в себя дополнительную информацию о видах, такую как морфологические характеристики (биолум, размеры и т. Д.), Формы жизни (подвижность, тип колоний и т. Д.) Или экологические особенности (чувствительность к загрязнению и т. Д.).
Биоинформатический анализ
[ редактировать ]Чтобы получить хорошо структурированные, чистые и интерпретируемые данные, необработанные данные секвенирования должны обрабатываться с использованием биоинформатического анализа. Файл FASTQ с данными секвенирования содержит два типа информации: последовательности, обнаруженные в выборке ( файл FASTA ) и качественный файл с качественными показателями ( оценки PHRED ), связанные с каждым нуклеотидом каждой последовательности ДНК. Оценки PHRED указывают на вероятность, с которой соответствующий нуклеотид был правильно оценен.
| 10 | 90% |
| 20 | 99% |
| 30 | 99.9% |
| 40 | 99.99% |
| 50 | 99.999% |
В целом, оценка PHRED уменьшается к концу каждой последовательности ДНК. Таким образом, некоторые биоинформатические трубопроводы просто сокращают конец последовательностей на определенном пороге.
Некоторые технологии секвенирования, такие как MiseQ, используют парное секвенирование, в течение которого секвенирование выполняется из обоих направлений, производящих более качественное качество. Перекрывающиеся последовательности затем выровнены в контиги и объединяются. Обычно несколько образцов объединяются за один пробег, и каждый образец характеризуется коротким фрагментом ДНК, меткой. На стадии демольтиплексирования последовательности отсортируются с использованием этих тегов, чтобы собрать отдельные образцы. Перед дальнейшим анализом теги и другие адаптеры удаляются из фрагмента ДНК штрих -последовательности. Во время обрезки последовательности плохого качества (низкие оценки PHRED) или последовательности, которые намного короче или длиннее целевого штрих -кода ДНК, удаляются. Следующим шагом дерепликации является процесс, в котором все последовательности, фильтрованные качеством, развернуты в наборе уникальных считываний (отдельные единицы последовательности ISU) с информацией о их изобилии в образцах. После этого химеры (т.е. составные последовательности, образованные из кусочков смешанного происхождения) обнаруживаются и удаляются. Наконец, последовательности сгруппированы в OTU (операционные таксономические единицы), используя одну из многих стратегий кластеризации. Наиболее часто используемое биоинформационное программное обеспечение включает Mothur, [ 40 ] Uparse, [ 41 ] Ценить, [ 42 ] Галактика, [ 43 ] Обитулс, [ 44 ] Но джем, японец. [ 45 ] Барк, [ 46 ] и дада2. [ 47 ]
Сравнение численности чтения, IE -последовательностей, между различными образцами по -прежнему является проблемой, поскольку общее количество чтения в выборке, так и относительное количество чтения для вида могут варьироваться между образцами, методами или другими переменными. Для сравнения, затем может затем уменьшить количество чтения каждого образца до минимального количества считываний сравниваемых образцов - процесс, называемый разреженным делом. Другой способ - использовать относительное изобилие чтения. [ 48 ]
Идентификация видов и таксономическое назначение
[ редактировать ]Таксономическое назначение OTU к видам достигается путем сопоставления последовательностей с эталонными библиотеками. Основной инструмент поиска локального выравнивания (BLAST) обычно используется для идентификации областей сходства между последовательностями путем сравнения считываний последовательностей из образца с последовательностями в эталонных базах данных. [ 49 ] Если эталонная база данных содержит последовательности соответствующих видов, то последовательности образцов могут быть идентифицированы до уровня видов. Если последовательность не может быть сопоставлена с существующей записью справочной библиотеки, для создания новой записи может использоваться штрих -кодирование ДНК.
В некоторых случаях из -за неполноты эталонных баз данных идентификация может быть достигнута только на более высоких таксономических уровнях, таких как назначение семье или классу. В некоторых группах организма, таких как бактерии, таксономическое назначение уровню видов часто невозможно. В таких случаях выборка может быть назначена конкретной оперативной таксономической единице (OTU) .
В некоторых случаях образцы с идентичными (COI) штрих -кодами ДНК явно принадлежат к различным видам, например, видам рода рыбы . [ 50 ]
Приложения
[ редактировать ]Применение штрих -кодирования ДНК включает идентификацию новых видов , оценку безопасности пищи, идентификация и оценка загадочных видов, обнаружение инопланетных видов, идентификация исчезающих и угрожаемых видов , [ 51 ] Связывание яичных и личиночных стадий со взрослыми видами, обеспечение прав интеллектуальной собственности для биорезорпов, составление глобальных планов управления стратегиями сохранения, выяснение нишей кормления, [ 52 ] и судебная наука. [ 53 ] Маркеры штрих -кода ДНК могут быть применены для решения основных вопросов в области систематики, экологии , эволюционной биологии и сохранения , включая сборку сообщества, сети взаимодействия видов , таксономическое открытие и оценку приоритетных областей для защиты окружающей среды .
Идентификация видов
[ редактировать ]Конкретные короткие последовательности ДНК или маркеры из стандартизированной области генома могут обеспечить штрих -код ДНК для идентификации видов. [ 54 ] Молекулярные методы особенно полезны, когда традиционные методы не применимы. Штрих -кодирование ДНК обладает большой применимостью в идентификации личинок, для которых, как правило, мало доступных диагностических признаков, и в связи с различными этапами жизни (например, личинки и взрослые) у многих животных. [ 55 ] Идентификация видов, перечисленных в Приложении Международной торговли исчезающими видами ( CITE ), с использованием методов штрих -кодирования используется для мониторинга незаконной торговли. [ 56 ]
Обнаружение инвазивных видов
[ редактировать ]Чужой виды могут быть обнаружены с помощью штрих -кодирования. [ 57 ] [ 58 ] Штрих -кодирование может быть подходящим для обнаружения видов в EG -границе, где быстрая и точная морфологическая идентификация часто невозможно из -за сходства между различными видами, отсутствием достаточных диагностических характеристик [ 57 ] и/или отсутствие таксономической экспертизы. Штрих -кодирование и метабардодирование также могут использоваться для скрининга экосистем для инвазивных видов и для различения инвазивных видов и местных, морфологически сходных видов. [ 59 ] Высокая эффективность идентификации ДНК показана относительно традиционного мониторинга биологических вторжений. [ 60 ]
Разграничение загадочных видов
[ редактировать ]ДНК штрих -кодирование обеспечивает идентификацию и распознавание загадочных видов . [ 61 ] Результаты анализа штрих -кодирования ДНК зависят, однако от выбора аналитических методов, поэтому процесс разграничения загадочных видов с использованием штрих -кодов ДНК может быть столь же субъективным, как и любая другая форма таксономии . Hebert et al. (2004) пришли к выводу, что бабочка астратат астратат-фургонт на северо-западе Коста-Рики фактически состоит из 10 различных видов. [ 62 ] Эти результаты, однако, впоследствии были оспорены Brower (2006), который указал на многочисленные серьезные недостатки в анализе и пришел к выводу, что первоначальные данные могут поддержать не более, чем возможность от трех до семи загадочных таксонов , а не десять загадочных видов. [ 63 ] Smith et al. (2007) использовали штрих -коды ДНК -оксидазы цитохрома С оксидазы I для идентификации видов 20 морфоспеципов бельвозийских паразитоидных мух ( Diptera : Tachinidae ), выращенных из густопилларов ( Lepidoptera ) в районе De Conservación Guanacaste (ACG), Северная ветерская костюра. Эти авторы обнаружили, что штрих-кодирование поднимает количество видов до 32, показывая, что каждый из трех паразитоидных видов, ранее считавшихся универсалами, на самом деле представляют собой массивы специфических для хозяина криптических видов. [ 64 ] Для 15 морфоспеципов полихэтов в глубоком антарктическом бентосе , изученном с помощью штрих -кодирования ДНК, загадочное разнообразие было обнаружено в 50% случаев. Кроме того, было обнаружено 10 ранее пропущенных морфоспекций, что увеличило общее видовое богатство в выборке на 233%. [ 65 ]

Анализ диеты и применение пищевой сети
[ редактировать ]ДНК штрих -кодирование и метабардодирование могут быть полезны в исследованиях анализа диеты, [ 66 ] и обычно используется, если образцы добычи не могут быть идентифицированы на основе морфологических признаков. [ 67 ] [ 68 ] Существует ряд подходов отбора проб при анализе диеты: ДНК метабардодирование может проводиться на содержании желудка, [ 69 ] фекалии, [ 68 ] [ 70 ] слюна [ 71 ] или анализ всего тела. [ 51 ] [ 72 ] В образцах фекалий или с высоким расщепленным содержанием желудка часто невозможно отличить ткани от отдельных видов, и, следовательно, вместо этого можно применять метабардодирование. [ 68 ] [ 73 ] Фекалии или слюна представляют собой неинвазивные подходы к отбору отбора проб, в то время как анализ всего тела часто означает, что индивидуума нужно убить в первую очередь. Для более мелких организмов секвенирование для содержания желудка затем часто выполняется путем секвенирования всего животного.
Штрих для безопасности пищевых продуктов
[ редактировать ]Шаткодирование ДНК представляет собой важный инструмент для оценки качества пищевых продуктов. Цель состоит в том, чтобы гарантировать прослеживаемость продовольствия, свести к минимуму пиратство пищевого питания и ценить местное и типичное производство агропродуктов. Другая цель - защитить общественное здравоохранение; Например, метабардодирование предлагает возможность идентифицировать группировщики , вызывающие отравление рыбой сигуатеры из остатков еды, [ 74 ] или отделить ядовитые грибы от съедобных (ссылка).
Биомониторинг и экологическая оценка
[ редактировать ]ДНК штрих -кодирование может использоваться для оценки присутствия исчезающих видов для усилий по сохранению (REF) или наличия индикаторных видов, отражающих специфические экологические условия (REF), например, избыточные питательные вещества или низкие уровни кислорода.
Судебная наука
[ редактировать ]ДНК штрих -кодирование часто используется для идентификации видов в криминалистики случаях . Неизвестные образцы животных или растений на сценах преступности могут быть найдены, собраны и определены в надежде связать его с подозреваемым и получить осуждение. [ 75 ] Браконьерство , убийство исчезающих видов и злоупотребление животными являются примерами преступлений, в которых используется штрих -кодирование ДНК, поскольку часто обнаруживается ДНК животных. [ 53 ] [ 76 ] С другой стороны, ДНК растений обычно используется в качестве следов, чтобы связать подозреваемого на месте преступления. [ 77 ]
Потенциалы и недостатки
[ редактировать ]Потенциал
[ редактировать ]Традиционные методы биологической оценки хорошо известны на международном уровне и хорошо обслуживают биомониторинг, как, например, для водной биологической оценки в директивах ЕС WFD и MSFD . Тем не менее, штрих -кодирование ДНК может улучшить традиционные методы по следующим причинам; Шаткодирование ДНК (i) может увеличить таксономическое разрешение и гармонизировать идентификацию таксонов, которые трудно идентифицировать или отсутствовать экспертов, (ii) может более точно/точно связать факторы окружающей среды с конкретными таксонами (iii) может повысить сопоставимость между регионами, (iv) Позволяет включить стадии раннего жизни и фрагментированные образцы, (v) позволяет разграничить загадочные /редкие виды (VI) допускает развитие новых индексов, например, Редкие/загадочные виды, которые могут быть чувствительными/толерантными к стрессовым факторам , (VII) увеличивает количество образцов, которые могут быть обработаны, и сокращает время обработки, что приводит к повышению знаний о экологии видов, (VIII) является неинвазивным способом мониторинга при использовании Эдна Методы. [ 78 ]
Время и стоимость
[ редактировать ]Шаткодирование ДНК быстрее, чем традиционные морфологические методы в течение всего обучения до таксономического назначения. Требуется меньше времени, чтобы получить опыт в методах ДНК, чем становятся экспертом в области таксономии. Кроме того, рабочий процесс штрих -кодирования ДНК (то есть от образца к результату), как правило, быстрее, чем традиционный морфологический рабочий процесс, и позволяет обрабатывать больше образцов.
Таксономическое разрешение
[ редактировать ]Штрих -кодирование ДНК позволяет разрешить таксоны от более высоких (например, семейства (например,) до более низких (например, видов), которые в остальном слишком сложно идентифицировать с использованием традиционных морфологических методов, таких как идентификация, например, с помощью микроскопии. Например, Chironomidae (не кусочная среда) широко распространены как в наземных, так и в пресноводных экосистемах. Их богатство и изобилие делают их важными для экологических процессов и сетей, и они являются одной из многих групп беспозвоночных, используемых при биомониторинге. Образцы беспозвоночных могут содержать до 100 видов хирономидов, которые часто составляют до 50% образца. Несмотря на это, они обычно не идентифицируются ниже уровня семьи из -за таксономической экспертизы и требуемого времени. [ 79 ] Это может привести к различным видам хирономидов с различными экологическими предпочтениями, сгруппированными вместе, что приводит к неточной оценке качества воды.
Шаткодирование ДНК дает возможность разрешить таксоны и напрямую связывать эффекты стрессора с конкретными таксонами, такими как отдельные виды хирономидов. Например, Beermann et al. (2018) ДНК шаркодировали Chironomidae, чтобы исследовать их ответ на множественные стрессоры; Снижение потока, увеличение тонкого седимента и повышенная соленость. [ 80 ] После штрих -кодирования было обнаружено, что выборка хирономида состояла из 183 оперативных таксономических единиц (OTU), т.е. штрих -коды (последовательности), которые часто эквивалентны морфологическим видам. Эти 183 OTU показали 15 типов ответов, а не ранее сообщенные [ 81 ] Два типа ответов, зарегистрированные, когда все хирономиды были сгруппированы вместе в одном и том же исследовании с множественным стрессором. Аналогичная тенденция была обнаружена в исследовании Macher et al. (2016), который обнаружил загадочное разнообразие в новозеландских видах Mayfly Deleatidium sp . Это исследование обнаружило, что различные паттерны ответа 12 молекулярных различных OTU на стрессоры, которые могут изменить консенсус о том, что этот майфлай чувствителен к загрязнению. [ 82 ]
Недостатки
[ редактировать ]Несмотря на преимущества, предлагаемые штрих -кодированием ДНК, было также высказано предположение, что штрих -кодирование ДНК лучше всего используется в качестве дополнения к традиционным морфологическим методам. [ 78 ] Эта рекомендация основана на многочисленных воспринимаемых проблемах.
Физические параметры
[ редактировать ]Не совсем просто соединять штрих -коды ДНК с экологическими предпочтениями рассматриваемого штрих -кодирования, о котором идет речь, как это необходимо, если для биомониторинга следует использовать штрих -кодирование. Например, обнаружение ДНК -мишени в водных системах зависит от концентрации молекул ДНК в месте, что, в свою очередь, может зависеть от многих факторов. Наличие молекул ДНК также зависит от дисперсии на месте, например, направления или силы токов. На самом деле не известно, как ДНК движется в потоках и озерах, что затрудняет выборку. Другим фактором может быть поведение целевого вида, например, рыба может иметь сезонные изменения движений, раки или мидии выпускают ДНК в больших количествах в определенные времена их жизни (линька, нерест). Для ДНК в почве, еще меньше известно о распределении, количестве или качестве.
Основным ограничением метода штрих -кодирования является то, что он опирается на справочные библиотеки штрих -кода для таксономической идентификации последовательностей. Таксономическая идентификация является точной, только если доступна надежная ссылка. Тем не менее, большинство баз данных по -прежнему неполны, особенно для небольших организмов, например, грибов, фитопланктона, нематоды и т. Д. Кроме того, текущие базы данных содержат ошибочные идентификации, ошибки правописания и другие ошибки. Существуют массовые усилия по курированию и завершению баз данных для всех необходимых организмов, включающих крупные проекты штрих -кодирования (например, проект IBOL для контрольной базы данных штрих -кода систем жизненных данных (BOLD)). [ 83 ] [ 84 ] Тем не менее, завершение и курирование сложны и трудоемки. Без ответных образцов не может быть уверенности в том, правильной ли последовательность, используемая в качестве эталона.
Базы данных последовательностей ДНК, такие как GenBank, содержат много последовательностей, которые не привязаны к вашнивым образцам (например, образцы гербария, культивируемые клеточные линии или иногда изображения). Это проблематично перед лицом таксономических вопросов, таких как, следует ли разделить или объединить несколько видов, или были ли прошлые идентификации. Повторное использование последовательностей, не привязанных к ваучерным образцам, изначально неверно идентифицированного организма может подтвердить неверные выводы и следует избегать. [ 85 ] Следовательно, наилучшей практикой штрих -кодирования ДНК является последовательность ваучерных образцов. [ 86 ] [ 87 ] Однако для многих таксонов может быть трудно получить эталонные образцы, например, с образцами, которые трудно поймать, доступные образцы являются плохо консервативными, или не хватает таксономической экспертизы. [ 85 ]
Важно отметить, что штрих -коды ДНК также могут использоваться для создания промежуточной таксономии, и в этом случае OTU могут использоваться в качестве заменителей традиционных латинских биномиалов, что значительно снижает зависимость от полностью заполненных эталонных баз данных. [ 88 ]
Технологический уклон
[ редактировать ]ДНК штрих -кодирование также несет методологическое смещение, от отбора проб до анализа данных биоинформатики . Помимо риска загрязнения образца ДНК ингибиторами ПЦР, смещение праймера является одним из основных источников ошибок в штрих -кодировании ДНК. [ 89 ] [ 90 ] Выделение эффективного маркера ДНК и конструкция праймеров является сложным процессом, и были предприняты значительные усилия для разработки праймеров для штрих -кодирования ДНК в различных таксономических группах. [ 91 ] Тем не менее, праймеры часто связываются преимущественно с некоторыми последовательностями, что приводит к дифференциальной эффективности и специфичности праймеров, а также оценке и инфляции непредотверждающих сообществ. [ 92 ] Таким образом, состав последовательностей сообществ выборки в основном изменяется на стадии ПЦР. Кроме того, репликация ПЦР часто требуется, но приводит к экспоненциальному увеличению риска загрязнения. Несколько исследований подчеркнули возможность использования образцов, обогащенных митохондриями [ 93 ] [ 94 ] или без ПЦР подходы, чтобы избежать этих смещений, но на сегодняшний день метод метабардодирования ДНК все еще основан на секвенировании ампликонов. [ 91 ] Другое смещение входит в картинку во время секвенирования и во время биоинформатической обработки последовательностей, таких как создание химеров.
Отсутствие стандартизации
[ редактировать ]Несмотря на то, что штрих -кодирование ДНК более широко используется и применяется, нет согласия, касающихся методов сохранения или экстракции ДНК, выбор маркеров ДНК и набора праймеров или протоколов ПЦР. Параметры биоинформатических трубопроводов (например, кластеризация OTU, алгоритмы таксономических назначений или пороговые значения и т. Д.) Имеются многочисленные дискуссии среди пользователей штрих -кодирования ДНК. [ 91 ] Технологии секвенирования также быстро развиваются, вместе с инструментами для анализа огромных объемов генерируемых данных ДНК, а стандартизация методов срочно необходима для обеспечения совместной работы и обмена данными в большей пространственной и временной масштабе. Эта стандартизация методов штрих-кодирования в европейском масштабе является частью целей европейского действия ДНКК-Нет [ 95 ] и также рассматривается CEN (Европейский комитет по стандартизации). [ 96 ]
Другая критика штрих -кодирования ДНК - это ограниченная эффективность для точной дискриминации ниже уровня видов (например, для различения разновидностей), для обнаружения гибридов, и на это может повлиять эволюционные скорости [ Цитация необходима ] .
Несоответствия между обычной (морфологической) и идентификацией на основе штрих -кодов
[ редактировать ]Важно знать, что списки таксонов, полученные с помощью традиционной (морфологической) идентификации, не являются и, возможно, никогда не будут, непосредственно сопоставимы с списками таксонов, полученных из идентификации на основе штрих -кода по нескольким причинам. Наиболее важной причиной является, вероятно, неполнота и отсутствие точности молекулярных эталонных баз данных, предотвращающих правильное таксономическое назначение последовательностей EDNA. Таксоны, отсутствующие в справочных базах данных, не будут найдены EDNA, а последовательности, связанные с неправильным именем, приведут к неправильной идентификации. [ 78 ] Другими известными причинами являются различная шкала выборки и размер между традиционным и молекулярным образцом, возможный анализ мертвых организмов, который может происходить по -разному для обоих методов в зависимости от группы организма и конкретного выбора идентификации в любом методе, т.е. Различная таксономическая экспертиза или возможность выявления определенных групп организма, соответственно, предвзятость праймеров, приводящих к потенциальному предвзятому анализу таксонов. [ 78 ]
Оценки богатства/разнообразия
[ редактировать ]Шаткодирование ДНК может привести к чрезмерному или недооценке видового богатства и разнообразия. Некоторые исследования показывают, что артефакты (идентификация видов, не присутствующих в сообществе), являются основной причиной завышенного биоразнообразия. [ 97 ] [ 98 ] Наиболее проблематичной проблемой являются таксоны, представленные низким количеством чтений секвенирования. Эти чтения обычно удаляются во время процесса фильтрации данных, поскольку различные исследования показывают, что большинство из этих низкочастотных чтений могут быть артефактами. [ 99 ] Тем не менее, настоящие редкие таксоны могут существовать среди этих чтений с низким содержанием. [ 100 ] Редкие последовательности могут отражать уникальные линии в сообществах, которые делают их информативными и ценными последовательностями. Таким образом, существует серьезная потребность в более надежных алгоритмах биоинформатики, которые позволяют дифференциацию между информативными чтениями и артефактами. Полные справочные библиотеки также позволили бы лучше проверить алгоритмы биоинформатики, позволяя лучшей фильтрации артефактов (то есть удаление последовательностей, в которых отсутствует аналог среди существующих видов), и, следовательно, можно было бы получить более точное назначение видов. [ 101 ] Загадочное разнообразие также может привести к раздутым биоразнообразию, поскольку один морфологический вид может фактически разделяться на многие различные молекулярные последовательности. [ 78 ] Это будет иметь большое значение для генерации эталонных данных ДНК, что имеет решающее значение для ДНК окружающей среды мониторинга биоразнообразия на основе .
Мегабардовая
[ редактировать ]Мегабардодирование -это термин, используемый для описания высокопроизводительного штрих-кодирования ДНК на основе образцов, где тысячи образцов могут одновременно штрих-кодировать для идентификации и обнаружения видов. [ 102 ] [ 103 ] [ 104 ] [ 105 ] [ 106 ]

Это включено путем использования платформ секвенирования третьего поколения, включая Pacbio (продолжение I/II) Pacific Biosciences и Minion, Promethion от Oxford Nanopore Technology . По сравнению с секвенированием Sanger , мегабардодирование более быстрее и дешевле, что позволяет создать крупномасштабную генерацию штрих-кодов ДНК для тысяч видов. [ 107 ]
Приложения
[ редактировать ]Мегабардодирование может помочь заполнить темные таксоны . ДНК штрих -кодовый промежуток данных для насекомых и ускоряет открытие видов, [ 108 ] [ 109 ] понимать видовые паттерны разнообразия, [ 110 ] [ 111 ] [ 112 ] оценить богатство видов, [ 113 ] генерировать быстрые запасы видов биоразнообразия, [ 114 ] отслеживать базовые сдвиги, [ 115 ] и сопоставление этапов жизни. [ 116 ]
Метабардодирование
[ редактировать ]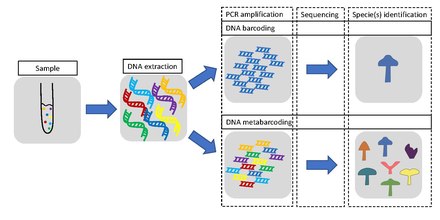
Метабардодирование определяется как штрих -кодирование ДНК или EDNA (ДНК окружающей среды), которое позволяет одновременно идентифицировать множество таксонов в одной и той же (экологической) выборке, однако часто в одной группе организма. Основное различие между подходами заключается в том, что метабардодирование, в отличие от штрих -кодирования, не фокусируется на одном специфическом организме, но вместо этого стремится определить состав видового состава в образце.
Методология
[ редактировать ]Процедура метабардодирования, такая как общее штрих -кодирование, охватывает стадии экстракции ДНК , амплификации ПЦР , секвенирования и анализа данных . Штрих -код состоит из короткой вариабельной области гена (например, см. Различные маркеры/штрих -коды ), которая полезна для таксономического назначения, окруженного высококонсервативными областями генов, которые можно использовать для проектирования праймера . [ 117 ] Различные гены используются в зависимости от того, если цель состоит в том, чтобы штрих -кодировать отдельные виды или метабардодирование нескольких видов. В последнем случае используется более универсальный ген. Метабардодирование не использует ДНК/РНК с одним видом в качестве отправной точки, но ДНК/РНК из нескольких различных организмов, полученных из одного экологического или объемного образца.
Приложения
[ редактировать ]Metabarcoding имеет потенциал для дополнения мер биоразнообразия и даже заменить их в некоторых случаях, особенно когда технология достигает и процедуры постепенно становятся дешевле, более оптимизированы и широко распространены. [ 118 ] [ 119 ]
Применение метабардодирования ДНК включает в себя мониторинг биоразнообразия в наземной и водной среде, палеонтологии и древних экосистемах, взаимодействиях растений-и- кар
Преимущества и проблемы
[ редактировать ]Общие преимущества и недостатки для штрих -кодирования, рассмотренные выше, также действительны для метабардодирования. Одним из конкретных недостатков для исследований метабардодирования является то, что еще нет консенсуса относительно оптимального экспериментального дизайна и критериев биоинформатики, которые будут применены в метабардодировании Эдны. [ 120 ] Тем не менее, существуют текущие объединенные попытки, например, например, сеть сети стоимости ЕС , чтобы продвинуться вперед, обмениваясь опытом и знаниями, чтобы установить стандарты наилучшего практики для биомонитора. [ 78 ]
Искусственная ДНК штрих -кодирование
[ редактировать ]В 2014 году исследователи из Eth Zurich предложили использовать искусственные штрих-коды ДНК размером с субмикрометр в качестве «невидимой масляной метки». Штрих -коды состоят из синтетических последовательностей ДНК внутри магнитно восстанавливаемых частиц кремнезема. Они могут быть добавлены в пищевое масло в очень небольшом количестве (вниз до 1 ppb) в качестве метки и могут быть извлечены в любое время для теста подлинности с помощью ПЦР/секвенирования. Этот метод может быть использован для тестирования оливкового масла для фальсификации. [ 121 ]
Смотрите также
[ редактировать ]Subtopics:
Связанные темы:
Также см. Навигацию по боковой панели в верхней части статьи.
Ссылки
[ редактировать ]- ^ "Что такое штрих -кодирование ДНК?" Полем ibol . Получено 2019-03-26 .
- ^ Кресс, В. Джон; Эриксон, Дэвид Л., ред. (2012). ДНК штрих -коды: методы и протоколы . Методы в молекулярной биологии. Тол. 858. Totowa, NJ: Humana Press. doi : 10.1007/978-1-61779-591-6 . ISBN 978-1-61779-590-9 Полем S2CID 3668979 .
- ^ Irinyi, L.; Лакнер, М.; de Hoog, GS; Мейер, В. (2015). «ДНК штрих -кодирование грибов, вызывающих инфекции у людей и животных» . Грибковая биология . 120 (2): 125–136. doi : 10.1016/j.funbio.2015.04.007 . PMID 26781368 .
- ^ Schoch, Conrad L.; Seifert, Keith A.; Huhndorf, Sabine; Роберт, Винсент; Spouge, John L.; Левеск, С. Андре; Чен, Вэнь; Грибковой штрих -консорциум (2012). «Ядерная рибосомальная внутренняя транскрибированная область спейсеров (ITS) в качестве универсального маркера штрих -кода ДНК для грибов» (PDF) . Труды Национальной академии наук . 109 (16): 6241–6246. doi : 10.1073/pnas.1117018109 . ISSN 0027-8424 . PMC 3341068 . PMID 22454494 .
- ^ Рабочая группа завода CBOL; Холлингсворт, премьер -министр; Forrest, LL; Spouge, JL; Hajibabaei, M.; Ratnasingham, S.; van der Bank, M.; Чейз, MW; Коуэн, Р.С. (2009-08-04). «Штрих -код ДНК для наземных растений» . Труды Национальной академии наук . 106 (31): 12794–12797. doi : 10.1073/pnas.0905845106 . ISSN 0027-8424 . PMC 2722355 . PMID 19666622 .
- ^ Полэй, Густав; Мейер, Кристофер П. (2005-11-29). «Шаткодирование ДНК: частота ошибок на основе комплексной выборки» . PLOS Биология . 3 (12): E422. doi : 10.1371/journal.pbio.0030422 . ISSN 1545-7885 . PMC 1287506 . PMID 16336051 .
- ^ Soininen, Eeva M; Валентини, Алиса; Койссак, Эрик; Микель, Кристиан; Гилли, Людович; Брохманн, Кристиан; Брайстнг, Энн К; Sønstebø, Jørn H; Ims, Rolf A (2009). «Анализ диеты мелких травоядных животных: эффективность штрих-кодирования ДНК в сочетании с высокопроизводительным пиросеквенированием для расшифровки состава сложных растительных смесей» . Границы в зоологии . 6 (1): 16. doi : 10.1186/1742-9994-6-16 . ISSN 1742-9994 . PMC 2736939 . PMID 19695081 .
- ^ Крир, Саймон; Дейнер, Кристи; Фрей, Серита ; Porazinska, Дорота; Таберлет, Пьер; Томас, В. Келли; Поттер, Кейтлин; Бик, Холли М. (2016). Фреклтон, Роберт (ред.). «Полевое руководство эколога по идентификации биоразнообразия на основе последовательностей» (PDF) . Методы экологии и эволюции . 7 (9): 1008–1018. doi : 10.1111/2041-210x.12574 . S2CID 87512991 .
- ^ Лиз, Флориан; и др. (Январь 2018). «Зачем нам нужны устойчивые сети, соединяющие страны, дисциплины, культуры и поколения для водного биомониторирования 2.0: перспектива, полученная в результате действия затрат на сеть DNAQUA». Достижения в области экологических исследований . 58 : 63–99. doi : 10.1016/bs.aecr.2018.01.001 . HDL : 1822/72852 . ISBN 9780128139493 .
- ^ Васселон, Валентин; Римет, Фредерик; Тапольчай, Калман; Bouchez, Agnès (2017). «Оценка экологического статуса с помощью метабардодирования ДНК DIATOMS: масштабирование в сети мониторинга WFD (остров Майотт, Франция)». Экологические индикаторы . 82 : 1–12. doi : 10.1016/j.ecolind.2017.06.024 . ISSN 1470-160x .
- ^ Woese, Carl R.; Кандлер, Отто; Wheelis, Mark L. (1990). «На пути к естественной системе организмов: предложение о доменах археи, бактерий и эурья» (PDF) . Труды Национальной академии наук . 87 (12): 4576–4579. Bibcode : 1990pnas ... 87.4576w . doi : 10.1073/pnas.87.12.4576 . OCLC 678728346 . PMC 54159 . PMID 2112744 .
- ^ Подпрыгнуть до: а беременный в Хеберт, Пол Д.Н.; Cywinska, Alina; Ball, Shelley L.; Деваард, Джереми Р. (2003-02-07). «Биологическая идентификация через штрих -коды ДНК» . Труды Королевского общества B: Биологические науки . 270 (1512): 313–321. doi : 10.1098/rspb.2002.2218 . ISSN 1471-2954 . PMC 1691236 . PMID 12614582 .
- ^ Фолмер, О.; Черный, м.; Hoeh, W.; Lutz, R.; Vrijenhoek, R. (октябрь 1994). «Праймеры ДНК для амплификации субъединицы I митохондриальной цитохрома с оксидазы от разнообразных метазоанских беспозвоночных». Молекулярная морская биология и биотехнология . 3 (5): 294–299. ISSN 1053-6426 . PMID 7881515 .
- ^ Jelger Herder, ДНК окружающей среды - обзор возможных применений для обнаружения (инвазивных) видов.
- ^ Schrader, C тогда это может быть так из -за ДНК.; Schielke, A.; Ellerbroek, L.; Johne, R. (2012). «Ингибиторы ПЦР - возникновение, свойства и удаление» . Журнал прикладной микробиологии . 113 (5): 1014–1026. doi : 10.1111/j.1365-2672.2012.05384.x . ISSN 1365-2672 . PMID 22747964 . S2CID 30892831 .
- ^ Саволайнен, Винсент ; Коуэн, Робин С; Фоглер, Алфрид П; Родерик, Джордж К; Лейн, Ричард (2005-10-29). «Направление энциклопедии жизни: введение в штрих -кодирование ДНК» . Философские транзакции Королевского общества B: биологические науки . 360 (1462): 1805–1811. doi : 10.1098/rstb.2005.1730 . ISSN 0962-8436 . PMC 1609222 . PMID 16214739 .
- ^ Пигготт, Максин П. (2016). «Оценка влияния лабораторных протоколов на вероятность обнаружения EDNA для исчезающих пресноводных рыб» . Экология и эволюция . 6 (9): 2739–2750. doi : 10.1002/ECE3.2083 . ISSN 2045-7758 . PMC 4798829 . PMID 27066248 .
- ^ MA, Hongjuan; Стюарт, Кэтрин; Лафейд, Стивен; Чжэн, Джиннг; Ван, Юсиан; Чжао, Цзянфу (2016). «Характеристика, оптимизация и валидация маркеров ДНК окружающей среды (EDNA) для обнаружения исчезающих водных млекопитающих». Сохранение генетики ресурсов . 8 (4): 561–568. doi : 10.1007/s12686-016-0597-9 . ISSN 1877-7252 . S2CID 1613649 .
- ^ Д'Амор, Розалинда; Ijaz, Умер Зишан; Ширмер, Мелани; Кенни, Джон Г.; Грегори, Ричард; Дарби, Алистер С.; Шакья, Мигун; Podar, Mircea; Куинс, Кристофер (2016-01-14). «Комплексное исследование протоколов и платформ секвенирования для профилирования сообщества 16S рРНК» . BMC Genomics . 17 (1): 55. doi : 10.1186/s12864-015-2194-9 . ISSN 1471-2164 . PMC 4712552 . PMID 26763898 .
- ^ Кресс, WJ; Эриксон, Д.Л. (2008-02-26). «Штрих -коды ДНК: гены, геномика и биоинформатика» . Труды Национальной академии наук . 105 (8): 2761–2762. Bibcode : 2008pnas..105.2761k . doi : 10.1073/pnas.0800476105 . ISSN 0027-8424 . PMC 2268532 . PMID 18287050 .
- ^ Подпрыгнуть до: а беременный Хеберт, Пол Д.Н.; Ратнасингем, Судживан; Де Ваард, Джереми Р. (2003-08-07). «Жизнь штрих -кодирования: субъединица цитохрома С оксидазы 1 среди близкородственных видов» . Труды Королевского общества B: Биологические науки . 270 (Suppl_1): S96-9. doi : 10.1098/rsbl.2003.0025 . ISSN 1471-2954 . PMC 1698023 . PMID 12952648 .
- ^ Blaxter, Mark L. (2004-04-29). Godfray, HCJ; Кнапп, С. (ред.). «Обещание таксономии ДНК» . Философские транзакции Королевского общества Лондона. Серия B: Биологические науки . 359 (1444): 669–679. doi : 10.1098/rstb.2003.1447 . ISSN 1471-2970 . PMC 1693355 . PMID 15253352 .
- ^ Фазекас, Арон Дж.; Берджесс, Кевин С.; Кесанакурти, Прасад Р.; Грэм, Шон В.; Newmaster, Steven G.; Муж, Брайан С.; Перси, Диана М.; Хаджибабей, Мехрддад; Barrett, Spencer CH (2008-07-30). Десалле, Роберт (ред.). «Многочисленные штрих -коды ДНК с мультилокусной ДНК из пластидного генома в равной степени различают виды растений» . Plos один . 3 (7): E2802. Bibcode : 2008ploso ... 3.2802f . doi : 10.1371/journal.pone.0002802 . ISSN 1932-6203 . PMC 2475660 . PMID 18665273 .
- ^ Кресс, В. Джон; Эриксон, Дэвид Л. (2007-06-06). Шиу, Шин-Хан (ред.). «Глобальный штрих-код ДНК с двумя локусами для наземных растений: кодирующий ген RBCL дополняет некодирующую область спейсера TRNH-PSBA» . Plos один . 2 (6): E508. Bibcode : 2007ploso ... 2..508k . doi : 10.1371/journal.pone.0000508 . ISSN 1932-6203 . PMC 1876818 . PMID 17551588 .
- ^ Janda, JM; Эббот, С.Л. (2007-09-01). «Секвенирование гена 16S рРНК для бактериальной идентификации в диагностической лаборатории: плюсы, опасности и ловушки» . Журнал клинической микробиологии . 45 (9): 2761–2764. doi : 10.1128/jcm.01228-07 . ISSN 0095-1137 . PMC 2045242 . PMID 17626177 .
- ^ Смит, М. Алекс; Бертран, Клаудия; Кросби, Кейт; Eveleigh, Eldon S.; Фернандес-Триана, Хосе; Фишер, Брайан Л.; Гиббс, Джейсон; Хаджибабей, Мехрддад; Hallwachs, Winnie (2012-05-02). Барсук, Джонатан Х. (ред.). « Вулбахия и штрих -кодирующие насекомые: закономерности, потенциал и проблемы» . Plos один . 7 (5): E36514. BIBCODE : 2012PLOSO ... 736514S . doi : 10.1371/journal.pone.0036514 . ISSN 1932-6203 . PMC 3342236 . PMID 22567162 .
- ^ Ссылки, Мэтью Г.; Dumonceaux, Tim J.; Хеммингсен, Шон М.; Хилл, Джанет Э. (2012-11-26). Нойфельд, Джош (ред.). «Универсальная мишень шаперонина-60 представляет собой штрих-код для бактерий, который позволяет сборке данных DE novo данных метагеномных последовательностей» . Plos один . 7 (11): E49755. BIBCODE : 2012PLOSO ... 749755L . doi : 10.1371/journal.pone.0049755 . ISSN 1932-6203 . PMC 3506640 . PMID 23189159 .
- ^ Дело, RJ; Boucher, Y.; Dahllof, я.; Holmsstrom, C.; Doolittle, WF; Kjelleberg, S. (2007-01-01). «Использование генов 16S рРНК и RPOB в качестве молекулярных маркеров для исследований микробной экологии» . Прикладная и экологическая микробиология . 73 (1): 278–288. Bibcode : 2007apenm..73..278c . doi : 10.1128/aem.01177-06 . ISSN 0099-2240 . PMC 1797146 . PMID 17071787 .
- ^ Беллемейн, Ева; Карлсен, Тор; Брохманн, Кристиан; Койссак, Эрик; Таберлет, Пьер; Kauserud, Håvard (2010). «Это как экологический штрих -код ДНК для грибов: подход в Silico выявляет потенциальные смещения ПЦР» . BMC Микробиология . 10 (1): 189. doi : 10.1186/1471-2180-10-189 . ISSN 1471-2180 . PMC 2909996 . PMID 20618939 .
- ^ Seifert, Ka; Самсон, Ра; Dewaard, Jr; Houbraken, J.; Левеск, Калифорния; Moncalvo, J.-M.; Louis-Seize, G.; Hebert, Pdn (2007-03-06). «Перспективы идентификации грибков с использованием штрих -кодов ДНК CO1, с Penicillium в качестве тестового примера» . Труды Национальной академии наук . 104 (10): 3901–3906. doi : 10.1073/pnas.0611691104 . ISSN 0027-8424 . PMC 1805696 . PMID 17360450 .
- ^ Dentinger, Bryn TM; Дидух, Мэрина Y.; Moncalvo, Jean-Marc (2011-09-22). Шираотер, Бернд (ред.). «Сравнивая COI и его маркеры штрих -кода ДНК для грибов и союзников (Agaricomycotina)» . Plos один . 6 (9): E25081. Bibcode : 2011ploso ... 625081d . doi : 10.1371/journal.pone.0025081 . ISSN 1932-6203 . PMC 3178597 . PMID 21966418 .
- ^ Хаунд, Полашри; Джоши, старший (октябрь 2014 г.). «Шаткодирование ДНК диких съедобных грибов, потребляемых этническими племенами Индии». Ген . 550 (1): 123–130. doi : 10.1016/j.gene.2014.08.027 . PMID 25130907 .
- ^ Вейганд, Ханна; Beermann, Arne J.; Чинасп, зловоние; Коста, Филипе О.; Csabai, Zoltán; Дуарте, София; Гейгер, Матиас Ф.; Grabowski, Michał; Rimet, Frédéric (2019-03-14). «Справочные библиотеки штрих-кода ДНК для мониторинга водной биоты в Европе: аализ GAP и рекомендации для будущей работы». Bioorxiv . 678 : 499–524. Bibcode : 2019 Scepten.678..499W . Doi : 10.1101/5765553 . HDL : 11250/2608962 . PMID 31077928 . S2CID 92160002 .
- ^ Gottschling M, J Chacón, A žerdoner čalasan, St Neuhaus, J Kretchmann, H Stibor & U John (2020): филогенетическое размещение последовательностей окружающей среды с использованием таксономически надежных баз данных помогает строго оценить биорацерность динофита в бабарных замыканиях (Германия). Freshw Biol 65: 193–208. Doi : 10.1111/fwb.13413
- ^ Rdmpage (2016), Международный проект Barcode of Life Project (IBOL) (набор данных), Институт биоразнообразия, здоровье животных и сравнительная медицина, Колледж медицинских, ветеринарных и жизненных наук, Университет Глазго, DOI : 10.15468/inygc6 , Получено 2019- 05-14
- ^ Ратнасингем, Судживан; Хеберт, Пол Д.Н. (2007-01-24). «Штрих -кодирование: жирный жирный кадр: штрих -код системы жизненных данных (http://www.barcodinglife.org): штрих -кодирование» . Молекулярная экология Примечания . 7 (3): 355–364. doi : 10.1111/j.1471-8286.2007.01678.x . PMC 1890991 . PMID 18784790 .
- ^ Нильссон, Рольф Хенрик; Ларссон, Карл-Хенрик; Тейлор, Энди Ф.С.; Бенгтссон-Палм, Йохан; Jeppesen, Thomas S.; Шигель, Дмитрий; Кеннеди, Питер; Пикард, Кэтрин; Glöckner, Frank Oliver (2019-01-08). «База данных UNITE для молекулярной идентификации грибов: обработка темных таксонов и параллельных таксономических классификаций» . Исследование нуклеиновых кислот . 47 (D1): D259 - D264. doi : 10.1093/nar/gky1022 . ISSN 0305-1048 . PMC 6324048 . PMID 30371820 .
- ^ Римет, Фредер; Гусев, Евгений; Калерт, Мэри; Келли, Мартин; Куливиски, Максим; Мальтев, Евхен; Манн, Дэвид; Пфаннкинкен, Мартин; Trobajo, Rose (2019-02–14). Открытая библиотека . data.fr (набор данных). Портал Дара Инра. doi : 10.15454/ tmbyz
- ^ Римет, Фредерик; Chaumeil, Philippe; Кек, Франсуа; Кермаррек, Ленаиг; Васселон, Валентин; Калерт, Мария; Франк, Ален; Bouchez, Agnès (2016). «R-Syst :: Diatom: открытые достоинства и кураторская база данных штрих-кода для диатомов и мониторинга пресной воды» . База данных . 2016 : BAW016. Doi : 10.1093/база данных/BAW016 . ISSN 1758-0463 . PMC 4795936 . PMID 26989149 .
- ^ Шлосс, Патрик Д.; Уэсткотт, Сара Л.; Рьябин, Томас; Холл, Джастин Р.; Хартманн, Мартин; Холлистер, Эмили Б.; Lesniewski, Ryan A.; Оукли, Брайан Б.; Паркс, Донован Х.; Робинсон, Кортни Дж.; Сахл, Джейсон В.; Stres, Blaž.; Thallinger, Gerhard G.; Хорн, Дэвид Дж.; фургон Вебер, Кэроли Ф. (2009). «Представление Mothur: программное обеспечение с открытым исходным кодом, независимое от платформы, для описания и сравнения микробных сообществ» . Прикладная и экологическая микробиология . 75 (23): 7537–41. Bibcode : 2009apenm..75.7537s . doi : 10.1128/aem.01541-09 . OCLC 780918718 . PMC 2786419 . PMID 19801464 .
- ^ Эдгар, Роберт С. (2013-08-18). «Uparse: Высокие точные последовательности OTU от микробного ампликона чтения». Природные методы . 10 (10): 996–998. doi : 10.1038/nmeth.2604 . ISSN 1548-7091 . PMID 23955772 . S2CID 7181682 .
- ^ Капорасо, Дж. Грегори; Кучински, Джастин; Stombaugh, Jesse; Биттингер, Кайл; Бушмен, Фредерик Д; Костелло, Элизабет К; Старше, Ной; Пенья, Антонио Гонсалес; Гудрич, Джулия К (май 2010 г.). «QIIME позволяет анализировать высокопроизводительные данные секвенирования сообщества» . Природные методы . 7 (5): 335–336. doi : 10.1038/nmeth.f.303 . ISSN 1548-7091 . PMC 3156573 . PMID 20383131 .
- ^ Афган, Энис; Бейкер, Даннон; Батут, Береняс; Ван Ден Бик, Мариус; Бувье, Дэйв; Чеч, Мартин; Чилтон, Джон; Клементс, Дэйв; Coraor, Nate (2018-07-02). «Платформа Galaxy для доступного, воспроизводимого и совместного биомедицинского анализа: обновление 2018 года» . Исследование нуклеиновых кислот . 46 (W1): W537 - W544. doi : 10.1093/nar/gky379 . ISSN 0305-1048 . PMC 6030816 . PMID 29790989 .
- ^ Бойер, Фрейдрик; Мерсье, Селин; Бонин, Аурели; Рука, Иван; Таберлет, Пьер; Coissac, Eric (2015-05-26). «Obitools: программный пакет, вдохновленный AUTIX для метабардодирования ДНК». Молекулярные экологические ресурсы . 16 (1): 176–182. Doi : 10.1111/1755-0998.12428 . ISSN 1755-098X . PMID 25959493 . S2CID 39412858 .
- ^ Elbrecht, Vasco (2019-04-30), Github-Vascoelbrecht/Jamp: Jamp: просто еще один трубопровод метабардодирования. Получено 2019-05-14
- ^ Нормандо, Эрик (2020-01-21), Github-Enormandeau/Barque: Barque: анализ метабардодирования ДНК окружающей среды. Получено 2020-01-21
- ^ Каллахан, Бенджамин Дж; МакМерди, Пол Дж; Розен, Майкл Дж; Хан, Эндрю У; Джонсон, Эми Джо А; Холмс, Сьюзен П (июль 2016 г.). «DADA2: выборочный вывод высокого разрешения из данных Illumina Amplicon» . Природные методы . 13 (7): 581–583. doi : 10.1038/nmeth.3869 . ISSN 1548-7091 . PMC 4927377 . PMID 27214047 .
- ^ McMurdie, Paul J.; Холмс, Сьюзен (2014). «Не хочу, не хочу: почему данные микробиома не подходят» . PLOS Computational Biology . 10 (4): E1003531. Arxiv : 1310.0424 . BIBCODE : 2014PLSCB..10E3531M . doi : 10.1371/journal.pcbi.1003531 . PMC 3974642 . PMID 24699258 .
- ^ Валенте, Габриэль; Янссон, Джеспер; Клементе, Хосе Карлос; Alons-Alemany, Daniel (2011-10-10). «Таксономическое назначение в метагеномике с танго» . Embne.journal . 17 (2): 16–20. doi : 10.14806/ej.17.2.237 . HDL : 2117/16286 . ISSN 2226-6089 .
- ^ Пайл, Ричард Л.; Эрл, Джон Л.; Грин, Брайан Д. (2008-01-01). «Пять новых видов самого самого рода (Perciformes: Labroidei: Pomacentridae) из глубоких коралловых рифов в тропической западной части Тихого океана» . Zootaxa . 1671 (1). doi : 10.11646/Zootaxa.1671.1.2 . ISSN 1175-5334 .
- ^ Подпрыгнуть до: а беременный Schnell, Ida Bærholm; Томсен, Филипп Фрэнсис; Уилкинсон, Николас; Расмуссен, Мортен; Дженсен, Ларс Р.Д.; Уиллерслев, Эске; Bertelsen, Mads f.; Гилберт, М. Томас П. (апрель 2012 г.). «Скрининг биоразнообразия млекопитающих с использованием ДНК от Леохес» . Текущая биология . 22 (8): R262 - R263. Doi : 10.1016/j.cub.2012.02.058 . PMID 22537625 . S2CID 18058748 .
- ^ Subrata., Triedi (2016). ДНК штрих -кодирование в морских перспективах: оценка и сохранение биоразнообразия . Ансари, Абид Али., Гош, Санкар К., Рехман, Хасибур. Cham: Springer International Publishing. ISBN 9783319418407 Полем OCLC 958384953 .
- ^ Подпрыгнуть до: а беременный Далтон, Дезире Ли; де Брюн, Марли; Томпсон, Тиа; Kotzé, Антуанетта (2020-12-01). «Оценка полезности штрих -кодирования ДНК в судебных случаях дикой природы, связанных с южноафриканской антилопой» . Forensic Science International: Отчеты . 2 : 100071. DOI : 10.1016/j.fsir.2020.100071 . ISSN 2665-9107 . S2CID 213926390 .
- ^ Хеберт, Пол Д.Н.; Stoeckle, Mark Y.; Землак, Тайлер С.; Фрэнсис, Чарльз М. (октябрь 2004 г.). «Идентификация птиц через штрих -коды ДНК» . PLOS Биология . 2 (10): E312. doi : 10.1371/journal.pbio.0020312 . ISSN 1545-7885 . PMC 518999 . PMID 15455034 .
- ^ Коста, Филипе О; Карвалью, Гэри Р (декабрь 2007 г.). «Инициатива штрих -кода жизни: синопсис и проспективные социальные последствия штрих -кодирования рыбы» . Геномика, общество и политика . 3 (2): 29. doi : 10.1186/1746-5354-3-2-29 . ISSN 1746-5354 . PMC 5425017 .
- ^ Lahaye, R.; van der Bank, M.; Богарин, Д.; Warner, J.; Пупулин, ф.; Gigot, G.; Маурин, О.; Duthoit, S.; Barraclough, TG (2008-02-26). «ДНК шаркодирует флоры горячих точек биоразнообразия» . Труды Национальной академии наук . 105 (8): 2923–2928. doi : 10.1073/pnas.0709936105 . ISSN 0027-8424 . PMC 2268561 . PMID 18258745 .
- ^ Подпрыгнуть до: а беременный Сюй, Сонг-Зхи; Ли, Чжэнь-Ю; Джин, Сяо-хуа (январь 2018 г.). «Шаткодирование ДНК инвазивных растений в Китае: ресурс для идентификации инвазивных растений». Молекулярные экологические ресурсы . 18 (1): 128–136. doi : 10.1111/1755-0998.12715 . PMID 28865184 . S2CID 24911390 .
- ^ Лю, Джуннинг; Цзян, Цзямей; Песня, Шули; Торнабен, Люк; Чабария, Райан; Нейлор, Гэвин Дж.П.; Ли, Ченхонг (декабрь 2017 г.). «Многолокусное штрих -кодирование ДНК - идентификация видов с помощью многолокусных данных» . Научные отчеты . 7 (1): 16601. Bibcode : 2017natsr ... 716601L . doi : 10.1038/s41598-017-16920-2 . ISSN 2045-2322 . PMC 5709489 . PMID 29192249 .
- ^ Нагоши, Родни Н.; Брамбила, Джульетта; Мигер, Роберт Л. (ноябрь 2011 г.). «Использование штрих -кодов ДНК для выявления инвазивных видов Spodoptera во Флориде во Флориде» . Журнал науки насекомых . 11 (154): 154. doi : 10.1673/031.011.15401 . ISSN 1536-2442 . PMC 3391933 . PMID 22239735 .
- ^ Бабанов, DP; Беккер, EI; Павлов, DD; Боровикова, EA; Кадудухова, YV; Коток, А.А. (1 февраля 2022 г.). «Новые наборы праймеров для идентификации ДНК некоренных видов рыб в бассейне Волга-Кама (Европейская Россия) » Вода 14 (3): 437. doi : 10.3390/ w140304 ISSN 2073-4
- ^ Тонгтам на Аюдхайя, Прадипунт; Муангмай, Наронгрит; Banjongsat, Nuwadee; Сингчат, Ворпонг; Janekitkarn, Sommai; Пейахокнагул, Сурин; Шрикулнатх, Корнсонн (июнь 2017 г.). «Раскрытие загадочного разнообразия родов -анемонов Амфиприона и Премнас (Percifformes: Pomacentridae) в Таиланде с штрих -кодами митохондрий ДНК» . Сельское хозяйство и природные ресурсы . 51 (3): 198–205. Doi : 10.1016/j.anres.2017.07.001 .
- ^ Хеберт, Pdn; Пентон, эх; Бернс, JM; Янзен, DH; Hallwachs, W. (2004-10-12). «Десять видов в одном: штрих -кодирование ДНК выявляет загадочные виды в неотропическом шкипере бабочка астрататора » . Труды Национальной академии наук . 101 (41): 14812–14817. Bibcode : 2004pnas..10114812H . doi : 10.1073/pnas.0406166101 . ISSN 0027-8424 . PMC 522015 . PMID 15465915 .
- ^ Brower, Andrew VZ (июнь 2006 г.). «Проблемы с штрих -кодами ДНК для разграничения видов:« десять видов » астраптов, переосмыслимый (Lepidoptera: Hesperiidae)». Систематика и биоразнообразие . 4 (2): 127–132. doi : 10.1017/s147720000500191x . ISSN 1477-2000 . S2CID 54687052 .
- ^ Смит, Массачусетс; Вудли, NE; Янзен, DH; Hallwachs, W.; Hebert, Pdn (2006-03-07). «Штрих-коды ДНК показывают специфичность загадочного хозяина в пределах предполагаемых полифагозных членов рода паразитоидных мух (Diptera: Tachinidae)» . Труды Национальной академии наук . 103 (10): 3657–3662. doi : 10.1073/pnas.0511318103 . ISSN 0027-8424 . PMC 1383497 . PMID 16505365 .
- ^ Брэзер, Мадлен Дж.; Виклунд, Хелена; Нил, Ленка; Джеффрис, Рэйчел; Линсе, Катрин; Руль, Генри; Гловер, Адриан Г. (ноябрь 2016 г.). «Шатрикодирование ДНК раскрывает загадочное разнообразие в 50% глубоководных антарктических полихет» . Королевское общество открыто наука . 3 (11): 160432. Bibcode : 2016rsos .... 360432b . doi : 10.1098/rsos.160432 . ISSN 2054-5703 . PMC 5180122 . PMID 28018624 .
- ^ Помпанон, Франсуа; Deagle, Bruce E.; Симондсон, Уильям ОК; Браун, Дэвид С.; Джарман, Саймон Н.; Таберлет, Пьер (апрель 2012 г.). «Кто едет что: оценка диеты с использованием секвенирования следующего поколения: анализ диеты NGS» . Молекулярная экология . 21 (8): 1931–1950. doi : 10.1111/j.1365-294x.2011.05403.x . PMID 22171763 . S2CID 10013333 .
- ^ Валентини, Алиса; Помпанон, Франсуа; Таберлет, Пьер (февраль 2009 г.). «Шадкодирование ДНК для экологов». Тенденции в экологии и эволюции . 24 (2): 110–117. doi : 10.1016/j.tree.2008.09.011 . PMID 19100655 .
- ^ Подпрыгнуть до: а беременный в Каунисто, Кари М.; Рослин, Томас; Sääksjärvi, Ilari E.; Vesterinen, Eero J. (октябрь 2017 г.). «Пеллеты доказывания: сначала взглянуть на пищевую композицию взрослых одонатов, что выявило метабардодирование фекалий» . Экология и эволюция . 7 (20): 8588–8598. doi : 10.1002/ECE3.3404 . PMC 5648679 . PMID 29075474 .
- ^ Harms-Tuohy, CA; Шиза, NV; Appeldoorn, RS (2016-10-25). «Использование метабардодирования ДНК для анализа содержания желудка в инвазивных левких рыбах Vterois Volitans в Пуэрто -Рико» . Серия прогресса в морской экологии . 558 : 181–191. Bibcode : 2016meps..558..181h . doi : 10.3354/meps11738 . ISSN 0171-8630 .
- ^ Ковальчик, Рафал; Таберлет, Пьер; Койссак, Эрик; Валентини, Алиса; Микель, Кристиан; Каминьски, Томаш; Wójcik, Jan M. (февраль 2011 г.). «Влияние практики управления на большую травоядную диету -пример европейского бизона в Биольежском первобытном лесу (Польша)». Лесная экология и управление . 261 (4): 821–828. Doi : 10.1016/j.foreco 2010.11.026 .
- ^ Николс, Рут против.; Cromsigt, Joris PGM; Спонг, Гёран (декабрь 2015 г.). «Использование EDNA для экспериментального тестирования копытных предпочтений просмотра» . Springerplus . 4 (1): 489. doi : 10.1186/s40064-015-1285-z . ISSN 2193-1801 . PMC 4565800 . PMID 26380165 .
- ^ Agusti, N.; Shayler, Sp; Харвуд, JD; Воган, IP; Sunderland, KD; Symondson, WOC (декабрь 2003 г.). «Коллембола как альтернативная добыча, поддерживающая пауки в пахотных экосистемах: обнаружение добычи в хищниках с использованием молекулярных маркеров» . Молекулярная экология . 12 (12): 3467–3475. doi : 10.1046/j.1365-294x.2003.02014.x . ISSN 0962-1083 . PMID 14629361 . S2CID 7985256 .
- ^ Валентини, Алиса; Микель, Кристиан; Наваз, Мухаммед Али; Беллемейн, Ева; Койссак, Эрик; Помпанон, Франсуа; Гилли, Людович; Круа, Коринн; Nascetti, Джузеппе (январь 2009 г.). «Новые перспективы анализа диеты, основанные на штрих -кодировании ДНК и параллельном пиросеквенировании: подход TRN L» . Молекулярные экологические ресурсы . 9 (1): 51–60. doi : 10.1111/j.1755-0998.2008.02352.x . PMID 21564566 . S2CID 5308081 .
- ^ Фридман, Мелисса; Фернандес, Мерседес; Сакер, Лорейн; Дики, Роберт; Бернштейн, Джеффри; Шранк, Кэтлин; Киблер, Стивен; Стефан, Венди; Гриббл, Мэтью (2017-03-14). «Обновленный обзор отравления рыбой сигуатер: клинический, эпидемиологический, экологический и управление здравоохранением» . Морские лекарства . 15 (3): 72. DOI : 10.3390/MD15030072 . ISSN 1660-3397 . PMC 5367029 . PMID 28335428 .
- ^ Хан, FM; Уильям, К.; Aruge, S.; Janjua, S.; Shah, SA (2018-03-04). «Незаконное производство продукта и экспорт из Пакистана: выявление фактических действий высоко обработанных образцов кожи дикой природы с помощью ДНК мини-бардодирования» . Нуклеозиды, нуклеотиды и нуклеиновые кислоты . 37 (3): 179–185. doi : 10.1080/15257770.2018.1450507 . PMID 29608392 . S2CID 4623232 .
- ^ Мвале, Моника; Далтон, желание L.; Янсен, Рэймонд; Де Брюн, Марли; Питерсен, Даррен; Mokgokong, осторожный с.; Kotzé, Антуанетта (март 2017 г.). Стейнке, Дирк (ред.). «Судебно -медицинское применение штрих -кодирования ДНК для выявления незаконно торгуемых африканских масштабов панголина» . Геном . 60 (3): 272–284. doi : 10.1139/gen 2016-0144 . HDL : 1807/75671 . ISSN 0831-2796 . PMID 28177847 . S2CID 207093202 .
- ^ Лю, Янлей; Сюй, Чао; Донг, Венпан; Ян, Xueying; Чжоу, Шилиан (2021-07-01). «Определение подозреваемого преступника с использованием технологии метабардодирования ДНК завода окружающей среды» . Forensic Science International . 324 : 110828. DOI : 10.1016/j.forsciint.2021.110828 . ISSN 0379-0738 . PMID 34000616 . S2CID 234768561 .
- ^ Подпрыгнуть до: а беременный в дюймовый и фон Павловский, Ян; Келли-Куинн, Мэри; Altermatt, Florian; Apothéloz-Perret-Gentil, Laure; Беха, Педро; Богда, Анжела; Борха, Ангел; Бушез, Агнес; Кордиер, Тристан (2018). «Будущее биотических индексов в эпогномическую эпоху: интеграция (E) метабардодирование ДНК в биологической оценке водных экосистем» . Наука общей среды . 637–638: 1295–1310. Bibcode : 2018scten.637.1295p . doi : 10.1016/j.scitotenv.2018.05.002 . HDL : 20.500.12327/138 . PMID 29801222 .
- ^ Армитаж, Патрик Д.; Крэнстон, Питер С.; Пиндер, LCV, ред. (1995). Chironomidae . Дордрехт: Спрингер Нидерланды. doi : 10.1007/978-94-011-0715-0 . ISBN 9789401043083 Полем S2CID 46138170 .
- ^ Beermann, Arne J.; Zizka, Vera Ma; Эльбрехт, Васко; Баранов, Виктор; Лиз, Флориан (2018-07-24). «ДНК -мебаркодирование выявляет сложные и скрытые реакции хрономидов на множественное стресс » Экологические науки Европа 30 (1): 26. doi : 10.1186/s12302-018-0157-x . ISSN 2190-4715 . S2CID 51802465
- ^ Берманн, Арне Дж.; Эльбрехт, Васко; Карнац, Св. Ма, Ли; Matthewi, Christophy D.; Пигготт, Джереми Дж.; Аренда, Флориан (2018). «Влияние с множественной ситри-ситриром на передачу макробеспозвоночных коммуникаций: эксперимент по мезокосмам, манипулирующий санитом, тонким осадком и потоком». Наука общей среды 610–611: 961–9 Bibcode : 2018scten.610..961b Doi : 10.1016/ j.scitetenv.2017.08.0 28830056PMID
- ^ Macher, Jan N.; Салис, Романа К.; Блакмор, Кэти С.; Толриан, Ральф; Matthaei, Christoph D.; Лиз, Флориан (2016). «Влияние множественного стресса на потоковые беспозвоночные: штрих-кодирование ДНК выявляет контрастные реакции загадочных видов Mayfly». Экологические индикаторы . 61 : 159–169. doi : 10.1016/j.ecolind.2015.08.024 .
- ^ «Международный консорциум штрих -кода жизни» . Международный штрих -код жизни . Получено 2019-03-29 .
- ^ «Смелые системы v4» . www.boldsystems.org . Получено 2019-04-02 .
- ^ Подпрыгнуть до: а беременный Огванг, Джоэл; Барихе, Мишель; Бос, Артур Р. (2020). «Генетическое разнообразие и филогенетические отношения ниточных лещных ( Nemipterus spp.) Из красного моря и восточного Средиземного моря» . Геном . 63 (3): 207–216. doi : 10.1139/gen 2019-0163 . PMID 32678985 .
- ^ Шандер, Кристоффер; Willassen, Endre (2005). «Что может сделать биологическое штрих -кодирование для морской биологии?» Полем Морская биологическая исследование . 1 (1): 79–83. doi : 10.1080/17451000510018962 . ISSN 1745-1000 . S2CID 84070971 .
- ^ Миллер, SE (2007-03-20). «Шаткодирование ДНК и эпохи Возрождения таксономии» . Труды Национальной академии наук . 104 (12): 4775–4776. Bibcode : 2007pnas..104.4775m . doi : 10.1073/pnas.0700466104 . ISSN 0027-8424 . PMC 1829212 . PMID 17363473 .
- ^ Ratnasingham, S. (2013). «Реестр на основе ДНК для всех видов животных: система индекса штрих-кода (BIN)» . Plos один . 8 (7): E66213. BIBCODE : 2013PLOSO ... 866213R . doi : 10.1371/journal.pone.0066213 . PMC 3704603 . PMID 23861743 .
- ^ Лиз, Флориан; Эльбрехт, Васко (2015-07-08). «Могут ли ДНК-экосистемные оценки количественно определить изобилие вида? Тестирование смещения праймеров и биомассы-взаимосвязи последовательности с инновационным протоколом метабардодирования» . Plos один . 10 (7): E0130324. BIBCODE : 2015PLOSO..1030324E . doi : 10.1371/journal.pone.0130324 . ISSN 1932-6203 . PMC 4496048 . PMID 26154168 .
- ^ Эльбрехт, Васко; Vamos, Ecaterina Edith; Мейснер, Кристиан; Aroviita, Jukka; Лиз, Флориан (2017). «Оценка сильных и слабых сторон ДНК на основе метабардодирования на основе макробеспозвоночных на основе ДНК для обычного мониторинга потока» . Методы экологии и эволюции . 8 (10): 1265–1275. doi : 10.1111/2041-210x.12789 . ISSN 2041-210x .
- ^ Подпрыгнуть до: а беременный в Pawlowski, J.; Kelly-Quinn, M.; Altermatt, F.; Апотетотический-пертерель-гентиле, L.; Beja, P.; Bogger, A.; Borja, A.; Музыка, а.; Шнур, т.; Domaizon, i.; Вера, MJ; Филипп, AF; Fornarli, R.; График, W.; Herder, J.; Ван Торн, Б.; Iwan Jones, J.; Sagova-Mareccova, M.; Мориц, C.; Barce, J.; Пиггер, JJ; Pinna, M.; Rime, F.; Ринквич, Б.; Такие святые, C.; Specch, v.; Trobajo, R.; Vasselon, v.; Visual, S.; и ал. (Октябрь 2018). «Будущее экономических отходов было: Интеграция (E) ДНК в биогенении . Общая наука 637–638: 1295–1310. BIBCODE : 2018ST.637,1295P . doi : 10.1016/ j.scytotenv.2018.05.0 HDL : 20 500,12327/ 1 29801222PMID
- ^ Quince, Кристофер; Слоан, Уильям Т.; Холл, Нил; Д'Амор, Розалинда; Ijaz, Umer Z.; Ширмер, Мелани (2015-03-31). «Понимание смещений и ошибок секвенирования для секвенирования ампликонов с платформой Illumina Miseq» . Исследование нуклеиновых кислот . 43 (6): E37. doi : 10.1093/nar/gku1341 . ISSN 0305-1048 . PMC 4381044 . PMID 25586220 .
- ^ Хуан, Кванфей; Ли, Джигуан; Фу, Рибей; Тан, мин; Чжоу, Лили; Su, Xu; Ян, Цин; Лю, Шанлин; Li, Yiyuan (2013-12-01). «Секвенирование ультрагибации обеспечивает высокую точную восстановление биоразнообразия для объемных образцов членистоногих без ПЦР-амплификации» . Гигаскаука . 2 (1): 4. doi : 10.1186/2047-217x-2-4 . PMC 3637469 . PMID 23587339 .
- ^ Мачер, Ян-Никлас; Zizka, Вера Мари Алида; Вейганд, Александр Мартин; Лиз, Флориан (2018). «Простой протокол центрифугирования для метагеномных исследований увеличивает выход митохондриальной ДНК на два порядка» . Методы экологии и эволюции . 9 (4): 1070–1074. doi : 10.1111/2041-210x.12937 . ISSN 2041-210x .
- ^ "ДНККАНЕ" . Получено 2019-03-29 .
- ^ CEN (2018) CEN/TC 230/Рабочая группа 2 - предложение о новой рабочей группе WG28 «ДНК и методы EDNA». План удовлетворения потребностей в стандартизации ДНК и EDNA в отношении законодательства ЕС в водной политике (предложение после решений 2017 года 2017 года. Берлинское собрание CEN/TC 230, его рабочие группы и представители затрат на EDNA)
- ^ Слоан, Уильям Т.; Читать, Л. Фиона; Голова, Ян М.; Нил Холл; Давенпорт, Рассел Дж.; Кертис, Томас П.; Ланзен, Андерс; Куинс, Кристофер (2009). «Точное определение микробного разнообразия из 454 данных пиросеквенирования». Природные методы . 6 (9): 639–641. doi : 10.1038/nmeth.1361 . HDL : 1956/6529 . ISSN 1548-7105 . PMID 19668203 . S2CID 1975660 .
- ^ Кунин, Виктор; Энгельбректсон, Анна; Охман, Говард; Hugenholtz, Philip (2010). «Морщины в редкой биосфере: ошибки пиросеквенирования могут привести к искусственному инфляции оценок разнообразия» . Экологическая микробиология . 12 (1): 118–123. doi : 10.1111/j.1462-2920.2009.02051.x . ISSN 1462-2920 . PMID 19725865 . S2CID 19870165 .
- ^ Роб Найт; Ридер, Дженс (2009). «Редкая биосфера»: проверка реальности ». Природные методы . 6 (9): 636–637. doi : 10.1038/nmeth0909-636 . ISSN 1548-7105 . PMID 19718016 . S2CID 5278501 .
- ^ Чжан, Айбин; Хулак, Мартин; Сильвестр, Франциско; Хуан, Сяотинг; Adebayo, Abisola A.; Эббот, Кэтрин Л.; Адамович, Сара Дж.; Хит, Даниэль Д.; Cristescu, Melania E. (2013). «Высокая чувствительность 454 пиросеквенирования для обнаружения редких видов в водных сообществах» . Методы экологии и эволюции . 4 (6): 558–565. doi : 10.1111/2041-210x.12037 . HDL : 11336/2674 . ISSN 2041-210x . S2CID 53576369 .
- ^ Чжан, Айбин; Он, песня; Браун, Эмили А.; Цепь, Frédéric JJ; Террио, Томас У.; Эббот, Кэтрин Л.; Хит, Даниэль Д.; Cristescu, Melania E.; Macisaac, Hugh J. (2014). «Воспроизводимость данных пиросеквенирования для оценки биоразнообразия в сложных сообществах» . Методы экологии и эволюции . 5 (9): 881–890. doi : 10.1111/2041-210x.12230 . ISSN 2041-210x .
- ^ Chua, Physionia ys; Бурлат, Сара Дж.; Фергюсон, Кэмерон; Корлевич, Петра; Чжао, Лея; Экрем, Торбьёрн; Мейер, Рудольф; Lawniczak, Mara Kn (10 марта 2023 г.). «Будущее мониторинга насекомых на основе ДНК» . Тенденции в генетике . 39 (7): 531–544. doi : 10.1016/j.tig.2023.02.012 . PMID 36907721 . S2CID 257470926 .
- ^ Шриватсан, Амрита; Хартоп, Эмили; Пуниаморти, Джаянти; Ли, Ван Тинг; Кутти, Суджата Нараянан; Курина, Олави; Мейер, Рудольф (декабрь 2019 г.). «Быстрое, крупномасштабное открытие видов у гипердиверных таксонов с использованием 1D-секвенирования миньона» . BMC Biology . 17 (1): 96. doi : 10.1186/s12915-019-0706-9 . PMC 6884855 . PMID 31783752 .
- ^ Шриватсан, Амрита; Ли, Лешон; Като, Казутака; Хартоп, Эмили; Кутти, Суджата Нараянан; Вонг, Джонтан; Yeo, Даррен; Мейер, Рудольф (декабрь 2021 г.). «Штрих -коды Ontbarcoder и Minion помогают всем, чтобы каждый, для всех» . BMC Biology . 19 (1): 217. DOI : 10.1186/S12915-021-01141-X . PMC 8479912 . PMID 34587965 .
- ^ Шриватсан, Амрита; Балоглу, Билгенур; Ван, Венди; Тан, Вей Х.; Бертран, Денис; Ng, amanda hq; Boey, Esther JH; Ко, Джейс Джи; Нагараджан, Ниранджан; Мейер, Рудольф (сентябрь 2018 г.). «Миньон ™ трубопровод для быстрого и экономически эффективного штрих-кодирования ДНК». Молекулярные экологические ресурсы . 18 (5): 1035–1049. doi : 10.1111/1755-0998.12890 . PMID 29673082 . S2CID 4982474 .
- ^ Мейер, Рудольф; Вонг, Кинга; Шриватсан, Амрита; Foo, Maosheng (февраль 2016 г.). «Штрих-коды ДНК за 1 доллар для реконструкции сложных явлений и поиска редких видов в образцах богатых образцами» . Кладистика . 32 (1): 100–110. doi : 10.1111/cla.12115 . PMID 34732017 . S2CID 83862072 .
- ^ Хеберт, Пол Д.Н.; Браукманн, Томас Ва; Проссер, Шон В.Дж.; Ратнасингем, Судживан; Dewaard, Джереми Р.; Иванова, Наталья В.; Янзен, Даниэль Х.; Hallwachs, Winnie; Найк, Суреш; Sones, Jayme E.; Захаров, Евгений В. (27 марта 2018 г.). «Продолжение Sanger: секвенирование Amplicon, которое масштабируется» . BMC Genomics . 19 (1): 219. DOI : 10.1186/S12864-018-4611-3 . PMC 5870082 . PMID 29580219 .
- ^ Шриватсан, Амрита; Ючен; Heraty, Джон М.; Hwang, Wei Song; Юсу, Ван Фа; Cutty, Sujatha Train; Puniamorthy, Jayanthhi; Да, Даррен; Рослин, Томас; Мейер, Рудольф (4 августа 2022 г.). Глобальное и наклонное насекомое . Biorxiv doi : 10.1101/ 2022.08.502512 S2CID 251369606 .
- ^ Фернандес-Триана, Хосе Л. (25 февраля 2022 г.). «Турбосономия подходов: уроки из прошлого и рекомендации на будущее, основанные на опыте с паразитоидными осами Braconidae (Hymenoptera)» . Zookeys (1087): 199–220. doi : 10.3897/Zookeys.1087.76720 . PMC 8897373 . PMID 35585942 .
- ^ Балоглу, Билгенур; КЛЕВС, Эстер; Мейер, Рудольф (декабрь 2018 г.). «Штрих -кодирование NGS выявляет высокую резистентность гипердиверной хирономидовой (Diptera) фауны против вторжения из прилегающих пресноводных водохранилищ» . Границы в зоологии . 15 (1): 31. DOI : 10.1186/S12983-018-0276-7 . PMC 6092845 . PMID 30127839 .
- ^ Yeo, Даррен; Шриватсан, Амрита; Punamoorthy, Jayanthhi; Maasheng, fo; Грурт, Патрик; Чан, Лена; Генард, Бенуа; Дамкен, Clas; Вахаб, Родзай А.; Ючен, Анг; Мейер, Рудольф (14 сентября 2021 года). «Мангровые леса - это непрерывная точка разнообразия насекомых, несмотря на низкое разнообразие растений » BMC Biology 19 (1): 202. DOI : 10.1186/S12915-021-01088- Z 8442405PMC 34521395PMID
- ^ Гейгер, Матиас; Мориньер, Джером; Хаусманн, Аксель; Haszprunar, Gerhard; Wägele, Wolfgang; Хеберт, Пол; Рулик, Бьорн (1 декабря 2016 года). «Тестирование глобальной программы ловушек Malaise - насколько хорошо текущая справочная библиотека штрих -кода идентифицирует летающих насекомых в Германии?» Полем Журнал данных о биоразнообразии . 4 (4): E10671. doi : 10.3897/bdj.4.e10671 . PMC 5136679 . PMID 27932930 .
- ^ Хеберт, Пол Д.Н.; Ратнасингем, Судживан; Захаров, Евгений В.; Телфорт, Анжела С.; Levesque-beaudin, Валери; Милтон, Меган А.; Педерсен, Стефани; Джаннетта, Пол; Деваард, Джереми Р. (5 сентября 2016 г.). «Подсчет видов животных с штрих -кодами ДНК: канадские насекомые» . Философские транзакции Королевского общества B: биологические науки . 371 (1702): 20150333. DOI : 10.1098/rstb.2015.0333 . PMC 4971185 . PMID 27481785 .
- ^ Телфер, Анжела; и др. (30 августа 2015 г.). «Инвентаризация биоразнообразия в высокой передаче: штрих -кодирование ДНК облегчает быстрое биотическое обследование умеренного природного заповедника» . Журнал данных о биоразнообразии . 3 (3): E6313. doi : 10.3897/bdj.3.e6313 . PMC 4568406 . PMID 26379469 .
- ^ Д'Суза, Мишель Л.; Ван дер Банк, Мишель; Shongwe, Zandisile; Rattray, Ryan D.; Стюарт, Росс; Ван Ройен, Джохандре; Говендер, Дэнни; Хеберт, Пол Д.Н. (апрель 2021 г.). «Базовые линии биоразнообразия: отслеживание насекомых в национальном парке Крюгера с штрих -кодами ДНК» . Биологическое сохранение . 256 : 109034. DOI : 10.1016/j.biocon.2021.109034 . HDL : 2263/81603 . S2CID 233489409 .
- ^ Yeo, Даррен; Пуниаморти, Джаянти; Нгиам, Робин Вэнь Цзян; Мейер, Рудольф (октябрь 2018 г.). «На пути к голоморфологии в энтомологии: быстрое и экономически эффективное сопоставление взрослых-ларва с использованием штрих-кодов NGS: сопоставление стадии жизни жизни с штрих-кодами NGS». Систематическая энтомология . 43 (4): 678–691. doi : 10.1111/syen.12296 . S2CID 49211569 .
- ^ Пьер, Таберлет (2018-02-02). ДНК окружающей среды: для исследования и мониторинга биоразнообразия . Бонин, Аурели, 1979-. Оксфорд. ISBN 9780191079993 Полем OCLC 1021883023 .
{{cite book}}: CS1 Maint: местоположение отсутствует издатель ( ссылка ) - ^ Рупперт, Криста М.; Клайн, Ричард Дж.; Рахман, доктор медицинских наук (январь 2019 г.). «Прошлые, настоящие и будущие перспективы метабардодирования ДНК окружающей среды (EDNA): систематический обзор методов, мониторинга и применения глобальной EDNA» . Глобальная экология и сохранение . 17 : E00547. doi : 10.1016/j.gecco.2019.e00547 .
- ^ Сток, Торстен; Frühe, Larissa; Форстер, Доминик; Кордиер, Тристан; Мартинс, Катарина IM; Pawlowski, январь (февраль 2018 г.). «Экологическая ДНК метабардодирование бентических бактериальных сообществ указывает на бентическую следование аквакультуры лосося» . Бюллетень загрязнения морской пехоты . 127 : 139–149. doi : 10.1016/j.marpolbul.2017.11.065 . PMID 29475645 .
- ^ Эванс, Даррен М.; Китсон, Джеймс Дж. Н. Lunt, David H.; Солома, Найджел А.; Покок, Майкл Джо (2016). «Слияние метабардодирования ДНК и анализа экологического сети, чтобы понять и создать устойчивые наземные экосистемы» (PDF) . Функциональная экология . 30 (12): 1904–1916. doi : 10.1111/1365-2435.12659 . ISSN 1365-2435 .
- ^ Puddu, M.; Paunescu, D.; Старк, WJ; Grass, RN (2014). «Магнитно восстанавливаемая, термостабильная, гидрофобная ДНК/кремнезем инкапсулирует, а их применение в виде невидимых меток масла». ACS Nano . 8 (3): 2677–2685. doi : 10.1021/nn4063853 . PMID 24568212 .