АльфаФолд
| Часть серии о |
| Искусственный интеллект |
|---|
AlphaFold — это программа искусственного интеллекта (ИИ), разработанная DeepMind , дочерней компанией Alphabet , которая выполняет прогнозирование структуры белков . [1] Программа спроектирована как система глубокого обучения . [2]
Программное обеспечение AlphaFold имело три основные версии. Команда исследователей, использовавшая AlphaFold 1 (2018), заняла первое место в общем рейтинге 13-й критической оценки прогнозирования структуры (CASP) в декабре 2018 года. Программа особенно успешно предсказала наиболее точную структуру для целей, оцененных как наиболее сложные. организаторами конкурса, где не было существующих матричных структур из белков с частично сходной последовательностью. Команда, использовавшая AlphaFold 2 (2020), повторила свое место в конкурсе CASP14 в ноябре 2020 года. [3] Команда достигла уровня точности, намного более высокого, чем любая другая группа. [2] [4] Он набрал более 90 баллов примерно для двух третей белков в глобальном дистанционном тесте CASP (GDT), тесте, который измеряет степень, в которой предсказанная вычислительной программой структура похожа на структуру, определенную в лабораторном эксперименте, причем 100 означает полное совпадение. в пределах расстояния отсечки, используемого для расчета GDT. [2] [5]
Результаты AlphaFold 2 на CASP14 были названы «поразительными». [6] и «трансформационный». [7] Некоторые исследователи отметили, что точность недостаточно высока для трети его предсказаний и что она не раскрывает механизм или правила сворачивания белка , чтобы проблему сворачивания белка можно было считать решенной. [8] [9] Тем не менее, это техническое достижение вызвало широкое уважение. 15 июля 2021 года статья AlphaFold 2 была опубликована в журнале Nature в качестве публикации с предварительным доступом вместе с программным обеспечением с открытым исходным кодом видов с возможностью поиска и базой данных протеомов . [10] [11] [12]
AlphaFold 3 был анонсирован 8 мая 2024 года. Он может предсказывать структуру комплексов белков с ДНК , РНК , различными лигандами и ионами . [13]
Фон
[ редактировать ]
Белки состоят из цепочек аминокислот , которые спонтанно сворачиваются , образуя трехмерные (3-D) структуры белков. Трехмерная структура имеет решающее значение для понимания биологической функции белка.
Структуры белков можно определить экспериментально с помощью таких методов, как рентгеновская кристаллография , криоэлектронная микроскопия и ядерный магнитный резонанс , которые являются дорогостоящими и отнимают много времени. [14] Такие усилия, используя экспериментальные методы, идентифицировали структуры около 170 000 белков за последние 60 лет, в то время как во всех формах жизни известно более 200 миллионов белков. [5]
На протяжении многих лет исследователи применяли многочисленные вычислительные методы для предсказания трехмерных структур белков по их аминокислотным последовательностям, но точность таких методов не приближалась к экспериментальным методам. Программа CASP , которая была запущена в 1994 году с целью бросить вызов научному сообществу предоставить наилучшие предсказания структуры белков, обнаружила, что по GDT . к 2016 году для наиболее сложных белков можно достичь лишь около 40 баллов из 100 [5] AlphaFold начала участвовать в CASP 2018 года, используя искусственного интеллекта (ИИ) технику глубокого обучения . [14]
Алгоритм
[ редактировать ]Известно, что DeepMind обучил программу более чем 170 000 белков из общедоступного хранилища белковых последовательностей и структур. В программе используется своего рода сеть внимания — метод глубокого обучения , который фокусируется на том, чтобы ИИ идентифицировал части более крупной проблемы, а затем соединял их воедино, чтобы получить общее решение. [2] Общее обучение проводилось на вычислительной мощности от 100 до 200 графических процессоров . [2]
АльфаФолд 1 (2018)
[ редактировать ]AlphaFold 1 (2018) был основан на работе, разработанной различными командами в 2010-х годах, работе, в которой рассматривались большие банки данных связанных последовательностей ДНК, которые теперь доступны из многих различных организмов (большинство из которых не имеют известных трехмерных структур), чтобы попытаться найти изменения в различных остатках. это, по-видимому, коррелировало, хотя остатки не были последовательными в основной цепи. Такие корреляции предполагают, что остатки могут быть близки друг к другу физически, даже если они не близки по последовательности, что позволяет карту контактов оценить . Основываясь на недавней работе, проведенной до 2018 года, AlphaFold 1 расширил это, чтобы оценить распределение вероятностей того, насколько близкими могут быть остатки, превратив карту контактов в карту вероятных расстояний. Для разработки выводов также использовались более продвинутые методы обучения, чем раньше. [15] [16]
АльфаФолд 2 (2020)
[ редактировать ]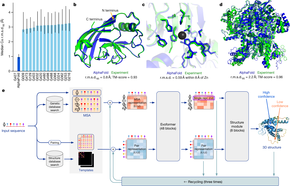

версия программы 2020 года ( AlphaFold 2 , 2020) существенно отличается от оригинальной версии, которая выиграла CASP 13 в 2018 году. По словам команды DeepMind, [18] [19]
Программное обеспечение, использованное в AlphaFold 1, содержало ряд модулей, каждый из которых обучался отдельно, которые использовались для создания направляющего потенциала, который затем объединялся с основанным на физике энергетическим потенциалом. AlphaFold 2 заменил это системой подсетей, объединенных в единую дифференцируемую сквозную модель, полностью основанную на распознавании образов, которая была интегрированно обучена как единая интегрированная структура. [19] [20] Локальная физика в форме энергетического уточнения на основе модели AMBER применяется только в качестве последнего этапа уточнения после того, как прогноз нейронной сети сходится, и лишь незначительно корректирует предсказанную структуру. [21]
Ключевой частью системы 2020 являются два модуля, предположительно основанные на конструкции преобразователя , которые используются для постепенного уточнения вектора информации для каждого отношения (или « ребра » в терминологии теории графов) между аминокислотным остатком белок и другой аминокислотный остаток (эти связи представлены массивом, показанным зеленым цветом); и между каждой позицией аминокислоты и каждой другой последовательностью при выравнивании входной последовательности (эти отношения представлены массивом, показанным красным). [20] Внутри эти уточняющие преобразования содержат слои, которые объединяют соответствующие данные и отфильтровывают ненужные данные («механизм внимания») для этих отношений контекстно-зависимым способом, полученным из обучающих данных. Эти преобразования повторяются, обновленная информация, выдаваемая на одном этапе, становится входной информацией для следующего, при этом уточненная информация об остатках/остатках подается в обновление информации об остатках/последовательности, а затем улучшенная информация об остатках/последовательности подается в обновление информация об остатке/остатке. [20] Согласно одному отчету, по ходу итерации «алгоритм внимания… имитирует способ, которым человек собирает головоломку: сначала соединяет части в небольшие комочки — в данном случае кластеры аминокислот — а затем ищет способы соединить их». сгустки в единое целое». [5] [ нужно обновить ]
Результаты этих итераций затем передаются окончательному модулю прогнозирования структуры: [20] где также используются трансформаторы, [22] и затем сам повторяется. В примере, представленном DeepMind, модуль прогнозирования структуры достиг правильной топологии целевого белка на первой итерации, получив GDT_TS 78, но с большим количеством (90%) стереохимических нарушений – т.е. нефизических валентных углов или длины. При последующих итерациях количество стереохимических нарушений уменьшалось. К третьей итерации GDT_TS прогноза приближался к 90, а к восьмой итерации количество стереохимических нарушений приближалось к нулю. [23]
Данные обучения изначально были ограничены отдельными пептидными цепями. Однако обновление за октябрь 2021 года, получившее название AlphaFold-Multimer, включило в данные обучения белковые комплексы. DeepMind заявила, что это обновление позволило примерно в 70% случаев точно предсказать белок-белковые взаимодействия. [24]
АльфаФолд 3 (2024)
[ редактировать ], анонсированный 8 мая 2024 года, AlphaFold 3 был разработан совместно Google DeepMind и Isomorphic Labs , дочерними компаниями Alphabet . AlphaFold 3 не ограничивается одноцепочечными белками, поскольку он также может предсказывать структуры белковых комплексов с ДНК , РНК , посттрансляционные модификации и выбранные лиганды и ионы . [25] [13]
AlphaFold 3 представляет «Pairformer», архитектуру глубокого обучения, вдохновленную трансформатором, которая считается похожей, но более простой, чем Evoformer, представленный в AlphaFold 2. [26] [27] Необработанные прогнозы из модуля Pairformer передаются в диффузионную модель , которая начинается с облака атомов и использует эти прогнозы для итеративного продвижения к трехмерному изображению молекулярной структуры. [13]
Сервер AlphaFold был создан для предоставления бесплатного доступа к AlphaFold 3 для некоммерческих исследований. [28]
Соревнования
[ редактировать ]
Красная линия тренда показывает, как несколько моделей, включая AlphaFold 1, достигли в 2018 году значительного скачка по сравнению с темпами прогресса, достигнутыми ранее, особенно в отношении белковых последовательностей, которые считаются наиболее трудно прогнозируемыми.
(Качественное улучшение было достигнуто и в более ранние годы, но только когда изменения приводят к тому, что структуры находятся в пределах 8 Å от их экспериментальных положений, они начинают влиять на показатель CASP GDS-TS).
Оранжевая линия тренда показывает, что к 2020 году серверы онлайн-прогнозирования смогли извлечь уроки из этой производительности и соответствовать ей, в то время как другие лучшие группы (зеленая кривая) в среднем смогли ее улучшить. Однако черная кривая тренда показывает, в какой степени AlphaFold 2 снова превзошел этот показатель в 2020 году по всем направлениям.
Подробный разброс точек данных указывает на степень согласованности или вариаций, достигнутую с помощью AlphaFold. Выбросы представляют собой несколько последовательностей, для которых не удалось сделать такой успешный прогноз.
CASP13
[ редактировать ]В декабре 2018 года AlphaFold компании DeepMind заняла первое место в общем рейтинге 13-й критической оценки методов прогнозирования структуры белка (CASP). [29] [30]
Программа особенно успешно предсказывала наиболее точную структуру для мишеней, которые организаторы конкурса считали наиболее сложными, где не было существующих матричных структур из белков с частично сходной последовательностью. AlphaFold дал лучший прогноз для 25 из 43 белков-мишеней этого класса. [30] [31] [32] CASP достижение среднего балла 58,9 по глобальному дистанционному тесту (GDT), опередив 52,5 и 52,4 у двух следующих лучших команд, [33] которые также использовали глубокое обучение для оценки контактных расстояний. [34] [35] В целом по всем целевым показателям программа получила оценку GDT 68,5. [36]
В январе 2020 года реализации и иллюстративный код AlphaFold 1 были опубликованы с открытым исходным кодом на GitHub . [37] [14] но, как указано в файле «Read Me» на этом веб-сайте: «Этот код нельзя использовать для прогнозирования структуры произвольной белковой последовательности. Его можно использовать для прогнозирования структуры только в наборе данных CASP13 (ссылки ниже). Код генерации функций тесно связан с нашей внутренней инфраструктурой, а также с внешними инструментами, поэтому мы не можем открыть его исходный код». Поэтому, по сути, депонированный код не пригоден для общего использования, а только для белков CASP13. Компания не объявила о планах сделать свой код общедоступным с 5 марта 2021 года.
CASP14
[ редактировать ]В ноябре 2020 года новая версия DeepMind AlphaFold 2 выиграла CASP14. [38] [39] В целом AlphaFold 2 дал лучший прогноз для 88 из 97 целей. [6]
По предпочтительному показателю точности глобального дистанционного теста (GDT) программа получила средний балл 92,4 (из 100), что означает, что более половины ее прогнозов были оценены на уровне выше 92,4% из-за того, что их атомы находятся в более или-менее подходящее место, [40] [41] Сообщается, что уровень точности сопоставим с экспериментальными методами, такими как рентгеновская кристаллография . [18] [7] [36] В 2018 году AlphaFold 1 достиг такого уровня точности только в двух из всех своих прогнозов. [6] 88% прогнозов в конкурсе 2020 года имели балл GDT_TS более 80. В группе целей, классифицированных как самые сложные, AlphaFold 2 достиг среднего балла 87.
Измеряется среднеквадратическим отклонением (RMS-D) расположения альфа-углеродных атомов основной цепи белка, в котором, как правило, преобладают характеристики наименее подходящих выбросов, 88% прогнозов AlphaFold 2. имело среднеквадратичное отклонение менее 4 Å для набора перекрывающихся атомов C-альфа. [6] 76% прогнозов были лучше, чем 3 Å, а 46% имели среднеквадратическую точность атома C-альфа лучше, чем 2 Å, [6] со средним среднеквадратичным отклонением в своих предсказаниях 2,1 Å для набора перекрывающихся атомов СА. [6] AlphaFold 2 также достиг точности моделирования поверхностных боковых цепей , которую можно назвать «действительно-выдающейся».
Чтобы дополнительно проверить AlphaFold-2, организаторы конференции обратились к четырем ведущим экспериментальным группам по поводу структур, которые они считали особенно сложными и которые не смогли определить. Во всех четырех случаях трехмерные модели, созданные AlphaFold 2, были достаточно точными, чтобы определить структуры этих белков путем молекулярной замены . В их число входила мишень T1100 (Af1503), небольшой мембранный белок, изучаемый экспериментаторами в течение десяти лет. [5]
Из трех структур, предсказание которых с помощью AlphaFold 2 имело наименьший успех, две были получены методами ЯМР белков , которые определяют структуру белка непосредственно в водном растворе, тогда как AlphaFold в основном обучался на белковых структурах в кристаллах . Третий существует в природе как мультидоменный комплекс, состоящий из 52 идентичных копий одного и того же домена , но AlphaFold не был запрограммирован учитывать эту ситуацию. Для всех мишеней с одним доменом, за исключением только одного очень большого белка и двух структур, определенных с помощью ЯМР, AlphaFold 2 достиг показателя GDT_TS более 80.
CASP15
[ редактировать ]В 2022 году DeepMind не участвовала в CASP15, но большинство участников использовали AlphaFold или инструменты, включающие AlphaFold. [42]
Прием
[ редактировать ]AlphaFold 2, набравший более 90 баллов в CASP глобальном дистанционном тесте (GDT), считается значительным достижением в области вычислительной биологии. [5] и большой прогресс в решении великой задачи биологии, которая существует уже несколько десятилетий. [7] Лауреат Нобелевской премии и структурный биолог Венки Рамакришнан назвал результат «ошеломляющим достижением в решении проблемы сворачивания белка». [5] добавив, что «это произошло на десятилетия раньше, чем могли предсказать многие люди в этой области. Будет интересно увидеть, как это фундаментально изменит биологические исследования». [38]
На основе пресс-релизов CASP и DeepMind, [43] [38] Успех AlphaFold 2 привлек широкое внимание средств массовой информации. [44] А также новости в специализированной научной прессе, такой как Nature , [7] Наука , [5] Обзор технологий MIT , [2] и Новый учёный , [45] [46] эта история широко освещалась в крупных национальных газетах. [47] [48] [49] [50] Частой темой было то, что способность точно предсказывать структуру белка на основе составляющей аминокислотной последовательности, как ожидается, будет иметь широкий спектр преимуществ в области наук о жизни, включая ускорение открытия передовых лекарств и обеспечение лучшего понимания болезней. [7] [51] Некоторые отмечают, что даже идеальный ответ на проблему прогнозирования белков все равно оставит вопросы о проблеме сворачивания белков — детальное понимание того, как процесс сворачивания на самом деле происходит в природе (и как иногда они могут сворачиваться неправильно ). [52]
В 2023 году Демис Хассабис и Джон Джампер получили премию за прорыв в области наук о жизни. [53] а также премию Альберта Ласкера за фундаментальные медицинские исследования за управление проектом AlphaFold. [54]
Исходный код
[ редактировать ]Открытый доступ к исходному коду нескольких версий AlphaFold (за исключением AlphaFold 3) был предоставлен DeepMind по запросам научного сообщества. [55] [56] [57] Ожидается, что полный исходный код AlphaFold-3 будет предоставлен в открытый доступ к концу 2024 года. [58] [59]
База данных моделей белков, созданная AlphaFold
[ редактировать ]| Содержание | |
|---|---|
| Типы данных захвачен | предсказание структуры белка |
| Организмы | все протеомы UniProt |
| Контакт | |
| Исследовательский центр | ЭМБЛ-ЭБИ |
| Первичное цитирование | [10] |
| Доступ | |
| Веб-сайт | https://www.alphafold.ebi.ac.uk/ |
| URL-адрес загрузки | да |
| Инструменты | |
| Интернет | да |
| Разнообразный | |
| Лицензия | CC-BY 4.0 |
| Политика курирования | автоматический |
База данных о структуре белков AlphaFold была запущена 22 июля 2021 года в результате совместных усилий AlphaFold и EMBL-EBI . На момент запуска база данных содержит предсказанные AlphaFold модели белковых структур почти полного UniProt протеома человека и 20 модельных организмов , насчитывающих более 365 000 белков. менее 16 и более 2700 В базу данных не включены белки с числом аминокислотных остатков . [60] но для людей они доступны во всем пакетном файле. [61] AlphaFold планировала добавить в коллекцию больше последовательностей, первоначальная цель (на начало 2022 года) заключалась в том, чтобы охватить большую часть набора UniRef90, состоящего из более чем 100 миллионов белков. По состоянию на 15 мая 2022 года было доступно 992 316 прогнозов. [62]
В июле 2021 года ЮниПрот-КБ и ИнтерПро [63] был обновлен, чтобы показывать прогнозы AlphaFold, когда они доступны. [64]
28 июля 2022 года команда загрузила в базу данных структуры около 200 миллионов белков 1 миллиона видов, охватывающие почти все известные белки на планете. [65]
Ограничения
[ редактировать ]AlphaFold имеет различные ограничения:
- AlphaFold DB предоставляет мономерные модели белков, а не их биологически значимые комплексы . [66]
- Многие белковые области прогнозируются с низкой степенью достоверности, включая внутренне неупорядоченные белковые области. [67]
- Афафолд-2 был проверен для прогнозирования структурных эффектов мутаций с ограниченным успехом. [68]
- Модель в некоторой степени полагается на информацию о коэволюции сходных белков и, следовательно, может неэффективно работать с синтетическими белками или белками с очень низкой гомологией с чем-либо в базе данных. [69]
- Способность модели создавать множественные нативные конформации белков ограничена.
- Версия AlphaFold 3 может предсказывать структуры белковых комплексов с очень ограниченным набором выбранных кофакторов и ко- и посттрансляционных модификаций . [70] От 50% до 70% структур протеома человека являются неполными без ковалентно связанных гликанов. [71] [66] AlphaFill, производная база данных, при необходимости добавляет кофакторы к моделям AlphaFold. [72]
- В алгоритме остатки перемещаются свободно, без каких-либо ограничений. Поэтому при моделировании целостность цепочки не сохраняется. В результате AlphaFold может давать топологически неверные результаты, например, структуры с произвольным количеством узлов. [73]
Приложения
[ редактировать ]AlphaFold использовался для прогнозирования структур белков SARS-CoV-2 , возбудителя COVID-19 . Структуры этих белков ожидали экспериментального обнаружения в начале 2020 года. [74] [7] Результаты были изучены учеными из Института Фрэнсиса Крика в Соединенном Королевстве, а затем опубликованы более широкому исследовательскому сообществу. Команда также подтвердила точные предсказания относительно экспериментально определенного белка-шипа SARS-CoV-2 , который был опубликован в Protein Data Bank , международной базе данных с открытым доступом, прежде чем опубликовать определенные с помощью вычислений структуры недостаточно изученных белковых молекул. [75] Команда признала, что, хотя эти белковые структуры, возможно, и не являются предметом текущих терапевтических исследований, они внесут вклад в понимание сообществом вируса SARS-CoV-2. [75] В частности, предсказание AlphaFold 2 структуры белка ORF3a было очень похоже на структуру, определенную исследователями из Калифорнийского университета в Беркли с помощью криоэлектронной микроскопии . Считается, что этот специфический белок помогает вирусу вырваться из клетки-хозяина после его репликации. Считается также, что этот белок играет роль в запуске воспалительной реакции на инфекцию. [76]
Опубликованные работы
[ редактировать ]- Эндрю В. Старший и др. (Декабрь 2019 г.), «Прогнозирование структуры белка с использованием нескольких глубоких нейронных сетей в 13-й критической оценке прогнозирования структуры белка (CASP13)» , Белки: структура, функции, биоинформатика 87 (12) 1141–1148 два : 10.1002/прот.25834
- Эндрю В. Старший и др. (15 января 2020 г.), «Улучшенное предсказание структуры белка с использованием потенциала глубокого обучения» , Nature 577 706–710. два : 10.1038/s41586-019-1923-7
- Джон Джампер и др. (декабрь 2020 г.), «Высокоточное предсказание структуры белка с использованием глубокого обучения», в Четырнадцатой критической оценке методов прогнозирования структуры белка (реферат) , стр. 22–24.
- Джон Джампер и др. (декабрь 2020 г.), « АльфаФолд 2 ». Презентация на CASP 14.
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ «АльфаФолд» . Глубокий разум . Проверено 30 ноября 2020 г. .
- ^ Jump up to: а б с д и ж «ИИ компании DeepMind, сворачивающий белки, решил грандиозную проблему биологии, возникшую 50 лет назад» . Обзор технологий Массачусетского технологического института . Проверено 30 ноября 2020 г.
- ^ Шид, Сэм (30 ноября 2020 г.). «DeepMind решает 50-летнюю «большую задачу» с помощью искусственного интеллекта по сворачиванию белков», CNBC . Проверено 30 ноября 2020 г.
- ^ Стоддарт, Шарлотта (1 марта 2022 г.). «Структурная биология: как белки оказались крупным планом» . Знающий журнал . doi : 10.1146/knowable-022822-1 . S2CID 247206999 . Проверено 25 марта 2022 г.
- ^ Jump up to: а б с д и ж г час Роберт Ф. Сервис: «Игра изменилась». ИИ побеждает в решении белковых структур , Наука , 30 ноября 2020 г.
- ^ Jump up to: а б с д и ж Мохаммед Аль-Кураиши, только что стали известны результаты CASP14, и они поразительны , Твиттер, 30 ноября 2020 г.
- ^ Jump up to: а б с д и ж Каллауэй, Юэн (30 ноября 2020 г.). « Это изменит все»: искусственный интеллект DeepMind совершает гигантский скачок в решении белковых структур». Природа . 588 (7837): 203–204. Бибкод : 2020Natur.588..203C . дои : 10.1038/d41586-020-03348-4 . ПМИД 33257889 . S2CID 227243204 .
- ^ Стивен Карри, Нет, DeepMind не решил проблему сворачивания белка , Reciprocal Space (блог), 2 декабря 2020 г.
- ^ Боллс, Филипп (9 декабря 2020 г.). «За ширмами AlphaFold» . Химический мир .
- ^ Jump up to: а б Джампер, Джон; Эванс, Ричард; Притцель, Александр; Грин, Тим; Фигурнов Михаил; Роннебергер, Олаф; Туньясувунакул, Кэтрин; Бейтс, Расс; Жидек, Августин; Потапенко, Анна; Бриджланд, Алекс; Мейер, Клеменс; Коль, Саймон А.А.; Баллард, Эндрю Дж; Коуи, Эндрю; Ромера-Паредес, Бернардино; Николов Станислав; Джайн, Ришуб; Адлер, Йонас; Назад, Тревор; Петерсен, Стиг; Рейман, Дэвид; Клэнси, Эллен; Зелински, Михал; Штайнеггер, Мартин; Пачольска, Михалина; Бергаммер, Томас; Боденштейн, Себастьян; Сильвер, Дэвид; Виньялс, Иволга; Старший, Эндрю В.; Кавукчуоглу, Корай; Кохли, Пушмит; Хассабис, Демис (15 июля 2021 г.). «Высокоточное предсказание структуры белка с помощью AlphaFold» . Природа . 596 (7873): 583–589. Бибкод : 2021Nature.596..583J . дои : 10.1038/s41586-021-03819-2 . ПМЦ 8371605 . PMID 34265844 .
- ^ «GitHub — deepmind/alphafold: открытый исходный код для AlphaFold » Гитхаб . Получено 2 июля 2021 г.
- ^ «База данных структуры белков AlphaFold» . Alphafold.ebi.ac.uk . Проверено 24 июля 2021 г.
- ^ Jump up to: а б с «AlphaFold 3 предсказывает структуру и взаимодействие всех молекул жизни» . Google . 08 мая 2024 г. Проверено 9 мая 2024 г.
- ^ Jump up to: а б с «AlphaFold: Использование ИИ для научных открытий» . Глубокий разум . 15 января 2020 г. Проверено 30 ноября 2020 г.
- ^ Мохаммед Аль-Кураиши (май 2019 г.), AlphaFold на CASP13 , Биоинформатика , 35 (22), 4862–4865 doi : 10.1093/биоинформатика/btz422 . См. также Мохаммед Аль-Курайши (9 декабря 2018 г.), AlphaFold @ CASP13: «Что только что произошло?» (сообщение в блоге).
Мохаммед Аль-Курайши (15 января 2020 г.), Переломный момент в предсказании структуры белков , Nature 577 , 627–628. два : 10.1038/d41586-019-03951-0 - ^ AlphaFold: Машинное обучение для прогнозирования структуры белков , Foldit , 31 января 2020 г.
- ^ Jump up to: а б Джампер, Джон; и др. (август 2021 г.). «Высокоточное предсказание структуры белка с помощью AlphaFold» . Природа . 596 (7873): 583–589. Бибкод : 2021Natur.596..583J . дои : 10.1038/s41586-021-03819-2 . ISSN 1476-4687 . ПМЦ 8371605 . PMID 34265844 .
- ^ Jump up to: а б «DeepMind отвечает на одну из самых больших проблем биологии» . Экономист . 2020-11-30. ISSN 0013-0613 . Проверено 30 ноября 2020 г.
- ^ Jump up to: а б Джереми Кан, Уроки прорыва DeepMind в области искусственного интеллекта, складывающего белки , Fortune , 1 декабря 2020 г.
- ^ Jump up to: а б с д См. блок-схему. Также Джон Джампер и др. (1 декабря 2020 г.), Презентация AlphaFold 2 , Слайд
- ^ Джон Джампер и др., тезисы конференции (декабрь 2020 г.)
- ^ Заявлено, что модуль структуры использует «3-мерную эквивариантную архитектуру преобразователя» (Джон Джампер и др. (1 декабря 2020 г.), презентация AlphaFold 2 , слайд 12).
Одна конструкция трансформаторной сети с SE (3) была эквивариантностью предложена в работе Fabian Fuchs et al SE (3)-Transformers: 3D Roto-Translation Equivariant Attention Networks , NeurIPS 2020; также веб-сайт . Неизвестно, насколько это может быть похоже или не похоже на то, что использовалось в AlphaFold.
См. также сообщение в блоге AlQuaraishi об этом или более подробное сообщение Фабиана Фукса. - ^ Джон Джампер и др. (1 декабря 2020 г.), презентация AlphaFold 2 , слайды с 12 по
- ^ Каллауэй, Юэн (13 апреля 2022 г.). «Что будет дальше с AlphaFold и революцией в складывании белков ИИ» . Природа . 604 (7905): 234–238. Бибкод : 2022Natur.604..234C . дои : 10.1038/d41586-022-00997-5 . ПМИД 35418629 . S2CID 248156195 .
- ^ Мец, Кейд (08 мая 2024 г.). «Google представляет искусственный интеллект для прогнозирования поведения молекул человека» . Нью-Йорк Таймс . ISSN 0362-4331 . Проверено 9 мая 2024 г.
- ^ Абрамсон, Джош; Адлер, Йонас; Дангер, Джек; Эванс, Ричард; Грин, Тим; Притцель, Александр; Роннебергер, Олаф; Уиллмор, Линдси; Баллард, Эндрю Дж.; Бамбрик, Джошуа; Боденштейн, Себастьян В.; Эванс, Дэвид А.; Хун, Чиа-Чун; О'Нил, Майкл; Рейман, Дэвид (08 мая 2024 г.). «Точное предсказание структуры биомолекулярных взаимодействий с помощью AlphaFold 3» . Природа : 1–3. doi : 10.1038/s41586-024-07487-w . ISSN 1476-4687 . ПМЦ 11168924 .
- ^ Точное предсказание структуры биомолекулярных взаимодействий с помощью AlphaFold 3 , pdf препринта статьи в журнале Nature.
- ^ Некоммерческий сервер AlphaFold-3.
- ^ Производительность группы на основе комбинированных z-показателей , CASP 13, декабрь 2018 г. (AlphaFold = Team 043: A7D)
- ^ Jump up to: а б Сэмпл, Ян (2 декабря 2018 г.). «DeepMind от Google предсказывает трехмерные формы белков» . Хранитель . Проверено 30 ноября 2020 г. .
- ^ «AlphaFold: Использование ИИ для научных открытий» . Глубокий разум . Проверено 30 ноября 2020 г. .
- ^ Сингх, Арунима (2020). «Глубокое обучение 3D-структур» . Природные методы . 17 (3): 249. doi : 10.1038/s41592-020-0779-y . ISSN 1548-7105 . ПМИД 32132733 . S2CID 212403708 .
- ^ См. таблицы данных CASP 13 для 043 A7D, 322 Zhang и 089 MULTICOM.
- ^ Вэй Чжэн и др ., Прогнозирование структуры белка на основе контактной карты глубокого обучения в CASP13 , Белки: структура, функции и биоинформатика , 87 (12) 1149–1164 дои : 10.1002/прот.25792 ; и слайды
- ^ Хоу, Цзе; Ву, Тяньци; Цао, Ренжи; Ченг, Цзяньлинь (25 апреля 2019 г.). «Моделирование третичной структуры белка на основе глубокого обучения и прогнозирования контактного расстояния в CASP13» . Белки: структура, функции и биоинформатика . 87 (12). Уайли: 1165–1178. bioRxiv 10.1101/552422 . дои : 10.1002/прот.25697 . ISSN 0887-3585 . ПМК 6800999 . ПМИД 30985027 .
- ^ Jump up to: а б «Прорыв DeepMind помогает понять, как болезни проникают в клетки» . Bloomberg.com . 2020-11-30 . Проверено 30 ноября 2020 г.
- ^ «глубокий разум/глубинное исследование» . Гитхаб . Проверено 30 ноября 2020 г.
- ^ Jump up to: а б с «AlphaFold: решение грандиозной проблемы биологии, возникшей 50 лет назад» . Глубокий разум . 30 ноября 2020 г. Проверено 30 ноября 2020 г. .
- ^ «ИИ компании DeepMind, сворачивающий белки, решил грандиозную проблему биологии, возникшую 50 лет назад» . Обзор технологий Массачусетского технологического института . Проверено 30 ноября 2020 г. .
- ^ Для используемой меры GDT_TS каждый атом в прогнозе получает четверть балла, если он находится в пределах 8 Å (0,80 нм) от экспериментального положения; половина точки, если она находится в пределах 4 Å, три четверти точки, если она находится в пределах 2 Å, и целая точка, если она находится в пределах 1 Å.
- ^ Чтобы получить оценку GDT_TS 92,5, математически не менее 70% структуры должны быть с точностью до 1 Å, а не менее 85% должны иметь точность с точностью до 2 Å,
- ^ Каллауэй, Юэн (13 декабря 2022 г.). «После AlphaFold: соревнование по сворачиванию белков стремится к следующему большому прорыву» . Природа . 613 (7942): 13–14. дои : 10.1038/d41586-022-04438-1 . ПМИД 36513827 . S2CID 254660427 .
- ^ Решение искусственного интеллекта для решения 50-летней научной проблемы может «революционизировать» медицинские исследования (пресс-релиз), CASP , 30 ноября 2020 г. Оргкомитет
- ^ Бриджит Нерлих, Сворачивание белка и научная коммуникация: между шумихой и смирением , Ноттингемского университета , 4 декабря 2020 г. блог
- ↑ Майкл Ле Пейдж, биолог DeepMind, занимающийся искусственным интеллектом, может разгадать секреты механизма жизни , New Scientist , 30 ноября 2020 г.
- ↑ Предсказания новейшего искусственного интеллекта DeepMind могут произвести революцию в медицине , New Scientist , 2 декабря 2020 г.
- ↑ Том Уиппл, Deepmind находит «Святой Грааль» биологии с ответом на проблему белка , The Times (онлайн), 30 ноября 2020 г.
Всего научный редактор Том Уиппл написал шесть статей на эту тему для «Таймс» в день появления новостей . ( нить ). - ↑ Кейд Мец , Лондонская лаборатория искусственного интеллекта заявляет о прорыве, который может ускорить открытие лекарств , New York Times , 30 ноября 2020 г.
- ^ Ян Сэмпл, ИИ DeepMind решает 50-летнюю проблему сворачивания белка , The Guardian , 30 ноября 2020 г.
- ^ Лиззи Робертс, «Раз в поколение вперед», как исследователи Google AI решают 50-летнюю биологическую задачу . Дейли Телеграф , 30 ноября 2020 г.
- ^ Тим Хаббард , Секрет жизни, часть 2: решение проблемы сворачивания белка. , medium.com , 30 ноября 2020 г.
- ^ например, Грег Боуман, Сворачивание белка и связанные с ним проблемы остаются нерешенными, несмотря на достижения AlphaFold , Folding@home , 8 декабря 2020 г. блог
- ^ Кнапп, Алекс. «Объявлены прорывные премии 2023 года: компания Deepmind Protein Folders получила 3 миллиона долларов» . Форбс . Проверено 9 мая 2024 г.
- ^ Сэмпл, Ян (21 сентября 2023 г.). «Команда программы искусственного интеллекта AlphaFold выиграла научную премию Ласкера» . Хранитель . ISSN 0261-3077 . Проверено 9 мая 2024 г.
- ^ Домингес, Нуньо (2 декабря 2020 г.). «Искусственный интеллект решает одну из важнейших проблем биологии» . Эль Паис (на испанском языке) . Проверено 12 мая 2024 г.
- ^ Бриггс, Дэвид (04 декабря 2020 г.). «Если Alphafold2 от Google действительно решил проблему сворачивания белков, им нужно продемонстрировать свою работу» . Скептик . Проверено 12 мая 2024 г.
- ^ Демис Хассабис , «Краткая информация о захватывающем прогрессе в #AlphaFold!» (твит), через Твиттер , 18 июня 2021 г.
- ^ AlphaFold3 - почему Nature опубликовала его без кода?
- ^ AlphaFold 3: Шаг в будущее прогнозирования структуры
- ^ «База данных структуры белков AlphaFold» . Alphafold.ebi.ac.uk . Проверено 29 июля 2021 г.
- ^ «База данных структуры белков AlphaFold» . Alphafold.ebi.ac.uk . Проверено 27 июля 2021 г.
- ^ «База данных структуры белков AlphaFold» . www.alphafold.ebi.ac.uk .
- ^ ИнтерПро (22 июля 2021 г.). «Предсказания альфафолдной структуры, доступные в Interpro» . протеиныwebteam.github.io . Проверено 29 июля 2021 г.
- ^ «Передача силы AlphaFold в руки всего мира» . Глубокий разум . 22 июля 2022 г.
- ^ Каллауэй, Юэн (28 июля 2022 г.). « Вся белковая вселенная»: ИИ предсказывает форму почти каждого известного белка» . Природа . 608 (7921): 15–16. Бибкод : 2022Natur.608...15C . дои : 10.1038/d41586-022-02083-2 . ПМИД 35902752 . S2CID 251159714 .
- ^ Jump up to: а б «Какие варианты использования не поддерживает AlphaFold?» . База данных структуры белков AlphaFold .
- ^ AlphaFold объявляет революцию, основанную на данных, в биологии и медицине , Джанет М. Торнтон , Роман А. Ласковски и Нира Боркакоти, Nature Medicine , том 12, страницы 1666–1669, 12 октября 2021 г.
- ^ Макбрайд, Джон М.; Полев, Константин; Абдирасулов, Амирбек; Рейнгарц, Владимир; Гжибовский, Бартош А.; Тлусти, Цви (20 ноября 2023 г.). «AlphaFold2 может предсказывать эффекты одиночной мутации» . Письма о физических отзывах . 131 (21): 218401. arXiv : 2204.06860 . Бибкод : 2023PhRvL.131u8401M . doi : 10.1103/PhysRevLett.131.218401 . ISSN 0031-9007 . ПМИД 38072605 .
- ^ «Последний прорыв DeepMind в области искусственного интеллекта может ускорить открытие новых лекарств» . Компания Фаст . ISSN 1085-9241 . Проверено 24 января 2023 г.
- ^ Багдонас, Гарольдас; Фогарти, Карл А.; Фадда, Элиза; Агирре, Джон (29 октября 2021 г.). «Дело в пользу постпрогнозированных модификаций в базе данных структуры белков AlphaFold» (PDF) . Структурная и молекулярная биология природы . 28 (11): 869–870. дои : 10.1038/s41594-021-00680-9 . ISSN 1545-9985 . ПМИД 34716446 . S2CID 240228913 .
- ^ Ан, Хён Джу; Фрелих, Джон В.; Лебрилла, Карлито Б (01 октября 2009 г.). «Определение сайтов гликозилирования и сайт-специфической гетерогенности в гликопротеинах» . Современное мнение в области химической биологии . Аналитические методы/механизмы. 13 (4): 421–426. дои : 10.1016/j.cbpa.2009.07.022 . ISSN 1367-5931 . ПМЦ 2749913 . ПМИД 19700364 .
- ^ Хекельман, Мартин Л.; де Врис, Ида; Йостен, Робби П.; Перракис, Анастасис (февраль 2023 г.). «AlphaFill: обогащение моделей AlphaFold лигандами и кофакторами » Природные методы 20 (2): 205–213. дои : 10.1038/s41592-022-01685-y . ПМЦ 9911346 . ПМИД 36424442 .
- ^ Домбровский-Туманский, Павел; Стасяк, Анджей (7 ноября 2023 г.). «Слепота AlphaFold к топологическим барьерам влияет на ее способность правильно предсказывать топологию белков» . Молекулы . 28 (22): 7462. doi : 10,3390/molecules28227462 . ПМЦ 10672856 . ПМИД 38005184 .
- ^ «ИИ может помочь ученым найти вакцину от Covid-19» . Проводной . ISSN 1059-1028 . Проверено 1 декабря 2020 г.
- ^ Jump up to: а б «Вычислительные прогнозы белковых структур, связанных с COVID-19» . Глубокий разум . 4 августа 2020 г. Проверено 1 декабря 2020 г.
- ^ «Как новый ИИ DeepMind, складывающий белки, уже помогает бороться с пандемией коронавируса» . Удача . Проверено 1 декабря 2020 г.
Дальнейшее чтение
[ редактировать ]- Карлос Оутейрал, CASP14: чего на самом деле удалось добиться с помощью AlphaFold 2 от Google DeepMind, и что это значит для сворачивания белков, биологии и биоинформатики , Oxford Protein Informatics Group. (3 декабря)
- Мохаммед Аль-Курайши, AlphaFold2 @ CASP14: «Такое ощущение, будто твой ребенок ушел из дома». (блог), 8 декабря 2020 г.
- Мохаммед Аль-Курайши, Документ по методу AlphaFold2: кладезь хороших идей (блог), 25 июля 2021 г.
Внешние ссылки
[ редактировать ]- Веб-сервер AlphaFold-3
- Код AlphaFold v2.1 и ссылки на модель на GitHub
- Открытый доступ к предсказаниям структуры белков протеома человека и 20 других ключевых организмов в Европейском институте биоинформатики (база данных структуры белков AlphaFold)
- CASP 14 Веб-сайт
- AlphaFold: Научный прорыв , DeepMind, через YouTube.
- ColabFold ( Мирдита, Милот; Шютце, Константин; Мориваки, Ёситака; Привет, Лим; Овчинников Сергей; Штайнеггер, Мартин (30 мая 2022 г.). «ColabFold: Делаем сворачивание белка доступным для всех» . Природные методы . 19 (6): 679–682. дои : 10.1038/s41592-022-01488-1 . ПМЦ 9184281 . ПМИД 35637307 . ), версия для предсказания гомоолигомеров и комплексов
