Циркулирующая опухоль ДНК
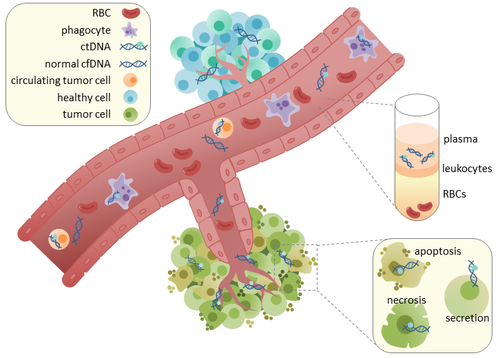
Циркулирующая опухолевая ДНК (ctDNA) представляет собой , полученную опухоли, фрагментированную дн -ДНК в кровотоке, которая не связана с клетками. CTDNA не следует путать с бесклеточной ДНК ( CFDNA ), более широким термином, который описывает ДНК, которая свободно циркулирует в кровотоке, но не обязательно имеет происхождение опухоли. Поскольку ctDNA может отражать весь геном опухоли , он приобрел тягу к своей потенциальной клинической полезности; « Жидкие биопсии » в виде привлечения крови могут быть взяты в различные моменты времени для мониторинга прогрессирования опухоли на протяжении всей режима лечения. [ 1 ] [ 2 ]
Недавние исследования заложили основу для вывода экспрессии генов из cfDNA (и ctDNA), а EPIC-Seq стала заметным достижением. [ 3 ] Этот метод существенно поднял планку для неинвазивного вывода уровней экспрессии отдельных генов, тем самым увеличивая применимость анализа в характеристике заболевания, гистологической классификации и мониторинга эффективности лечения. [ 3 ] [ 4 ] [ 5 ]
CTDNA происходит непосредственно из опухоли или из циркулирующих опухолевых клеток (CTCS), [ 6 ] который описывает жизнеспособные, неповрежденные опухолевые клетки, которые теряются от первичных опухолей и попадают в кровоток или лимфатическую систему. Точный механизм высвобождения ctDNA неясен. Биологические процессы, поступившие участие в высвобождении ctDNA, включают апоптоз и некроз из умирающих клеток или активное высвобождение из жизнеспособных опухолевых клеток. [ 7 ] [ 8 ] [ 9 ] [ 10 ] [ 11 ] Исследования как у людей (здоровые и раковые пациенты) [ 12 ] и ксенокрасные мыши [ 13 ] Покажите, что размер фрагментированной cfDNA преобладает длиной 166 п.н., что соответствует длине ДНК, обернутой вокруг нуклеосомы, плюс линкер. Фрагментация этой длины может указывать на апоптотическую фрагментацию ДНК , что позволяет предположить, что апоптоз может быть основным методом высвобождения ctDNA. Фрагментация cfDNA изменяется в плазме раковых пациентов. [ 14 ] [ 15 ] В здоровых тканях инфильтрирующие фагоциты ответственны за клиренс апоптотического или некротического клеточного мусора, который включает в себя cfDNA. [ 16 ] CTDNA у здоровых пациентов присутствует только на низких уровнях, но более высокие уровни CTDNA у пациентов с раком могут быть обнаружены с увеличением размеров опухоли. [ 17 ] Это, возможно, происходит из -за неэффективной инфильтрации иммунных клеток к сайтам опухоли, что снижает эффективное клиренс ctDNA из кровотока. [ 16 ] Сравнение мутаций в CTDNA и ДНК, экстрагированных из первичных опухолей тех же пациентов, выявило наличие идентичных генетических изменений, связанных с раком,. [ 18 ] [ 19 ] Это привело к возможности использования CTDNA для более раннего обнаружения рака и лечения. [ 20 ]
Методы
[ редактировать ]Преаналитические соображения
[ редактировать ]Когда кровь собирается в пробирках ЭДТА и сохраняется, лейкоциты начинают лизировать и высвобождать геномную ДНК дикого типа в образце в количествах, как правило, складывается выше, чем в ctDNA. [ 21 ] Это усложняет обнаружение мутаций или других биомаркеров CTDNA. [ 22 ] Использование коммерчески доступных клеточных стабилизационных труб может предотвратить или задержать лизис белых клеток, тем самым уменьшая эффект разбавления CTDNA. [ 23 ] Sherwood et al. продемонстрировал превосходное обнаружение мутаций KRAS в подходящих образцах, собранных как в EDTA K3, так и в трубках BCT Streck. [ 23 ] Преимущества трубок стабилизации клеток могут быть реализованы в ситуации, когда кровь не может быть обработана в плазму немедленно.
Другие процедуры могут также уменьшить количество «загрязняющей» ДНК дикого типа и сделать обнаружение ctDNA более осуществимым: [ 23 ]
- Никогда не заморозить образец крови перед извлечением плазмы для анализа CTDNA
- Обработайте образец в плазму в течение 2–4 часов (если собрано в пробирке ЭДТА)
- Никогда не используйте гепаринизированные трубки, гепарин ингибирует ПЦР, имитируя спиральную структуру ДНК
- Выполните двойную центрифугирование (центрифуга кровь для извлечения плазмы, затем повторите на плазме, чтобы удалить из мусора в нижней части трубки), чтобы удалить более клеточный мусор перед экстракцией ДНК.
- Плазма лучше, чем сыворотка для восстановления ctdna [ 24 ]
Экстракция ctDNA
[ редактировать ]Основная привлекательность анализа CTDNA заключается в том, что он извлекается неинвазивным образом с помощью сбора крови. Приобретение CFDNA или CTDNA обычно требует сбора приблизительно 3 мл крови в пробирки с EDTA . Использование EDTA важно для снижения коагуляции крови. Фракции плазмы и сыворотки крови могут быть разделены на стадию центрифугирования. КтДНК или cfDNA могут быть впоследствии извлечены из этих фракций. Хотя сыворотка имеет тенденцию иметь более высокие уровни cfDNA, это в первую очередь объясняется ДНК из лимфоцитов. [ 25 ] Высокие уровни загрязняющих cfDNA являются неоптимальными, поскольку это может снизить чувствительность обнаружения CTDNA. Следовательно, большинство исследований используют плазму для выделения ctDNA. Затем плазма снова обрабатывается центрифугированием для удаления остаточных интактных клеток крови. Супернатант используется для экстракции ДНК, которая может быть выполнена с использованием коммерчески доступных наборов. [ Цитация необходима ]
Анализ CTDNA
[ редактировать ]Анализ CTDNA после экстракции требует использования различных методов амплификации и секвенирования. Эти методы могут быть разделены на две основные группы на основе того, является ли цель, чтобы опросить все гены в нецелевом подходе, или если цель состоит в том, чтобы контролировать определенные гены и мутации в целевом подходе. [ Цитация необходима ]
Неточенные подходы
[ редактировать ]Для обнаружения новых мутаций в опухолевой ДНК при мониторинге бремени заболевания или отслеживания лекарственной устойчивости могут быть необходимы целый геном или целое экзом подходы секвенирования. [ 26 ] Непредсказуемые подходы также полезны в исследованиях для наблюдения за гетерогенностью опухоли или для обнаружения новых лекарственных целей. Тем не менее, хотя нецелевые методы могут быть необходимы в определенных приложениях, они дороже и имеют более низкое разрешение. Это затрудняет обнаружение редких мутаций или в ситуациях, когда присутствуют низкие уровни ctDNA (такие как минимальное остаточное заболевание). Кроме того, могут возникнуть проблемы между ДНК от опухолевых клеток и ДНК от нормальных клеток с использованием целого генома. [ Цитация необходима ]
Секвенирование всего генома или экзома обычно использует технологии секвенирования высокой пропускной способности . Ограничение секвенирования только весь экзом вместо этого может снизить расходы и увеличить скорость, но за счет потери информации о мутациях в некодирующих регуляторных областях ДНК. [ 27 ] При простом взгляде на полиморфизмы ДНК посредством секвенирования не дифференцирует ДНК от опухоли или нормальных клеток, эта проблема может быть решена путем сравнения с контрольным образцом нормальной ДНК (например, ДНК, полученной через буйный мазынет .) Важно, целый геном и целый Секвенирование экзома полезно для начального обнаружения мутации. Это предоставляет информацию для использования более чувствительных целевых методов, которые затем можно использовать для целей мониторинга заболеваний.
Секвенирование целого генома позволяет восстановить структурные свойства cfDNA, размер фрагментов и их фрагментацию. Эти уникальные закономерности могут быть важным источником информации для улучшения обнаружения ctDNA или локализации ткани происхождения этих фрагментов. [ 28 ] Выбор размеров коротких фрагментов (<150BP) с помощью in vitro или в силико-методах может улучшить восстановление мутаций и аберрации числа копий. [ 15 ]
Цифровое кариотипирование
[ редактировать ]Этот метод был первоначально разработан лабораторией Берта Фогельштейна , Луиса Диаса и Виктора Велкулеску в Университете Джона Хопкинса . [ 29 ] В отличие от нормального кариотипирования , где краситель используется для окрашивания хромосомных полос для визуализации хромосом, цифровое кариотипирование использует последовательности ДНК локусов по всему геному для расчета изменения числа копий . [ 29 ] Изменения числа копий распространены при раке и описывают ситуации, когда потеря гетерозиготности гена может привести к снижению функции из -за более низкой экспрессии или дублирования гена, что приводит к сверхэкспрессии.
Персонализированный анализ перестроенных концов
[ редактировать ]После того, как весь геном секвенировался с использованием метода секвенирования высокой пропускной способности, такого как Illumina hiseq, индивидуальный анализ перестроенных концов (PARE) применяется к данным для анализа хромосомных перестройств и транслокаций. Этот метод был первоначально разработан для анализа твердой опухольной ДНК, но была модифицирована для применений CTDNA. [ 29 ]
Метилирование ДНК и гидроксиметилирование
[ редактировать ]Правильная эпигенетическая маркировка необходима для нормальной экспрессии генов, а функция клеток, а аберрантные изменения в эпигенетических паттернах являются отличительной чертой рака. [ 30 ] Нормальный эпигенетический статус поддерживается в клетке, по крайней мере, частично посредством метилирования ДНК . [ 31 ] Измерение паттернов аберрантного метилирования в ctDNA возможно из -за стабильного метилирования областей ДНК, называемых « островами CPG ». Метилирование ctDNA может быть обнаружено посредством лечения бисульфитом . Бисульфитная обработка химически превращает неметилированные цитозины в урацил, оставляя метилированные цитозины немодифицированными. ДНК впоследствии секвенирована, и любые изменения в схеме метилирования ДНК могут быть идентифицированы. Гидроксиметилирование ДНК является аналогичной ассоциированной меткой, которая, как было показано, является прогностическим маркером здоровых и больных состояний в cfDNA, включая рак. [ 32 ] [ 33 ] )
Целевые подходы
[ редактировать ]В целевом подходе секвенирование ctDNA может быть направлено на генетическую панель, построенную на основе мутационных горячих точек для интересующего рака. Это особенно важно для информирования лечения в ситуациях, когда мутации выявляются в лекарствах. [ 27 ] Персонализация целенаправленного анализа CTDNA для каждого пациента также возможна путем объединения биопсии жидкости со стандартной первичной биопсией тканей. Секвенирование целого генома или целого экзома первичной биопсии опухоли позволяет открывать генетические мутации, специфичные для опухоли пациента, и может использоваться для последующего целевого секвенирования ctDNA пациента. Самая высокая чувствительность обнаружения ctDNA достигается посредством целевого секвенирования специфических одноклеотидных полиморфизмов (SNP). Обычно мутированные гены, такие как онкогены, которые обычно имеют мутации горячей точки, являются хорошими кандидатами для подходов к целевым секвенированию. И наоборот, большинство генов -супрессоров опухолей имеют широкий спектр возможной потери мутаций функции по всему гену, и, как таковые, не подходят для целевого секвенирования. [ Цитация необходима ]
Целевые подходы имеют преимущество амплификации ctDNA с помощью полимеразных цепных реакций (ПЦР) или цифровой ПЦР . Это особенно важно при анализе CTDNA не только потому, что в кровотоке существуют относительно низкие уровни ДНК, но также потому, что ctDNA составляет небольшую долю общей бесклеточной ДНК, извлеченной. [ 27 ] Следовательно, амплификация интересующих областей может резко повысить чувствительность обнаружения CTDNA. Тем не менее, амплификация через ПЦР может ввести ошибки, учитывая неотъемлемая частота ошибок ДНК -полимеразы. Ошибки, введенные во время секвенирования, также могут снизить чувствительность обнаружения мутаций ctDNA. [ Цитация необходима ]
Цифровая полимеразная цепная реакция капель
[ редактировать ]Цифровая ПЦР Droplet (DDPCR) получена из цифровой полимеразной цепной реакции , первоначально названной группой Берта Фогельштейна в Университете Джона Хопкинса . Цифровая ПЦР Droplet использует генератор капель для разделения отдельных кусочков ДНК на капли с использованием масляной/водной эмульсии. Затем в каждой капельке возникают отдельные полимеразные цепные реакции, используя выбранные праймеры против областей ctDNA и переходят к конечной точке. Присутствие интересующих последовательностей измеряется флуоресцентными зондами, которые связываются с амплифицированной областью. DDPCR допускает высоко количественную оценку частот аллеля и мутантов в CTDNA, но ограничена количеством флуоресцентных зондов, которые можно использовать в одном анализе (до 5). [ 34 ] Чувствительность анализа может варьироваться в зависимости от количества проанализированной ДНК и составляет около 1 на 10 000. [ 34 ] Специфичность должна быть увеличена за счет использования либо незначительных модифицированных зондов, измененных (MGB), либо альтернативы, такой как блокированные нуклеиновые кислоты (LNA).
Бусы, эмульгирование, усиление и магнетика
[ редактировать ]Бусины, эмульгирование, амплификация и магнетики (пучок) представляют собой метод, который строится на капельной цифровой ПЦР, чтобы идентифицировать мутации в ctDNA с использованием проточной цитометрии. [ 35 ] После того, как CTDNA экстрагируется из крови, ПЦР выполняется с праймерами, предназначенными для нацеливания на интересующие области. Эти праймеры также содержат специфические последовательности ДНК или теги. Амплифицированная ДНК смешивается с покрытыми стрептавидином магнитных шариков и эмульгируется в капли. Биотинилированные праймеры, предназначенные для связывания с метками, используются для амплификации ДНК. Биотинилирование позволяет амплифицированной ДНК связываться с магнитными шариками, которые покрыты стрептавидином. После того, как ПЦР завершена, гранулы из ДНК разделяются с использованием магнита. ДНК на шариках затем денатурируется и позволяет гибридизоваться с флуоресцентными олигонуклеотидами, специфичными для каждого матрица ДНК. Полученные комплексы боссовой ДНК затем анализируются с использованием проточной цитометрии. Этот метод способен захватывать частоты аллеля и мутаций из -за сочетания с DDPCR. Однако, в отличие от DDPCR, большее количество последовательностей ДНК может быть опробовано из -за гибкости использования флуорестно связанных зондов. Другое преимущество этой системы заключается в том, что изолированная ДНК также может использоваться для последующего секвенирования. [ 36 ] Чувствительность составляет 1,6 в 10 4 до 4.3 в 10 5 . [ 34 ]
Персонализированное профилирование рака путем глубокого секвенирования
[ редактировать ]Персонализированное профилирование рака с помощью глубокого секвенирования (CAPP-Seq) было первоначально описано группами Ash Alizadeh и Maximilian Diehn в Стэнфордском университете . В этом методе используются биотинилированные олигонуклеотидные зонды для целевых последовательностей ДНК, относящихся к обнаружению CTDNA. [ 37 ] Общедоступные базы данных рака были использованы для построения библиотеки зондов против рецидивирующих мутаций при раке путем расчета их индекса рецидивов. Протокол был оптимизирован для низких уровней ДНК, наблюдаемых в сборе ctDNA. Затем изолированная ДНК подвергается глубокому секвенированию для повышения чувствительности. Этот метод допускает опрос сотен областей ДНК. Сообщается, что чувствительность обнаружения CTDNA CAPP-Seq составляет 2,5 молекулы в 1 000 000. [ 38 ]
Tagged Amplicon Deep Sequencing
[ редактировать ]Глубокое секвенирование Amplicon Tagged (TAM-Seq) позволяет целевое секвенирование целых генов для обнаружения мутаций в ctDNA. [ 39 ] Сначала общий этап усиления выполняется с использованием праймеров, которые охватывают весь интерес, представляющий интерес в 150-200BP. Затем для прикрепленных адаптеров с уникальным идентификатором к каждому ампликону используется микрофлюидическая система для дальнейшего усиления ДНК в параллельных реакциях с одним шагом. Было показано, что этот метод успешно идентифицирует мутации, разбросанные в ген -супрессоре опухоли TP53 у пациентов с раком прогрессирующих яичников. Чувствительность этой техники составляет 1 из 50.
Безопасное секвенирование
[ редактировать ]Безопасное секвенирование (Safe-seq) было первоначально описано Бертом Фогельштейном и его группой в Университете Джона Хопкинса . Safe-seq уменьшает частоту ошибок массового параллельного секвенирования, чтобы повысить чувствительность к редким мутантам. [ 40 ] Это достигает этого путем добавления уникальной последовательности идентификатора (UID) к каждому шаблону ДНК. Затем ДНК амплифицируется с использованием добавленных UID и секвенирования. Все молекулы ДНК с одним и тем же UID (семейство UID) должны иметь одинаковую последовательность ДНК, поскольку они были амплифицированы из одной молекулы. Тем не менее, мутации могут быть введены посредством усиления, или неверные базовые назначения могут быть вызваны на этапах секвенирования и анализа. Наличие UID позволяет отделить эти методологические ошибки от истинных мутаций ctDNA. Мутация считается «супермутантом», если 95% секвенированных чтений согласуются. Чувствительность этого подхода составляет 9 на 1 миллион. [ 34 ]
Дуплексное секвенирование
[ редактировать ]Дуплексное секвенирование -это улучшение на единых UID, добавленных в технике Safe-seq. [ 41 ] В дуплексном секвенировании рандомизированная двухцепочечная ДНК действует как уникальные теги и прикрепляется к инвариантной проставке. Теги прикрепляются к обоим концам фрагмента ДНК (α и β -метки), что приводит к двум уникальным шаблонам для ПЦР - одна цепь с α -меткой на 5 'конце и β на 3' конец, а другая цепь с β -меткой на 5 -дюймовом конце и α -меткой на 3 'конец. Эти фрагменты ДНК затем усиливаются праймерами против инвариантных последовательностей тегов. Амплифицированная ДНК секвенирована и анализируется. ДНК с дуплексными адаптерами сравниваются, а мутации принимаются только в том случае, если существует консенсус между обеими нитями. Этот метод учитывает как ошибки из -за секвенирования, так и ошибок в результате амплификации ПЦР на ранней стадии. Чувствительность подхода к обнаружению мутантов составляет 1 из 10^7.
Интегрированная цифровая пострадавшая ошибка, усиленная CAPP-seq
[ редактировать ]Интегрированное подавление цифровых ошибок (IDES) улучшает анализ CAPP-Seq CTDNA, чтобы уменьшить ошибку и, следовательно, повысить чувствительность обнаружения. [ 38 ] Сообщается в 2016 году, IDES объединяет CAPP-Seq с технологией секвенирования дуплексного штрих-кодирования и с помощью вычислительного алгоритма, который устраняет стереотипные ошибки, связанные со стадией гибридизации CAPP-Seq. Метод также интегрирует дуплексное секвенирование, где это возможно, и включает в себя методы для более эффективного дуплексного восстановления от свободной ДНК. Чувствительность этой улучшенной версии CAPP-Seq составляет 4 из 100 000 экземпляров.
Секвенирование всего генома
[ редактировать ]Исследования секвенирования всего генома были проведены на CTDNA, присутствующей у разных пациентов с резистентным к лечению рака предстательной железы (подавляющее большинство, а в некоторых случаях, метастатические ), рак мочевого пузыря и контрольные пациенты, которые не представляли эту ДНК, включая соматические мутации и структурные Перестановки в их геномах.
Эта новая и многообещающая методика предоставила информацию о резистентности к лечению рецепторов андрогенов ингибиторами передачи сигналов , внутриопухолевой гетерогенности (благодаря филогенетической эволюции и молекулярной хронологии), хромосомной нестабильности , вклад CTDNA в метастазирование с помощью глобальных транскриптомных схем (с учетом с учетом ядерных ядер. Начальные сайты (TSSS) и AR-связывающие сайты (ARBS). Различия в фенотипе рака и, чтобы увидеть, как терапия влияет на пациентов.
Это делает CTDNA мощным новым инструментом для обнаружения генетических мутаций в геномной шкале у пациентов, страдающих метастатическим раком, чтобы наблюдать за клинической значимостью клонального состава этих опухолей для понимания лучшего контроля рака. Эта субклональная реконструкция, основанная на CTDNA, благодаря секвенированию всего генома создает уникальный набор проблем и возможностей для научных исследований в онкологии. Кроме того, серийная ctDNA выявляет отбор для лечения для Увеличение андрогенового рецептора, потому что он увеличивает размерность данных.
Необходима дальнейшая работа, чтобы понять, как метастатическое местоположение и размер в отношении нагрузки на опухоль влияют на циркулирующую опухолевую ДНК и выбор новых методов для выбора других поражений, которые отражают клинически доминирующее заболевание. [ 42 ]
Соображения
[ редактировать ]«Нормальный» против обнаружения ДНК опухоли
[ редактировать ]Одной из проблем при использовании ctDNA в качестве биомаркера рака является то, можно ли отличить кфДНК с CFDNA от нормальных клеток. CFDNA высвобождается незлокачественными клетками во время нормального клеточного оборота, но также во время таких процедур, как хирургия , лучевая терапия или химиотерапия . Считается, что лейкоциты являются основными участниками CFDNA в сыворотке. [ 27 ]
Исследовать
[ редактировать ]ctdna в скрининге рака
[ редактировать ]Клиническая полезность CTDNA для обнаружения первичных заболеваний частично ограничена чувствительностью современной технологии для обнаружения небольших опухолей с низкими уровнями присутствующей ctDNA и априорными неизвестными соматическими мутациями. [ 17 ] [ 34 ]
ctdna в мониторинге рака
[ редактировать ]Свидетельство заболевания традиционными методами визуализации, таких как КТ , ПЭТ или МРТ, могут отсутствовать после резекции опухоли. Следовательно, анализ CTDNA создает потенциальный путь для выявления минимального остаточного заболевания (MRD) и, следовательно, вероятности рецидива опухоли, в тех случаях, когда объемные опухоли отсутствуют обычными методами визуализации. [ 17 ] Сравнение обнаружения MRD с помощью КТ -визуализации по сравнению с CTDNA было ранее проводилось у людей с раком толстой кишки II стадии; В этом исследовании исследователи смогли обнаружить ctDNA у людей, которые не показали признаков клинического злокачественного новообразования с помощью компьютерной томографии, что позволяет предположить, что обнаружение CTDNA обладает большей чувствительностью к оценке MRD. [ 27 ] Однако авторы признают, что анализ CTDNA не без ограничений; Собранные образцы плазмы после операции были способны предсказать рецидив только через 36 месяцев в 48% случаев. [ 27 ] Впоследствии были разработаны анализы CTDNA для обоих колоректального рака [ 43 ] и меланома . [ 44 ]
ctdna как прогностический биомаркер
[ редактировать ]Вопрос о том, можно ли использовать измерение количества или качеств CTDNA для определения результатов у людей с раком, было предметом исследования. По состоянию на 2015 год это было очень неуверенно. [ 45 ] Хотя некоторые исследования показали тенденцию к более высоким уровням CTDNA у людей с высокой стадией метастатического рака, бремя ctDNA не всегда коррелирует с традиционной постановкой рака. [ 34 ] По состоянию на 2013 год маловероятно, что ctdna будет иметь клиническую полезность в качестве единственного предиктора прогноза. [ 46 ]
РАНКА
[ редактировать ]Появление устойчивых к лекарственным средствам опухолей из-за внутри- и межопухолевой гетерогенности проблема в эффективности лечения. Незначительный генетический клон в опухоли может расширяться после лечения, если он несет мутацию, устойчивую к лекарствам. Начальные биопсии могут пропустить эти клоны из -за низкой частоты или пространственного разделения клеток в опухоли. Например, поскольку биопсия только пробует небольшую часть опухоли, клоны, которые находятся в другом месте, могут остаться незамеченными. Это может ввести в заблуждение исследования, посвященные изучению роли гетерогенности опухоли в прогрессировании и рецидиве рака. Использование CTDNA в исследованиях может облегчить эти проблемы, поскольку оно может обеспечить более репрезентативный «скриншот» генетического разнообразия рака как в первичных, так и в метастатических местах. Например, было показано, что CTDNA полезна при изучении клональной эволюции рака пациента до и после схем лечения. [ 47 ] Раннее обнаружение рака все еще является сложным, но недавний прогресс в анализе эпигенетических признаков CFDNA или разблокировки фрагментации улучшает чувствительность биопсии жидкости. [ 28 ] Кроме того, анализ CTDNA является новым инструментом для понимания клональной составы метастатических опухолей, обнаружения различных мутаций в геномной масштабе, изучая разнообразие субклонального, которое влияет на прогноз заболевания как различные резистентные фенотипы и появление новых механизмов геномического и транскриптомная устойчивость к лечению. [ 42 ]
Проблемы для реализации
[ редактировать ]Внедрение CTDNA в клинической практике в значительной степени препятствует отсутствию стандартизированных методов обработки и анализа CTDNA. Стандартизация методов сбора образцов (включая время сбора), нижестоящую обработку (экстракция и амплификация ДНК), количественная оценка и валидация должны быть установлены до того, как анализ CTDNA может стать обычным клиническим анализом. Кроме того, может потребоваться создание панели «стандартных» биомаркеров, ассоциированных с опухолью, с учетом разрешения текущих методов секвенирования и обнаружения CtDNA. Секвенирование опухоли-специфических аберраций из образцов плазмы также может помочь исключить загрязнение CFDNA из анализа; Повышенные уровни CFDNA из нормальных клеток могут быть отнесены к причинам, не связанным с раковинами. [ 27 ] Эти методы секвенирования также могут определять клональную эволюцию рака, гетерогенности опухоли и механизмов лекарственной устойчивости, участвующих в раке. [ 42 ]
Смотрите также
[ редактировать ]Ссылки
[ редактировать ]- ^ Wan J, Massie C, Garcia-Corbacho J, Mouliere F, Brenton J, Caldas C, PACESY S, Baird R, Rosenfeld N (апрель 2017 г.). «Жидкая биопсия достигает совершеннолетия: к реализации циркулирующей опухолевой ДНК» . Природа рецензирует рак . 17 (4): 223–238. doi : 10.1038/nrc.2017.7 . PMID 28233803 . S2CID 4561229 .
- ^ Нонака, т; Вонг, DTW (13 июня 2022 года). «Диагностика слюны» . Ежегодный обзор аналитической химии . 15 (1): 107–121. Bibcode : 2022Arac ... 15..107n . doi : 10.1146/annurev-anchem-061020123959 . PMC 9348814 . PMID 35696523 .
- ^ Jump up to: а беременный Эсфахани, Мохаммад Шахрох; Гамильтон, Эмили Дж.; Мехрмохамади, Маха; и др. (Апрель 2022 г.). «Вывод экспрессии генов из профилей фрагментации ДНК без клеток» . Nature Biotechnology . 40 (4): 585–597. doi : 10.1038/s41587-022-01222-4 . PMC 9337986 . PMID 35361996 .
- ^ Бормотать, Юрик А; Шахрох Эсфахани, Мохаммед; Шроерс-Мартин, Джозеф; и др. (28 ноября 2023 г.). «Предполагаемая экспрессия генов с помощью бесклеточного профилирования ДНК обеспечивает неинвазивную классификацию лимфомы» . Кровь . 142 (Дополнение 1): 245–245. doi : 10.1182/blood-201023-186853 .
- ^ Алиг, Стефан К.; Шахрох Эсфахани, Мохаммед; Гарофало, Андреа; и др. (25 января 2024 г.). «Отчетливые подтипы лимфомы Ходжкина, определяемые неинвазивным геномным профилированием» . Природа . 625 (7996): 778–787. doi : 10.1038/s41586-023-06903-x . PMC 11293530 . PMID 38081297 .
- ^ Акка Х., Демирай А., Ярен А., Бир Ф., Коселер А., Ивакава Р., Багси Г., Йокота Дж (март 2013 г.). «Полезность сывороточной ДНК и пиросеквенирования для обнаружения мутаций EGFR при немелкоклеточном раке легких». Рак генетика . 206 (3): 73–80. doi : 10.1016/j.cancergen.2013.01.005 . PMID 23491080 .
- ^ Schwarzenbach H, Hoon DS, Pantel K (июнь 2011 г.). «Бесклеточные нуклеиновые кислоты как биомаркеры у больных раком». Природные обзоры. Рак . 11 (6): 426–37. doi : 10.1038/nrc3066 . PMID 21562580 . S2CID 6061607 .
- ^ Стрюн М., Анкер П (июль 1972 г.). «Нуклеиновые кислоты спонтанно высвобождаются живыми ушными ракушками лягушки» . Биохимический журнал . 128 (3): 100p - 101p. doi : 10.1042/bj1280100pb . PMC 1173871 . PMID 4634816 .
- ^ Стрюн М., Льятеи Дж., Ледерри С., Олсон-Санд А., Анкер П (ноябрь 2001 г.). «О возможном происхождении и механизме циркулирующего апоптоза ДНК и активного высвобождения ДНК». Clinica Chimica Acta; Международный журнал клинической химии . 313 (1–2): 139–42. doi : 10.1016/s0009-8981 (01) 00665-9 . PMID 11694251 .
- ^ Anker P, Stroun M, Maurice PA (сентябрь 1975 г.). «Спонтанное высвобождение ДНК лимфоцитами крови человека, как показано в системе in vitro» . РАНКА . 35 (9): 2375–82. PMID 1149042 .
- ^ Rogers JC, Boldt D, Kornfeld S, Skinner A, Valeri CR (июль 1972 г.). «Исключение дезоксирибонуклеиновой кислоты лимфоцитами стимулировано фитогемагглютинином или антигеном» . Труды Национальной академии наук Соединенных Штатов Америки . 69 (7): 1685–9. Bibcode : 1972pnas ... 69.1685r . doi : 10.1073/pnas.69.7.1685 . PMC 426778 . PMID 4505646 .
- ^ Heitzer E, Auer M, Hoffmann EM, Pichler M, Gasch C, Ulz P, Lax S, Waldispuehl-Geigl J, Mauermann O, Mohan S, Pristauz G, Laxner C, Höfler G, Eisner F, Petru E, Sill H, Самонигг Х., Пантель К., Ритдорф С., Бауернхофер Т., Гейгл Дж.Б., Спейхер М.Р. (июль 2013 г.). «Создание опухоли-специфических изменений числа копий от плазменной ДНК пациентов с раком» . Международный журнал рака . 133 (2): 346–56. doi : 10.1002/ijc.28030 . PMC 3708119 . PMID 23319339 .
- ^ Thierry AR, Mouliere F, Gongora C, Ollier J, Robert B, Ychou M, Del Rio M, Molina F (октябрь 2010 г.). «Происхождение и количественная оценка циркулирующей ДНК у мышей с ксенотрансплантатами о колоректальном раке человека» . Исследование нуклеиновых кислот . 38 (18): 6159–75. doi : 10.1093/nar/gkq421 . PMC 2952865 . PMID 20494973 .
- ^ Mouliere F, Robert B, Arnau Peyrotte E, Del Rio M, Ychou M, Molina F, Gongora C, Thierry AR (2011). «Высокая фрагментация характеризует циркулирующую ДНК, полученную из опухоля» . Plos один . 6 (9): E23418. Bibcode : 2011ploso ... 623418m . doi : 10.1371/journal.pone.0023418 . PMC 3167805 . PMID 21909401 .
- ^ Jump up to: а беременный Mouliere F, Chandrananda D, Piskorz AM, Moore EK, Morris J, Ahlborn LB, et al. (Ноябрь 2018). «Усиленное обнаружение циркулирующей опухолевой ДНК с помощью анализа размеров фрагмента» . Sci Transl Med . 10 (466). doi : 10.1126/scitranslmed.aat4921 . PMC 6483061 . PMID 30404863 .
- ^ Jump up to: а беременный Pisetsky DS, Fairhurst AM (июнь 2007 г.). «Происхождение внеклеточной ДНК во время очистки мертвых и умирающих клеток». Аутоиммунитет . 40 (4): 281–4. doi : 10.1080/08916930701358826 . PMID 17516210 . S2CID 11499768 .
- ^ Jump up to: а беременный в Avanzini S, Kurtz DM, Chabon JJ, Moding EJ, Hori SS, Gambhir SS, Alizadeh AA, Diehn M, Reiter JG (декабрь 2020 г.). «Математическая модель сливания ctDNA предсказывает размер обнаружения опухоли» . Наука достижения . 6 (50): EABC4308. Бибкод : 2020Scia .... 6.4308a . doi : 10.1126/sciadv.abc4308 . PMC 7732186 . PMID 33310847 . S2CID 228096858 .
- ^ Васиухин В., Анкер П., Морис П., Ляутеи Дж., Ледерри С., Страун М. (апрель 1994 г.). «Точечные мутации гена N-Ras в ДНК плазмы крови пациентов с миелодиспластическим синдромом или острым миелогенным лейкозом». Британский журнал гематологии . 86 (4): 774–779. doi : 10.1111/j.1365-2141.1994.tb04828.x . PMID 7918071 . S2CID 26365875 .
- ^ Васиухин В., Страун М., Морис П., Ляутеи Дж., Ледерри С., Анкер П (май 1994). Мутации точечной точки K-RAS в ДНК плазмы крови пациентов с колоректальными опухолями в проблемах современной медицины . Тол. 5. С. 141–150.
- ^ Yong E (июль 2014 г.). «Биомаркеры рака: написано в крови» . Природа . 511 (7511): 524–526. Bibcode : 2014natur.511..524y . doi : 10.1038/511524a . PMID 25079538 . S2CID 4445938 .
- ^ Xue X, Tepe MD, Holen I, Zhu YM, Woll PJ (июнь 2009 г.). «Оптимизация урожайности и полезности циркулирующей бесклеточной ДНК из плазмы и сыворотки» (PDF) . Clinica Chimica Acta; Международный журнал клинической химии . 404 (2): 100–4. doi : 10.1016/j.cca.2009.02.018 . PMID 19281804 .
- ^ Norton SE, Lechner JM, Williams T, Fernando MR (октябрь 2013 г.). «Стабилизирующий реагент предотвращает бесклеточное загрязнение ДНК клеточной ДНК в плазме во время хранения образцов крови и доставки, как определено цифровой ПЦР» . Клиническая биохимия . 46 (15): 1561–5. doi : 10.1016/j.clinbiochem.2013.06.002 . PMID 23769817 .
- ^ Jump up to: а беременный в Шервуд Дж.Л., Коркоран С., Браун Х, Шарп А.Д., Мусилов М., Колманн А (2016). «Оптимизированные преаналитические методы улучшают обнаружение мутаций KRAS в ДНК циркулирующей опухоли (CTDNA) от пациентов с немелкоклеточным раком легких (NSCLC)» . Plos один . 11 (2): E0150197. BIBCODE : 2016PLOSO..1150197S . doi : 10.1371/journal.pone.0150197 . PMC 4769175 . PMID 26918901 .
- ^ Vallée A, Marcq M, Bizieux A, Kouri CE, Lacroix H, Bennouna J, Douillard Jy, Denis Mg (ноябрь 2013). «Плазма является лучшим источником циркулирующей бесклеточной ДНК, полученной из опухоли, чем сыворотка для обнаружения изменений EGFR у пациентов с опухолью легких». Рак легких . 82 (2): 373–4. doi : 10.1016/j.lungcan.2013.08.014 . PMID 24007628 .
- ^ Lee TH, Montalvo L, Chrebtow V, Busch MP (февраль 2001 г.). «Количественное определение геномной ДНК в образцах плазмы и сыворотки: более высокие концентрации геномной ДНК, обнаруженные в сыворотке, чем в плазме». Переливание . 41 (2): 276–82. doi : 10.1046/j.1537-2995.2001.4102020276.x . PMID 11239235 . S2CID 45714834 .
- ^ Qin Z, Ljubimov VA, Zhou C, Tong Y, Liang J (апрель 2016 г.). «Бесклеточная циркулирующая опухолевая ДНК при раке» . Китайский журнал рака . 35 : 36. DOI : 10.1186/S40880-016-0092-4 . PMC 4823888 . PMID 27056366 .
- ^ Jump up to: а беременный в дюймовый и фон глин Heitzer E, Ulz P, Geigl JB (январь 2015 г.). «Циркулирующая опухолевая ДНК как жидкая биопсия для рака» . Клиническая химия . 61 (1): 112–23. doi : 10.1373/clinchem.2014.222679 . PMID 25388429 .
- ^ Jump up to: а беременный van der Pol Y, Mouliere F (октябрь 2019 г.). «На пути к раннему обнаружению рака декодировал эпигенетические и экологические отпечатки пальцев бесклеточной ДНК» . Раковая клетка . 36 (4): 350–368. doi : 10.1016/j.ccell.2019.09.003 . PMID 31614115 .
- ^ Jump up to: а беременный в Лири Р.Дж., Саусен М., Кинде И., Пападопулос Н., Карптен Д.Д., Крейг Д., О'Шонесси Дж., Кинцлер К.В., Пармиджиани Г., Фогельштейн Б., Диаз Л.А., Велкулеску (ноябрь 2012 г.). «Обнаружение хромосомных изменений в кровообращении больных раком с секвенированием всего генома» . Научная трансляционная медицина . 4 (162): 162Ra154. doi : 10.1126/scitranslmed.3004742 . PMC 3641759 . PMID 23197571 .
- ^ Preobrazhenski BS (1966). «[Современные перспективы и метод систематического лечения кохлеарного неврита и хронической лабиринтопатии]». Вестник otorinolaringologii (на русском языке). 28 (1): 3–11. PMID 5988180 .
- ^ Бомонт Г., Доббинс С., Латта Д., Макмиллин В.П. (май 1990). «Мекитазин в лечении сеного». Британский журнал клинической практики . 44 (5): 183–8. PMID 1975200 .
- ^ Li W, Zhang X, Lu X, You L, Song Y, Luo Z, et al. (Октябрь 2017). «5-гидроксиметилцитозиновые сигнатуры в циркулирующей бесклеточной ДНК в качестве диагностических биомаркеров для рака человека» . Клеточные исследования . 27 (10): 1243–1257. doi : 10.1038/cr.2017.121 . PMC 5630683 . PMID 28925386 .
- ^ Песня CX, Yin S, Ma L, Wheeler A, Chen Y, Zhang Y, Liu B, Xiong J, Zhang W, Hu J, Zhou Z, Dong B, Tian Z, Jeffrey SS, Chua MS, So S, Li W , Wei Y, Diao J, Xie D, Quake Sr (октябрь 2017 г.). «5-гидроксиметилцитозиновые сигнатуры в бесклеточной ДНК предоставляют информацию о типах и этапах опухоли» . Клеточные исследования . 27 (10): 1231–1242. doi : 10.1038/cr.2017.106 . PMC 5630676 . PMID 28820176 .
- ^ Jump up to: а беременный в дюймовый и фон Батлер Т.М., Спеллман П.Т., Грей Дж (февраль 2017 г.). «ДНК циркулирующей точки как раннее обнаружение и диагностический инструмент». Текущее мнение в области генетики и развития . 42 : 14–21. doi : 10.1016/j.gde.2016.12.003 . PMID 28126649 .
- ^ Dressman D, Yan H, Traverso G, Kinzler KW, Vogelstein B (июль 2003 г.). «Преобразование молекул отдельных ДНК в флуоресцентные магнитные частицы для обнаружения и подсчета генетических вариаций» . Труды Национальной академии наук Соединенных Штатов Америки . 100 (15): 8817–22. Bibcode : 2003pnas..100.8817d . doi : 10.1073/pnas.1133470100 . PMC 166396 . PMID 12857956 .
- ^ Diehl F, Li M, He Y, Kinzler KW, Vogelstein B, Dressman D (июль 2006 г.). «Берег: ПЦР с одной молекулой на микрочастицах в эмульсиях с водой в масло». Природные методы . 3 (7): 551–9. doi : 10.1038/nmeth898 . PMID 16791214 . S2CID 7059151 .
- ^ Ньюман А.М., Братман С.В., Дж., Винн Дж.Ф., Эклов Н.К., Модлин Л.А., Лю К.Л., Нил Дж.В., Вейли Х.А., Мерритт Р.Е., Шрагер Дж. Б., Лу Б.В., Ализаде А.А., Дейн М (май 2014 г.). «Ультрачувствительный метод количественной циркулирующей опухолевой ДНК с широким охватом пациента» . Природная медицина . 20 (5): 548–54. doi : 10.1038/nm.3519 . PMC 4016134 . PMID 24705333 .
- ^ Jump up to: а беременный Ньюман А.М., Лавджой А.Ф., Класс Д.М., Курц Д.М., Шабон Дж.Дж., Шерер Ф. и др. (Май 2016 г.). «Интегрированное подавление цифровых ошибок для улучшения обнаружения циркулирующей опухолевой ДНК» . Nature Biotechnology . 34 (5): 547–555. doi : 10.1038/nbt.3520 . PMC 4907374 . PMID 27018799 .
- ^ Forshew T, Murtaza M, Parkinson C, Gale D, Tsui DW, Kaper F, Dawson SJ, Piskorz AM, Jimenez-Linan M, Bentley D, Hadfield J, May AP, Caldas C, Brenton JD, Rosenfeld N (май 2012 г.) Полем «Неинвазивная идентификация и мониторинг раковых мутаций путем целенаправленного глубокого секвенирования плазменной ДНК». Научная трансляционная медицина . 4 (136): 136RA68. doi : 10.1126/scitranslmed.3003726 . PMID 22649089 . S2CID 34723244 .
- ^ КОНДЕ I, WU J, Papadopoulos N, Kinzler KW, Vogelstein B (июнь 2011 г.). «Обнаружение и количественное определение редких мутаций с массовой параллельной секвенированием» . Труды Национальной академии наук Соединенных Штатов Америки . 108 (23): 9530–5. Bibcode : 2011pnas..108.9530K . doi : 10.1073/pnas.1105422108 . PMC 3111315 . PMID 21586637 .
- ^ Кеннеди С.Р., Шмитт М.В., Фокс Э.Дж., Корн Б.Ф., Салк Дж.Дж., Ан Э.Х., Принл М.Дж., Куонг К.Дж., Шен Дж.С., Риски Р.А., Лоеб Л.А. (ноябрь 2014). «Обнаружение ультраловочастотных мутаций с помощью дуплексного секвенирования» . Природные протоколы . 9 (11): 2586–606. doi : 10.1038/nprot.2014.170 . PMC 4271547 . PMID 25299156 .
- ^ Jump up to: а беременный в Гербертс, Кэмерон; Аннала, Матти; Сипола, Джунатан; Нг, Сара У.С.; Чен, Синьи Э.; Нурминен, Анси; Korhonen, Olga v.; Munzur, Aslı D.; Бежа, Кевин; Шёнлау, Елена; Берналес, Сесили Q.; Ритч, Эли; Бекон, Джек VW; Недостаток, Натан А.; Никтер, Матти (август 2022 г.). «Глубокая хронология CtDNA в целом геноме рака простаты устойчивого к лечению» . Природа . 608 (7921): 199–208. Bibcode : 2022natur.608..199H . doi : 10.1038/s41586-022-04975-9 . ISSN 1476-4687 . PMID 35859180 . S2CID 250730778 .
- ^ Зу, Донхуи; День, Роберт; Кокадиз, Джуди А; Паракал, Сара; Митчелл, Уилсон; Черный, Майкл А; Лоуренс, Бен; Фицджеральд, Сандра; Печать, Кристин; Джексон, Кристофер; Гилфорд, Парри (2020-11-13). «Циркулирующая опухолевая ДНК является чувствительным маркером для обычного мониторинга ответа на лечение при распространенном колоректальном раке» . Канцерогенез . 41 (11): 1507–1517. doi : 10.1093/carcin/bgaa102 . ISSN 0143-3334 . PMID 32955091 .
- ^ Фицджеральд, Сандра; Бленкирон, Чери; Стивенс, Розали; Мати, Джон А.; Сомерс-Эдгар, Тиффани; Рольф, Гилл; Мартин, Ричард; Джексон, Кристофер; Эклс, Майкл; Робб, Тамсин; Роджер, Юань; Лоуренс, Бен; Гилфорд, Парри; Лашам, Аннет; Печать, Кристин Г. (2023). «Динамическая мутационная сложность ctDNA у пациентов с меланомой, получающей иммунотерапию» . Молекулярная диагностика и терапия . 27 (4): 537–550. doi : 10.1007/s40291-023-00651-4 . ISSN 1177-1062 . PMC 10131510 . PMID 37099071 .
- ^ Rapisuwon S, Vietch Ee, Wellstein A (2016). «Циркулирующие биомаркеры для мониторинга прогрессирования и лечения рака» . Вычислительный и структурный биотехнологический журнал . 14 : 211–22. doi : 10.1016/j.csbj.2016.05.004 . PMC 4913179 . PMID 27358717 .
- ^ Crowley E, Di Nicolantonio F, Loupakis F, Bardelli A (август 2013). «Жидкая биопсия: мониторинг генетики рака в крови» . Природные обзоры. Клиническая онкология . 10 (8): 472–84. doi : 10.1038/nrclinonc.2013.110 . PMID 23836314 . S2CID 25537784 .
- ^ Murtaza M, Dawson SJ, Pogrebniak K, Rueda OM, Provenzano E, Grant J, Chin SF, Tsui DW, Marass F, Gale D, Ali HR, Shah P, Contece-Cuomo T, Farahani H, Shumansky K, Kingsbury Z, Humphray S, Bentley D, Shah SP, Wallis M, Rosenfeld N, Caldas C (ноябрь 2015). «Многофокальная клональная эволюция, характеризующаяся с использованием циркулирующей опухолевой ДНК в случае метастатического рака молочной железы» . Природная связь . 6 (1): 8760. Bibcode : 2015natco ... 6,8760 м . doi : 10.1038/ncomms9760 . PMC 4659935 . PMID 26530965 .
Дальнейшее чтение
[ редактировать ]- Применение метилирования ДНК циркулирующей опухоли при диагностике рака легких в мае 2019 г.
- Циркулирующая опухолевая ДНК: новое поколение биомаркеров рака февраль 2014 г.
- Ctdna «жидкая биопсия» может революционизировать лечение рака ноябрь 2014
- Карачалиу Н., Майо-де-Лас-Касас С., Молина-Вила М.А., Розелл Р (март 2015 г.). «Жидкая биопсия в реальном времени становится реальностью в лечении рака» . Анналы трансляционной медицины . 3 (3): 36. doi : 10.3978/j.issn.2305-5839.2015.01.16 . PMC 4356857 . PMID 25815297 .
- Марусина, Кейт (8 февраля 2018 г.). «Дразнить циркулирующую опухолевую ДНК» . Clinicalomics . Получено 5 марта 2018 года .
- Du-Bois, Asante (2019). «Жидкая биопсия при раке яичников с использованием циркулирующей опухолевой ДНК и клеток: готово к прайм -тайм?» Полем Раковые письма . 468 : 59–71. doi : 10.1016/j.canlet.2019.10.014 . PMID 31610267 .
- NUCPOSDB: база данных о позиционировании нуклеосомы in vivo и нуклеосомной ДНК без клеток