Вирофаг
| Лавидавирусиды | |
|---|---|

| |
| Спутник-вирофаг | |
| Классификация вирусов | |
| (без рейтинга): | Вирус |
| Область : | Вариднавирия |
| Королевство: | Бэмфордвире |
| Тип: | Преплазмовирус |
| Сорт: | Маверивирицетес |
| Заказ: | Относится к вирусам |
| Семья: | Лавидавирусиды |
| Роды и виды | |

(Б) Когда клетка-хозяин одновременно инфицирована гигантским вирусом и его вирофагом, последний паразитирует на гигантской вирусной фабрике. Присутствие вирофагов может серьезно повлиять на инфекционность гигантского вируса, снизив эффективность его репликации и увеличив выживаемость клетки-хозяина.
(C) Когда геном гигантского вируса паразитирует провирофагом, последний экспрессируется во время репликации гигантского вируса. Вирофаг производится на гигантской вирусной фабрике и ингибирует репликацию гигантского вируса, тем самым увеличивая выживаемость клеток-хозяев.

(B) Концепция сателлитного вируса предполагает, что вирус инициирует экспрессию и репликацию своего генома в ядре, используя механизм клетки-хозяина, а затем переходит в цитоплазму. В цитоплазме сателлитный вирус захватывает механизм морфогенеза своего вируса-помощника, чтобы произвести свое потомство.
Вирофаги представляют собой небольшие вирусные фаги с двухцепочечной ДНК, которые требуют совместной инфекции с другим вирусом. Вирусы, вызывающие коинфекцию, обычно представляют собой гигантские вирусы . Для собственной репликации вирофаги полагаются на фабрику вирусной репликации коинфицирующего гигантского вируса. Одной из характеристик вирофагов является то, что они имеют паразитные отношения с коинфицирующим вирусом. Их зависимость от гигантского вируса для репликации часто приводит к дезактивации гигантских вирусов. Вирофаг может улучшить восстановление и выживаемость организма хозяина.
Все известные вирофаги объединены в семейство Lavidaviridae (от «большие вирусозависимые или ассоциированные» + -viridae ). [ 2 ]
Открытие
[ редактировать ]Первый вирофаг был обнаружен в градирне в Париже в 2008 году. Он был обнаружен вместе с коинфицирующим его гигантским вирусом Acanthamoeba castellanii mamavirus (ACMV). Вирофаг получил название «Спутник» , и его репликация полностью зависела от совместной инфекции ACMV и его цитоплазматического механизма репликации. Также было обнаружено, что «Спутник» оказывает ингибирующее действие на ACMV и улучшает выживаемость хозяина. Другие охарактеризованные вирофаги включают Спутник 2, Спутник 3, Замилон и Мавирус . [ 3 ] [ 4 ] [ 5 ] [ 6 ]
Большинство этих вирофагов обнаруживаются путем анализа наборов метагеномных данных. В метагеномном анализе последовательности ДНК проходят через множество биоинформатических алгоритмов, которые выявляют определенные важные закономерности и характеристики. В этих наборах данных присутствуют гигантские вирусы и вирофаги. Их разделяют путем поиска последовательностей длиной от 17 до 20 т.п.н. , которые имеют сходство с уже секвенированными вирофагами. Эти вирофаги могут иметь линейные или кольцевые геномы двухцепочечной ДНК. [ 7 ] Известные вирофаги в культуре имеют икосаэдрические частицы капсида длиной от 40 до 80 нанометров. [ 8 ] а частицы вирофагов настолько малы, что для их просмотра необходимо использовать электронную микроскопию. Анализ метагеномных последовательностей был использован для предсказания около 57 полных и частичных геномов вирофагов. [ 9 ] а в декабре 2019 года — идентифицировать 328 высококачественных (полных или почти полных) геномов из различных сред обитания, включая кишечник человека, ризосферу растений и недра Земли, из 27 различных таксономических клад. [ 10 ]
Диапазон хостов и репликация
[ редактировать ]Для репликации вирофагам необходим коинфицирующий вирус. Вирофаги не имеют необходимых ферментов для самостоятельной репликации. Вирофаги используют гигантский механизм репликации вирусов для репликации собственных геномов и продолжения своего существования. Диапазон хозяев вирофагов включает гигантские вирусы с геномами двухцепочечной ДНК. Вирофаги используют транскрипционный аппарат этих гигантских вирусов для собственной репликации вместо транскрипционного аппарата хозяина. Например, открытие вирофага, связанного с вирусом Самба, снизило концентрацию вируса в организме хозяина, пока вирофаг размножался с использованием гигантского вируса. Амеба-хозяин также продемонстрировала частичное выздоровление от заражения вирусом Самба. [ 7 ]
Геном
[ редактировать ]двухцепочечной ДНК Вирофаги имеют небольшие геномы , имеющие кольцевую или линейную форму. Размер этих геномов может варьироваться в зависимости от гигантского вируса, который он заражает. Большинство вирофагов имеют геномы размером около 17–30 т.п.н. (килобазовые пары). [ 8 ] [ 9 ] Их геном защищен икосаэдрическим капсидом длиной примерно 40–80 нм. [ 8 ] Напротив, их коинфицирующие гигантские вирусные аналоги могут иметь геномы размером до 1–2 Мбит/с (мегабазовые пары). [ 7 ] Некоторые из крупнейших геномов вирофагов аналогичны размеру генома аденовируса. [ 8 ]
| Геном
Размер (кбит/с) |
Размер частиц
(диаметр, в нм) | |
|---|---|---|
| Вирус : Полиовирус | 7 | 30 |
| Вирус: Аденовирусиды. | 26–48 | 90–100 |
| Вирофаг: Замилон Вирофаг | 17 | 50–60 |
| Вирофаг: Спутник Вирофаг | 18 | 74 |
| Гигантский вирус : вирус Cafeteria roenbergensis. | 700 | 75 |
| Гигантский вирус : Мимивирус | 1,181 | 400–800 |
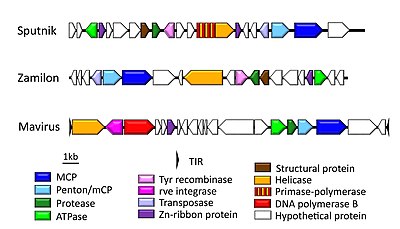
Все известные на данный момент вирофаги имеют четыре основных гена. Это специфичные для вирофага основные и второстепенные капсидные белки (MCP и mCP), PRO ( цистеиновая протеаза , упаковывающая ДНК ) и АТФаза . Два капсида почти всегда встречаются в консервативном блоке. [ 10 ] MCP имеет два вертикальных складчатых домена желейного рулона, типичных для Bamfordvirae , тогда как mCP (пентон) имеет регулярный складчатый домен желейного рулона. [ 11 ]
Таксономия
[ редактировать ]Семейство Lavidaviridae, состоящее из двух родов, Sputnikvirus и Mavirus , было создано Международным комитетом по таксономии вирусов для классификации вирофагов. Это единственное семейство отряда Priklausovirales (от литовского priklausomas , «зависимый»), который, в свою очередь, является единственным отрядом класса Maveriviricetes (от транспозонов Maverick ). [ 8 ] [ 12 ]
- Семейство Lavidaviridae
- Неназначенный род
Кроме того, геномы вирофагов, идентифицированные из метагеномов, были классифицированы вместе с изолированными вирофагами на 27 различных клад с постоянной длиной генома, содержанием генов и распределением среды обитания. [ 10 ] Некоторые фрагментарные последовательности вирофагов были дополнительно обнаружены в метагеноме Замка Локи . [ 13 ]


Ссылки
[ редактировать ]- ^ Jump up to: а б с д Мугари С., Сахми-Бунсиар Д., Левассер А., Колсон П. и Ла Скола Б. (2019) «Вирофаги гигантских вирусов: обновление одиннадцати». Вирусы , 11 (8): 733. два : 10.3390/v11080733 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0.
- ^ Дюпоншель, С; Фишер, М.Г. (март 2019 г.). «Вива лавидавирусы! Пять особенностей вирофагов, паразитирующих на гигантских ДНК-вирусах» . ПЛОС Патогены . 15 (3): e1007592. дои : 10.1371/journal.ppat.1007592 . ПМЦ 6428243 . ПМИД 30897185 .
- ^ Фишер М.Г., Саттл, Калифорния (апрель 2011 г.). «Вирофаг – источник больших ДНК-транспозонов». Наука . 332 (6026): 231–4. Бибкод : 2011Sci...332..231F . дои : 10.1126/science.1199412 . ПМИД 21385722 . S2CID 206530677 .
- ^ Фишер М.Г., Хакл (декабрь 2016 г.). «Интеграция генома хозяина и гигантская вирус-индуцированная реактивация мавируса вирофага». Природа . 540 (7632): 288–91. Бибкод : 2016Natur.540..288F . дои : 10.1038/nature20593 . ПМИД 27929021 . S2CID 4458402 .
- ^ таксономия. «Браузер таксономии (Lavidaviridae)» . www.ncbi.nlm.nih.gov . Проверено 20 сентября 2023 г.
- ^ таксономия. «Таксономический браузер (Preplasmiviricota)» . www.ncbi.nlm.nih.gov . Проверено 20 сентября 2023 г.
- ^ Jump up to: а б с Кацуракис, Арис; Асвад, Амр (2014). «Происхождение гигантских вирусов, вирофагов и их родственников в геномах хозяев» . БМК Биология . 12 : 2–3. дои : 10.1186/s12915-014-0051-y . ПМК 4096385 . ПМИД 25184667 .
- ^ Jump up to: а б с д и Крупович, Март; Кун, Йенс; Фишер, Меттиас (осень 2015 г.). «Система классификации вирофагов и сателлитных вирусов» (PDF) . Архив вирусологии . 161 (1): 233–247. дои : 10.1007/s00705-015-2622-9 . ПМИД 26446887 . S2CID 14196910 – через Springer.
- ^ Jump up to: а б Ру, Саймон; Чан, Леонг-Кит; Иган, Роб; Мальмстрем, Рекс Р.; МакМахон, Кэтрин Д.; Салливан, Мэтью Б. (11 октября 2017 г.). «Экогеномика вирофагов и их гигантских вирусных хозяев, оцененная с помощью метагеномики временных рядов» . Природные коммуникации . 8 (1): 858. Бибкод : 2017NatCo...8..858R . дои : 10.1038/s41467-017-01086-2 . ISSN 2041-1723 . ПМЦ 5636890 . ПМИД 29021524 .
- ^ Jump up to: а б с Паес-Эспино, Дэвид; Чжоу, Цзинли; Ру, Саймон; Найфач, Стивен; Павлопулос, Георгиос А.; Шульц, Фредерик; МакМахон, Кэтрин Д.; Уолш, Дэвид; Войке, Таня; Иванова Наталья Н.; Элоэ-Фадрош, Эмили А.; Тринге, Сюзанна Г.; Кирпидес, Никос К. (10 декабря 2019 г.). «Разнообразие, эволюция и классификация вирофагов, обнаруженные с помощью глобальной метагеномики» . Микробиом . 7 (1): 157. дои : 10.1186/s40168-019-0768-5 . ПМК 6905037 . ПМИД 31823797 .
- ^ Борн, Д; Рейтер, Л; Мерсдорф, У; Мюллер, М; Фишер, МГ; Мейнхарт, А; Райнштейн, Дж. (10 июля 2018 г.). «Структура капсидного белка, самосборка и процессинг раскрывают морфогенез морского вирофага мавируса» . Труды Национальной академии наук Соединенных Штатов Америки . 115 (28): 7332–7337. Бибкод : 2018PNAS..115.7332B . дои : 10.1073/pnas.1805376115 . ПМК 6048507 . ПМИД 29941605 .
- ^ Кунин Е.В., Доля В.В., Крупович М., Варсани А., Вольф Ю.И., Ютин Н., Зербини М., Кун Дж.Х. (октябрь 2019 г.). «Создать мегатаксономическую структуру, заполнив все основные таксономические ранги, для ДНК-вирусов, кодирующих основные капсидные белки типа вертикального желеобразного рулона» . Предложение ICTV (Taxoprop) : 2019.003G. дои : 10.13140/RG.2.2.14886.47684 .
- ^ Бэкстрём Д., Ютин Н., Йоргенсен С.Л., Дхарамши Дж., Хома Ф., Заремба-Недведска К., Спанг А., Вольф Ю.И., Кунин Е.В., Эттема Т.Дж. (2019). «Вирусные геномы из глубоководных отложений расширяют океанский мегавиром и поддерживают независимое происхождение вирусного гигантизма» . мБио 10 (2):e02497-1 дои : 10.1128/mBio.02497-18 . ПМК 6401483 . ПМИД 30837339 . PDF
- ^ Дюпоншель, С. и Фишер, М.Г. (2019) «Вива лавидавирусы! Пять особенностей вирофагов, паразитирующих на гигантских ДНК-вирусах». PLoS-возбудители , 15 (3). дои : 10.1371/journal.ppat.1007592 .
 Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0 .
Материал был скопирован из этого источника, который доступен по международной лицензии Creative Commons Attribution 4.0 .
