Вироме
| Часть серии о |
| Микробиомы |
|---|
 |
Виром – это совокупность вирусов. [1] [2] который часто исследуется и описывается с помощью метагеномного секвенирования вирусных нуклеиновых кислот. [3] которые обнаружены связанными с определенной экосистемой, организмом или холобионтом . Это слово часто используется для описания метагеномов вирусов окружающей среды . Вирусы , включая бактериофаги , встречаются во всех средах, а исследования вирома дали представление о круговороте питательных веществ . [4] [5] развитие иммунитета, [6] и основной источник генов посредством лизогенной конверсии . [7] человеческий виром был охарактеризован в девяти органах (толстая кишка, печень, легкие, сердце, мозг, почки, кожа, кровь, волосы) 31 финна Кроме того, с использованием методологий qPCR и NGS . [8]
История
[ редактировать ]Первые комплексные исследования виромов были проведены методом секвенирования с помощью дробовика. [9] которую часто называют метагеномикой. В 2000-х годах лаборатория Ровера секвенировала виромы из морской воды. [9] [10] морские отложения, [11] стул взрослого человека, [12] детский человеческий стул, [13] земля, [14] и кровь. [15] Эта группа также выполнила первый РНК-виром совместно с сотрудниками Института геномики Сингапура. [16] На основании этих ранних работ был сделан вывод, что большая часть геномного разнообразия содержится в глобальном вироме и что большая часть этого разнообразия остается нехарактерной. [17] Эта точка зрения была поддержана отдельными проектами геномного секвенирования, особенно фага микобактерий. [18]
К концу 2010-х годов достижения в технологиях секвенирования позволили провести глубокое исследование виромов. [19] В результате этих достижений виром кишечника человека, в частности, привлек повышенное внимание. [20] [21]
Методы исследования
[ редактировать ]Для изучения вирома вирусоподобные частицы отделяют от клеточных компонентов, обычно используя комбинацию фильтрации, центрифугирования плотности и ферментативной обработки для избавления от свободных нуклеиновых кислот. [22] Затем нуклеиновые кислоты секвенируют и анализируют с использованием метагеномных методов. Альтернативно, существуют недавние вычислительные методы, которые используют непосредственно метагеномные собранные последовательности для обнаружения вирусов. [23]
Global Ocean Viromes (GOV) представляет собой набор данных, состоящий из глубокого секвенирования более чем 150 образцов, собранных в мировом океане в течение двух периодов исследований международной командой. [24]
Вирусные хосты
[ редактировать ]
Вирусы являются наиболее распространенными биологическими объектами на Земле, но проблемы с обнаружением, изоляцией и классификацией неизвестных вирусов помешали провести исчерпывающие исследования глобального вирома. [25] более 5 ТБ данных метагеномных последовательностей из 3042 географически разнообразных образцов. Для оценки глобального распространения, филогенетического разнообразия и специфичности вирусов к хозяину было использовано [25]
В августе 2016 года более 125 000 частичных ДНК вирусных геномов, включая самый крупный из когда-либо идентифицированных фагов, увеличили количество известных вирусных генов в 16 раз. [25] Для идентификации предполагаемых связей вируса-хозяина использовался набор вычислительных методов. [25] Информация о хозяине изолята вируса была проецирована на группу, в результате чего хозяин был назначен для 2,4% вирусных групп. [25]
Затем прокариотическая иммунная система CRISPR -Cas содержит «библиотеку» фрагментов генома фагов (протоспейсеров), ранее заразивших хозяина. [25] Спейсеры из изолированных микробных геномов, соответствующие метагеномным вирусным контигам (мВК), были идентифицированы для 4,4% вирусных групп и 1,7% одиночных вирусов. [25] Была исследована гипотеза о том, что гены вирусной транспортной РНК (тРНК) происходят от хозяина. [25]
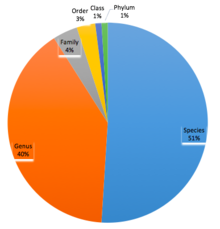
Вирусные тРНК, идентифицированные в 7,6% мВК, были сопоставлены для выделения геномов одного вида или рода. [25] Специфичность распределения вируса-хозяина на основе тРНК была подтверждена совпадением спейсеров CRISPR-Cas, показавшим согласие на 94% на уровне рода. Эти подходы выявили 9992 предполагаемых ассоциации хозяин-вирус, что позволило отнести хозяина к 7,7% mVC. [25] Большинство этих связей ранее были неизвестны и включают хозяев из 16 типов прокариот, для которых ранее не были идентифицированы вирусы. [25]
Многие вирусы специализируются на заражении родственных хостов. [25] Могут существовать вирусы широкого профиля, которые заражают хозяев разных таксономических порядков. [25] Большинство совпадений спейсеров CRISPR были связаны с вирусными последовательностями и хозяевами одного вида или рода. [25] Некоторые mVC были связаны с несколькими хозяевами из более высоких таксонов. Вирусная группа, состоящая из макинтошей из пероральных образцов человека, содержала три различных фотоспейсера, почти точно соответствующих спейсерам у Actionbacteria и Bacillota . [25]
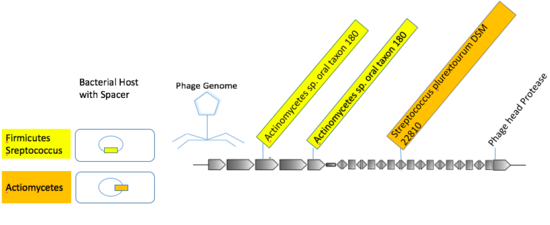
В январе 2017 года система IMG/VR [26] - крупнейшая интерактивная общедоступная вирусная база данных, содержащая 265 000 метагеномных вирусных последовательностей и изолятов вирусов. В ноябре 2018 года это число превысило 760 000 (IMG/VR v.2.0). [27] Системы IMG/VR служат отправной точкой для анализа последовательностей вирусных фрагментов, полученных из метагеномных образцов.
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Андерсон Н.Г., Герин Дж.Л., Андерсон Н.Л. (июль 2003 г.). «Глобальный скрининг вирусных патогенов человека» . Новые инфекционные заболевания . 9 (7): 768–774. дои : 10.3201/eid0907.030004 . ПМК 3023425 . ПМИД 12890315 .
- ^ Сарате С., Табоада Б., Йокупичио-Монрой М., Ариас К.Ф. (ноябрь 2017 г.). «Человеческий виром» Архивы медицинских исследований . 48 (8): 701–716. doi : 10.1016/j.arcmed.2018.01.005 . ПМИД 29398104 .
- ^ МакДэниел Л., Брейтбарт М. , Мобберли Дж., Лонг А., Хейнс М., Ровер Ф., Пол Дж. Х. (сентябрь 2008 г.). «Метагеномный анализ лизогении в заливе Тампа: значение для экспрессии генов профагов» . ПЛОС ОДИН . 3 (9): е3263. Бибкод : 2008PLoSO...3.3263M . дои : 10.1371/journal.pone.0003263 . ПМЦ 2533394 . ПМИД 18810270 .
- ^ Вильгельм С.В., Саттл, Калифорния (1999). «Вирусы и циклы питательных веществ в море» . Бионаука . 49 (10): 781–788. дои : 10.2307/1313569 . ISSN 1525-3244 . JSTOR 1313569 .
- ^ Уэгли Л., Эдвардс Р., Родригес-Брито Б., Лю Х., Ровер Ф. (ноябрь 2007 г.). «Метагеномный анализ микробного сообщества, связанного с кораллами Porites astreoides». Экологическая микробиология . 9 (11): 2707–2719. Бибкод : 2007EnvMi...9.2707W . дои : 10.1111/j.1462-2920.2007.01383.x . ПМИД 17922755 .
- ^ Барр Дж.Дж., Ауро Р., Фурлан М., Уайтсон К.Л., Эрб М.Л., Польяно Дж. и др. (июнь 2013 г.). «Бактериофаг, прикрепляющийся к слизи, обеспечивает иммунитет, не связанный с хозяином» . Труды Национальной академии наук Соединенных Штатов Америки . 110 (26): 10771–10776. Бибкод : 2013PNAS..11010771B . дои : 10.1073/pnas.1305923110 . ПМК 3696810 . ПМИД 23690590 .
- ^ Шарон И., Батчикова Н., Аро Э.М., Джильоне С., Мейннел Т., Глейзер Ф. и др. (июль 2011 г.). «Сравнительная метагеномика микробных признаков океанических вирусных сообществ» . Журнал ISME . 5 (7): 1178–1190. Бибкод : 2011ISMEJ...5.1178S . дои : 10.1038/ismej.2011.2 . ПМК 3146289 . ПМИД 21307954 .
- ^ Пьёрия Л., Пратас Д., Топпинен М., Хедман К., Саянтила А., Пердомо М.Ф. (2023). «Разоблачение вирома эукариотической ДНК, находящегося в тканях человека» . Исследования нуклеиновых кислот . 51 (7): 3223–3239. дои : 10.1093/nar/gkad199 . ПМЦ 10123123 . ПМИД 36951096 .
- ^ Jump up to: а б Брейтбарт М., Саламон П., Андресен Б., Махаффи Дж.М., Сигалл А.М., Мид Д. и др. (октябрь 2002 г.). «Геномный анализ некультивируемых морских вирусных сообществ» . Труды Национальной академии наук Соединенных Штатов Америки . 99 (22): 14250–14255. Бибкод : 2002PNAS...9914250B . дои : 10.1073/pnas.202488399 . ПМЦ 137870 . ПМИД 12384570 .
- ^ Энгли Ф.Е., Фелтс Б., Брейтбарт М., Саламон П., Эдвардс Р.А., Карлсон С. и др. (ноябрь 2006 г.). «Морские виромы четырех океанических регионов» . ПЛОС Биология . 4 (11): е368. doi : 10.1371/journal.pbio.0040368 . ПМК 1634881 . ПМИД 17090214 .
- ^ Брейтбарт М., Фелтс Б., Келли С., Махаффи Дж. М., Налтон Дж., Саламон П., Ровер Ф. (март 2004 г.). «Разнообразие и популяционная структура прибрежного вирусного сообщества морских отложений» . Слушания. Биологические науки . 271 (1539): 565–574. дои : 10.1098/rspb.2003.2628 . ПМЦ 1691639 . ПМИД 15156913 .
- ^ Брейтбарт М., Хьюсон И., Фелтс Б., Махаффи Дж. М., Налтон Дж., Саламон П., Ровер Ф. (октябрь 2003 г.). «Метагеномный анализ некультивируемого вирусного сообщества из человеческих фекалий» . Журнал бактериологии . 185 (20): 6220–6223. дои : 10.1128/jb.185.20.6220-6223.2003 . ПМК 225035 . ПМИД 14526037 .
- ^ Брейтбарт М., Хейнс М., Келли С., Энгли Ф., Эдвардс Р.А., Фелтс Б. и др. (июнь 2008 г.). «Вирусное разнообразие и динамика в кишечнике младенца» . Исследования в области микробиологии . 159 (5): 367–373. дои : 10.1016/j.resmic.2008.04.006 . ПМИД 18541415 .
- ^ Фирер Н., Брейтбарт М., Нултон Дж., Саламон П., Лозупон С., Джонс Р. и др. (ноябрь 2007 г.). «Метагеномный анализ и анализ рРНК малых субъединиц выявляет генетическое разнообразие бактерий, архей, грибов и вирусов в почве» . Прикладная и экологическая микробиология . 73 (21): 7059–7066. Бибкод : 2007ApEnM..73.7059F . дои : 10.1128/aem.00358-07 . ПМК 2074941 . ПМИД 17827313 .
- ^ Брейтбарт М., Ровер Ф (ноябрь 2005 г.). «Метод обнаружения новых ДНК-вирусов в крови с использованием отбора вирусных частиц и дробового секвенирования» . БиоТехники . 39 (5): 729–736. дои : 10.2144/000112019 . ПМИД 16312220 .
- ^ Чжан Т., Брейтбарт М., Ли В.Х., Ран Дж.К., Вэй К.Л., Со С.В. и др. (январь 2006 г.). «РНК-вирусное сообщество в фекалиях человека: распространенность фитопатогенных вирусов» . ПЛОС Биология . 4 (1): e3. doi : 10.1371/journal.pbio.0040003 . ПМК 1310650 . ПМИД 16336043 .
- ^ Эдвардс Р.А., Ровер Ф. (июнь 2005 г.). «Вирусная метагеномика». Обзоры природы. Микробиология . 3 (6): 504–510. дои : 10.1038/nrmicro1163 . ПМИД 15886693 . S2CID 8059643 .
- ^ Ровер Ф (апрель 2003 г.). «Глобальное разнообразие фагов» . Клетка . 113 (2): 141. doi : 10.1016/s0092-8674(03)00276-9 . ПМИД 12705861 .
- ^ Гармаева С., Синха Т., Курильщиков А., Фу Дж., Вейменга С., Жернакова А. (октябрь 2019 г.). «Изучение кишечного вирома в эпоху метагеномики: проблемы и перспективы» . БМК Биология . 17 (1): 84. дои : 10.1186/s12915-019-0704-y . ПМК 6819614 . ПМИД 31660953 .
- ^ Шкопоров А.Н., Клуни А.Г., Саттон Т.Д., Райан Ф.Дж., Дейли К.М., Нолан Дж.А. и др. (октябрь 2019 г.). «Виром кишечника человека очень разнообразен, стабилен и индивидуален» . Клетка-хозяин и микроб . 26 (4): 527–541.е5. дои : 10.1016/j.chom.2019.09.009 . ПМИД 31600503 . S2CID 204242937 .
- ^ Клуни А.Г., Саттон Т.Д., Шкопоров А.Н., Холохан Р.К., Дейли К.М., О'Риган О. и др. (декабрь 2019 г.). «Анализ цельного вирома проливает свет на вирусную темную материю при воспалительных заболеваниях кишечника» . Клетка-хозяин и микроб . 26 (6): 764–778.e5. дои : 10.1016/j.chom.2019.10.009 . ПМИД 31757768 . S2CID 208234961 .
- ^ Тербер Р.В., Хейнс М., Брейтбарт М., Уэгли Л., Ровер Ф. (2009). «Лабораторные процедуры по созданию вирусных метагеномов». Протоколы природы . 4 (4): 470–483. дои : 10.1038/nprot.2009.10 . ПМИД 19300441 . S2CID 205464352 .
- ^ Паес-Эспино Д., Павлопулос Г.А., Иванова Н.Н., Кирпидес Н.К. (август 2017 г.). «Конвейер обнаружения нецелевых последовательностей вирусов и кластеризация вирусов для метагеномных данных» . Протоколы природы . 12 (8): 1673–1682. дои : 10.1038/нпрот.2017.063 . ПМИД 28749930 . S2CID 2127494 .
- ^ Тан Л (июль 2019 г.). «Океанские виромы от полюса до полюса» . Основные моменты исследования. Природные методы (статья). 16 (7): 575. дои : 10.1038/s41592-019-0480-1 . ПМИД 31249411 . – через Springer Nature (требуется подписка)
- ^ Jump up to: а б с д и ж г час я дж к л м н тот Паес-Эспино Д., Элое-Фадрош Е.А., Павлопулос Г.А., Томас А.Д., Хантеманн М., Михайлова Н. и др. (август 2016 г.). «Открытие земного вирома » Природа 536 (7617): 425–430. Бибкод : 2016Nature.536..425P . дои : 10.1038/nature19094 . ПМИД 27533034 . S2CID 4466854 .
- ^ Паес-Эспино Д., Чен И.А., Паланиаппан К., Ратнер А., Чу К., Сето Е. и др. (январь 2017 г.). «IMG/VR: база данных культивируемых и некультивируемых ДНК-вирусов и ретровирусов» . Исследования нуклеиновых кислот . 45 (Д1): Д457–Д465. дои : 10.1093/nar/gkw1030 . ПМК 5210529 . ПМИД 27799466 .
- ^ Паес-Эспино Д., Ру С., Чен И.А., Паланиаппан К., Ратнер А., Чу К. и др. (январь 2019 г.). «IMG/VR v.2.0: интегрированная система управления и анализа данных для культивируемых и находящихся в окружающей среде вирусных геномов» . Исследования нуклеиновых кислот . 47 (Д1): Д678–Д686. дои : 10.1093/nar/gky1127 . ПМК 6323928 . ПМИД 30407573 .
