Транскриптом
Транскриптом — совокупность всех транскриптов РНК , включая кодирующие и некодирующие , в индивидууме или популяции клеток . Этот термин также иногда может использоваться для обозначения всех РНК или только мРНК , в зависимости от конкретного эксперимента. Термин «транскриптом» представляет собой сочетание слов «транскрипт» и «геном» ; это связано с процессом производства транскриптов во время биологического процесса транскрипции .
Ранние этапы аннотаций транскриптома начались с библиотек кДНК , опубликованных в 1980-х годах. Впоследствии появление высокопроизводительных технологий привело к более быстрым и эффективным способам получения данных о транскриптоме. Для изучения транскриптома используются два биологических метода, а именно микрочип ДНК , метод, основанный на гибридизации, и RNA-seq , подход, основанный на последовательностях. [ 1 ] РНК-секвенирование является предпочтительным методом и доминирующим методом транскриптомики с 2010-х годов. Транскриптомика отдельных клеток позволяет отслеживать изменения транскриптов с течением времени внутри отдельных клеток.
Данные, полученные из транскриптома, используются в исследованиях, чтобы получить представление о таких процессах, как клеточная дифференциация , канцерогенез , регуляция транскрипции и открытие биомаркеров , среди других. Данные, полученные с помощью транскриптома, также находят применение при установлении филогенетических связей в процессе эволюции и при экстракорпоральном оплодотворении . Транскриптом тесно связан с другими -оме биологическими областями исследований, основанными на ; он дополняет протеом и метаболом и включает в себя транслатом , экзом , мейом и танатотранскриптом , которые можно рассматривать как некоторые поля, изучающие определенные типы транскриптов РНК. Между транскриптомом и другими -омами существуют количественные и консервативные отношения, и данные транскриптомики можно эффективно использовать для прогнозирования других молекулярных видов, таких как метаболиты. [ 2 ] Существует множество общедоступных баз данных транскриптомов.
Этимология и история
[ редактировать ]Слово «транскриптом» представляет собой сочетание слов «транскрипт» и «геном» . Он появился вместе с другими неологизмами, образованными с помощью суффиксов -ome и -omics для обозначения всех исследований, проводимых в полногеномном масштабе в области наук о жизни и технологий. Таким образом, транскриптом и транскриптомика были одними из первых слов, появившихся наряду с геномом и протеомом. [ 3 ] Первое исследование, в котором представлен случай коллекции библиотеки мРНК кДНК шелкопряда , было опубликовано в 1979 году. [ 4 ] Первое плодотворное исследование, в котором упоминается и исследуется транскриптом организма, было опубликовано в 1997 году и описало 60 633 транскрипта, экспрессируемых в S. cerevisiae, с использованием серийного анализа экспрессии генов (SAGE). [ 5 ] С появлением высокопроизводительных технологий и биоинформатики и последующим увеличением вычислительной мощности стало все более эффективно и легко характеризовать и анализировать огромные объемы данных. [ 3 ] Попытки охарактеризовать транскриптом стали более заметными с появлением автоматизированного секвенирования ДНК в 1980-х годах. [ 6 ] В 1990-е годы экспрессированных последовательностей . для идентификации генов и их фрагментов использовалось секвенирование [ 7 ] За этим последовали такие методы, как серийный анализ экспрессии генов (SAGE), анализ экспрессии генов (CAGE) и массово-параллельное сигнатурное секвенирование (MPSS).
Транскрипция
[ редактировать ]Транскриптом включает в себя все транскрипты рибонуклеиновой кислоты (РНК), присутствующие в данном организме или экспериментальном образце. [ 8 ] РНК — основной носитель генетической информации, отвечающий за процесс преобразования ДНК в фенотип организма. Ген может дать начало одноцепочечной информационной РНК (мРНК) посредством молекулярного процесса, известного как транскрипция ; эта мРНК комплементарна цепи ДНК, из которой она произошла. [ 6 ] Фермент РНК-полимераза II прикрепляется к матричной цепи ДНК и катализирует добавление рибонуклеотидов к 3'-концу растущей последовательности транскрипта мРНК. [ 9 ]
Чтобы инициировать свою функцию, РНК-полимераза II должна распознать последовательность промотора , расположенную выше (5') гена. У эукариот этот процесс опосредован факторами транскрипции , в первую очередь фактором транскрипции II D (TFIID), который распознает ТАТА-бокс и помогает позиционировать РНК-полимеразу в соответствующем стартовом сайте. Чтобы завершить образование транскрипта РНК, обычно происходит терминация на расстоянии нескольких сотен нуклеотидов от терминирующей последовательности и происходит расщепление. [ 9 ] Этот процесс происходит в ядре клетки вместе с процессингом РНК, посредством которого молекулы мРНК кэпируются , сплайсируются и полиаденилируются для повышения их стабильности перед последующим попаданием в цитоплазму. МРНК дает начало белкам в процессе трансляции , происходящей в рибосомах .
Типы транскриптов РНК
[ редактировать ]Почти все функциональные транскрипты происходят от известных генов. Единственным исключением является небольшое количество транскриптов, которые могут играть непосредственную роль в регуляции экспрессии генов вблизи суфлеров известных генов. (См. Энхансерная РНК .)
Гены занимают большую часть геномов прокариот, поэтому большая часть их геномов транскрибируется. Геномы многих эукариот очень велики, и известные гены могут занимать лишь часть генома. Например, у млекопитающих известные гены составляют лишь 40–50% генома. [ 10 ] Тем не менее, идентифицированные транскрипты часто картируются на гораздо большей части генома, что позволяет предположить, что транскриптом содержит ложные транскрипты, не происходящие от генов. Известно, что некоторые из этих транскриптов нефункциональны, поскольку они картируются с транскрибируемыми псевдогенами или дегенеративными транспозонами и вирусами. Другие соответствуют неопознанным участкам генома, которые могут быть мусорной ДНК.
Ложная транскрипция очень распространена у эукариот, особенно у тех, у кого большие геномы, которые могут содержать много мусорной ДНК . [ 11 ] [ 12 ] [ 13 ] [ 14 ] Некоторые ученые утверждают, что если транскрипт не был присвоен известному гену, то по умолчанию должно быть предположение, что это мусорная РНК, пока не будет доказано, что она функциональна. [ 11 ] [ 15 ] Это означало бы, что большая часть транскриптома у видов с большими геномами, вероятно, представляет собой мусорную РНК. (См. Некодирующая РНК )
Транскриптом включает в себя транскрипты генов, кодирующих белок (мРНК плюс интроны), а также транскрипты некодирующих генов (функциональные РНК плюс интроны).
- Рибосомальная РНК /рРНК: обычно самая распространенная РНК в транскриптоме.
- Длинная некодирующая РНК /днРНК: транскрипты некодирующей РНК длиной более 200 нуклеотидов. Члены этой группы составляют наибольшую часть некодирующего транскриптома, кроме интронов. Неизвестно, сколько из этих транскриптов являются функциональными, а сколько — мусорными РНК.
- транспортная РНК /тРНК
- микроРНК /миРНК: длина 19-24 нуклеотида (нт). МикроРНК повышают или понижают уровень экспрессии мРНК посредством процесса РНК-интерференции на посттранскрипционном уровне. [ 3 ]
- малая интерферирующая РНК /миРНК: 20-24 нт.
- малая ядрышковая РНК /мяРНК
- Piwi-взаимодействующая РНК /piRNA: 24-31 нт. Они взаимодействуют с белками Piwi семейства Argonaute и выполняют функцию нацеливания и расщепления транспозонов . [ 16 ]
- энхансерная РНК /еРНК: [ 3 ]
Объем исследования
[ редактировать ]В геноме человека все гены транскрибируются в РНК, потому что именно так определяется молекулярный ген. (См. Ген .) Транскриптом состоит из кодирующих областей мРНК плюс некодирующие НТО, интроны, некодирующие РНК и ложные нефункциональные транскрипты.
Несколько факторов затрудняют определение содержания транскриптома. К ним относятся , среди прочего, альтернативный сплайсинг , редактирование РНК и альтернативная транскрипция. [ 17 ] Кроме того, методы транскриптома способны фиксировать транскрипцию, происходящую в образце в определенный момент времени, хотя содержание транскриптома может меняться во время дифференцировки. [ 6 ] Основными целями транскриптомики являются следующие: «каталогизировать все виды транскриптов, включая мРНК, некодирующие РНК и малые РНК; определить транскрипционную структуру генов с точки зрения их стартовых сайтов, 5'- и 3'-концов, сплайсинга паттерны и другие посттранскрипционные модификации, а также количественно оценить изменяющиеся уровни экспрессии каждого транскрипта во время развития и в различных условиях». [ 1 ]
Этот термин может применяться к общему набору транскриптов в данном организме или к определенному подмножеству транскриптов, присутствующих в определенном типе клеток. В отличие от генома , который примерно фиксирован для данной клеточной линии (исключая мутации ), транскриптом может меняться в зависимости от внешних условий среды. Поскольку он включает в себя все транскрипты мРНК в клетке, транскриптом отражает гены , которые активно экспрессируются в любой момент времени, за исключением явлений деградации мРНК, таких как ослабление транскрипции . Исследование транскриптомики (которое включает в себя профилирование экспрессии , анализ вариантов сплайсинга и т. д.) изучает уровень экспрессии РНК в данной клеточной популяции, часто сосредотачиваясь на мРНК, но иногда включая и другие, такие как тРНК и мРНК.
Методы строительства
[ редактировать ]Транскриптомика — это количественная наука, которая включает в себя присвоение списка строк («чтений») объекту («транскриптам» в геноме). Для расчета силы экспрессии подсчитывается плотность прочтений, соответствующих каждому объекту. [ 18 ] Первоначально транскриптомы анализировали и изучали с использованием библиотек экспрессированных меток последовательностей , а также серийного и кэп-анализа экспрессии генов (SAGE).
В настоящее время два основных метода транскриптомики включают микрочипы ДНК и RNA-Seq . Оба метода требуют выделения РНК с помощью методов экстракции РНК с последующим ее отделением от других клеточных компонентов и обогащением мРНК. [ 19 ] [ 20 ]
Существует два основных метода определения последовательностей транскриптома. Один подход сопоставляет считывание последовательности с эталонным геномом либо самого организма (транскриптом которого изучается), либо близкородственного вида. Другой подход, de novo сборка транскриптома , использует программное обеспечение для вывода транскриптов непосредственно из считывания коротких последовательностей и используется в организмах с геномами, которые не секвенированы. [ 21 ]
ДНК-микрочипы
[ редактировать ]
Первые исследования транскриптома были основаны на методах микрочипов (также известных как ДНК-чипы). Микрочипы состоят из тонких стеклянных слоев с пятнами, на которых олигонуклеотиды расположены , известные как «зонды»; каждое пятно содержит известную последовательность ДНК. [ 22 ]
При проведении микрочипового анализа мРНК собирают из контрольного и экспериментального образца, последний обычно представляет заболевание. Интересующую РНК преобразуют в кДНК для повышения ее стабильности и маркируют флуорофорами двух цветов, обычно зеленого и красного, для двух групп. кДНК наносится на поверхность микрочипа, где она гибридизуется с олигонуклеотидами на чипе, а для сканирования используется лазер. Интенсивность флуоресценции в каждом пятне микрочипа соответствует уровню экспрессии генов, и по цвету выбранных флуорофоров можно определить, какой из образцов демонстрирует более высокие уровни интересующей мРНК. [ 7 ]
Один микрочип обычно содержит достаточно олигонуклеотидов, чтобы представить все известные гены; однако данные, полученные с помощью микрочипов, не дают информации о неизвестных генах. В 2010-е годы микрочипы были почти полностью заменены методами нового поколения, основанными на секвенировании ДНК.
секвенирование РНК
[ редактировать ]Секвенирование РНК — это технология секвенирования нового поколения ; как таковой для него требуется лишь небольшое количество РНК и никаких предварительных знаний о геноме. [ 3 ] Он позволяет проводить как качественный, так и количественный анализ транскриптов РНК: первый позволяет обнаруживать новые транскрипты, а второй позволяет измерить относительное количество транскриптов в образце. [ 16 ]
Три основных этапа секвенирования транскриптомов любых биологических образцов включают очистку РНК, синтез библиотеки РНК или кДНК и секвенирование библиотеки. [ 16 ] Процесс очистки РНК различен для коротких и длинных РНК. [ 16 ] За этим шагом обычно следует оценка качества РНК, чтобы избежать таких загрязнений, как ДНК, или технических загрязнений, связанных с обработкой образцов. Качество РНК измеряют с помощью УФ-спектрометрии с пиком поглощения 260 нм. [ 23 ] Целостность РНК также можно анализировать количественно, сравнивая соотношение и интенсивность 28S РНК и 18S РНК, указанные в шкале целостности РНК (RIN). [ 23 ] Поскольку мРНК представляет собой вид, представляющий интерес, и она составляет только 3% от ее общего содержания, образец РНК следует обработать для удаления рРНК и тРНК, а также тканеспецифичных транскриптов РНК. [ 23 ]
Этап подготовки библиотеки с целью получения коротких фрагментов кДНК начинается с фрагментации РНК до транскриптов длиной от 50 до 300 пар оснований . Фрагментация может быть ферментативной ( эндонуклеазы РНК ), химической (буфер на основе трисмагниевой соли, химический гидролиз ) или механической ( обработка ультразвуком , распыление). [ 24 ] Обратная транскрипция используется для преобразования матриц РНК в кДНК, и для этого можно использовать три метода прайминга, включая олиго-DT, использование случайных праймеров или лигирование специальных адаптерных олигонуклеотидов.
Транскриптомика одноклеточных клеток
[ редактировать ]Транскрипцию также можно изучать на уровне отдельных клеток с помощью одноклеточной транскриптомики . Секвенирование одноклеточной РНК (scRNA-seq) — это недавно разработанный метод, позволяющий анализировать транскриптом отдельных клеток, включая бактерии . [ 25 ] При транскриптомике отдельных клеток также принимаются во внимание субпопуляции типов клеток, составляющих интересующую ткань. [ 26 ] Этот подход позволяет определить, связаны ли изменения в экспериментальных образцах с фенотипическими клеточными изменениями, а не с пролиферацией, при которой определенный тип клеток может сверхэкспрессироваться в образце. [ 27 ] Кроме того, при оценке клеточной прогрессии посредством дифференцировки средние профили экспрессии способны упорядочить клетки только по времени, а не по стадии их развития, и, следовательно, не могут показать тенденции в уровнях экспрессии генов, специфичные для определенных стадий. [ 28 ] Транскриптомные методы отдельных клеток использовались для характеристики популяций редких клеток, таких как циркулирующие опухолевые клетки , раковые стволовые клетки в солидных опухолях и эмбриональные стволовые клетки (ЭСК) в бластоцистах млекопитающих . [ 29 ]
Хотя не существует стандартизированных методов транскриптомики отдельных клеток, необходимо предпринять несколько шагов. Первый шаг включает изоляцию клеток, которую можно выполнить с использованием методов с низкой и высокой пропускной способностью. За этим следует этап qPCR, а затем одноклеточная RNAseq, где интересующая РНК преобразуется в кДНК. Новые разработки в области транскриптомики одиночных клеток позволяют сохранять тканевую и субклеточную локализацию посредством криодеструкции тонких срезов тканей и секвенирования транскриптома в каждом срезе. Другой метод позволяет визуализировать отдельные транскрипты под микроскопом, сохраняя при этом пространственную информацию каждой отдельной клетки, в которой они экспрессируются. [ 29 ]
Анализ
[ редактировать ]Был создан и аннотирован ряд баз данных транскриптомов, специфичных для организма, чтобы помочь в идентификации генов, которые дифференциально экспрессируются в различных популяциях клеток.
РНК-секвенирование становится (2013 г.) методом выбора для измерения транскриптомов организмов, хотя более старый метод микрочипов ДНК все еще используется. [ 1 ] RNA-seq измеряет транскрипцию конкретного гена путем преобразования длинных РНК в библиотеку фрагментов кДНК . Затем фрагменты кДНК секвенируются с использованием технологии высокопроизводительного секвенирования и выравниваются с эталонным геномом или транскриптомом, который затем используется для создания профиля экспрессии генов. [ 1 ]
Приложения
[ редактировать ]Млекопитающие
[ редактировать ]Транскриптомы стволовых и раковых клеток представляют особый интерес для исследователей, стремящихся понять процессы клеточной дифференцировки и канцерогенеза . Конвейер, использующий данные РНК-секвенирования или массива генов, может использоваться для отслеживания генетических изменений, происходящих в стволовых клетках и клетках-предшественниках , и требует как минимум трех независимых данных об экспрессии генов из первого типа клеток и зрелых клеток. [ 30 ]
Анализ транскриптомов ооцитов и эмбрионов человека используется для понимания молекулярных механизмов и сигнальных путей, контролирующих раннее эмбриональное развитие, и теоретически может стать мощным инструментом для правильного отбора эмбрионов при экстракорпоральном оплодотворении . [ нужна ссылка ] Анализ содержания транскриптома плаценты в первом триместре беременности при экстракорпоральном оплодотворении и переносе эмбрионов (ИВТ-ЭТ) выявил различия в генетической экспрессии, которые связаны с более высокой частотой неблагоприятных перинатальных исходов. Такое понимание можно использовать для оптимизации практики. [ 31 ] Транскриптомный анализ также можно использовать для оптимизации криоконсервации ооцитов за счет снижения травм, связанных с этим процессом. [ 32 ]
Транскриптомика – это новая и постоянно развивающаяся область поиска биомаркеров , которые можно использовать при оценке безопасности лекарств или оценке химического риска . [ 33 ]
Транскриптомы также можно использовать для вывода о филогенетических отношениях между людьми или для обнаружения эволюционных закономерностей сохранения транскриптома. [ 34 ]
Анализ транскриптома использовался для выявления частоты антисмысловой транскрипции, их роли в экспрессии генов посредством взаимодействия с окружающими генами и их распространенности в различных хромосомах. [ 35 ] RNA-seq также использовался, чтобы показать, как изоформы РНК, транскрипты, происходящие от одного и того же гена, но с разной структурой, могут создавать сложные фенотипы из ограниченных геномов. [ 21 ]
Растения
[ редактировать ]Транскриптомный анализ использовался для изучения процесса эволюции и разнообразия видов растений. В 2014 году был завершен проект «1000 геномов растений» , в рамках которого были секвенированы транскриптомы 1124 видов растений из семейств viridiplantae , glaucophyta и rhodophyta . В дальнейшем кодирующие белки последовательности сравнивали, чтобы сделать вывод о филогенетических отношениях между растениями и охарактеризовать время их диверсификации в процессе эволюции. [ 36 ] Исследования транскриптома использовались для характеристики и количественной оценки экспрессии генов в зрелой пыльце . Было обнаружено, что гены, участвующие в метаболизме клеточной стенки и цитоскелете, сверхэкспрессируются. Транскриптомные подходы также позволили отслеживать изменения в экспрессии генов на разных стадиях развития пыльцы, от микроспор до зрелых пыльцевых зерен; кроме того, такие стадии можно было бы сравнить у разных видов растений, включая арабидопсис , рис и табак . [ 37 ]
Связь с другими ome-полями
[ редактировать ]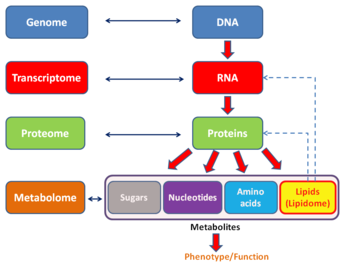
Подобно другим технологиям, основанным на -оме , анализ транскриптома позволяет использовать беспристрастный подход при экспериментальной проверке гипотез. Этот подход также позволяет открыть новые медиаторы в сигнальных путях. [ 18 ] Как и в случае с другими технологиями, основанными на -омике, транскриптом можно анализировать в рамках подхода мультиомики . Он дополняет метаболомику, прямую связь между транскриптом и метаболитом но, в отличие от протеомики, не может установить .
Существует несколько полей -ome, которые можно рассматривать как подкатегории транскриптома. Экзом . отличается от транскриптома тем, что он включает только те молекулы РНК, обнаруженные в определенной популяции клеток, и обычно включает количество или концентрацию каждой молекулы РНК в дополнение к молекулярным особенностям Кроме того, транскритпома также отличается от транслатома , который представляет собой набор РНК, подвергающихся трансляции.
Термин мейом используется в функциональной геномике для описания мейотического транскриптома или набора транскриптов РНК, образующихся в процессе мейоза . [ 38 ] Мейоз является ключевой особенностью эукариот , размножающихся половым путем , и включает в себя спаривание гомологичных хромосом , синапсов и рекомбинацию. Поскольку мейоз у большинства организмов происходит в течение короткого периода времени, профилирование мейотических транскриптов затруднено из-за проблемы выделения (или обогащения) мейотических клеток ( мейоцитов ). Как и в случае с анализом транскриптома, мейом можно изучать на уровне всего генома с использованием крупномасштабных транскриптомных методов. [ 39 ] Мейом хорошо охарактеризован у млекопитающих и дрожжевых систем и несколько менее подробно охарактеризован у растений. [ 40 ]
Танатотранскриптом состоит из всех транскриптов РНК , которые продолжают экспрессироваться или начинают реэкспрессироваться во внутренних органах мертвого тела через 24–48 часов после смерти. Некоторые гены включают те, которые ингибируются после развития плода . Если танатотранскриптом связан с процессом запрограммированной гибели клеток ( апоптоз ), его можно назвать апоптотическим танатотранскриптомом. Анализы танатотранскриптома используются в судебной медицине . [ 41 ]
Картирование eQTL можно использовать для дополнения геномики транскриптомикой; генетические варианты на уровне ДНК и меры экспрессии генов на уровне РНК. [ 42 ]
Связь с протеомом
[ редактировать ]Транскриптом можно рассматривать как подмножество протеома , то есть весь набор белков, экспрессируемых геномом.
Однако анализ относительных уровней экспрессии мРНК может быть осложнен тем фактом, что относительно небольшие изменения экспрессии мРНК могут вызывать большие изменения общего количества соответствующего белка, присутствующего в клетке. Один из методов анализа, известный как анализ обогащения набора генов , идентифицирует корегулируемые генные сети, а не отдельные гены, активность которых повышена или понижена в различных клеточных популяциях. [1]
Хотя исследования на микрочипах могут выявить относительное количество различных мРНК в клетке, уровни мРНК не прямо пропорциональны уровню экспрессии белков, которые они кодируют. [ 43 ] Количество белковых молекул, синтезируемых с использованием данной молекулы мРНК в качестве матрицы, сильно зависит от особенностей инициации трансляции последовательности мРНК; в частности, способность последовательности инициации трансляции является ключевым фактором, определяющим рекрутирование рибосом для трансляции белка .
Базы данных транскриптомов
[ редактировать ]См. также
[ редактировать ]Примечания
[ редактировать ]- ^ Jump up to: а б с д Ван, Чжун; Герштейн, Марк; Снайдер, Майкл (январь 2009 г.). «RNA-Seq: революционный инструмент для транскриптомики» . Обзоры природы Генетика . 10 (1): 57–63. дои : 10.1038/nrg2484 . ПМЦ 2949280 . ПМИД 19015660 .
- ^ Кавиччиоли, Мария Виттория; Санторсола, Мариангела; Бальбони, Никола; Меркателли, Даниэле; Джорджи, Федерико Мануэль (январь 2022 г.). «Прогнозирование метаболических профилей на основе данных транскриптомики в линиях раковых клеток человека» . Международный журнал молекулярных наук . 23 (7): 3867. doi : 10.3390/ijms23073867 . ISSN 1422-0067 . ПМЦ 8998886 . ПМИД 35409231 .
- ^ Jump up to: а б с д и Хименес-Чильярон, Хосеп К.; Диас, Рубен; Рамон-Крауэль, Марта (2014). «Глава 4 — Инструменты Omics для полногеномного анализа метилирования и модификаций гистонов» . Комплексная аналитическая химия . 64 : 81–110. дои : 10.1016/B978-0-444-62651-6.00004-0 . ISBN 9780444626516 . Проверено 25 апреля 2020 г.
- ^ ГК, Сим; ФК, Кафатос; CW, Джонс; доктор медицинских наук, Келер; А, Эфстратиадис; Т., Маниатис (декабрь 1979 г.). «Использование библиотеки кДНК для исследований эволюции и экспрессии в развитии мультигенных семейств хориона» . Клетка . 8 (4): 1303–16. дои : 10.1016/0092-8674(79)90241-1 . ПМИД 519770 .
- ^ Э Велкулеску, Виктор; Чжан, Линь; Чжоу, Вэй; Фогельштейн, Джейкоб; А Басрай, Мунира; Э. Бассетт-младший, Дуглас; Хитер, Фил; Фогельштейн, Берт; В. Кинцлер, Кеннет (1997). «Характеристика дрожжевого транскриптома» . Ячейка 2 (88): 243–51. дои : 10.1016/S0092-8674(00) 81845-0 ПМИД 9008165 . S2CID 11430660 .
- ^ Jump up to: а б с Перальта, Михаэла (2012). «Человеческий транскриптом: незаконченная история» . Гены . 3 (3): 344–360. дои : 10.3390/genes3030344 . ПМЦ 3422666 . ПМИД 22916334 .
- ^ Jump up to: а б Говиндараджан, Раджешвар; Дурайян, Джияпрадха; Калияппан, Карунакаран; Паланисами, Муругесан (2012). «Микрочип и его применение» . Журнал фармации и биологических наук . 4 (6): С310-2. дои : 10.4103/0975-7406.100283 . ПМЦ 3467903 . ПМИД 23066278 .
- ^ Браун, Т.А. (2018). «Глава 12: Транскриптомика». Геномы 4 . Нью-Йорк, штат Нью-Йорк, США: Garland Science. ISBN 9780815345084 .
- ^ Jump up to: а б Клэнси, Сюзанна (2008). «Транскрипция ДНК» . Природное образование . 1 (11): 41.
- ^ Фрэнсис В.Р., Верхайде Г. (июнь 2017 г.). «Аналогичные соотношения интронов и межгенной последовательности в геномах животных» . Геномная биология и эволюция . 9 (6): 1582–1598. дои : 10.1093/gbe/evx103 . ПМЦ 5534336 . ПМИД 28633296 .
- ^ Jump up to: а б ван Бакель Х., Нислоу С., Бленкоу Б.Дж. и Хьюз Т.Р. (2011). «Ответ на «реальность повсеместной транскрипции» . PLOS Biology . 9 (7): e1001102. : 10.1371 /journal.pbio.1001102 . PMC 3134445. . S2CID 15680321 doi
- ^ Дженсен Т.Х., Жакье А. и Либри Д. (2013). «Работа с повсеместной транскрипцией» . Молекулярная клетка . 52 (4): 473–484. дои : 10.1016/j.molcel.2013.10.032 . ПМИД 24267449 .
- ^ Свердлов, Евгений (2017). «Переписанный мусор остается мусором, если он не приобрел выбранную функцию в эволюции». Биоэссе . 39 (12): 1700164. doi : 10.1002/bies.201700164 . ПМИД 29071727 . S2CID 35346807 .
- ^ Уэйд Дж.Т. и Грейнджер, округ Колумбия (2018). «Ложная транскрипция и ее влияние на функцию клеток» . Транскрипция . 9 (3): 182–189. дои : 10.1080/21541264.2017.1381794 . ПМК 5927700 . ПМИД 28980880 .
- ^ Палаццо AF и Ли ES (2015). «Некодирующая РНК: что функционально, а что мусор?» . Границы генетики . 6 :2. дои : 10.3389/fgene.2015.00002 . ПМК 4306305 . ПМИД 25674102 .
- ^ Jump up to: а б с д Челлерино и Кровь 2018 , с. 12
- ^ У. Адамс, Джилл (2008). «Транскриптом: связь генома с функцией гена» . Природное образование . 1 (1): 195.
- ^ Jump up to: а б Челлерино и Кровь 2018 , с. предисловие
- ^ Брайант С., Мэннинг Д.Л. (1998). «Выделение информационной РНК». Протоколы выделения и характеристики РНК . Методы молекулярной биологии. Том. 86. стр. 61–4. дои : 10.1385/0-89603-494-1:61 . ISBN 978-0-89603-494-5 . ПМИД 9664454 .
- ^ Хомчинский П., Сакки Н. (апрель 1987 г.). «Одностадийный метод выделения РНК путем кислой экстракции тиоцианатом гуанидиния, фенола и хлороформа». Аналитическая биохимия . 162 (1): 156–9. дои : 10.1016/0003-2697(87)90021-2 . ПМИД 2440339 .
- ^ Jump up to: а б Тачибана, Крис (31 июля 2015 г.). «Транскриптомика сегодня: микрочипы, секвенирование РНК и многое другое» . Научный журнал . 349 (6247): 544. Бибкод : 2015Sci...349..544T . Проверено 2 мая 2020 г.
- ^ Шена, М.; Шалон, Д.; Дэвис, RW; Браун, ПО (20 октября 1995 г.). «Количественный мониторинг закономерностей экспрессии генов с помощью комплементарного микрочипа ДНК». Наука . 270 (5235). Нью-Йорк, штат Нью-Йорк: 467–470. Бибкод : 1995Sci...270..467S . дои : 10.1126/science.270.5235.467 . ISSN 0036-8075 . ПМИД 7569999 . S2CID 6720459 .
- ^ Jump up to: а б с Челлерино и Кровь 2018 , с. 13
- ^ Челлерино и Сангуанини 2018 , с. 18
- ^ Толедо-Арана А, Ласа I (март 2020 г.). «Достижения в понимании бактериального транскриптома: от перекрывающейся транскрипции к концепции исключения» . Мол Микробиол . 113 (3): 593–602. дои : 10.1111/mmi.14456 . ПМЦ 7154746 . ПМИД 32185833 .
- ^ Кантер, Итамар; Калиски, Томер (10 марта 2015 г.). «Одноклеточная транскриптомика: методы и приложения» . Границы онкологии . 5 : 53. doi : 10.3389/fonc.2015.00053 . ISSN 2234-943X . ПМЦ 4354386 . ПМИД 25806353 .
- ^ Стегл, Оливер; А. Тейхманн, Сара; К. Мариони, Джон (2015). «Вычислительные и аналитические задачи транскриптомики одиночных клеток» . Обзоры природы Генетика . 16 (3): 133–45. дои : 10.1038/nrg3833 . ПМИД 25628217 . S2CID 205486032 .
- ^ Трапнелл, Коул (1 октября 2015 г.). «Определение типов и состояний клеток с помощью геномики отдельных клеток» . Геномные исследования . 25 (10): 1491–1498. дои : 10.1101/гр.190595.115 . ISSN 1088-9051 . ПМЦ 4579334 . ПМИД 26430159 .
- ^ Jump up to: а б Кантер, Итамар; Калиски, Томер (2015). «Одноклеточная транскриптомика: методы и приложения» . Границы онкологии . 5 (13): 53. doi : 10.3389/fonc.2015.00053 . ПМЦ 4354386 . ПМИД 25806353 .
- ^ Годой, Патрисио; Шмидт-Хек, Вольфганг; Хельвиг, Бирте; Нелл, Патрик; Фейерборн, Дэвид; Раненфюрер Йорг; Каттлер, Кэтрин; Вальтер, Йорн; Блютген, Нильс; Г. Хенгстлер, Ян (5 июля 2018 г.). «Оценка дифференцировки стволовых клеток на основе полногеномных профилей экспрессии» . Философские труды Королевского общества Б. 373 (1750): 20170221. doi : 10.1098/rstb.2017.0221 . ПМЦ 5974444 . ПМИД 29786556 .
- ^ Чжао, Л; Чжэн, X; Лю, Дж; Чжэн, Р; Ян, Р; Ван, Ю; Вс, Л (1 июля 2019 г.). «На плацентарный транскриптом плаценты первого триместра влияет экстракорпоральное оплодотворение и перенос эмбрионов» . Репродуктивная биология и эндокринология . 17 (1): 50. дои : 10.1186/s12958-019-0494-7 . ПМК 6604150 . ПМИД 31262321 .
- ^ Эроглу, Биннур; А. Шурек, Эдита; Шалл, Питер; Э. Лэтэм, Кейт; Эроглу, Али (6 апреля 2020 г.). «Изучение стойких криоповреждений транскриптома ооцитов-эмбрионов» . ПЛОС ОДИН . 15 (4): e0231108. Бибкод : 2020PLoSO..1531108E . дои : 10.1371/journal.pone.0231108 . ПМК 7135251 . ПМИД 32251418 .
- ^ Сабо, Дэвид (2014). «Транскриптомные биомаркеры в оценке безопасности и риска химических веществ». Транскриптомные биомаркеры в оценке безопасности и риска химических веществ. В Рамеше Гупте, редакторы: Гупта - Биомаркеры в токсикологии, Оксфорд: Академическая пресса . стр. 1033–1038. дои : 10.1016/B978-0-12-404630-6.00062-2 . ISBN 978-0-12-404630-6 . S2CID 89396307 .
- ^ Дрост, Хайк-Георг; Габель, Александр; Гросс, Иво; Квинт, Марсель; Гроссе, Иво (01 мая 2018 г.). «myTAI: эволюционная транскриптомика с R» . Биоинформатика . 34 (9): 1589–1590. дои : 10.1093/molbev/msv012 . ISSN 0737-4038 . ПМЦ 5925770 . ПМИД 29309527 .
- ^ С, Катаяма; и др. (2005). «Антисмысловая транскрипция в транскриптоме млекопитающих» . Наука . 309 (5740): 1564–6. Бибкод : 2005Sci...309.1564R . дои : 10.1126/science.1112009 . ПМИД 16141073 . S2CID 34559885 .
- ^ Инициатива «Тысяча транскриптомов растений» (23 октября 2019 г.). «Тысяча растительных транскриптомов и филогеномика зеленых растений» . Природа . 574 (7780): 679–685. дои : 10.1038/s41586-019-1693-2 . ПМК 6872490 . ПМИД 31645766 .
- ^ Ратли, Николас; Твелл, Дэвид (12 марта 2015 г.). «Десятилетие транскриптомики пыльцы» . Размножение растений . 28 (2): 73–89. дои : 10.1007/s00497-015-0261-7 . ПМЦ 4432081 . ПМИД 25761645 .
- ^ Крисмани, Уэйн; Бауманн, Юте; Саттон, Тим; Ширли, Нил; Вебстер, Трейси; Шпангенберг, немец; Лэнгридж, Питер; Способный, Джейсон (2006). «Микрочиповый анализ экспрессии мейоза и микроспорогенеза у гексаплоидной мягкой пшеницы» . БМК Геномика . 7 (267): 267. дои : 10.1186/1471-2164-7-267 . ПМЦ 1647286 . ПМИД 17052357 .
- ^ Д. Бовилл, Уильям; Девешвар, Приянка; Капур, Санджай; А. Эйбл, Джейсон (2009). «Полногеномные подходы к идентификации кандидатов в гены раннего мейоза в злаках». Функциональная и интегративная геномика . 9 (2): 219–29. дои : 10.1007/s10142-008-0097-4 . ПМИД 18836753 . S2CID 22854431 .
- ^ Девешвар, Приянка; Д. Бовилл, Уильям; Шарма, Рита; Способный, Джейсон; Капур, Санджай (9 мая 2011 г.). «Анализ транскриптомов пыльников для выявления генов, способствующих мейозу и развитию мужских гаметофитов риса» . Биология растений BMC . 11 (78): 78. дои : 10.1186/1471-2229-11-78 . ПМК 3112077 . ПМИД 21554676 .
- ^ Джаван, GT; Могу, И.; Финли, С.Дж.; Сони, С (2015). «Апоптозный танатотранскриптом, связанный с печенью трупов». Судебная медицина, медицина и патология . 11 (4): 509–516. дои : 10.1007/s12024-015-9704-6 . ПМИД 26318598 . S2CID 21583165 .
- ^ Манцони, Клаудия; Киа, Демис; Вандровцова, Яна; Харди, Джон; Вуд, Николас; Льюис, Патрик; Феррари, Рафаэле (март 2018 г.). «Геном, транскриптом и протеом: появление данных омики и их интеграция в биомедицинские науки» . Брифинги по биоинформатике . 19 (2): 286–302. дои : 10.1093/нагрудник/bbw114 . ПМК 6018996 . ПМИД 27881428 .
- ^ Шванхойссер, Бьёрн; и др. (май 2011 г.). «Глобальная количественная оценка контроля экспрессии генов млекопитающих» (PDF) . Природа . 473 (7347): 337–342. Бибкод : 2011Natur.473..337S . дои : 10.1038/nature10098 . ПМИД 21593866 . S2CID 205224972 .
Ссылки
[ редактировать ]- Челлерино, А; Сангуанини, М. (2018), Транскриптомный анализ: введение и примеры из нейронаук , doi : 10.1007/978-88-7642-642-1 , ISBN 978-88-7642-641-4
Дальнейшее чтение
[ редактировать ]- ^ Субраманиан А, Тамайо П, Мута ВК, Мукерджи С, Эберт БЛ, Джилетт М.А., Паулович А, Помрой С.Л., Голуб Т.Р., Ландер Э.С., Месиров Дж.П. (2005). Анализ обогащения набора генов: основанный на знаниях подход для интерпретации профилей экспрессии в масштабе всего генома. Proc Natl Acad Sci USA 102(43):15545-50.
- ^ Лауле О, Хирш-Хоффманн М, Хруз Т, Груиссем В и П. Циммерманн. (2006)Интернет-анализ транскриптома мыши с использованием Genevestigator. БМК Биоинформатика 7:311
- ^ Ассу, С.; Бумела, И.; Хаузи, Д.; Анахори, Т.; Дешо, Х.; Де Вос, Дж.; Хамама, С. (2010). «Динамические изменения в экспрессии генов во время раннего развития эмбриона человека: от фундаментальных аспектов к клиническому применению» . Обновление репродукции человека . 17 (2): 272–290. дои : 10.1093/humupd/dmq036 . ПМК 3189516 . ПМИД 20716614 .
- ^ Огородников А; Каргаполова Ю.; Данквардт, С. (2016). «Процессинг и расширение транскриптома на 3'-конце мРНК в норме и при болезни: поиск правильного конца» . Eur J Physiol . 468 (6): 993–1012. дои : 10.1007/s00424-016-1828-3 . ПМЦ 4893057 . ПМИД 27220521 .