микроРНК

МикроРНК ( миРНК ) представляют собой небольшие одноцепочечные некодирующие молекулы РНК, содержащие от 21 до 23 нуклеотидов . [ 1 ] МикроРНК, обнаруженные в растениях, животных и некоторых вирусах, участвуют в подавлении РНК и посттранскрипционной регуляции экспрессии генов . [ 2 ] [ 3 ] микроРНК пара оснований к комплементарным последовательностям в молекулах мРНК , [ 4 ] затем заставляют молчать указанные молекулы мРНК с помощью одного или нескольких из следующих процессов: [ 1 ] [ 5 ]
- Расщепление цепи мРНК на две части.
- Дестабилизация мРНК за счет укорочения ее поли(А)-хвоста , или
- Снижение трансляции мРНК в белки.
В клетках человека и других животных микроРНК в первую очередь действуют путем дестабилизации мРНК. [ 6 ] [ 7 ]
miRNAs resemble the small interfering RNAs (siRNAs) of the RNA interference (RNAi) pathway, except miRNAs derive from regions of RNA transcripts that fold back on themselves to form short hairpins, whereas siRNAs derive from longer regions of double-stranded RNA.[2] The human genome may encode over 1900 miRNAs,[8][9] However, only about 500 human miRNAs represent bona fide miRNAs in the manually curated miRNA gene database MirGeneDB.[10]
miRNAs are abundant in many mammalian cell types[11][12] miRNAs appear to target about 60% of the genes of humans and other mammals.[13][14] Many miRNAs are evolutionarily conserved, which implies that they have important biological functions.[15][1] For example, 90 families of miRNAs have been conserved since at least the common ancestor of mammals and fish, and most of these conserved miRNAs have important functions, as shown by studies in which genes for one or more members of a family have been knocked out in mice.[1]
History
[edit]The first miRNA was discovered in the early 1990s.[16] However, miRNAs were not recognized as a distinct class of biological regulators until the early 2000s.[17][18][19][20][21] miRNA research revealed different sets of miRNAs expressed in different cell types and tissues[12][22] and multiple roles for miRNAs in plant and animal development and in many other biological processes.[23][24][25][26][27][28][29] Aberrant miRNA expression are implicated in disease states. MiRNA-based therapies are under investigation.[30][31][32][33]
The first miRNA was discovered in 1993 by a group led by Ambros and including Lee and Feinbaum. However, additional insight into its mode of action required simultaneously published work by Ruvkun's team, including Wightman and Ha.[16][34] These groups published back-to-back papers on the lin-4 gene, which was known to control the timing of C. elegans larval development by repressing the lin-14 gene. When Lee et al. isolated the lin-4 miRNA, they found that instead of producing an mRNA encoding a protein, it produced short non-coding RNAs, one of which was a ~22-nucleotide RNA that contained sequences partially complementary to multiple sequences in the 3' UTR of the lin-14 mRNA.[16] This complementarity was proposed to inhibit the translation of the lin-14 mRNA into the LIN-14 protein. At the time, the lin-4 small RNA was thought to be a nematode idiosyncrasy.
In 2000, a second small RNA was characterized: let-7 RNA, which represses lin-41 to promote a later developmental transition in C. elegans.[17] The let-7 RNA was found to be conserved in many species, leading to the suggestion that let-7 RNA and additional "small temporal RNAs" might regulate the timing of development in diverse animals, including humans.[18]
A year later, the lin-4 and let-7 RNAs were found to be part of a large class of small RNAs present in C. elegans, Drosophila and human cells.[19][20][21] The many RNAs of this class resembled the lin-4 and let-7 RNAs, except their expression patterns were usually inconsistent with a role in regulating the timing of development. This suggested that most might function in other types of regulatory pathways. At this point, researchers started using the term "microRNA" to refer to this class of small regulatory RNAs.[19][20][21]
The first human disease associated with deregulation of miRNAs was chronic lymphocytic leukemia. In this disorder, the miRNAs have a dual role working as both tumor suppressors and oncogenes.[35]
Nomenclature
[edit]Under a standard nomenclature system, names are assigned to experimentally confirmed miRNAs before publication.[36][37] The prefix "miR" is followed by a dash and a number, the latter often indicating order of naming. For example, miR-124 was named and likely discovered prior to miR-456. A capitalized "miR-" refers to the mature form of the miRNA, while the uncapitalized "mir-" refers to the pre-miRNA and the pri-miRNA.[38] The genes encoding miRNAs are also named using the same three-letter prefix according to the conventions of the organism gene nomenclature. For examples, the official miRNAs gene names in some organisms are "mir-1 in C. elegans and Drosophila, Mir1 in Rattus norvegicus and MIR25 in human.
miRNAs with nearly identical sequences except for one or two nucleotides are annotated with an additional lower case letter. For example, miR-124a is closely related to miR-124b. For example:
- hsa-miR-181a: aacauucaACgcugucggugAgu
- hsa-miR-181b: aacauucaUUgcugucggugGgu
Pre-miRNAs, pri-miRNAs and genes that lead to 100% identical mature miRNAs but that are located at different places in the genome are indicated with an additional dash-number suffix. For example, the pre-miRNAs hsa-mir-194-1 and hsa-mir-194-2 lead to an identical mature miRNA (hsa-miR-194) but are from genes located in different genome regions.
Species of origin is designated with a three-letter prefix, e.g., hsa-miR-124 is a human (Homo sapiens) miRNA and oar-miR-124 is a sheep (Ovis aries) miRNA. Other common prefixes include "v" for viral (miRNA encoded by a viral genome) and "d" for Drosophila miRNA (a fruit fly commonly studied in genetic research).
When two mature microRNAs originate from opposite arms of the same pre-miRNA and are found in roughly similar amounts, they are denoted with a -3p or -5p suffix. (In the past, this distinction was also made with "s" (sense) and "as" (antisense)). However, the mature microRNA found from one arm of the hairpin is usually much more abundant than that found from the other arm,[2] in which case, an asterisk following the name indicates the mature species found at low levels from the opposite arm of a hairpin. For example, miR-124 and miR-124* share a pre-miRNA hairpin, but much more miR-124 is found in the cell.
Targets
[edit]Plant miRNAs usually have near-perfect pairing with their mRNA targets, which induces gene repression through cleavage of the target transcripts.[23][39] In contrast, animal miRNAs are able to recognize their target mRNAs by using as few as 6–8 nucleotides (the seed region) at the 5' end of the miRNA,[13][40][41] which is not enough pairing to induce cleavage of the target mRNAs.[4] Combinatorial regulation is a feature of miRNA regulation in animals.[4][42] A given miRNA may have hundreds of different mRNA targets, and a given target might be regulated by multiple miRNAs.[14][43]
Estimates of the average number of unique messenger RNAs that are targets for repression by a typical miRNA vary, depending on the estimation method,[44] but multiple approaches show that mammalian miRNAs can have many unique targets. For example, an analysis of the miRNAs highly conserved in vertebrates shows that each has, on average, roughly 400 conserved targets.[14] Likewise, experiments show that a single miRNA species can reduce the stability of hundreds of unique messenger RNAs.[45] Other experiments show that a single miRNA species may repress the production of hundreds of proteins, but that this repression often is relatively mild (much less than 2-fold).[46][47]
Biogenesis
[edit]
As many as 40% of miRNA genes may lie in the introns or even exons of other genes.[48] These are usually, though not exclusively, found in a sense orientation,[49][50] and thus usually are regulated together with their host genes.[48][51][52]
The DNA template is not the final word on mature miRNA production: 6% of human miRNAs show RNA editing (IsomiRs), the site-specific modification of RNA sequences to yield products different from those encoded by their DNA. This increases the diversity and scope of miRNA action beyond that implicated from the genome alone.
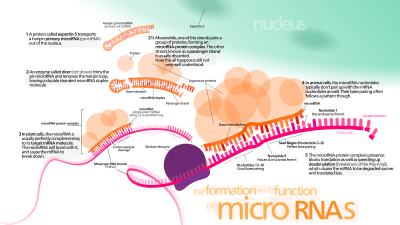
Transcription
[edit]miRNA genes are usually transcribed by RNA polymerase II (Pol II).[53][54] The polymerase often binds to a promoter found near the DNA sequence, encoding what will become the hairpin loop of the pre-miRNA. The resulting transcript is capped with a specially modified nucleotide at the 5' end, polyadenylated with multiple adenosines (a poly(A) tail),[53][49] and spliced. Animal miRNAs are initially transcribed as part of one arm of an ~80 nucleotide RNA stem-loop that in turn forms part of a several hundred nucleotide-long miRNA precursor termed a pri-miRNA.[53][49] When a stem-loop precursor is found in the 3' UTR, a transcript may serve as a pri-miRNA and a mRNA.[49] RNA polymerase III (Pol III) transcribes some miRNAs, especially those with upstream Alu sequences, transfer RNAs (tRNAs), and mammalian wide interspersed repeat (MWIR) promoter units.[55]
Nuclear processing
[edit]
A single pri-miRNA may contain from one to six miRNA precursors. These hairpin loop structures are composed of about 70 nucleotides each. Each hairpin is flanked by sequences necessary for efficient processing.
The double-stranded RNA (dsRNA) structure of the hairpins in a pri-miRNA is recognized by a nuclear protein known as DiGeorge Syndrome Critical Region 8 (DGCR8 or "Pasha" in invertebrates), named for its association with DiGeorge Syndrome. DGCR8 associates with the enzyme Drosha, a protein that cuts RNA, to form the Microprocessor complex.[56][57] In this complex, DGCR8 orients the catalytic RNase III domain of Drosha to liberate hairpins from pri-miRNAs by cleaving RNA about eleven nucleotides from the hairpin base (one helical dsRNA turn into the stem).[58][59] The product resulting has a two-nucleotide overhang at its 3' end; it has 3' hydroxyl and 5' phosphate groups. It is often termed as a pre-miRNA (precursor-miRNA). Sequence motifs downstream of the pre-miRNA that are important for efficient processing have been identified.[60][61][62]
Pre-miRNAs that are spliced directly out of introns, bypassing the Microprocessor complex, are known as "mirtrons."[63] Mirtrons have been found in Drosophila, C. elegans, and mammals.[64][65]
As many as 16% of pre-miRNAs may be altered through nuclear RNA editing.[66][67][68] Most commonly, enzymes known as adenosine deaminases acting on RNA (ADARs) catalyze adenosine to inosine (A to I) transitions. RNA editing can halt nuclear processing (for example, of pri-miR-142, leading to degradation by the ribonuclease Tudor-SN) and alter downstream processes including cytoplasmic miRNA processing and target specificity (e.g., by changing the seed region of miR-376 in the central nervous system).[66]
Nuclear export
[edit]
Pre-miRNA hairpins are exported from the nucleus in a process involving the nucleocytoplasmic shuttler Exportin-5. This protein, a member of the karyopherin family, recognizes a two-nucleotide overhang left by the RNase III enzyme Drosha at the 3' end of the pre-miRNA hairpin. Exportin-5-mediated transport to the cytoplasm is energy-dependent, using guanosine triphosphate (GTP) bound to the Ran protein.[69]
Cytoplasmic processing
[edit]In the cytoplasm, the pre-miRNA hairpin is cleaved by the RNase III enzyme Dicer.[70] This endoribonuclease interacts with 5' and 3' ends of the hairpin[71] and cuts away the loop joining the 3' and 5' arms, yielding an imperfect miRNA:miRNA* duplex about 22 nucleotides in length.[70] Overall hairpin length and loop size influence the efficiency of Dicer processing. The imperfect nature of the miRNA:miRNA* pairing also affects cleavage.[70][72] Some of the G-rich pre-miRNAs can potentially adopt the G-quadruplex structure as an alternative to the canonical stem-loop structure. For example, human pre-miRNA 92b adopts a G-quadruplex structure which is resistant to the Dicer mediated cleavage in the cytoplasm.[73] Although either strand of the duplex may potentially act as a functional miRNA, only one strand is usually incorporated into the RNA-induced silencing complex (RISC) where the miRNA and its mRNA target interact.
While the majority of miRNAs are located within the cell, some miRNAs, commonly known as circulating miRNAs or extracellular miRNAs, have also been found in extracellular environment, including various biological fluids and cell culture media.[74][75]
Biogenesis in plants
[edit]miRNA biogenesis in plants differs from animal biogenesis mainly in the steps of nuclear processing and export. Instead of being cleaved by two different enzymes, once inside and once outside the nucleus, both cleavages of the plant miRNA are performed by a Dicer homolog, called Dicer-like1 (DL1). DL1 is expressed only in the nucleus of plant cells, which indicates that both reactions take place inside the nucleus. Before plant miRNA:miRNA* duplexes are transported out of the nucleus, its 3' overhangs are methylated by a RNA methyltransferaseprotein called Hua-Enhancer1 (HEN1). The duplex is then transported out of the nucleus to the cytoplasm by a protein called Hasty (HST), an Exportin 5 homolog, where they disassemble and the mature miRNA is incorporated into the RISC.[76]
RNA-induced silencing complex
[edit]The mature miRNA is part of an active RNA-induced silencing complex (RISC) containing Dicer and many associated proteins.[77] RISC is also known as a microRNA ribonucleoprotein complex (miRNP);[78] A RISC with incorporated miRNA is sometimes referred to as a "miRISC."
Dicer processing of the pre-miRNA is thought to be coupled with unwinding of the duplex. Generally, only one strand is incorporated into the miRISC, selected on the basis of its thermodynamic instability and weaker base-pairing on the 5' end relative to the other strand.[79][80][81] The position of the stem-loop may also influence strand choice.[82] The other strand, called the passenger strand due to its lower levels in the steady state, is denoted with an asterisk (*) and is normally degraded. In some cases, both strands of the duplex are viable and become functional miRNA that target different mRNA populations.[83]
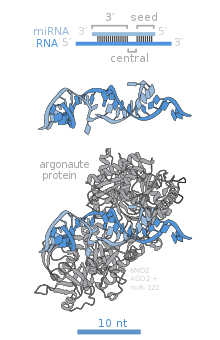
Members of the Argonaute (Ago) protein family are central to RISC function. Argonautes are needed for miRNA-induced silencing and contain two conserved RNA binding domains: a PAZ domain that can bind the single stranded 3' end of the mature miRNA and a PIWI domain that structurally resembles ribonuclease-H and functions to interact with the 5' end of the guide strand. They bind the mature miRNA and orient it for interaction with a target mRNA. Some argonautes, for example human Ago2, cleave target transcripts directly; argonautes may also recruit additional proteins to achieve translational repression.[84] The human genome encodes eight argonaute proteins divided by sequence similarities into two families: AGO (with four members present in all mammalian cells and called E1F2C/hAgo in humans), and PIWI (found in the germline and hematopoietic stem cells).[78][84]
Additional RISC components include TRBP [human immunodeficiency virus (HIV) transactivating response RNA (TAR) binding protein],[85] PACT (protein activator of the interferon-induced protein kinase), the SMN complex, fragile X mental retardation protein (FMRP), Tudor staphylococcal nuclease-domain-containing protein (Tudor-SN), the putative DNA helicase MOV10, and the RNA recognition motif containing protein TNRC6B.[69][86][87]
Mode of silencing and regulatory loops
[edit]Gene silencing may occur either via mRNA degradation or preventing mRNA from being translated. For example, miR16 contains a sequence complementary to the AU-rich element found in the 3'UTR of many unstable mRNAs, such as TNF alpha or GM-CSF.[88] It has been demonstrated that given complete complementarity between the miRNA and target mRNA sequence, Ago2 can cleave the mRNA and lead to direct mRNA degradation. In the absence of complementarity, silencing is achieved by preventing translation.[45] The relation of miRNA and its target mRNA can be based on the simple negative regulation of a target mRNA, but it seems that a common scenario is the use of a "coherent feed-forward loop", "mutual negative feedback loop" (also termed double negative loop) and "positive feedback/feed-forward loop". Some miRNAs work as buffers of random gene expression changes arising due to stochastic events in transcription, translation and protein stability. Such regulation is typically achieved by the virtue of negative feedback loops or incoherent feed-forward loop uncoupling protein output from mRNA transcription.
Turnover
[edit]Turnover of mature miRNA is needed for rapid changes in miRNA expression profiles. During miRNA maturation in the cytoplasm, uptake by the Argonaute protein is thought to stabilize the guide strand, while the opposite (* or "passenger") strand is preferentially destroyed. In what has been called a "Use it or lose it" strategy, Argonaute may preferentially retain miRNAs with many targets over miRNAs with few or no targets, leading to degradation of the non-targeting molecules.[89]
Decay of mature miRNAs in Caenorhabditis elegans is mediated by the 5'-to-3' exoribonuclease XRN2, also known as Rat1p.[90] In plants, SDN (small RNA degrading nuclease) family members degrade miRNAs in the opposite (3'-to-5') direction. Similar enzymes are encoded in animal genomes, but their roles have not been described.[89]
Several miRNA modifications affect miRNA stability. As indicated by work in the model organism Arabidopsis thaliana (thale cress), mature plant miRNAs appear to be stabilized by the addition of methyl moieties at the 3' end. The 2'-O-conjugated methyl groups block the addition of uracil (U) residues by uridyltransferase enzymes, a modification that may be associated with miRNA degradation. However, uridylation may also protect some miRNAs; the consequences of this modification are incompletely understood. Uridylation of some animal miRNAs has been reported. Both plant and animal miRNAs may be altered by addition of adenine (A) residues to the 3' end of the miRNA. An extra A added to the end of mammalian miR-122, a liver-enriched miRNA important in hepatitis C, stabilizes the molecule and plant miRNAs ending with an adenine residue have slower decay rates.[89]
Cellular functions
[edit]
The function of miRNAs appears to be in gene regulation. For that purpose, a miRNA is complementary to a part of one or more messenger RNAs (mRNAs). Animal miRNAs are usually complementary to a site in the 3' UTR whereas plant miRNAs are usually complementary to coding regions of mRNAs.[92] Perfect or near perfect base pairing with the target RNA promotes cleavage of the RNA.[93] This is the primary mode of plant miRNAs.[94] In animals the match-ups are imperfect.
For partially complementary microRNAs to recognise their targets, nucleotides 2–7 of the miRNA (its 'seed region'[13][40]) must be perfectly complementary.[95] Animal miRNAs inhibit protein translation of the target mRNA[96] (this is present but less common in plants).[94] Partially complementary microRNAs can also speed up deadenylation, causing mRNAs to be degraded sooner.[97] While degradation of miRNA-targeted mRNA is well documented, whether or not translational repression is accomplished through mRNA degradation, translational inhibition, or a combination of the two is hotly debated. Recent work on miR-430 in zebrafish, as well as on bantam-miRNA and miR-9 in Drosophila cultured cells, shows that translational repression is caused by the disruption of translation initiation, independent of mRNA deadenylation.[98][99]
miRNAs occasionally also cause histone modification and DNA methylation of promoter sites, which affects the expression of target genes.[100][101]
Nine mechanisms of miRNA action are described and assembled in a unified mathematical model:[91]
- Cap-40S initiation inhibition;
- 60S Ribosomal unit joining inhibition;
- Elongation inhibition;
- Ribosome drop-off (premature termination);
- Co-translational nascent protein degradation;
- Sequestration in P-bodies;
- mRNA decay (destabilisation);
- mRNA cleavage;
- Transcriptional inhibition through microRNA-mediated chromatin reorganization followed by gene silencing.
It is often impossible to discern these mechanisms using experimental data about stationary reaction rates. Nevertheless, they are differentiated in dynamics and have different kinetic signatures.[91]
Unlike plant microRNAs, the animal microRNAs target diverse genes.[40] However, genes involved in functions common to all cells, such as gene expression, have relatively fewer microRNA target sites and seem to be under selection to avoid targeting by microRNAs.[102] There is a strong correlation between ITPR gene regulations and mir-92 and mir-19.[103]
dsRNA can also activate gene expression, a mechanism that has been termed "small RNA-induced gene activation" or RNAa. dsRNAs targeting gene promoters can induce potent transcriptional activation of associated genes. This was demonstrated in human cells using synthetic dsRNAs termed small activating RNAs (saRNAs),[104] but has also been demonstrated for endogenous microRNA.[105]
Interactions between microRNAs and complementary sequences on genes and even pseudogenes that share sequence homology are thought to be a back channel of communication regulating expression levels between paralogous genes (genes having a similar structure indicating divergence from a common ancestral gene). Given the name "competing endogenous RNAs" (ceRNAs), these microRNAs bind to "microRNA response elements" on genes and pseudogenes and may provide another explanation for the persistence of non-coding DNA.[106]
miRNAs are also found as extracellular circulating miRNAs.[107] Circulating miRNAs are released into body fluids including blood and cerebrospinal fluid and have the potential to be available as biomarkers in a number of diseases.[107][108]Some researches show that mRNA cargo of exosomes may have a role in implantation, they can savage an adhesion between trophoblast and endometrium or support the adhesion by down regulating or up regulating expression of genes involved in adhesion/invasion.[109]
Moreover, miRNA as miR-183/96/182 seems to play a key role in circadian rhythm.[110]
Evolution
[edit]miRNAs are well conserved in both plants and animals, and are thought to be a vital and evolutionarily ancient component of gene regulation.[111][112][113][114][115] While core components of the microRNA pathway are conserved between plants and animals, miRNA repertoires in the two kingdoms appear to have emerged independently with different primary modes of action.[116][117]
microRNAs are useful phylogenetic markers because of their apparently low rate of evolution.[118] microRNAs' origin as a regulatory mechanism developed from previous RNAi machinery that was initially used as a defense against exogenous genetic material such as viruses.[119] Their origin may have permitted the development of morphological innovation, and by making gene expression more specific and 'fine-tunable', permitted the genesis of complex organs[120] and perhaps, ultimately, complex life.[115] Rapid bursts of morphological innovation are generally associated with a high rate of microRNA accumulation.[118][120]
New microRNAs are created in multiple ways. Novel microRNAs can originate from the random formation of hairpins in "non-coding" sections of DNA (i.e. introns or intergene regions), but also by the duplication and modification of existing microRNAs.[121] microRNAs can also form from inverted duplications of protein-coding sequences, which allows for the creation of a foldback hairpin structure.[122] The rate of evolution (i.e. nucleotide substitution) in recently originated microRNAs is comparable to that elsewhere in the non-coding DNA, implying evolution by neutral drift; however, older microRNAs have a much lower rate of change (often less than one substitution per hundred million years),[115] suggesting that once a microRNA gains a function, it undergoes purifying selection.[121] Individual regions within an miRNA gene face different evolutionary pressures, where regions that are vital for processing and function have higher levels of conservation.[123] At this point, a microRNA is rarely lost from an animal's genome,[115] although newer microRNAs (thus presumably non-functional) are frequently lost.[121] In Arabidopsis thaliana, the net flux of miRNA genes has been predicted to be between 1.2 and 3.3 genes per million years.[124] This makes them a valuable phylogenetic marker, and they are being looked upon as a possible solution to outstanding phylogenetic problems such as the relationships of arthropods.[125] On the other hand, in multiple cases microRNAs correlate poorly with phylogeny, and it is possible that their phylogenetic concordance largely reflects a limited sampling of microRNAs.[126]
microRNAs feature in the genomes of most eukaryotic organisms, from the brown algae[127] to the animals. However, the difference in how these microRNAs function and the way they are processed suggests that microRNAs arose independently in plants and animals.[128]
Focusing on the animals, the genome of Mnemiopsis leidyi[129] appears to lack recognizable microRNAs, as well as the nuclear proteins Drosha and Pasha, which are critical to canonical microRNA biogenesis. It is the only animal thus far reported to be missing Drosha. MicroRNAs play a vital role in the regulation of gene expression in all non-ctenophore animals investigated thus far except for Trichoplax adhaerens, the first known member of the phylum Placozoa.[130]
Across all species, in excess of 5000 different miRNAs had been identified by March 2010.[131] Whilst short RNA sequences (50 – hundreds of base pairs) of a broadly comparable function occur in bacteria, bacteria lack true microRNAs.[132]
Experimental detection and manipulation
[edit]While researchers focused on miRNA expression in physiological and pathological processes, various technical variables related to microRNA isolation emerged. The stability of stored miRNA samples has been questioned.[75] microRNAs degrade much more easily than mRNAs, partly due to their length, but also because of ubiquitously present RNases. This makes it necessary to cool samples on ice and use RNase-free equipment.[133]
microRNA expression can be quantified in a two-step polymerase chain reaction process of modified RT-PCR followed by quantitative PCR. Variations of this method achieve absolute or relative quantification.[134] miRNAs can also be hybridized to microarrays, slides or chips with probes to hundreds or thousands of miRNA targets, so that relative levels of miRNAs can be determined in different samples.[135] microRNAs can be both discovered and profiled by high-throughput sequencing methods (microRNA sequencing).[136] The activity of an miRNA can be experimentally inhibited using a locked nucleic acid (LNA) oligo, a Morpholino oligo[137][138] or a 2'-O-methyl RNA oligo.[139] A specific miRNA can be silenced by a complementary antagomir. microRNA maturation can be inhibited at several points by steric-blocking oligos.[140][141] The miRNA target site of an mRNA transcript can also be blocked by a steric-blocking oligo.[142] For the "in situ" detection of miRNA, LNA[143] or Morpholino[144] probes can be used. The locked conformation of LNA results in enhanced hybridization properties and increases sensitivity and selectivity, making it ideal for detection of short miRNA.[145]
High-throughput quantification of miRNAs is error prone, for the larger variance (compared to mRNAs) that comes with methodological problems. mRNA-expression is therefore often analyzed to check for miRNA-effects in their levels (e.g. in[146]). Databases can be used to pair mRNA- and miRNA-data that predict miRNA-targets based on their base sequence.[147][148] While this is usually done after miRNAs of interest have been detected (e. g. because of high expression levels), ideas for analysis tools that integrate mRNA- and miRNA-expression information have been proposed.[149][150]
Human and animal diseases
[edit]Just as miRNA is involved in the normal functioning of eukaryotic cells, so has dysregulation of miRNA been associated with disease. A manually curated, publicly available database, miR2Disease, documents known relationships between miRNA dysregulation and human disease.[151]
Inherited diseases
[edit]A mutation in the seed region of miR-96 causes hereditary progressive hearing loss.[152]
A mutation in the seed region of miR-184 causes hereditary keratoconus with anterior polar cataract.[153]
Deletion of the miR-17~92 cluster causes skeletal and growth defects.[154]
Cancer
[edit]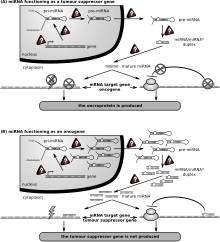
The first human disease known to be associated with miRNA deregulation was chronic lymphocytic leukemia.[155] Many other miRNAs also have links with cancer and accordingly are sometimes referred to as "oncomirs".[156] In malignant B cells miRNAs participate in pathways fundamental to B cell development like B-cell receptor (BCR) signalling, B-cell migration/adhesion, cell-cell interactions in immune niches and the production and class-switching of immunoglobulins. MiRNAs influence B cell maturation, generation of pre-, marginal zone, follicular, B1, plasma and memory B cells.[157]
Another role for miRNA in cancers is to use their expression level for prognosis. In NSCLC samples, low miR-324a levels may serve as an indicator of poor survival.[158] Either high miR-185 or low miR-133b levels may correlate with metastasis and poor survival in colorectal cancer.[159]
Furthermore, specific miRNAs may be associated with certain histological subtypes of colorectal cancer. For instance, expression levels of miR-205 and miR-373 have been shown to be increased in mucinous colorectal cancers and mucin-producing Ulcerative Colitis-associated colon cancers, but not in sporadic colonic adenocarcinoma that lack mucinous components.[160] In-vitro studies suggested that miR-205 and miR-373 may functionally induce different features of mucinous-associated neoplastic progression in intestinal epithelial cells.[160]
Hepatocellular carcinoma cell proliferation may arise from miR-21 interaction with MAP2K3, a tumor repressor gene.[161] Optimal treatment for cancer involves accurately identifying patients for risk-stratified therapy. Those with a rapid response to initial treatment may benefit from truncated treatment regimens, showing the value of accurate disease response measures. Cell-free circulating miRNAs (cimiRNAs) are highly stable in blood, are overexpressed in cancer and are quantifiable within the diagnostic laboratory. In classical Hodgkin lymphoma, plasma miR-21, miR-494, and miR-1973 are promising disease response biomarkers.[162] Circulating miRNAs have the potential to assist clinical decision making and aid interpretation of positron emission tomography combined with computerized tomography. They can be performed at each consultation to assess disease response and detect relapse.
MicroRNAs have the potential to be used as tools or targets for treatment of different cancers.[163] The specific microRNA, miR-506 has been found to work as a tumor antagonist in several studies. A significant number of cervical cancer samples were found to have downregulated expression of miR-506. Additionally, miR-506 works to promote apoptosis of cervical cancer cells, through its direct target hedgehog pathway transcription factor, Gli3.[164][165]
DNA repair and cancer
[edit]Many miRNAs can directly target and inhibit cell cycle genes to control cell proliferation. A new strategy for tumor treatment is to inhibit tumor cell proliferation by repairing the defective miRNA pathway in tumors.[166] Cancer is caused by the accumulation of mutations from either DNA damage or uncorrected errors in DNA replication.[167] Defects in DNA repair cause the accumulation of mutations, which can lead to cancer.[168] Several genes involved in DNA repair are regulated by microRNAs.[169]
Germline mutations in DNA repair genes cause only 2–5% of colon cancer cases.[170] However, altered expression of microRNAs, causing DNA repair deficiencies, are frequently associated with cancers and may be an important causal factor. Among 68 sporadic colon cancers with reduced expression of the DNA mismatch repair protein MLH1, most were found to be deficient due to epigenetic methylation of the CpG island of the MLH1 gene.[171] However, up to 15% of MLH1-deficiencies in sporadic colon cancers appeared to be due to over-expression of the microRNA miR-155, which represses MLH1 expression.[172]
In 29–66%[173][174] of glioblastomas, DNA repair is deficient due to epigenetic methylation of the MGMT gene, which reduces protein expression of MGMT. However, for 28% of glioblastomas, the MGMT protein is deficient, but the MGMT promoter is not methylated.[173] In glioblastomas without methylated MGMT promoters, the level of microRNA miR-181d is inversely correlated with protein expression of MGMT and the direct target of miR-181d is the MGMT mRNA 3'UTR (the three prime untranslated region of MGMT mRNA).[173] Thus, in 28% of glioblastomas, increased expression of miR-181d and reduced expression of DNA repair enzyme MGMT may be a causal factor.
HMGA proteins (HMGA1a, HMGA1b and HMGA2) are implicated in cancer, and expression of these proteins is regulated by microRNAs. HMGA expression is almost undetectable in differentiated adult tissues, but is elevated in many cancers. HMGA proteins are polypeptides of ~100 amino acid residues characterized by a modular sequence organization. These proteins have three highly positively charged regions, termed AT hooks, that bind the minor groove of AT-rich DNA stretches in specific regions of DNA. Human neoplasias, including thyroid, prostatic, cervical, colorectal, pancreatic and ovarian carcinomas, show a strong increase of HMGA1a and HMGA1b proteins.[175] Transgenic mice with HMGA1 targeted to lymphoid cells develop aggressive lymphoma, showing that high HMGA1 expression is associated with cancers and that HMGA1 can act as an oncogene.[176] HMGA2 protein specifically targets the promoter of ERCC1, thus reducing expression of this DNA repair gene.[177] ERCC1 protein expression was deficient in 100% of 47 evaluated colon cancers (though the extent to which HGMA2 was involved is not known).[178]
Single Nucleotide polymorphisms (SNPs) can alter the binding of miRNAs on 3'UTRs for example the case of hsa-mir181a and hsa-mir181b on the CDON tumor suppressor gene.[179]
Heart disease
[edit]The global role of miRNA function in the heart has been addressed by conditionally inhibiting miRNA maturation in the murine heart. This revealed that miRNAs play an essential role during its development.[180][181] miRNA expression profiling studies demonstrate that expression levels of specific miRNAs change in diseased human hearts, pointing to their involvement in cardiomyopathies.[182][183][184] Furthermore, animal studies on specific miRNAs identified distinct roles for miRNAs both during heart development and under pathological conditions, including the regulation of key factors important for cardiogenesis, the hypertrophic growth response and cardiac conductance.[181][185][186][187][188][189] Another role for miRNA in cardiovascular diseases is to use their expression levels for diagnosis, prognosis or risk stratification.[190] miRNA's in animal models have also been linked to cholesterol metabolism and regulation.
miRNA-712
[edit]Murine microRNA-712 is a potential biomarker (i.e. predictor) for atherosclerosis, a cardiovascular disease of the arterial wall associated with lipid retention and inflammation.[191] Non-laminar blood flow also correlates with development of atherosclerosis as mechanosenors of endothelial cells respond to the shear force of disturbed flow (d-flow).[192] A number of pro-atherogenic genes including matrix metalloproteinases (MMPs) are upregulated by d-flow,[192] mediating pro-inflammatory and pro-angiogenic signals. These findings were observed in ligated carotid arteries of mice to mimic the effects of d-flow. Within 24 hours, pre-existing immature miR-712 formed mature miR-712 suggesting that miR-712 is flow-sensitive.[192] Coinciding with these results, miR-712 is also upregulated in endothelial cells exposed to naturally occurring d-flow in the greater curvature of the aortic arch.[192]
Origin
[edit]Pre-mRNA sequence of miR-712 is generated from the murine ribosomal RN45s gene at the internal transcribed spacer region 2 (ITS2).[192] XRN1 is an exonuclease that degrades the ITS2 region during processing of RN45s.[192] Reduction of XRN1 under d-flow conditions therefore leads to the accumulation of miR-712.[192]
Mechanism
[edit]MiR-712 targets tissue inhibitor of metalloproteinases 3 (TIMP3).[192] TIMPs normally regulate activity of matrix metalloproteinases (MMPs) which degrade the extracellular matrix (ECM). Arterial ECM is mainly composed of collagen and elastin fibers, providing the structural support and recoil properties of arteries.[193] These fibers play a critical role in regulation of vascular inflammation and permeability, which are important in the development of atherosclerosis.[194] Expressed by endothelial cells, TIMP3 is the only ECM-bound TIMP.[193] A decrease in TIMP3 expression results in an increase of ECM degradation in the presence of d-flow. Consistent with these findings, inhibition of pre-miR712 increases expression of TIMP3 in cells, even when exposed to turbulent flow.[192]
TIMP3 also decreases the expression of TNFα (a pro-inflammatory regulator) during turbulent flow.[192] Activity of TNFα in turbulent flow was measured by the expression of TNFα-converting enzyme (TACE) in blood. TNFα decreased if miR-712 was inhibited or TIMP3 overexpressed,[192] suggesting that miR-712 and TIMP3 regulate TACE activity in turbulent flow conditions.
Anti-miR-712 effectively suppresses d-flow-induced miR-712 expression and increases TIMP3 expression.[192] Anti-miR-712 also inhibits vascular hyperpermeability, thereby significantly reducing atherosclerosis lesion development and immune cell infiltration.[192]
Human homolog microRNA-205
[edit]The human homolog of miR-712 was found on the RN45s homolog gene, which maintains similar miRNAs to mice.[192] MiR-205 of humans share similar sequences with miR-712 of mice and is conserved across most vertebrates.[192] MiR-205 and miR-712 also share more than 50% of the cell signaling targets, including TIMP3.[192]
When tested, d-flow decreased the expression of XRN1 in humans as it did in mice endothelial cells, indicating a potentially common role of XRN1 in humans.[192]
Kidney disease
[edit]Targeted deletion of Dicer in the FoxD1-derived renal progenitor cells in a murine model resulted in a complex renal phenotype including expansion of nephron progenitors, fewer renin cells, smooth muscle arterioles, progressive mesangial loss and glomerular aneurysms.[195] High throughput whole transcriptome profiling of the FoxD1-Dicer knockout mouse model revealed ectopic upregulation of pro-apoptotic gene, Bcl2L11 (Bim) and dysregulation of the p53 pathway with increase in p53 effector genes including Bax, Trp53inp1, Jun, Cdkn1a, Mmp2, and Arid3a. p53 protein levels remained unchanged, suggesting that FoxD1 stromal miRNAs directly repress p53-effector genes. Using a lineage tracing approach followed by Fluorescent-activated cell sorting, miRNA profiling of the FoxD1-derived cells not only comprehensively defined the transcriptional landscape of miRNAs that are critical for vascular development, but also identified key miRNAs that are likely to modulate the renal phenotype in its absence. These miRNAs include miRs‐10a, 18a, 19b, 24, 30c, 92a, 106a, 130a, 152, 181a, 214, 222, 302a, 370, and 381 that regulate Bcl2L11 (Bim) and miRs‐15b, 18a, 21, 30c, 92a, 106a, 125b‐5p, 145, 214, 222, 296‐5p and 302a that regulate p53-effector genes. Consistent with the profiling results, ectopic apoptosis was observed in the cellular derivatives of the FoxD1 derived progenitor lineage and reiterates the importance of renal stromal miRNAs in cellular homeostasis.[195]
Nervous system
[edit]MiRNAs are crucial for the healthy development and function of the nervous system.[196] Previous studies demonstrate that miRNAs can regulate neuronal differentiation and maturation at various stages.[197] MiRNAs also play important roles in synaptic development[198] (such as dendritogenesis or spine morphogenesis) and synaptic plasticity[199] (contributing to learning and memory). Elimination of miRNA formation in mice by experimental silencing of Dicer has led to pathological outcomes, such as reduced neuronal size, motor abnormalities (when silenced in striatal neurons[200]), and neurodegeneration (when silenced in forebrain neurons[201]). Altered miRNA expression has been found in neurodegenerative diseases (such as Alzheimer's disease, Parkinson's disease, and Huntington's disease[202]) as well as many psychiatric disorders (including epilepsy,[203] schizophrenia, major depression, bipolar disorder, and anxiety disorders[204][205][206]).
Stroke
[edit]According to the Center for Disease Control and Prevention, Stroke is one of the leading causes of death and long-term disability in America. 87% of the cases are ischemic strokes, which results from blockage in the artery of the brain that carries oxygen-rich blood. The obstruction of the blood flow means the brain cannot receive necessary nutrients, such as oxygen and glucose, and remove wastes, such as carbon dioxide.[207][208] miRNAs plays a role in posttranslational gene silencing by targeting genes in the pathogenesis of cerebral ischemia, such as the inflammatory, angiogenesis, and apoptotic pathway.[209]
Alcoholism
[edit]The vital role of miRNAs in gene expression is significant to addiction, specifically alcoholism.[210] Chronic alcohol abuse results in persistent changes in brain function mediated in part by alterations in gene expression.[210] miRNA global regulation of many downstream genes deems significant regarding the reorganization or synaptic connections or long term neural adaptations involving the behavioral change from alcohol consumption to withdrawal and/or dependence.[211] Up to 35 different miRNAs have been found to be altered in the alcoholic post-mortem brain, all of which target genes that include the regulation of the cell cycle, apoptosis, cell adhesion, nervous system development and cell signaling.[210] Altered miRNA levels were found in the medial prefrontal cortex of alcohol-dependent mice, suggesting the role of miRNA in orchestrating translational imbalances and the creation of differentially expressed proteins within an area of the brain where complex cognitive behavior and decision making likely originate.[212]
miRNAs can be either upregulated or downregulated in response to chronic alcohol use. miR-206 expression increased in the prefrontal cortex of alcohol-dependent rats, targeting the transcription factor brain-derived neurotrophic factor (BDNF) and ultimately reducing its expression. BDNF plays a critical role in the formation and maturation of new neurons and synapses, suggesting a possible implication in synapse growth/synaptic plasticity in alcohol abusers.[213] miR-155, important in regulating alcohol-induced neuroinflammation responses, was found to be upregulated, suggesting the role of microglia and inflammatory cytokines in alcohol pathophysiology.[214] Downregulation of miR-382 was found in the nucleus accumbens, a structure in the basal forebrain significant in regulating feelings of reward that power motivational habits. miR-382 is the target for the dopamine receptor D1 (DRD1), and its overexpression results in the upregulation of DRD1 and delta fosB, a transcription factor that activates a series of transcription events in the nucleus accumbens that ultimately result in addictive behaviors.[215] Alternatively, overexpressing miR-382 resulted in attenuated drinking and the inhibition of DRD1 and delta fosB upregulation in rat models of alcoholism, demonstrating the possibility of using miRNA-targeted pharmaceuticals in treatments.[215]
Obesity
[edit]miRNAs play crucial roles in the regulation of stem cell progenitors differentiating into adipocytes.[216] Studies to determine what role pluripotent stem cells play in adipogenesis, were examined in the immortalized human bone marrow-derived stromal cell line hMSC-Tert20.[217] Decreased expression of miR-155, miR-221, and miR-222, have been found during the adipogenic programming of both immortalized and primary hMSCs, suggesting that they act as negative regulators of differentiation. Conversely, ectopic expression of the miRNAs 155, 221, and 222 significantly inhibited adipogenesis and repressed induction of the master regulators PPARγ and CCAAT/enhancer-binding protein alpha (CEBPA).[218] This paves the way for possible genetic obesity treatments.
Another class of miRNAs that regulate insulin resistance, obesity, and diabetes, is the let-7 family. Let-7 accumulates in human tissues during the course of aging.[219] When let-7 was ectopically overexpressed to mimic accelerated aging, mice became insulin-resistant, and thus more prone to high fat diet-induced obesity and diabetes.[220] In contrast when let-7 was inhibited by injections of let-7-specific antagomirs, mice become more insulin-sensitive and remarkably resistant to high fat diet-induced obesity and diabetes. Not only could let-7 inhibition prevent obesity and diabetes, it could also reverse and cure the condition.[221] These experimental findings suggest that let-7 inhibition could represent a new therapy for obesity and type 2 diabetes.
Hemostasis
[edit]miRNAs also play crucial roles in the regulation of complex enzymatic cascades including the hemostatic blood coagulation system.[222] Large scale studies of functional miRNA targeting have recently uncovered rationale therapeutic targets in the hemostatic system.[223][224] They have been directly linked to Calcium homeostasis in the endoplasmic reticulum, which is critical in cell differentiation in early development.[225]
Plants
[edit]miRNAs are considered to be key regulators of many developmental, homeostatic, and immune processes in plants.[226] Their roles in plant development include shoot apical meristem development, leaf growth, flower formation, seed production, or root expansion.[227][228][229][230] In addition, they play a complex role in responses to various abiotic stresses comprising heat stress, low-temperature stress, drought stress, light stress, or gamma radiation exposure.[226]
Viruses
[edit]Viral microRNAs play an important role in the regulation of gene expression of viral and/or host genes to benefit the virus. Hence, miRNAs play a key role in host–virus interactions and pathogenesis of viral diseases.[231][232] The expression of transcription activators by human herpesvirus-6 DNA is believed to be regulated by viral miRNA.[233]
Target prediction
[edit]miRNAs can bind to target messenger RNA (mRNA) transcripts of protein-coding genes and negatively control their translation or cause mRNA degradation. It is of key importance to identify the miRNA targets accurately.[234] A comparison of the predictive performance of eighteen in silico algorithms is available.[235] Large scale studies of functional miRNA targeting suggest that many functional miRNAs can be missed by target prediction algorithms.[223]
See also
[edit]- Anti-miRNA oligonucleotides
- Gene expression
- List of miRNA gene prediction tools
- List of miRNA target prediction tools
- MicroDNA
- MicroRNA Biosensors
- MiRNEST
- MIR222
- miR-324-5p
- RNA interference
- Small interfering RNA
- Small nucleolar RNA-derived microRNA
- C19MC miRNA cluster
References
[edit]- ^ Перейти обратно: а б с д Бартель Д.П. (март 2018 г.). «МикроРНК многоклеточных животных» . Клетка . 173 (1): 20–51. дои : 10.1016/j.cell.2018.03.006 . ПМК 6091663 . ПМИД 29570994 .
- ^ Перейти обратно: а б с Бартель Д.П. (январь 2004 г.). «МикроРНК: геномика, биогенез, механизм и функции» . Клетка . 116 (2): 281–297. дои : 10.1016/S0092-8674(04)00045-5 . ПМИД 14744438 .
- ^ Куреши А., Тхакур Н., Монга И., Тхакур А., Кумар М. (1 января 2014 г.). «VIRmiRNA: комплексный ресурс экспериментально подтвержденных вирусных микроРНК и их мишеней» . База данных . 2014 : бау103. дои : 10.1093/база данных/bau103 . ПМК 4224276 . ПМИД 25380780 .
- ^ Перейти обратно: а б с Бартель Д.П. (январь 2009 г.). «МикроРНК: целевое распознавание и регуляторные функции» . Клетка . 136 (2): 215–33. дои : 10.1016/j.cell.2009.01.002 . ПМЦ 3794896 . ПМИД 19167326 .
- ^ Йонас С., Изаурральде Э. (июль 2015 г.). «На пути к молекулярному пониманию молчания генов, опосредованного микроРНК». Обзоры природы. Генетика . 16 (7): 421–433. дои : 10.1038/nrg3965 . ПМИД 26077373 . S2CID 24892348 .
- ^ Йонас С., Изаурральде Э. (июль 2015 г.). «На пути к молекулярному пониманию молчания генов, опосредованного микроРНК». Обзоры природы. Генетика . 16 (7): 421–433. дои : 10.1038/nrg3965 . ПМИД 26077373 . S2CID 24892348 .
- ^ Го Х, Инголия НТ, Вайсман Дж.С., Бартель Д.П. (август 2010 г.). «МикроРНК млекопитающих преимущественно снижают уровни целевой мРНК» . Природа . 466 (7308): 835–840. Бибкод : 2010Natur.466..835G . дои : 10.1038/nature09267 . hdl : 1721.1/72447 . ПМК 2990499 . ПМИД 20703300 .
- ^ МикроРНК Homo sapiens в miRBase в Манчестерском университете
- ^ Аллес Дж., Фельманн Т., Фишер У., Бакес С., Галата В., Минет М. и др. (апрель 2019 г.). «Оценка общего количества истинных микроРНК человека» . Исследования нуклеиновых кислот . 47 (7): 3353–3364. дои : 10.1093/nar/gkz097 . ПМК 6468295 . ПМИД 30820533 .
- ^ Фромм Б., Доманска Д., Хойе Е., Овчинников В., Канг В., Апарисио-Пуэрта Е. и др. (январь 2020 г.). «MirGeneDB 2.0: комплемент микроРНК многоклеточных животных» . Исследования нуклеиновых кислот . 48 (Д1): Д132–Д141. дои : 10.1093/nar/gkz885 . ПМК 6943042 . ПМИД 31598695 .
- ^ Лим Л.П., Лау Н.К., Вайнштейн Э.Г., Абдельхаким А., Йекта С., Роудс М.В. и др. (апрель 2003 г.). «МикроРНК Caenorhabditis elegans» . Гены и развитие . 17 (8): 991–1008. дои : 10.1101/gad.1074403 . ЧВК 196042 . ПМИД 12672692 .
- ^ Перейти обратно: а б Лагос-Кинтана М., Раухут Р., Ялчин А., Мейер Дж., Лендекель В., Тушль Т. (апрель 2002 г.). «Идентификация тканеспецифичных микроРНК у мышей» . Современная биология . 12 (9): 735–9. Бибкод : 2002CBio...12..735L . дои : 10.1016/S0960-9822(02)00809-6 . ПМИД 12007417 .
- ^ Перейти обратно: а б с Льюис Б.П., Бердж CB, Бартель Д.П. (январь 2005 г.). «Консервативные пары семян, часто окруженные аденозинами, указывают на то, что тысячи человеческих генов являются мишенями микроРНК» . Клетка . 120 (1): 15–20. дои : 10.1016/j.cell.2004.12.035 . ПМИД 15652477 .
- ^ Перейти обратно: а б с Фридман Р.К., Фарх К.К., Бердж CB, Бартель Д.П. (январь 2009 г.). «Большинство мРНК млекопитающих являются консервативными мишенями микроРНК» . Геномные исследования . 19 (1): 92–105. дои : 10.1101/гр.082701.108 . ПМЦ 2612969 . ПМИД 18955434 .
- ^ Фромм Б., Биллипп Т., Пек Л.Е., Йохансен М., Тарвер Дж.Э., Кинг Б.Л. и др. (2015). «Единая система аннотации генов микроРНК позвоночных и эволюция микроРНК человека» . Ежегодный обзор генетики . 49 : 213–42. doi : 10.1146/annurev-genet-120213-092023 . ПМЦ 4743252 . ПМИД 26473382 .
- ^ Перейти обратно: а б с Ли Р.К., Фейнбаум Р.Л., Амброс В. (декабрь 1993 г.). «Гетерохронный ген lin-4 C. elegans кодирует малые РНК с антисмысловой комплементарностью lin-14» . Клетка . 75 (5): 843–54. дои : 10.1016/0092-8674(93)90529-Y . ПМИД 8252621 .
- ^ Перейти обратно: а б Рейнхарт Б.Дж., Слэк Ф.Дж., Бассон М., Паскинелли А.Е., Беттингер Дж.К., Ругви А.Е. и др. (февраль 2000 г.). «21-нуклеотидная РНК let-7 регулирует сроки развития Caenorhabditis elegans». Природа . 403 (6772): 901–6. Бибкод : 2000Natur.403..901R . дои : 10.1038/35002607 . ПМИД 10706289 . S2CID 4384503 .
- ^ Перейти обратно: а б Паскинелли А.Е., Рейнхарт Б.Дж., Слэк Ф., Мартиндейл М.К., Курода М.И., Маллер Б. и др. (ноябрь 2000 г.). «Сохранение последовательности и временной экспрессии гетерохронной регуляторной РНК let-7». Природа . 408 (6808): 86–9. Бибкод : 2000Natur.408...86P . дои : 10.1038/35040556 . ПМИД 11081512 . S2CID 4401732 .
- ^ Перейти обратно: а б с Лагос-Кинтана М., Раухут Р., Лендекель В., Тушль Т. (октябрь 2001 г.). «Идентификация новых генов, кодирующих малые экспрессируемые РНК». Наука . 294 (5543): 853–8. Бибкод : 2001Sci...294..853L . дои : 10.1126/science.1064921 . hdl : 11858/00-001M-0000-0012-F65F-2 . ПМИД 11679670 . S2CID 18101169 .
- ^ Перейти обратно: а б с Лау, Северная Каролина, Лим Л.П., Вайнштейн Э.Г., Бартель Д.П. (октябрь 2001 г.). «Обильный класс крошечных РНК, играющих вероятную регуляторную роль у Caenorhabditis elegans». Наука . 294 (5543): 858–62. Бибкод : 2001Sci...294..858L . дои : 10.1126/science.1065062 . ПМИД 11679671 . S2CID 43262684 .
- ^ Перейти обратно: а б с Ли Р.К., Амброс В. (октябрь 2001 г.). «Обширный класс малых РНК Caenorhabditis elegans». Наука . 294 (5543): 862–4. Бибкод : 2001Sci...294..862L . дои : 10.1126/science.1065329 . ПМИД 11679672 . S2CID 33480585 .
- ^ Винхолдс Е., Клоостерман В.П., Миска Е., Альварес-Сааведра Е., Березиков Е., де Брёйн Е. и др. (июль 2005 г.). «Экспрессия микроРНК в эмбриональном развитии рыбок данио». Наука . 309 (5732): 310–1. Бибкод : 2005Sci...309..310W . дои : 10.1126/science.1114519 . ПМИД 15919954 . S2CID 38939571 .
- ^ Перейти обратно: а б Джонс-Роудс М.В., Бартель Д.П., Бартель Б. (2006). «МикроРНК и их регуляторная роль в растениях». Ежегодный обзор биологии растений . 57 : 19–53. doi : 10.1146/annurev.arplant.57.032905.105218 . ПМИД 16669754 .
- ^ Бреннеке Дж., Хипфнер Д.Р., Старк А., Рассел Р.Б., Коэн С.М. (апрель 2003 г.). «Бантам кодирует микроРНК, регулируемую в процессе развития, которая контролирует пролиферацию клеток и регулирует проапоптотический ген, скрытый у дрозофилы» . Клетка . 113 (1): 25–36. дои : 10.1016/S0092-8674(03)00231-9 . ПМИД 12679032 .
- ^ Куэльяр Т.Л., Макманус М.Т. (декабрь 2005 г.). «МикроРНК и эндокринная биология» . Журнал эндокринологии . 187 (3): 327–32. дои : 10.1677/joe.1.06426 . ПМИД 16423811 .
- ^ Пой М.Н., Элиассон Л., Круцфельдт Дж., Кувадзима С., Ма Х., Макдональд П.Е. и др. (ноябрь 2004 г.). «МикроРНК, специфичная для островков поджелудочной железы, регулирует секрецию инсулина». Природа . 432 (7014): 226–30. Бибкод : 2004Natur.432..226P . дои : 10.1038/nature03076 . ПМИД 15538371 . S2CID 4415988 .
- ^ Чен Ч.З., Ли Л., Лодиш Х.Ф., Бартель Д.П. (январь 2004 г.). «МикроРНК модулируют дифференцировку гемопоэтических линий». Наука . 303 (5654): 83–6. Бибкод : 2004Sci...303...83C . дои : 10.1126/science.1091903 . hdl : 1721.1/7483 . ПМИД 14657504 . S2CID 7044929 .
- ^ Уилфред БР, Ван В.С., Нельсон П.Т. (июль 2007 г.). «Энергичные исследования микроРНК: обзор роли микроРНК в метаболизме липидов с прогнозом, что микроРНК-103/107 регулирует метаболические пути человека» . Молекулярная генетика и обмен веществ . 91 (3): 209–17. дои : 10.1016/j.ymgme.2007.03.011 . ПМК 1978064 . ПМИД 17521938 .
- ^ Харф Б.Д., Макманус М.Т., Мэнсфилд Дж.Х., Хорнштейн Э., Табин С.Дж. (август 2005 г.). «Фермент РНКаза III Dicer необходим для морфогенеза, но не для формирования паттерна конечностей позвоночных» . Труды Национальной академии наук Соединенных Штатов Америки . 102 (31): 10898–903. Бибкод : 2005PNAS..10210898H . дои : 10.1073/pnas.0504834102 . ПМК 1182454 . ПМИД 16040801 .
- ^ Транг П., Вайдхас Дж.Б., Слак Ф.Дж. (декабрь 2008 г.). «МикроРНК как потенциальные средства лечения рака» . Онкоген . 27 (Приложение 2): S52–7. дои : 10.1038/onc.2009.353 . ПМЦ 10033140 . ПМИД 19956180 .
- ^ Ли С., Фэн Ю., Кукос Г., Чжан Л. (декабрь 2009 г.). «Терапевтические стратегии микроРНК при раке человека» . Журнал AAPS . 11 (4): 747–57. дои : 10.1208/s12248-009-9145-9 . ПМК 2782079 . ПМИД 19876744 .
- ^ Фасанаро П., Греко С., Иван М., Капогросси М.К., Мартелли Ф. (январь 2010 г.). «микроРНК: новые терапевтические мишени при острых ишемических заболеваниях». Фармакология и терапия . 125 (1): 92–104. doi : 10.1016/j.pharmthera.2009.10.003 . ПМИД 19896977 .
- ^ Гидбринг П., Бадалян-Вери Дж. (август 2013 г.). «Клиническое применение микроРНК» . F1000Исследования . 2 : 136. doi : 10.12688/f1000research.2-136.v2 . ПМЦ 3917658 . ПМИД 24627783 .
- ^ Вайтман Б., Ха И, Рувкун Г. (декабрь 1993 г.). «Посттранскрипционная регуляция гетерохронного гена lin-14 с помощью lin-4 опосредует формирование временного паттерна у C. elegans» . Клетка . 75 (5): 855–62. дои : 10.1016/0092-8674(93)90530-4 . ПМИД 8252622 .
- ^ Гиза Д.Э., Калин Г.А. (2015). «МикроРНК и хронический лимфоцитарный лейкоз». МикроРНК: Рак . Достижения экспериментальной медицины и биологии. Том. 889. стр. 23–40. дои : 10.1007/978-3-319-23730-5_2 . ISBN 978-3-319-23729-9 . ПМИД 26658994 .
- ^ Амброс В., Бартель Б., Бартель Д.П., Бердж С.Б., Кэррингтон Дж.К., Чен X и др. (март 2003 г.). «Единая система аннотации микроРНК» . РНК . 9 (3): 277–9. дои : 10.1261/rna.2183803 . ПМЦ 1370393 . ПМИД 12592000 .
- ^ Гриффитс-Джонс С., Грокок Р.Дж., ван Донген С., Бейтман А., Энрайт А.Дж. (январь 2006 г.). «miRBase: последовательности микроРНК, мишени и номенклатура генов» . Исследования нуклеиновых кислот . 34 (Проблема с базой данных): D140–4. дои : 10.1093/нар/gkj112 . ПМЦ 1347474 . ПМИД 16381832 .
- ^ Райт М.В., Бруфорд Э.А. (январь 2011 г.). «Именование «мусора»: номенклатура генов небелковой РНК (нкРНК) человека» . Геномика человека . 5 (2): 90–8. дои : 10.1186/1479-7364-5-2-90 . ПМК 3051107 . ПМИД 21296742 .
- ^ Хант М., Банерджи С., Сурана П., Лю М., Фюрст Г., Матиони С. и др. (2019). «Открытие малых РНК при взаимодействии ячменя и возбудителя мучнистой росы» . БМК Геномика . 20 (1): 19–53. дои : 10.1186/s12864-019-5947-z . ПМК 6657096 . ПМИД 31345162 .
- ^ Перейти обратно: а б с Льюис Б.П., Ши И.Х., Джонс-Роудс М.В., Бартель Д.П., Бердж CB (декабрь 2003 г.). «Прогнозирование целей микроРНК млекопитающих» . Клетка . 115 (7): 787–98. дои : 10.1016/S0092-8674(03)01018-3 . ПМИД 14697198 .
- ^ Эльвангер, округ Колумбия, Бюттнер Ф.А., Мьюс Х.В., Штюмпфлен В. (май 2011 г.). «Достаточный минимальный набор типов семян микроРНК» . Биоинформатика . 27 (10): 1346–50. doi : 10.1093/биоинформатика/btr149 . ПМК 3087955 . ПМИД 21441577 .
- ^ Раевский Н. (июнь 2006 г.). «Предсказания целевых микроРНК у животных». Природная генетика . 38 (6с): С8–13. дои : 10.1038/ng1798 . ПМИД 16736023 . S2CID 23496396 .
- ^ Крек А., Грюн Д., Пой М.Н., Вольф Р., Розенберг Л., Эпштейн Э.Дж. и др. (май 2005 г.). «Комбинаторные предсказания целей микроРНК». Природная генетика . 37 (5): 495–500. дои : 10.1038/ng1536 . ПМИД 15806104 . S2CID 22672750 .
- ^ Томсон Д.В., Бракен С.П., Гудолл Дж.Дж. (сентябрь 2011 г.). «Экспериментальные стратегии идентификации мишеней микроРНК» . Исследования нуклеиновых кислот . 39 (16): 6845–53. дои : 10.1093/nar/gkr330 . ПМК 3167600 . ПМИД 21652644 .
- ^ Перейти обратно: а б Лим Л.П., Лау Н.К., Гарретт-Энгеле П., Гримсон А., Шелтер Дж.М., Касл Дж. и др. (февраль 2005 г.). «Анализ микрочипов показывает, что некоторые микроРНК подавляют большое количество целевых мРНК». Природа . 433 (7027): 769–73. Бибкод : 2005Natur.433..769L . дои : 10.1038/nature03315 . ПМИД 15685193 . S2CID 4430576 .
- ^ Зельбах М., Шванхойссер Б., Тирфельдер Н., Фанг З., Ханин Р., Раевски Н. (сентябрь 2008 г.). «Широко распространенные изменения в синтезе белка, вызванные микроРНК». Природа . 455 (7209): 58–63. Бибкод : 2008Natur.455...58S . дои : 10.1038/nature07228 . ПМИД 18668040 . S2CID 4429008 .
- ^ Пэк Д., Виллен Дж., Шин С., Камарго Ф.Д., Гиги С.П., Бартель Д.П. (сентябрь 2008 г.). «Влияние микроРНК на выход белка» . Природа . 455 (7209): 64–71. Бибкод : 2008Natur.455...64B . doi : 10.1038/nature07242 (неактивен 26 апреля 2024 г.). ПМК 2745094 . ПМИД 18668037 .
{{cite journal}}: CS1 maint: DOI неактивен по состоянию на апрель 2024 г. ( ссылка ) - ^ Перейти обратно: а б Родригес А., Гриффитс-Джонс С., Ашерст Дж.Л., Брэдли А. (октябрь 2004 г.). «Идентификация генов-хозяев микроРНК млекопитающих и единиц транскрипции» . Геномные исследования . 14 (10А): 1902–10. дои : 10.1101/гр.2722704 . ПМК 524413 . ПМИД 15364901 .
- ^ Перейти обратно: а б с д Кай X, Хагедорн CH, Каллен BR (декабрь 2004 г.). «Человеческие микроРНК производятся из кэпированных полиаденилированных транскриптов, которые также могут функционировать как мРНК» . РНК . 10 (12): 1957–66. дои : 10.1261/rna.7135204 . ПМК 1370684 . ПМИД 15525708 .
- ^ Вебер М.Дж. (январь 2005 г.). «Новые гены микроРНК человека и мыши, обнаруженные методом поиска гомологии» . Журнал ФЭБС . 272 (1): 59–73. дои : 10.1111/j.1432-1033.2004.04389.x . ПМИД 15634332 . S2CID 32923462 .
- ^ Ким Ю.К., Ким В.Н. (февраль 2007 г.). «Процессинг интронных микроРНК» . Журнал ЭМБО . 26 (3): 775–83. дои : 10.1038/sj.emboj.7601512 . ПМЦ 1794378 . ПМИД 17255951 .
- ^ Баскервиль С., Бартель Д.П. (март 2005 г.). «Профилирование микроРНК на микрочипах выявило частую коэкспрессию с соседними микроРНК и генами хозяина» . РНК . 11 (3): 241–7. дои : 10.1261/rna.7240905 . ПМК 1370713 . ПМИД 15701730 .
- ^ Перейти обратно: а б с Ли Ю, Ким М, Хан Дж, Ём К.Х., Ли С., Пэк Ш. и др. (октябрь 2004 г.). «Гены микроРНК транскрибируются РНК-полимеразой II» . Журнал ЭМБО . 23 (20): 4051–60. дои : 10.1038/sj.emboj.7600385 . ПМК 524334 . ПМИД 15372072 .
- ^ Чжоу X, Жуань Дж, Ван Г, Чжан В (март 2007 г.). «Характеристика и идентификация основных промоторов микроРНК у четырех модельных видов» . PLOS Вычислительная биология . 3 (3): е37. Бибкод : 2007PLSCB...3...37Z . дои : 10.1371/journal.pcbi.0030037 . ПМЦ 1817659 . ПМИД 17352530 .
- ^ Фаллер М., Го Ф (ноябрь 2008 г.). «Биогенез микроРНК: есть несколько способов снять шкуру с кошки» . Biochimica et Biophysica Acta (BBA) - Механизмы регуляции генов . 1779 (11): 663–7. дои : 10.1016/j.bbagrm.2008.08.005 . ПМЦ 2633599 . ПМИД 18778799 .
- ^ Ли Ю, Ан С, Хан Дж, Чой Х, Ким Дж, Йим Дж и др. (сентябрь 2003 г.). «Ядерная РНКаза III Дроша инициирует процессинг микроРНК». Природа . 425 (6956): 415–9. Бибкод : 2003Natur.425..415L . дои : 10.1038/nature01957 . ПМИД 14508493 . S2CID 4421030 .
- ^ Грегори Р.И., Чендримада Т.П., Шихаттар Р. (2006). «Биогенез микроРНК: выделение и характеристика микропроцессорного комплекса». Протоколы микроРНК . Методы молекулярной биологии. Том. 342. стр. 33–47. дои : 10.1385/1-59745-123-1:33 . ISBN 978-1-59745-123-9 . ПМИД 16957365 .
- ^ Хан Дж., Ли Ю., Ём К.Х., Ким Ю.К., Джин Х., Ким В.Н. (декабрь 2004 г.). «Комплекс Дроша-DGCR8 в первичном процессинге микроРНК» . Гены и развитие . 18 (24): 3016–27. дои : 10.1101/gad.1262504 . ПМК 535913 . ПМИД 15574589 .
- ^ Хан Дж., Ли Ю., Йом К.Х., Нам Дж.В., Хо И., Ри Дж.К. и др. (июнь 2006 г.). «Молекулярные основы распознавания первичных микроРНК комплексом Дроша-DGCR8» . Клетка . 125 (5): 887–901. дои : 10.1016/j.cell.2006.03.043 . ПМИД 16751099 .
- ^ Конрад Т., Марсико А., Гере М., Ором У.А. (октябрь 2014 г.). «Активность микропроцессора контролирует дифференциальный биогенез микроРНК в Vivo» . Отчеты по ячейкам . 9 (2): 542–54. дои : 10.1016/j.celrep.2014.09.007 . ПМИД 25310978 .
- ^ Ауён В.К., Улицкий И., Макгири С.Э., Бартель Д.П. (февраль 2013 г.). «Помимо вторичной структуры: детерминанты первичной последовательности лицензируют шпильки при-миРНК для обработки» . Клетка . 152 (4): 844–58. дои : 10.1016/j.cell.2013.01.031 . ПМК 3707628 . ПМИД 23415231 .
- ^ Али П.С., Гошдастидер У., Хоффманн Дж., Брутши Б., Филипек С. (ноябрь 2012 г.). «Распознавание предшественника микроРНК let-7g человеческим Lin28B» . Письма ФЭБС . 586 (22): 3986–90. дои : 10.1016/j.febslet.2012.09.034 . ПМИД 23063642 . S2CID 28899778 .
- ^ Руби Дж.Г., Ян CH, Бартель Д.П. (июль 2007 г.). «Интронные предшественники микроРНК, обходящие процессинг Дроши» . Природа . 448 (7149): 83–86. Бибкод : 2007Natur.448...83R . дои : 10.1038/nature05983 . ПМЦ 2475599 . ПМИД 17589500 .
- ^ Руби Дж.Г., Ян CH, Бартель Д.П. (июль 2007 г.). «Интронные предшественники микроРНК, обходящие процессинг Дроши» . Природа . 448 (7149): 83–86. Бибкод : 2007Natur.448...83R . дои : 10.1038/nature05983 . ПМЦ 2475599 . ПМИД 17589500 .
- ^ Березиков Э., Чанг В.Дж., Уиллис Дж., Куппен Э., Лай Э.К. (октябрь 2007 г.). «Миртронные гены млекопитающих» . Молекулярная клетка . 28 (2): 328–336. doi : 10.1016/j.molcel.2007.09.028 . ПМЦ 2763384 . ПМИД 17964270 .
- ^ Перейти обратно: а б Кавахара Ю., Меграу М., Крайдер Э., Иисаса Х., Валенте Л., Хацигеоргиу А.Г. и др. (сентябрь 2008 г.). «Частота и судьба редактирования микроРНК в мозге человека» . Исследования нуклеиновых кислот . 36 (16): 5270–80. дои : 10.1093/нар/gkn479 . ПМЦ 2532740 . ПМИД 18684997 .
- ^ Винтер Дж., Юнг С., Келлер С., Грегори Р.И., Дидерихс С. (март 2009 г.). «Многие пути к зрелости: пути биогенеза микроРНК и их регуляция». Природная клеточная биология . 11 (3): 228–34. дои : 10.1038/ncb0309-228 . ПМИД 19255566 . S2CID 205286318 .
- ^ Оман М. (октябрь 2007 г.). «Претендент или союзник процесса редактирования микроРНК». Биохимия . 89 (10): 1171–6. дои : 10.1016/j.biochi.2007.06.002 . ПМИД 17628290 .
- ^ Перейти обратно: а б Мерчисон Э.П. , Хэннон Дж.Дж. (июнь 2004 г.). «МиРНК в движении: биогенез микроРНК и механизм РНКи». Современное мнение в области клеточной биологии . 16 (3): 223–9. дои : 10.1016/j.ceb.2004.04.003 . ПМИД 15145345 .

- ^ Перейти обратно: а б с Лунд Э., Дальберг Дж. Э. (2006). «Субстратная селективность экспортина 5 и Dicer в биогенезе микроРНК» . Симпозиумы Колд-Спринг-Харбор по количественной биологии . 71 : 59–66. дои : 10.1101/sqb.2006.71.050 . ПМИД 17381281 .
- ^ Пак Дж.Э., Хо И., Тянь Ю., Симаншу Д.К., Чанг Х., Джи Д. и др. (июль 2011 г.). «Dicer распознает 5'-конец РНК для эффективной и точной обработки» . Природа . 475 (7355): 201–5. дои : 10.1038/nature10198 . ПМЦ 4693635 . ПМИД 21753850 .
- ^ Джи Икс (2008). «Механизм действия РНКазы III: как Dicer Dices». РНК-интерференция . Актуальные темы микробиологии и иммунологии. Том. 320. стр. 99–116. дои : 10.1007/978-3-540-75157-1_5 . ISBN 978-3-540-75156-4 . ПМИД 18268841 .
- ^ Мирихана Араччилаге Дж., Дассанаяке AC, Басу С. (февраль 2015 г.). «Зависимый от ионов калия структурный переключатель РНК регулирует созревание пре-миРНК 92b человека» . Химия и биология . 22 (2): 262–72. doi : 10.1016/j.chembiol.2014.12.013 . ПМИД 25641166 .
- ^ Сохель М.Х. (2016). «Внеклеточные/циркулирующие микроРНК: механизмы высвобождения, функции и проблемы» . Достижения в области наук о жизни . 10 (2): 175–186. дои : 10.1016/j.als.2016.11.007 .
- ^ Перейти обратно: а б Бекель Й.Н., Рейс С.М., Лейстнер Д., Томе К.Э., Зейхер А.М., Фихтльшерер С. и др. (апрель 2014 г.). «От сердца до пят: вклад сердца в уровень периферических микроРНК». Международный журнал кардиологии . 172 (3): 616–7. doi : 10.1016/j.ijcard.2014.01.082 . ПМИД 24508494 .
- ^ Леландэ-Брьер С., Сорин С., Деклерк М., Бенслиман А., Креспи М., Хартманн С. (март 2010 г.). «Разнообразие малых РНК у растений и его влияние на развитие» . Современная геномика . 11 (1): 14–23. дои : 10.2174/138920210790217918 . ПМЦ 2851111 . ПМИД 20808519 .
- ^ Рана ТМ (январь 2007 г.). «Осветляя тишину: понимание структуры и функции малых РНК». Nature Reviews Молекулярно-клеточная биология . 8 (1): 23–36. дои : 10.1038/nrm2085 . ПМИД 17183358 . S2CID 8966239 .
- ^ Перейти обратно: а б Шварц Д.С., Замор П.Д. (май 2002 г.). «Почему микроРНК живут в микроРНК?» . Гены и развитие . 16 (9): 1025–31. дои : 10.1101/gad.992502 . ПМИД 12000786 .
- ^ Крол Дж., Собчак К., Вильчинска У., Драт М., Ясинска А., Качиньска Д. и др. (октябрь 2004 г.). «Структурные особенности предшественников микроРНК (миРНК) и их значение для биогенеза микроРНК и дизайна малых интерферирующих РНК/коротких шпилек РНК» . Журнал биологической химии . 279 (40): 42230–9. дои : 10.1074/jbc.M404931200 . ПМИД 15292246 .
- ^ Хворова А., Рейнольдс А., Джаясена С.Д. (октябрь 2003 г.). «Функциональные миРНК и микроРНК демонстрируют смещение цепи» . Клетка . 115 (2): 209–16. дои : 10.1016/S0092-8674(03)00801-8 . ПМИД 14567918 .
- ^ Шварц Д.С., Хутвагнер Г., Ду Т., Сюй З., Аронин Н., Замор П.Д. (октябрь 2003 г.). «Асимметрия сборки ферментного комплекса РНКи» . Клетка . 115 (2): 199–208. дои : 10.1016/S0092-8674(03)00759-1 . ПМИД 14567917 .
- ^ Линь С.Л., Чанг Д., Ин С.Ю. (август 2005 г.). «Асимметрия интронных структур пре-миРНК в функциональной сборке RISC» . Джин . 356 : 32–8. дои : 10.1016/j.gene.2005.04.036 . ПМК 1788082 . ПМИД 16005165 .
- ^ Окамура К., Чунг В.Дж., Лай Э.К. (сентябрь 2008 г.). «Длинные и короткие гены с инвертированными повторами у животных: микроРНК, миртроны и шпильковые РНК» . Клеточный цикл . 7 (18): 2840–5. дои : 10.4161/cc.7.18.6734 . ПМК 2697033 . ПМИД 18769156 .
- ^ Перейти обратно: а б Пратт А.Дж., Макрей И.Дж. (июль 2009 г.). «Комплекс молчания, индуцированный РНК: универсальная машина для подавления генов» . Журнал биологической химии . 284 (27): 17897–901. дои : 10.1074/jbc.R900012200 . ПМК 2709356 . ПМИД 19342379 .
- ^ Макрей И.Дж., Ма Э., Чжоу М., Робинсон К.В., Дудна Дж.А. (январь 2008 г.). «Восстановление in vitro человеческого RISC-загрузочного комплекса» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (2): 512–7. Бибкод : 2008PNAS..105..512M . дои : 10.1073/pnas.0710869105 . ПМК 2206567 . ПМИД 18178619 .
- ^ Мурелатос З., Дости Дж., Паушкин С., Шарма А., Шарру Б., Абель Л. и др. (март 2002 г.). «МиРНП: новый класс рибонуклеопротеинов, содержащий многочисленные микроРНК» . Гены и развитие . 16 (6): 720–8. дои : 10.1101/gad.974702 . ПМК 155365 . ПМИД 11914277 .
- ^ Мейстер Г., Ландталер М., Петерс Л., Чен П.Ю., Урлауб Х., Люрманн Р. и др. (декабрь 2005 г.). «Идентификация новых белков, связанных с аргонавтами» . Современная биология . 15 (23): 2149–55. Бибкод : 2005CBio...15.2149M . дои : 10.1016/j.cub.2005.10.048 . hdl : 11858/00-001M-0000-0012-E763-B . ПМИД 16289642 .
- ^ Цзин Ц, Хуан С., Гут С., Зарубин Т., Мотояма А., Чен Дж. и др. (март 2005 г.). «Участие микроРНК в нестабильности мРНК, опосредованной AU-богатыми элементами» . Клетка . 120 (5): 623–34. дои : 10.1016/j.cell.2004.12.038 . PMID 15766526 .
- ^ Перейти обратно: а б с Кай З.С., Паскинелли А.Е. (январь 2010 г.). «Убийцы микроРНК: факторы, регулирующие исчезновение микроРНК» . Структурная и молекулярная биология природы . 17 (1): 5–10. дои : 10.1038/nsmb.1762 . ПМК 6417416 . ПМИД 20051982 .
- ^ Чаттерджи С., Гроссханс Х. (сентябрь 2009 г.). «Активный оборот модулирует активность зрелых микроРНК у Caenorhabditis elegans». Природа . 461 (7263): 546–9. Бибкод : 2009Natur.461..546C . дои : 10.1038/nature08349 . ПМИД 19734881 . S2CID 4414841 .
- ^ Перейти обратно: а б с Морозова Н., Зиновьев А., Нонне Н., Притчард Л.Л., Горбан А.Н., Харель-Беллан А. (сентябрь 2012 г.). «Кинетические признаки действия микроРНК» . РНК . 18 (9): 1635–55. дои : 10.1261/rna.032284.112 . ПМЦ 3425779 . ПМИД 22850425 .
- ^ Ван XJ, Рейес Дж.Л., Чуа Н.Х., Гаастерланд Т. (2004). «Прогнозирование и идентификация микроРНК Arabidopsis thaliana и их мишеней мРНК» . Геномная биология . 5 (9): 65 р. дои : 10.1186/gb-2004-5-9-r65 . ПМК 522872 . ПМИД 15345049 .
- ^ Кавасаки Х, Тайра К (2004). «МикроРНК-196 ингибирует экспрессию HOXB8 при миелоидной дифференцировке клеток HL60» . Серия симпозиумов по нуклеиновым кислотам . 48 (1): 211–2. дои : 10.1093/насс/48.1.211 . ПМИД 17150553 .
- ^ Перейти обратно: а б Моксон С., Цзин Р., Шиттия Г., Швах Ф., Рашолм Пилчер Р.Л., Моултон В. и др. (октябрь 2008 г.). «Глубокое секвенирование коротких РНК томатов идентифицирует микроРНК, нацеленные на гены, участвующие в созревании фруктов» . Геномные исследования . 18 (10): 1602–9. дои : 10.1101/гр.080127.108 . ПМК 2556272 . ПМИД 18653800 .
- ^ Мазьер П., Энрайт А.Дж. (июнь 2007 г.). «Прогнозирование мишеней микроРНК». Открытие наркотиков сегодня . 12 (11–12): 452–8. дои : 10.1016/j.drudis.2007.04.002 . ПМИД 17532529 .
- ^ Уильямс А.Е. (февраль 2008 г.). «Функциональные аспекты микроРНК животных» . Клеточные и молекулярные науки о жизни . 65 (4): 545–62. дои : 10.1007/s00018-007-7355-9 . ПМЦ 11131689 . ПМИД 17965831 . S2CID 5708394 .
- ^ Эулалио А., Ханцингер Э., Нишихара Т., Ревинкель Дж., Фаузер М., Изаурральде Э. (январь 2009 г.). «Деаденилирование является широко распространенным эффектом регуляции микроРНК» . РНК . 15 (1): 21–3 дои : 10.1261/rna.1399509 . ПМЦ 2612776 . ПМИД 19029310 .
- ^ Баззини А.А., Ли М.Т., Хиральдес А.Дж. (апрель 2012 г.). «Профилирование рибосом показывает, что миР-430 снижает трансляцию, прежде чем вызвать распад мРНК у рыбок данио» . Наука . 336 (6078): 233–7. Бибкод : 2012Sci...336..233B . дои : 10.1126/science.1215704 . ПМЦ 3547538 . ПМИД 22422859 .
- ^ Джуранович С., Нахви А., Грин Р. (апрель 2012 г.). «миРНК-опосредованное замалчивание генов путем репрессии трансляции с последующим деаденилированием и распадом мРНК» . Наука . 336 (6078): 237–40. Бибкод : 2012Sci...336..237D . дои : 10.1126/science.1215691 . ПМЦ 3971879 . ПМИД 22499947 .
- ^ Тан Ю, Чжан Б, Ву Т, Скогербё Г, Чжу X, Го X и др. (февраль 2009 г.). «Транскрипционное ингибирование экспрессии Hoxd4 с помощью миРНК-10а в клетках рака молочной железы человека» . BMC Молекулярная биология . 10 (1): 12. дои : 10.1186/1471-2199-10-12 . ПМК 2680403 . ПМИД 19232136 .
- ^ Хокинс П.Г., Моррис К.В. (март 2008 г.). «РНК и транскрипционная модуляция экспрессии генов» . Клеточный цикл . 7 (5): 602–7. дои : 10.4161/cc.7.5.5522 . ПМЦ 2877389 . ПМИД 18256543 .
- ^ Старк А., Бреннеке Дж., Бушати Н., Рассел Р.Б., Коэн С.М. (декабрь 2005 г.). «МикроРНК животных придают устойчивость экспрессии генов и оказывают значительное влияние на эволюцию 3'UTR» . Клетка . 123 (6): 1133–46. дои : 10.1016/j.cell.2005.11.023 . ПМИД 16337999 .
- ^ Он Л., Хэннон Г.Дж. (июль 2004 г.). «МикроРНК: малые РНК, играющие большую роль в регуляции генов». Обзоры природы. Генетика . 5 (7): 522–531. дои : 10.1038/nrg1379 . ПМИД 15211354 . S2CID 5270062 .
- ^ Ли Л.К. (2008). «Активация генов, опосредованная малыми РНК» . В Моррисе К.В. (ред.). РНК и регуляция экспрессии генов: скрытый уровень сложности . Горизонт Научная Пресса. ISBN 978-1-904455-25-7 .
- ^ Плейс РФ, Ли Л.К., Пукот Д., Нунан Э.Дж., Дахия Р. (февраль 2008 г.). «МикроРНК-373 индуцирует экспрессию генов с комплементарными последовательностями промотора» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (5): 1608–13. Бибкод : 2008PNAS..105.1608P . дои : 10.1073/pnas.0707594105 . ПМК 2234192 . ПМИД 18227514 . (Ошибка: два : 10.1073/pnas.1803343115 , PMID 29555737 , Часы втягивания )
- ^ Салмена Л., Полисено Л., Тай Ю., Кац Л., Пандольфи П.П. (август 2011 г.). «Гипотеза ceRNA: Розеттский камень скрытого языка РНК?» . Клетка . 146 (3): 353–8. дои : 10.1016/j.cell.2011.07.014 . ПМЦ 3235919 . ПМИД 21802130 .
- ^ Перейти обратно: а б Кумар С., Редди П.Х. (сентябрь 2016 г.). «Являются ли циркулирующие микроРНК периферическими биомаркерами болезни Альцгеймера?» . Биохим Биофиз Акта . 1862 (9): 1617–27. дои : 10.1016/j.bbadis.2016.06.001 . ПМК 5343750 . ПМИД 27264337 .
- ^ ван ден Берг М.М., Краускопф Дж., Рамаекерс Дж.Г. и др. (февраль 2020 г.). «Циркулирующие микроРНК как потенциальные биомаркеры психиатрических и нейродегенеративных расстройств» . Прога Нейробиол . 185 : 101732. doi : 10.1016/j.pneurobio.2019.101732 . ПМИД 31816349 .
- ^ Куман С., Ван Синдерен М., Гантье М.П., Раинчук К., Сорби К., Ромбоутс Л. и др. (октябрь 2015 г.). «Секретируемые бластоцистой человека микроРНК регулируют адгезию эпителиальных клеток эндометрия» . Электронная биомедицина . 2 (10): 1528–1535. дои : 10.1016/j.ebiom.2015.09.003 . ПМЦ 4634783 . ПМИД 26629549 .
- ^ Чжоу Л., Миллер С., Миралья Л.Дж., Ромеро А., Муре Л.С., Панда С. и др. (январь 2021 г.). «Полногеномный скрининг микроРНК идентифицирует кластер микроРНК-183/96/182 как модулятор циркадных ритмов» . Труды Национальной академии наук Соединенных Штатов Америки . 118 (1): e2020454118. Бибкод : 2021PNAS..11820454Z . дои : 10.1073/pnas.2020454118 . ПМЦ 7817116 . ПМИД 33443164 . S2CID 230713808 .
- «МикроРНК играют ключевую роль в регуляции циркадных ритмов» . Новости науки . 6 января 2021 г.
- ^ Axtell MJ, Bartel DP (июнь 2005 г.). «Древность микроРНК и их мишени в наземных растениях» . Растительная клетка . 17 (6): 1658–73. дои : 10.1105/tpc.105.032185 . ПМК 1143068 . ПМИД 15849273 .
- ^ Танцер А., Штадлер ПФ (май 2004 г.). «Молекулярная эволюция кластера микроРНК». Журнал молекулярной биологии . 339 (2): 327–35. CiteSeerX 10.1.1.194.1598 . дои : 10.1016/j.jmb.2004.03.065 . ПМИД 15136036 .
- ^ Чен К., Раевски Н. (февраль 2007 г.). «Эволюция регуляции генов с помощью факторов транскрипции и микроРНК». Обзоры природы Генетика . 8 (2): 93–103. дои : 10.1038/nrg1990 . ПМИД 17230196 . S2CID 174231 .
- ^ Ли К.Т., Рисом Т., Штраус В.М. (апрель 2007 г.). «Эволюционная консервация регуляторных цепей микроРНК: исследование сложности генов микроРНК и консервативных взаимодействий микроРНК-мишень в филогении многоклеточных животных». ДНК и клеточная биология . 26 (4): 209–18. дои : 10.1089/dna.2006.0545 . PMID 17465887 .
- ^ Перейти обратно: а б с д Петерсон К.Дж., Дитрих М.Р., МакПик М.А. (июль 2009 г.). «МикроРНК и макроэволюция многоклеточных животных: понимание канализации, сложности и кембрийского взрыва» . Биоэссе . 31 (7): 736–47. doi : 10.1002/bies.200900033 . ПМИД 19472371 . S2CID 15364875 .
- ^ Шабалина С.А., Кунин Е.В. (октябрь 2008 г.). «Происхождение и эволюция эукариотической РНК-интерференции» . Тенденции в экологии и эволюции . 23 (10): 578–87. дои : 10.1016/j.tree.2008.06.005 . ПМЦ 2695246 . ПМИД 18715673 .
- ^ Экстелл М.Дж., Вестхольм Дж.О., Лай Э.К. (2011). «Vive la différence: биогенез и эволюция микроРНК у растений и животных» . Геномная биология . 12 (4): 221. doi : 10.1186/gb-2011-12-4-221 . ПМК 3218855 . ПМИД 21554756 .
- ^ Перейти обратно: а б Уилер Б.М., Хаймберг А.М., Мой В.Н., Сперлинг Е.А., Хольштейн Т.В., Хибер С. и др. (2009). «Глубокая эволюция микроРНК многоклеточных животных». Эволюция и развитие . 11 (1): 50–68. дои : 10.1111/j.1525-142X.2008.00302.x . ПМИД 19196333 . S2CID 14924603 .
- ^ Пашковский ПП, Рязанский СС (июнь 2013 г.). «Биогенез, эволюция и функции растительных микроРНК». Биохимия. Биохимия . 78 (6): 627–37. дои : 10.1134/S0006297913060084 . ПМИД 23980889 . S2CID 12025420 .
- ^ Перейти обратно: а б Хеймберг А.М., Семпере Л.Ф., Мой В.Н., Донохью ПК, Петерсон К.Дж. (февраль 2008 г.). «МикроРНК и появление морфологической сложности позвоночных» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (8): 2946–50. Бибкод : 2008PNAS..105.2946H . дои : 10.1073/pnas.0712259105 . ПМК 2268565 . ПМИД 18287013 .
- ^ Перейти обратно: а б с Нозава М., Миура С., Ней М. (июль 2010 г.). «Происхождение и эволюция генов микроРНК у видов дрозофилы» . Геномная биология и эволюция . 2 : 180–9. дои : 10.1093/gbe/evq009 . ПМК 2942034 . ПМИД 20624724 .
- ^ Аллен Э., Се З., Густафсон А.М., Сунг Г.Х., Спатафора Дж.В., Кэррингтон Дж.К. (декабрь 2004 г.). «Эволюция генов микроРНК путем инвертированного дупликации последовательностей целевых генов у Arabidopsis thaliana». Природная генетика . 36 (12): 1282–90. дои : 10.1038/ng1478 . ПМИД 15565108 . S2CID 11997028 .
- ^ Вартманн Н., Дас С., Ланц С., Вайгель Д. (май 2008 г.). «Сравнительный анализ локуса микроРНК MIR319a у Arabidopsis и родственных Brassicaceae» . Молекулярная биология и эволюция . 25 (5): 892–902. дои : 10.1093/molbev/msn029 . ПМИД 18296705 .
- ^ Фальгрен Н., Джогдео С., Касшау К.Д., Салливан С.М., Чепмен Э.Дж., Лаубингер С. и др. (апрель 2010 г.). «Эволюция генов микроРНК у Arabidopsis lyrata и Arabidopsis thaliana» . Растительная клетка . 22 (4): 1074–89. дои : 10.1105/tpc.110.073999 . ПМЦ 2879733 . ПМИД 20407027 .
- ^ Каравас Дж., Фридрих М. (июнь 2010 г.). «О клещах и многоножках: недавний прогресс в разрешении основания дерева членистоногих». Биоэссе . 32 (6): 488–95. doi : 10.1002/bies.201000005 . ПМИД 20486135 . S2CID 20548122 .
- ^ Кенни Нью-Джерси, Намигай Э.К., Марлетаз Ф., Хуэй Дж.Х., Шимелд С.М. (декабрь 2015 г.). «Проект сборок генома и прогнозируемые комплементы микроРНК приливных лофотрохозозов Patella vulgata (Mollusca, Patellogastropoda) и Spirobranchus (Pomatoceros) lamarcki (Annelida, Serpulida)» . Морская геномика . 24 (2): 139–46. Бибкод : 2015Март..24..139К . дои : 10.1016/j.margen.2015.07.004 . ПМИД 26319627 .
- ^ Кок Дж.М., Стерк Л., Рузе П., Скорнет Д., Аллен А.Е., Амуциас Г. и др. (июнь 2010 г.). «Геном Ectocarpus и независимая эволюция многоклеточности бурых водорослей» . Природа . 465 (7298): 617–21. Бибкод : 2010Natur.465..617C . дои : 10.1038/nature09016 . ПМИД 20520714 .
- ^ Куперус Дж. Т., Фальгрен Н., Кэррингтон Дж. К. (февраль 2011 г.). «Эволюция и функциональное разнообразие генов MIRNA» . Растительная клетка . 23 (2): 431–42. дои : 10.1105/tpc.110.082784 . ПМК 3077775 . ПМИД 21317375 .
- ^ Райан Дж. Ф., Панг К., Шницлер С. Э., Нгуен А. Д., Морленд РТ, Симмонс Д. К. и др. (декабрь 2013 г.). «Геном гребневика Mnemiopsis leidyi и его значение для эволюции типов клеток» . Наука . 342 (6164): 1242592. doi : 10.1126/science.1242592 . ПМЦ 3920664 . ПМИД 24337300 .
- ^ Максвелл Э.К., Райан Дж.Ф., Шницлер К.Э., Браун В.Е., Баксеванис А.Д. (декабрь 2012 г.). «МикроРНК и важные компоненты механизма обработки микроРНК не закодированы в геноме гребневика Mnemiopsis leidyi» . БМК Геномика . 13 (1): 714. дои : 10.1186/1471-2164-13-714 . ПМЦ 3563456 . ПМИД 23256903 .
- ^ Даймонд П.Ф. (15 марта 2010 г.). «Терапевтический потенциал микроРНК» . Новости генной инженерии и биотехнологии . 30 (6): 1. Архивировано из оригинала 19 июля 2010 года . Проверено 10 июля 2010 г.
- ^ Тьяден Б., Гудвин С.С., Опдайк Дж.А., Гильер М., Фу Д.С., Готтесман С. и др. (2006). «Целевой прогноз для малых некодирующих РНК в бактериях» . Исследования нуклеиновых кислот . 34 (9): 2791–802. дои : 10.1093/нар/gkl356 . ПМЦ 1464411 . ПМИД 16717284 .
- ^ Лю К.Г., Калин Г.А., Волиния С., Кроче С.М. (2008). «Профилирование экспрессии микроРНК с использованием микрочипов». Протоколы природы . 3 (4): 563–78. дои : 10.1038/nprot.2008.14 . ПМИД 18388938 . S2CID 2441105 .
- ^ Чен С., Ридзон Д.А., Брумер А.Дж., Чжоу З., Ли Д.Х., Нгуен Дж.Т. и др. (ноябрь 2005 г.). «Количественное определение микроРНК в реальном времени с помощью RT-PCR на основе стволовой петли» . Исследования нуклеиновых кислот . 33 (20): e179. дои : 10.1093/nar/gni178 . ПМК 1292995 . ПМИД 16314309 .
- ^ Шингара Дж., Кейгер К., Шелтон Дж., Лаосинчай-Вольф В., Пауэрс П., Конрад Р. и др. (сентябрь 2005 г.). «Оптимизированная платформа выделения и маркировки для точного профилирования экспрессии микроРНК» . РНК . 11 (9): 1461–70. дои : 10.1261/rna.2610405 . ПМЦ 1370829 . ПМИД 16043497 .
- ^ Буерманс Х.П., Ариюрек Ю., ван Оммен Г., ден Даннен Дж.Т., 'т Хоэн П.А. (декабрь 2010 г.). «Новые методы определения профиля экспрессии микроРНК на основе секвенирования следующего поколения» . БМК Геномика . 11 : 716. дои : 10.1186/1471-2164-11-716 . ПМК 3022920 . ПМИД 21171994 .
- ^ Клоостерман В.П., Винхольдс Э., Кеттинг Р.Ф., Пластерк Р.Х. (2004). «Требования к субстрату для функционирования let-7 в развивающемся эмбрионе рыбки данио» . Исследования нуклеиновых кислот . 32 (21): 6284–91. дои : 10.1093/nar/gkh968 . ПМК 535676 . ПМИД 15585662 .
- ^ Флинт А.С., Ли Н., Тэтчер Э.Дж., Солника-Крезель Л., Паттон Дж.Г. (февраль 2007 г.). «МиР-214 рыбки данио модулирует передачу сигналов Hedgehog, чтобы определить судьбу мышечных клеток» . Природная генетика . 39 (2): 259–63. дои : 10.1038/ng1953 . ПМЦ 3982799 . ПМИД 17220889 .
- ^ Мейстер Г., Ландталер М., Дорсетт Ю., Тушл Т. (март 2004 г.). «Последовательность-специфическое ингибирование молчания РНК, индуцированного микроРНК и миРНК» . РНК . 10 (3): 544–50. дои : 10.1261/rna.5235104 . ПМК 1370948 . ПМИД 14970398 .
- ^ Клоостерман В.П., Лагендейк А.К., Кеттинг Р.Ф., Моултон Дж.Д., Пластерк Р.Х. (август 2007 г.). «Направленное ингибирование созревания миРНК с помощью морфолино раскрывает роль миР-375 в развитии островков поджелудочной железы» . ПЛОС Биология . 5 (8): е203. doi : 10.1371/journal.pbio.0050203 . ЧВК 1925136 . ПМИД 17676975 . (В настоящее время в этом документе выражается обеспокоенность , см. дои : 10.1371/journal.pbio.3001631 , ПМИД 35486652 )
- ^ Геберт Л.Ф., Ребхан М.А., Кривелли С.Е., Дензлер Р., Стоффель М., Холл Дж. (январь 2014 г.). «Миравирсен (SPC3649) может ингибировать биогенез миР-122» . Исследования нуклеиновых кислот . 42 (1): 609–21. дои : 10.1093/нар/gkt852 . ПМЦ 3874169 . ПМИД 24068553 .
- ^ Чой Вайоминг, Хиральдес А.Дж., Шир А.Ф. (октябрь 2007 г.). «Защитники мишени демонстрируют подавление и балансировку агониста и антагониста узла с помощью миР-430» . Наука . 318 (5848): 271–4. Бибкод : 2007Sci...318..271C . дои : 10.1126/science.1147535 . ПМИД 17761850 . S2CID 30461594 .
- ^ Ю Ю, Морейра Б.Г., Бельке М.А., Овзарзи Р. (май 2006 г.). «Разработка МШУ-зондов, улучшающих распознавание несоответствий» . Исследования нуклеиновых кислот . 34 (8): е60. дои : 10.1093/нар/gkl175 . ПМЦ 1456327 . ПМИД 16670427 .
- ^ Лагендейк А.К., Моултон Дж.Д., Баккерс Дж. (июнь 2012 г.). «Показательные детали: протокол гибридизации in situ микроРНК целиком для эмбрионов рыбок данио и взрослых тканей» . Биология Открытая . 1 (6): 566–9. дои : 10.1242/bio.2012810 . ПМК 3509442 . ПМИД 23213449 .
- ^ Каур Х., Арора А., Венгель Дж., Маити С. (июнь 2006 г.). «Термодинамические, противоионные и гидратационные эффекты для включения заблокированных нуклеотидов нуклеиновой кислоты в дуплексы ДНК». Биохимия . 45 (23): 7347–55. дои : 10.1021/bi060307w . ПМИД 16752924 .
- ^ Нильсен Дж.А., Лау П., Марик Д., Баркер Дж.Л., Хадсон Л.Д. (август 2009 г.). «Интеграция профилей экспрессии микроРНК и мРНК предшественников нейронов для выявления регуляторных сетей, лежащих в основе начала коркового нейрогенеза» . BMC Нейронаука . 10:98 . дои : 10.1186/1471-2202-10-98 . ПМЦ 2736963 . ПМИД 19689821 .
- ^ Гримсон А., Фарх К.К., Джонстон В.К., Гаррет-Энгеле П., Лим Л.П., Бартель Д.П. (июль 2007 г.). «Специфичность нацеливания микроРНК у млекопитающих: детерминанты, выходящие за рамки спаривания семян» . Молекулярная клетка . 27 (1): 91–105. doi : 10.1016/j.molcel.2007.06.017 . ПМК 3800283 . ПМИД 17612493 .
- ^ Гриффитс-Джонс С., Сайни Х.К., ван Донген С., Энрайт А.Дж. (январь 2008 г.). «miRBase: инструменты для геномики микроРНК» . Исследования нуклеиновых кислот . 36 (Проблема с базой данных): D154–8. дои : 10.1093/нар/gkm952 . ПМК 2238936 . ПМИД 17991681 .
- ^ Нам С., Ли М., Чой К., Балч С., Ким С., Племянник К.П. (июль 2009 г.). «Интегрированный анализ микроРНК и мРНК (MMIA): веб-инструмент для изучения биологических функций экспрессии микроРНК» . Исследования нуклеиновых кислот . 37 (проблема с веб-сервером): W356–62. дои : 10.1093/nar/gkp294 . ПМК 2703907 . ПМИД 19420067 .
- ^ Артманн С., Юнг К., Блекманн А., Байсбарт Т. (2012). Проверо П. (ред.). «Обнаружение одновременных групповых эффектов в экспрессии микроРНК и связанных с ней наборах целевых генов» . ПЛОС ОДИН . 7 (6): e38365. Бибкод : 2012PLoSO...738365A . дои : 10.1371/journal.pone.0038365 . ПМЦ 3378551 . ПМИД 22723856 .
- ^ Цзян Ц, Ван Ю, Хао Ю, Хуан Л, Тенг М, Чжан Икс и др. (январь 2009 г.). «miR2Disease: созданная вручную база данных для дерегуляции микроРНК при заболеваниях человека» . Исследования нуклеиновых кислот . 37. 37 (Проблема с базой данных): D98–104. дои : 10.1093/нар/gkn714 . ПМЦ 2686559 . ПМИД 18927107 .
- ^ Менсия А., Модамио-Хойбьор С., Редшоу Н., Морин М., Майо-Мерино Ф., Олаварриета Л. и др. (май 2009 г.). «Мутации в семенной области человеческой миР-96 ответственны за несиндромальную прогрессирующую потерю слуха». Природная генетика . 41 (5): 609–13. дои : 10.1038/ng.355 . ПМИД 19363479 . S2CID 11113852 .
- ^ Хьюз А.Е., Брэдли Д.Т., Кэмпбелл М., Лехнер Дж., Дэш Д.П., Симпсон Д.А. и др. (ноябрь 2011 г.). «Мутация, изменяющая семенную область миР-184, вызывает семейный кератоконус с катарактой» . Американский журнал генетики человека . 89 (5): 628–33. дои : 10.1016/j.ajhg.2011.09.014 . ПМЦ 3213395 . ПМИД 21996275 .
- ^ де Понтуаль Л., Яо Э., Калье П., Фавр Л., Друэн В., Кариу С. и др. (сентябрь 2011 г.). «Удаление зародышевой линии кластера миР-17~92 вызывает дефекты скелета и роста у людей» . Природная генетика . 43 (10): 1026–30. дои : 10.1038/ng.915 . ПМК 3184212 . ПМИД 21892160 .
- ^ Калин Г.А., Лю К.Г., Севиньяни С., Феррацин М., Фелли Н., Думитру К.Д. и др. (август 2004 г.). «Профилирование микроРНК выявляет отчетливые признаки В-клеточного хронического лимфоцитарного лейкоза» . Труды Национальной академии наук Соединенных Штатов Америки . 101 (32): 11755–11760. Бибкод : 2004PNAS..10111755C . дои : 10.1073/pnas.0404432101 . ПМК 511048 . ПМИД 15284443 .
- ^ Велле А., Песенти С., Грасси Т., Бельтрам Л., Мартини П., Якони М. и др. (май 2023 г.). «Комплексное исследование гистотип-специфичных микроРНК и их вариантов при эпителиальном раке яичников I стадии» . Международный журнал рака . 152 (9): 1989–2001. дои : 10.1002/ijc.34408 . ПМИД 36541726 . S2CID 255034585 .
- ^ Кириакидис И, Кириакидис К, Цезу А (август 2022 г.). «МикроРНК и диагностика острого лимфобластного лейкоза у детей: систематический обзор, метаанализ и повторный анализ с использованием новых инструментов секвенирования малых РНК» . Раки . 14 (16): 3976. doi : 10.3390/cancers14163976 . ПМЦ 9406077 . ПМИД 36010971 .
- ^ Выса У, Воодер Т, Кольде Р, Фишер К, Вялк К, Тыниссон Н и др. (октябрь 2011 г.). «Идентификация миР-374a как прогностического маркера выживаемости у пациентов с немелкоклеточным раком легкого на ранней стадии» . Гены, хромосомы и рак . 50 (10): 812–22. дои : 10.1002/gcc.20902 . ПМИД 21748820 . S2CID 9746594 .
- ^ Акчакая П., Экелунд С., Колосенко И., Карамута С., Озата Д.М., Се Х. и др. (август 2011 г.). «Нарушение регуляции миР-185 и миР-133b связано с общей выживаемостью и метастазированием при колоректальном раке» . Международный журнал онкологии . 39 (2): 311–8. дои : 10.3892/ijo.2011.1043 . ПМИД 21573504 .
- ^ Перейти обратно: а б Эйкинг А., Рейс Х., Франк М., Геркен Г., Шмид К.В., Карио Э. (2016). «МиР-205 и МиР-373 связаны с агрессивным муцинозным колоректальным раком человека» . ПЛОС ОДИН . 11 (6): e0156871. Бибкод : 2016PLoSO..1156871E . дои : 10.1371/journal.pone.0156871 . ПМЦ 4894642 . ПМИД 27271572 .
- ^ МикроРНК-21 способствует пролиферации клеток гепатоцеллюлярной карциномы HepG2 посредством репрессии митоген-активируемой протеинкиназы-киназы 3. Guangxian Xu et al., 2013
- ^ Джонс К., Нурс Дж.П., Кин С., Бхатнагар А., Ганди М.К. (январь 2014 г.). «МикроРНК плазмы являются биомаркерами реакции на заболевание при классической лимфоме Ходжкина» . Клинические исследования рака . 20 (1): 253–64. дои : 10.1158/1078-0432.CCR-13-1024 . ПМИД 24222179 .
- ^ Хоссейнали Н., Агапур М., Дуйф П.Х., Барадаран Б. (август 2018 г.). «Лечение рака заместительной терапией микроРНК: обзор литературы» . Журнал клеточной физиологии . 233 (8): 5574–5588. дои : 10.1002/jcp.26514 . ПМИД 29521426 . S2CID 3766576 .
- ^ Лю Дж., Сунь Ю., Цзи П., Ли Х., Когделл Д., Ян Д. и др. (июль 2014 г.). «МиР-506 подавляет пролиферацию и индуцирует старение, напрямую воздействуя на ось CDK4/6-FOXM1 при раке яичников» . Журнал патологии . 233 (3): 308–18. дои : 10.1002/путь.4348 . ПМК 4144705 . ПМИД 24604117 .
- ^ Вэнь С.Ю., Линь Ю., Ю Ю.К., Цао С.Дж., Чжан Р., Ян С.М. и др. (февраль 2015 г.). «МиР-506 действует как супрессор опухоли, напрямую воздействуя на фактор транскрипции Gli3 пути hedgehog при раке шейки матки человека» . Онкоген . 34 (6): 717–25. дои : 10.1038/onc.2014.9 . ПМИД 24608427 . S2CID 20603801 .
- ^ Пекинский университет (13 сентября 2022 г.). «Ученые разрабатывают новое мощное оружие для борьбы с раком» . Клетка . 185 (11). SciTech Daily : 1888–1904.e24. дои : 10.1016/j.cell.2022.04.030 . ПМИД 35623329 . S2CID 249070106 . Проверено 15 сентября 2022 г.
- ^ Леб К.Р., Леб Л.А. (март 2000 г.). «Значение множественных мутаций при раке» . Канцерогенез . 21 (3): 379–385. дои : 10.1093/carcin/21.3.379 . PMID 10688858 .
- ^ Лодиш Х., Берк А., Кайзер К.А., Кригер М., Бретшер А., Плох Х. и др. (2016). Молекулярно-клеточная биология (8-е изд.). Нью-Йорк: WH Freeman and Company. п. 203. ИСБН 978-1-4641-8339-3 .
- ^ Ху Х, Гатти Р.А. (июнь 2011 г.). «МикроРНК: новые игроки в реакции на повреждение ДНК» . Журнал молекулярно-клеточной биологии . 3 (3): 151–158. дои : 10.1093/jmcb/mjq042 . ПМК 3104011 . ПМИД 21183529 .
- ^ Джасперсон К.В., Туохи Т.М., Некласон Д.В., Берт Р.В. (июнь 2010 г.). «Наследственный и семейный рак толстой кишки» . Гастроэнтерология . 138 (6): 2044–58. дои : 10.1053/j.gastro.2010.01.054 . ПМК 3057468 . ПМИД 20420945 .
- ^ Трунингер К., Менигатти М., Луз Дж., Рассел А., Хайдер Р., Гебберс Дж.О. и др. (май 2005 г.). «Иммуногистохимический анализ выявляет высокую частоту дефектов PMS2 при колоректальном раке» . Гастроэнтерология . 128 (5): 1160–71. дои : 10.1053/j.gastro.2005.01.056 . ПМИД 15887099 .
- ^ Валери Н., Гаспарини П., Фаббри М., Бракони С., Веронезе А., Ловат Ф. и др. (апрель 2010 г.). «Модуляция репарации ошибочных спариваний и стабильности генома с помощью миР-155» . Труды Национальной академии наук Соединенных Штатов Америки . 107 (15): 6982–7. Бибкод : 2010PNAS..107.6982V . дои : 10.1073/pnas.1002472107 . ПМЦ 2872463 . ПМИД 20351277 .
- ^ Перейти обратно: а б с Чжан В., Чжан Дж., Ходли К., Кушваха Д., Рамакришнан В., Ли С. и др. (июнь 2012 г.). «миР-181d: прогнозирующий биомаркер глиобластомы, который подавляет экспрессию MGMT» . Нейроонкология . 14 (6): 712–9. дои : 10.1093/neuonc/nos089 . ПМЦ 3367855 . ПМИД 22570426 .
- ^ Шпигль-Крейнекер С., Пиркер С., Филипитс М., Лётч Д., Бухройтнер Дж., Пихлер Дж. и др. (январь 2010 г.). «Экспрессия белка O6-метилгуанин-ДНК-метилтрансферазы в опухолевых клетках предсказывает результат терапии темозоломидом у пациентов с глиобластомой» . Нейроонкология . 12 (1): 28–36. дои : 10.1093/neuonc/nop003 . ПМЦ 2940563 . ПМИД 20150365 .
- ^ Сгарра Р., Рустиги А., Тессари М.А., Ди Бернардо Дж., Альтамура С., Фуско А. и др. (сентябрь 2004 г.). «Ядерные фосфопротеины HMGA и их связь со структурой хроматина и раком». Письма ФЭБС . 574 (1–3): 1–8. дои : 10.1016/j.febslet.2004.08.013 . ПМИД 15358530 . S2CID 28903539 .
- ^ Сюй Ю, Самтер Т.Ф., Бхаттачарья Р., Тесфай А., Фукс Э.Дж., Вуд Л.Дж. и др. (май 2004 г.). «Онкоген HMG-I вызывает высоко проникающие агрессивные лимфоидные злокачественные новообразования у трансгенных мышей и сверхэкспрессируется при лейкемии человека» . Исследования рака . 64 (10): 3371–5. дои : 10.1158/0008-5472.CAN-04-0044 . ПМИД 15150086 .
- ^ Боррманн Л., Шванбек Р., Хейдук Т., Зеебек Б., Рогалла П., Буллердик Дж. и др. (декабрь 2003 г.). «Белок группы А2 с высокой подвижностью и его производные связывают конкретную область промотора гена репарации ДНК ERCC1 и модулируют его активность» . Исследования нуклеиновых кислот . 31 (23): 6841–51. дои : 10.1093/nar/gkg884 . ПМК 290254 . ПМИД 14627817 .
- ^ Фациста А., Нгуен Х., Льюис С., Прасад А.Р., Рэмси Л., Зайтлин Б. и др. (апрель 2012 г.). «Недостаточная экспрессия ферментов репарации ДНК на ранней стадии развития спорадического рака толстой кишки» . Целостность генома . 3 (1): 3. дои : 10.1186/2041-9414-3-3 . ПМК 3351028 . ПМИД 22494821 .
- ^ Жибер Б., Деллуа-Буржуа С., Гаттолья Ш., Меретт О., Ле Герневель С., Фомбонн Дж. и др. (ноябрь 2014 г.). «Регуляция с помощью семейства miR181 опухолевой супрессорной активности рецептора зависимости CDON при нейробластоме» . Журнал Национального института рака . 106 (11). дои : 10.1093/jnci/dju318 . ПМИД 25313246 .
- ^ Чен Дж.Ф., Мерчисон Э.П. , Тан Р., Каллис Т.Э., Тацугучи М., Дэн З. и др. (февраль 2008 г.). «Направленное удаление Dicer в сердце приводит к дилатационной кардиомиопатии и сердечной недостаточности» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (6): 2111–6. Бибкод : 2008PNAS..105.2111C . дои : 10.1073/pnas.0710228105 . ПМК 2542870 . ПМИД 18256189 .
- ^ Перейти обратно: а б Чжао Ю., Рэнсом Дж. Ф., Ли А., Ведантам В., фон Дреле М., Мут А.Н. и др. (апрель 2007 г.). «Нарушение регуляции кардиогенеза, сердечной проводимости и клеточного цикла у мышей, лишенных микроРНК-1-2» . Клетка . 129 (2): 303–17. дои : 10.1016/j.cell.2007.03.030 . ПМИД 17397913 .
- ^ Тум Т., Галуппо П., Вольф С., Фидлер Дж., Кнейтц С., ван Лааке Л.В. и др. (июль 2007 г.). «МикроРНК в сердце человека: ключ к перепрограммированию генов плода при сердечной недостаточности» . Тираж . 116 (3): 258–67. doi : 10.1161/CIRCULATIONAHA.107.687947 . ПМИД 17606841 .
- ^ ван Рой Э., Сазерленд Л.Б., Лю Н., Уильямс А.Х., Макэналли Дж., Джерард Р.Д. и др. (ноябрь 2006 г.). «Характерный образец стресс-чувствительных микроРНК, которые могут вызвать гипертрофию сердца и сердечную недостаточность» . Труды Национальной академии наук Соединенных Штатов Америки . 103 (48): 18255–60. Бибкод : 2006PNAS..10318255V . дои : 10.1073/pnas.0608791103 . ПМЦ 1838739 . ПМИД 17108080 .
- ^ Тацугучи М., Сок Х.И., Каллис Т.Э., Томсон Дж.М., Чен Дж.Ф., Ньюман М. и др. (июнь 2007 г.). «Экспрессия микроРНК динамически регулируется во время гипертрофии кардиомиоцитов» . Журнал молекулярной и клеточной кардиологии . 42 (6): 1137–41. дои : 10.1016/j.yjmcc.2007.04.004 . ЧВК 1934409 . ПМИД 17498736 .
- ^ Чжао Ю, Самал Э, Шривастава Д (июль 2005 г.). «Фактор ответа сыворотки регулирует специфическую для мышц микроРНК, которая нацелена на Hand2 во время кардиогенеза». Природа . 436 (7048): 214–20. Бибкод : 2005Natur.436..214Z . дои : 10.1038/nature03817 . ПМИД 15951802 . S2CID 4340449 .
- ^ Сяо Дж., Луо Икс, Линь Х., Чжан Ю., Лу Ю., Ван Н. и др. (апрель 2007 г.). «МикроРНК миР-133 подавляет экспрессию канала HERG K+, способствуя удлинению интервала QT в диабетическом сердце» . Журнал биологической химии . 282 (17): 12363–7. дои : 10.1074/jbc.C700015200 . ПМК 3151106 . ПМИД 17344217 . (Отозвано, см. дои : 10.1074/jbc.A111.700015 , PMID 21961173 , Часы втягивания )
- ^ Ян Б., Линь Х., Сяо Дж., Лу Ю., Луо Икс., Ли Б. и др. (апрель 2007 г.). «Мышечная специфичная микроРНК миР-1 регулирует сердечный аритмогенный потенциал, воздействуя на GJA1 и KCNJ2». Природная медицина . 13 (4): 486–91. дои : 10.1038/nm1569 . ПМИД 17401374 . S2CID 1935811 .
- ^ Каре А., Каталуччи Д., Феличетти Ф., Бончи Д., Аддарио А., Галло П. и др. (май 2007 г.). «МикроРНК-133 контролирует гипертрофию сердца». Природная медицина . 13 (5): 613–8. дои : 10.1038/nm1582 . ПМИД 17468766 . S2CID 10097893 .
- ^ ван Рой Э., Сазерленд Л.Б., Ци Икс, Ричардсон Дж.А., Хилл Дж., Олсон Э.Н. (апрель 2007 г.). «Контроль стресс-зависимого роста сердца и экспрессии генов с помощью микроРНК». Наука . 316 (5824): 575–9. Бибкод : 2007Sci...316..575V . дои : 10.1126/science.1139089 . ПМИД 17379774 . S2CID 1927839 .
- ^ Келлер Т., Бекель Дж.Н., Гросс С., Клотше Дж., Палапис Л., Лейстнер Д. и др. (июль 2017 г.). «Улучшенная стратификация риска при профилактике за счет использования панели выбранных циркулирующих микроРНК» . Научные отчеты . 7 (1): 4511. Бибкод : 2017НатСР...7.4511К . дои : 10.1038/s41598-017-04040-w . ПМЦ 5495799 . ПМИД 28674420 .
- ^ Инсулл В. (январь 2009 г.). «Патология атеросклероза: развитие бляшек и реакция бляшек на лечение». Американский медицинский журнал . 122 (1 дополнение): S3 – S14. дои : 10.1016/j.amjmed.2008.10.013 . ПМИД 19110086 .
- ^ Перейти обратно: а б с д и ж г час я дж к л м н тот п д Сон Д.Д., Кумар С., Такабэ В., Ким К.В., Ни К.В., Альбертс-Гриль Н. и др. (2013). «Атипичная механочувствительная микроРНК-712, полученная из прерибосомальной РНК, вызывает воспаление эндотелия и атеросклероз» . Природные коммуникации . 4 : 3000. Бибкод : 2013NatCo...4.3000S . дои : 10.1038/ncomms4000 . ПМЦ 3923891 . ПМИД 24346612 .
- ^ Перейти обратно: а б Басу Р., Фан Д., Кандалам В., Ли Дж., Дас С.К., Ван Х и др. (декабрь 2012 г.). «Потеря гена Timp3 приводит к образованию аневризмы брюшной аорты в ответ на ангиотензин II» . Журнал биологической химии . 287 (53): 44083–96. дои : 10.1074/jbc.M112.425652 . ПМЦ 3531724 . ПМИД 23144462 .
- ^ Либби П. (2002). «Воспаление при атеросклерозе». Природа . 420 (6917): 868–74. Бибкод : 2002Natur.420..868L . дои : 10.1038/nature01323 . ПМИД 12490960 . S2CID 407449 .
- ^ Перейти обратно: а б Фуа Ю.Л., Чу Дж.И., Марроне А.К., Боднар А.Дж., Симс-Лукас С., Хо Дж. (октябрь 2015 г.). «Почечные стромальные микроРНК необходимы для нормального нефрогенеза и выживания гломерулярной мезангии» . Физиологические отчеты . 3 (10): е12537. дои : 10.14814/phy2.12537 . ПМЦ 4632944 . ПМИД 26438731 .
- ^ Цао Д.Д., Ли Л., Чан Вайоминг (май 2016 г.). «МикроРНК: ключевые регуляторы центральной нервной системы и их значение при неврологических заболеваниях» . Международный журнал молекулярных наук . 17 (6): 842. doi : 10.3390/ijms17060842 . ПМЦ 4926376 . ПМИД 27240359 .
- ^ Ланг М.Ф., Ши Ю (2012). «Динамическая роль микроРНК в нейрогенезе» . Границы в неврологии . 6 : 71. дои : 10.3389/fnins.2012.00071 . ПМК 3356852 . ПМИД 22661924 .
- ^ Шратт Дж. (декабрь 2009 г.). «микроРНК в синапсе». Обзоры природы. Нейронаука . 10 (12): 842–849. дои : 10.1038/nrn2763 . ПМИД 19888283 . S2CID 3507952 .
- ^ Луо М, Ли Л, Дин М, Ню Ю, Сюй Х, Ши Х и др. (19 января 2023 г.). «С помощью секвенирования микроРНК и биоинформатического анализа были выявлены микроРНК, регулирующие долговременное потенцирование и депрессию при нарушениях обучения и памяти, вызванных бисфенолом А» . ПЛОС ОДИН . 18 (1): e0279029. Бибкод : 2023PLoSO..1879029L . дои : 10.1371/journal.pone.0279029 . ПМЦ 9851566 . PMID 36656826 .
- ^ Куэльяр Т.Л., Дэвис Т.Х., Нельсон П.Т., Леб Г.Б., Харф Б.Д., Уллиан Э. и др. (апрель 2008 г.). «Потеря Dicer в нейронах полосатого тела приводит к поведенческим и нейроанатомическим фенотипам при отсутствии нейродегенерации» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (14): 5614–5619. Бибкод : 2008PNAS..105.5614C . дои : 10.1073/pnas.0801689105 . ПМК 2291142 . ПМИД 18385371 .
- ^ Эбер С.С., Пападопулу А.С., Смит П., Галас М.К., Планель Э., Силахтароглу А.Н. и др. (октябрь 2010 г.). «Генетическая абляция Dicer в нейронах переднего мозга взрослых приводит к аномальному гиперфосфорилированию тау и нейродегенерации» . Молекулярная генетика человека . 19 (20): 3959–3969. дои : 10.1093/hmg/ddq311 . ПМИД 20660113 .
- ^ Рой Б., Ли Э., Ли Т., Рамперсауд М. (февраль 2022 г.). «Роль микроРНК в нейродегенерации: от причины заболевания к инструментам открытия биомаркеров и терапии» . Гены . 13 (3): 425. doi : 10.3390/genes13030425 . ПМЦ 8951370 . ПМИД 35327979 .
- ^ Хеншолл Д.К., Хамер Х.М., Пастеркамп Р.Дж., Гольдштейн Д.Б., Кджемс Дж., Прен Дж.Х. и др. (декабрь 2016 г.). «МикроРНК при эпилепсии: патофизиология и клиническое применение» . «Ланцет». Неврология . 15 (13): 1368–1376. дои : 10.1016/S1474-4422(16)30246-0 . ПМИД 27839653 .
- ^ Хоммерс Л.Г., Домшке К., Декерт Дж. (январь 2015 г.). «Гетерогенность и индивидуальность: микроРНК при психических расстройствах» (PDF) . Журнал нейронной передачи . 122 (1): 79–97. дои : 10.1007/s00702-014-1338-4 . ПМИД 25395183 . S2CID 25088900 . Архивировано (PDF) из оригинала 23 мая 2022 года.
- ^ Фэн Дж., Сунь Г., Ян Дж., Нолтнер К., Ли В., Бузин CH. и др. (июль 2009 г.). Рейф А (ред.). «Доказательства Х-хромосомной шизофрении, связанной с изменениями микроРНК» . ПЛОС ОДИН . 4 (7): е6121. Бибкод : 2009PLoSO...4.6121F . дои : 10.1371/journal.pone.0006121 . ПМК 2699475 . ПМИД 19568434 .
- ^ Беверидж, Нью-Джерси, Гардинер Э., Кэрролл А.П., Туни П.А., Кэрнс М.Дж. (декабрь 2010 г.). «Шизофрения связана с увеличением биогенеза кортикальных микроРНК» . Молекулярная психиатрия . 15 (12): 1176–1189. дои : 10.1038/mp.2009.84 . ПМК 2990188 . ПМИД 19721432 .
- ^ «Факты об инсульте» . Центры по контролю и профилактике заболеваний . 15 марта 2019 года . Проверено 5 декабря 2019 г.
- ^ Ринк С., Ханна С. (май 2011 г.). «МикроРНК в этиологии и патологии ишемического инсульта» . Физиологическая геномика . 43 (10): 521–528. doi : 10.1152/физиологгеномика.00158.2010 . ПМК 3110894 . ПМИД 20841499 .
- ^ Оуян Ю.Б., Старый К.М., Ян Г.И., Гиффард Р. (январь 2013 г.). «МикроРНК: инновационные мишени для лечения ишемии головного мозга и инсульта» . Текущие цели по борьбе с наркотиками . 14 (1): 90–101. дои : 10.2174/138945013804806424 . ПМЦ 3673881 . ПМИД 23170800 .
- ^ Перейти обратно: а б с Левол Дж. М., Нуньес Ю. О., Додд П. Р., Тивари Г. Р., Харрис Р. А., Мэйфилд Р. Д. (ноябрь 2011 г.). «Повышающая регуляция микроРНК в мозгу алкоголиков» . Алкоголизм: клинические и экспериментальные исследования . 35 (11): 1928–37. дои : 10.1111/j.1530-0277.2011.01544.x . ПМК 3170679 . ПМИД 21651580 .
- ^ Тапочик Дж.Д., Соломон М., Фланиган М., Мейнхардт М., Барбье Э., Шанк Дж.Р. и др. (июнь 2013 г.). «Координированная дисрегуляция мРНК и микроРНК в медиальной префронтальной коре крыс после алкогольной зависимости» . Журнал «Фармакогеномика» . 13 (3): 286–96. дои : 10.1038/tpj.2012.17 . ПМЦ 3546132 . ПМИД 22614244 .
- ^ Горини Дж., Нуньес Ю.О., Мэйфилд Р.Д. (2013). «Интеграция микроРНК и профилирование белков выявляет скоординированные нейроадаптации в мозге зависимых от алкоголя мышей» . ПЛОС ОДИН . 8 (12): е82565. Бибкод : 2013PLoSO...882565G . дои : 10.1371/journal.pone.0082565 . ПМЦ 3865091 . ПМИД 24358208 .
- ^ Тапокик Дж.Д., Барбье Э., Фланиган М., Соломон М., Пинкус А., Пиллинг А. и др. (март 2014 г.). «МикроРНК-206 в медиальной префронтальной коре крыс регулирует экспрессию BDNF и употребление алкоголя» . Журнал неврологии . 34 (13): 4581–8. doi : 10.1523/JNEUROSCI.0445-14.2014 . ПМЦ 3965783 . ПМИД 24672003 .
- ^ Липпай Д., Бала С., Чак Т., Курт-Джонс Э.А., Сабо Г. (2013). «Хроническая микроРНК-155, индуцированная алкоголем, способствует нейровоспалению TLR4-зависимым образом у мышей» . ПЛОС ОДИН . 8 (8): е70945. Бибкод : 2013PLoSO...870945L . дои : 10.1371/journal.pone.0070945 . ПМЦ 3739772 . ПМИД 23951048 .
- ^ Перейти обратно: а б Ли Дж, Ли Дж, Лю Икс, Цинь С, Гуань Ю, Лю Ю и др. (сентябрь 2013 г.). «Профиль экспрессии микроРНК и функциональный анализ показывают, что миР-382 является критически важным новым геном алкогольной зависимости» . ЭМБО Молекулярная медицина . 5 (9): 1402–14. дои : 10.1002/emmm.201201900 . ПМК 3799494 . ПМИД 23873704 .
- ^ Ромао Дж. М., Джин В., Додсон М. В., Хаусман Г. Дж., Мур С. С., Гуан Л. Л. (сентябрь 2011 г.). «Регуляция микроРНК в адипогенезе млекопитающих». Экспериментальная биология и медицина . 236 (9): 997–1004. дои : 10.1258/ebm.2011.011101 . ПМИД 21844119 . S2CID 30646787 .
- ^ Скорн М., Намлёс Х.М., Нордхус П., Ван М.Ю., Меза-Сепеда Л.А., Миклебост О. (апрель 2012 г.). «Дифференцировка адипоцитов стромальных клеток костного мозга человека модулируется микроРНК-155, микроРНК-221 и микроРНК-222». Стволовые клетки и развитие . 21 (6): 873–83. дои : 10.1089/scd.2010.0503 . hdl : 10852/40423 . ПМИД 21756067 .
- ^ Цзо Ю, Цян Л, фермер-старший (март 2006 г.). «Активация экспрессии CCAAT/энхансер-связывающего белка (C/EBP) альфа с помощью C/EBP бета во время адипогенеза требует активируемой пролифератором пероксисомы гамма-ассоциированной репрессии HDAC1 на промоторе гена C/ebp альфа» . Журнал биологической химии . 281 (12): 7960–7. дои : 10.1074/jbc.M510682200 . ПМИД 16431920 .
- ^ Джун-Хао ET, Гупта Р.Р., Шых-Чанг Н. (март 2016 г.). «Lin28 и let-7 в метаболической физиологии старения». Тенденции в эндокринологии и обмене веществ . 27 (3): 132–141. дои : 10.1016/j.tem.2015.12.006 . ПМИД 26811207 . S2CID 3614126 .
- ^ Чжу Х., Шых-Чанг Н., Сегре А.В., Шинода Г., Шах С.П., Эйнхорн В.С. и др. (сентябрь 2011 г.). «Ось Lin28/let-7 регулирует метаболизм глюкозы» . Клетка . 147 (1): 81–94. дои : 10.1016/j.cell.2011.08.033 . ПМЦ 3353524 . ПМИД 21962509 .
- ^ Фрост Р.Дж., Олсон Э.Н. (декабрь 2011 г.). «Контроль гомеостаза глюкозы и чувствительности к инсулину с помощью семейства микроРНК Let-7» . Труды Национальной академии наук Соединенных Штатов Америки . 108 (52): 21075–80. Бибкод : 2011PNAS..10821075F . дои : 10.1073/pnas.1118922109 . ПМЦ 3248488 . ПМИД 22160727 .
- ^ Теруэль-Монтойя Р., Розендал Ф.Р., Мартинес К. (февраль 2015 г.). «МикроРНК в гемостазе» . Журнал тромбозов и гемостаза . 13 (2): 170–181. дои : 10.1111/jth.12788 . ПМИД 25400249 .
- ^ Перейти обратно: а б Нурс Дж., Браун Дж., Лакнер К., Хюттельмайер С., Данквардт С. (ноябрь 2018 г.). «Крупномасштабная идентификация функционального нацеливания микроРНК выявляет совместную регуляцию системы гемостаза» . Журнал тромбозов и гемостаза . 16 (11): 2233–2245. дои : 10.1111/jth.14290 . ПМИД 30207063 .
- ^ Нурс Дж., Данквардт С. (февраль 2021 г.). «Новое обоснование нацеливания на FXI: данные о гемостатическом мишени микроРНК для новых стратегий антикоагулянтов» . Фармакология и терапия . 218 : 107676. doi : 10.1016/j.pharmthera.2020.107676 . ПМИД 32898547 .
- ^ Берарди Э., Пуес М., Торрес Л., Сампаолеси М. (октябрь 2012 г.). «миРНК в дифференцировке ЭСК». Американский журнал физиологии. Физиология сердца и кровообращения . 303 (8): H931–H939. дои : 10.1152/ajpheart.00338.2012 . ПМИД 22886416 . S2CID 6402014 .
- ^ Перейти обратно: а б Борчерс А., Пилер Т. (ноябрь 2010 г.). «Программирование плюрипотентных клеток-предшественников, полученных из эмбрионов Xenopus, для создания специфических тканей и органов» . Гены . 1 (3): 413–426. дои : 10.3390/ijms232314755 . ПМК 9740008 . ПМИД 36499082 .
- ^ Кураба Дж., Сприггс А., Тейлор Дж., Ли З., Хелливелл С. (июль 2012 г.). «Регуляция микроРНК на раннем этапе развития семян ячменя» . Биология растений BMC . 12 (1): 120. дои : 10.1186/1471-2229-12-120 . ПМК 3443071 . ПМИД 22838835 .
- ^ Чоудхари А., Кумар А., Каур Х., Каур Н. (май 2021 г.). «МиРНК: надсмотрщик растительного мира». Биология . 76 (5): 1551–1567. Бибкод : 2021Биолг..76.1551С . дои : 10.1007/s11756-021-00720-1 . ISSN 1336-9563 . S2CID 233685660 .
- ^ Вахид С., Цзэн Л. (март 2020 г.). «Критическая роль микроРНК в регуляции времени цветения и развития цветов» . Гены . 11 (3): 319. doi : 10.3390/genes11030319 . ПМК 7140873 . ПМИД 32192095 .
- ^ Вонг CE, Чжао Ю.Т., Ван XJ, Крофт Л., Ван Ч.Х., Хаэризаде Ф. и др. (май 2011 г.). «МикроРНК в апикальной меристеме побега сои». Журнал экспериментальной ботаники . 62 (8): 2495–2506. дои : 10.1093/jxb/erq437 . hdl : 10536/DRO/DU:30106047 . ПМИД 21504877 .
- ^ Куреши А., Тхакур Н., Монга И., Тхакур А., Кумар М. (1 января 2014 г.). «VIRmiRNA: комплексный ресурс экспериментально подтвержденных вирусных микроРНК и их мишеней» . База данных . 2014 : бау103. дои : 10.1093/база данных/bau103 . ПМК 4224276 . ПМИД 25380780 .
- ^ Кумар М. «ВИРмиРНК» . Ресурс экспериментальных вирусных микроРНК и их мишеней . Центр биоинформатики, ЦНИР-ИМТЕХ.
- ^ Тадденхэм Л., Юнг Дж.С., Чейн-Вун-Минг Б., Долкен Л., Пфеффер С. (февраль 2012 г.). «Глубокое секвенирование малых РНК идентифицирует микроРНК и другие малые некодирующие РНК вируса герпеса человека 6B» . Журнал вирусологии . 86 (3): 1638–49. дои : 10.1128/JVI.05911-11 . ПМК 3264354 . ПМИД 22114334 .
- ^ Чжэн Х., Фу Р., Ван Дж.Т., Лю Ц., Чэнь Х., Цзян С.В. (апрель 2013 г.). «Достижения в методах прогнозирования мишеней микроРНК» . Международный журнал молекулярных наук . 14 (4): 8179–87. дои : 10.3390/ijms14048179 . ПМЦ 3645737 . ПМИД 23591837 .
- ^ Агарвал В., Белл Г.В., Нам Дж.В., Бартель Д.П. (август 2015 г.). «Прогнозирование эффективных целевых участков микроРНК в мРНК млекопитающих» . электронная жизнь . 4 : e05005. doi : 10.7554/eLife.05005 . ПМЦ 4532895 . ПМИД 26267216 .
Дальнейшее чтение
[ редактировать ]- Определение и классификация микроРНК: Амброс В., Бартель Б., Бартель Д.П., Бердж С.Б., Кэррингтон Дж.К., Чен X и др. (март 2003 г.). «Единая система аннотации микроРНК» . РНК . 9 (3): 277–9. дои : 10.1261/rna.2183803 . ПМЦ 1370393 . ПМИД 12592000 .
- Научный обзор малых РНК: Баулкомб Д. (сентябрь 2002 г.). «ДНК-события. Микрокосм РНК». Наука . 297 (5589): 2002–3. дои : 10.1126/science.1077906 . ПМИД 12242426 . S2CID 82531727 .
- Открытие lin-4 , первой обнаруженной микроРНК: Ли Р.К., Фейнбаум Р.Л., Амброс В. (декабрь 1993 г.). «Гетерохронный ген lin-4 C. elegans кодирует малые РНК с антисмысловой комплементарностью lin-14» . Клетка . 75 (5): 843–54. дои : 10.1016/0092-8674(93)90529-Y . ПМИД 8252621 .
Внешние ссылки
[ редактировать ]- База данных miRBase
- miRTarBase — экспериментально подтвержденная база данных взаимодействий микроРНК-мишени.
- semirna , веб-приложение для поиска микроРНК в геноме растений.
- ONCO.IO : Интегративный ресурс для анализа микроРНК и факторов транскрипции при раке.
- MirOB Архивировано 4 марта 2014 г. в Wayback Machine : MicroRNA нацелена на базу данных, инструмент для анализа и визуализации данных.
- База данных ChIPBase : база данных с открытым доступом для расшифровки факторов транскрипции , которые участвовали в транскрипции микроРНК или влияли на нее, из данных ChIP-seq.
- Анимированное видео процесса биогенеза микроРНК .
- Реагенты для модуляции микроРНК , обеспечивающие усиление или подавление функции эндогенной зрелой микроРНК.