Гаплогруппа R1a
| Гаплогруппа R1a | |
|---|---|
 | |
| Возможное время происхождения | 22,000 [1] до 25 000 [2] много лет назад |
| Возможное место происхождения | Евразия |
| Предок | Гаплогруппа R1 |
| Потомки | R1a-Z282, R1a-Z93 |
| Определение мутаций |
|
| Самые высокие частоты | См. Список частот R1a по населению. |

Гаплогруппа R1a , или гаплогруппа R-M420 , представляет собой гаплогруппу ДНК Y-хромосомы человека , которая распространена в большом регионе Евразии , простирающемся от Скандинавии и Центральной Европы до Центральной Азии , Южной Сибири и Южной Азии . [3] [2]
Хотя одно генетическое исследование показывает, что R1a возник 25 000 [2] лет назад его субклад M417 (R1a1a1) диверсифицировал ок. 5800 лет назад. [4] Место происхождения субклада играет роль в споре о происхождении протоиндоевропейцев .
Мутация SNP R-M420 была обнаружена после R-M17 (R1a1a), что привело к реорганизации линии, в частности к созданию новой парагруппы (обозначенной R-M420*) для относительно редких линий, не входящих в R-SRY10831. .2 (R1a1) ответвление, ведущее к R-M17.
Происхождение
[ редактировать ]R1a origins
[edit]The genetic divergence of R1a (M420) is estimated to have occurred 25,000[2] years ago, which is the time of the last glacial maximum. A 2014 study by Peter A. Underhill et al., using 16,244 individuals from over 126 populations from across Eurasia, concluded that there was "a compelling case for the Middle East, possibly near present-day Iran, as the geographic origin of hg R1a".[2] The ancient DNA record has shown the first R1a during the Mesolithic in Eastern Hunter-Gatherers (from Eastern Europe, c. 13,000 years ago),[5][6] and the earliest case of R* among Upper Paleolithic Ancient North Eurasians,[7] from which the Eastern Hunter-Gatherers predominantly derive their ancestry.[8]
Diversification of R1a1a1 (M417) and ancient migrations
[edit]
According to Underhill et al. (2014), the downstream R1a-M417 subclade diversified into Z282 and Z93 circa 5,800 years ago "in the vicinity of Iran and Eastern Turkey".[4][note 1] Even though R1a occurs as a Y-chromosome haplogroup among various languages such as Slavic and Indo-Iranian, the question of the origins of R1a1a is relevant to the ongoing debate concerning the urheimat of the Proto-Indo-European people, and may also be relevant to the origins of the Indus Valley civilization. R1a shows a strong correlation with Indo-European languages of Southern and Western Asia, Central and Eastern Europe and to Scandinavia[10][3] being most prevalent in Eastern Europe, West Asia, and South Asia. In Europe, Z282 is prevalent particularly while in Asia Z93 dominates. The connection between Y-DNA R-M17 and the spread of Indo-European languages was first noted by T. Zerjal and colleagues in 1999.[11]
Indo-European relation
[edit]Proposed steppe dispersal of R1a1a
[edit]Semino et al. (2000) proposed Ukrainian origins, and a postglacial spread of the R1a1 haplogroup during the Late Glacial Maximum, subsequently magnified by the expansion of the Kurgan culture into Europe and eastward.[12] Spencer Wells proposes Central Asian origins, suggesting that the distribution and age of R1a1 points to an ancient migration corresponding to the spread by the Kurgan people in their expansion from the Eurasian steppe.[13] According to Pamjav et al. (2012), R1a1a diversified in the Eurasian Steppes or the Middle East and Caucasus region:
Inner and Central Asia is an overlap zone for the R1a1-Z280 and R1a1-Z93 lineages [which] implies that an early differentiation zone of R1a1-M198 conceivably occurred somewhere within the Eurasian Steppes or the Middle East and Caucasus region as they lie between South Asia and Central- and Eastern Europe.[14]
Three genetic studies in 2015 gave support to the Kurgan theory of Gimbutas regarding the Indo-European Urheimat. According to those studies, haplogroups R1b and R1a, now the most common in Europe (R1a is also common in South Asia) would have expanded from the Pontic–Caspian steppes, along with the Indo-European languages; they also detected an autosomal component present in modern Europeans which was not present in Neolithic Europeans, which would have been introduced with paternal lineages R1b and R1a, as well as Indo-European languages.[15][16][17]
Silva et al. (2017) noted that R1a in South Asia most "likely spread from a single Central Asian source pool, there do seem to be at least three and probably more R1a founder clades within the Indian subcontinent, consistent with multiple waves of arrival."[18] According to Martin P. Richards, co-author of Silva et al. (2017), the prevalence of R1a in India was "very powerful evidence for a substantial Bronze Age migration from central Asia that most likely brought Indo-European speakers to India."[19][note 2]
Possible Yamnaya or Corded Ware origins
[edit]

David Anthony considers the Yamnaya culture to be the Indo-European Urheimat.[20][21] According to Haak et al. (2015), a massive migration from the Yamnaya culture northwards took place c. 2,500 BCE, accounting for 75% of the genetic ancestry of the Corded Ware culture, noting that R1a and R1b may have "spread into Europe from the East after 3,000 BCE".[22] Yet, all their seven Yamnaya samples belonged to the R1b-M269 subclade,[22] but no R1a1a has been found in their Yamnaya samples. This raises the question where the R1a1a in the Corded Ware culture came from, if it was not from the Yamnaya culture.[23]
According to Marc Haber, the absence of haplogroup R1a-M458 in Afghanistan does not support a Pontic-Caspian steppe origin for the R1a lineages in modern Central Asian populations.[24]
According to Leo Klejn, the absence of haplogroup R1a in Yamnaya remains (despite its presence in Eneolithic Samara and Eastern Hunter Gatherer populations) makes it unlikely that Europeans inherited haplogroup R1a from Yamnaya.[25]
Archaeologist Barry Cunliffe has said that the absence of haplogroup R1a in Yamnaya specimens is a major weakness in Haak's proposal that R1a has a Yamnaya origin.[26]
Semenov & Bulat (2016) do argue for a Yamnaya origin of R1a1a in the Corded Ware culture, noting that several publications point to the presence of R1a1 in the Comb Ware culture.[27][note 3]
Proposed South Asian origins
[edit]Kivisild et al. (2003) have proposed either South or West Asia,[28][note 4] while Mirabal et al. (2009) see support for both South and Central Asia.[10] Sengupta et al. (2006) have proposed Indian origins.[29] Thanseem et al. (2006) have proposed either South or Central Asia.[30] Sahoo et al. (2006) have proposed either South or West Asia.[31] Thangaraj et al. (2010) have also proposed a South Asian origin.[32] Sharma et al.(2009) theorizes the existence of R1a in India beyond 18,000 years to possibly 44,000 years in origin.[1]
A number of studies from 2006 to 2010 concluded that South Asian populations have the highest STR diversity within R1a1a,[33][34][10][3][1][35] and subsequent older TMRCA datings.[note 5] R1a1a is present among both higher (Brahmin) castes and lower castes, and while the frequency is higher among Brahmin castes, the oldest TMRCA datings of the R1a haplogroup occur in the Saharia tribe, a scheduled caste of the Bundelkhand region of Central India.[1][35]
From these findings some researchers argued that R1a1a originated in South Asia,[34][1][note 6] excluding a more recent, yet minor, genetic influx from Indo-European migrants in northwestern regions such as Afghanistan, Balochistan, Punjab, and Kashmir.[34][33][3][note 7]
The conclusion that R1a originated in India has been questioned by more recent research,[18][37][note 8] offering proof that R1a arrived in India with multiple waves of migration.[18][38]
Proposed Transcaucasia and West Asian origins and possible influence on Indus Valley Civilization
[edit]Haak et al. (2015) found that part of the Yamnaya ancestry derived from the Middle East and that neolithic techniques probably arrived at the Yamnaya culture from the Balkans.[note 9] The Rössen culture (4,600–4,300 BC), which was situated on Germany and predates the Corded Ware culture, an old subclade of R1a, namely L664, can still be found.[note 10]
Part of the South Asian genetic ancestry derives from west Eurasian populations, and some researchers have implied that Z93 may have come to India via Iran[40] and expanded there during the Indus Valley civilization.[2][41]
Mascarenhas et al. (2015) proposed that the roots of Z93 lie in West Asia, and proposed that "Z93 and L342.2 expanded in a southeasterly direction from Transcaucasia into South Asia",[40] noting that such an expansion is compatible with "the archeological records of eastward expansion of West Asian populations in the 4th millennium BCE culminating in the so-called Kura-Araxes migrations in the post-Uruk IV period."[40] Yet, Lazaridis noted that sample I1635 of Lazaridis et al. (2016), their Armenian Kura-Araxes sample, carried Y-haplogroup R1b1-M415(xM269)[note 11] (also called R1b1a1b-CTS3187).[42][unreliable source?]
According to Underhill et al. (2014) the diversification of Z93 and the "early urbanization within the Indus Valley ... occurred at [5,600 years ago] and the geographic distribution of R1a-M780 (Figure 3d[note 12]) may reflect this."[2][note 13] Poznik et al. (2016) note that "striking expansions" occurred within R1a-Z93 at c. 4,500–4,000 years ago, which "predates by a few centuries the collapse of the Indus Valley Civilisation."[41][note 14]
However, according to Narasimhan et al. (2018), steppe pastoralists are a likely source for R1a in India.[44][note 15]
Phylogeny
[edit]The R1a family tree now has three major levels of branching, with the largest number of defined subclades within the dominant and best known branch, R1a1a (which will be found with various names such as "R1a1" in relatively recent but not the latest literature).
Topology
[edit]The topology of R1a is as follows (codes [in brackets] non-isogg codes):[9][45][verification needed][46][2][47] Tatiana et al. (2014) "rapid diversification process of K-M526 likely occurred in Southeast Asia, with subsequent westward expansions of the ancestors of haplogroups R and Q."[48]
- P P295/PF5866/S8 (also known as K2b2).
- R (R-M207)[46][9]
- R*
- R1 (R-M173)
- R1*[46]
- R1a (M420)[46] (Eastern Europe, Asia)[2]
- R1a*[9]
- R1a1[46] (M459/PF6235,[46] SRY1532.2/SRY10831.2[46])
- R1a1 (M459)[46][9]
- R1a1a (M17, M198)[46]
- R1a1a1 (M417, page7)[46]
- R1a1a1a (CTS7083/L664/S298)[46]
- R1a1a1b (S224/Z645, S441/Z647)[46]
- R1a1a1b1 (PF6217/S339/Z283)[46]
- R1a1a1b1a (Z282)[46] [R1a1a1a*] (Z282) [49] (Eastern Europe)
- R1a1a1b1a1[46] [The old topological code is R1a1a1b*,which is outdated and might lead to some confusion.][49] (M458)[46][49] [R1a1a1g] (M458)[47]
- R1a1a1b1a2[46] (S466/Z280, S204/Z91)[46]
- R1a1a1b1a2a[46]
- R1a1a1b1a2b (CTS1211)[46] [R1a1a1c*] (M558)[49] [R-CTS1211] (V2803/CTS3607/S3363/M558, CTS1211/S3357, Y34/FGC36457)[9]
- R1a1a1b1a2b3* (M417+, Z645+, Z283+, Z282+, Z280+, CTS1211+, CTS3402, Y33+, CTS3318+, Y2613+) (Gwozdz's Cluster K)[45][verification needed]
- R1a1a1b1a2b3a (L365/S468)[46]
- R1a1a1b1a3 (Z284)[46] [R1a1a1a1] (Z284)[49]
- R1a1a1b1a (Z282)[46] [R1a1a1a*] (Z282) [49] (Eastern Europe)
- R1a1a1b2 (F992/S202/Z93)[46] [R1a1a2*] (Z93, M746)[49] (Central Asia, South Asia and West Asia)
- R1a1a1b1 (PF6217/S339/Z283)[46]
- [R1a1a1c] (M64.2, M87, M204)[47]
- [R1a1a1d] (P98)[47]
- [R1a1a1d2a][50]
- [R1a1a1e] (PK5)[47]
- R1a1a1 (M417, page7)[46]
- R1b (M343) (Western Europe)
- R2 (India)
Haplogroup R
[edit]| Haplogroup R phylogeny |
R-M173 (R1)
[edit]R1a is distinguished by several unique markers, including the M420 mutation. It is a subclade of Haplogroup R-M173 (previously called R1). R1a has the sister-subclades Haplogroup R1b-M343, and the paragroup R-M173*.
R-M420 (R1a)
[edit]R-M420, defined by the mutation M420, has two branches: R-SRY1532.2, defined by the mutation SRY1532.2, which makes up the vast majority; and R-M420*, the paragroup, defined as M420 positive but SRY1532.2 negative. (In the 2002 scheme, this SRY1532.2 negative minority was one part of the relatively rare group classified as the paragroup R1*.) Mutations understood to be equivalent to M420 include M449, M511, M513, L62, and L63.[3][51]
Only isolated samples of the new paragroup R-M420* were found by Underhill 2009, mostly in the Middle East and Caucasus: 1/121 Omanis, 2/150 Iranians, 1/164 in the United Arab Emirates, and 3/612 in Turkey. Testing of 7224 more males in 73 other Eurasian populations showed no sign of this category.[3]
This paragroup is now known as R1a2 (R-YP4141). It then has two branches R1a2a (R-YP5018) and R1a2b (R-YP4132).
R-SRY1532.2 (R1a1)
[edit]R1a1 is defined by SRY1532.2 or SRY10831.2 (understood to always include SRY10831.2, M448, L122, M459, and M516[3][52]). This family of lineages is dominated by M17 and M198. In contrast, paragroup R-SRY1532.2* lacks either the M17 or M198 markers.
The R-SRY1532.2* paragroup is apparently less rare than R1*, but still relatively unusual, though it has been tested in more than one survey. Underhill et al. (2009) reported 1/51 in Norway, 3/305 in Sweden, 1/57 Greek Macedonians, 1/150 Iranians, 2/734 ethnic Armenians, and 1/141 Kabardians.[3] Sahoo et al. (2006) reported R-SRY1532.2* for 1/15 Himachal Pradesh Rajput samples.[34]
R-M17/M198 (R1a1a)
[edit]The following SNPs are associated with R1a1a:
| SNP | Mutation | Y-position (NCBI36) | Y-position (GRCh37) | RefSNP ID |
|---|---|---|---|---|
| M17 | INS G | 20192556 | 21733168 | rs3908 |
| M198 | C->T | 13540146 | 15030752 | rs2020857 |
| M512 | C->T | 14824547 | 16315153 | rs17222146 |
| M514 | C->T | 17884688 | 19375294 | rs17315926 |
| M515 | T->A | 12564623 | 14054623 | rs17221601 |
| L168 | A->G | 14711571 | 16202177 | - |
| L449 | C->T | 21376144 | 22966756 | - |
| L457 | G->A | 14946266 | 16436872 | rs113195541 |
| L566 | C->T | - | - | - |
R-M417 (R1a1a1)
[edit]R1a1a1 (R-M417) is the most widely found subclade, in two variations which are found respectively in Europe (R1a1a1b1 (R-Z282) ([R1a1a1a*] (R-Z282) (Underhill 2014)[2]) and Central and South Asia (R1a1a1b2 (R-Z93) ([R1a1a2*] (R-Z93) Underhill 2014)[2]).
R-Z282 (R1a1a1b1a) (Eastern Europe)
[edit]This large subclade appears to encompass most of the R1a1a found in Europe.[14]
- R1a1a1b1a [R1a1a1a* (Underhill (2014))] (R-Z282*) occurs in northern Ukraine, Belarus, and Russia at a frequency of c. 20%.[2]
- R1a1a1b1a3 [R1a1a1a1 (Underhill (2014))] (R-Z284) occurs in Northwest Europe and peaks at c. 20% in Norway.[2]
- R1a1a1c (M64.2, M87, M204) is apparently rare: it was found in 1 of 117 males typed in southern Iran.[53]
R-M458 (R1a1a1b1a1)
[edit]
R-M458 is a mainly Slavic SNP, characterized by its own mutation, and was first called cluster N. Underhill et al. (2009) found it to be present in modern European populations roughly between the Rhine catchment and the Ural Mountains and traced it to "a founder effect that ... falls into the early Holocene period, 7.9±2.6 KYA." (Zhivotovsky speeds, 3x overvalued)[3] M458 was found in one skeleton from a 14th-century grave field in Usedom, Mecklenburg-Vorpommern, Germany.[54] The paper by Underhill et al. (2009) also reports a surprisingly high frequency of M458 in some Northern Caucasian populations (18% among Ak Nogai,[55] 7.8% among Qara Nogai and 3.4% among Abazas).[56]
R-L260 (R1a1a1b1a1a)
[edit]R1a1a1b1a1a (R-L260), commonly referred to as West Slavic or Polish, is a subclade of the larger parent group R-M458, and was first identified as an STR cluster by Pawlowski et al. 2002. In 2010 it was verified to be a haplogroup identified by its own mutation (SNP).[57] It apparently accounts for about 8% of Polish men, making it the most common subclade in Poland. Outside of Poland it is less common.[58] In addition to Poland, it is mainly found in the Czech Republic and Slovakia, and is considered "clearly West Slavic". The founding ancestor of R-L260 is estimated to have lived between 2000 and 3000 years ago, i.e. during the Iron Age, with significant population expansion less than 1,500 years ago.[59]
R-M334
[edit]R-M334 ([R1a1a1g1],[47] a subclade of [R1a1a1g] (M458)[47] c.q. R1a1a1b1a1 (M458)[46]) was found by Underhill et al. (2009) only in one Estonian man and may define a very recently founded and small clade.[3]
R1a1a1b1a2 (S466/Z280, S204/Z91)
[edit]R1a1a1b1a2b3* (Gwozdz's Cluster K)
[edit]R1a1a1b1a2b3* (M417+, Z645+, Z283+, Z282+, Z280+, CTS1211+, CTS3402, Y33+, CTS3318+, Y2613+) (Gwozdz's Cluster K)[45][verification needed] is a STR based group that is R-M17(xM458). This cluster is common in Poland but not exclusive to Poland.[59]
R1a1a1b1a2b3a (R-L365)
[edit]R1a1a1b1a2b3a (R-L365)[46] was early called Cluster G.[citation needed]
R1a1a1b2 (R-Z93) (Asia)
[edit]| Region | People | N | R-M17 | R-M434 | ||
|---|---|---|---|---|---|---|
| Number | Freq. (%) | Number | Freq. (%) | |||
| Pakistan | Baloch | 60 | 9 | 15% | 5 | 8% |
| Pakistan | Makrani | 60 | 15 | 25% | 4 | 7% |
| Middle East | Oman | 121 | 11 | 9% | 3 | 2.5% |
| Pakistan | Sindhi | 134 | 65 | 49% | 2 | 1.5% |
| Table only shows positive sets from N = 3667 derived from 60 Eurasian populations sample.[3] | ||||||
This large subclade appears to encompass most of the R1a1a found in Asia, being related to Indo-European migrations (including Scythians, Indo-Aryan migrations and so on).[14]
- R-Z93* or R1a1a1b2* (R1a1a2* in Underhill (2014)) is most common (>30%) in the South Siberian Altai region of Russia, cropping up in Kyrgyzstan (6%) and in all Iranian populations (1-8%).[2]
- R-Z2125 occurs at highest frequencies in Kyrgyzstan and in Afghan Pashtuns (>40%). At a frequency of >10%, it is also observed in other Afghan ethnic groups and in some populations in the Caucasus and Iran.[2]
- R-M560 is very rare and was only observed in four samples: two Burushaski speakers (north Pakistan), one Hazara (Afghanistan), and one Iranian Azerbaijani.[2]
- R-M780 occurs at high frequency in South Asia: India, Pakistan, Afghanistan, and the Himalayas. Turkey share R1a (12.1%) sublineages.[60] Roma from Slovakia share 3% of R1a[61] The group also occurs at >3% in some Iranian populations and is present at >30% in Roma from Croatia and Hungary.[2]
Geographic distribution of R1a1a
[edit]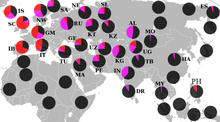
Pre-historical
[edit]In Mesolithic Europe, R1a is characteristic of Eastern Hunter-Gatherers (EHGs).[62] A male EHG of the Veretye culture buried at Peschanitsa near Lake Lacha in Arkhangelsk Oblast, Russia c. 10,700 BCE was found to be a carrier of the paternal haplogroup R1a5-YP1301 and the maternal haplogroup U4a.[63][64][62] A male, named PES001, from Peschanitsa in northwestern Russia was found to carry R1a5, and dates to at least 10,600 years ago.[5] More examples include the males Minino II (V) and Minino II (I/1), with the former carrying R1a1 and the latter R1a respectively, with the former being at 10,600 years old and the latter at least 10,400 years old respectively, both from Minino in northwestern Russia.[65] A Mesolithic male from Karelia c. 8,800 BCE to 7950 BCE has been found to be carrying haplogroup R1a.[66] A Mesolithic male buried at Deriivka c. 7000 BCE to 6700 BCE carried the paternal haplogroup R1a and the maternal U5a2a.[17] Another male from Karelia from c. 5,500 to 5,000 BC, who was considered an EHG, carried haplogroup R1a.[15] A male from the Comb Ceramic culture in Kudruküla c. 5,900 BCE to 3,800 BCE has been determined to be a carrier of R1a and the maternal U2e1.[67] According to archaeologist David Anthony, the paternal R1a-Z93 was found at the Oskol river near a no longer existing kolkhoz "Alexandria", Ukraine c. 4000 BCE, "the earliest known sample to show the genetic adaptation to lactase persistence (13910-T)."[68] R1a has been found in the Corded Ware culture,[69][70] in which it is predominant.[71] Examined males of the Bronze Age Fatyanovo culture belong entirely to R1a, specifically subclade R1a-Z93.[62][63][72]
Haplogroup R1a has later been found in ancient fossils associated with the Urnfield culture;[73] as well as the burial of the remains of the Sintashta,[16] Andronovo,[74] the Pazyryk,[75] Tagar,[74] Tashtyk,[74] and Srubnaya cultures, the inhabitants of ancient Tanais,[76] in the Tarim mummies,[77] and the aristocracy of Xiongnu.[78] The skeletal remains of a father and his two sons, from an archaeological site discovered in 2005 near Eulau (in Saxony-Anhalt, Germany) and dated to about 2600 BCE, tested positive for the Y-SNP marker SRY10831.2. The Ysearch number for the Eulau remains is 2C46S. The ancestral clade was thus present in Europe at least 4600 years ago, in association with one site of the widespread Corded Ware culture.[69]
Europe
[edit]In Europe, the R1a1 sub-clade is found at highest levels among peoples of Central and Eastern European descent, with results ranging from 35% to 65% among Czechs, Hungarians, Poles, Slovaks, western Ukrainians, Rusyns, Belarusians, Moldovans, and Russians.[79][80][12] In the Baltics, R1a1a frequencies decrease from Lithuania (45%) to Estonia (around 30%).[81][82][83][12][84]
There is a significant presence in peoples of Scandinavian descent, with highest levels in Norway and Iceland, where between 20 and 30% of men are in R1a1a.[85][86] Vikings and Normans may have also carried the R1a1a lineage further out, accounting for at least part of the small presence in the British Isles, the Canary Islands, and Sicily.[87][88] In East Germany, Haplogroup R1a1a averages between 20 and 30%, with a peak in Rostock at 31.3%, it.[89]
In Southern Europe R1a1a is not common, but significant levels have been found in pockets, such as in the Pas Valley in Northern Spain, areas of Venice, and Calabria in Italy.[90][better source needed] The Balkans shows wide variation between areas with significant levels of R1a1a, for example 36–39% in Slovenia,[91] 27–34% in Croatia,[82][92][93][94][95] and over 30% in Greek Macedonia, but less than 10% in Albania, Kosovo and parts of Greece south of Olympus gorge.[96][83][12]
R1a is virtually composed only of the Z284 subclade in Scandinavia. In Slovenia, the main subclade is Z282 (Z280 and M458), although the Z284 subclade was found in one sample of a Slovenian. There is a negligible representation of Z93 in Turkey, 12,1%[60][2] West Slavs and Hungarians are characterized by a high frequency of the subclade M458 and a low Z92, a subclade of Z280. Hundreds of Slovenian samples and Czechs lack the Z92 subclade of Z280, while Poles, Slovaks, Croats and Hungarians only show a very low frequency of Z92.[2] The Balts, East Slavs, Serbs, Macedonians, Bulgarians and Romanians demonstrate a ratio Z280>M458 and a high, up to a prevailing share of Z92.[2] Balts and East Slavs have the same subclades and similar frequencies in a more detailed phylogeny of the subclades.[97][98] The Russian geneticist Oleg Balanovsky speculated that there is a predominance of the assimilated pre-Slavic substrate in the genetics of East and West Slavic populations, according to him the common genetic structure which contrasts East Slavs and Balts from other populations may suggest the explanation that the pre-Slavic substrate of the East and West Slavs consisted most significantly of Baltic-speakers, which at one point predated the Slavs in the cultures of the Eurasian steppe according to archaeological and toponymic references.[note 16]
Asia
[edit]Central Asia
[edit]Zerjal et al. (2002) found R1a1a in 64% of a sample of the Tajiks of Tajikistan and 63% of a sample of the Kyrgyz of Kyrgyzstan.[99]
Haber et al. (2012) found R1a1a-M17 in 26.0% (53/204) of a set of samples from Afghanistan, including 60% (3/5) of a sample of Nuristanis, 51.0% (25/49) of a sample of Pashtuns, 30.4% (17/56) of a sample of Tajiks, 17.6% (3/17) of a sample of Uzbeks, 6.7% (4/60) of a sample of Hazaras, and in the only sampled Turkmen individual.[100]
Di Cristofaro et al. (2013) found R1a1a-M198/M17 in 56.3% (49/87) of a pair of samples of Pashtuns from Afghanistan (including 20/34 or 58.8% of a sample of Pashtuns from Baghlan and 29/53 or 54.7% of a sample of Pashtuns from Kunduz), 29.1% (37/127) of a pool of samples of Uzbeks from Afghanistan (including 28/94 or 29.8% of a sample of Uzbeks from Jawzjan, 8/28 or 28.6% of a sample of Uzbeks from Sar-e Pol, and 1/5 or 20% of a sample of Uzbeks from Balkh), 27.5% (39/142) of a pool of samples of Tajiks from Afghanistan (including 22/54 or 40.7% of a sample of Tajiks from Balkh, 9/35 or 25.7% of a sample of Tajiks from Takhar, 4/16 or 25.0% of a sample of Tajiks from Samangan, and 4/37 or 10.8% of a sample of Tajiks from Badakhshan), 16.2% (12/74) of a sample of Turkmens from Jawzjan, and 9.1% (7/77) of a pair of samples of Hazara from Afghanistan (including 7/69 or 10.1% of a sample of Hazara from Bamiyan and 0/8 or 0% of a sample of Hazara from Balkh).[101]
Malyarchuk et al. (2013) found R1a1-SRY10831.2 in 30.0% (12/40) of a sample of Tajiks from Tajikistan.[102]
Ashirbekov et al. (2017) found R1a-M198 in 6.03% (78/1294) of a set of samples of Kazakhs from Kazakhstan. R1a-M198 was observed with greater than average frequency in the study's samples of the following Kazakh tribes: 13/41 = 31.7% of a sample of Suan, 8/29 = 27.6% of a sample of Oshaqty, 6/30 = 20.0% of a sample of Qozha, 4/29 = 13.8% of a sample of Qypshaq, 1/8 = 12.5% of a sample of Tore, 9/86 = 10.5% of a sample of Jetyru, 4/50 = 8.0% of a sample of Argyn, 1/13 = 7.7% of a sample of Shanyshqyly, 8/122 = 6.6% of a sample of Alimuly, 3/46 = 6.5% of a sample of Alban. R1a-M198 also was observed in 5/42 = 11.9% of a sample of Kazakhs of unreported tribal affiliation.[103]
South Asia
[edit]In South Asia, R1a1a has often been observed in a number of demographic groups.[34][33]
In India, high frequencies of this haplogroup is observed in West Bengal Brahmins (72%)[33] to the east, Gujarat Lohanas (60%) [3] to the west, Punjab/Haryana Khatris (67%)[3] Ahirs(63%)[104]in the north and Karnataka Medars (39%)[105] in the south. It has also been found in several South Indian Dravidian-speaking Adivasis including the Chenchu (26%) of Andhra Pradesh and Kota of Andhra Pradesh (22.58%)[106] and the Kallar of Tamil Nadu suggesting that R1a1a is widespread in Tribal Southern Indians.[28]
Besides these, studies show high percentages in regionally diverse groups such as Manipuris (50%)[3] to the extreme North East and among Punjabis (47%)[28] to the extreme North West.
In Pakistan it is found at 71% among the Mohanna tribe in Sindh province to the south and 46% among the Baltis of Gilgit-Baltistan to the north.[3] Among the Sinhalese of Sri Lanka, 23% were found to be R1a1a (R-SRY1532) positive.[107] Hindus of Chitwan District in the Terai region Nepal show it at 69%.[108]
East Asia
[edit]The frequency of R1a1a is comparatively low among some Turkic-speaking groups like Yakuts, yet levels are higher (19 to 28%) in certain Turkic or Mongolic-speaking groups of Northwestern China, such as the Bonan, Dongxiang, Salar, and Uyghurs.[13][109][110]
A Chinese paper published in 2018 found R1a-Z94 in 38.5% (15/39) of a sample of Keriyalik Uyghurs from Darya Boyi / Darya Boye Village, Yutian County, Xinjiang (于田县达里雅布依乡), R1a-Z93 in 28.9% (22/76) of a sample of Dolan Uyghurs from Horiqol township, Awat County, Xinjiang (阿瓦提县乌鲁却勒镇), and R1a-Z93 in 6.3% (4/64) of a sample of Loplik Uyghurs from Karquga / Qarchugha Village, Yuli County, Xinjiang (尉犁县喀尔曲尕乡). R1a(xZ93) was observed only in one of 76 Dolan Uyghurs.[111] Note that Darya Boyi Village is located in a remote oasis formed by the Keriya River in the Taklamakan Desert. A 2011 Y-DNA study found Y-dna R1a1 in 10% of a sample of southern Hui people from Yunnan, 1.6% of a sample of Tibetan people from Xizang (Tibet Autonomous Region), 1.6% of a sample of Xibe people from Xinjiang, 3.2% of a sample of northern Hui from Ningxia, 9.4% of a sample of Hazak (Kazakhs) from Xinjiang, and rates of 24.0%, 22.2%, 35.2%, 29.2% in 4 different samples of Uyghurs from Xinjiang, 9.1% in a sample of Mongols from Inner Mongolia. A different subclade of R1 was also found in 1.5% of a sample of northern Hui from Ningxia.[112] in the same study there were no cases of R1a detected at all in 6 samples of Han Chinese in Yunnan, 1 sample of Han in Guangxi, 5 samples of Han in Guizhou, 2 samples of Han in Guangdong, 2 samples of Han in Fujian, 2 samples of Han in Zhejiang, 1 sample of Han in Shanghai, 1 samples of Han in Jiangxi, 2 samples of Han in Hunan, 1 sample of Han in Hubei, 2 samples of Han in Sichuan, 1 sample of Han in Chongqing, 3 samples of Han in Shandong, 5 samples of Han in Gansu, 3 samples of Han in Jilin and 2 samples of Han in Heilongjiang.[113] 40% of Salars, 45.2% of Tajiks of Xinjiang, 54.3% of Dongxiang, 60.6% of Tatars and 68.9% of Kyrgyz in Xinjiang in northwestern China tested in one sample had R1a1-M17. Bao'an (Bonan) had the most haplogroup diversity of 0.8946±0.0305 while the other ethnic minorities in northwestern China had a high haplogroup diversity like Central Asians, of 0.7602±0.0546.[114]
In Eastern Siberia, R1a1a is found among certain indigenous ethnic groups including Kamchatkans and Chukotkans, and peaking in Itel'man at 22%.[115]
Southeast Asia
[edit]Y-haplogroups R1a-M420 and R2-M479 are found in Ede (8.3% and 4.2%) and Giarai (3.7% and 3.7%) peoples in Vietnam. The Cham additionally have haplogroups R-M17 (13.6%) and R-M124 (3.4%).
R1a1a1b2a2a (R-Z2123) and R1a1 are found in Khmer peoples from Thailand (3.4%) and Cambodia (7.2%) respectively. Haplogroup R1a1a1b2a1b (R-Y6) is also found among Kuy peoples (5%).
According to Changmai et. al (2022), these haplogroup frequencies originate from South Asians, who left a cultural and genetic legacy in Southeast Asia since the first millennium CE.[116]
West Asia
[edit]R1a1a has been found in various forms, in most parts of Western Asia, in widely varying concentrations, from almost no presence in areas such as Jordan, to much higher levels in parts of Kuwait and Iran. The Shimar (Shammar) Bedouin tribe in Kuwait show the highest frequency in the Middle East at 43%.[117][118][119]
Wells 2001, noted that in the western part of the country, Iranians show low R1a1a levels, while males of eastern parts of Iran carried up to 35% R1a1a. Nasidze et al. 2004 found R1a1a in approximately 20% of Iranian males from the cities of Tehran and Isfahan. Regueiro 2006 in a study of Iran, noted much higher frequencies in the south than the north.
A newer study has found 20.3% R-M17* among Kurdish samples which were taken in the Kurdistan Province in western Iran, 19% among Azerbaijanis in West Azerbaijan, 9.7% among Mazandaranis in North Iran in the province of Mazandaran, 9.4% among Gilaks in province of Gilan, 12.8% among Persian and 17.6% among Zoroastrians in Yazd, 18.2% among Persians in Isfahan, 20.3% among Persians in Khorasan, 16.7% Afro-Iranians, 18.4% Qeshmi "Gheshmi", 21.4% among Persian Bandari people in Hormozgan and 25% among the Baloch people in Sistan and Baluchestan Province.[120]
Di Cristofaro et al. (2013) found haplogroup R1a in 9.68% (18/186) of a set of samples from Iran, though with a large variance ranging from 0% (0/18) in a sample of Iranians from Tehran to 25% (5/20) in a sample of Iranians from Khorasan and 27% (3/11) in a sample of Iranians of unknown provenance. All Iranian R1a individuals carried the M198 and M17 mutations except one individual in a sample of Iranians from Gilan (n=27), who was reported to belong to R1a-SRY1532.2(xM198, M17).[101]
Малярчук и др. (2013) обнаружили R1a1-SRY10831.2 у 20,8% (16/77) выборки персов, собранной в провинциях Хорасан и Керман на востоке Ирана, но не нашли ни одного члена этой гаплогруппы в выборке из 25 курдов. собраны в провинции Керманшах на западе Ирана. [102]
С другой стороны, к северу от этих регионов Западной Азии уровни R1a1a начинают повышаться на Кавказе , опять же неравномерно. В нескольких изученных популяциях не было обнаружено никаких признаков R1a1a, в то время как самые высокие уровни, обнаруженные в регионе, по-видимому, принадлежат носителям карачаево -балкарского языка, среди которых около четверти протестированных мужчин имеют гаплогруппу R1a1a. [3]
Историческое название R1a
[ редактировать ]Историческая система именования, обычно используемая для R1a, была противоречивой в разных опубликованных источниках, поскольку она часто менялась; это требует некоторого объяснения.
В 2002 году Консорциум Y-хромосомы (YCC) предложил новую систему наименования гаплогрупп ( YCC 2002 ), которая теперь стала стандартной. В этой системе имена в формате «R1» и «R1a» являются « филогенетическим » именем, предназначенным для обозначения позиций в генеалогическом древе. Названия мутаций SNP также можно использовать для обозначения клад или гаплогрупп. Например, поскольку M173 в настоящее время является определяющей мутацией R1, R1 также является R-M173, «мутационным» названием клады. Когда обнаруживается новое ветвление в дереве, некоторые филогенетические названия изменятся, но по определению все мутационные названия останутся прежними.
Широко распространенная гаплогруппа, определенная мутацией M17, была известна под различными названиями, такими как «Eu19», которые использовались в ( Semino et al. 2000 ) в старых системах именования. Предложение YCC 2002 года присвоило имя R1a гаплогруппе, определенной мутацией SRY1532.2. Сюда входил Eu19 (т.е. R-M17) в качестве субклада, поэтому Eu19 был назван R1a1. Обратите внимание: SRY1532.2 также известен как SRY10831.2. [ нужна ссылка ] Открытие M420 в 2009 году привело к изменению этих филогенетических названий. ( Underhill et al. 2009 и ISOGG 2012 ) R1a теперь определяется мутацией M420: в этом обновленном дереве субклад, определенный SRY1532.2, переместился из R1a. к R1a1 и Eu19 (R-M17) от R1a1 до R1a1a.
Более поздние обновления, записанные на справочной веб-странице ISOGG, касаются ветвей R-M17, включая одну основную ветку, R-M417.
| Схема 2002 г., предложенная в ( YCC 2002 ). | Схема 2009 г. согласно ( Underhill et al. 2009 ) | Дерево ISOGG по состоянию на январь 2011 г. [ нужна ссылка ] | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
|
См. также
[ редактировать ]Субклады Y-ДНК R-M207
[ редактировать ]Основополагающее дерево Y-ДНК
[ редактировать ]Примечания
[ редактировать ]- ^ Согласно Семейному древу, [ ВОЗ? ] они диверсифицировали c. 5000 лет назад. [9]
- ^ См. также: « Рассредоточение населения по Индийскому субконтиненту с «сильной половой предвзятостью» (Сильва и др., 2017)» . Блог Еврогенеса . 28 марта 2017 г. [ самостоятельно опубликованный источник? ]
- ^ Семенов и Булат (2016) ссылаются на следующие публикации:
- Хаак, Вольфганг (2015). «Массовая миграция из степи является источником индоевропейских языков в Европе» . Природа . 522 (7555): 207–211. arXiv : 1502.02783 . Бибкод : 2015Natur.522..207H . bioRxiv 10.1101/013433 . дои : 10.1038/NATURE14317 . ПМК 5048219 . ПМИД 25731166 .
- Мэтисон, Иэн (2015). «Восемь тысяч лет естественного отбора в Европе». биоRxiv 10.1101/016477 .
- Чекунова Е.М., Ярцева Н.В., Чекунов М.К., Мазуркевич А.Н. Первые результаты генотипирования аборигенов и человеческих костных остатков археологических памятников Верхнего Подвинья. // Археология озерных поселений IV—II тысячелетий до нашей эры: Хронология культур и природной среды и климатических ритмов. Материалы международной конференции, посвященной 50-летнему исследованию свайных поселений Северо-Запада России. Санкт-Петербург, 13–15 ноября 2014 г.
- Джонс, скорая помощь; Гонсалес-Фортес, Г; Коннелл, С; Сиска, В; Эрикссон, А; Мартиниано, Р; Маклафлин, РЛ; Гальего Льоренте, М; Кэссиди, LM; Гамба, К; Мешвелиани, Т; Бар-Йосеф, О; Мюллер, В; Белфер-Коэн, А; Мацкевич З.; Джейкли, Н; Хайэм, штат Техас; Куррат, М; Лордкипанидзе, Д; Хофрейтер, М; Маника, А; Пинхаси, Р; Брэдли, генеральный директор (2015). «Геномы верхнего палеолита раскрывают глубокие корни современных евразийцев» . Нат Коммун . 6 : 8912. Бибкод : 2015NatCo...6.8912J . дои : 10.1038/ncomms9912 . ПМК 4660371 . ПМИД 26567969 .
- ^ Кивисилд и др. (2003): «Гаплогруппа R1a, ранее связанная с предполагаемым индоарийским вторжением, была обнаружена с самой высокой частотой в Пенджабе, но также с относительно высокой частотой (26%) в племени Ченчу. Это открытие вместе с более высоким R1a Связанное с этим разнообразие коротких тандемных повторов в Индии и Иране по сравнению с Европой и Центральной Азией позволяет предположить, что источником этой гаплогруппы могла быть Южная и Западная Азия». [28]
- ^ Сенгупта (2006) : «Мы обнаружили, что влияние Центральной Азии на ранее существовавший генофонд было незначительным. Возраст накопленных микросателлитных вариаций в большинстве индийских гаплогрупп превышает 10 000–15 000 лет, что свидетельствует о древности региональных Таким образом, наши данные не поддерживают модели, которые ссылаются на явно выраженный недавний генетический вклад из Центральной Азии для объяснения наблюдаемой генетической изменчивости в Южной Азии».
- ^ Южноазиатское происхождение:
* Саху и др. (2006) : «...следует ожидать резко более низкой генетической изменчивости среди индийских линий Rla. На самом деле все наоборот: разнообразие гаплотипов STR на фоне R1a в Центральной Азии (а также в Восточной Европе) уже было показано, что он ниже, чем в Индии (6). Скорее, высокая частота встречаемости R1* и Rla в популяциях Центральной Азии и Европы (в большинстве случаев без R2 и R*) более экономно объясняется потоком генов в противоположном направлении. , возможно, с ранним эффектом основателя в Южной или Западной Азии. [36]
* Шарма и др. (2009) : «Своеобразное наблюдение самой высокой частоты (до 72,22%) Y-гаплогруппы R1a1* у брахманов намекало на ее присутствие в качестве линии-основателя этой кастовой группы. Кроме того, наблюдение R1a1* в различных племенных группах населения , существование Y-гаплогруппы R1a* у предков и расширенный филогенетический анализ объединенного набора данных 530 индийцев, 224 пакистанцев и 276 выходцев из Центральной Азии и Евразии, несущих гаплогруппу R1a1*, подтвердили автохтонное происхождение линии R1a1 в Индии и племенную связь с индийцами. Однако важно обнаружить новый бинарный маркер Y-хромосомы для более высокого разрешения R1a1* и подтвердить сделанные выводы». - ^ Хотя Сенгупта (2006) признал, что «[R1a1 и R2] могли действительно прибыть в южную Индию из исходного региона на юго-западе Азии несколько раз». Полностью: «Широкое географическое распространение HG R1a1-M17 по всей Евразии и отсутствие в настоящее время информативных подразделений, определяемых бинарными маркерами, оставляют неясным географическое происхождение HG R1a1-M17. Однако контурная карта дисперсии R1a1-M17 показывает самый высокий вариация в северо-западном регионе Индии... Остается вопрос, насколько различна история L1 по отношению к некоторым или всем представителям R1a1 и R2. Эта неопределенность нейтрализует предыдущие выводы о вторжении HGs R1a1 и R2 с северо-запада в дравидский период. -говорящие южные племена связаны с одним недавним событием [R1a1 и R2], которое могло фактически прибыть в южную Индию из исходного региона юго-западной Азии несколько раз, причем некоторые эпизоды существуют значительно раньше, чем другие. Существуют значительные археологические свидетельства присутствия мезолита. народы Индии (Кеннеди, 2000), некоторые из которых могли проникнуть на субконтинент с северо-запада в эпоху позднего плейстоцена. Высокая дисперсия R1a1 в Индии (таблица 12), пространственное распределение частот микросателлитных дисперсионных клинов R1a1 (рис. 4) и время расширения (таблица 11) подтверждают эту точку зрения». [33]
- ^ Лалуэса-Фокс: «Несколько лет назад местные ученые поддержали точку зрения, что существование Y-хромосомы R1a не связано с потоком чужеродных генов, а вместо этого связано с тем, что эта линия возникла на субконтиненте и распространилась оттуда. Но филогенетическая реконструкция этой гаплогруппы не подтвердили эту точку зрения». [37]
- ^ Тем не менее, Хаак и др. также прямо заявляют: «тип ближневосточного происхождения, отличный от того, который был привнесен ранними фермерами». [ нужны разъяснения ] [39]
- ^ Согласно ДНК Генеалогического древа, L664 образовался 4700 лн, то есть 2700 г. до н.э. [9]
- ↑ Лазаридис, Твиттер, 18 июня 2016 г .: «I1635 (Armenia_EBA) — это R1b1-M415(xM269). Мы обязательно включим в редакцию. Спасибо человеку, который заметил! #ILovePreprints». [ ненадежный источник? ]
См. также «Большое дело 2016 года: территория современного Ирана не может быть индоевропейской родиной» . Блог Еврогенеса . 26 ноября 2016 г., [ ненадежный источник? ] для обсуждения той же темы. - ^ См. карту распространения M780 в блоге Dieneke's Anthropology Blog, крупную новую статью о глубоком происхождении Y-гаплогруппы R1a (Underhill et al. 2014). [43]
- ↑ Согласно генеалогическому древу ДНК, M780 образовался 4700 лн. [9] Эта датировка совпадает с движением на восток между 2800 и 2600 гг. до н. э. ямной культуры в область полтавской культуры , предшественницы синташтинской культуры , от которой произошли индоиранцы. М780 сосредоточен в долине Ганга, месте проживания классического ведического общества .
- ^ Позник и др. (2016) рассчитывают со временем генерации 30 лет; время генерации в 20 лет дает другие результаты.
- ^ «Доказательства того, что кластер Steppe_MLBA [средний и поздний бронзовый век] является вероятным источником степного происхождения в Южной Азии, также подтверждается данными Y-хромосомы, поскольку гаплогруппа R1a относится к подтипу Z93, распространенному сегодня в Южной Азии [Андерхилл et al. (2014), Silva et al. (2017)] была высокой частотой в Steppe_MLBA (68%) (16), но редко в Steppe_EMBA [от раннего до среднего бронзового века] (отсутствует в наших данных)». [44]
- ^ Балановский (2015) , p. 208 (in Russian) Прежде всего, это преобладание в славянских популяциях дославянского субстрата — двух ассимилированных ими генетических компонентов – восточноевропейского для западных и восточных славян и южноевропейского для южных славян...Можно с осторожностью предположить, что ассимилированный субстратмог быть представлен по преимуществу балтоязычными популяциями. Действительно, археологические данные указыва ют на очень широкое распространение балтских групп перед началом расселения славян. Балтскийсубстрату славян (правда, наряду с финно-угорским) выявляли и антропологи. Полученные нами генетические данные — и на графиках генетических взаимоотношений, и по доле общих фрагментов генома — указывают, что современные балтские народы являются ближайшими генетически ми соседями восточных славян. При этом балты являются и лингвистически ближайшими род ственниками славян. И можно полагать, что к моменту ассимиляции их генофонд не так сильно отличался от генофонда начавших свое широкое расселение славян. Поэтому если предположить,что расселяющиеся на восток славяне ассимилировали по преимуществу балтов, это может объяснить и сходство современных славянских и балтских народов друг с другом, и их отличия от окружающих их не балто-славянских групп Европы...В работе высказывается осторожное предположение, что ассимилированный субстрат мог быть представлен по преимуществу балтоязычными популяциями. Действительно, археологические данные указывают на очень широкое распространение балтских групп перед началом расселения славян. Балтский субстрат у славян (правда, наряду с финно-угорским) выявляли и антропологи. Полученные в этой работе генетические данные — и на графиках генетических взаимоотношений, и по доле общих фрагментов генома — указывают, что современные балтские народы являются ближайшими генетическими соседями восточных славян.
Ссылки
[ редактировать ]- ^ Перейти обратно: а б с д и Шарма и др. 2009 .
- ^ Перейти обратно: а б с д и ж г час я дж к л м н тот п д р с т в Андерхилл и др. 2014 .
- ^ Перейти обратно: а б с д и ж г час я дж к л м н тот п д р Андерхилл и др. 2009 .
- ^ Перейти обратно: а б Андерхилл и др. 2014 , с. 130.
- ^ Перейти обратно: а б Сааг, Лехти; Васильев Сергей Владимирович; Варул, Лийви; Косорукова Наталья Владимировна; Герасимов Дмитрий В.; Ошибкина Светлана Владимировна; Гриффит, Сэмюэл Дж.; Сольник, Ану; Сааг, Лаури; Д'Атанасио, Евгения; Мецпалу, Эне (январь 2021 г.). «Изменения генетического происхождения при переходе от каменного века к бронзовому веку на восточноевропейской равнине» . Достижения науки . 7 (4): eabd6535. Бибкод : 2021SciA....7.6535S . дои : 10.1126/sciadv.abd6535 . ПМЦ 7817100 . ПМИД 33523926 .
- ^ Хаак, Вольфганг; Лазаридис, Иосиф; Паттерсон, Ник; Роланд, Надин; Маллик, Свапан; Ламас, Бастьен; Брандт, Гвидо; Норденфельт, Сюзанна; Харни, Эдаоин; Стюардсон, Кристин; Фу, Цяомэй (10 февраля 2015 г.). «Массовая миграция из степи является источником индоевропейских языков в Европе» . bioRxiv : 013433. arXiv : 1502.02783 . дои : 10.1101/013433 . S2CID 196643946 . Архивировано из оригинала 23 декабря 2019 года . Проверено 8 февраля 2021 г.
- ^ Рагхаван, Маанаса; Скоглунд, Понт; Граф, Келли Э.; Мецпалу, Мейт; Альбрехцен, Андерс; Мольтке, Ида; Расмуссен, Саймон; Стаффорд-младший, Томас В.; Орландо, Людовик; Мецпалу, Эне; Кармин, Моника (январь 2014 г.). «Геном Сибири верхнего палеолита свидетельствует о двойном происхождении коренных американцев» . Природа . 505 (7481): 87–91. Бибкод : 2014Natur.505...87R . дои : 10.1038/nature12736 . ПМК 4105016 . ПМИД 24256729 .
- ^ Нарасимхан, Вагиш М.; Паттерсон, Ник; Мурджани, Прия; Роланд, Надин; Бернардос, Ребекка; Маллик, Свапан; Лазаридис, Иосиф; Накацука, Натан; Олальде, Иньиго; Липсон, Марк; Ким, Александр М. (6 сентября 2019 г.). «Формирование человеческих популяций в Южной и Центральной Азии» . Наука . 365 (6457): eaat7487. дои : 10.1126/science.aat7487 . ПМЦ 6822619 . ПМИД 31488661 .
Гаплогруппы Y-хромосомы типов R1b или R1a, не представленные в Иране и Туране в этот период...
- ^ Перейти обратно: а б с д и ж г час я дж «Дерево R1a» . YПолный . Архивировано из оригинала 19 августа 2016 года . Проверено 15 июля 2016 г.
- ^ Перейти обратно: а б с Mirabal et al. 2009Мирабал и др. 2009
- ^ Зержал, Т.; и др. (1999). «Использование вариаций Y-хромосомной ДНК для изучения истории населения: недавнее распространение самцов в Азии и Европе». В Папихе, СС; Дека Р. и Чакраборти Р. (ред.). Геномное разнообразие: применение в популяционной генетике человека . Нью-Йорк: Издательство Kluwer Academic/Plenum. стр. 91–101. ISBN 978-0-3064-6295-5 .
- ^ Перейти обратно: а б с д Семино и др. 2000 .
- ^ Перейти обратно: а б Уэллс 2001 .
- ^ Перейти обратно: а б с Памджав и др. 2012 .
- ^ Перейти обратно: а б Хаак и др. 2015 .
- ^ Перейти обратно: а б Аллентофт и др. 2015 .
- ^ Перейти обратно: а б Мэтисон и др. 2015 .
- ^ Перейти обратно: а б с Silva et al. 2017Сильва и др. 2017
- ^ Джозеф, Тони (16 июня 2017 г.). «Как генетика разрешает дебаты об арийской миграции» . Индус . Архивировано из оригинала 4 октября 2023 года . Проверено 2 июня 2019 г.
- ^ Энтони 2007 .
- ^ Энтони и Ринге, 2015 .
- ^ Перейти обратно: а б Хаак и др. 2015 , с. 5.
- ^ Semenov & Bulat 2016 .
- ^ Хабер и др. 2012 «R1a1a7-M458 отсутствовал в Афганистане, что позволяет предположить, что R1a1a-M17 не поддерживает, как считалось ранее [47], экспансию из Понтийской степи [3], приносящую индоевропейские языки в Среднюю Азию и Индию».
- ^ Клейн, Лео С. (22 апреля 2017 г.). «Степная гипотеза индоевропейского происхождения еще не доказана» . Акта Археологика . 88 (1): 193–204. дои : 10.1111/j.1600-0390.2017.12184.x . ISSN 0065-101X . Архивировано из оригинала 25 декабря 2022 года . Проверено 23 ноября 2022 г. «Что касается Y-хромосомы, то уже в Хааке, Лазаридисе и др. (2015) отмечалось, что ямная из Самары имела Y-хромосомы, которые принадлежали R-M269, но не принадлежали к распространенной в Западной Европе кладе (p .46 приложения). Кроме того, в Ямной нет ни одного R1a, в отличие от шнуровой керамики (с преобладанием R1a)».
- ^ Кох, Джон Т.; Канлифф, Барри (2016). Кельтский с Запада 3: Атлантическая Европа в эпоху металла . Книги Оксбоу. п. 634. ИСБН 978-1-78570-228-0 . Архивировано из оригинала 23 ноября 2022 года . Проверено 23 ноября 2022 г.
- ^ Semenov & Bulat 2016 , p. 41.
- ^ Перейти обратно: а б с д Кивисилд и др. 2003 .
- ^ Сенгупта С., Животовский Л.А., Кинг Р., Мехди С.К., Эдмондс К.А., Чоу К.Э. и др. (февраль 2006 г.). «Полярность и временность распределения Y-хромосомы в Индии с высоким разрешением определяют как местное, так и экзогенное распространение и выявляют незначительное генетическое влияние скотоводов Центральной Азии» . Американский журнал генетики человека . 78 (2): 202–221. дои : 10.1086/499411 . ПМК 1380230 . ПМИД 16400607 . «Хотя значительное культурное влияние на социальную иерархию и язык в Южной Азии связано с приходом кочевых скотоводов из Центральной Азии, генетические данные (митохондриальные и Y-хромосомные) привели к крайне противоречивым выводам о генетическом происхождении племен и каст Южной Азии. Мы стремились разрешить этот конфликт, используя данные высокого разрешения по 69 информативным бинарным маркерам Y-хромосомы и 10 микросателлитным маркерам из большого набора географически, социально и лингвистически репрезентативных этнических групп Южной Азии. ранее существовавший генофонд был незначительным. Возраст накопленных микросателлитных вариаций в большинстве индийских гаплогрупп превышает 10–15 тыс. лет, что свидетельствует о древности региональной дифференциации. Поэтому наши данные не подтверждают модели, предполагающие выраженную новейшую генетическую дифференциацию. данные из Центральной Азии, объясняющие наблюдаемую генетическую изменчивость в Южной Азии. Гаплогруппы R1a1 и R2 указывают на демографическую сложность, несовместимую с недавней единой историей. ПОЛУОСТРОВ ИНДИЙСКИЙ РЕГИОН».
- ^ Тансим И., Тангарадж К., Чауби Дж., Сингх В.К., Бхаскар Л.В., Редди Б.М. и др. (август 2006 г.). «Генетическое сходство среди низших каст и племенных групп Индии: вывод на основе Y-хромосомы и митохондриальной ДНК» . БМК Генетика . 7:42 . дои : 10.1186/1471-2156-7-42 . ПМЦ 1569435 . ПМИД 16893451 .
- ^ Саху С., Сингх А., Химабинду Г., Банерджи Дж., Ситалакшими Т., Гайквад С. и др. (январь 2006 г.). «Предыстория индийских Y-хромосом: оценка сценариев демического распространения» . Труды Национальной академии наук Соединенных Штатов Америки . 103 (4): 843–848. Бибкод : 2006PNAS..103..843S . дои : 10.1073/pnas.0507714103 . ПМЦ 1347984 . ПМИД 16415161 .
- ^ Тангарадж К., Найду Б.П., Кривелларо Ф., Таманг Р., Упадьяй С., Шарма В.К. и др. (декабрь 2010 г.). Кордо Р. (ред.). «Влияние естественных барьеров на формирование генетической структуры популяций Махараштры» . ПЛОС ОДИН . 5 (12): e15283. Бибкод : 2010PLoSO...515283T . дои : 10.1371/journal.pone.0015283 . ПМК 3004917 . ПМИД 21187967 .
- ^ Перейти обратно: а б с д и Сенгупта 2006 .
- ^ Перейти обратно: а б с д и Саху и др. 2006 год .
- ^ Перейти обратно: а б Тангарадж и др. 2010 .
- ^ Саху и др. 2006 , с. 845-846.
- ^ Перейти обратно: а б Лалуэса-Фокс, К. (2022). Неравенство: генетическая история . МТИ Пресс. стр. 81–82. ISBN 978-0-262-04678-7 . Архивировано из оригинала 16 июля 2023 года . Проверено 16 июля 2023 г.
- ^ Нарасимхан и др. 2019 .
- ^ Хаак и др. 2015 , с. 4.
- ^ Перейти обратно: а б с Маскареньяс и др. 2015 , с. 9.
- ^ Перейти обратно: а б Позник и др. 2016 , с. 5.
- ↑ Английский блог Араме, Y-ДНК с древнего Ближнего Востока. Архивировано 27 ноября 2016 г., в Wayback Machine.
- ^ «Блог антропологии Диенекеса: большая новая статья о глубоком происхождении Y-гаплогруппы R1a (Underhill et al. 2014)» . 27 марта 2014 года. Архивировано из оригинала 20 декабря 2019 года . Проверено 20 декабря 2019 г. [ ненадежный источник? ]
- ^ Перейти обратно: а б Нарасимхан и др. 2018 .
- ^ Перейти обратно: а б с "О нас" . ДНК генеалогического древа . Архивировано из оригинала 15 августа 2019 года . Проверено 20 декабря 2019 г.
- ^ Перейти обратно: а б с д и ж г час я дж к л м н тот п д р с т в v В х и С аа «ISOGG 2017 Гаплогруппа R Y-ДНК» . isogg.org . Архивировано из оригинала 10 февраля 2007 года . Проверено 20 декабря 2019 г.
- ^ Перейти обратно: а б с д и ж г час я дж к «Гаплогруппа R (Y-ДНК) — SNPedia» . www.snpedia.com . Архивировано из оригинала 5 мая 2018 года . Проверено 20 декабря 2019 г.
- ^ Карафет и др. 2014 .
- ^ Перейти обратно: а б с д и ж г час я дж к л м Андерхилл и др. 2014 , с. 125.
- ^ «R1a в Ямной» . Блог Еврогенеса . 21 марта 2016. Архивировано из оригинала 5 мая 2018 года . Проверено 20 декабря 2019 г.
- ^ «Гаплогруппа R Y-ДНК и ее субклады» . Международное общество генетической генеалогии (ISOGG). Архивировано из оригинала 30 марта 2019 года . Проверено 8 января 2011 г.
- ^ Кран, Томас. «Проект дерева Y-хромосомы» . ДНК генеалогического древа . Архивировано из оригинала 26 мая 2013 года . Проверено 7 декабря 2012 г.
- ^ Стрим 2006 .
- ^ Фредер, Джанин (2010). Средневековые скелеты Узедома: антропологическое исследование с особым учетом этнического происхождения [ Антропологическое исследование с учетом этнического происхождения ] (Диссертация) (на немецком языке). Свободный университет Берлина. п. 86. дои : 10.17169/refubium-8995 .
- ^ https://cyberleninka.ru/article/n/tyurki-kavkaza-sravnitelnyy-analiz-genofondov-po-dannym-o-y-hromosome Archived November 7, 2023, at the Wayback Machine "высокая частота R1a среди кубанских ногайцев (субветвь R1a1a1g-M458 забирает 18%"
- ^ Андерхилл, Пенсильвания; и др. (2009). «Разделение постледникового происхождения европейских и азиатских y-хромосом внутри гаплогруппы R1a» . Европейский журнал генетики человека . 18 (4): 479–484. дои : 10.1038/ejhg.2009.194 . ПМЦ 2987245 . ПМИД 19888303 .
- ^ Гвоздз, Питер (6 августа 2018 г.). «Польские клады Y-ДНК» . Архивировано из оригинала 15 июля 2016 года . Проверено 15 июля 2016 г.
- ^ Павловский и др. 2002 .
- ^ Перейти обратно: а б Гвоздь 2009 .
- ^ Перейти обратно: а б Карс, Мэн; Башак, АН; Онат, О.Э.; Билгувар, К.; Чой, Дж.; Итан, Ю.; Чаглар, К.; Палвадо, Р.; Казанова, Дж.Л.; Купер, Д.Н.; Стенсон, доктор медицинских наук; Явуз, А.; Булуш, Х.; Гюнель, М.; Фридман, Дж. М.; Озчелик, Т. (2021). «Генетическая структура турецкого населения демонстрирует высокий уровень изменчивости и примеси» . Труды Национальной академии наук Соединенных Штатов Америки . 118 (36): e2026076118. Бибкод : 2021PNAS..11826076K . дои : 10.1073/pnas.2026076118 . ПМЦ 8433500 . ПМИД 34426522 .
- ^ Петрейчикова, Ева; Сотак, Мирослав; Бернасовска, Ярмила; Бернасовский, Иван; Совикова, Адриана; Бозикова, Александра; Боронова, Ивета; Швикова, Петра; Габрикова, Дана; МаЦекова, Сона (2009). «Частоты Y-гаплогруппы в населении словацких цыган» . Антропологическая наука . 117 (2): 89–94. дои : 10.1537/ase.080422 .
- ^ Перейти обратно: а б с Сааг и др. 2020 , с. 5.
- ^ Перейти обратно: а б Сааг и др. 2020 , с. 29, таблица 1.
- ^ Сааг и др. 2020 г. , Дополнительные данные 2, строка 4.
- ^ Пост, Козимо; Ю, Хэ; Галичи, Айшин; Ружье, Элен; Кревкер, Изабель; Хуан, Илей; Рингбауэр, Харальд; Рорлах, Адам Б.; Нэгеле, Катрин; Вильяльба-Моуко, Ванесса; Радзевичюте, Рита; Феррас, Тьяго; Стессель, Александр; Тухбатова, Резеда; Друкер, Дороти Г. (1 марта 2023 г.). «Палеогеномика от верхнего палеолита до неолита европейских охотников-собирателей» . Природа . 615 (7950): 117–126. дои : 10.1038/s41586-023-05726-0 . hdl : 10256/23099 . ISSN 1476-4687 .
- ^ Фу и др. 2016 .
- ^ Сааг и др. 2017
- ^ Энтони 2019 , стр. 16, 17.
- ^ Перейти обратно: а б Хаак и др. 2008 .
- ^ Брандит и др. 2013 .
- ^ Мальмстрем и др. 2019 , стр. 2.
- ^ Сааг и др. 2020 г. , Дополнительные данные 2, строки 5–49.
- ^ Швейцер, Д. (23 марта 2008 г.). «Анализ данных пещеры Лихтенштейн» (PDF) . dirkschweitzer.net. Архивировано из оригинала (PDF) 14 августа 2011 года. Краткое изложение на английском языке Schilz (2006) .
- ^ Перейти обратно: а б с Кейзер и др. 2009 .
- ^ Рико и др. 2004
- ^ Korniyenko, I. V.; Vodolazhsky D. I. "Использование нерекомбинантных маркеров Y-хромосомы в исследованиях древних популяций (на примере поселения Танаис)" [The use of non-recombinant markers of the Y-chromosome in the study of ancient populations (on the example of the settlement of Tanais)]. Материалы Донских антропологических чтений [Materials of the Don Anthropological Readings]. Rostov-on-Don: Rostov Research Institute of Oncology, 2013.
- ^ Чуньсян Ли и др. 2010 .
- ^ Ким и др. 2010 .
- ^ Балановский и др. 2008 год .
- ^ Бехар и др. 2003 .
- ^ Касперавичюте, Кучинскас и Стоункинг 2005 .
- ^ Перейти обратно: а б Батталья и др. 2008 год .
- ^ Перейти обратно: а б Rosser et al. 2000Россер и др. 2000
- ^ Также и др. 2004 .
- ^ Боуден и др. 2008 год .
- ^ Дюпюи и др. 2005 .
- ^ Пассарино и др. 2002
- ^ Капелли и др. 2003 .
- ^ Кайзер и др. 2005 .
- ^ Скоццари и др. 2001 .
- ^ Андерхилл, Питер А. (1 января 2015 г.). «Филогенетическая и географическая структура гаплогруппы R1a Y-хромосомы» . Европейский журнал генетики человека . 23 (1): 124–131. дои : 10.1038/ejhg.2014.50 . ПМК 4266736 . ПМИД 24667786 .
- ^ Л. Барах; и др. (2003). «Y-хромосомное наследие хорватского населения и его островных изолятов» (PDF) . Европейский журнал генетики человека . 11 (7): 535–42. дои : 10.1038/sj.ejhg.5200992 . ПМИД 12825075 . S2CID 15822710 . Архивировано из оригинала (PDF) 17 декабря 2012 года . Проверено 10 сентября 2009 г.
- ^ С. Роотси; и др. (2004). «Филогеография гаплогруппы I Y-хромосомы раскрывает различные области доисторического потока генов в Европе» (PDF) . Американский журнал генетики человека . 75 (1): 128–137. дои : 10.1086/422196 . ПМК 1181996 . ПМИД 15162323 . Архивировано из оригинала (PDF) 5 сентября 2020 г. Проверено 13 февраля 2021 г.
- ^ М. Перичич; и др. (2005). «Филогенетический анализ Юго-Восточной Европы с высоким разрешением прослеживает основные эпизоды потока отцовских генов среди славянских популяций» . Молекулярная биология и эволюция . 22 (10): 1964–75. дои : 10.1093/molbev/msi185 . ПМИД 15944443 .
- ^ М. Перичич; и др. (2005). «Обзор хорватского генетического наследия, выявленного с помощью митохондриальной ДНК и Y-хромосомных линий». Хорватский медицинский журнал . 46 (4): 502–513. ПМИД 16100752 .
- ^ Перичич и др. 2005
- ^ «Без названия» . pereformat.ru (на русском языке). Архивировано из оригинала 15 марта 2016 года . Проверено 29 мая 2017 г.
- ^ «Без названия» . www.rodstvo.ru . Архивировано из оригинала 16 сентября 2021 года . Проверено 29 мая 2017 г.
- ^ Зержал и др. 2002 .
- ^ Хабер и др. 2012 .
- ^ Перейти обратно: а б Ди Кристофаро и др. 2013 .
- ^ Перейти обратно: а б Malyarchuk et al. 2013 .
- ^ Ashirbekov et al. 2017 .
- ^ Андерхилл, Питер А.; Майрес, Натали М.; Роотси, Сиири; Мецпалу, Мейт; Животовский Лев А.; Кинг, Рой Дж.; Лин, Алиса А.; Чоу, Шерил-Эмилиан Т.; Семино, Орнелла; Батталья, Винченца; Кутуев, Ильдус; Ярве, Мари; Чаубей, Гьянешвер; Аюб, Касим; Мохьюддин, Аиша (апрель 2010 г.). «Разделение постледникового происхождения европейских и азиатских Y-хромосом в пределах гаплогруппы R1a» . Европейский журнал генетики человека . 18 (4): 479–484. дои : 10.1038/ejhg.2009.194 . ISSN 1476-5438 . ПМЦ 2987245 .
- ^ Шах 2011 .
- ^ Арункумар 2012 .
- ^ Тоомас Кивисилд; Сиири Роотси; Мейт Метспалу; Эне Мецпалу; Юрий Парик; Катрин Калдма; Эсиен Усанга; Сарабджит Мастана; Суриндер С. Папиха; Ричард Виллемс. «Генетика языка и распространения сельского хозяйства в Индии» (PDF) . В П. Белввуде; К. Ренфрю (ред.). Исследование гипотезы расселения сельского хозяйства/языка . Монографии Института Макдональда. Кембриджский университет. стр. 215–222. Архивировано из оригинала (PDF) 19 февраля 2006 г. Проверено 20 декабря 2019 г.
- ^ Форнарино и др. 2009
- ^ Ван и др. 2003 .
- ^ Чжоу и др. 2007 .
- ^ Лю Шу-ху и др. 2018 .
- ^ Чжун и др. 2011 .
- ^ Чжун, Хуа; Ши, Хун; Ци, Сюэ-Бин; Дуань, Цзы-Юань; Тан, Пин-Пин; Джин, Ли; Су, Бинг; Ма, Ранлин З. (2011). «Расширенное исследование Y-хромосомы предполагает постледниковую миграцию современного человека в Восточную Азию по Северному маршруту» . Молекулярная биология и эволюция . 28 (1): 717–727. дои : 10.1093/molbev/msq247 . ПМИД 20837606 .
- ^ Шоу, Вэй-Хуа; Цяо, Внь-Фа; Вэй, Чуан-Юй; Донг, Юн-Ли; Тан, Си-Цзе; Ши, Хун; Тан, Вэнь-Ру; Сяо, Чун-Цзе (2010). «Распределение Y-хромосомы среди популяций Северо-Западного Китая указывает на значительный вклад скотоводов Центральной Азии и меньшее влияние западных евразийцев» . Журнал генетики человека . 55 (5): 314–322. дои : 10.1038/jhg.2010.30 . ПМИД 20414255 . S2CID 23002493 .
- ^ Лелль и др. 2002
- ^ Чангмай, Пия; Джайсамут, Китипонг; Кампуансай, Ятуполь; и др. (2022). «Индийское генетическое наследие в популяциях Юго-Восточной Азии» . ПЛОС Генетика . 18 (2): e1010036. дои : 10.1371/journal.pgen.1010036 . ПМЦ 8853555 . ПМИД 35176016 .
- ^ Мохаммад и др. 2009 .
- ^ Насидзе и др. 2004 .
- ^ Насидзе и др. 2005 .
- ^ Груни и др. 2012 .
Источники
[ редактировать ]- Аллентофт, Мортен Э.; Сикора, Мартин; Шегрен, Карл-Горан; Расмуссен, Саймон; Расмуссен, Мортен; Стендерап, Йеспер; Дамгаард, Питер Б.; Шредер, Ханнес; и др. (2015). «Популяционная геномика Евразии бронзового века» . Природа . 522 (7555): 167–172. Бибкод : 2015 Природа.522..167А . дои : 10.1038/nature14507 . ПМИД 26062507 . S2CID 4399103 . Архивировано из оригинала 13 июля 2019 года . Проверено 23 июля 2019 г.
- Энтони, Дэвид В. (2007), Лошадь, колесо и язык. Как всадники бронзового века из евразийских степей сформировали современный мир , Princeton University Press
- Энтони, Дэвид (весна – лето 2019 г.). «Археология, генетика и язык в степях: комментарий к Бомхарду» . Журнал индоевропейских исследований . 47 (1–2). Архивировано из оригинала 3 мая 2024 года . Проверено 9 января 2020 г.
- Энтони, Дэвид; Ринге, Дон (2015), «Индоевропейская родина с лингвистической и археологической точек зрения», Annual Review of Linguistics , 1 : 199–219, doi : 10.1146/annurev-linguist-030514-124812
- Шах, AM; Таманг, Р.; Мурджани, П.; Рани, Д.С.; Говиндарадж, П.; Кулкарни, Г.; Бхаттачарья, Т.; Мустак, М.С.; Бхаскар, ЛВКС; Редди, AG; Гадви, Д.; Гай, ПБ; Чаубей, Г.; Паттерсон, Н.; Райх, Д.; Тайлер-Смит, К.; Сингх, Л.; Тангарадж, К. (2011). «Индийские сидди: африканские потомки с индийской примесью» . Американский журнал генетики человека . 89 (1): 154–61. дои : 10.1016/j.ajhg.2011.05.030 . ПМК 3135801 . ПМИД 21741027 .
- Арун Кумар, Г; Сория-Эрнанц, DF; Кавита, виджей; Арун, В.С.; Шьяма, А; Ашокан, Канзас (2012). «Дифференциация населения мужских линий Южной Индии коррелирует с сельскохозяйственной экспансией, предшествовавшей кастовой системе» . ПЛОС ОДИН . 7 (11): е50269. Бибкод : 2012PLoSO...750269A . дои : 10.1371/journal.pone.0050269 . ПМК 3508930 . ПМИД 23209694 .
- Аширбеков Э.Э.; и др. (2017). «Распространение гаплогрупп Y-хромосомы казахов Южно-Казахстанской, Жамбылской и Алматинской областей» (PDF) . Доклады Национальной академии наук Республики Казахстан . 6 (316): 85–95. Архивировано из оригинала (PDF) 23 сентября 2021 года . Проверено 4 июня 2020 г.
- Балановский О, Роотси С, Пшеничнов А, Кивисилд Т, Чурносов М, Евсеева И, Почешхова Е, Болдырева М и др. (2008). «Два источника русского отцовского наследия в евразийском контексте» . Американский журнал генетики человека . 82 (1): 236–250. дои : 10.1016/j.ajhg.2007.09.019 . ПМК 2253976 . ПМИД 18179905 .
- Балановский, О. П. (November 30, 2015). Генофонд Европы (in Russian). KMK Scientific Press. ISBN 9785990715707 . Архивировано из оригинала 3 мая 2024 года . Проверено 8 августа 2020 г.
- Батталья В., Форнарино С., Аль-Захери Н., Оливьери А., Пала М., Майрес Н.М., Кинг Р.Дж., Рутси С. и др. (2008). «Y-хромосомные свидетельства культурного распространения сельского хозяйства в Юго-Восточной Европе» . Европейский журнал генетики человека . 17 (6): 820–30. дои : 10.1038/ejhg.2008.249 . ПМК 2947100 . ПМИД 19107149 .
- Бехар Д., Томас М.Г., Скорецки К., Хаммер М.Ф., Булыгина Е., Розенгартен Д., Джонс А.Л., Хелд К. и др. (2003). «Множественное происхождение ашкеназских левитов: свидетельства Y-хромосомы как ближневосточного, так и европейского происхождения» (PDF) . Американский журнал генетики человека . 73 (4): 768–779. дои : 10.1086/378506 . ПМК 1180600 . ПМИД 13680527 . Архивировано (PDF) из оригинала 17 апреля 2007 г. Проверено 23 апреля 2007 г.
- Боуден Г.Р., Балареск П., Кинг Т.Э., Хансен З., Ли А.С., Пергл-Уилсон Г., Херли Э., Робертс С.Дж. и др. (2008). «Раскопки прошлых структур населения путем выборки на основе фамилий: генетическое наследие викингов в северо-западной Англии» . Молекулярная биология и эволюция . 25 (2): 301–309. дои : 10.1093/molbev/msm255 . ПМЦ 2628767 . ПМИД 18032405 .
- Брандит, Г.; и др. (Генографический консорциум) (2013). «Древняя ДНК раскрывает ключевые этапы формирования генетического разнообразия митохондрий Центральной Европы» . Наука . 342 (6155): 257–261. Бибкод : 2013Sci...342..257B . дои : 10.1126/science.1241844 . ПМК 4039305 . ПМИД 24115443 .
- Капелли С., Редхед Н., Абернети Дж.К., Гратрикс Ф., Уилсон Дж.Ф., Моен Т., Хервиг Т., Ричардс М. и др. (2003). «Перепись хромосом AY на Британских островах» (PDF) . Современная биология . 13 (11): 979–84. Бибкод : 2003CBio...13..979C . дои : 10.1016/S0960-9822(03)00373-7 . ПМИД 12781138 . S2CID 526263 . Архивировано (PDF) из оригинала 8 июля 2021 г. Проверено 8 августа 2020 г. также в «Университетский колледж Лондона» (PDF) . Архивировано (PDF) из оригинала 17 января 2017 г. Проверено 29 января 2007 г.
- Чуньсян Ли; Хунцзе Ли; Иньцю Цуй; Чэнчжи Се; Давэй Цай; Вэньин Ли; Виктор Х Майр; Чжи Сюй; и др. (2010). «Свидетельства того, что смешанное население Запада и Востока жило в Таримской котловине еще в раннем бронзовом веке» . БМК Биология . 8 (1): 15. дои : 10.1186/1741-7007-8-15 . ПМЦ 2838831 . ПМИД 20163704 .
- Ди Кристофаро Дж., Пеннарун Э., Мазьер С., Майрес Н.М., Лин А.А., Темори С.А., Метспалу М., Метспалу Е. и др. (2013). «Афганский Гиндукуш: место, где сходятся потоки генов Евразийского субконтинента» . ПЛОС ОДИН . 8 (10). е76748. Бибкод : 2013PLoSO...876748D . дои : 10.1371/journal.pone.0076748 . ПМЦ 3799995 . ПМИД 24204668 .
- Дюпюи Б.М., Стенерсен М., Лу Т.Т., Олайсен Б. (2005). «Географическая гетерогенность линий Y-хромосомы в Норвегии» (PDF) . Международная судебно-медицинская экспертиза . 164 (1): 10–19. doi : 10.1016/j.forsciint.2005.11.009 . ПМИД 16337760 . Архивировано (PDF) из оригинала 22 июля 2012 г. Проверено 18 ноября 2009 г.
- Форнарино, Симона; Пала, Мария; Батталья, Винченца; Маранта, Рамона; Ахилли, Алессандро; Модиано, Гвидо; Торрони, Антонио; Семино, Орнелла; и др. (2009). «Митохондриальное и Y-хромосомное разнообразие Таруса (Непал): резервуар генетических вариаций» . Эволюционная биология BMC . 9 (1): 154. Бибкод : 2009BMCEE...9..154F . дои : 10.1186/1471-2148-9-154 . ПМК 2720951 . ПМИД 19573232 .
- Фу, Цяомэй; и др. (2 мая 2016 г.). «Генетическая история Европы ледникового периода» . Природа . 534 (7606): 200–205. Бибкод : 2016Natur.534..200F . дои : 10.1038/nature17993 . hdl : 10211.3/198594 . ПМЦ 4943878 . ПМИД 27135931 .
- Гругни В., Батталья В., Кашани Б.Х., Пароло С., Аль-Захери Н., Ахилли А., Оливьери А., Гандини Ф., Хоушманд М., Санати М.Х., Торрони А., Семино О (2012). «Древние миграционные события на Ближнем Востоке: новые данные по Y-хромосомной вариации современных иранцев» . ПЛОС ОДИН . 7 (7). е41252. Бибкод : 2012PLoSO...741252G . дои : 10.1371/journal.pone.0041252 . ПМК 3399854 . ПМИД 22815981 .
- Гвоздз (2009). «Горы Y-STR в гаплопространстве, Часть II: Приложение к общим польским кладам» (PDF) . Журнал генетической генеалогии . 5 (2). Архивировано (PDF) из оригинала 21 июля 2011 г. Проверено 28 ноября 2009 г.
- Хаак, В.; Брандт, Г.; Джонг, HN d.; Мейер, К.; Гансмейер, Р.; Хейд, В.; Хоксворт, К.; Пайк, AWG; и др. (2008). «Древняя ДНК, изотопы стронция и остеологический анализ проливают свет на социальную и родственную организацию позднего каменного века» . Труды Национальной академии наук . 105 (47): 18226–18231. Бибкод : 2008PNAS..10518226H . дои : 10.1073/pnas.0807592105 . ПМК 2587582 . ПМИД 19015520 .
- Хаак, Вольфганг; Лазаридис, Иосиф; Паттерсон, Ник; Роланд, Надин; Маллик, Свапан; Ламас, Бастьен; Брандт, Гвидо; Норденфельт, Сюзанна; и др. (2015). «Массовая миграция из степи является источником индоевропейских языков в Европе» . Природа . 522 (7555): 207–211. arXiv : 1502.02783 . Бибкод : 2015Natur.522..207H . bioRxiv 10.1101/013433 . дои : 10.1038/NATURE14317 . ПМК 5048219 . ПМИД 25731166 .
- Хабер М., Платт Д.Э., Ашрафиан Бонаб М., Юханна С.К., Сориа-Эрнанц Д.Ф., Мартинес-Крус Б., Дуайхи Б., Гассибе-Саббах М. и др. (2012). «Этнические группы Афганистана имеют общее Y-хромосомное наследие, структурированное историческими событиями» . ПЛОС ОДИН . 7 (3). е34288. Бибкод : 2012PLoSO...734288H . дои : 10.1371/journal.pone.0034288 . ПМЦ 3314501 . ПМИД 22470552 .
- Карафет Татьяна М.; Мендес, Фернандо Л.; Судойо, Херавати; Лансинг, Дж. Стивен; Хаммер, Майкл Ф. (2014). «Улучшенное филогенетическое разрешение и быстрая диверсификация гаплогруппы Y-хромосомы K-M526 в Юго-Восточной Азии» . Природа . 23 (3): 369–373. дои : 10.1038/ejhg.2014.106 . ПМЦ 4326703 . ПМИД 24896152 .
- Касперавичюте, Д.; Кучинскас, В.; Стоункинг, М. (2005). «Вариации Y-хромосомы и митохондриальной ДНК у литовцев». Анналы генетики человека . 68 (5): 438–452. дои : 10.1046/j.1529-8817.2003.00119.x . ПМИД 15469421 . S2CID 26562505 .
- Кайзер М., Лао О., Анслингер К., Августин С., Баргель Г., Эдельманн Дж., Элиас С., Генрих М. и др. (2005). «Значительная генетическая дифференциация между Польшей и Германией соответствует современным политическим границам, как показал анализ Y-хромосомы» (PDF) . Генетика человека . 117 (5): 428–443. дои : 10.1007/s00439-005-1333-9 . ПМИД 15959808 . S2CID 11066186 . Архивировано из оригинала (PDF) 4 марта 2009 г.
- Кейзер, Кристина; Буаказе, Кэролайн; Крубези, Эрик; Николаев Валерий Георгиевич; Монтаньон, Даниэль; Рейс, Татьяна; Людес, Бертран (2009). «Древняя ДНК дает новое представление об истории южносибирских курганцев». Генетика человека . 126 (3): 395–410. дои : 10.1007/s00439-009-0683-0 . ПМИД 19449030 . S2CID 21347353 .
- Ким, Киджон; Бреннер, Чарльз Х.; Майр, Виктор Х.; Ли, Кван-Хо; Ким, Джэ Хён; Гелегдорж, Эрегзен; Батболд, Нацаг; Сон, И-Чунг; и др. (2010). «Западно-евразийский мужчина найден на 2000-летнем элитном кладбище хунну на северо-востоке Монголии». Американский журнал физической антропологии . 142 (3): 429–440. дои : 10.1002/ajpa.21242 . ПМИД 20091844 .
- Кивисилд, Т; Рутси, С; Метспалу, М; Мастана, С; Калдма, К; Парик, Дж; Метспалу, Э; Адохаан, М; и др. (2003). «Генетическое наследие первых поселенцев сохраняется как в индийских племенных, так и в кастовых популяциях» . АДЖХГ . 72 (2): 313–32. дои : 10.1086/346068 . ПМК 379225 . ПМИД 12536373 .
- Лазаридис, Иосиф; и др. (2016). «Геномный взгляд на происхождение земледелия на древнем Ближнем Востоке» . Природа . 536 (7617): 419–424. Бибкод : 2016Natur.536..419L . дои : 10.1038/nature19310 . ПМЦ 5003663 . ПМИД 27459054 .
- Лелл Дж.Т., Сукерник Р.И., Стариковская Ю.Б., Су Б., Джин Л., Шурр Т.Г., Андерхилл П.А., Уоллес округ Колумбия (2002). «Двойное происхождение и сибирское сходство Y-хромосом коренных американцев» (PDF) . Американский журнал генетики человека . 70 (1): 192–206. дои : 10.1086/338457 . ПМЦ 384887 . ПМИД 11731934 . Архивировано из оригинала (PDF) 22 апреля 2003 г.
- Лю Шу-ху; Низам Илихаму; Рабияму Баке; Абдукерам Бупатима; Долкун Матюсуп (2018). «Исследование генетического разнообразия трех изолированных популяций в Синьцзяне с использованием Y-SNP». Acta Anthropologica Sinica . 37 (1): 146–156.
- Карлос Килес (10 сентября 2018 г.). «Исследование генетического разнообразия трех изолированных популяций в Синьцзяне с использованием Y-SNP» . Индоевропейский . Архивировано из оригинала 20 сентября 2020 года . Проверено 24 июля 2020 г.
- Мальмстрем, Хелена; Гюнтер, Торстен; Свенссон, Эмма М.; Юрас, Анна; Фрейзер, Магдалена; Мунтерс, Ариэль Р.; Поспешный, Лукаш; Тырв, Мари; и др. (9 октября 2019 г.). «Геномное происхождение людей скандинавской культуры боевых топоров и их связь с более широким горизонтом шнуровой керамики» . Труды Королевского общества Б. 286 (1912). дои : 10.1098/rspb.2019.1528 . ПМК 6790770 . ПМИД 31594508 .
- Малярчук Борис; Деренко, Мирослава; Возняк, Марцин; Гжибовский, Томаш (2013). «Вариация Y-хромосомы у таджиков и иранцев». Анналы биологии человека . 40 (1): 48–54. дои : 10.3109/03014460.2012.747628 . ПМИД 23198991 . S2CID 2752490 .
- Маскареньяс, Десмонд Д.; Райна, Анупума; Астон, Кристофер Э.; Сангера, Дхарабир К. (2015). «Генетическая и культурная реконструкция миграции древней линии» . БиоМед Исследования Интернэшнл . 2015 : 651415. doi : 10.1155/2015/651415 . ПМК 4605215 . ПМИД 26491681 .
- Мэтисон, Иэн; Лазаридис, Иосиф; Роланд, Надин; Маллик, Свапан; Паттерсон, Ник; Алпаслан Руденберг, Сонгул; Харни, Эдаоин; Стюардсон, Кристин; и др. (2015). «Восемь тысяч лет естественного отбора в Европе». биоRxiv 10.1101/016477 .
- Мирабал, Шейла; Регейро, М; Каденас, AM; Кавалли-Сфорца, LL; Андерхилл, Пенсильвания; Вербенко Д.А.; Лимборска, ЮАР; Эррера, Р.Дж.; и др. (2009). «Распространение Y-хромосомы в геолингвистическом ландшафте северо-запада России» . Европейский журнал генетики человека . 17 (10): 1260–1273. дои : 10.1038/ejhg.2009.6 . ПМЦ 2986641 . ПМИД 19259129 .
- Мохаммад Т., Сюэ Ю., Эвисон М., Тайлер-Смит С. (2009). «Генетическая структура кочевых бедуинов из Кувейта» . Наследственность . 103 (5): 425–433. дои : 10.1038/hdy.2009.72 . ПМК 2869035 . ПМИД 19639002 .
- Нарасимхан, Вагиш М.; Энтони, Дэвид; Мэллори, Джеймс; Райх, Дэвид (2018). «Геномное формирование Южной и Центральной Азии». bioRxiv 10.1101/292581 .
- Нарасимхан, Вагиш М.; Паттерсон, Нью-Джерси; Мурджани, Прия; Роланд, Надин; и др. 365 Science, , PMID eaat7487 ( 6457): doi : 10.1126 , PMC 6822619 , science.aat7487 /
- Насидзе И., Линг Е.Ю., Куинке Д., Дюпанлуп И., Кордо Р., Рычков С., Наумова О., Жукова О. и др. (2004). «Митохондриальная ДНК и вариации Y-хромосомы на Кавказе» (PDF) . Анналы генетики человека . 68 (Часть 3): 205–221. дои : 10.1046/j.1529-8817.2004.00092.x . ПМИД 15180701 . S2CID 27204150 . Архивировано из оригинала (PDF) 30 октября 2004 г.
- Насидзе И., Куинке Д., Озтюрк М., Бендукидзе Н., Стоункинг М. (2005). «Вариации мтДНК и Y-хромосомы в курдских группах» (PDF) . Анналы генетики человека . 69 (Часть 4): 401–412. дои : 10.1046/j.1529-8817.2005.00174.x . ПМИД 15996169 . S2CID 23771698 . Архивировано из оригинала (PDF) 23 августа 2009 г.
- Памджав, Хоролма; Фехер, Тибор; Немет, Эндре; Падар, Жолт (2012), «Краткое сообщение: новые бинарные маркеры Y-хромосомы улучшают филогенетическое разрешение в гаплогруппе R1a1», Американский журнал физической антропологии , 149 (4): 611–615, doi : 10.1002/ajpa.22167 , PMID 23115110 , S2CID 4820868
- Пассарино Г., Каваллери Г.Л., Лин А.А., Кавалли-Сфорца Л.Л., Борресен-Дейл А.Л., Андерхилл (2002). «Различные генетические компоненты норвежской популяции, выявленные с помощью анализа полиморфизмов мтДНК и Y-хромосомы» . Европейский журнал генетики человека . 10 (9): 521–529. дои : 10.1038/sj.ejhg.5200834 . ПМИД 12173029 .
- Патхак, Аджай К.; Кадиан, Анураг; Кушнеревич Алена; Монтинаро, Франческо; Мондал, Маюх; Онгаро, Линда; Сингх, Манвендра; Кумар, Прамод; и др. (6 декабря 2018 г.). «Генетическое происхождение современного населения долины Инда из северо-западной Индии» . Американский журнал генетики человека . 103 (6): 918–929. дои : 10.1016/j.ajhg.2018.10.022 . ПМК 6288199 . ПМИД 30526867 .
- Павловский, Р; Детлафф-Какол, А; Макиеевска, А; Пашковска, Р; Райхерт, М; Езерски, Г. (2002). «Популяционная генетика 9 локусов STR Y-хромосомы в Северной Польше». Арх. Мед. Садова Крыминол . 52 (4): 261–277. ПМИД 14669672 .
- Перичич М., Лаук Л.Б., Кларич И.М., Роотси С., Яничиевич Б., Рудан И., Терзич Р., Чолак И. и др. (2005). «Филогенетический анализ Юго-Восточной Европы с высоким разрешением прослеживает основные эпизоды потока отцовских генов среди славянских популяций» . Мол. Биол. Эвол . 22 (10): 1964–75. дои : 10.1093/molbev/msi185 . ПМИД 15944443 .
- Позник Г.Д. и др. (2016). «Периодические всплески демографии мужского пола, выявленные на основе 1244 последовательностей Y-хромосомы по всему миру» . Природная генетика . 48 (6): 593–599. дои : 10.1038/ng.3559 . hdl : 11858/00-001M-0000-002A-F024-C . ПМЦ 4884158 . ПМИД 27111036 .
- Регейро, М; Каденас, AM; Гайден, Т; Андерхилл, Пенсильвания; Эррера, Р.Дж. (2006). «Иран: трехконтинентальная связь для миграции, вызванной Y-хромосомой». Хум Херед . 61 (3): 132–143. дои : 10.1159/000093774 . ПМИД 16770078 . S2CID 7017701 .
- Рико Ф., Кейзер-Тракки С., Буржуа I, Крубези Э., Людес Б. (2004). «Генетический анализ скифо-сибирского скелета и его значение для древних миграций в Центральной Азии». Биология человека . 76 (1): 109–25. дои : 10.1353/hub.2004.0025 . ПМИД 15222683 . S2CID 35948291 .
- Россер Ж.Х., Зержал Т., Хёрлес М.Е., Адохаан М., Алавантик Д., Аморим А., Амос В., Арментерос М. и др. (2000). «Разнообразие Y-хромосомы в Европе является клинальным и зависит в первую очередь от географии, а не от языка» . Американский журнал генетики человека . 67 (6): 1526–1543. дои : 10.1086/316890 . ПМЦ 1287948 . ПМИД 11078479 .
- Сааг, Лехти; Варул, Лийви; Шейб, Кристиана Лин; Стендерап, Йеспер; Аллентофт, Мортен Э.; Сааг, Лаури; Пагани, Лука; Рейдла, Маере; и др. (24 июля 2017 г.). «Экстенсивное земледелие в Эстонии началось благодаря миграции из степи по признаку пола» . Современная биология . 27 (14). Cell Press : 2185–2193. Бибкод : 2017CBio...27E2185S . дои : 10.1016/j.cub.2017.06.022 . ПМИД 28712569 .
- Saag, Lehti; Vasilyev, Sergey V.; Varul, Liivi; Kosorukova, Natalia V.; Gerasimov, Dmitri V.; Oshibkina, Svetlana V.; Griffith, Samuel J.; Solnik, Anu; et al. (July 3, 2020). "Genetic ancestry changes in Stone to Bronze Age transition in the East European plain". bioRxiv 10.1101/2020.07.02.184507 .
- Саху, С; Сингх, А; Химабинду, Г; Банерджи, Дж; Ситалакшими, Т; Гайквад, С; Триведи, Р; Эндикотт, П; и др. (2006). «Предыстория индийских Y-хромосом: оценка сценариев демического распространения» . Труды Национальной академии наук . 103 (4): 843–848. Бибкод : 2006PNAS..103..843S . дои : 10.1073/pnas.0507714103 . ПМЦ 1347984 . ПМИД 16415161 .
- Скоццари Р., Кручиани Ф., Панграцио А., Сантоламазца П., Вона Дж., Морал П., Латини В., Варези Л. и др. (2001). «Вариации Y-хромосомы человека в районе Западного Средиземноморья: последствия для заселения региона» (PDF) . Иммунология человека . 62 (9): 871–84. CiteSeerX 10.1.1.408.4857 . дои : 10.1016/S0198-8859(01)00286-5 . ПМИД 11543889 . Архивировано из оригинала (PDF) 17 декабря 2012 года . Проверено 15 марта 2009 г.
- Семенов Александр С.; Булат, Владимир В. (2016), «Древняя палео-ДНК Северо-Восточной Европы домедного века: установление следов миграции гаплогруппы Y-ДНК R1a1», Европейский журнал молекулярной биотехнологии , 11 (1): 40–54 , doi : 10.13187/ejmb.2016.11.40 , S2CID 172131289
- Семино, О; Пассарино, Дж; Оефнер, П.Дж.; Лин, А.А.; Арбузова С; Бекман, Ле; Де Бенедиктис, Дж; Франкалаччи, П; и др. (2000). «Генетическое наследие палеолитического Homo sapiens sapiens у современных европейцев: хромосомная перспектива AY» (PDF) . Наука . 290 (5494): 1155–1159. Бибкод : 2000Sci...290.1155S . дои : 10.1126/science.290.5494.1155 . ПМИД 11073453 . Архивировано из оригинала (PDF) 25 ноября 2003 г.
- Сенгупта, С; Животовский, Л.А.; Кинг, Р; Мехди, SQ; Эдмондс, Калифорния; Чоу, CE; Лин, А.А.; Митра, М; и др. (2006). «Полярность и временность распределения Y-хромосомы с высоким разрешением в Индии определяют как местное, так и экзогенное распространение и выявляют незначительное генетическое влияние скотоводов Центральной Азии» . Американский журнал генетики человека . 78 (2): 202–21. дои : 10.1086/499411 . ПМК 1380230 . ПМИД 16400607 .
- Шарма, С; Рай, Э; Шарма, П; Йена, М; Сингх, С; Дарвиши, К; Бхат, АК; Бханвер, AJ; и др. (2009). «Индийское происхождение отцовской гаплогруппы R1a1(*) обосновывает автохтонное происхождение брахманов и кастовой системы» . Журнал генетики человека . 54 (1): 47–55. дои : 10.1038/jhg.2008.2 . ПМИД 19158816 .
- Шильц, Феликс (2006). Анализ молекулярно-генетического родства доисторического коллектива скелетов пещеры Лихтенштейн [ Молекулярно-генетический анализ родства доисторического коллектива скелетов пещеры Лихтенштейн ] (PDF) (Диссертация) (на немецком языке). Геттинген: Факультет математики и естественных наук Университета Георга Августа. Архивировано (PDF) из оригинала 8 июня 2011 г. Проверено 9 ноября 2009 г.
- Сильва, Марина; и др. (2017). «Генетическая хронология Индийского субконтинента указывает на сильное расселение по признаку пола» . Эволюционная биология BMC . 17 (1): 88. Бибкод : 2017BMCEE..17...88S . дои : 10.1186/s12862-017-0936-9 . ПМК 5364613 . ПМИД 28335724 .
- Тамбетс К., Роотси С., Кивисилд Т., Хелп Х., Серк П., Лугвяли Э.Л., Толк Х.В., Рейдла М. и др. (2004). «Западные и восточные корни саамов - история генетических «выбросов», рассказанная митохондриальной ДНК и Y-хромосомами» . Американский журнал генетики человека . 74 (4): 661–682. дои : 10.1086/383203 . ПМК 1181943 . ПМИД 15024688 .
- Тангарадж, Кумарасами; Найду, Б. Пратап; Кривелларо, Федерика; Таманг, Ракеш; Упадхьяй, Шашанк; Шарма, Варун Кумар; Редди, Алла Г.; Валимбе, СР; и др. (2010). Кордо, Ричард (ред.). «Влияние естественных барьеров на формирование генетической структуры популяций Махараштры» . ПЛОС ОДИН . 5 (12): e15283. Бибкод : 2010PLoSO...515283T . дои : 10.1371/journal.pone.0015283 . ПМК 3004917 . ПМИД 21187967 .
- Андерхилл, Пенсильвания; Майрес, Нью-Мексико; Рутси, С; Метспалу, М; Животовский, Л.А.; Кинг, Р.Дж.; Лин, А.А.; Чоу, CE; и др. (4 ноября 2009 г.). «Разделение постледникового происхождения европейских и азиатских Y-хромосом в пределах гаплогруппы R1a» . Европейский журнал генетики человека . 18 (4) (опубликовано в апреле 2010 г.): 479–84. дои : 10.1038/ejhg.2009.194 . ПМЦ 2987245 . ПМИД 19888303 .
- Андерхилл, Питер А.; и др. (26 марта 2014 г.). «Филогенетическая и географическая структура гаплогруппы R1a Y-хромосомы» . Европейский журнал генетики человека . 23 (1) (опубликовано в январе 2015 г.): 124–131. дои : 10.1038/ejhg.2014.50 . ПМК 4266736 . ПМИД 24667786 . «ПДФ» (PDF) . Архивировано из оригинала (PDF) 16 августа 2016 года . Проверено 12 июня 2016 г.
- Ван, Вэй; Мудрая, Шерил; Барик, Том; Блэк, Майкл Л.; Биттлс, Алан Х. (2003). «Происхождение и генетическая структура трех совместно проживающих в Китае мусульманских популяций: Салар, Боань и Дунсян». Генетика человека . 113 (3): 244–52. дои : 10.1007/s00439-003-0948-y . ПМИД 12759817 . S2CID 11138499 .
- Уэллс, Р.С. (2001), «Евразийский центр: континентальный взгляд на разнообразие Y-хромосомы», Труды Национальной академии наук США , 98 (18): 10244–10249, Бибкод : 2001PNAS...9810244W , doi : 10.1073/pnas.171305098 , PMC 56946 , PMID 11526236
- Ян, Ши; Ван, Чуан-Чао; Чжэн, Хун-Сян; Ван, Вэй; Цинь, Чжэнь-Дун; Вэй, Лан-Хай; Ван, Йи; Пан, Сюэ-Донг; и др. (29 августа 2014 г.). «Y-хромосомы 40% китайцев происходят от трех неолитических супердедушек» . ПЛОС ОДИН . 9 (8). е105691. arXiv : 1310.3897 . Бибкод : 2014PLoSO...9j5691Y . дои : 10.1371/journal.pone.0105691 . ПМК 4149484 . ПМИД 25170956 .
- Консорциум Y-хромосомы «YCC» (2002). «Система номенклатуры дерева бинарных гаплогрупп Y-хромосомы человека» . Геномные исследования . 12 (2): 339–348. дои : 10.1101/гр.217602 . ПМК 155271 . ПМИД 11827954 .
- Зержал, Татьяна; Уэллс, Р. Спенсер; Юлдашева, Надира; Рузибакиев Руслан; Тайлер-Смит, Крис (2002). «Генетический ландшафт, измененный недавними событиями: взгляд на Y-хромосому в Центральной Азии» . Американский журнал генетики человека . 71 (3): 466–82. дои : 10.1086/342096 . ПМК 419996 . ПМИД 12145751 .
- Чжун Х, Ши Х, Ци XB, Дуань Ю, Тан П.П., Цзинь Л, СУ Б, Ма РЗ (январь 2011 г.). «Расширенное исследование Y-хромосомы позволяет предположить, что современные люди мигрировали в Восточную Азию северным путем после ледникового периода» . Молекулярная биология и эволюция . 28 (1): 717–27. дои : 10.1093/molbev/msq247 . ПМИД 20837606 .
- Чжоу, Жуйся; Ан, Лиже; Ван, Сюньлин; Шао, Вэй; Линь, Гунхуа; Ю, Вэйпин; Йи, Лин; Сюй, Шицзянь; и др. (2007). «Проверка гипотезы о древнеримском солдатском происхождении народа Лицянь на северо-западе Китая: взгляд на Y-хромосому» . Журнал генетики человека . 52 (7): 584–91. дои : 10.1007/s10038-007-0155-0 . ПМИД 17579807 .
Дальнейшее чтение
[ редактировать ]- Гимбутас (1970). Индоевропейцы и индоевропейцы . Филадельфия, Пенсильвания: Univ. из Пенсильвании Пресс. стр. 155–195.
- Патриция Маласпина; Андрей Иванович Козлов; Фульвио Кручиани; Пьеро Сантоламазца; Неджат Акар; Димитр Ковачев; Керимова Марина Георгиевна; Юрий Парик; Ричард Виллемс; Розана Скоццари; Андреа Новеллетто (2003). «Анализ вариаций Y-хромосомы в современных популяциях на европейско-азиатской границе» (PDF) . У К. Бойля; К. Ренфрю; М. Левин (ред.). Древние взаимодействия: Восток и Запад в Евразии . Монографии Института Макдональда. Кембридж: Издательство Кембриджского университета. стр. 309–313. Архивировано из оригинала (PDF) 24 июня 2023 года . Проверено 10 сентября 2009 г.
- Парпола, Аско (2015). Корни индуизма. Ранние арийцы и цивилизация Инда . Издательство Оксфордского университета.
- Шарма, С. (2007). «1344/T: Автохтонное происхождение и племенная связь индийских браминов: оценка с помощью молекулярно-генетических маркеров». 57-е ежегодное собрание Американского общества генетики человека, 23–27 октября 2007 г.; Сан-Диего, Калифорния (PDF) (Аннотация). п. 273. Архивировано из оригинала (PDF) 26 июня 2008 г.
- Траутманн, Томас (2005). Арийские дебаты . Издательство Оксфордского университета.
- Варзари, Александр (2006). Популяционная история Днестровско-Карпат: данные по вставке Alu и полиморфизму Y-хромосомы (PDF) (Диссертация). Мюнхен: Университет Людвига-Максимилиана. Архивировано (PDF) из оригинала 28 октября 2008 г. Проверено 30 октября 2008 г.
- Уэллс, Спенсер (2002). Путешествие человека: генетическая одиссея . Издательство Принстонского университета. ISBN 978-0-691-11532-0 .
журнальные статьи |
|---|
Внешние ссылки
[ редактировать ]- ДНК-дерево
- Проект гаплогруппы FTDNA R1a Y-хромосомы
- Проект R1a1a1 и субкладов Y-ДНК - Фоновое генеалогическое древо ДНК R1a1a1
- ТМРЦА
- Различный
- Датский региональный проект ДНК Демеса: Гаплогруппа Y-ДНК R1a. Архивировано 20 августа 2008 г. в Wayback Machine.
- Блог Eurogenes, Полтовский выброс
- Авотайну Онлайн, Отпечаток Y-ДНК Шполера Зейды, цадика, коснувшегося мира