ДНК-метилтрансфераза
| ДНК-метилаза N-6 | |||
|---|---|---|---|
 кристаллическая структура белка рестрикции i типа ecoki m (ec 2.1.1.72) (m.ecoki) | |||
| Идентификаторы | |||
| Символ | N6_Mtase | ||
| Пфам | PF02384 | ||
| Пфам Клан | CL0063 | ||
| ИнтерПро | ИПР003356 | ||
| PROSITE | PDOC00087 | ||
| |||
| N-концевой домен HsdM | |||
|---|---|---|---|
| Идентификаторы | |||
| Символ | HsdM_N | ||
| Пфам | PF12161 | ||
| |||
| C-5 цитозинспецифическая ДНК-метилаза | |||
|---|---|---|---|
 структура человеческого dnmt2, загадочного гомолога ДНК-метилтрансферазы | |||
| Идентификаторы | |||
| Символ | ДНК_метилаза | ||
| Пфам | PF00145 | ||
| Пфам Клан | CL0063 | ||
| ИнтерПро | ИПР001525 | ||
| PROSITE | PDOC00089 | ||
| СКОП2 | 1хмы / ОБЛАСТЬ ПРИМЕНЕНИЯ / СУПФАМ | ||
| CDD | cd00315 | ||
| |||
| ДНК-метилаза | |||
|---|---|---|---|
 кристаллическая структура метилтрансферазы mboia (moraxella bovis) | |||
| Идентификаторы | |||
| Символ | N6_N4_Mtase | ||
| Пфам | PF01555 | ||
| Пфам Клан | CL0063 | ||
| ИнтерПро | ИПР002941 | ||
| PROSITE | PDOC00088 | ||
| СКОП2 | 1boo / SCOPe / СУПФАМ | ||
| |||
В биохимии ДНК -метилтрансферазы ( ДНК-МТаза , DNMT семейство ферментов ) катализирует перенос метильной группы на ДНК . Метилирование ДНК выполняет широкий спектр биологических функций. Все известные ДНК-метилтрансферазы используют S-аденозилметионин (SAM) в качестве донора метила.
Классификация
[ редактировать ]Субстрат
[ редактировать ]МТазы можно разделить на три различные группы в зависимости от химических реакций, которые они катализируют:
- m6A - генерирующие N6-метиладенин КЭ 2.1.1.72
- m4C - генерирующие N4-метилцитозин КЭ 2.1.1.113
- m5C - генерирующие С5-метилцитозин EC 2.1.1.37
Метилтрансферазы m6A и m4C обнаружены преимущественно у прокариот (хотя недавние данные свидетельствуют о том, что m6A в изобилии встречается у эукариот). [1] ). Метилтрансферазы m5C обнаружены у некоторых низших эукариот, у большинства высших растений и у животных, начиная с иглокожих .
Метилтрансферазы m6A (N-6-аденин-специфическая ДНК-метилаза) (A-Mtase) представляют собой ферменты , которые специфически метилируют аминогруппу в положении C-6 аденинов в ДНК. Они обнаружены в трех существующих типах бактериальных рестрикционно-модификационных систем (в системе I типа А-Мтаза является продуктом гена hsdM, а в типе III — продуктом гена mod). Эти ферменты ответственны за метилирование определенных последовательностей ДНК , чтобы предотвратить переваривание хозяином собственного генома с помощью ферментов рестрикции . Эти метилазы обладают той же специфичностью последовательности , что и соответствующие им ферменты рестрикции. Эти ферменты содержат консервативный мотив Asp / Asn - Pro -Pro- Tyr / Phe в своем N-концевом участке , этот консервативный участок может участвовать в субстрата связывании или в каталитической активности. [2] [3] [4] [5] Структура N6-MTase TaqI ( M.TaqI разрешена до 2,4 А. ) Молекула сайты складывается в 2 домена: N-концевой каталитический домен, который содержит связывания каталитического и кофактора , и включает центральный 9-нитевой бета-лист, окруженный 5 спиралями; и С-концевой домен распознавания ДНК, который образован 4 небольшими бета-листами и 8 альфа-спиралями . N- и C-концевые домены образуют щель, в которой размещается ДНК- субстрат . [6] Была предложена классификация N-MTases, основанная на расположении консервативных мотивов (CM). [5] Согласно этой классификации, N6-Мтазы, имеющие мотив DPPY (CM II), расположенный после мотива FxGxG (CM I), обозначаются N6-адениновыми МТазами класса D12. Система рестрикции и модификации типа I состоит из трех полипептидов M (hsdM) и S R, M и S. Субъединицы вместе образуют метилтрансферазу , которая метилирует два адениновых остатка в комплементарных цепях двудольной последовательности распознавания ДНК . В присутствии субъединицы R комплекс также может действовать как эндонуклеаза , связываясь с той же целевой последовательностью , но разрезая ДНК на некотором расстоянии от этого сайта. Будет ли ДНК разрезана или модифицирована, зависит от состояния метилирования целевой последовательности . Когда целевой сайт не модифицирован, ДНК разрезается. Когда целевой сайт гемиметилирован, комплекс действует как поддерживающая метилтрансфераза, модифицируя ДНК так, что обе цепи становятся метилированными . hsdM содержит альфа-спиральный домен на N-конце , N-концевой домен HsdM. [7]
Среди m6A-метилтрансфераз (N-6-аденин-специфическая ДНК-метилаза) существует группа орфанных МТаз, не участвующих в бактериальной системе рестрикции/метилирования. [8] Эти ферменты играют регулирующую роль в экспрессии генов и регуляции клеточного цикла. EcoDam из кишечной палочки [9] и CcrM из Caulobacter crescentus [10] являются хорошо охарактеризованными членами этого семейства. Совсем недавно было показано, что CamA из Clostridioides difficile играет ключевую функциональную роль в споруляции , образовании биопленок и адаптации к хозяину. [11]
Метилтрансферазы m4C (специфичные для цитозина ДНК-метилазы N-4) представляют собой ферменты , которые специфически метилируют аминогруппу в положении C-4 цитозинов в ДНК. [5] Такие ферменты встречаются как компоненты систем рестрикции-модификации типа II у прокариот . Такие ферменты распознают определенную последовательность ДНК и метилируют цитозин в этой последовательности . Этим действием они защищают ДНК от расщепления ферментами рестрикции типа II, которые распознают ту же последовательность. [ нужна ссылка ]
Метилтрансферазы m5C (цитозин-специфическая ДНК-метилаза C-5) (C5 Mtase) представляют собой ферменты, которые специфически метилируют углерод в C- 5 цитозинов ДНК с образованием C5-метилцитозина . [12] [13] [14] В клетках млекопитающих цитозинспецифические метилтрансферазы метилируют определенные последовательности CpG , которые, как полагают, модулируют экспрессию генов и дифференцировку клеток . У бактерий эти ферменты являются компонентом систем рестрикции-модификации и служат ценными инструментами для манипулирования ДНК. [13] [15] Структура домен метилтрансферазы HhaI (M.HhaI) разрешена до 2,5 А : молекула складывается в 2 домена - более крупный каталитический , содержащий сайты связывания каталитических и кофакторов , и меньший домен узнавания ДНК. [16]
Сообщалось о высококонсервативных ДНК-метилтрансферазах типов m4C, m5C и m6A. [17] которые представляются многообещающими целями для разработки новых эпигенетических ингибиторов для борьбы с бактериальной вирулентностью, устойчивостью к антибиотикам, а также для других биомедицинских применений.
Опять против. обслуживание
[ редактировать ]Метилтрансферазы de novo распознают в ДНК что-то, что позволяет им заново метилировать цитозины. Они экспрессируются главным образом на ранних стадиях развития эмбриона и определяют структуру метилирования. Метилтрансферазы de novo также активны, когда клетка, реагирующая на сигнал, такая как нейрон , должна изменить экспрессию белка. [18] Например, когда обусловленность страхом создает у крысы новые воспоминания , 9,17% генов в геноме нейронов гиппокампа крысы дифференциально метилированы. [19]
Поддерживающие метилтрансферазы добавляют метилирование к ДНК, когда одна цепь уже метилирована. Они работают на протяжении всей жизни организма, поддерживая структуру метилирования, установленную метилтрансферазами de novo. [ нужна ссылка ]
млекопитающее
[ редактировать ]У млекопитающих идентифицированы по крайней мере четыре ДНК-метилтрансферазы различной активности. Они называются DNMT1 , [20] две изоформы, транскрибируемые из гена DNMT3a : DNMT3a1 и DNMT3a2, [21] и DNMT3b . [22] Недавно был обнаружен еще один фермент DNMT3c, специфически экспрессирующийся в мужской зародышевой линии мышей. [23]
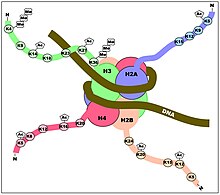
[ нужна ссылка ]

Манзо и др. [24] наблюдали различия в геномном связывании DNMT3a1, DNMT3a2 и DNMT3b. Они обнаружили 3970 областей, обогащенных исключительно DNMT3a1, 3838, обогащенных исключительно DNMT3a2, и 3432, обогащенных исключительно DNMT3b.
Ферменты DNMT регулируются не только в местах их метилирования в геноме, но и в том месте, где они связываются с ДНК. [24] но они также регулируются посттрансляционными модификациями гистоновых которых белков нуклеосом, вокруг обернута геномная ДНК (см. рисунки). Роуз и Клозе [25] рассмотрели взаимосвязь между метилированием ДНК и гистонов метилированием лизина . Например, они указали, что H3K4me3, по-видимому, блокирует метилирование ДНК, тогда как H3K9me3 играет роль в стимулировании метилирования ДНК.
ДНМТ3Л [26] представляет собой белок, тесно связанный с DNMT3a и DNMT3b по структуре и имеющий решающее значение для метилирования ДНК, но, по-видимому, сам по себе неактивен.
ДНМТ1
[ редактировать ]DNMT1 является наиболее распространенной ДНК-метилтрансферазой в клетках млекопитающих и считается ключевой поддерживающей метилтрансферазой у млекопитающих . Он преимущественно метилирует гемиметилированные CpG динуклеотиды в геноме млекопитающих. Мотив узнавания человеческого фермента включает только три основания в паре динуклеотидов CpG: C на одной цепи и CpG на другой. Это смягченное требование к специфичности субстрата позволяет ему метилировать необычные структуры, такие как промежуточные соединения проскальзывания ДНК, со скоростью de novo, которая равна скорости его поддержания. [27] Как и другие ДНК-цитозин-5-метилтрансферазы, человеческий фермент распознает перевернутые цитозины в двухцепочечной ДНК и действует по механизму нуклеофильной атаки. [28] В раковых клетках человека DNMT1 отвечает как за метилирование de novo , так и за поддерживающее метилирование генов-супрессоров опухоли. [29] [30] Фермент аминокислот имеет длину около 1620 . Первые 1100 аминокислот составляют регуляторный домен фермента, а остальные остатки составляют каталитический домен. К ним присоединяется Гли – повторяет Лис . Оба домена необходимы для каталитической функции DNMT1. [ нужна ссылка ]
DNMT1 имеет несколько изоформ : соматическую DNMT1, сплайсинговый вариант (DNMT1b) и ооцит -специфическую изоформу (DNMT1o). DNMT1o синтезируется и хранится в цитоплазме ооцита и транслоцируется в ядро клетки на ранних стадиях эмбрионального развития, тогда как соматический DNMT1 всегда обнаруживается в ядре соматической ткани. [ нужна ссылка ]
с нулевой мутацией DNMT1 Эмбриональные стволовые клетки были жизнеспособны и содержали небольшой процент метилированной ДНК и активности метилтрансферазы. Мышиные эмбрионы, гомозиготные по делеции Dnmt1, погибают на 10–11 день беременности. [31]
ТРДМТ1
[ редактировать ]Хотя этот фермент имеет сильное сходство последовательностей с 5-метилцитозинметилтрансферазами как прокариот, так и эукариот, в 2006 году было показано, что фермент метилирует положение 38 в транспортной РНК аспарагиновой кислоты и не метилирует ДНК. [32] Название этой метилтрансферазы было изменено с DNMT2 на TRDMT1 (тРНК метилтрансфераза аспарагиновой кислоты 1), чтобы лучше отражать ее биологическую функцию. [33] TRDMT1 — первая РНК-цитозинметилтрансфераза, идентифицированная в клетках человека.
ДНМТ3
[ редактировать ]DNMT3 представляет собой семейство ДНК- метилтрансфераз, которые могут метилировать гемиметилированные и неметилированные CpG с одинаковой скоростью. Архитектура ферментов DNMT3 аналогична архитектуре DNMT1, с регуляторной областью, прикрепленной к каталитическому домену. Существует как минимум пять членов семейства DNMT3: DNMT3a1, DNMT3a2, 3b, 3c и 3L. [ нужна ссылка ]
DNMT3a1, DNMT3a2 и DNMT3b могут опосредовать метилирование сайтов CpG в промоторах генов, что приводит к репрессии генов . Эти ДНК-метилтрансферазы могут также метилировать сайты CpG в кодирующих областях генов, где такое метилирование может увеличивать транскрипцию генов. [34] Работа с DNMT3a1 показала, что он преимущественно локализован на CpG-островках, бивалентно маркированных H3K4me3 (метка, способствующая транскрипции) и H3K27me3 (метка, репрессирующая транскрипцию), совпадающих с промоторами многих факторов транскрипции . Работа с DNMT3a2 в нейронах показала, что изменения метилирования ДНК, вызванные DNMT3a2, преимущественно происходят в межгенных и интронных областях. Считалось, что эти межгенные и интронные метилирования ДНК, вероятно, регулируют активность энхансеров , альтернативный сплайсинг или экспрессию некодирующих РНК . [35]
DNMT3a1 может совместно локализоваться с белком гетерохроматина (HP1) и метил-CpG-связывающим белком (MeCBP), а также с рядом других факторов. [36] Они также могут взаимодействовать с DNMT1, что может быть сопутствующим событием во время метилирования ДНК. DNMT3a предпочитает CpG метилирование метилированию CpA, CpT и CpC, хотя, по-видимому, существует некоторое предпочтение последовательностей метилирования для DNMT3a и DNMT3b. DNMT3a метилирует сайты CpG со скоростью, намного меньшей, чем DNMT1, но большей, чем DNMT3b.
Экспрессия DNMT3a2 отличается от DNMT3a1 и DNMT3b, поскольку экспрессия DNMT3a2 происходит по образцу непосредственно раннего гена . Экспрессия DNMT3a2 в нейронах индуцируется, например, за счет новой активности нейронов. [37] [35] Это может иметь значение для формирования долговременной памяти . [38] У крыс высокие уровни метилирования новой ДНК в нейронах гиппокампа возникают после того, как крысе было навязано запоминающееся событие, такое как контекстуальное обусловливание страха . [19] Байрактар и Крейц [39] обнаружили, что ингибиторы DNMT, применяемые в мозге, предотвращают формирование долговременных воспоминаний.
DNMT3L содержит мотивы ДНК-метилтрансферазы и необходим для установления материнских геномных отпечатков , несмотря на то, что он каталитически неактивен. DNMT3L экспрессируется во время гаметогенеза , когда геномный импринтинг происходит . Потеря DNMT3L приводит к биаллельной экспрессии генов, обычно не экспрессируемых материнским аллелем. DNMT3L взаимодействует с DNMT3a и DNMT3b и совместно локализуется в ядре. Хотя DNMT3L, по-видимому, неспособен к метилированию , он может участвовать в транскрипции репрессии .
Клиническое значение
[ редактировать ]Ингибиторы ДНМТ
[ редактировать ]Из-за эпигенетических эффектов семейства DNMT некоторые ингибиторы DNMT исследуются для лечения некоторых видов рака: [40]
- Видаза ( азацитидин ) в III фазе исследований по лечению миелодиспластических синдромов и ОМЛ
- Дакоген ( децитабин ) в III фазе исследований по лечению ОМЛ и ХМЛ . ЕС одобрил в 2012 году борьбу с отмыванием денег. [41]
- гуадецитабин, экспериментальный препарат, разрабатываемый компаниями Astex Pharmaceuticals и Otsuka Pharmaceutical. Ему не удалось достичь основных конечных точек в исследовании III фазы AML в 2018 году.
См. также
[ редактировать ]Ссылки
[ редактировать ]- ^ Айер Л.М., Чжан Д., Аравинд Л. (январь 2016 г.). «Метилирование аденина у эукариот: понимание сложной эволюционной истории и функционального потенциала эпигенетической модификации» . Биоэссе . 38 (1): 27–40. doi : 10.1002/bies.201500104 . ПМЦ 4738411 . ПМИД 26660621 .
- ^ Лоенен В.А., Дэниел А.С., Браймер Х.Д., Мюррей Н.Е. (ноябрь 1987 г.). «Организация и последовательность генов hsd Escherichia coli K-12». Журнал молекулярной биологии . 198 (2): 159–70. дои : 10.1016/0022-2836(87)90303-2 . ПМИД 3323532 .
- ^ Нарва К.Э., Ван Эттен Дж.Л., Слатко Б.Е., Беннер Дж.С. (декабрь 1988 г.). «Аминокислотная последовательность эукариотической ДНК [N6-аденин] метилтрансферазы, M.CviBIII, имеет области сходства с прокариотическим изошизомером M.TaqI и другими ДНК [N6-аденин] метилтрансферазами». Джин . 74 (1): 253–9. дои : 10.1016/0378-1119(88)90298-3 . ПМИД 3248728 .
- ^ Лаустер Р. (март 1989 г.). «Эволюция ДНК-метилтрансфераз типа II. Модель дупликации генов». Журнал молекулярной биологии . 206 (2): 313–21. дои : 10.1016/0022-2836(89)90481-6 . ПМИД 2541254 .
- ^ Перейти обратно: а б с Тиминскас А, Буткус В, Янулайтис А (май 1995 г.). «Мотивы последовательности, характерные для ДНК [цитозин-N4] и ДНК [аденин-N6] метилтрансфераз. Классификация всех ДНК-метилтрансфераз». Джин . 157 (1–2): 3–11. дои : 10.1016/0378-1119(94)00783-О . ПМИД 7607512 .
- ^ Лабан Дж., Гранзин Дж., Шлюкебир Г., Робинсон Д.П., Джек В.Е., Шильдкраут И., Сенгер В. (ноябрь 1994 г.). «Трехмерная структура аденинспецифической ДНК-метилтрансферазы M.Taq I в комплексе с кофактором S-аденозилметионином» . Труды Национальной академии наук Соединенных Штатов Америки . 91 (23): 10957–61. дои : 10.1073/pnas.91.23.10957 . ПМК 45145 . ПМИД 7971991 .
- ^ Келлехер Дж.Э., Дэниел А.С., Мюррей Н.Е. (сентябрь 1991 г.). «Мутации, которые придают активность de novo поддерживающей метилтрансферазе». Журнал молекулярной биологии . 221 (2): 431–40. дои : 10.1016/0022-2836(91)80064-2 . ПМИД 1833555 .
- ^ Адхикари С., Кертис П.Д. (сентябрь 2016 г.). «ДНК-метилтрансферазы и эпигенетическая регуляция у бактерий» . Обзоры микробиологии FEMS . 40 (5): 575–91. дои : 10.1093/femsre/fuw023 . ПМИД 27476077 .
- ^ Чахар С., Эльсави Х., Рагозин С., Елч А. (январь 2010 г.). «Изменение специфичности распознавания ДНК ДНК-(аденин-N6)-метилтрансферазы EcoDam путем направленной эволюции». Журнал молекулярной биологии . 395 (1): 79–88. дои : 10.1016/j.jmb.2009.09.027 . ПМИД 19766657 .
- ^ Майер Дж.А., Албу Р.Ф., Юрковски Т.П., Елч А. (декабрь 2015 г.). «Исследование С-концевого домена бактериальной ДНК-(аденин N6)-метилтрансферазы CcrM». Биохимия . 119 : 60–7. дои : 10.1016/j.biochi.2015.10.011 . ПМИД 26475175 .
- ^ Оливейра П.Х., Рибис Дж.В., Гарретт Э.М., Трзилова Д., Ким А., Секулович О. и др. (январь 2020 г.). «Эпигеномная характеристика Clostridioides difficile выявила консервативную ДНК-метилтрансферазу, которая опосредует споруляцию и патогенез» . Природная микробиология . 5 (1): 166–180. дои : 10.1038/s41564-019-0613-4 . ПМК 6925328 . ПМИД 31768029 .
- ^ Посфаи Дж., Бхагват А.С., Робертс Р.Дж. (декабрь 1988 г.). «Мотивы последовательности, специфичные для цитозинметилтрансфераз». Джин . 74 (1): 261–5. дои : 10.1016/0378-1119(88)90299-5 . ПМИД 3248729 .
- ^ Перейти обратно: а б Кумар С., Ченг Х., Климасаускас С., Ми С., Посфаи Дж., Робертс Р.Дж., Уилсон Г.Г. (январь 1994 г.). «ДНК (цитозин-5) метилтрансферазы» . Исследования нуклеиновых кислот . 22 (1): 1–10. дои : 10.1093/нар/22.1.1 . ПМК 307737 . ПМИД 8127644 .
- ^ Лаустер Р., Траутнер Т.А., Нойер-Вейднер М. (март 1989 г.). «Цитозинспецифические ДНК-метилтрансферазы типа II. Консервативное ядро фермента с вариабельными доменами, распознающими мишень». Журнал молекулярной биологии . 206 (2): 305–12. дои : 10.1016/0022-2836(89)90480-4 . ПМИД 2716049 .
- ^ Ченг Икс (февраль 1995 г.). «Модификация ДНК метилтрансферазами». Современное мнение в области структурной биологии . 5 (1): 4–10. дои : 10.1016/0959-440X(95)80003-J . ПМИД 7773746 .
- ^ Ченг X, Кумар С., Посфаи Дж., Пфлуграт Дж.В., Робертс Р.Дж. (июль 1993 г.). «Кристаллическая структура ДНК-метилтрансферазы HhaI в комплексе с S-аденозил-L-метионином». Клетка . 74 (2): 299–307. дои : 10.1016/0092-8674(93)90421-L . ПМИД 8343957 . S2CID 54238106 .
- ^ Оливейра П.Х., Фанг Дж. (май 2020 г.). «Консервативные ДНК-метилтрансферазы: окно в фундаментальные механизмы эпигенетической регуляции у бактерий» . Тенденции в микробиологии . 29 (1): 28–40. дои : 10.1016/j.tim.2020.04.007 . ПМК 7666040 . ПМИД 32417228 .
- ^ МакКлунг, Калифорния, Нестлер Э.Дж. (январь 2008 г.). «Нейропластичность, опосредованная измененной экспрессией генов» . Нейропсихофармакология . 33 (1): 3–17. дои : 10.1038/sj.npp.1301544 . ПМИД 17728700 . S2CID 18241370 .
- ^ Перейти обратно: а б Дьюк К.Г., Кеннеди Эй.Дж., Гэвин К.Ф., Дэй Дж.Дж., Суэтт Дж.Д. (июль 2017 г.). «Эпигеномная реорганизация в гиппокампе, зависящая от опыта» . Изучите Мем . 24 (7): 278–288. дои : 10.1101/lm.045112.117 . ПМК 5473107 . ПМИД 28620075 .
- ^ «ДНМТ1» . Отчет о генных символах . Комитет по генной номенклатуре Хьюго . Архивировано из оригинала 6 октября 2012 г. Проверено 27 сентября 2012 г.
- ^ Чен Т, Уэда Ю, Се С, Ли Э (октябрь 2002 г.). «Новая изоформа Dnmt3a, полученная с помощью альтернативного промотора, локализуется в эухроматине, и ее экспрессия коррелирует с активным метилированием de novo» . J Биол Хим . 277 (41): 38746–54. дои : 10.1074/jbc.M205312200 . ПМИД 12138111 .
- ^ «ДНМТ3Б» . Отчет о генных символах . Комитет по генной номенклатуре Хьюго . Архивировано из оригинала 20 ноября 2012 г. Проверено 27 сентября 2012 г.
- ^ Барау Дж., Тейсандье А., Самудио Н., Рой С., Налессо В., Эро И. и др. (ноябрь 2016 г.). «ДНК-метилтрансфераза DNMT3C защищает мужские половые клетки от активности транспозонов». Наука . 354 (6314): 909–912. Бибкод : 2016Sci...354..909B . дои : 10.1126/science.aah5143 . ПМИД 27856912 . S2CID 30907442 .
- ^ Перейти обратно: а б Манзо М., Вирц Дж., Амбрози С., Вилласеньор Р., Рошицкий Б., Баубек Т. (декабрь 2017 г.). «Изоформ-специфическая локализация DNMT3A регулирует точность метилирования ДНК на двухвалентных CpG-островках» . ЭМБО Дж . 36 (23): 3421–3434. дои : 10.15252/embj.201797038 . ПМЦ 5709737 . ПМИД 29074627 .
- ^ Роуз Н.Р., Клозе Р.Дж. (декабрь 2014 г.). «Понимание связи между метилированием ДНК и метилированием лизина гистонов» . Биохим Биофиз Акта . 1839 (12): 1362–72. дои : 10.1016/j.bbagrm.2014.02.007 . ПМК 4316174 . ПМИД 24560929 .
- ^ «ДНМТ3Л» . Отчет о генных символах . Комитет по генной номенклатуре Хьюго . Проверено 27 сентября 2012 г.
- ^ Хо М.Р., Бейкер Д.Д., Лааюн А., Смит С.С. (январь 1998 г.). «Остановка метилтрансферазы ДНК человека (цитозин-5) на одноцепочечных конформерах из места динамической мутации». Журнал молекулярной биологии . 275 (1): 67–79. дои : 10.1006/jmbi.1997.1430 . ПМИД 9451440 .
- ^ Смит С.С., Каплан Б.Е., Сауэрс Л.К., Ньюман Э.М. (май 1992 г.). «Механизм метил-направленной ДНК-метилтрансферазы человека и точность метилирования цитозина» . Труды Национальной академии наук Соединенных Штатов Америки . 89 (10): 4744–8. Бибкод : 1992PNAS...89.4744S . дои : 10.1073/pnas.89.10.4744 . ПМК 49160 . ПМИД 1584813 .
- ^ Джаир К.В., Бахман К.Е., Сузуки Х., Тинг А.Х., Ри И., Йен Р.В. и др. (январь 2006 г.). «Метилирование CpG-островков de novo в раковых клетках человека». Исследования рака . 66 (2): 682–92. doi : 10.1158/0008-5472.CAN-05-1980 . ПМИД 16423997 .
- ^ Тинг А.Х., Джаир К.В., Шубель К.Е., Байлин С.Б. (январь 2006 г.). «Дифференциальная потребность в ДНК-метилтрансферазе 1 для поддержания гиперметилирования промотора гена раковых клеток человека». Исследования рака . 66 (2): 729–35. дои : 10.1158/0008-5472.CAN-05-1537 . ПМИД 16424002 .
- ^ Ли Э., Бестор Т.Х., Джениш Р. (июнь 1992 г.). «Направленная мутация гена ДНК-метилтрансферазы приводит к эмбриональной смертности». Клетка . 69 (6): 915–26. дои : 10.1016/0092-8674(92)90611-F . ПМИД 1606615 . S2CID 19879601 .
- ^ Голл М.Г., Кирпекар Ф., Маггерт К.А., Йодер Дж.А., Се С.Л., Чжан X и др. (январь 2006 г.). «Метилирование тРНКАсп гомологом ДНК-метилтрансферазы Dnmt2» . Наука . 311 (5759): 395–8. Бибкод : 2006Sci...311..395G . дои : 10.1126/science.1120976 . ПМИД 16424344 . S2CID 39089541 .
- ^ «TRDMT1 тРНК метилтрансфераза 1 аспарагиновой кислоты (Homo sapiens)» . Энтрез Джин . НКБИ. 01.11.2010 . Проверено 7 ноября 2010 г.
- ^ Ян X, Хан Х, Де Карвалью Д.Д., Лэй Ф.Д., Джонс П.А., Лян Г. (октябрь 2014 г.). «Метилирование тел генов может изменить экспрессию генов и является терапевтической мишенью при раке» . Раковая клетка . 26 (4): 577–90. дои : 10.1016/j.ccr.2014.07.028 . ПМК 4224113 . ПМИД 25263941 .
- ^ Перейти обратно: а б Караджа К.Г., Купке Дж., Брито Д.В., Цейх Б., Томе С., Вейхенхан Д., Луцик П., Пласс С., Оливейра А.М. (январь 2020 г.). «Метилирование ДНК, специфичное для нейронных ансамблей, усиливает стабильность энграмм» . Нат Коммун . 11 (1): 639. Бибкод : 2020NatCo..11..639G . дои : 10.1038/s41467-020-14498-4 . ПМК 6994722 . ПМИД 32005851 .
- ^ Хегде М., Джоши М.Б. (апрель 2021 г.). «Комплексный анализ регуляции изоформ ДНК-метилтрансферазы в опухолях молочной железы человека» . J Cancer Res Clin Oncol . 147 (4): 937–971. дои : 10.1007/s00432-021-03519-4 . ПМЦ 7954751 . PMID 33604794 .
- ^ Оливейра А.М., Хемстедт Т.Дж., Бадинг Х. (июль 2012 г.). «Спасение связанного со старением снижения экспрессии Dnmt3a2 восстанавливает когнитивные способности». Нат Нейроски . 15 (8): 1111–3. дои : 10.1038/nn.3151 . ПМИД 22751036 . S2CID 10590208 .
- ^ Бернштейн С (2022). «Метилирование ДНК и установление памяти» . Эпигенетические идеи . 15 : 25168657211072499. дои : 10.1177/25168657211072499 . ПМЦ 8793415 . ПМИД 35098021 .
- ^ Байрактар Г., Кройц М.Р. (2018). «Роль деметилирования ДНК, зависящего от активности, в мозге взрослого человека и при неврологических расстройствах» . Фронт Мол Нейроски . 11 : 169. дои : 10.3389/fnmol.2018.00169 . ПМЦ 5975432 . ПМИД 29875631 .
- ^ Мак Г.С. (декабрь 2010 г.). «До избирательности и не только». Природная биотехнология . 28 (12): 1259–66. дои : 10.1038/nbt.1724 . ПМИД 21139608 . S2CID 11480326 .
- ^ «ЕС утверждает разрешение на продажу препарата DACOGEN для лечения острого миелоидного лейкоза» . 28 сентября 2012 г. Проверено 28 сентября 2012 г.
Дальнейшее чтение
[ редактировать ]- Смит СС (1994). Биологическое значение механизма действия ДНК (цитозин-5)метилтрансферазы человека . Прогресс в исследованиях нуклеиновых кислот и молекулярной биологии. Том. 49. стр. 65–111. дои : 10.1016/s0079-6603(08)60048-3 . ISBN 9780125400497 . ПМИД 7863011 .
- Прадхан С., Эстев П.О. (октябрь 2003 г.). «ДНК (цитозин-5) метилтрансферазы млекопитающих и их экспрессия». Клиническая иммунология . 109 (1): 6–16. дои : 10.1016/S1521-6616(03)00204-3 . ПМИД 14585271 .
- Голл М.Г., Бестор Т.Х. (2005). «Эукариотические цитозинметилтрансферазы». Ежегодный обзор биохимии . 74 : 481–514. doi : 10.1146/annurev.biochem.74.010904.153721 . ПМИД 15952895 . S2CID 32123961 .
Внешние ссылки
[ редактировать ]- Информация о ДНК-метилтрансферазах и метилировании ДНК на сайте epigeneticstation.com.
- Данные для антитела к ДНК-метилтрансферазе (DNMT)
- ДНК + Модификация + Метилтрансферазы в Национальной медицинской библиотеке США по медицинским предметным рубрикам (MeSH)